Method Article
Imagerie et analyse pour la quantification des phénotypes de stress abiotique du maïs (Zea mays)
Dans cet article
Résumé
Une méthode d’imagerie à faible coût des plantes pour mesurer les phénotypes est présentée, ainsi que les meilleures pratiques pour la capture d’images et un pipeline d’analyse d’images pour quantifier les caractéristiques des plantes. Ces méthodes ont été appliquées pour mesurer les phénotypes du maïs (Zea mays) dans des conditions de chaleur, de sécheresse et de stress abiotique combiné.
Résumé
La mesure quantitative des phénotypes végétaux, ou caractères, est essentielle pour comprendre les réponses des plantes aux facteurs environnementaux, y compris les stress biotiques et abiotiques. Cependant, les méthodes traditionnelles de mesure des plantes peuvent prendre du temps, être imprécises et destructrices, en particulier lorsqu’il s’agit d’évaluer des caractéristiques telles que la biomasse et la hauteur sur une base individuelle. L’analyse basée sur l’image offre la possibilité de réduire la main-d’œuvre, d’améliorer l’exactitude et la précision, et de permettre des mesures répétées de la même plante sur plusieurs points temporels. Bien que les installations de phénotypage à haut débit offrent une solution, leur coût est souvent prohibitif et leur disponibilité est limitée dans le monde entier. Une méthode est décrite pour capturer des images d’usine à l’aide d’ordinateurs monocartes peu coûteux et d’appareils photo numériques dans une configuration de studio photo, accompagnée d’un pipeline d’analyse d’images basé sur le package gratuit et open-source PlantCV. Cette méthode offre un protocole rapide et précis pour la capture, l’étiquetage et le transfert d’images, ainsi que les meilleures pratiques pour garantir une acquisition d’images de haute qualité. Grâce à l’analyse d’images, plus de 10 phénotypes ont été quantifiés simultanément pour toutes les images de l’expérience, y compris des traits tels que la taille, la hauteur et la couleur de la plante. Ces méthodes ont été utilisées pour caractériser la réponse de la variété consanguine B73 de Zea mays (maïs) à la chaleur, à la sécheresse et aux conditions combinées de chaleur et de stress abiotique de sécheresse.
Introduction
La mesure des traits des plantes, ou phénotypes, est essentielle pour répondre aux questions fondamentales et appliquées de la science végétale. Par exemple, un chercheur cherchant à comprendre l’impact d’une mutation génétique sur la taille d’une plante devra mesurer quantitativement la hauteur de la plante et la surface foliaire1. Un chercheur appliquant un stress biotique (tel qu’une infection bactérienne, virale ou fongique) ou un stress abiotique (tel que la sécheresse, la chaleur, le froid ou l’épuisement des nutriments) devra étudier l’effet du stress sur la couleur des plantes, ou « verdure », pour mesurer la chlorose 2,3.
Bien que la mesure de ces traits soit essentielle à la biologie végétale de base ainsi qu’à la sélection végétale appliquée, les mesures de ces traits à la main peuvent prendre du temps, être imprécises et inexactes 4,5,6,7. Par exemple, la mesure de la hauteur des plantes comprend l’utilisation manuelle d’une règle pour chaque plante, souvent dans des conditions environnementales chaudes ou humides, et est sujette à des erreurs du point de vue de la personne qui mesure, ainsi que du point considéré comme le « sommet » de la plante. De plus, les mesures telles que la biomasse et la teneur en pigments sont destructrices, et la même plante ne peut pas être mesurée pour détecter les changements au fil du temps ou utilisée pour plusieurs échantillons, car elle doit être détruite au cours du processus de réalisation deces mesures. La nature chronophage et sujette aux erreurs de ces mesures entrave les progrès dans l’évaluation des plantes pour leurs réponses phénotypiques et physiologiques au stress.
La capture d’images de plantes et la mesure des traits quantitatifs à partir de l’image fournissent des solutions à ces problèmes 4,5,6,7. L’analyse d’image améliore l’exactitude et la précision, tout en réduisant le temps dont dispose un chercheur pour effectuer les mesures, et la même plante peut être imagée plusieurs fois ou utilisée pour d’autres échantillonnages grâce à la quantification non destructive des traits 4,5,6,7. De plus, les images peuvent être stockées et réanalysées longtemps après la fin de l’expérience et l’élimination des plantes 4,5,6,7. L’analyse d’images peut être réalisée individuellement à l’aide d’outils d’édition d’images à faible débit, tels que ImageJ (Fiji)8, ou à haut débit à l’aide de la vision par ordinateur et de l’apprentissage automatique avec des plateformes telles que PlantCV9. PlantCV est une plate-forme d’analyse d’images gratuite et open-source qui permet aux chercheurs de masquer (ou de séparer) d’abord une plante ou un autre objet de l’arrière-plan d’une image, et d’effectuer des mesures de taille, de morphologie, de couleur et plus encore de cette plante ou de cet objet9. À l’aide d’un flux de travail modulaire, un chercheur crée d’abord un flux de travail flexible (ou pipeline) de différents modules avec une image d’échantillon, puis soumet les images restantes au flux de travail à haut débit sans interagir avec chaque image individuelle9. Les traits quantitatifs sont produits et analysés pour déterminer les différences statistiquement significatives entre les groupes. Des pipelines de Deep Learning peuvent être développés pour une analyse d’image efficace et automatisée une fois qu’un nombre suffisant d’images ont été capturées et étiquetées avec des mesures de caractéristiques ou des métadonnées appropriées10.
Le phénotypage « à haut débit » peut augmenter la vitesse et réduire le travail humain lors de la collecte de données et/ou de l’analyse des données 4,5,6,7. Les installations de phénotypage à haut débit ont permis l’automatisation de la collecte d’images dans des environnements contrôlés, où les plantes se déplacent sur des tapis roulants ou via des robots vers une station d’imagerie, et sont automatiquement étiquetées avec le nom (c’est-à-dire l’étiquette avec les métadonnées) associé à l’usine 6,11,12,13,14 . Bien que ces bandes transporteuses ou ces installations robotisées permettent de capturer et d’étiqueter de grandes quantités d’images sans intervention humaine, il y en a peu dans le monde et leur construction peut coûter des millions de dollars, ce qui les rend inaccessibles à de nombreux chercheurs. Pour appliquer ces concepts d’imagerie automatisée à moindre coût, les Raspberry Pi (ici appelés ordinateurs monocartes, SBC) sont des ordinateurs à faible coût (moins de 35 $ chacun) qui ont été utilisés pour capturer, nommer, stocker et transférer des images de plantes cultivées dans des environnements contrôlés. Ces configurations capturent des images à des intervalles automatisés et chronométrés à l’aide d’un réseau de 72 Raspberry Pi 12,15,16 au maximum. Bien qu’elles soient plus rentables que les systèmes de bande transporteuse qui peuvent coûter des millions de dollars, les chambres de croissance SBC décrites précédemment sont plus applicables à une vue descendante de la culture de petites plantes et nécessitent toujours de nombreux SBC15. Dans les environnements de terrain, les portiques, les UAS (systèmes aériens sans équipage) tels que les drones et les satellites capturent tous des images pour l’analyse des caractéristiques des plantes17,18. Les systèmes de terrain et les installations à environnement contrôlé à haut débit peuvent être prohibitifs en termes de coût initial, de maintenance et de connaissances spécialisées nécessaires à la construction et à la maintenance des systèmes, ainsi qu’à l’analyse des images17,18. Les téléphones portables et les appareils photo numériques utilisés seuls pour la photographie des plantes posent des défis chronophages liés au transfert et à la dénomination des images avec des métadonnées, ce qui est essentiel pour l’analyse des images en aval.
Les méthodes décrites ici présentent une solution peu coûteuse pour l’imagerie végétale pour les défis non relevés par d’autres systèmes. Une configuration photographique, où les plantes sont transportées individuellement au studio, permet de mesurer efficacement des plantes de toutes tailles dans des pots. À l’aide d’un SBC connecté à un appareil photo numérique, cette méthode fournit d’abord une capture, un étiquetage et un transfert d’images standardisés. L’utilisation d’un clavier ou d’un lecteur de codes QR/codes-barres pour nommer les images au fur et à mesure de leur capture permet d’éviter l’activité chronophage et sujette aux erreurs consistant à renommer les images avec des métadonnées une fois qu’elles ont été capturées et stockées. La composition de l’image et les paramètres recommandés de l’appareil photo sont importants pour permettre l’analyse d’image en aval, et des directives sont détaillées ici. Enfin, un pipeline d’analyse d’images dans PlantCV est fourni pour extraire des caractéristiques quantitatives des images capturées, telles que la surface des feuilles, la hauteur de la plante, la largeur et la couleur.
Par rapport aux systèmes de bande transporteuse, cette méthode est peu coûteuse et nécessite un seul SBC. La configuration de phénotypage décrite ici a été conçue pour éliminer les contraintes et les coûts d’une bande transporteuse ou de systèmes robotisés - n’importe quelle taille de plante peut être mesurée car la configuration peut être séparée de l’espace de croissance, un seul SBC est nécessaire et il fonctionne avec de nombreux types de caméras pour une flexibilité et un prix maximum abordables. Des approches similaires à celle présentée ici incluent la PhenoBox ou le PhenoRig12,19. Par rapport aux mesures manuelles, cette méthode permet de gagner du temps, de réduire les erreurs et de ne pas détruire les plantes 4,12,15,19.
Protocole
Les détails sur les réactifs et l’équipement utilisés dans ce protocole sont répertoriés dans la table des matériaux.
1. Préparation du studio photo
- Accrochez un tissu noir de qualité photographique à un mur ou au plafond pour une imagerie de côté. Drapez le tissu sur au moins 0,5 m sur le sol de manière à ce que la plante soit placée sur le tissu (figure 1).
REMARQUE : Le tissu doit être au moins 100 cm plus haut et plus large que la plante à imager. - Placez une carte de couleur sur la toile de fond, appuyée contre un objet tel qu’une boîte afin qu’elle soit visible dans le cadre (Figure 1). Pour les plantes de plus de 30 cm de haut ou de large, utilisez une carte de couleurs d’au moins 20 cm de large. Pour les plantes de moins de 30 cm de haut ou de large, utilisez une carte de couleurs de 4 à 8 cm de large.
- Branchez une carte SD sur un ordinateur portable ou de bureau. Téléchargez Raspberry Pi Imager à partir de la section logiciel du site Web (https://www.raspberrypi.com/software/), et téléchargez le fichier raspberrypi-image-capture.img.gz fourni (https://github.com/danforthcenter/photo-maize-paper) sur l’ordinateur portable ou de bureau.
- Lancez l’imageur en ouvrant le fichier (.exe) téléchargé. Cliquez sur le bouton Choisir le système d’exploitation et sélectionnez le bouton Utiliser personnalisé en bas. Sélectionnez le fichier image-capture.img.gz.
- Ensuite, cliquez sur le bouton Choisir le stockage et sélectionnez la carte SD branchée sur l’ordinateur. Cliquez sur le bouton Écrire .
REMARQUE : Assurez-vous que la bonne carte SD a été sélectionnée, car ce processus supprimera toutes les données stockées sur la carte.
- Ensuite, cliquez sur le bouton Choisir le stockage et sélectionnez la carte SD branchée sur l’ordinateur. Cliquez sur le bouton Écrire .
- Connectez la dernière version de SBC à un clavier à l’aide d’un câble USB, à un moniteur externe à l’aide d’un câble HDMI et à une multiprise à l’aide d’un cordon d’alimentation avec un interrupteur. Connectez le moniteur à la multiprise à l’aide du cordon d’alimentation fourni avec le moniteur. Placez la carte SD dans le SBC. Reportez-vous à la figure 1 pour un schéma étiqueté de la configuration.
- Allumez la multiprise en appuyant sur l’interrupteur de la barre, allumez le moniteur à l’aide du bouton du moniteur, puis allumez le SBC en basculant l’interrupteur du cordon d’alimentation sur « on ».
- Connectez le SBC à l’Internet local à l’aide du WIFI ou d’un câble Ethernet.
- Si les photos doivent être transférées directement vers un serveur ou un stockage en nuage :
- Modifiez le fichier « CAPTURE-PHOTO.py » sur le SBC pour qu’il contienne le chemin d’accès au fichier pour terminer le transfert.
- Modifiez le fichier win-credentials avec le chemin d’accès et le mot de passe du serveur ou du stockage dans le cloud à l’aide de la commande suivante dans le terminal : sudo nano /etc/win-credentials.
REMARQUE : Contactez l’administrateur du système Internet pour vous assurer que le dossier est autorisé à transférer des fichiers.
- Connectez un lecteur de codes-barres sans fil au SBC en suivant les instructions du fabricant fournies avec le lecteur de codes-barres, à l’aide d’un port USB ou Bluetooth.
- Éteignez le SBC en cliquant sur le bouton Raspberry Pi en haut à gauche de l’écran, en cliquant sur Déconnexion, puis en cliquant sur Arrêter pour éteindre le SBC en toute sécurité. Après l’arrêt, allumez l’interrupteur d’alimentation SBC, le cordon d’alimentation sur « off » et la multiprise sur « off ».
2. Croissance des plantes et traitement du stress
REMARQUE : Toute plante convient à cette méthode de phénotypage ; Ces méthodes de croissance des plantes sont applicables aux résultats représentatifs présentés ici.
- Procurez-vous des graines de B73 ou d’une autre variété de maïs auprès d’un centre de stockage.
- Préparez le sol et les conditions de croissance :
- Remplissez des pots de 3,5 pouces (0,7 L) avec un mélange de 1:1 en volume de Turface MVP et de Berger BM7-35 avec 3 lb/verge cube d’engrais à libération lente 14-14-14 (N-P-K). Placez les pots dans un bac troué et arrosez abondamment avec de l’eau osmosée (OI).
- Réglez une chambre de culture dans les conditions environnementales suivantes : 30 °C le jour/20 °C la nuit, 16 h de durée diurne, 60 % d’humidité et 500 mmol.m-2 s-1 de lumière au niveau du pot pendant la journée. Installez une chambre de culture séparée dans les mêmes conditions, à l’exception de 38 °C le jour et de 28 °C la nuit pour le stress thermique.
- Plantez 20 graines de maïs, chacune dans un pot de 3,5 pouces, en plaçant ~2 cm de profondeur dans le sol et en le recouvrant de la terre déplacée. Arrosez abondamment avec de l’eau osmosée et placez-le dans la chambre de croissance témoin. Arrosez quotidiennement avec de l’engrais liquide (mélangé selon les instructions du fabricant ; voir le tableau des matériaux).
REMARQUE : La plantation de plantes supplémentaires nécessaires pour l’expérience tiendra compte de la mauvaise germination et du manque d’uniformité avant le début d’une application de stress. Le taux de germination varie selon la variété. - Après 21 jours de croissance, repiquez 16 plantes des plus uniformes (n = 4 plantes par traitement) dans des pots de 3,79 L remplis du même terreau de terreau, pré-mouillés avec de l’eau RO. Remettez les plantes dans la chambre de croissance témoin.
- Le jour 25 (4 jours après le repiquage), déplacez la moitié des plantes (n = 8) dans la chambre de stress thermique au moment où les lumières s’allument dans la chambre. Arrêtez d’arroser les plantes stressées par la sécheresse dans les deux chambres (n = 4 par chambre).
REMARQUE : Les plantes de stress hydrique peuvent avoir besoin d’être arrosées pour qu’elles survivent jusqu’à la fin de l’expérience ; Ici, les plantes de sécheresse ont été arrosées le jour 31 et le jour 37 avec de l’eau d’engrais. - Le jour 38 (13 jours après le début du stress), retirez les plantes des chambres de croissance pour l’imagerie.
3. Imagerie végétale
- Démarrez le SBC (voir Figure 2) :
- Allumez la multiprise à l’aide de l’interrupteur situé sur la multiprise.
- Allumez le moniteur à l’aide du bouton situé sur le moniteur.
REMARQUE : Si le SBC est allumé avant que le moniteur ne soit allumé, il ne démarrera pas correctement. - Mettez l’interrupteur du cordon d’alimentation SBC sur « on ».
REMARQUE : Cela allumera le SBC et il démarrera sur le moniteur ; il n’y a pas d’interrupteur « on » sur le SBC lui-même, seulement sur le cordon d’alimentation.
- Placez l’appareil photo sur un trépied ou une table stable.
REMARQUE : L’ensemble de la plante et de la carte de couleur doit être visible sur l’écran de l’appareil photo. La caméra doit être perpendiculaire au sol et avoir une vue directe (sans angle) du centre de la plante. L’appareil photo doit être compatible avec le paquet gphoto2, et une liste peut être trouvée ici : http://www.gphoto.org/proj/libgphoto2/support.php. - Alimentez l’appareil photo via un cordon d’alimentation avec l’appareil photo ou des piles. Allumez l’appareil photo à l’aide du bouton de l’appareil photo.
- Connectez l’appareil photo au port USB du SBC à l’aide du câble de transfert de données fourni avec l’appareil photo.
- Disposez la plante dans la configuration de la photographie :
- Placez un morceau de ruban adhésif de couleur foncée sur le tissu de qualité photographique où le pot sera placé pour vous assurer que le placement est le même pour chaque plante. Assurez-vous que la carte de couleur est alignée avec le morceau de ruban adhésif.
REMARQUE : Placez la carte de couleur à au moins 10 cm du bord de la plante. Lors du contrôle qualité dans le flux de travail PlantCV (ci-dessous), l’image doit pouvoir être recadrée pour inclure uniquement la plante et le pot, mais pas la carte de couleur pour s’assurer que les pastilles de couleur ne sont pas incluses dans l’extraction finale du caractère. Voir la figure 2 pour des exemples d’images de haute et de basse qualité. - Placez le pot sur le morceau de ruban adhésif. Pour les plantes qui poussent dans un avion, comme le maïs (Zea mays) et le sorgho (Sorghum bicolor), faites face à l’angle le plus large de la plante vers la caméra. Pour les plantes qui ne poussent pas dans un avion, capturez les angles de 0 et de 90 degrés de la plante et calculez la moyenne des mesures quantitatives résultantes une fois l’analyse de l’image terminée.
- Placez un morceau de ruban adhésif de couleur foncée sur le tissu de qualité photographique où le pot sera placé pour vous assurer que le placement est le même pour chaque plante. Assurez-vous que la carte de couleur est alignée avec le morceau de ruban adhésif.
- Sur l’écran du bureau SBC affiché sur le moniteur, ouvrez le terminal en cliquant sur le bouton du terminal dans la barre de menu supérieure gauche.
- Si vous enregistrez directement sur un serveur ou un stockage cloud, montez le stockage à l’aide de la commande suivante en modifiant le chemin de stockage cloud nécessaire (cloud-storage-path dans l’exemple ci-dessous) et le mot de passe, en tapant la ligne de code dans la fenêtre de terminal ouverte et en appuyant sur la touche Entrée du clavier. S’il n’est pas modifié par rapport aux paramètres d’usine, l’ID utilisateur (UID) sera « pi ».
NOTE:
sudo mount -t cifs -o credentials=/etc/win-credentials,uid=pi //chemin-de-stockage-cloud /mnt/stockage-cloud - Tapez le code suivant dans le terminal et appuyez sur Entrée sur le clavier. Une nouvelle fenêtre apparaîtra.
./RUN-PHOTOSTUDIO-SHELL.sh
REMARQUE : Le fichier RUN-PHOTOSTUDIO-SHELL.sh appelle le fichier CAPTURE-PHOTO.py, qui est préchargé sur le pi s’il a été chargé à l’aide de la méthode ci-dessus. Alternativement, ces scripts ont été fournis sur GitHub pour être mis sur une carte SD Raspberry Pi existante. - Une fenêtre d’interface utilisateur graphique s’ouvrira. Tapez le nom de l’image, ou scannez le code-barres ou le code QR de l’usine à l’aide d’un lecteur de codes-barres.
- Sélectionnez une option pour enregistrer l’image localement ou sur un serveur/stockage cloud monté.
- Cliquez ici pour prendre une photo ! bouton.
- Si le stockage local a été choisi, ouvrez le dossier PHOTOS sur le bureau pour examiner l’image. Si un serveur ou un stockage dans le cloud a été choisi, ouvrez l’image à cet emplacement.
- Si nécessaire, ajustez les paramètres de l’appareil photo en le débranchant du SBC, en l’ajustant à l’aide des instructions du fabricant et en le rebranchant sur le SBC.
REMARQUE : Des paramètres tels que l’exposition, la luminosité, le contraste, etc., peuvent devoir être ajustés en fonction de l’éclairage de la pièce et de l’objet imagé. Une erreur courante est de surexposer les images. Pour vérifier l’exposition d’une image de test, procédez à l’image de test via le flux de travail d’analyse d’image PlantCV, qui comprend un contrôle qualité de l’exposition. Ajustez les paramètres de l’appareil photo si nécessaire pour réduire ou augmenter l’exposition jusqu’à ce que les histogrammes soient correctement répartis. - Une fois toutes les images capturées, transférez les images du SBC vers un ordinateur local ou un stockage cloud à l’aide de la méthode préférée, telle que le stockage USB, le navigateur Internet ou le transfert SSH.
4. Extraction de traits à partir d’images
- Téléchargez les fichiers suivants sur un ordinateur ou un serveur local : « photo-studio-SV-notebook.ipynb », « workflow.py » et « photo-studio-SV-config.json ». Tous les fichiers sont disponibles sur GitHub (https://github.com/danforthcenter/photo-maize-paper).
- Installez PlantCV sur l’ordinateur local ou le serveur en suivant les étapes décrites (https://plantcv.readthedocs.io/en/latest/installation/).
- Ouvrez le fichier photo-studio-SV-notebook.ipynb à l’aide de l’éditeur de code préféré, tel que JupyterLab ou Visual Studio Code.
- Exécutez chaque bloc de code, en modifiant si nécessaire compte tenu des paramètres décrits dans le bloc de code pour obtenir un masque propre de la plante uniquement dans l’image.
- Lorsque vous êtes satisfait de l’analyse de l’image d’exemple, ouvrez le fichier workflow.py dans l’éditeur de code de votre choix. Modifiez ce fichier avec tous les paramètres modifiés dans « photo-studio-SV-notebook.ipynb » et enregistrez-le.
- Ouvrez le photo-studio-SV-config.json de fichiers et modifiez les chemins d’accès aux fichiers pour les diriger vers un dossier d’images d’entrée, un dossier de sortie souhaité, etc., comme indiqué dans le fichier.
- Ouvrez un terminal et exécutez le code suivant pour activer PlantCV. Pour ce faire, tapez le code conda activate plantcv dans le terminal et appuyez sur Entrée sur le clavier.
- Remplacez la ligne de code suivante par le chemin d’accès au fichier « photo-studio-SV-config.json » (c’est la seule modification à apporter dans le code ci-dessous).
- Dans le terminal, tapez le code modifié et appuyez sur la touche Entrée du clavier. Cela exécutera le flux de travail sur toutes les images capturées dans l’expérience.
NOTE:
plantcv-run-workflow --config /Utilisateurs/NOM_D’UTILISATEUR/photo-studio-SV-config.json
- Dans le terminal, tapez le code modifié et appuyez sur la touche Entrée du clavier. Cela exécutera le flux de travail sur toutes les images capturées dans l’expérience.
- Inspectez les images de sortie pour déterminer si le code était approprié pour toutes les images.
REMARQUE : Le contour bleu et rose de la plante doit être sur les bords de la plante et il ne doit pas y avoir d’autres objets dans l’image. Si nécessaire, retournez sur le « photo-studio-SV-analysis.ipynb » et testez les images problématiques et éditez le code, en particulier les étapes de masquage et la réduction du bruit, pour obtenir les résultats souhaités. Voir la figure 3 pour des exemples de segmentation d’image élevée, acceptable et de faible qualité. - Le fichier de sortie se présente sous la forme d’un fichier .json. Retournez au terminal (assurez-vous que conda activate plantcv a déjà été exécuté), tapez le code suivant et appuyez sur la touche Entrée du clavier pour convertir le fichier en .csv pour une analyse statistique en aval, en remplaçant le chemin du fichier par celui du fichier « results-photo-studio.json ».
plantcv-utils json2csv -j /Utilisateurs/NOM_D’UTILISATEUR/results-photo-studio.json -c /Utilisateurs/NOM_D’UTILISATEUR/results-photo-studio.csv
REMARQUE : Pour convertir les mesures des pixels en une unité de mesure standard, telle que cm, utilisez la conversion fournie par la fonction find_color_card dans le flux de travail PlantCV. - Effectuez une analyse statistique des caractères quantitatifs de sortie pour déterminer les différences entre les phénotypes des plantes dues aux différentes espèces de plantes, aux différentes variétés, aux différents traitements et aux différents points temporels.
REMARQUE : Le package pcvr de R Studio a été formulé pour analyser les sorties de PlantCV pour la signification statistique et le tracé des résultats20. Voir un résumé et une représentation visuelle des traits recueillis dans la figure 4.
Résultats
Pour mettre au point cette méthode, les plants de maïs B73 ont été évalués pour des phénotypes quantitatifs à des températures contrôlées, des températures chaudes, bien arrosées et des conditions de sécheresse, ainsi que leurs combinaisons, pour un total de quatre traitements. B73 est une variété de maïs consanguin bien connue avec une richesse en données génétiques21.
L’analyse d’images dans PlantCV a permis de corriger les couleurs des images, d’étiqueter les résultats quantitatifs avec les métadonnées de la plante et de segmenter (en d’autres termes, de séparer) la plante de l’arrière-plan, comme le montre la figure 3. Une analyse infructueuse fournirait des images de sortie qui ne montrent pas le contour bleu ou la forme rose autour de l’extérieur de la plante, mais qui incluent plutôt du bruit supplémentaire ou des morceaux manquants de la plante (figure 3, exemple de faible qualité). Un seuil à double canal était nécessaire pour inclure autant de plante que possible dans le masque (voir les stratégies alternatives à un seul canal dans le « photo-studio-SV-notebook.ipynb »). Cependant, certaines plantes, en particulier celles dont les tiges sont de couleur violette, n’ont pas eu une segmentation parfaite, mais ont été suffisantes pour l’analyse en aval (figure 3).
L’analyse PlantCV a fourni 16 caractères quantitatifs à valeur unique, résumés dans la figure 4. Un caractère à valeur unique fournit une valeur par plante - par exemple, la surface des feuilles, la hauteur, la largeur et la moyenne circulaire de la teinte. La surface foliaire a été prise en compte à l’aide de la fonction « analyze.shape » (figure 4) et de la plante entière. La hauteur et la largeur, cependant, ont été prises en compte à l’aide de la « fonction analyze.bound_horizontal », avec une borne tracée à la ligne du sol de la plante. En effet, les plantes qui s’affaissent sur le côté du pot auront une plus grande hauteur si l’on considère la plante comme un objet entier, et ne devraient mesurer qu’à partir de la base de la plante où elle rencontre le sol. En plus des caractères à valeur unique, PlantCV produit des caractères à valeurs multiples, qui sont des histogrammes, en particulier des valeurs de couleur, représentant chaque pixel de la plante. La moyenne circulaire de la teinte est un caractère à valeur unique qui est la valeur moyenne de la teinte pour l’ensemble de la plante.
Pour déterminer quels traits sont les plus intéressants, la variance expliquée par le traitement pour chaque caractère a été calculée, ainsi que la corrélation entre les différents traits à l’aide de la fonction « frem » dans le package pcvr R Studio20 (Figure 5A). La surface, la hauteur, la largeur et la moyenne circulaire de la teinte des feuilles ont été sélectionnées pour l’analyse en aval parce qu’elles expliquaient plus de 50 % de la variance due au traitement et qu’elles sont des mesures pertinentes de la croissance (superficie, hauteur et largeur) et de la santé (teinte) des plantes. Des images représentatives de la plante avec la plus grande et la plus petite surface foliaire montrent la variation des phénotypes collectés dans cette expérience (figures 5B et C, respectivement). La plante dont la surface foliaire était la plus grande avait également la teinte moyenne la plus élevée et était une plante B73, bien arrosée et soumise à un stress thermique (figure 5B). La plus petite surface foliaire avait la plus petite teinte moyenne (figure 5C) et était une plante B73, stressée par la sécheresse et la chaleur.
Le traitement de l’eau a eu un effet significatif sur la détermination de la surface foliaire (F(1,13) = 226,5, p = 1,32 x 10-9), de la hauteur (F(1,13) = 21,1, p = 0,0005), de la largeur (F(1,13) = 75,5, p = 8,92 x 10-7) et de la moyenne circulaire de la teinte (F(1,13) = 27,8, p = 0,0002), déterminée à l’aide d’un modèle de régression linéaire pour l’effet du traitement et d’une ANOVA à deux facteurs dans R Studio (résultats complets dans le tableau 1). Le traitement thermique a eu un effet significatif sur la hauteur (F(1,13) = 5,94, p = 0,03), mais pas sur la surface, la largeur ou la teinte des feuilles, la moyenne circulaire (p > 0,05, tableau 1). L’interaction entre la température et l’état de l’eau n’était un facteur significatif dans aucun des caractères mesurés (p > 0,05, tableau 1).
Ensuite, les traitements individuels ont été comparés au témoin et les uns aux autres à l’aide d’une régression linéaire et d’un test post-hoc (moyennes marginales estimées) avec une correction de Sidak pour les comparaisons multiples (figure 6). La surface foliaire, la hauteur et la largeur de la plante et la moyenne circulaire de la teinte ont été considérablement réduites chez les plantes stressées par la sécheresse par rapport aux plantes bien arrosées dans les deux conditions de température (p < 0,05, figure 6). Le stress thermique lorsqu’il est bien arrosé ne réduit que la hauteur de la plante (p < 0,05, figure 6), mais ne réduit pas significativement la surface des feuilles, la largeur de la plante ou la moyenne circulaire de la teinte. Cette différence de teinte peut être étudiée plus en détail en évaluant la teinte du trait de couleur à valeurs multiples, tracée sous forme d’histogramme dans la figure 7, plutôt que d’une simple moyenne. La réduction de la teinte moyenne due à la sécheresse était due à un passage des pixels verts aux pixels jaunes, connu sous le nom de chlorose (Figure 7). Bien que la chaleur n’ait pas montré de différence significative dans la teinte moyenne, elle a montré une réduction et une augmentation en raison du jaunissement (chlorose) et de l’assombrissement de la couleur verte (figure 7).
Après l’imagerie, les plantes ont été coupées à la jonction racine-pousse, et le poids des tissus aériens a été enregistré. Les racines ont été soigneusement lavées et séchées avec une serviette, puis pesées, afin de calculer la biomasse végétale totale (également appelée poids) à des fins de comparaison avec la méthode de phénotypage basée sur l’image décrite. La surface foliaire mesurée par analyse d’images est fortement corrélée à la biomasse végétale (R2 = 0,84, Figure 8).

Figure 1 : Image de l’installation du studio photo avec les composants étiquetés pour la collecte d’images de plantes. (A) Tissu de qualité photographie, (B) carte couleur, (C) ligne de ruban adhésif pour placer les pots uniformément, (D) appareil photo numérique, (E) ordinateur monocarte, (F) moniteur affichant le processus de capture d’image. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Vue d’ensemble du flux de travail d’imagerie. Une image représentative de « haute qualité » et de « basse qualité » est fournie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
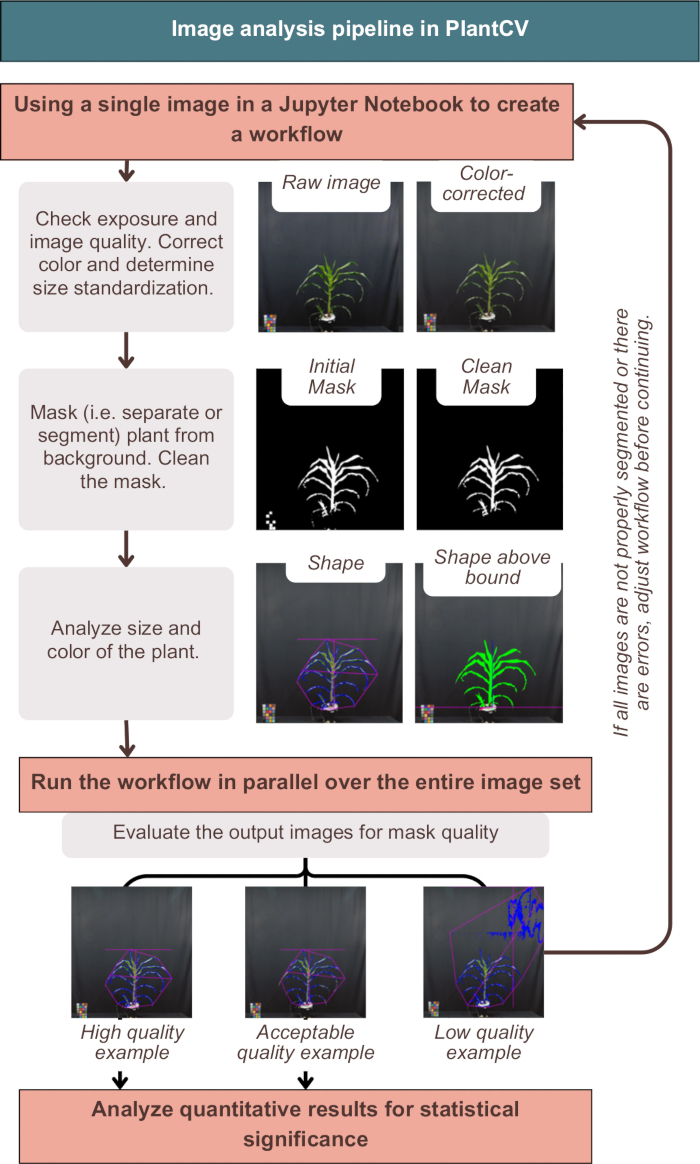
Figure 3 : Vue d’ensemble du flux de travail d’analyse d’images dans PlantCV. Des images représentatives de la segmentation de qualité « élevée », « acceptable » et « faible » sont présentées ; Les traits PlantCV sont représentés à l’aide d’un contour rose de la forme de la plante entière et d’un contour bleu de la surface de la plante. Veuillez cliquer ici pour voir une version agrandie de cette figure.
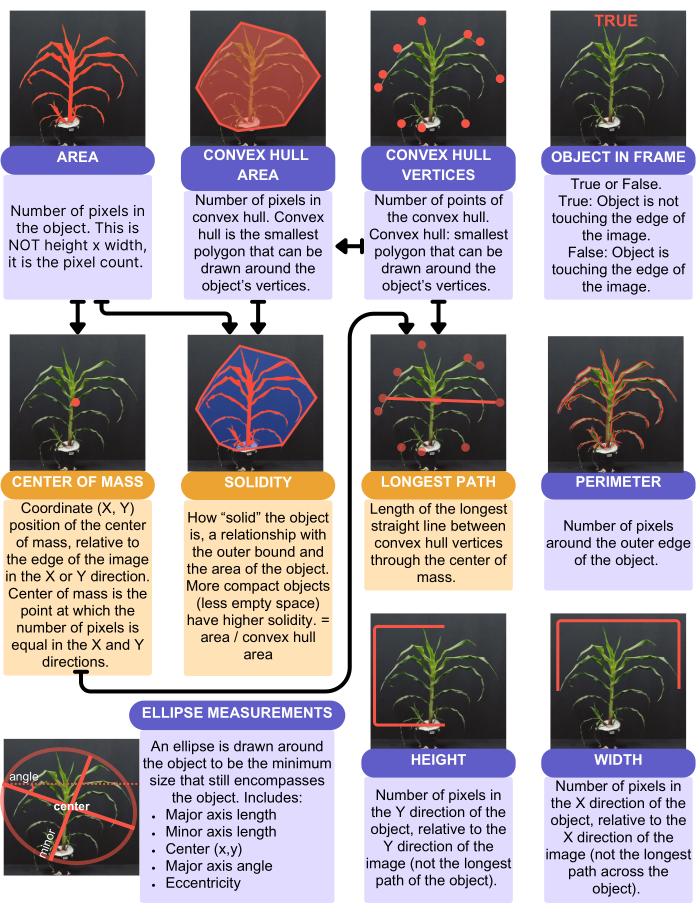
Figure 4 : Résumé et représentation visuelle des traits de forme capturés à partir de l’analyse. Fonction de forme dans PlantCV. Les cases violettes représentent des traits qui n’utilisent pas un autre trait dans leur calcul. Les cases orange représentent les caractères qui utilisent d’autres caractères dans leur calcul ; Les flèches indiquent les traits utilisés dans ces calculs. Veuillez cliquer ici pour voir une version agrandie de cette figure.
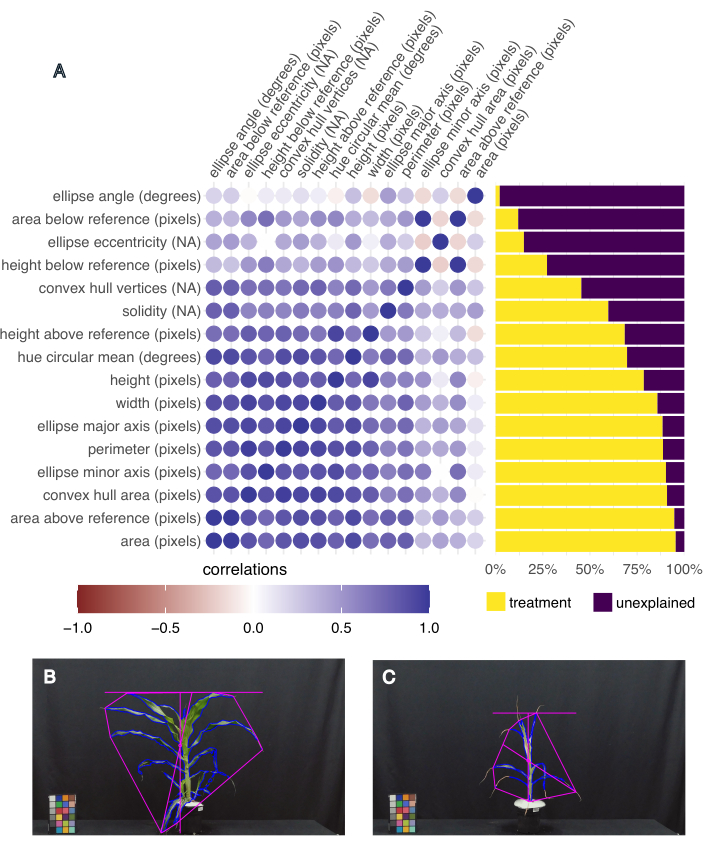
Figure 5 : Le phénotypage basé sur l’image capture la variance phénotypique et les traits corrélés. (A) Variance des traits expliquée par le traitement (à droite) et corrélation des traits les uns aux autres (à gauche). Chaque trait est étiqueté, suivi de son unité de mesure ; « NA » signifie qu’il s’agit d’une mesure sans unité. (B) Image de la plante qui avait la surface foliaire la plus élevée et la teinte la plus élevée (degrés) en moyenne. (C) Image de la plante qui avait la plus petite surface foliaire et la teinte la plus basse moyenne circulaire (degrés). Veuillez cliquer ici pour voir une version agrandie de cette figure.
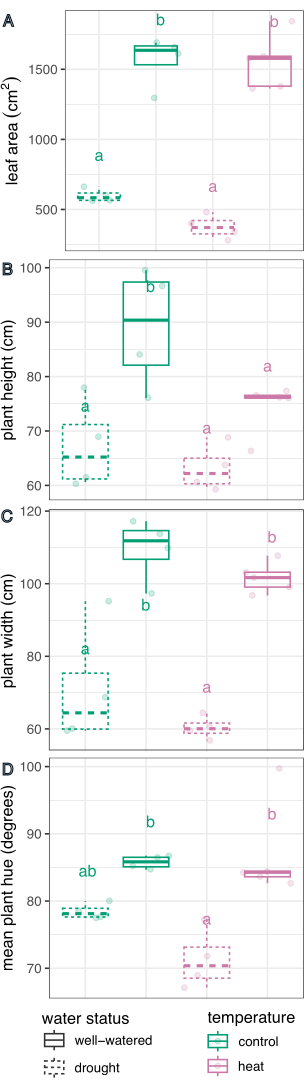
Figure 6 : Comparaison en boîte à moustaches des effets du traitement sur les phénotypes végétaux. La surface des feuilles (A), la hauteur (B), la largeur (C) et la moyenne circulaire de la teinte (D) sont comparées pour l’effet de la sécheresse, de la chaleur et des contraintes combinées. n = 4. Les lettres représentent des différences statistiquement significatives entre les traitements (p < 0,05) ; les traitements ont été comparés à l’aide d’une régression linéaire et d’un test post-hoc (moyennes marginales estimées) avec une correction de Sidak pour les comparaisons multiples. Veuillez cliquer ici pour voir une version agrandie de cette figure.
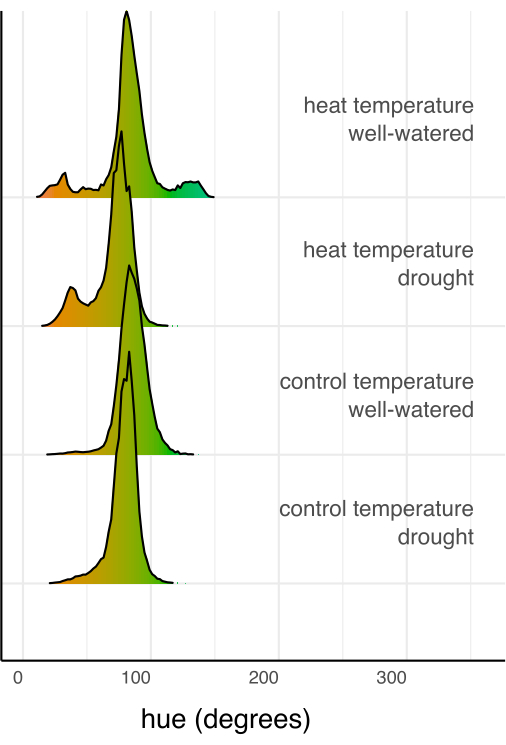
Figure 7 : Histogrammes de couleur (teinte) additionnés de tous les pixels de chaque combinaison de traitement, normalisés par le nombre total de pixels dans la combinaison de traitement. Les histogrammes sont colorés en fonction de la valeur de couleur du degré de teinte. Veuillez cliquer ici pour voir une version agrandie de cette figure.
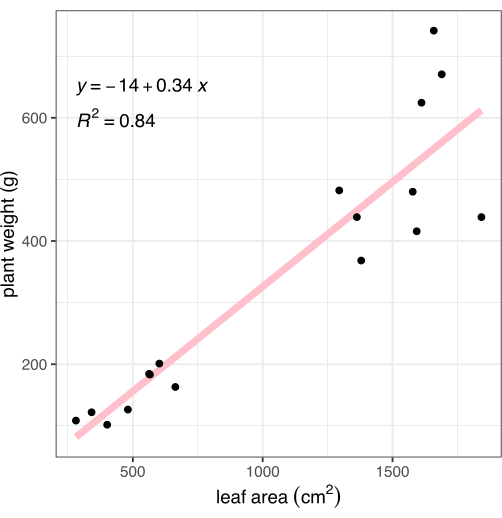
Figure 8 : Corrélation de la surface foliaire mesurée à partir des images à la biomasse de la plante entière. Chaque point noir représente une plante. La ligne rose représente la corrélation linéaire entre la surface foliaire (X) et la biomasse (Y). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Trait | Facteur | degrés de liberté | Valeur F | Valeur p |
| Surface foliaire (cm^2) | Température | 1 | 0.5372 | 0.4766 |
| Surface foliaire (cm^2) | Eau | 1 | 226.5318 | 1.32E-09 |
| Surface foliaire (cm^2) | Température :Eau | 1 | 2.1526 | 0.1661 |
| Surface foliaire (cm^2) | Résidus | 13 | ||
| Hauteur (cm) | Température | 1 | 5.9426 | 0.0298935 |
| Hauteur (cm) | Eau | 1 | 21.107 | 0.0005029 |
| Hauteur (cm) | Température :Eau | 1 | 2.1752 | 0.1640545 |
| Hauteur (cm) | Résidus | 13 | ||
| Largeur (cm) | Température | 1 | 2.2495 | 0.1575 |
| Largeur (cm) | Eau | 1 | 75.5474 | 8.92E-07 |
| Largeur (cm) | Température :Eau | 1 | 0.0839 | 0.7766 |
| Largeur (cm) | Résidus | 13 | ||
| teinte circulaire moyenne (degrés) | Température | 1 | 0.9154 | 0.3561432 |
| teinte circulaire moyenne (degrés) | Eau | 1 | 27.7988 | 0.0001509 |
| teinte circulaire moyenne (degrés) | Température :Eau | 1 | 3.4792 | 0.0848724 |
| teinte circulaire moyenne (degrés) | Résidus | 13 |
Tableau 1 : Résultats du modèle de régression linéaire et des analyses ANOVA à deux facteurs examinant les effets du traitement de l’eau, du traitement de la température et de leur interaction sur les caractéristiques des plantes.
Discussion
Le phénotypage basé sur l’image fournit des mesures quantitatives précises, rapides et précises des phénotypesvégétaux 4,5,6,7. Cette méthode est nettement plus rapide, plus précise, plus exacte et fournit un plus grand nombre de mesures que l’utilisation manuelle d’une règle pour enregistrer la hauteur des plantes, ou des mesures qualitatives des caractéristiques de la couleur des plantes qui peuvent être liées à la santé 4,5,6,7. On a constaté que la surface foliaire ici, et dans des recherches antérieures10, était fortement corrélée à la biomasse végétale (figure 8), ce qui suggère que cette mesure pourrait remplacer de manière fiable le processus long, destructeur et sujet aux erreurs de pesage manuel de la biomasse végétale entière. Bien qu’une seule caméra puisse être utilisée pour capturer les images, l’ajout du SBC à faible coût a augmenté le débit grâce à l’automatisation de la collecte d’images et de l’étiquetage des images avec les métadonnées nécessaires15. Cette méthode convient pour un à ~300 plantes qui sont imagées une fois par jour (ou moins fréquemment). Pour un plus grand nombre d’installations dans une expérience, un système d’imagerie basé sur un convoyeur, un portique aérien ou un robot est plus approprié, car le temps nécessaire pour déplacer les plantes individuellement pour l’imagerie peut être plus long qu’une journée de travail11,13. Pour des moments plus fréquents (par exemple, une image toutes les heures plutôt qu’une fois par jour ou moins fréquemment), les configurations de caméra à l’intérieur d’une chambre de culture sont plus appropriées car elles capturent des images automatiquement, mais sont limitées par la taille des plantes et nécessitent un SBC15 supplémentaire.
Bien que PlantCV soit présenté ici comme l’option d’analyse d’images, d’autres logiciels existent. ImageJ (Fidji) est un progiciel gratuit d’analyse d’images qui peut également être utilisé pour des images de plantes via une interface utilisateur graphique (GUI)8 ; cependant, il n’est pas particulièrement développé pour l’analyse d’images d’une plante entière, est moins adaptable à l’informatique à haut débit et est moins facilement reproductible que les pipelines déjà fonctionnels comme ceux de PlantCV qui peuvent être facilement appliqués à de nouvelles images9. Les méthodes d’apprentissage profond (telles que BioDock, voir la table des matériaux) et Segment Anything22) sont attrayantes pour leur capacité à segmenter facilement les plantes à partir de l’arrière-plan, et pourraient être utilisées pour les tâches de segmentation décrites ici. Cependant, ces modèles d’apprentissage profond ne fournissent pas les mesures de phénotypes végétaux décrites ici10, et pour effectuer ces mesures plus complexes de phénotypes quantitatifs, il peut être nécessaire d’utiliser une grande quantité d’images d’entraînement (souvent supérieure à 10 000) et une puissance de calcul supérieure à celle d’un ordinateur portable ou de bureaude base 10. Ces modèles nécessitent également une mémoire informatique et une puissance de traitement importantes, qui ne sont pas disponibles pour de nombreux chercheurs10.
L’étape la plus critique du protocole consiste à organiser la configuration de l’imagerie de la plante et à s’assurer qu’il n’y a pas d’objets (tels que d’autres plantes, une carte de couleur ou des débris de plantes) à proximité de la plante d’intérêt14. Si les images ne sont pas configurées de manière appropriée ou si une carte de couleur n’est pas utilisée, elles seront impossibles à analyser ultérieurement car il n’existe aucun mécanisme de normalisation. Bien qu’aucune étape d’étalonnage ne soit nécessaire dans ce protocole, la correction des couleurs et la standardisation de la taille pendant le traitement sont essentielles. Assurez-vous toujours que toute la carte de couleurs est dans le cadre, mais qu’elle ne chevauche pas la plante (Figure 2). Un autre piège courant est l’éclairage inégal, qui peut provoquer des ombres ou des photos lumineuses ; nous vous recommandons d’utiliser une lumière zénithale vive et diffuse et de couvrir les fenêtres de la pièce pour éviter un éclairage inégal (Figure 2). Des lampes supplémentaires peuvent être ajoutées à un angle de face à la plante si les plafonniers provoquent des ombres. Le type d’ampoule n’est pas un problème, étant donné que la couleur sera corrigée dans l’image à l’aide d’une carte de couleurs. Les images floues ne sont pas acceptables pour l’analyse - utilisez la mise au point automatique sur l’appareil photo ou les instructions du fabricant pour la mise au point manuelle pour vous assurer que les images sont claires.
De même, s’assurer que les images ne sont pas surexposées (une erreur courante) permet une analyse appropriée (Figure 2) ; Les images surexposées ne peuvent pas être corrigées lors d’une correction colorimétrique lors de l’analyse d’image, car les valeurs de pixels sont au-delà de la plage de détection de l’appareil photo avec les paramètres actuelsde 14. Pour éviter la surexposition, évaluez les histogrammes de couleur ou les valeurs RVB d’une image de test à l’aide du flux de travail PlantCV décrit ici (Figure 3), et réduisez le réglage de la luminosité de la caméra si le test d’exposition détermine que l’image est surexposée. Les valeurs RVB (et donc les histogrammes) ne doivent pas avoir de valeurs à 0 ou à 255, ce qui est la plage de valeurs, car il est possible que ces valeurs soient supérieures ou inférieures à la plage de détection et ne puissent enregistrer que le minimum ou le maximum plutôt que la valeur réelle. Une image d’essai doit toujours être évaluée à l’aide de ces méthodes pour détecter la surexposition (figures 2 et 3) ; nous vous suggérons d’utiliser le contrôle de la qualité d’exposition PlantCV fourni dans cette publication comme une vérification, qui détermine qu’une image de qualité a moins de 5 % de pixels à 0 ou 255 pour chaque couleur. En règle générale, les caméras et les logiciels d’analyse fournissent des histogrammes, qui doivent avoir des queues d’histogramme égales et complètes ; Les valeurs coupées inférieures à 0 (sous-exposée) ou supérieures à 255 (surexposée) doivent être reprises avec les paramètres ajustés de l’appareil photo. Nous vous suggérons de choisir un endroit avec un éclairage artificiel vif et uniforme sans fenêtres pour l’éclairage, car les fenêtres peuvent être trop lumineuses, inégales et changer fréquemment l’exposition à la lumière de l’espace. Les images doivent apparaître uniformément éclairées et les carrés gris d’une carte de couleurs ne doivent pas apparaître blancs, ce qui suggère une surexposition.
Un dépannage peut être nécessaire pour le code de capture d’image en fonction de l’adresse IP et des paramètres de sécurité de l’établissement de l’utilisateur. Pour résoudre le problème, contactez le service informatique pour les problèmes de connectivité Internet et de transfert de fichiers. Une erreur courante consiste à utiliser une carte SD de la mauvaise taille pour le SBC ou à allumer le SBC avant que le moniteur connecté ne soit allumé. Pour résoudre le problème, suivez scrupuleusement le protocole ci-dessus et rechargez le logiciel SBC. Les forums en ligne sont une excellente ressource pour le dépannage et la recherche de solutions pour l’appareil photo spécifique de votre choix lors de l’utilisation de gphoto2. Pour résoudre les problèmes d’analyse d’images PlantCV et utiliser la version la plus récente, utilisez la page de documentation (https://plantcv.readthedocs.io/en/latest/) ou publiez un problème sur GitHub (https://github.com/danforthcenter/plantcv/issues). En particulier, si la méthode de seuillage de ce protocole ne sépare pas suffisamment la plante de l’arrière-plan, utilisez d’autres méthodes de seuillage fournies dans « photo-studio-SV-notebook.ipynb » ou sur la page de documentation.
Cette méthode peut être appliquée à toutes les espèces de plantes et à tous les traitements qui peuvent être transportés vers l’installation de photographie assemblée décrite. Par exemple, il ne s’applique pas aux plantes qui poussent dans un champ, car elles ne peuvent pas être transportées vers l’installation de photographie sans déterrer la plante. Dans cette situation, les plantes pourraient être coupées et ensuite imagées à l’aide du protocole, bien que la mesure soit destructrice. Plusieurs plantes peuvent être capturées dans une seule image et analysées, mais un utilisateur doit veiller à ce que les plantes ne se touchent pas (Figure 2), et les plantes sont placées au même endroit sur la toile de fond à chaque fois pour s’assurer que le flux de travail peut être exécuté automatiquement sur l’ensemble des images. Si une vue de haut en bas est nécessaire pour des plantes telles qu’Arabidopsis thaliana, utilisez un trépied pour déplacer l’appareil photo vers le haut des plantes placées sur le tissu de qualité photographique ; Les autres méthodes de capture et d’analyse d’images restent les mêmes.
Comme démontré ici avec le maïs, cette méthode est importante à la fois pour la sélection végétale et pour des connaissances fondamentales sur la façon dont les plantes tolèrent les stress abiotiques, tels que la chaleur et la sécheresse. La chaleur et la sécheresse contribuent à une perte de rendement de 15 à 20 % du maïs chaque année dans le monde23. Les pertes de rendement ne devraient qu’augmenter avec le changement climatique mondial, car les températures devraient augmenter de 1,4 à 4,4 °C d’ici 210024. L’évaluation de la diversité du maïs pour la tolérance au stress, ainsi que la détermination des mécanismes phénotypiques, physiologiques et moléculaires de la tolérance au stress abiotique, sont des objectifs de recherche essentiels pour la sécurité alimentaire mondiale25. Pour répondre à ces questions, les chercheurs de tous les domaines adjacents - physiologistes, généticiens, sélectionneurs, biologistes moléculaires, etc. - doivent être en mesure d’évaluer rapidement, efficacement et souvent de manière non destructive les phénotypes des plants de maïs sous stress abiotique25. La méthode et les exemples de résultats présentés ici répondent à ce besoin et constituent un outil important pour les chercheurs afin de relever les défis agricoles du monde réel.
Les phénotypes observés ici ont été émis dans l’hypothèse, compte tenu d’une multitude de recherches antérieures - le stress hydrique réduit la taille et la verdure des plantes (mesurée par la moyenne circulaire de teinte)26 (figure 6, figure 7 et tableau 1). Le phénotype du stress thermique avec un bon arrosage n’a pas réduit de manière significative la surface foliaire, bien qu’avec plus de répétitions, cette relation ait pu être mieux évaluée statistiquement, et la hauteur a été réduite comme prévu (figure 6). Bien que la moyenne circulaire de la teinte n’ait pas été modifiée en raison de la chaleur, le profil de couleur a en effet été modifié pour inclure à la fois plus de pixels jaunes et verts plus foncés, ce qui montre que l’évaluation de la teinte moyenne est insuffisante et que l’histogramme de couleur à plusieurs valeurs doit être pris en compte (Figure 7). Ces changements phénotypiques décrivent la réponse d’une plante à l’environnement, et des techniques supplémentaires telles que les mesures des échanges gazeux, l’imagerie par microscopie et d’autres techniques pourraient être utilisées pour évaluer les changements physiologiques qui se produisent en réponse au stress. Ce résultat démontre la puissance de mesures quantitatives précises qui seraient autrement manquées en utilisant des mesures manuelles ou qualitatives de la santé des plantes. Ces informations sont essentielles pour la sélection et l’ingénierie des cultures résistantes au changement climatique, qui réduit considérablement les rendements mondiaux des cultures et devrait s’aggraver dans les années à venir.
Déclarations de divulgation
Aucun.
Remerciements
Ce travail a été mené dans la plateforme de phénotypage du Donald Danforth Plant Science Center (RRID : SCR_019049), qui a fourni un soutien financier à K.M.M. et J.G.D. Ce travail a été soutenu par la subvention de l’Institut de recherche sur la location de voiture d’entreprise du Donald Danforth Plant Science Center. D.S. a été financé par la subvention globale du Taylor Geospatial Institute et une subvention (ED21HDQ0240072) du US Economic Development Administration Center for AgTech and Applied Location Science and Technology (CATALST). Nous remercions Kevin Reilly et Kris Haines (Integrated Plant Growth Facility, Donald Danforth Plant Science Center, RRID : SCR_024902) pour leur expertise et leur aide dans la croissance des plantes. Nous remercions Noah Fahlgren, Malia Gehan et Haley Schuhl pour leurs conseils sur PlantCV.
matériels
| Name | Company | Catalog Number | Comments |
| 15W USB-C Power Supply | CanaKit | DCAR-RSP-3A5-C | |
| 25604 UC-E6 USB Cable | Nikon | 18208256044 | |
| 3.5 inch pots | Hummert International | 13006000 | |
| Berger BM7 35% Bark HP | Hummert International | 10121500 | |
| Biodock 2024 | AI Software Platform | www.biodock.ai. | |
| Code editing software | Jupyter | https://jupyter.org/install | |
| Code editing software | Visual Studio Code | https://code.visualstudio.com/download | |
| Color Card ColorChecker Classic | Calibrite | CCC | |
| Color Card ColorChecker Passport | Calibrite | CCPP2 | |
| Conviron Growth Chamber | Conviron | BDW80 model | |
| Dell 22 Monitor | Dell | SE2222H | |
| EH-67 AC Adapter | Nikon | AZ185 | |
| Image analysis software | PlantCV | https://plantcv.readthedocs.io/en/latest/installation/ | |
| Jack's Water Soluble Fertilizer 15-5-15 | Hummert International | 7590200 | liquid fertilizer |
| Keyboard and Mouse | Logitech | 1440638 | MK270 wireless keyboard and mouse |
| Maize seed | USDA-ARS Germplasm Resources Information Network (GRIN) | B73 | https://www.ars-grin.gov/ |
| Micro HDMI to HDMI cable | Amazon | B07KSDB25X | |
| Nikon Coolpix B500 camera | Nikon | ||
| Nikon D7200 camera | Nikon | ||
| Osmocote 14-14-14 | Hummert International | 7630000 | |
| Photography-grade fabric | Yayoya | Polyester Fabric Black Photo Backdrop for Photo Video Studio Televison | |
| Raspberry Pi 4 Case with heatsink | iUniker | B07ZVKN262 | |
| Raspberry Pi 4 Model B Rev 1.1 | Raspberry Pi Foundation | RP4B4GB_BP | |
| Raspberry Pi 4 PiSwitch (USB-C) | CanaKit | RSP-PISWITCH-USBC | |
| Raspberry Pi software | Raspberry Pi Imager | https://www.raspberrypi.com/software/ | |
| Statistical analysis software | Rstudio | https://posit.co/downloads/ | |
| Tripod | K&F Concept | T255A3+BH-28L | |
| Turface MVP | Hummert International | 10240000 | |
| Wireless Barcode Scanner | Netumscan | B08X4NMX2M | 1D and 2D barcode scanner |
Références
- Xiao, Q., Bai, X., Zhang, C., He, Y. Advanced high-throughput plant phenotyping techniques for genome-wide association studies: A review. J Adv Res. 35, 215-230 (2022).
- Mutka, A. M., et al. image-based phenotyping methods provide insight into spatial and temporal dimensions of plant disease. Plant Physiol. 172 (2), 650-660 (2016).
- Mutka, A. M., Bart, R. S. Image-based phenotyping of plant disease symptoms. Front Plant Sci. 5, 734 (2014).
- Fahlgren, N., Gehan, M. A., Baxter, I. Lights, camera, action: High-throughput plant phenotyping is ready for a close-up. Curr Opin Plant Biol. 24 (2), 93-99 (2015).
- Das Choudhury, S., Samal, A., Awada, T. Leveraging image analysis for high-throughput plant phenotyping. Front Plant Sci. 10, 508 (2019).
- Yang, W., et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives. Mol Plant. 13 (2), 187-214 (2020).
- Omari, M. K., et al. Digital image-based plant phenotyping: A review. Korean J Agric Sci. 47 (1), 119-130 (2020).
- Schindelin, J., et al. Fiji: An open-source platform for biolFiogical-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gehan, M. A., et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping. PeerJ. 5, e4088 (2017).
- Murphy, K. M., Ludwig, E., Gutierrez, J., Gehan, M. A. Deep learning in image-based plant phenotyping. Annu Rev Plant Biol. 75 (1), 771-795 (2024).
- Fahlgren, N., et al. A versatile phenotyping system and analytics platform reveals diverse temporal responses to water availability in Setaria. Mol Plant. 8 (3), 1520-1535 (2015).
- Yu, L., et al. Development of a mobile, high-throughput, and low-cost image-based plant growth phenotyping system. bioRxiv. , (2023).
- Atefi, A., Ge, Y., Pitla, S., Schnable, J. Robotic technologies for high-throughput plant phenotyping: Contemporary reviews and future perspectives. Front Plant Sci. 12, 611940 (2021).
- Berry, J. C., Fahlgren, N., Pokorny, A. A., Bart, R. S., Veley, K. M. An automated, high-throughput method for standardizing image color profiles to improve image-based plant phenotyping. PeerJ. 6, e5727 (2018).
- Tovar, J. C., et al. Raspberry Pi-powered imaging for plant phenotyping. Appl Plant Sci. 6, e1031 (2018).
- Jolles, J. W. Broad-scale applications of the Raspberry Pi: A review and guide for biologists. Methods Ecol Evol. 12 (5), 1562-1579 (2021).
- Watt, M., et al. Phenotyping: New windows into the plant for breeders. Annu Rev Plant Biol. 71 (1), 689-712 (2020).
- Xie, C., Yang, C. A review on plant high-throughput phenotyping traits using UAV-based sensors. Comput Electron Agric. 178, 105731 (2020).
- Czedik-Eysenberg, A., et al. The "PhenoBox", a flexible, automated, open-source plant phenotyping solution. New Phytol. 219 (4), 808-823 (2018).
- Sumner, J., Fahlgren, N., Murphy, K. M. PCVR: An R package and tutorials for guided statistical analysis of plant phenotyping data. Authorea Preprints. , (2023).
- Schnable, P. S., et al. The B73 maize genome: complexity, diversity, and dynamics. Science. 326 (5956), 1112-1115 (2009).
- Kirillov, A., et al. Segment Anything. , 3992-4003 (2023).
- Gabaldón-Leal, C., et al. Modelling the impact of heat stress on maize yield formation. Field Crops Res. 198 (3), 226-237 (2016).
- Intergovernmental Panel on Climate Change. . SYNTHESIS REPORT OF THE IPCC SIXTH ASSESSMENT REPORT (AR6). , (2023).
- Quiñones, A., et al. Optimized methods for applying and assessing heat, drought, and nutrient stress of maize seedlings in controlled environment experiments. Cold Spring Harb Protoc. , (2024).
- Murphy, K. M., et al. Maize abiotic stress treatments in controlled environments. Cold Spring Harb Protoc. , (2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon