Method Article
Bildgebung und Analyse zur Quantifizierung der abiotischen Stressphänotypen von Mais (Zea mays)
In diesem Artikel
Zusammenfassung
Es wird eine Methode für die kostengünstige Bildgebung von Pflanzen zur Messung von Phänotypen vorgestellt, zusammen mit Best Practices für die Bilderfassung und einer Bildanalyse-Pipeline zur Quantifizierung von Pflanzenmerkmalen. Diese Methoden wurden angewendet, um die Phänotypen von Mais (Zea mays) unter Hitze-, Trockenheits- und kombinierten abiotischen Stressbedingungen zu messen.
Zusammenfassung
Die Messung quantitativer Pflanzenphänotypen oder -merkmale ist unerlässlich, um die Reaktionen von Pflanzen auf Umweltfaktoren, einschließlich biotischer und abiotischer Stressfaktoren, zu verstehen. Herkömmliche Methoden zur Messung von Pflanzen können jedoch zeitaufwändig, ungenau und zerstörerisch sein, insbesondere wenn es darum geht, Merkmale wie Biomasse und Höhe individuell zu bewerten. Die bildbasierte Analyse bietet die Möglichkeit, den Arbeitsaufwand zu reduzieren, die Genauigkeit und Präzision zu verbessern und wiederholte Messungen derselben Anlage über mehrere Zeitpunkte hinweg zu ermöglichen. Hochdurchsatz-Phänotypisierungseinrichtungen bieten zwar eine Lösung, sind aber oft unerschwinglich teuer und weltweit nur begrenzt verfügbar. Es wird ein Verfahren zur Aufnahme von Pflanzenbildern mit kostengünstigen Einplatinencomputern und Digitalkameras innerhalb eines Fotostudio-Setups beschrieben, begleitet von einer Bildanalyse-Pipeline auf Basis des kostenlosen Open-Source-Pakets PlantCV. Diese Methode bietet ein schnelles und genaues Protokoll für die Bilderfassung, Beschriftung und Übertragung sowie Best Practices für die Sicherstellung einer qualitativ hochwertigen Bildaufnahme. Durch die Bildanalyse wurden mehr als 10 Phänotypen für alle Bilder im Experiment gleichzeitig quantifiziert, darunter Merkmale wie Pflanzengröße, -größe und -farbe. Diese Methoden wurden verwendet, um die Reaktion der Zea mays (Mais) Inzuchtsorte B73 auf Hitze, Trockenheit und kombinierte Hitze und Trockenheit unter abiotischen Stressbedingungen zu charakterisieren.
Einleitung
Die Messung von Pflanzenmerkmalen oder Phänotypen ist entscheidend für die Beantwortung grundlegender und angewandter Fragen in der Pflanzenwissenschaft. Zum Beispiel muss ein Forscher, der die Auswirkungen einer genetischen Mutation auf die Pflanzengröße verstehen möchte, die Pflanzenhöhe und die Blattfläche quantitativ messen1. Ein Forscher, der einen biotischen Stress (wie eine bakterielle, virale oder Pilzinfektion) oder einen abiotischen Stress (wie Dürre, Hitze, Kälte oder Nährstoffmangel) anwendet, muss die Auswirkungen des Stresses auf die Pflanzenfarbe oder das "Grün" untersuchen, um die Chlorosezu messen 2,3.
Während die Messung dieser Merkmale sowohl für die grundlegende Pflanzenbiologie als auch für die angewandte Pflanzenzüchtung unerlässlich ist, kann die Messung dieser Merkmale von Hand zeitaufwändig, ungenau und ungenau sein 4,5,6,7. Zum Beispiel beinhaltet das Messen der Höhe von Pflanzen die manuelle Verwendung eines Lineals für jede Pflanze, oft unter heißen oder feuchten Umgebungsbedingungen, und unterliegt Fehlern aus der Sicht der messenden Person sowie dem Punkt, der als "Spitze" der Pflanze angesehen wird. Darüber hinaus sind Messungen wie Biomasse und Pigmentgehalt zerstörerisch, und dieselbe Pflanze kann nicht auf Veränderungen im Laufe der Zeit gemessen oder für Mehrfachprobenahmen verwendet werden, da sie bei der Durchführung dieser Messungen zerstört werden muss 4,5,6,7. Die zeitaufwändige und fehleranfällige Natur dieser Messungen behindert den Fortschritt bei der Bewertung von Pflanzen auf ihre phänotypischen und physiologischen Stressreaktionen.
Das Aufnehmen von Bildern von Pflanzen und das Messen quantitativer Merkmale aus dem Bild bietet Lösungen für diese Probleme 4,5,6,7. Die Bildanalyse verbessert die Genauigkeit und Präzision und reduziert gleichzeitig die Zeit, die ein Forscher für die Messungen benötigt, und dieselbe Pflanze kann aufgrund der zerstörungsfreien Merkmalsquantifizierung mehrmals abgebildet oder für andere Probenahmen verwendet werden 4,5,6,7. Darüber hinaus können Bilder gespeichert und erneut analysiert werden, lange nachdem ein Experiment abgeschlossen ist und die Pflanzen entsorgt wurden 4,5,6,7. Die Bildanalyse kann individuell mit Bildbearbeitungswerkzeugen mit niedrigem Durchsatz wie ImageJ (Fidschi)8 oder mit höherem Durchsatz mit Computer Vision und maschinellem Lernen mit Plattformen wie PlantCV9 durchgeführt werden. PlantCV ist eine kostenlose Open-Source-Bildanalyseplattform, die es Forschern ermöglicht, eine Pflanze oder ein anderes Objekt zunächst vom Hintergrund eines Bildes zu maskieren (oder zu trennen) und Messungen von Größe, Morphologie, Farbe und mehr dieser Pflanze oder dieses Objektsvorzunehmen 9. Unter Verwendung eines modularen Workflows erstellt ein Forscher zunächst einen flexiblen Workflow (oder eine Pipeline) aus verschiedenen Modulen mit einem Beispielbild und unterzieht dann die verbleibenden Bilder dem Hochdurchsatz-Workflow, ohne mit jedem einzelnen Bild zu interagieren9. Quantitative Merkmale werden ausgegeben und auf statistisch signifikante Unterschiede zwischen den Gruppen analysiert. Deep-Learning-Pipelines können für eine effiziente, automatisierte Bildanalyse entwickelt werden, nachdem genügend Bilder erfasst und mit geeigneten Merkmalsmessungen oder Metadaten gekennzeichnet wurden10.
Die "Hochdurchsatz"-Phänotypisierung kann die Geschwindigkeit erhöhen und den menschlichen Arbeitsaufwand während der Datenerfassung und/oder während der Datenanalyse reduzieren 4,5,6,7. Hochdurchsatz-Phänotypisierungsanlagen haben die Automatisierung der Bilderfassung in kontrollierten Umgebungen ermöglicht, in denen sich Pflanzen auf Förderbändern oder über Roboter zu einer Bildgebungsstation bewegen und automatisch mit dem Namen (d. h. Etikett mit Metadaten) versehen werden, der der Pflanzezugeordnet ist 6,11,12,13,14 . Während diese Förderbänder oder robotergestützten Einrichtungen die Erfassung und Beschriftung großer Mengen von Bildern ohne menschliches Eingreifen ermöglichen, gibt es nur wenige auf der Welt, und ihr Bau kann Millionen von Dollar kosten, was sie für viele Forscher unzugänglich macht. Um diese Konzepte der automatisierten Bildgebung zu geringeren Kosten anzuwenden, sind Raspberry Pi (hier als Einplatinencomputer, SBC bezeichnet) kostengünstige Computer (weniger als 35 US-Dollar pro Stück), die zum Erfassen, Benennen, Speichern und Übertragen von Bildern von Pflanzen verwendet wurden, die in kontrollierten Umgebungen angebaut wurden. Diese Setups erfassen Bilder in automatisierten, zeitgesteuerten Intervallen mit einem Array von bis zu 72 Raspberry Pi 12,15,16. Obwohl sie kostengünstiger sind als Förderbandsysteme, die Millionen von Dollar kosten können, eignen sich die zuvor beschriebenen SBC-Wachstumskammern am besten für eine Top-Down-Ansicht des Anbaus kleiner Pflanzen und erfordern immer noch zahlreiche SBC15. In Feldumgebungen erfassen Portale, UAS (unbemannte Flugsysteme) wie Drohnen und Satelliten Bilder zur Analyse von Pflanzenmerkmalen17,18. Sowohl Feldsysteme als auch Einrichtungen mit kontrollierter Umgebung mit hohem Durchsatz können in Bezug auf die Anschaffungskosten, die Wartung und das Expertenwissen, die für den Aufbau und die Wartung der Systeme sowie für die Analyse von Bildern erforderlich sind, unerschwinglich sein17,18. Mobiltelefone und Digitalkameras, die allein für die Pflanzenfotografie verwendet werden, stellen zeitaufwändige Herausforderungen bei der Bildübertragung und Benennung mit Metadaten dar, die für die nachgelagerte Bildanalyse unerlässlich sind.
Die hier beschriebenen Methoden stellen eine kostengünstige Lösung für die Pflanzenbildgebung für die Herausforderungen dar, die von anderen Systemen nicht gelöst werden. Ein Fotografie-Setup, bei dem die Pflanzen einzeln ins Studio transportiert werden, ermöglicht die effiziente Vermessung von Pflanzen aller Größen in Töpfen. Bei Verwendung eines SBC, der an eine Digitalkamera angeschlossen ist, ermöglicht diese Methode zunächst die standardisierte Aufnahme, Beschriftung und Übertragung von Bildern. Durch die Verwendung einer Tastatur oder eines QR-/Barcode-Scanners zur Benennung von Bildern während der Erfassung wird die zeitaufwändige und fehleranfällige Aufgabe des Umbenennens von Bildern mit Metadaten nach der Erfassung und Speicherung umgangen. Die Komposition des Bildes und die empfohlenen Kameraeinstellungen sind wichtig, um eine nachgelagerte Bildanalyse zu ermöglichen. Schließlich wird eine Bildanalyse-Pipeline in PlantCV bereitgestellt, um quantitative Merkmale wie Blattfläche, Pflanzenhöhe, -breite und -farbe aus den aufgenommenen Bildern zu extrahieren.
Im Vergleich zu Förderbandsystemen ist diese Methode kostengünstig und erfordert einen einzigen SBC. Das hier beschriebene Phänotypisierungs-Setup wurde entwickelt, um die Einschränkungen und Kosten eines Förderbandes oder von Robotersystemen zu beseitigen - Pflanzen jeder Größe können gemessen werden, da das Setup vom Wachstumsraum getrennt werden kann, nur ein einziger SBC benötigt wird und es mit zahlreichen Kameratypen für maximale Flexibilität und Erschwinglichkeit funktioniert. Ähnliche Ansätze wie der hier vorgestellte sind die PhenoBox oder das PhenoRig12,19. Im Vergleich zu manuellen Messungen spart diese Methode Zeit, reduziert Fehler und zerstört die Pflanzen nicht 4,12,15,19.
Protokoll
Einzelheiten zu den Reagenzien und der in diesem Protokoll verwendeten Ausrüstung sind in der Materialtabelle aufgeführt.
1. Vorbereitung des Fotostudios
- Hängen Sie schwarzen Stoff in Fotoqualität an eine Wand oder Decke, um Bilder in der Seitenansicht zu machen. Drapieren Sie den Stoff mindestens 0,5 m auf den Boden, so dass die Pflanze auf dem Stoff platziert wird (Abbildung 1).
HINWEIS: Der Stoff sollte mindestens 100 cm höher und breiter sein als die abzubildende Pflanze. - Platzieren Sie eine Farbkarte auf dem Hintergrund, die gegen ein Objekt wie eine Box gelehnt ist, sodass sie im Rahmen sichtbar ist (Abbildung 1). Für Pflanzen, die größer als 30 cm hoch oder breit sind, verwenden Sie eine Farbkarte, die mindestens 20 cm breit ist. Für Pflanzen, die kleiner als 30 cm hoch oder breit sind, verwenden Sie eine Farbkarte, die zwischen 4 und 8 cm breit ist.
- Schließen Sie eine SD-Karte an einen Laptop oder Desktop-Computer an. Laden Sie den Raspberry Pi Imager aus dem Softwarebereich auf der Website herunter (https://www.raspberrypi.com/software/) und laden Sie die bereitgestellte raspberrypi-image-capture.img.gz Datei (https://github.com/danforthcenter/photo-maize-paper) auf den Laptop oder Desktop-Computer herunter.
- Starten Sie den Imager, indem Sie die heruntergeladene Imager-Datei (.exe) öffnen. Klicken Sie auf die Schaltfläche Betriebssystem auswählen und wählen Sie unten die Schaltfläche Benutzerdefiniert verwenden . Wählen Sie die image-capture.img.gz Datei aus.
- Klicken Sie anschließend auf die Schaltfläche Speicher auswählen und wählen Sie die SD-Karte aus, die an den Computer angeschlossen ist. Klicken Sie auf die Schaltfläche Schreiben .
HINWEIS: Stellen Sie sicher, dass die richtige SD-Karte ausgewählt wurde, da bei diesem Vorgang alle auf der Karte gespeicherten Daten gelöscht werden.
- Klicken Sie anschließend auf die Schaltfläche Speicher auswählen und wählen Sie die SD-Karte aus, die an den Computer angeschlossen ist. Klicken Sie auf die Schaltfläche Schreiben .
- Schließen Sie die neueste Version von SBC über ein USB-Kabel an eine Tastatur, über ein HDMI-Kabel an einen externen Monitor und über ein Netzkabel mit einem Schalter an eine Steckdosenleiste an. Schließen Sie den Monitor über das mit dem Monitor gelieferte Netzkabel an die Steckdosenleiste an. Legen Sie die SD-Karte in den SBC ein. In Abbildung 1 finden Sie ein beschriftetes Schema des Setups.
- Schalten Sie die Steckdosenleiste ein, indem Sie den Schalter an der Steckdosenleiste drücken, schalten Sie den Monitor mit der Taste am Monitor ein und schalten Sie dann den SBC ein, indem Sie den Schalter am Netzkabel auf "Ein" stellen.
- Verbinden Sie den SBC über WLAN oder ein Ethernet-Kabel mit dem lokalen Internet.
- Wenn Fotos direkt auf einen Server oder Cloud-Speicher übertragen werden sollen:
- Bearbeiten Sie die Datei "CAPTURE-PHOTO.py" auf dem SBC so, dass sie den Dateipfad enthält, um die Übertragung abzuschließen.
- Bearbeiten Sie die Datei win-credentials mit dem Pfad und dem Passwort für den Server oder Cloud-Speicher mit dem folgenden Befehl im Terminal: sudo nano /etc/win-credentials.
HINWEIS: Wenden Sie sich an den Internet-Systemadministrator, um sicherzustellen, dass der Ordner über die Berechtigung zum Übertragen von Dateien verfügt.
- Verbinden Sie einen drahtlosen Barcode-Scanner mit dem SBC gemäß den Anweisungen des Herstellers, die mit dem Barcode-Scanner geliefert werden, entweder über USB oder Bluetooth.
- Schalten Sie den SBC aus, indem Sie auf die Schaltfläche Raspberry Pi oben links auf dem Bildschirm klicken, auf Abmelden klicken und dann auf Herunterfahren klicken, um den SBC sicher auszuschalten. Schalten Sie nach dem Herunterfahren den SBC-Netzschalter auf "ein", das Netzkabel auf "aus" und die Steckdosenleiste auf "aus".
2. Pflanzenwachstum und Stressbehandlung
HINWEIS: Jede Pflanze ist für diese Methode der Phänotypisierung geeignet; Diese Pflanzenwachstumsmethoden sind auf die hier vorgestellten repräsentativen Ergebnisse anwendbar.
- Besorgen Sie sich Saatgut von B73 oder einer anderen Maissorte von einem Lagercenter.
- Bereiten Sie Bodenmedien und Wachstumsbedingungen vor:
- Füllen Sie 3,5-Zoll-Töpfe (0,7 l) mit einer 1:1-Volumenmischung aus Turface MVP und Berger BM7-35 mit 3 lb/Kubikmeter Langzeitdünger 14-14-14 (N-P-K). Stellen Sie die Töpfe in ein Tablett mit Löchern und gießen Sie es gründlich mit Umkehrosmosewasser (RO).
- Stellen Sie eine Wachstumskammer auf die folgenden Umgebungsbedingungen ein: 30 °C am Tag/20 °C in der Nacht, 16 h Tageslänge, 60 % Luftfeuchtigkeit und tagsüber 500 mmol.m-2 s-1 Licht auf Höhe des Topfes. Stellen Sie eine separate Wachstumskammer mit den gleichen Bedingungen ein, außer 38 °C am Tag / 28 °C in der Nacht für Hitzestress.
- Pflanzen Sie 20 Maissamen, jedes in einen 3,5-Zoll-Topf, indem Sie ~2 cm tief in die Erde stecken und sie mit der verdrängten Erde bedecken. Gießen Sie gründlich mit RO-Wasser und stellen Sie es in die Kontrollwachstumskammer. Täglich mit Flüssigdünger gießen (gemischt nach Herstellerangabe; siehe Materialtabelle).
HINWEIS: Das Pflanzen zusätzlicher Pflanzen, die für das Experiment benötigt werden, führt zu einer schlechten Keimung und mangelnder Gleichmäßigkeit vor Beginn einer Stressanwendung. Die Keimrate variiert je nach Sorte. - Nach 21 Tagen Wachstum verpflanzen Sie 16 der gleichmäßigsten Pflanzen (n = 4 Pflanzen pro Behandlung) in 3,79-Liter-Töpfe, die mit der gleichen Blumenerdemischung gefüllt und mit RO-Wasser vorbefeuchtet wurden. Bringen Sie die Pflanzen in die Kontrollwachstumskammer zurück.
- Am 25. Tag (4 Tage nach dem Umpflanzen) bringst Du die Hälfte der Pflanzen (n = 8) in die Hitzestresskammer, wenn die Lichter in der Kammer angehen. Hören Sie auf, die trockengestressten Pflanzen in beiden Kammern zu gießen (n = 4 pro Kammer).
HINWEIS: Pflanzen mit Trockenstress müssen möglicherweise gegossen werden, damit die Pflanzen bis zum Ende des Versuchs überleben. Hier wurden Trockenpflanzen an Tag 31 und Tag 37 mit Düngerwasser bewässert. - Am 38. Tag (13 Tage nach Beginn des Stresses) nehmen Sie die Pflanzen aus den Wachstumskammern für die Bildgebung.
3. Bildgebung von Pflanzen
- Starten Sie den SBC (siehe Abbildung 2):
- Schalten Sie die Steckdosenleiste mit dem Schalter an der Steckdosenleiste ein.
- Schalten Sie den Monitor mit der Taste am Monitor ein.
HINWEIS: Wenn der SBC eingeschaltet wird, bevor der Monitor eingeschaltet wird, wird er nicht korrekt gestartet. - Drehen Sie den Schalter am SBC-Netzkabel auf "Ein".
HINWEIS: Dadurch wird der SBC eingeschaltet und er wird auf dem Monitor hochgefahren. Am SBC selbst befindet sich kein "Ein"-Schalter, sondern nur am Netzkabel.
- Stellen Sie die Kamera auf ein Stativ oder einen stabilen Tisch.
HINWEIS: Die gesamte Pflanzen- und Farbkarte sollte auf dem Kamerabildschirm sichtbar sein. Die Kamera sollte senkrecht zum Boden stehen und einen direkten Blick (kein Winkel) auf das Zentrum der Pflanze haben. Die Kamera muss mit dem gphoto2-Paket kompatibel sein, und eine Liste finden Sie hier: http://www.gphoto.org/proj/libgphoto2/support.php. - Versorgen Sie die Kamera über ein Netzkabel mit der Kamera oder den Batterien mit Strom. Schalten Sie die Kamera mit der Taste an der Kamera ein.
- Schließen Sie die Kamera über das mit der Kamera gelieferte Datenübertragungskabel an den USB-Anschluss des SBC an.
- Ordnen Sie die Pflanze in der Fotografie an:
- Lege ein Stück dunkles Klebeband auf den Stoff in Fotoqualität, wo der Topf platziert wird, um sicherzustellen, dass die Platzierung für jede Pflanze gleich ist. Stellen Sie sicher, dass die Farbkarte mit dem Klebeband übereinstimmt.
HINWEIS: Platzieren Sie die Farbkarte mindestens 10 cm vom Rand der Pflanze entfernt. Während der Qualitätskontrolle im PlantCV-Workflow (unten) sollte das Bild so beschnitten werden können, dass es nur die Pflanze und den Topf, aber nicht die Farbkarte enthält, um sicherzustellen, dass die Farbchips nicht in die endgültige Merkmalsextraktion einbezogen werden. In Abbildung 2 finden Sie Beispiele für Bilder mit hoher und niedriger Qualität. - Stellen Sie den Topf auf das Stück Klebeband. Bei Pflanzen, die in einer Ebene wachsen, wie z. B. Mais (Zea mays) und Sorghum (Sorghum bicolor), richten Sie die Pflanze aus dem weitesten Winkel in Richtung Kamera. Bei Pflanzen, die nicht in einer Ebene wachsen, erfassen Sie 0- und 90-Grad-Winkel der Pflanze und mitteln Sie die resultierenden quantitativen Messungen nach Abschluss der Bildanalyse.
- Lege ein Stück dunkles Klebeband auf den Stoff in Fotoqualität, wo der Topf platziert wird, um sicherzustellen, dass die Platzierung für jede Pflanze gleich ist. Stellen Sie sicher, dass die Farbkarte mit dem Klebeband übereinstimmt.
- Öffnen Sie auf dem SBC-Desktop-Bildschirm, der auf dem Monitor angezeigt wird, das Terminal, indem Sie auf die Terminalschaltfläche in der oberen linken Menüleiste klicken.
- Wenn Sie direkt auf einem Server oder Cloud-Speicher speichern, mounten Sie den Speicher mit dem folgenden Befehl, indem Sie den erforderlichen Cloud-Speicherpfad (cloud-storage-path im folgenden Beispiel) und das Kennwort eingeben, die Codezeile in das geöffnete Terminalfenster eingeben und die Eingabetaste auf der Tastatur drücken. Wenn sie gegenüber den Werkseinstellungen unverändert ist, lautet die Benutzer-ID (UID) "pi".
ANMERKUNG:
sudo mount -t cifs -o credentials=/etc/win-credentials,uid=pi //cloud-storage-path /mnt/cloud-storage - Geben Sie den folgenden Code in das Terminal ein und drücken Sie die Eingabetaste auf der Tastatur. Ein neues Fenster wird angezeigt.
./RUN-PHOTOSTUDIO-SHELL.sh
HINWEIS: Die RUN-PHOTOSTUDIO-SHELL.sh Datei ruft die CAPTURE-PHOTO.py Datei auf, die vorinstalliert auf den Pi geladen wird, wenn sie mit der obigen Methode geladen wurde. Alternativ wurden diese Skripte auf GitHub bereitgestellt, um sie auf eine vorhandene Raspberry Pi SD-Karte zu legen. - Es öffnet sich ein Fenster mit der grafischen Benutzeroberfläche. Geben Sie den Namen des Bildes ein oder scannen Sie den Barcode oder QR-Code der Anlage mit einem Barcode-Scanner.
- Wählen Sie eine Option aus, um das Bild lokal oder auf einem bereitgestellten Server/Cloud-Speicher zu speichern.
- Klicken Sie hier, um ein Foto zu machen! Knopf.
- Wenn Sie den lokalen Speicher ausgewählt haben, öffnen Sie den Ordner PHOTOS auf dem Desktop, um das Bild zu überprüfen. Wenn Sie einen Server oder Cloud-Speicher ausgewählt haben, öffnen Sie das Bild an diesem Speicherort.
- Passen Sie bei Bedarf die Einstellungen an der Kamera an, indem Sie sie vom SBC trennen, die Anpassung gemäß den Anweisungen des Herstellers vornehmen und wieder an den SBC anschließen.
HINWEIS: Einstellungen wie Belichtung, Helligkeitskontrast usw. müssen je nach Beleuchtung im Raum und dem abzubildenden Objekt möglicherweise angepasst werden. Ein häufiger Fehler besteht darin, die Bilder zu überbelichten. Um die Belichtung eines Testbildes zu überprüfen, durchlaufen Sie das Testbild durch den PlantCV-Bildanalyse-Workflow, der eine Qualitätskontrolle für die Belichtung umfasst. Passen Sie die Kameraeinstellungen nach Bedarf an, um die Belichtung zu verringern oder zu erhöhen, bis die Histogramme richtig verteilt sind. - Nachdem alle Bilder erfasst wurden, übertragen Sie die Bilder vom SBC auf einen lokalen Computer oder Cloud-Speicher, indem Sie die bevorzugte Methode verwenden, z. B. USB-Speicher, Internetbrowser oder SSH-Übertragung.
4. Extraktion von Merkmalen aus Bildern
- Laden Sie die folgenden Dateien auf einen lokalen Computer oder Server herunter: "photo-studio-SV-notebook.ipynb", "workflow.py" und "photo-studio-SV-config.json". Alle Dateien sind auf GitHub (https://github.com/danforthcenter/photo-maize-paper) verfügbar.
- Installieren Sie PlantCV auf dem lokalen Computer oder Server mit den beschriebenen Schritten (https://plantcv.readthedocs.io/en/latest/installation/).
- Öffnen Sie die Datei photo-studio-SV-notebook.ipynb mit dem bevorzugten Code-Editor, z. B. JupyterLab oder Visual Studio Code.
- Führen Sie jeden Codeblock aus und bearbeiten Sie ihn nach Bedarf unter Berücksichtigung der im Codeblock beschriebenen Parameter, um eine saubere Maske nur der Pflanze im Bild zu erhalten.
- Wenn Sie mit der Analyse des Beispielbilds zufrieden sind, öffnen Sie die Datei workflow.py im bevorzugten Code-Editor. Bearbeiten Sie diese Datei mit den in "photo-studio-SV-notebook.ipynb" geänderten Parametern und speichern Sie sie.
- Öffnen Sie die Datei photo-studio-SV-config.json und bearbeiten Sie die Dateipfade, um zu einem Ordner mit Eingabebildern, einem gewünschten Ausgabeordner usw. zu leiten, wie in der Datei angegeben.
- Öffnen Sie ein Terminal und führen Sie den folgenden Code aus, um PlantCV zu aktivieren. Geben Sie dazu den Code conda activate plantcv in das Terminal ein und drücken Sie die Eingabetaste auf der Tastatur.
- Ändern Sie die folgende Codezeile in den Dateipfad der Datei "photo-studio-SV-config.json" (dies ist die einzige Änderung, die im folgenden Code vorgenommen werden muss).
- Geben Sie im Terminal den bearbeiteten Code ein und drücken Sie die Eingabetaste auf der Tastatur. Dadurch wird der Workflow für alle Bilder ausgeführt, die im Experiment erfasst wurden.
ANMERKUNG:
plantcv-run-workflow --config /Benutzer/BENUTZERNAME/photo-studio-SV-config.json
- Geben Sie im Terminal den bearbeiteten Code ein und drücken Sie die Eingabetaste auf der Tastatur. Dadurch wird der Workflow für alle Bilder ausgeführt, die im Experiment erfasst wurden.
- Überprüfen Sie die Ausgabebilder, um festzustellen, ob der Code für alle Bilder geeignet war.
HINWEIS: Der blaue und rosa Umriss der Pflanze sollte sich an den Rändern der Pflanze befinden und es sollten keine anderen Objekte auf dem Bild zu sehen sein. Gehen Sie bei Bedarf zurück zur "photo-studio-SV-analysis.ipynb" und testen Sie problematische Bilder und bearbeiten Sie den Code, insbesondere die Maskierungsschritte und die Rauschunterdrückung, um die gewünschten Ergebnisse zu erzielen. In Abbildung 3 finden Sie Beispiele für eine Bildsegmentierung mit hoher, akzeptabler und niedriger Qualität. - Die Ausgabedatei liegt in Form einer .json Datei vor. Gehen Sie zurück zum Terminal (stellen Sie sicher, dass conda activate plantcv bereits ausgeführt wurde), geben Sie den folgenden Code ein und drücken Sie die Eingabetaste auf der Tastatur, um die Datei in .csv für die nachgelagerte statistische Analyse zu konvertieren, und ändern Sie den Dateipfad in den der Datei "results-photo-studio.json".
plantcv-utils json2csv -j /Benutzer/BENUTZERNAME/results-photo-studio.json -c /Benutzer/BENUTZERNAME/results-photo-studio.csv
HINWEIS: Um die Maße von Pixeln in eine Standardmaßeinheit wie cm umzurechnen, verwenden Sie die Umrechnung, die über die Funktion find_color_card im PlantCV-Workflow bereitgestellt wird. - Führen Sie eine statistische Analyse der quantitativen Ausgabemerkmale durch, um Unterschiede in den Pflanzenphänotypen aufgrund verschiedener Pflanzenarten, Sorten, Behandlungen und Zeitpunkte zu bestimmen.
HINWEIS: Das pcvr-Paket in R Studio wurde entwickelt, um die Ausgaben von PlantCV auf statistische Signifikanz und Darstellung der Ergebnissezu analysieren 20. In Abbildung 4 finden Sie eine Zusammenfassung und visuelle Darstellung der in Abbildung 4 gesammelten Merkmale.
Ergebnisse
Um diese Methode zu entwickeln, wurden B73-Maispflanzen in insgesamt vier Behandlungen auf quantitative Phänotypen unter Kontrolltemperaturen, Hitzetemperaturen, gut bewässerten und Trockenheitsbedingungen sowie deren Kombinationen untersucht. B73 ist eine bekannte Inzuchtmaissorte mit einer Fülle genetischer Daten21.
Bei der Bildanalyse in PlantCV wurden die Bilder erfolgreich farbkorrigiert, die quantitativen Ergebnisse mit den Pflanzenmetadaten gekennzeichnet und die Pflanze vom Hintergrund segmentiert (d. h. vom Hintergrund getrennt), wie in Abbildung 3 zu sehen ist. Eine erfolglose Analyse würde Ausgabebilder liefern, die nicht den blauen Umriss oder die rosa Form um die Außenseite der Pflanze zeigen, sondern zusätzliches Rauschen oder fehlende Teile der Pflanze enthalten (Abbildung 3, Beispiel mit geringer Qualität). Ein Zweikanal-Schwellenwert war notwendig, um so viel wie möglich von der Pflanze in die Maske aufzunehmen (siehe alternative Einkanal-Strategien in der "photo-studio-SV-notebook.ipynb"). Einige Pflanzen, insbesondere solche mit violett gefärbten Stängeln, wiesen jedoch keine perfekte Segmentierung auf, waren aber für eine nachgelagerte Analyse ausreichend (Abbildung 3).
Die PlantCV-Analyse lieferte 16 quantitative, einwertige Merkmale, die in Abbildung 4 zusammengefasst sind. Ein Einzelwertmerkmal liefert einen Wert pro Pflanze - zum Beispiel die Blattfläche, die Höhe, die Breite und den kreisförmigen Farbton. Die Blattfläche wurde mit der Funktion "analyze.shape" (Abbildung 4) betrachtet und die gesamte Pflanze berücksichtigt. Höhe und Breite wurden jedoch mit der "analyze.bound_horizontal-Funktion" berücksichtigt, wobei eine Grenze an der Bodenlinie der Pflanze gezeichnet wurde. Das liegt daran, dass Pflanzen, die über die Seite des Topfes hängen, eine größere Höhe haben, wenn man die Pflanze als Ganzes betrachtet, und stattdessen nur von der Pflanzenbasis aus messen sollten, wo sie auf die Erde trifft. Zusätzlich zu den einwertigen Merkmalen gibt PlantCV mehrwertige Merkmale aus, bei denen es sich um Histogramme handelt, insbesondere der Farbwerte, die jedes Pixel der Pflanze darstellen. Der zirkuläre Mittelwert des Farbtons ist ein Einzelwertmerkmal, das den durchschnittlichen Farbtonwert für die gesamte Pflanze darstellt.
Um zu bestimmen, welche Merkmale am interessantesten sind, wurde die Varianz, die durch die Behandlung für jedes Merkmal erklärt wird, sowie die Korrelation zwischen den verschiedenen Merkmalen mit Hilfe der Funktion "frem" im pcvr R Studio Paket20 berechnet (Abbildung 5A). Blattfläche, Höhe, Breite und kreisförmiger Mittelwert des Farbtons wurden für die nachgelagerte Analyse ausgewählt, da sie mehr als 50 % der Varianz aufgrund der Behandlung erklären und relevante Maße für das Pflanzenwachstum (Fläche, Höhe und Breite) und die Gesundheit (Farbton) sind. Repräsentative Bilder der Pflanze mit der größten und kleinsten Blattfläche zeigen die Variation der Phänotypen, die in diesem Experiment gesammelt wurden (Abbildung 5B, C). Die Pflanze mit der größten Blattfläche hatte auch den höchsten mittleren Farbton und war eine gut bewässerte, hitzegestresste Pflanze B73 (Abbildung 5B). Die kleinste Blattfläche hatte den kleinsten mittleren Farbton (Abbildung 5C) und war eine B73-Pflanze, die durch Trockenheit und Hitze gestresst war.
Die Wasseraufbereitung hatte einen signifikanten Einfluss auf die Bestimmung der Blattfläche (F(1,13) = 226,5, p = 1,32 x 10-9), der Höhe (F(1,13) = 21,1, p = 0,0005), der Breite (F(1,13) = 75,5, p = 8,92 x 10-7) und des zirkulären Mittelwerts des Farbtons (F(1,13) = 27,8, p = 0,0002), bestimmt unter Verwendung eines linearen Regressionsmodells für den Behandlungseffekt und einer bidirektionalen ANOVA in R Studio (vollständige Ergebnisse in Tabelle 1). Die Temperaturbehandlung hatte einen signifikanten Einfluss auf die Höhe (F(1,13) = 5,94, p = 0,03), aber nicht auf die Blattfläche, die Breite oder den kreisförmigen Farbton (p > 0,05, Tabelle 1). Die Wechselwirkung zwischen Temperatur und Wasserstatus war bei keinem der gemessenen Merkmale ein signifikanter Faktor (p > 0,05, Tabelle 1).
Anschließend wurden die einzelnen Behandlungen mittels linearer Regression und Post-hoc-Test (geschätzte marginale Mittelwerte) mit einer Sidak-Korrektur für Mehrfachvergleiche mit der Kontrolle und untereinander verglichen (Abbildung 6). Die Blattfläche, die Pflanzenhöhe, die Pflanzenbreite und der zirkuläre Mittelwert des Farbtons waren bei trockengestressten Pflanzen im Vergleich zu gut bewässerten Pflanzen unter beiden Temperaturbedingungen signifikant reduziert (p < 0,05, Abbildung 6). Hitzestress bei guter Bewässerung reduzierte nur die Pflanzenhöhe (p < 0,05, Abbildung 6), reduzierte aber nicht signifikant die Blattfläche, die Pflanzenbreite oder den kreisförmigen Mittelwert des Farbtons. Dieser Unterschied im Farbton kann weiter untersucht werden, indem der mehrwertige Farbton bewertet wird, der in Abbildung 7 als Histogramm und nicht nur als Durchschnitt dargestellt ist. Die Verringerung des mittleren Farbtons aufgrund von Trockenheit war auf eine Verschiebung von grünen zu gelben Pixeln zurückzuführen, die als Chlorose bekannt ist (Abbildung 7). Während die Hitze keinen signifikanten Unterschied im durchschnittlichen Farbton zeigte, zeigte sie eine Abnahme und Zunahme sowohl aufgrund der Vergilbung (Chlorose) als auch der Verdunkelung der grünen Farbe (Abbildung 7).
Nach der Bildgebung wurden die Pflanzen an der Wurzel-Spross-Verbindung geschnitten und das Gewicht des oberirdischen Gewebes erfasst. Die Wurzeln wurden vorsichtig gewaschen und mit einem Handtuch getrocknet und dann gewogen, um die gesamte pflanzliche Biomasse (auch als Gewicht bezeichnet) für den Vergleich mit der beschriebenen bildbasierten Phänotypisierungsmethode zu berechnen. Die mittels Bildanalyse gemessene Blattfläche korrelierte stark mit der pflanzlichen Biomasse (R2 = 0,84, Abbildung 8).

Abbildung 1: Bild der Einrichtung des Fotostudios mit beschrifteten Komponenten zum Sammeln von Pflanzenbildern. (A) Stoff in Fotoqualität, (B) Farbkarte, (C) Klebeband zum gleichmäßigen Platzieren von Töpfen, (D) Digitalkamera, (E) Einplatinencomputer, (F) Monitor mit Anzeige des Bildaufnahmeprozesses. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Überblick über den Imaging-Workflow. Es wird ein repräsentatives Bild "hohe Qualität" und "niedrige Qualität" bereitgestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
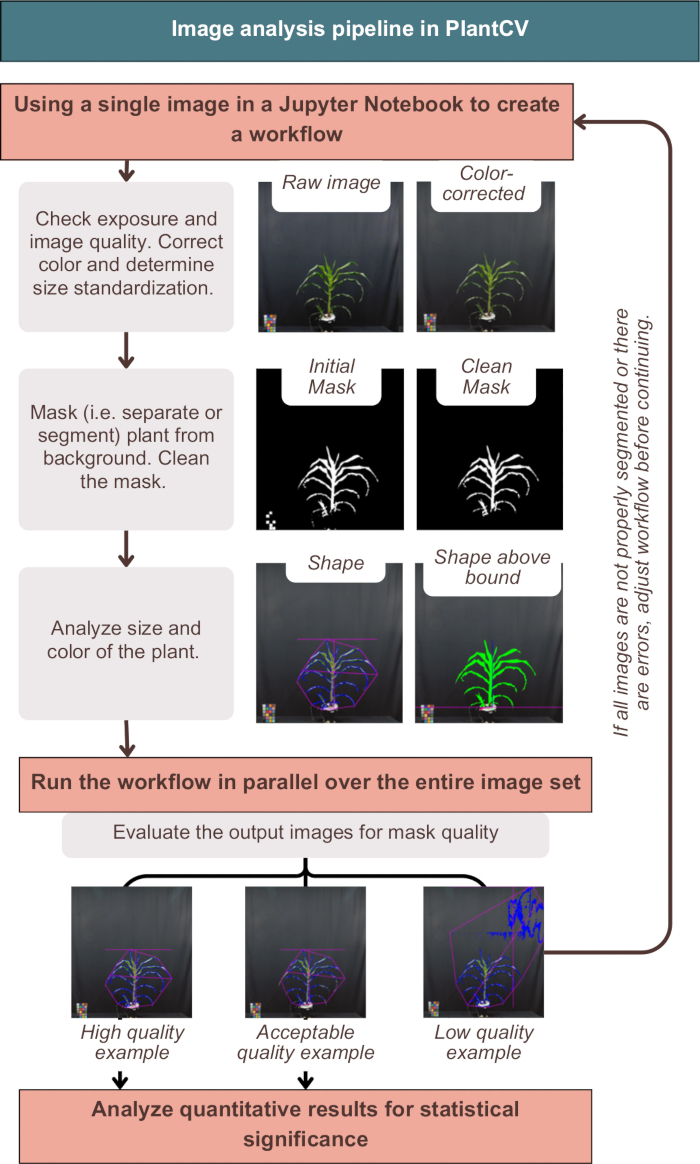
Abbildung 3: Überblick über den Workflow der Bildanalyse in PlantCV. Es werden repräsentative Bilder der Segmentierung "hohe", "akzeptable" und "niedrige" Qualität präsentiert; PlantCV-Merkmale werden durch einen rosa Umriss der gesamten Pflanzenform und einen blauen Umriss der Pflanzenfläche dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
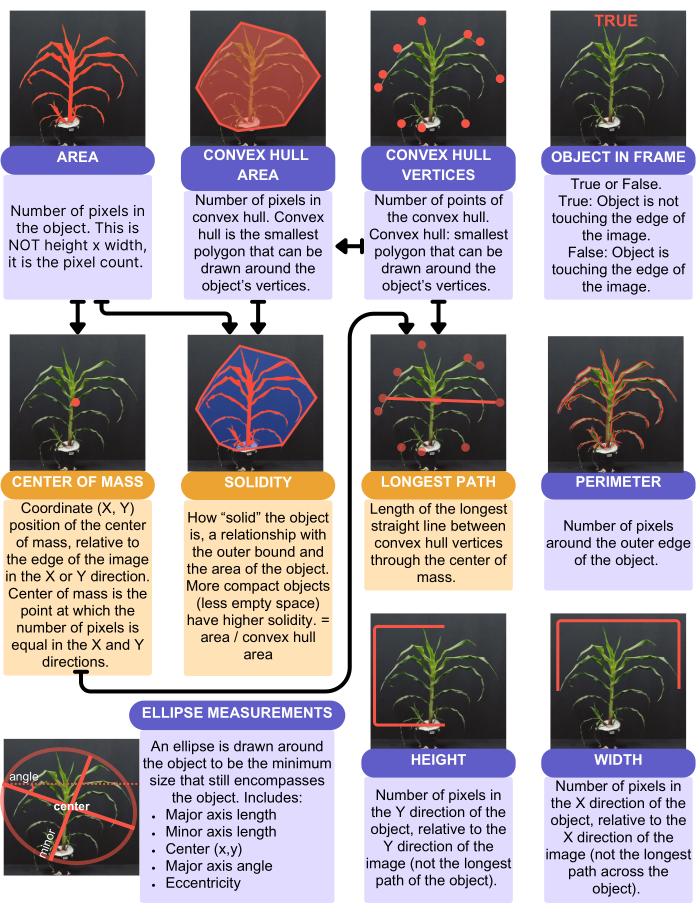
Abbildung 4: Zusammenfassung und visuelle Darstellung der aus der Analyse erfassten Formmerkmale. Shape-Funktion in PlantCV. Lila Kästchen stellen Merkmale dar, die keine andere Eigenschaft in ihrer Berechnung verwenden. Orangefarbene Kästchen stellen Merkmale dar, die andere Merkmale in ihrer Berechnung verwenden. Pfeile zeigen an, welche Merkmale in diesen Berechnungen verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
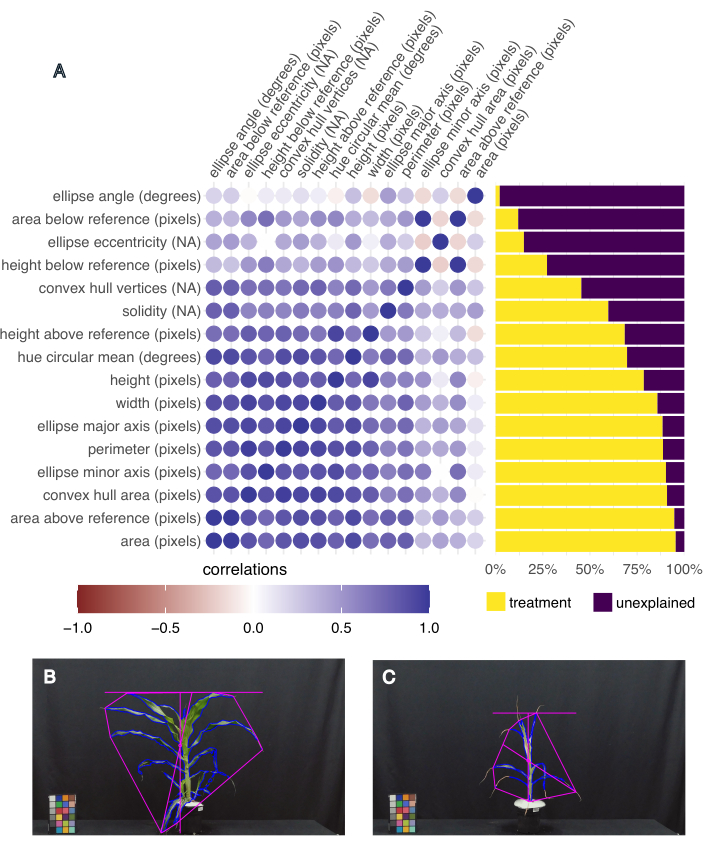
Abbildung 5: Die bildbasierte Phänotypisierung erfasst phänotypische Varianz und korrelierte Merkmale. (A) Varianz der Merkmale, die durch die Behandlung erklärt werden (rechts), und Korrelation der Merkmale untereinander (links). Jedes Merkmal ist gekennzeichnet, gefolgt von seiner Maßeinheit; "NA" bedeutet, dass es sich um ein einheitenloses Maß handelt. (B) Bild der Pflanze mit der größten Blattfläche und dem höchsten kreisförmigen Mittelwert (Grad). (C) Bild der Pflanze mit der kleinsten Blattfläche und dem geringsten Farbton im kreisförmigen Mittelwert (Grad). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
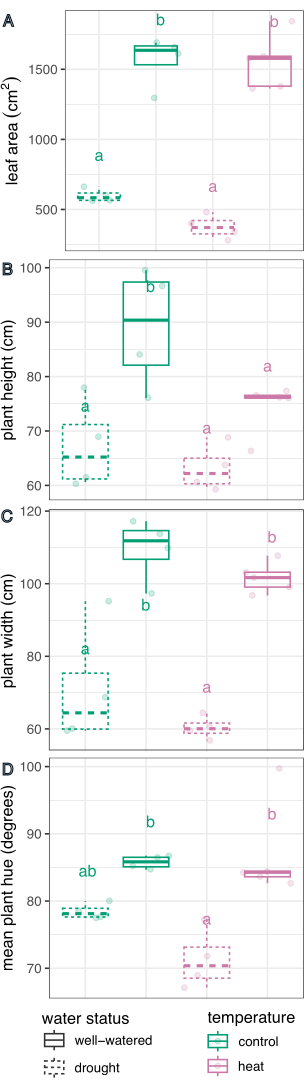
Abbildung 6: Boxplot-Vergleich der Behandlungseffekte auf pflanzliche Phänotypen. Die Blattfläche (A), die Höhe (B), die Breite (C) und der kreisförmige Mittelwert des Farbtons (D) werden für den Einfluss von Trockenheit, Hitze und den kombinierten Spannungen verglichen. n = 4. Buchstaben stellen statistisch signifikante Unterschiede zwischen den Behandlungen dar (p < 0,05); Die Behandlungen wurden mittels linearer Regression und Post-hoc-Test (geschätzte marginale Mittelwerte) mit einer Sidak-Korrektur für Mehrfachvergleiche verglichen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
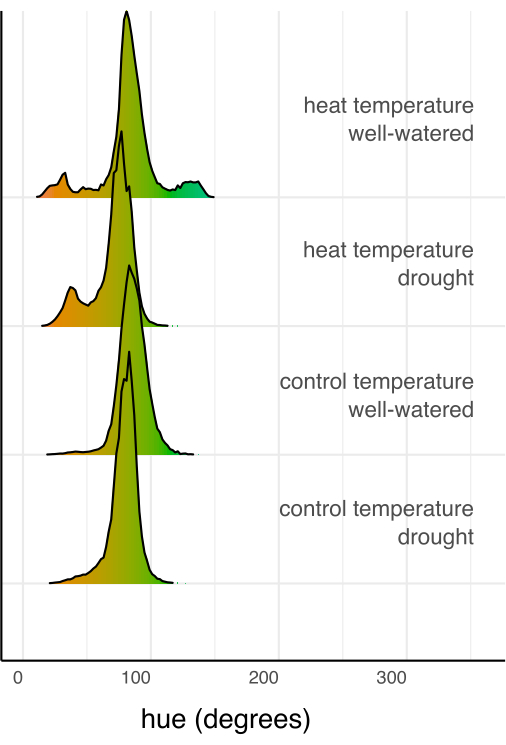
Abbildung 7: Histogramme der Farbe (Farbton), summiert aller Pixel jeder Behandlungskombination, normiert durch die Gesamtzahl der Pixel in der Behandlungskombination. Histogramme werden durch den Farbwert für den Farbtongrad eingefärbt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
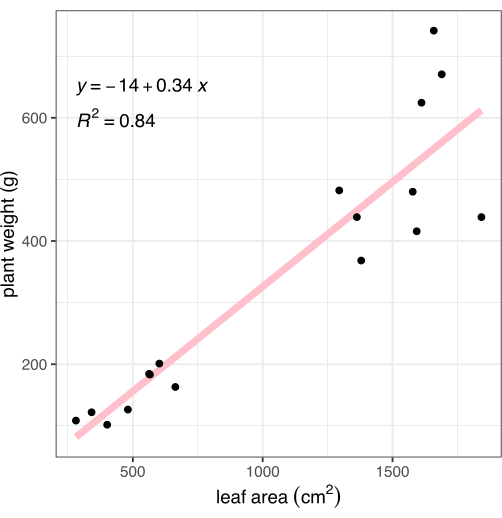
Abbildung 8: Korrelation der anhand von Bildern gemessenen Blattfläche mit der Biomasse der ganzen Pflanze. Jeder Schwarzpunkt steht für eine Pflanze. Die rosa Linie stellt die lineare Korrelation zwischen Blattfläche (X) und Biomasse (Y) dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Charakterzug | Faktor | Freiheitsgrade | F-Wert | p-Wert |
| Blattfläche (cm^2) | Temperatur | 1 | 0.5372 | 0.4766 |
| Blattfläche (cm^2) | Wasser | 1 | 226.5318 | Nr. 1.32E-09 |
| Blattfläche (cm^2) | Temperatur: Wasser | 1 | 2.1526 | 0.1661 |
| Blattfläche (cm^2) | Reste | 13 | ||
| Höhe (cm) | Temperatur | 1 | 5.9426 | 0.0298935 |
| Höhe (cm) | Wasser | 1 | 21.107 | 0.0005029 |
| Höhe (cm) | Temperatur: Wasser | 1 | 2.1752 | 0.1640545 |
| Höhe (cm) | Reste | 13 | ||
| Breite (cm) | Temperatur | 1 | 2.2495 | 0.1575 |
| Breite (cm) | Wasser | 1 | 75.5474 | 8.92E-07 |
| Breite (cm) | Temperatur: Wasser | 1 | 0.0839 | 0.7766 |
| Breite (cm) | Reste | 13 | ||
| Farbton kreisförmiger Mittelwert (Grad) | Temperatur | 1 | 0.9154 | 0.3561432 |
| Farbton kreisförmiger Mittelwert (Grad) | Wasser | 1 | 27.7988 | 0.0001509 |
| Farbton kreisförmiger Mittelwert (Grad) | Temperatur: Wasser | 1 | 3.4792 | 0.0848724 |
| Farbton kreisförmiger Mittelwert (Grad) | Reste | 13 |
Tabelle 1: Ergebnisse des linearen Regressionsmodells und der bidirektionalen ANOVA-Analysen, die die Auswirkungen von Wasseraufbereitung, Temperaturbehandlung und deren Wechselwirkung auf Pflanzeneigenschaften untersuchen.
Diskussion
Die bildbasierte Phänotypisierung ermöglicht genaue, schnelle und quantitative Messungen von Pflanzenphänotypen 4,5,6,7. Diese Methode ist wesentlich schneller, präziser, genauer und bietet eine größere Anzahl von Messungen als die manuelle Verwendung eines Lineals zur Aufzeichnung der Pflanzenhöhe oder qualitative Messungen von Pflanzenfarbmerkmalen, die sich auf die Gesundheit beziehen können 4,5,6,7. Die Blattfläche korrelierte hier und in früheren Forschungen10 stark mit der pflanzlichen Biomasse (Abbildung 8), was darauf hindeutet, dass dieses Maß den zeitaufwändigen, zerstörerischen und fehleranfälligen Prozess des manuellen Wiegens von ganzpflanzlicher Biomasse zuverlässig ersetzen könnte. Während eine Kamera allein für die Aufnahme der Bilder verwendet werden konnte, erhöhte die Hinzufügung des kostengünstigen SBC den Durchsatz durch die Automatisierung der Bilderfassung und der Bildbeschriftung mit den erforderlichen Metadaten15. Diese Methode eignet sich für eine bis ~300 Pflanzen, die einmal täglich (oder seltener) abgebildet werden. Für eine größere Anzahl von Pflanzen in einem Experiment ist ein förderbandbasiertes, über Kopfportal oder robotergestütztes Bildgebungssystem besser geeignet, da die Zeit, um die Pflanzen einzeln für die Bildgebung zu bewegen, länger als ein Arbeitstag sein kann11,13. Für häufigere Zeitpunkte (z. B. stündlich statt einmal pro Tag oder seltener) sind Kamera-Setups in einer Wachstumskammer besser geeignet, da sie Bilder automatisch aufnehmen, aber durch die Größe der Pflanzen begrenzt sind und zusätzlichen SBC15 benötigen.
Während PlantCV hier als Option zur Bildanalyse vorgestellt wird, gibt es auch andere Software. ImageJ (Fiji) ist ein kostenloses Softwarepaket zur Bildanalyse, das über eine grafische Benutzeroberfläche (GUI) auch für Bilder von Pflanzen verwendet werden kann8; Es ist jedoch nicht speziell für die Bildanalyse ganzer Anlage entwickelt, weniger anpassungsfähig für Hochdurchsatz-Computing und weniger leicht wiederholbar als bereits funktionierende Pipelines wie die in PlantCV, die leicht auf neue Bilder angewendet werden können9. Deep Learning (wie BioDock, siehe Table of Materials) und Segment Anything22) Methoden sind attraktiv, weil sie Pflanzen einfach aus dem Hintergrund segmentieren können und könnten für die hier beschriebenen Segmentierungsaufgaben verwendet werden. Diese Deep-Learning-Modelle liefern jedoch nicht die hier beschriebenen Messungen des Pflanzenphänotyps10, und um diese komplexeren Messungen quantitativer Phänotypen durchzuführen, ist möglicherweise eine große Menge an Trainingsbildern (oft mehr als 10.000) und eine Rechenleistung erforderlich, die über einen einfachen Laptop oder Desktop-Computerhinausgeht 10. Diese Modelle erfordern auch einen erheblichen Arbeitsspeicher und eine beträchtliche Rechenleistung, die vielen Forschern nicht zur Verfügung stehen10.
Der kritischste Schritt des Protokolls besteht darin, die Einrichtung der Pflanzenbildgebung zu arrangieren und sicherzustellen, dass sich keine Objekte (wie andere Pflanzen, eine Farbkarte oder Pflanzenreste) in der Nähe der interessierenden Pflanzebefinden 14. Wenn Bilder nicht richtig eingestellt sind oder keine Farbkarte verwendet wird, können sie später nicht analysiert werden, da es keinen Mechanismus für die Standardisierung gibt. Während in diesem Protokoll keine Kalibrierungsschritte erforderlich sind, sind die Farbkorrektur und die Größenstandardisierung während der Verarbeitung unerlässlich. Stellen Sie immer sicher, dass sich die gesamte Farbkarte im Rahmen befindet, aber nicht mit der Pflanze überlappt (Abbildung 2). Ein weiterer häufiger Fallstrick ist die ungleichmäßige Beleuchtung, die Schatten oder helle Aufnahmen verursachen kann. Wir empfehlen die Verwendung von hellem, diffusem Deckenlicht und abdeckenden Fenstern im Raum, um eine ungleichmäßige Beleuchtung zu vermeiden (Abbildung 2). Zusätzliche Lampen können in einem frontalen Winkel zur Pflanze angebracht werden, wenn Deckenleuchten Schatten verursachen. Die Art der Glühbirne spielt keine Rolle, da die Farbe im Bild mit einer Farbkarte korrigiert wird. Unscharfe Bilder sind für die Analyse nicht akzeptabel - verwenden Sie den Autofokus an der Kamera oder die Anweisungen des Herstellers für den manuellen Fokus, um sicherzustellen, dass die Bilder klar sind.
Ebenso ermöglicht die Sicherstellung, dass Bilder nicht überbelichtet sind (ein häufiger Fehler), eine angemessene Analyse (Abbildung 2). Überbelichtete Bilder können bei der Bildanalyse nicht in einer Farbkorrektur korrigiert werden, da die Pixelwerte mit den aktuellen Einstellungen14 außerhalb des Erfassungsbereichs der Kamera liegen. Um eine Überbelichtung zu vermeiden, werten Sie die Farbhistogramme oder RGB-Werte für ein Testbild mit dem hier beschriebenen PlantCV-Workflow aus (Abbildung 3) und reduzieren Sie die Helligkeitseinstellung der Kamera, wenn der Belichtungstest feststellt, dass das Bild überbelichtet ist. Die RGB-Werte (und damit die Histogramme) sollten keine Werte bei 0 oder 255 haben, was dem Wertebereich entspricht, da diese Werte möglicherweise über oder unter dem Erfassungsbereich liegen und nur das Minimum oder Maximum und nicht den wahren Wert aufzeichnen können. Ein Prüfbild sollte immer mit diesen Methoden auf Überbelichtung untersucht werden (Abbildung 2 und Abbildung 3); Wir empfehlen, die in dieser Veröffentlichung bereitgestellte PlantCV-Belichtungsqualitätskontrolle als Überprüfung zu verwenden, die feststellt, dass ein Qualitätsbild weniger als 5 Prozent der Pixel bei 0 oder 255 für jede Farbe hat. Als Faustregel gilt, dass Kameras und Analysesoftware Histogramme liefern, die gleiche und vollständige Histogrammschwänze aufweisen sollten. Werte, die unter 0 (unterbelichtet) oder über 255 (überbelichtet) liegen, sollten mit angepassten Kameraeinstellungen erneut aufgenommen werden. Wir empfehlen, einen Ort mit hellem, gleichmäßigem Kunstlicht ohne Fenster für die Beleuchtung zu wählen, da die Fenster zu hell und uneben sein können und die Lichteinstrahlung häufig verändern. Bilder sollten gleichmäßig beleuchtet erscheinen, und die grauen Quadrate einer Farbkarte sollten nicht weiß erscheinen, was auf eine Überbelichtung hindeutet.
Abhängig von der IP- und Sicherheitseinstellungen der Institution des Benutzers kann eine Problembehandlung für den Bilderfassungscode erforderlich sein. Wenden Sie sich zur Problembehandlung an die IT-Abteilung, wenn es um Probleme mit der Internetverbindung und der Dateiübertragung geht. Ein häufiger Fehler ist die Verwendung der SD-Karte in der falschen Größe für den SBC oder das Einschalten des SBC, bevor der angeschlossene Monitor eingeschaltet wird. Befolgen Sie zur Fehlerbehebung das obige Protokoll genau und laden Sie die SBC-Software neu. Online-Foren sind eine hervorragende Ressource für die Fehlerbehebung und die Suche nach Lösungen für die Kamera der Wahl bei der Verwendung von gphoto2. Um Fehler bei der PlantCV-Bildanalyse zu beheben und die aktuellste Versionierung zu verwenden, verwenden Sie die Dokumentationsseite (https://plantcv.readthedocs.io/en/latest/) oder posten Sie ein Problem auf GitHub (https://github.com/danforthcenter/plantcv/issues). Insbesondere wenn die Schwellenwertmethode in diesem Protokoll die Anlage nicht ausreichend vom Hintergrund trennt, verwenden Sie alternative Schwellenwertmethoden, die in "photo-studio-SV-notebook.ipynb" oder auf der Dokumentationsseite bereitgestellt werden.
Diese Methode kann auf jede Pflanzenart und Behandlung angewendet werden, die zu dem beschriebenen zusammengesetzten Phototographie-Aufbau transportiert werden kann. Zum Beispiel ist es nicht auf Pflanzen anwendbar, die auf einem Feld wachsen, da sie nicht zum Foto-Setup transportiert werden können, ohne die Pflanze auszugraben. In dieser Situation könnten Pflanzen abgeschnitten und anschließend mit dem Protokoll abgebildet werden, obwohl die Messung zerstörerisch wäre. Mehrere Pflanzen können in einem einzigen Bild erfasst und analysiert werden, aber ein Benutzer sollte darauf achten, dass sich die Pflanzen nicht gegenseitig berühren (Abbildung 2), und die Pflanzen werden jedes Mal an der gleichen Stelle auf dem Hintergrund platziert, um sicherzustellen, dass der Arbeitsablauf automatisch über den gesamten Satz von Bildern ausgeführt werden kann. Wenn für Pflanzen wie Arabidopsis thaliana eine Draufsicht erforderlich ist, verwenden Sie ein Stativ, um die Kamera an die Spitze der Pflanzen zu bringen, die auf dem Stoff für Fotografien platziert sind. Die übrigen Methoden zur Bilderfassung und -analyse bleiben gleich.
Wie hier am Beispiel von Mais gezeigt, ist diese Methode sowohl für die Pflanzenzüchtung als auch für grundlegende Erkenntnisse darüber, wie Pflanzen mit abiotischen Stressfaktoren wie Hitze und Trockenheit umgehen, wichtig. Hitze- und Trockenstress zusammen tragen weltweit zu jährlichen Ertragsverlusten von 15 % bis 20 % bei Maisbei 23. Es wird erwartet, dass die Ertragsverluste mit dem globalen Klimawandel noch zunehmen werden, da die Temperaturen bis zum Jahr 2100 um 1,4 bis 4,4 °C steigen dürften24. Die Bewertung der Maisvielfalt im Hinblick auf Stresstoleranz sowie die Bestimmung der phänotypischen, physiologischen und molekularen Mechanismen der abiotischen Stresstoleranz sind wichtige Forschungsziele für die globale Ernährungssicherheit25. Um diese Fragen zu beantworten, müssen Forscher in allen angrenzenden Bereichen - Physiologen, Genetiker, Züchter, Molekularbiologen usw. - in der Lage sein, die Phänotypen von Maispflanzen unter abiotischem Stress schnell, effektiv und oft zerstörungsfrei zu bewerten25. Die hier gezeigte Methode und die Beispielergebnisse adressieren diesen Bedarf und sind ein wichtiges Instrument für Forscher, um reale landwirtschaftliche Herausforderungen anzugehen.
Die hier beobachteten Phänotypen wurden unter Berücksichtigung einer Fülle früherer Forschungen hypothetisch aufgestellt - Trockenstress reduziert die Pflanzengröße und das Grün (gemessen am kreisförmigen Mittelwert des Farbtons)26 (Abbildung 6, Abbildung 7 und Tabelle 1). Der Phänotyp des Hitzestresses bei guter Bewässerung reduzierte unerwarteterweise die Blattfläche nicht signifikant, obwohl dieser Zusammenhang mit mehr Replikaten statistisch besser bewertet werden konnte und die Höhe wie erwartet reduziert wurde (Abbildung 6). Während der zirkuläre Mittelwert des Farbtons aufgrund der Hitze nicht verändert wurde, wurde das Farbprofil tatsächlich so verändert, dass es sowohl mehr gelbe als auch dunklere grüne Pixel umfasste, was zeigt, dass die Bewertung nur des mittleren Farbtons unzureichend ist und das mehrwertige Merkmalshistogramm der Farbe berücksichtigt werden sollte (Abbildung 7). Diese phänotypischen Veränderungen beschreiben die Reaktion einer Pflanze auf die Umwelt, und zusätzliche Techniken wie Messungen des Gasaustauschs, mikroskopische Bildgebung und mehr könnten eingesetzt werden, um die physiologischen Veränderungen zu bewerten, die als Reaktion auf Stress auftreten. Dieses Ergebnis zeigt die Leistungsfähigkeit präziser, quantitativer Messungen, die sonst mit manuellen oder qualitativen Messungen der Pflanzengesundheit übersehen würden. Diese Erkenntnisse sind entscheidend für die Züchtung und den Anbau von Kulturpflanzen, die gegen den Klimawandel resistent sind, der die globalen Ernteerträge erheblich reduziert und sich in den kommenden Jahren voraussichtlich verschlechtern wird.
Offenlegungen
Nichts.
Danksagungen
Diese Arbeit wurde in der Phenotyping Core Facility des Donald Danforth Plant Science Center (RRID: SCR_019049) durchgeführt, die K.M.M. und J.G.D. finanziell unterstützte. Diese Arbeit wurde durch den Donald Danforth Plant Science Center Enterprise Rent-A-Car Research Institute Grant unterstützt. D.S. wurde durch den Taylor Geospatial Institute Block Grant und einen Zuschuss des US Economic Development Administration Center for AgTech and Applied Location Science and Technology (CATALST) (ED21HDQ0240072) finanziert. Wir danken Kevin Reilly und Kris Haines (Integrated Plant Growth Facility, Donald Danforth Plant Science Center, RRID: SCR_024902) für ihre Expertise und Unterstützung beim Pflanzenwachstum. Wir danken Noah Fahlgren, Malia Gehan und Haley Schuhl für ihre Ratschläge zu PlantCV.
Materialien
| Name | Company | Catalog Number | Comments |
| 15W USB-C Power Supply | CanaKit | DCAR-RSP-3A5-C | |
| 25604 UC-E6 USB Cable | Nikon | 18208256044 | |
| 3.5 inch pots | Hummert International | 13006000 | |
| Berger BM7 35% Bark HP | Hummert International | 10121500 | |
| Biodock 2024 | AI Software Platform | www.biodock.ai. | |
| Code editing software | Jupyter | https://jupyter.org/install | |
| Code editing software | Visual Studio Code | https://code.visualstudio.com/download | |
| Color Card ColorChecker Classic | Calibrite | CCC | |
| Color Card ColorChecker Passport | Calibrite | CCPP2 | |
| Conviron Growth Chamber | Conviron | BDW80 model | |
| Dell 22 Monitor | Dell | SE2222H | |
| EH-67 AC Adapter | Nikon | AZ185 | |
| Image analysis software | PlantCV | https://plantcv.readthedocs.io/en/latest/installation/ | |
| Jack's Water Soluble Fertilizer 15-5-15 | Hummert International | 7590200 | liquid fertilizer |
| Keyboard and Mouse | Logitech | 1440638 | MK270 wireless keyboard and mouse |
| Maize seed | USDA-ARS Germplasm Resources Information Network (GRIN) | B73 | https://www.ars-grin.gov/ |
| Micro HDMI to HDMI cable | Amazon | B07KSDB25X | |
| Nikon Coolpix B500 camera | Nikon | ||
| Nikon D7200 camera | Nikon | ||
| Osmocote 14-14-14 | Hummert International | 7630000 | |
| Photography-grade fabric | Yayoya | Polyester Fabric Black Photo Backdrop for Photo Video Studio Televison | |
| Raspberry Pi 4 Case with heatsink | iUniker | B07ZVKN262 | |
| Raspberry Pi 4 Model B Rev 1.1 | Raspberry Pi Foundation | RP4B4GB_BP | |
| Raspberry Pi 4 PiSwitch (USB-C) | CanaKit | RSP-PISWITCH-USBC | |
| Raspberry Pi software | Raspberry Pi Imager | https://www.raspberrypi.com/software/ | |
| Statistical analysis software | Rstudio | https://posit.co/downloads/ | |
| Tripod | K&F Concept | T255A3+BH-28L | |
| Turface MVP | Hummert International | 10240000 | |
| Wireless Barcode Scanner | Netumscan | B08X4NMX2M | 1D and 2D barcode scanner |
Referenzen
- Xiao, Q., Bai, X., Zhang, C., He, Y. Advanced high-throughput plant phenotyping techniques for genome-wide association studies: A review. J Adv Res. 35, 215-230 (2022).
- Mutka, A. M., et al. image-based phenotyping methods provide insight into spatial and temporal dimensions of plant disease. Plant Physiol. 172 (2), 650-660 (2016).
- Mutka, A. M., Bart, R. S. Image-based phenotyping of plant disease symptoms. Front Plant Sci. 5, 734 (2014).
- Fahlgren, N., Gehan, M. A., Baxter, I. Lights, camera, action: High-throughput plant phenotyping is ready for a close-up. Curr Opin Plant Biol. 24 (2), 93-99 (2015).
- Das Choudhury, S., Samal, A., Awada, T. Leveraging image analysis for high-throughput plant phenotyping. Front Plant Sci. 10, 508 (2019).
- Yang, W., et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives. Mol Plant. 13 (2), 187-214 (2020).
- Omari, M. K., et al. Digital image-based plant phenotyping: A review. Korean J Agric Sci. 47 (1), 119-130 (2020).
- Schindelin, J., et al. Fiji: An open-source platform for biolFiogical-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gehan, M. A., et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping. PeerJ. 5, e4088 (2017).
- Murphy, K. M., Ludwig, E., Gutierrez, J., Gehan, M. A. Deep learning in image-based plant phenotyping. Annu Rev Plant Biol. 75 (1), 771-795 (2024).
- Fahlgren, N., et al. A versatile phenotyping system and analytics platform reveals diverse temporal responses to water availability in Setaria. Mol Plant. 8 (3), 1520-1535 (2015).
- Yu, L., et al. Development of a mobile, high-throughput, and low-cost image-based plant growth phenotyping system. bioRxiv. , (2023).
- Atefi, A., Ge, Y., Pitla, S., Schnable, J. Robotic technologies for high-throughput plant phenotyping: Contemporary reviews and future perspectives. Front Plant Sci. 12, 611940 (2021).
- Berry, J. C., Fahlgren, N., Pokorny, A. A., Bart, R. S., Veley, K. M. An automated, high-throughput method for standardizing image color profiles to improve image-based plant phenotyping. PeerJ. 6, e5727 (2018).
- Tovar, J. C., et al. Raspberry Pi-powered imaging for plant phenotyping. Appl Plant Sci. 6, e1031 (2018).
- Jolles, J. W. Broad-scale applications of the Raspberry Pi: A review and guide for biologists. Methods Ecol Evol. 12 (5), 1562-1579 (2021).
- Watt, M., et al. Phenotyping: New windows into the plant for breeders. Annu Rev Plant Biol. 71 (1), 689-712 (2020).
- Xie, C., Yang, C. A review on plant high-throughput phenotyping traits using UAV-based sensors. Comput Electron Agric. 178, 105731 (2020).
- Czedik-Eysenberg, A., et al. The "PhenoBox", a flexible, automated, open-source plant phenotyping solution. New Phytol. 219 (4), 808-823 (2018).
- Sumner, J., Fahlgren, N., Murphy, K. M. PCVR: An R package and tutorials for guided statistical analysis of plant phenotyping data. Authorea Preprints. , (2023).
- Schnable, P. S., et al. The B73 maize genome: complexity, diversity, and dynamics. Science. 326 (5956), 1112-1115 (2009).
- Kirillov, A., et al. Segment Anything. , 3992-4003 (2023).
- Gabaldón-Leal, C., et al. Modelling the impact of heat stress on maize yield formation. Field Crops Res. 198 (3), 226-237 (2016).
- Intergovernmental Panel on Climate Change. . SYNTHESIS REPORT OF THE IPCC SIXTH ASSESSMENT REPORT (AR6). , (2023).
- Quiñones, A., et al. Optimized methods for applying and assessing heat, drought, and nutrient stress of maize seedlings in controlled environment experiments. Cold Spring Harb Protoc. , (2024).
- Murphy, K. M., et al. Maize abiotic stress treatments in controlled environments. Cold Spring Harb Protoc. , (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten