Method Article
Imágenes y análisis para cuantificar los fenotipos de estrés abiótico del maíz (Zea mays)
En este artículo
Resumen
Se presenta un método para la obtención de imágenes de plantas de bajo costo para medir los fenotipos, junto con las mejores prácticas para la captura de imágenes y una línea de análisis de imágenes para cuantificar los rasgos de las plantas. Estos métodos se aplicaron para medir los fenotipos del maíz (Zea mays) en condiciones de calor, sequía y estrés abiótico combinado.
Resumen
La medición cuantitativa de los fenotipos o rasgos de las plantas es esencial para comprender las respuestas de las plantas a los factores ambientales, incluidos los estreses bióticos y abióticos. Sin embargo, los métodos tradicionales de medición de plantas pueden llevar mucho tiempo, ser inexactos y destructivos, especialmente cuando se evalúan rasgos como la biomasa y la altura de forma individual. El análisis basado en imágenes ofrece la oportunidad de reducir la mano de obra, mejorar la exactitud y la precisión, y permitir mediciones repetidas de la misma planta en múltiples puntos de tiempo. Si bien las instalaciones de fenotipado de alto rendimiento proporcionan una solución, a menudo son prohibitivamente caras y tienen una disponibilidad limitada en todo el mundo. Se describe un método para capturar imágenes de plantas utilizando computadoras de placa única de bajo costo y cámaras digitales dentro de una configuración de estudio fotográfico, acompañadas de una línea de análisis de imágenes basada en el paquete gratuito y de código abierto PlantCV. Este método ofrece un protocolo rápido y preciso para la captura, el etiquetado y la transferencia de imágenes, junto con las mejores prácticas para garantizar la adquisición de imágenes de alta calidad. A través del análisis de imágenes, se cuantificaron más de 10 fenotipos para todas las imágenes del experimento simultáneamente, incluidos rasgos como el tamaño, la altura y el color de la planta. Estos métodos se utilizaron para caracterizar la respuesta de la variedad endogámica B73 de Zea mays (maíz) a condiciones de calor, sequía y estrés abiótico combinado por calor y sequía.
Introducción
La medición de los rasgos o fenotipos de las plantas es fundamental para responder a preguntas fundamentales y aplicadas en la ciencia de las plantas. Por ejemplo, un investigador que busque comprender el impacto de una mutación genética en el tamaño de la planta necesitará medir cuantitativamente la altura de la planta y el área foliar1. Un investigador que aplique un estrés biótico (como una infección bacteriana, viral o fúngica) o un estrés abiótico (como sequía, calor, frío o agotamiento de nutrientes) tendrá que investigar el efecto del estrés en el color de la planta, o "verdor", para medir la clorosis 2,3.
Si bien la medición de estos rasgos es esencial para la biología básica de las plantas, así como para el fitomejoramiento aplicado, las mediciones manuales de estos rasgos pueden llevar mucho tiempo, ser imprecisas e inexactas 4,5,6,7. Por ejemplo, la medición de la altura de las plantas incluye el uso manual de una regla para cada planta, a menudo en condiciones ambientales cálidas o húmedas, y está sujeta a errores desde el punto de vista de la persona que mide, así como el punto que se considera la "cima" de la planta. Además, mediciones como la biomasa y el contenido de pigmentos son destructivas, y la misma planta no puede medirse para cambiar a lo largo del tiempo ni utilizarse para múltiples muestreos, ya que debe destruirse en el proceso de realización de estas mediciones 4,5,6,7. La naturaleza lenta y propensa a errores de estas mediciones dificulta el progreso en la evaluación de las plantas para sus respuestas fenotípicas y fisiológicas al estrés.
La captura de imágenes de plantas y la medición de rasgos cuantitativos a partir de la imagen proporciona soluciones a estos problemas 4,5,6,7. El análisis de imágenes mejora la exactitud y la precisión, al tiempo que reduce el tiempo que un investigador tiene para realizar las mediciones, y la misma planta puede ser fotografiada varias veces o utilizada para otros muestreos debido a la cuantificación no destructiva de rasgos 4,5,6,7. Además, las imágenes pueden almacenarse y volver a analizarse mucho después de que se complete un experimento y se eliminen las plantas 4,5,6,7. El análisis de imágenes se puede completar individualmente con herramientas de edición de imágenes de bajo rendimiento, como ImageJ (Fiji)8, o en un rendimiento más alto utilizando la visión artificial y el aprendizaje automático con plataformas como PlantCV9. PlantCV es una plataforma de análisis de imágenes gratuita y de código abierto que permite a los investigadores enmascarar (o separar) primero una planta u otro objeto del fondo de una imagen, y realizar mediciones de tamaño, morfología, color y más de esa plantau objeto. Utilizando un flujo de trabajo modular, un investigador primero crea un flujo de trabajo flexible (o canalización) de diferentes módulos con una imagen de muestra, luego somete las imágenes restantes al flujo de trabajo de alto rendimiento sin interactuar con cada imagen individual9. Se generan rasgos cuantitativos y se analizan las diferencias estadísticamente significativas entre los grupos. Las canalizaciones de Deep Learning se pueden desarrollar para un análisis de imágenes eficiente y automatizado después de que se hayan capturado y etiquetado suficientes imágenes con mediciones de rasgos o metadatos adecuados10.
El fenotipado de "alto rendimiento" puede aumentar la velocidad y reducir el trabajo humano durante la recolección de datos y/o durante el análisis de datos 4,5,6,7. Las instalaciones de fenotipado de alto rendimiento han permitido la automatización de la recolección de imágenes en entornos controlados, donde las plantas se mueven en cintas transportadoras o a través de robots a una estación de imágenes, y se etiquetan automáticamente con el nombre (es decir, etiqueta con metadatos) asociado con la planta 6,11,12,13,14 . Si bien estas cintas transportadoras o instalaciones habilitadas para robots permiten la captura y el etiquetado de grandes cantidades de imágenes sin intervención humana, hay pocas en el mundo y su construcción puede costar millones de dólares, lo que las hace inaccesibles para muchos investigadores. Para aplicar estos conceptos de imágenes automatizadas a un costo menor, Raspberry Pi (aquí referidas como computadoras de placa única, SBC) son computadoras de bajo costo (menos de $ 35 cada una) que se han utilizado para capturar, nombrar, almacenar y transferir imágenes de plantas cultivadas en entornos controlados. Estas configuraciones capturan imágenes en intervalos automatizados y cronometrados utilizando una matriz de hasta 72 Raspberry Pi 12,15,16. Si bien son más rentables que los sistemas de cintas transportadoras que pueden costar millones de dólares, las cámaras de crecimiento SBC descritas anteriormente son más aplicables a una vista de arriba hacia abajo del crecimiento de plantas pequeñas y aún requieren numerosos SBC15. En entornos de campo, los pórticos, los UAS (sistemas aéreos no tripulados) como los drones y los satélites capturan imágenes para el análisis de las características de las plantas17,18. Tanto los sistemas de campo como las instalaciones de entorno controlado de alto rendimiento pueden ser prohibitivos en cuanto a su costo inicial, mantenimiento y conocimiento experto necesarios para construir y mantener los sistemas, así como para analizar imágenes17,18. Los teléfonos celulares y las cámaras digitales que se usan solos para la fotografía de plantas presentan desafíos que requieren mucho tiempo en la transferencia de imágenes y la nomenclatura con metadatos, lo cual es esencial para el análisis posterior de imágenes.
Los métodos descritos aquí presentan una solución de bajo costo para la obtención de imágenes de plantas para los desafíos no abordados por otros sistemas. Una configuración de fotografía, en la que las plantas se transportan individualmente al estudio, permite la medición eficiente de plantas de todos los tamaños en macetas. Utilizando un SBC conectado a una cámara digital, este método proporciona primero captura, etiquetado y transferencia de imágenes estandarizadas. El uso de un teclado o un escáner QR/código de barras para nombrar las imágenes a medida que se capturan evita la actividad lenta y propensa a errores de cambiar el nombre de las imágenes con metadatos después de que se hayan capturado y almacenado. La composición de la imagen y los ajustes recomendados de la cámara son importantes para hacer posible el análisis de imágenes posteriores, y aquí se detallan las directrices. Por último, se proporciona una canalización de análisis de imágenes en PlantCV para extraer rasgos cuantitativos de las imágenes capturadas, como el área de la hoja, la altura, el ancho y el color de la planta.
En relación con los sistemas de cintas transportadoras, este método es de bajo costo y requiere un solo SBC. La configuración de fenotipado descrita aquí se construyó para eliminar las limitaciones y los costos de una cinta transportadora o sistemas robóticos: se puede medir una planta de cualquier tamaño porque la configuración se puede separar del espacio de crecimiento, solo se necesita un solo SBC y funciona con numerosos tipos de cámaras para obtener la máxima flexibilidad y asequibilidad. Enfoques similares al que se presenta aquí incluyen el PhenoBox o el PhenoRig12,19. En comparación con las mediciones manuales, este método ahorra tiempo, reduce los errores y no destruye las plantas 4,12,15,19.
Protocolo
Los detalles sobre los reactivos y el equipo utilizado en este protocolo se enumeran en la Tabla de Materiales.
1. Preparación del estudio fotográfico
- Cuelga tela negra de calidad fotográfica de la pared o el techo para obtener imágenes de vista lateral. Coloque la tela al menos 0,5 m en el piso para que la planta se coloque encima de la tela (Figura 1).
NOTA: La tela debe ser al menos 100 cm más alta y ancha que la planta que se va a fotografiar. - Coloque una tarjeta de color en el fondo, apoyada contra un objeto como una caja para que sea visible en el marco (Figura 1). Para plantas de más de 30 cm de alto o ancho, use una tarjeta de colores que tenga al menos 20 cm de ancho. Para plantas de menos de 30 cm de alto o ancho, use una tarjeta de colores que tenga entre 4 y 8 cm de ancho.
- Conecte una tarjeta SD a una computadora portátil o de escritorio. Descargue Raspberry Pi Imager desde la sección de software del sitio web (https://www.raspberrypi.com/software/) y descargue el archivo de raspberrypi-image-capture.img.gz proporcionado (https://github.com/danforthcenter/photo-maize-paper) en la computadora portátil o de escritorio.
- Inicie el generador de imágenes abriendo el archivo de generador de imágenes (.exe) descargado. Haga clic en el botón Elegir sistema operativo y seleccione el botón Usar personalizado en la parte inferior. Seleccione el archivo image-capture.img.gz.
- A continuación, haga clic en el botón Elegir almacenamiento y seleccione la tarjeta SD conectada a la computadora. Haga clic en el botón Escribir .
NOTA: Asegúrese de haber seleccionado la tarjeta SD correcta, ya que este proceso eliminará todos los datos almacenados en la tarjeta.
- A continuación, haga clic en el botón Elegir almacenamiento y seleccione la tarjeta SD conectada a la computadora. Haga clic en el botón Escribir .
- Conecte la última versión de SBC a un teclado mediante un cable USB, a un monitor externo mediante un cable HDMI y a una regleta de enchufes mediante un cable de alimentación con un interruptor. Conecte el monitor a la regleta de enchufes mediante el cable de alimentación suministrado con el monitor. Coloca la tarjeta SD en el SBC. Consulte la Figura 1 para obtener un esquema etiquetado de la configuración.
- Encienda la regleta presionando el interruptor en la regleta, encienda el monitor usando el botón en el monitor, luego encienda el SBC girando el interruptor del cable de alimentación a "encendido".
- Conecte el SBC a Internet local mediante WIFI o un cable Ethernet.
- Si las fotos se van a transferir directamente a un servidor o almacenamiento en la nube:
- Edite el archivo "CAPTURE-PHOTO.py" en el SBC para que contenga la ruta del archivo para completar la transferencia.
- Edite el archivo win-credentials con la ruta y la contraseña para el servidor o el almacenamiento en la nube utilizando el siguiente comando en el terminal: sudo nano /etc/win-credentials.
NOTA: Póngase en contacto con el administrador del sistema de Internet para asegurarse de que la carpeta tenga permiso para transferir archivos.
- Conecte un escáner de código de barras inalámbrico al SBC siguiendo las instrucciones del fabricante proporcionadas con el escáner de código de barras, ya sea mediante un USB o Bluetooth.
- Apague el SBC haciendo clic en el botón Raspberry Pi en la parte superior izquierda de la pantalla, haga clic en Cerrar sesión y luego haga clic en Apagar para apagar el SBC de forma segura. Después del apagado, encienda el interruptor de alimentación SBC, el cable de alimentación a "apagado" y la regleta de enchufes a "apagado".
2. Crecimiento de las plantas y tratamiento del estrés
NOTA: Cualquier planta es apta para este método de fenotipado; Estos métodos de crecimiento de plantas son aplicables a los resultados representativos presentados aquí.
- Obtenga semillas de B73 u otra variedad de maíz en un centro de almacenamiento.
- Prepare los medios del suelo y las condiciones de crecimiento:
- Llene macetas de 3.5 pulgadas (0.7 L) con una mezcla 1:1 por volumen de Turface MVP y Berger BM7-35 con 3 lb/yarda cúbica de fertilizante de liberación lenta 14-14-14 (N-P-K). Coloque las macetas en una bandeja con agujeros y riegue abundantemente con agua de ósmosis inversa (RO).
- Ajuste una cámara de crecimiento a las siguientes condiciones ambientales: 30 °C día/20 °C noche, 16 h de día, 60% de humedad y 500 mmol.m-2 s-1 de luz al nivel de la maceta durante el día. Establezca una cámara de crecimiento separada con las mismas condiciones, excepto 38 °C de día y 28 °C de noche para el estrés por calor.
- Plante 20 semillas de maíz, cada una en una maceta de 3.5 pulgadas, colocando ~ 2 cm de profundidad en el suelo y cubriéndolo con la tierra desplazada. Riegue a fondo con agua de ósmosis inversa y colóquelo en la cámara de crecimiento de control. Riegue diariamente con fertilizante líquido (mezclado de acuerdo con las instrucciones del fabricante; consulte la Tabla de materiales).
NOTA: Plantar plantas adicionales que se necesitan para el experimento tendrá en cuenta la mala germinación y la falta de uniformidad antes del inicio de una aplicación de estrés. La tasa de germinación variará según la variedad. - Después de 21 días de crecimiento, trasplante 16 de las plantas más uniformes (n = 4 plantas por tratamiento) a macetas de 3,79 L llenas de la misma mezcla de tierra para macetas, previamente humedecida con agua de ósmosis inversa. Regrese las plantas a la cámara de control de crecimiento.
- El día 25 (4 días después del trasplante) mueva la mitad de las plantas (n = 8) a la cámara de estrés por calor en el momento en que se encienden las luces de la cámara. Deje de regar las plantas estresadas por la sequía en ambas cámaras (n = 4 por cámara).
NOTA: Es posible que las plantas con estrés por sequía deban regarse para que las plantas sobrevivan hasta el final del experimento; Aquí, las plantas de sequía se regaron el día 31 y el día 37 con agua fertilizante. - El día 38 (13 días después del inicio del estrés), retire las plantas de las cámaras de crecimiento para obtener imágenes.
3. Imágenes de plantas
- Inicie el SBC (consulte la Figura 2):
- Encienda la regleta con el interruptor de la regleta de enchufes.
- Encienda el monitor con el botón del monitor.
NOTA: Si el SBC se enciende antes de encender el monitor, no se iniciará correctamente. - Coloque el interruptor del cable de alimentación SBC en "on".
NOTA: Esto encenderá el SBC y se iniciará en el monitor; no hay un interruptor de "encendido" en el SBC en sí, solo en el cable de alimentación.
- Coloque la cámara en un trípode o mesa estable.
NOTA: Toda la planta y la carta de colores deben estar a la vista en la pantalla de la cámara. La cámara debe ser perpendicular al suelo y tener una vista directa (sin ángulo) del centro de la planta. La cámara debe ser compatible con el paquete gphoto2, y se puede encontrar una lista aquí: http://www.gphoto.org/proj/libgphoto2/support.php. - Proporcione energía a la cámara a través de un cable de alimentación con la cámara o las baterías. Encienda la cámara con el botón de la cámara.
- Conecte la cámara al puerto USB del SBC mediante el cable de transferencia de datos suministrado con la cámara.
- Organizar la planta en la configuración de la fotografía:
- Coloque un trozo de cinta adhesiva de color oscuro sobre la tela de grado fotográfico donde se colocará la maceta para asegurarse de que la ubicación sea la misma para cada planta. Asegúrese de que la tarjeta de color esté alineada con el trozo de cinta.
NOTA: Coloque la carta de colores al menos a 10 cm del borde de la planta. Durante el control de calidad en el flujo de trabajo de PlantCV (a continuación), la imagen debe poder recortarse para incluir solo la planta y la maceta, pero no la tarjeta de color para garantizar que las fichas de color no se incluyan en la extracción de rasgos final. Consulte la Figura 2 para ver ejemplos de imágenes de alta y baja calidad. - Coloca la olla sobre el pedazo de cinta adhesiva. En el caso de las plantas que crecen en un plano, como el maíz (Zea mays) y el sorgo (Sorghum bicolor), mire el ángulo más amplio de la planta hacia la cámara. En el caso de las plantas que no crecen en un plano, capture ángulos de 0 y 90 grados de la planta y promedie las mediciones cuantitativas resultantes una vez finalizado el análisis de la imagen.
- Coloque un trozo de cinta adhesiva de color oscuro sobre la tela de grado fotográfico donde se colocará la maceta para asegurarse de que la ubicación sea la misma para cada planta. Asegúrese de que la tarjeta de color esté alineada con el trozo de cinta.
- En la pantalla de escritorio de SBC que se muestra en el monitor, abra el terminal haciendo clic en el botón de terminal en la barra de menú superior izquierda.
- Si guarda directamente en un servidor o almacenamiento en la nube, monte el almacenamiento usando el siguiente comando editando la ruta de almacenamiento en la nube necesaria (cloud-storage-path en el ejemplo a continuación) y la contraseña, escribiendo la línea de código en la ventana abierta del terminal y presionando enter en el teclado. Si no se cambia con respecto a la configuración de fábrica, el ID de usuario (UID) será "pi".
NOTA:
sudo mount -t cifs -o credentials=/etc/win-credentials,uid=pi //ruta-de-almacenamiento-de-la nube /mnt/almacenamiento-en--la nube - Escriba el siguiente código en el terminal y presione enter en el teclado. Aparecerá una nueva ventana.
./RUN-PHOTOSTUDIO-SHELL.sh
NOTA: El archivo RUN-PHOTOSTUDIO-SHELL.sh llama al archivo CAPTURE-PHOTO.py, que están precargados en el pi si se cargó utilizando el método anterior. Alternativamente, estos scripts se han proporcionado en GitHub para colocarlos en una tarjeta SD Raspberry Pi existente. - Se abrirá una ventana gráfica de la interfaz gráfica de usuario. Escriba el nombre de la imagen o escanee el código de barras o el código QR de la planta con un escáner de código de barras.
- Seleccione una opción para guardar la imagen localmente o en un servidor montado/almacenamiento en la nube.
- ¡Haga clic aquí para tomar una foto! botón.
- Si se eligió el almacenamiento local, abra la carpeta FOTOS en el escritorio para examinar la imagen. Si se eligió un servidor o almacenamiento en la nube, abra la imagen en esa ubicación.
- Si es necesario, ajuste la configuración de la cámara desconectándola del SBC, ajustándola siguiendo las instrucciones del fabricante y volviéndola a conectar al SBC.
NOTA: Es posible que sea necesario ajustar ajustes como la exposición, el brillo, el contraste, etc., en función de la iluminación de la habitación y del objeto que se está fotografiando. Un error común es sobreexponer las imágenes. Para comprobar la exposición de una imagen de prueba, proceda con la imagen de prueba a través del flujo de trabajo de análisis de imágenes de PlantCV, que incluye una comprobación de control de calidad para la exposición. Ajuste la configuración de la cámara según sea necesario para reducir o aumentar la exposición hasta que los histogramas se distribuyan correctamente. - Una vez que se hayan capturado todas las imágenes, transfiera las imágenes del SBC a una computadora local o almacenamiento en la nube utilizando el método preferido, como almacenamiento USB, navegador de Internet o transferencia SSH.
4. Extracción de rasgos de las imágenes
- Descargue los siguientes archivos en un equipo o servidor local: "photo-studio-SV-notebook.ipynb", "workflow.py" y "photo-studio-SV-config.json". Todos los archivos están disponibles en GitHub (https://github.com/danforthcenter/photo-maize-paper).
- Instale PlantCV en el equipo o servidor local siguiendo los pasos descritos (https://plantcv.readthedocs.io/en/latest/installation/).
- Abra el archivo photo-studio-SV-notebook.ipynb con el editor de código preferido, como JupyterLab o Visual Studio Code.
- Ejecute cada bloque de código, editando según sea necesario dados los parámetros descritos en el bloque de código para obtener una máscara limpia de solo la planta en la imagen.
- Cuando esté satisfecho con el análisis de la imagen de muestra, abra el archivo workflow.py en el editor de código preferido. Edite este archivo con los parámetros modificados en "photo-studio-SV-notebook.ipynb" y guárdelo.
- Abra el archivo photo-studio-SV-config.json y edite las rutas de archivo para dirigirlas a una carpeta de imágenes de entrada, una carpeta de salida deseada, etc., como se indica en el archivo.
- Abra un terminal y ejecute el siguiente código para activar PlantCV. Para ello, escribe el código conda activate plantcv en el terminal y pulsa enter en el teclado.
- Cambie la siguiente línea de código a la ruta del archivo "photo-studio-SV-config.json" (este es el único cambio que se puede realizar en el código siguiente).
- En el terminal, escriba el código editado y presione enter en el teclado. Esto ejecutará el flujo de trabajo en todas las imágenes capturadas en el experimento.
NOTA:
plantcv-run-workflow --config /usuarios/nombre de usuario/photo-studio-SV-config.json
- En el terminal, escriba el código editado y presione enter en el teclado. Esto ejecutará el flujo de trabajo en todas las imágenes capturadas en el experimento.
- Inspeccione las imágenes de salida para determinar si el código era adecuado para todas las imágenes.
NOTA: El contorno azul y rosa de la planta debe estar alrededor de los bordes de la planta y no debe haber otros objetos en la imagen. Si es necesario, vuelva a "photo-studio-SV-analysis.ipynb" y pruebe las imágenes problemáticas y edite el código, específicamente los pasos de enmascaramiento y reducción de ruido, para obtener los resultados deseados. Consulte la Figura 3 para ver ejemplos de segmentación de imágenes de alta calidad, aceptable y baja. - El archivo de salida tendrá la forma de un archivo .json. Regrese a la terminal (asegúrese de que conda activate plantcv ya se haya ejecutado), escriba el siguiente código y presione enter en el teclado para convertir el archivo a .csv para el análisis estadístico posterior, cambiando la ruta del archivo a la del archivo "results-photo-studio.json".
plantcv-utils json2csv -j /usuarios/NOMBRE DE USUARIO/results-photo-studio.json -c /usuarios/NOMBRE DE USUARIO/results-photo-studio.csv
NOTA: Para convertir las medidas de píxeles a una unidad de medida estándar, como cm, utilice la conversión proporcionada desde la función find_color_card en el flujo de trabajo de PlantCV. - Realizar un análisis estadístico de los rasgos cuantitativos de salida para determinar las diferencias en los fenotipos de las plantas debido a diferentes especies de plantas, variedades, tratamientos y puntos temporales.
NOTA: El paquete pcvr de R Studio se ha formulado para analizar los resultados de PlantCV en busca de significación estadística y representación gráfica de los resultados20. Vea un resumen y una representación visual de los rasgos recopilados en la Figura 4.
Resultados
Para desarrollar este método, se evaluaron plantas de maíz B73 para fenotipos cuantitativos bajo temperaturas controladas, temperaturas de calor, condiciones de buen riego y sequía, así como sus combinaciones, para un total de cuatro tratamientos. B73 es una conocida variedad endogámica de maíz con una gran cantidad de datos genéticos21.
El análisis de imágenes en PlantCV corrigió con éxito el color de las imágenes, etiquetó los resultados cuantitativos con los metadatos de la planta y segmentó (en otras palabras, separó) la planta del fondo, como se muestra en la Figura 3. Un análisis fallido proporcionaría imágenes de salida que no muestran el contorno azul o la forma rosada alrededor del exterior de la planta, sino que incluyen ruido adicional o piezas faltantes de la planta (Figura 3, ejemplo de baja calidad). Era necesario un umbral de doble canal para incluir la mayor cantidad posible de la planta en la máscara (ver estrategias alternativas de un solo canal en el "photo-studio-SV-notebook.ipynb"). Sin embargo, algunas plantas, particularmente aquellas con tallos de color púrpura, no tenían una segmentación perfecta, pero eran suficientes para el análisis posterior (Figura 3).
El análisis de PlantCV proporcionó 16 rasgos cuantitativos de un solo valor, resumidos en la Figura 4. Un rasgo de un solo valor proporciona un valor por planta, por ejemplo, el área de la hoja, la altura, el ancho y la media circular del tono. El área foliar se consideró utilizando la función "analyze.shape" (Figura 4) y se consideró toda la planta. Sin embargo, la altura y la anchura se consideraron utilizando la "función analyze.bound_horizontal", con un límite trazado en la línea del suelo de la planta. Esto se debe a que las plantas que caen sobre el costado de la maceta tendrán una mayor altura considerando la planta como un objeto completo, y en su lugar solo deben medirse desde la base de la planta donde se encuentra con el suelo. Además de los rasgos de un solo valor, PlantCV genera rasgos de varios valores, que son histogramas, en particular de los valores de color, que representan cada píxel de la planta. La media circular de tono es un rasgo de un solo valor que es el valor de tono promedio para toda la planta.
Para determinar qué rasgos son los más interesantes, se calculó la varianza explicada por tratamiento para cada rasgo, así como la correlación entre los diferentes rasgos utilizando la función "frem" en el paquete pcvr R Studio20 (Figura 5A). El área foliar, la altura, el ancho y la media circular del tono se seleccionaron para el análisis posterior porque explicaban más del 50% de la varianza debida al tratamiento, y son medidas relevantes del crecimiento de la planta (área, altura y ancho) y la salud (tono). Las imágenes representativas de la planta con el área foliar más grande y más pequeña demuestran la variación en los fenotipos recolectados en este experimento (Figura 5B, C, respectivamente). La planta con la mayor área foliar también tenía el tono medio más alto y era una planta B73, bien regada y estresada por calor (Figura 5B). El área foliar más pequeña tenía el tono medio más pequeño (Figura 5C) y era una planta B73, estresada por sequía y calor.
El tratamiento del agua tuvo un efecto significativo en la determinación del área foliar (F(1,13) = 226.5, p = 1.32 x 10-9), la altura (F(1,13) = 21.1, p = 0.0005), el ancho (F(1,13) = 75.5, p = 8.92 x 10-7) y la media circular de tono (F(1,13) = 27.8, p = 0.0002), determinados utilizando un modelo de regresión lineal para el efecto del tratamiento y un ANOVA de dos factores en R Studio (resultados completos en la Tabla 1). El tratamiento con temperatura tuvo un efecto significativo sobre la altura (F(1,13) = 5.94, p = 0.03), pero no sobre el área foliar, el ancho o la media circular de tono (p > 0.05, Tabla 1). La interacción entre la temperatura y el estado del agua no fue un factor significativo en ninguno de los rasgos medidos (p > 0.05, Tabla 1).
A continuación, se compararon los tratamientos individuales con el control y entre sí mediante regresión lineal y prueba post-hoc (medias marginales estimadas) con una corrección de Sidak para comparaciones múltiples (Figura 6). El área foliar, la altura de la planta, el ancho de la planta y la media circular del tono se redujeron significativamente en las plantas estresadas por sequía en comparación con las bien regadas en ambas condiciones de temperatura (p < 0.05, Figura 6). El estrés por calor mientras se regaba bien solo redujo la altura de la planta (p < 0.05, Figura 6), pero no redujo significativamente el área foliar, el ancho de la planta o la media circular del tono. Esta diferencia en el tono se puede investigar más a fondo evaluando el tono del rasgo de color de múltiples valores, trazado como un histograma en la Figura 7, en lugar de solo un promedio. La reducción en el tono medio debido a la sequía se debió a un cambio de píxeles verdes a amarillos, conocido como clorosis (Figura 7). Si bien el calor no mostró una diferencia significativa en el tono promedio, sí mostró una reducción y un aumento debido tanto al amarillamiento (clorosis) como al oscurecimiento del color verde (Figura 7).
Después de la toma de imágenes, las plantas se cortaron en la unión raíz-brote y se registró el peso del tejido aéreo. Las raíces se lavaron suavemente y se secaron con una toalla, luego se pesaron, para calcular la biomasa total de la planta (también conocida como peso) para compararla con el método de fenotipado basado en imágenes descrito. El área foliar medida mediante análisis de imágenes se correlacionó fuertemente con la biomasa vegetal (R2 = 0.84, Figura 8).

Figura 1: Imagen de la configuración del estudio fotográfico con componentes etiquetados para recolectar imágenes de plantas. (A) Tela de grado fotográfico, (B) tarjeta de color, (C) línea de cinta para colocar macetas de manera uniforme, (D) cámara digital, (E) computadora de placa única, (F) monitor que muestra el proceso de captura de imágenes. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Descripción general del flujo de trabajo de imágenes. Se proporciona una imagen representativa de "alta calidad" y "baja calidad". Haga clic aquí para ver una versión más grande de esta figura.
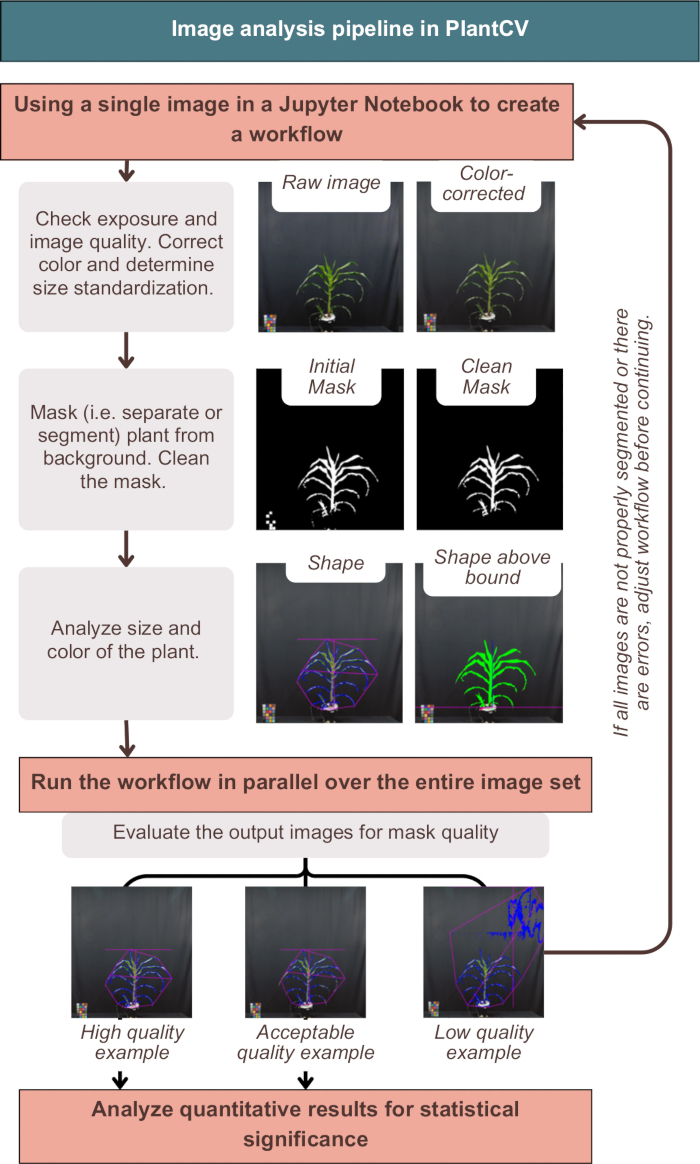
Figura 3: Descripción general del flujo de trabajo de análisis de imágenes en PlantCV. Se presentan imágenes representativas de segmentación de "alta", "aceptable" y "baja" calidad; Los rasgos de PlantCV se representan con un contorno rosa de la forma de toda la planta y un contorno azul del área de la planta. Haga clic aquí para ver una versión más grande de esta figura.
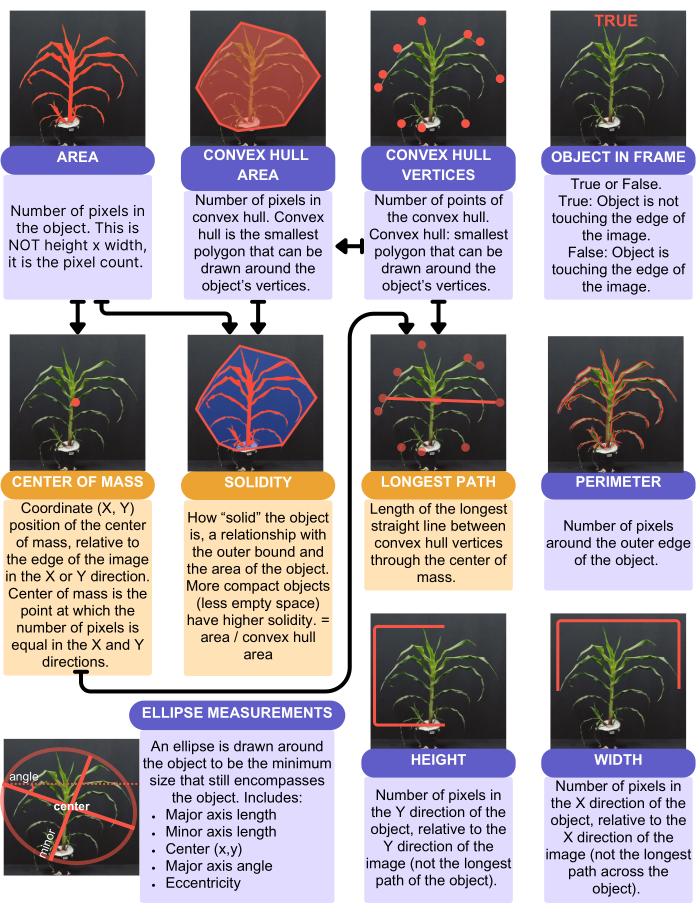
Figura 4: Resumen y representación visual de los rasgos de forma capturados a partir del análisis. Función de forma en PlantCV. Las cajas moradas representan rasgos que no utilizan otro rasgo en su cálculo. Las cajas naranjas representan rasgos que utilizan otros rasgos en su cálculo; Las flechas indican qué características se utilizan en estos cálculos. Haga clic aquí para ver una versión más grande de esta figura.
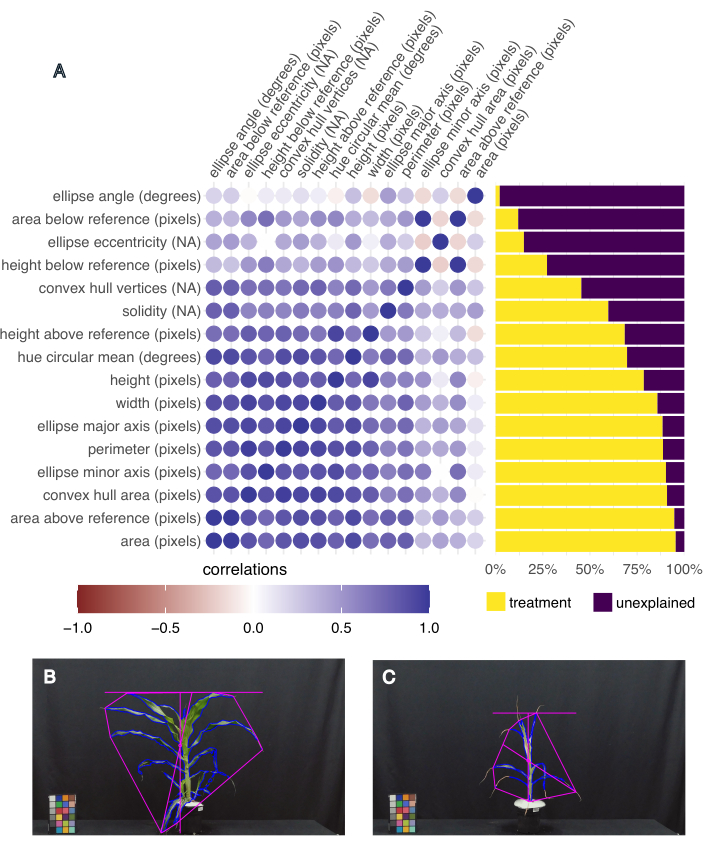
Figura 5: El fenotipado basado en imágenes captura la varianza fenotípica y los rasgos correlacionados. (A) Varianza de los rasgos explicados por el tratamiento (derecha) y correlación de los rasgos entre sí (izquierda). Cada rasgo está etiquetado, seguido de su unidad de medida; "NA" significa que es una medida sin unidades. (B) Imagen de la planta que tenía el área foliar más alta y la media circular de tono más alta (grados). (C) Imagen de la planta que tenía el área foliar más pequeña y la media circular de tono más bajo (grados). Haga clic aquí para ver una versión más grande de esta figura.
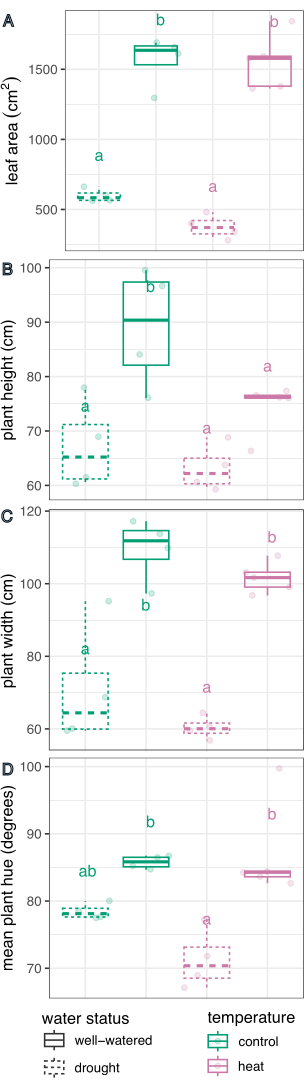
Figura 6: Comparación de los efectos del tratamiento en los fenotipos de las plantas. El área foliar (A), la altura (B), la anchura (C) y la media circular del tono (D) se comparan para determinar el efecto de la sequía, el calor y las tensiones combinadas. n = 4. Las letras representan diferencias estadísticamente significativas entre tratamientos (p < 0,05); los tratamientos se compararon mediante regresión lineal y prueba post-hoc (medias marginales estimadas) con una corrección de Sidak para comparaciones múltiples. Haga clic aquí para ver una versión más grande de esta figura.
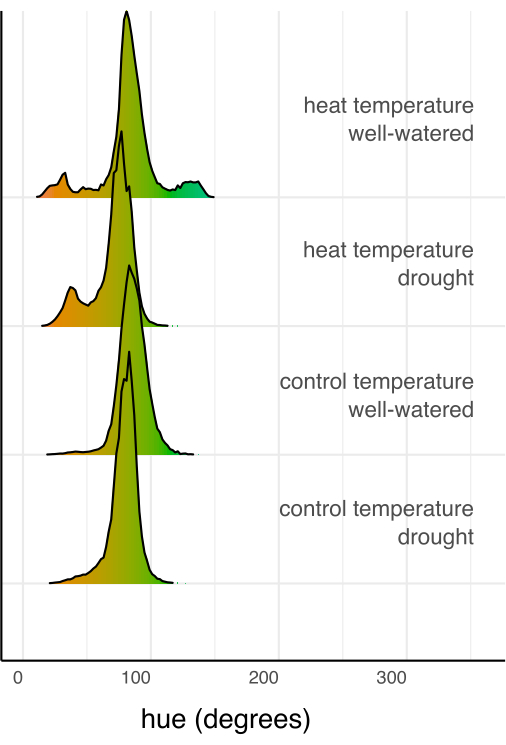
Figura 7: Histogramas de color (tono) sumados de todos los píxeles de cada combinación de tratamiento, normalizados por el número total de píxeles en la combinación de tratamiento. Los histogramas se colorean según el valor de color para el grado de tono. Haga clic aquí para ver una versión más grande de esta figura.
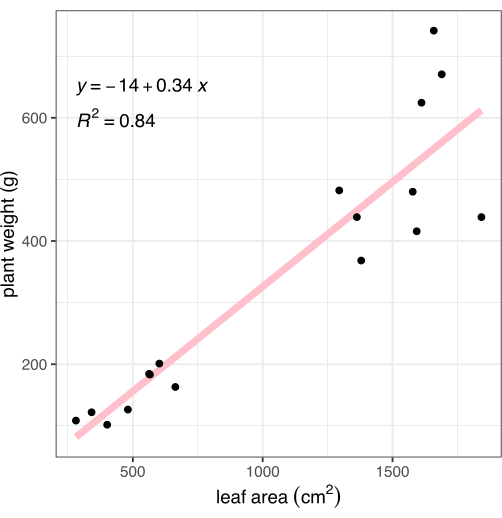
Figura 8: Correlación del área foliar medida a partir de imágenes con la biomasa de toda la planta. Cada punto negro representa una planta. La línea rosa representa la correlación lineal entre el área foliar (X) y la biomasa (Y). Haga clic aquí para ver una versión más grande de esta figura.
| Rasgo | Factor | Grados de libertad | Valor F | Valor p |
| Área foliar (cm^2) | Temperatura | 1 | 0.5372 | 0.4766 |
| Área foliar (cm^2) | Agua | 1 | 226.5318 | 1.32E-09 |
| Área foliar (cm^2) | Temperatura:Agua | 1 | 2.1526 | 0.1661 |
| Área foliar (cm^2) | Residuos | 13 | ||
| Altura (cm) | Temperatura | 1 | 5.9426 | 0.0298935 |
| Altura (cm) | Agua | 1 | 21.107 | 0.0005029 |
| Altura (cm) | Temperatura:Agua | 1 | 2.1752 | 0.1640545 |
| Altura (cm) | Residuos | 13 | ||
| Ancho (cm) | Temperatura | 1 | 2.2495 | 0.1575 |
| Ancho (cm) | Agua | 1 | 75.5474 | 8.92E-07 |
| Ancho (cm) | Temperatura:Agua | 1 | 0.0839 | 0.7766 |
| Ancho (cm) | Residuos | 13 | ||
| Tono medio circular (grados) | Temperatura | 1 | 0.9154 | 0.3561432 |
| Tono medio circular (grados) | Agua | 1 | 27.7988 | 0.0001509 |
| Tono medio circular (grados) | Temperatura:Agua | 1 | 3.4792 | 0.0848724 |
| Tono medio circular (grados) | Residuos | 13 |
Tabla 1: Resultados del modelo de regresión lineal y análisis de ANOVA de dos vías que examinan los efectos del tratamiento del agua, el tratamiento de la temperatura y su interacción en las características de las plantas.
Discusión
El fenotipado basado en imágenes proporciona mediciones cuantitativas, rápidas y precisas de los fenotipos de las plantas 4,5,6,7. Este método es significativamente más rápido, más preciso, más exacto y proporciona un mayor número de mediciones que el uso manual de una regla para registrar la altura de la planta, o medidas cualitativas de las características del color de la planta que pueden relacionarse con la salud 4,5,6,7. Se observó que el área foliar aquí, y en investigaciones anteriores10, se correlaciona fuertemente con la biomasa de la planta (Figura 8), lo que sugiere que esta medida podría reemplazar de manera confiable el proceso lento, destructivo y propenso a errores de pesar manualmente la biomasa de la planta entera. Si bien solo se podía usar una cámara para capturar las imágenes, la adición del SBC de bajo costo aumentó el rendimiento a través de la automatización de la recopilación de imágenes y el etiquetado de imágenes con los metadatos necesarios15. Este método es adecuado para una a ~ 300 plantas que se visualizan una vez al día (o con menos frecuencia). Para un mayor número de plantas en un experimento, es más adecuado un sistema de imágenes basado en una cinta transportadora, un pórtico aéreo o un sistema de imágenes habilitado para robots, ya que el tiempo para mover las plantas individualmente para obtener imágenes puede ser más largo que un día de trabajo11,13. Para puntos de tiempo más frecuentes (por ejemplo, una imagen cada hora en lugar de una vez al día o con menos frecuencia), las configuraciones de cámara dentro de una cámara de crecimiento son más adecuadas porque capturan imágenes automáticamente, pero están limitadas por el tamaño de las plantas y necesitanSBC 15 adicionales.
Si bien PlantCV se presenta como la opción de análisis de imágenes aquí, existe otro software. ImageJ (Fiji) es un paquete de software libre para el análisis de imágenes que también se puede utilizar para imágenes de plantas a través de una interfaz gráfica de usuario (GUI)8; sin embargo, no está particularmente desarrollado para el análisis de imágenes de toda la planta, es menos adaptable a la computación de alto rendimiento y es menos fácilmente repetible que las tuberías que ya funcionan, como las de PlantCV, que se pueden aplicar fácilmente a nuevas imágenes9. Los métodos de Deep Learning (como BioDock, ver Tabla de Materiales) y Segment Anything22) son atractivos por su capacidad para segmentar fácilmente las plantas desde el fondo, y podrían usarse para las tareas de segmentación descritas aquí. Sin embargo, estos modelos de Deep Learning no proporcionan las mediciones de fenotipos de plantas descritas aquí10, y para realizar estas mediciones más complejas de fenotipos cuantitativos puede requerir una gran cantidad de imágenes de entrenamiento (a menudo superiores a 10.000) y potencia de cálculo más allá de una computadora portátil o de escritorio básica10. Estos modelos también requieren una cantidad significativa de memoria de computadora y potencia de procesamiento, que no están disponibles para muchos investigadores10.
El paso más crítico del protocolo es organizar la configuración de imágenes de la planta y asegurarse de que no haya objetos (como otras plantas, una tarjeta de colores o restos de plantas) cerca de la plantade interés. Si las imágenes no se configuran adecuadamente, o no se utiliza una tarjeta de color, serán imposibles de analizar posteriormente porque no existe un mecanismo de estandarización. Si bien no son necesarios pasos de calibración en este protocolo, la corrección de color y la estandarización del tamaño durante el procesamiento son esenciales. Asegúrese siempre de que toda la carta de colores esté en el marco, pero que no se superponga con la planta (Figura 2). Otro error común es la iluminación desigual, que puede causar sombras o tomas brillantes; recomendamos utilizar una luz superior brillante y difusa y cubrir las ventanas de la habitación para evitar una iluminación desigual (Figura 2). Se pueden agregar lámparas adicionales en un ángulo frontal a la planta si las luces del techo están causando sombras. El tipo de bombilla no es una preocupación, dado que el color se corregirá en la imagen mediante una tarjeta de colores. Las imágenes borrosas no son aceptables para el análisis: utilice el enfoque automático de la cámara o las instrucciones del fabricante para el enfoque manual para asegurarse de que las imágenes sean claras.
Del mismo modo, asegurarse de que las imágenes no estén sobreexpuestas (un error común) permite un análisis adecuado (Figura 2); Las imágenes sobreexpuestas no se pueden corregir en una corrección de color durante el análisis de imagen porque los valores de píxel están más allá del rango de detección de la cámara con la configuración actual14. Para evitar la sobreexposición, evalúe los histogramas de color o los valores RGB de una imagen de prueba utilizando el flujo de trabajo de PlantCV descrito aquí (Figura 3) y reduzca la configuración de brillo de la cámara si la prueba de exposición determina que la imagen está sobreexpuesta. Los valores RGB (y, por lo tanto, los histogramas) no deben tener valores en 0 o 255, que es el rango de valores, ya que es posible que esos valores estén por encima o por debajo del rango de detección y solo puedan registrar el valor mínimo o máximo en lugar del valor verdadero. Una imagen de prueba siempre debe evaluarse utilizando estos métodos para la sobreexposición (Figura 2 y Figura 3); sugerimos utilizar el control de calidad de exposición PlantCV proporcionado en esta publicación como verificación, que determina que una imagen de calidad tiene menos del 5 por ciento de píxeles en 0 o 255 para cada color. Como regla general, las cámaras y el software de análisis proporcionan histogramas, que deben tener colas de histograma iguales y completas; Los valores cortados por debajo de 0 (subexpuestos) o por encima de 255 (sobreexpuestos) deben volver a tomarse con la configuración de la cámara ajustada. Sugerimos elegir un lugar con iluminación artificial brillante y uniforme sin ventanas para la iluminación, ya que las ventanas pueden ser demasiado brillantes, desiguales y cambiar con frecuencia la exposición a la luz en el espacio. Las imágenes deben aparecer iluminadas uniformemente y los cuadrados grises de una tarjeta de color no deben aparecer blancos, lo que sugiere una sobreexposición.
Es posible que sea necesario solucionar problemas para el código de captura de imagen en función de la IP y la configuración de seguridad de la institución del usuario. Para solucionar problemas, póngase en contacto con el departamento de TI para problemas de conectividad a Internet y transferencia de archivos. Un error común es usar una tarjeta SD de tamaño incorrecto para el SBC o encender el SBC antes de que se encienda el monitor conectado. Para solucionar problemas, siga el protocolo anterior con precisión y vuelva a cargar el software SBC. Los foros en línea son un excelente recurso para solucionar problemas y encontrar soluciones para la cámara específica de elección cuando se usa gphoto2. Para solucionar problemas de análisis de imágenes de PlantCV y usar el control de versiones más actualizado, utilice la página de documentación (https://plantcv.readthedocs.io/en/latest/) o publique un problema en GitHub (https://github.com/danforthcenter/plantcv/issues). En particular, si el método de umbralización en este protocolo no separa suficientemente la planta del fondo, utilice métodos de umbral alternativos proporcionados en "photo-studio-SV-notebook.ipynb" o en la página de documentación.
Este método se puede aplicar a cualquier especie de planta y tratamiento que se pueda transportar a la configuración de fototografía ensamblada descrita. Por ejemplo, no es aplicable a las plantas que crecen en un campo, ya que no se pueden transportar a la configuración de fotografía sin desenterrar la planta. En esta situación, las plantas podrían ser taladas y posteriormente fotografiadas utilizando el protocolo, aunque la medición sería destructiva. Se pueden capturar y analizar varias plantas en una sola imagen, pero el usuario debe tener cuidado de evitar que las plantas se toquen entre sí (Figura 2), y las plantas se colocan en la misma ubicación en el fondo cada vez para garantizar que el flujo de trabajo se pueda ejecutar automáticamente en todo el conjunto de imágenes. Si es necesaria una vista de arriba hacia abajo para plantas como Arabidopsis thaliana, use un trípode para mover la cámara a la parte superior de las plantas colocadas en la tela de grado fotográfico; El resto de los métodos de captura y análisis de imágenes siguen siendo los mismos.
Como se demostró aquí con el maíz, este método es importante tanto para el mejoramiento de plantas como para los conocimientos fundamentales sobre cómo las plantas toleran el estrés abiótico, como el calor y la sequía. El estrés por calor y sequía combinados contribuye a una pérdida de rendimiento anual de entre el 15% y el 20% en maíz en todo el mundo23. Se espera que las pérdidas de rendimiento aumenten con el cambio climático global, ya que se espera que las temperaturas aumenten entre 1,4 y 4,4 °C para el año 210024. La evaluación de la diversidad de maíz para determinar la tolerancia al estrés, así como la determinación de los mecanismos fenotípicos, fisiológicos y moleculares de la tolerancia al estrés abiótico, son objetivos de investigación fundamentales para la seguridad alimentaria mundial25. Para responder a estas preguntas, los investigadores de todos los campos adyacentes (fisiólogos, genetistas, mejoradores, biólogos moleculares, etc.) deben ser capaces de evaluar de manera rápida, eficaz y, a menudo, no destructiva los fenotipos de las plantas de maíz sometidas a estrés abiótico25. El método y los ejemplos de resultados que se muestran aquí abordan esta necesidad y son una herramienta importante para que los investigadores aborden los desafíos agrícolas del mundo real.
Los fenotipos observados aquí fueron hipotetizados, dada la gran cantidad de investigaciones previas: el estrés por sequía reduce el tamaño de la planta y el verdor (medido por la media circular del tono)26 (Figura 6, Figura 7 y Tabla 1). El fenotipo de estrés calórico con riego de pozo inesperadamente no redujo significativamente el área foliar, aunque con más repeticiones, esta relación pudo evaluarse mejor estadísticamente, y la altura se redujo como se esperaba (Figura 6). Si bien la media circular del tono no se modificó debido al calor, el perfil de color se alteró para incluir píxeles más amarillos y verdes más oscuros, lo que demuestra que la evaluación solo del tono medio es insuficiente y se debe considerar el histograma de rasgos de color de múltiples valores (Figura 7). Estos cambios fenotípicos describen la respuesta de una planta al medio ambiente, y se podrían emplear técnicas adicionales, como mediciones de intercambio de gases, imágenes de microscopía, etc., para evaluar los cambios fisiológicos que ocurren en respuesta al estrés. Este resultado demuestra el poder de las mediciones precisas y cuantitativas que, de otro modo, se pasarían por alto utilizando medidas manuales o cualitativas de la salud de las plantas. Estos conocimientos son fundamentales para el mejoramiento y la ingeniería de cultivos resistentes al cambio climático, que reduce significativamente el rendimiento mundial de los cultivos y se prevé que empeore en los próximos años.
Divulgaciones
Ninguno.
Agradecimientos
Este trabajo se llevó a cabo en el Centro Central de Fenotipado del Centro de Ciencias Vegetales Donald Danforth (RRID: SCR_019049), que proporcionó apoyo financiero a K.M.M. y J.G.D. Este trabajo fue apoyado por la subvención del Instituto de Investigación de Alquiler de Coches del Centro de Ciencias de las Plantas Donald Danforth. D.S. fue financiado por la Subvención en Bloque del Instituto Geoespacial Taylor y una subvención del Centro de la Administración de Desarrollo Económico de los Estados Unidos para la Tecnología y la Ciencia y Tecnología de Ubicación Aplicada (CATALST) (ED21HDQ0240072). Agradecemos a Kevin Reilly y Kris Haines (Centro Integrado de Crecimiento de Plantas, Centro de Ciencias de las Plantas Donald Danforth, RRID: SCR_024902) por su experiencia y asistencia en el crecimiento de las plantas. Agradecemos a Noah Fahlgren, Malia Gehan y Haley Schuhl por sus consejos sobre PlantCV.
Materiales
| Name | Company | Catalog Number | Comments |
| 15W USB-C Power Supply | CanaKit | DCAR-RSP-3A5-C | |
| 25604 UC-E6 USB Cable | Nikon | 18208256044 | |
| 3.5 inch pots | Hummert International | 13006000 | |
| Berger BM7 35% Bark HP | Hummert International | 10121500 | |
| Biodock 2024 | AI Software Platform | www.biodock.ai. | |
| Code editing software | Jupyter | https://jupyter.org/install | |
| Code editing software | Visual Studio Code | https://code.visualstudio.com/download | |
| Color Card ColorChecker Classic | Calibrite | CCC | |
| Color Card ColorChecker Passport | Calibrite | CCPP2 | |
| Conviron Growth Chamber | Conviron | BDW80 model | |
| Dell 22 Monitor | Dell | SE2222H | |
| EH-67 AC Adapter | Nikon | AZ185 | |
| Image analysis software | PlantCV | https://plantcv.readthedocs.io/en/latest/installation/ | |
| Jack's Water Soluble Fertilizer 15-5-15 | Hummert International | 7590200 | liquid fertilizer |
| Keyboard and Mouse | Logitech | 1440638 | MK270 wireless keyboard and mouse |
| Maize seed | USDA-ARS Germplasm Resources Information Network (GRIN) | B73 | https://www.ars-grin.gov/ |
| Micro HDMI to HDMI cable | Amazon | B07KSDB25X | |
| Nikon Coolpix B500 camera | Nikon | ||
| Nikon D7200 camera | Nikon | ||
| Osmocote 14-14-14 | Hummert International | 7630000 | |
| Photography-grade fabric | Yayoya | Polyester Fabric Black Photo Backdrop for Photo Video Studio Televison | |
| Raspberry Pi 4 Case with heatsink | iUniker | B07ZVKN262 | |
| Raspberry Pi 4 Model B Rev 1.1 | Raspberry Pi Foundation | RP4B4GB_BP | |
| Raspberry Pi 4 PiSwitch (USB-C) | CanaKit | RSP-PISWITCH-USBC | |
| Raspberry Pi software | Raspberry Pi Imager | https://www.raspberrypi.com/software/ | |
| Statistical analysis software | Rstudio | https://posit.co/downloads/ | |
| Tripod | K&F Concept | T255A3+BH-28L | |
| Turface MVP | Hummert International | 10240000 | |
| Wireless Barcode Scanner | Netumscan | B08X4NMX2M | 1D and 2D barcode scanner |
Referencias
- Xiao, Q., Bai, X., Zhang, C., He, Y. Advanced high-throughput plant phenotyping techniques for genome-wide association studies: A review. J Adv Res. 35, 215-230 (2022).
- Mutka, A. M., et al. image-based phenotyping methods provide insight into spatial and temporal dimensions of plant disease. Plant Physiol. 172 (2), 650-660 (2016).
- Mutka, A. M., Bart, R. S. Image-based phenotyping of plant disease symptoms. Front Plant Sci. 5, 734 (2014).
- Fahlgren, N., Gehan, M. A., Baxter, I. Lights, camera, action: High-throughput plant phenotyping is ready for a close-up. Curr Opin Plant Biol. 24 (2), 93-99 (2015).
- Das Choudhury, S., Samal, A., Awada, T. Leveraging image analysis for high-throughput plant phenotyping. Front Plant Sci. 10, 508 (2019).
- Yang, W., et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives. Mol Plant. 13 (2), 187-214 (2020).
- Omari, M. K., et al. Digital image-based plant phenotyping: A review. Korean J Agric Sci. 47 (1), 119-130 (2020).
- Schindelin, J., et al. Fiji: An open-source platform for biolFiogical-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gehan, M. A., et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping. PeerJ. 5, e4088 (2017).
- Murphy, K. M., Ludwig, E., Gutierrez, J., Gehan, M. A. Deep learning in image-based plant phenotyping. Annu Rev Plant Biol. 75 (1), 771-795 (2024).
- Fahlgren, N., et al. A versatile phenotyping system and analytics platform reveals diverse temporal responses to water availability in Setaria. Mol Plant. 8 (3), 1520-1535 (2015).
- Yu, L., et al. Development of a mobile, high-throughput, and low-cost image-based plant growth phenotyping system. bioRxiv. , (2023).
- Atefi, A., Ge, Y., Pitla, S., Schnable, J. Robotic technologies for high-throughput plant phenotyping: Contemporary reviews and future perspectives. Front Plant Sci. 12, 611940 (2021).
- Berry, J. C., Fahlgren, N., Pokorny, A. A., Bart, R. S., Veley, K. M. An automated, high-throughput method for standardizing image color profiles to improve image-based plant phenotyping. PeerJ. 6, e5727 (2018).
- Tovar, J. C., et al. Raspberry Pi-powered imaging for plant phenotyping. Appl Plant Sci. 6, e1031 (2018).
- Jolles, J. W. Broad-scale applications of the Raspberry Pi: A review and guide for biologists. Methods Ecol Evol. 12 (5), 1562-1579 (2021).
- Watt, M., et al. Phenotyping: New windows into the plant for breeders. Annu Rev Plant Biol. 71 (1), 689-712 (2020).
- Xie, C., Yang, C. A review on plant high-throughput phenotyping traits using UAV-based sensors. Comput Electron Agric. 178, 105731 (2020).
- Czedik-Eysenberg, A., et al. The "PhenoBox", a flexible, automated, open-source plant phenotyping solution. New Phytol. 219 (4), 808-823 (2018).
- Sumner, J., Fahlgren, N., Murphy, K. M. PCVR: An R package and tutorials for guided statistical analysis of plant phenotyping data. Authorea Preprints. , (2023).
- Schnable, P. S., et al. The B73 maize genome: complexity, diversity, and dynamics. Science. 326 (5956), 1112-1115 (2009).
- Kirillov, A., et al. Segment Anything. , 3992-4003 (2023).
- Gabaldón-Leal, C., et al. Modelling the impact of heat stress on maize yield formation. Field Crops Res. 198 (3), 226-237 (2016).
- Intergovernmental Panel on Climate Change. . SYNTHESIS REPORT OF THE IPCC SIXTH ASSESSMENT REPORT (AR6). , (2023).
- Quiñones, A., et al. Optimized methods for applying and assessing heat, drought, and nutrient stress of maize seedlings in controlled environment experiments. Cold Spring Harb Protoc. , (2024).
- Murphy, K. M., et al. Maize abiotic stress treatments in controlled environments. Cold Spring Harb Protoc. , (2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados