Method Article
Imaging e analisi per la quantificazione dei fenotipi di stress abiotico del mais (Zea mays)
In questo articolo
Riepilogo
Viene presentato un metodo per l'imaging a basso costo delle piante per misurare i fenotipi, insieme alle migliori pratiche per l'acquisizione delle immagini e una pipeline di analisi delle immagini per quantificare i tratti delle piante. Questi metodi sono stati applicati per misurare i fenotipi del mais (Zea mays) in condizioni di caldo, siccità e stress abiotico combinato.
Abstract
La misurazione quantitativa dei fenotipi delle piante, o tratti, è essenziale per comprendere le risposte delle piante ai fattori ambientali, compresi gli stress biotici e abiotici. Tuttavia, i metodi tradizionali di misurazione degli impianti possono richiedere molto tempo, essere imprecisi e distruttivi, in particolare quando si valutano tratti come la biomassa e l'altezza su base individuale. L'analisi basata su immagini offre l'opportunità di ridurre la manodopera, migliorare l'accuratezza e la precisione e consentire misurazioni ripetute dello stesso impianto in più punti temporali. Sebbene le strutture di fenotipizzazione ad alto rendimento forniscano una soluzione, sono spesso proibitive e limitate in termini di disponibilità in tutto il mondo. Viene descritto un metodo per l'acquisizione di immagini di impianti utilizzando computer a scheda singola a basso costo e fotocamere digitali all'interno di una configurazione di studio fotografico, accompagnata da una pipeline di analisi delle immagini basata sul pacchetto gratuito e open source PlantCV. Questo metodo offre un protocollo rapido e accurato per l'acquisizione, l'etichettatura e il trasferimento delle immagini, insieme alle best practice per garantire un'acquisizione delle immagini di alta qualità. Attraverso l'analisi delle immagini, sono stati quantificati più di 10 fenotipi per tutte le immagini dell'esperimento contemporaneamente, inclusi tratti come le dimensioni, l'altezza e il colore delle piante. Questi metodi sono stati utilizzati per caratterizzare la risposta della varietà consanguinea B73 di Zea mays (mais) al caldo, alla siccità e alle condizioni di stress abiotico combinato di calore e siccità.
Introduzione
La misurazione dei tratti delle piante, o fenotipi, è fondamentale per rispondere a domande fondamentali e applicate nella scienza delle piante. Ad esempio, un ricercatore che cerca di comprendere l'impatto di una mutazione genetica sulle dimensioni delle piante dovrà misurare quantitativamente l'altezza delle piante e l'area fogliare1. Un ricercatore che applica uno stress biotico (come un'infezione batterica, virale o fungina) o uno stress abiotico (come siccità, caldo, freddo o esaurimento dei nutrienti) dovrà studiare l'effetto dello stress sul colore delle piante, o "verde", per misurare la clorosi 2,3.
Sebbene la misurazione di questi tratti sia essenziale per la biologia vegetale di base e per il miglioramento genetico applicato, le misurazioni manuali di questi tratti possono richiedere molto tempo, essere imprecise e imprecise 4,5,6,7. Ad esempio, la misurazione dell'altezza delle piante include l'uso manuale di un righello per ogni pianta, spesso in condizioni ambientali calde o umide, ed è soggetta a errori dal punto di vista della persona che misura, nonché da quale punto è considerato la "cima" della pianta. Inoltre, misurazioni come il contenuto di biomassa e pigmenti sono distruttive e la stessa pianta non può essere misurata per il cambiamento nel tempo o utilizzata per campionamenti multipli perché deve essere distrutta nel processo di effettuazione di queste misurazioni 4,5,6,7. La natura dispendiosa in termini di tempo e soggetta a errori di queste misurazioni ostacola i progressi nella valutazione delle piante per le loro risposte fenotipiche e fisiologiche allo stress.
L'acquisizione di immagini di piante e la misurazione dei tratti quantitativi dall'immagine forniscono soluzioni a questi problemi 4,5,6,7. L'analisi delle immagini migliora l'accuratezza e la precisione, riducendo al contempo il tempo che un ricercatore ha per effettuare le misurazioni, e la stessa pianta può essere ripresa più volte o utilizzata per altri campionamenti grazie alla quantificazione non distruttiva dei tratti 4,5,6,7. Inoltre, le immagini possono essere memorizzate e rianalizzate molto tempo dopo che un esperimento è stato completato e le piante vengono smaltite 4,5,6,7. L'analisi delle immagini può essere completata singolarmente con strumenti di modifica delle immagini a bassa produttività, come ImageJ (Fiji)8, o in una velocità di produzione più elevata utilizzando la visione artificiale e l'apprendimento automatico con piattaforme come PlantCV9. PlantCV è una piattaforma di analisi delle immagini gratuita e open source che consente ai ricercatori di mascherare (o separare) prima una pianta o un altro oggetto dallo sfondo di un'immagine e di effettuare misurazioni di dimensioni, morfologia, colore e altro ancora di quella pianta o oggetto9. Utilizzando un flusso di lavoro modulare, un ricercatore crea prima un flusso di lavoro flessibile (o pipeline) di diversi moduli con un'immagine di esempio, quindi sottopone le immagini rimanenti al flusso di lavoro ad alto rendimento senza interagire con ogni singola immagine9. I tratti quantitativi vengono prodotti e analizzati per differenze statisticamente significative tra i gruppi. Le pipeline di deep learning possono essere sviluppate per un'analisi efficiente e automatizzata delle immagini dopo che un numero sufficiente di immagini è stato acquisito ed etichettato con misurazioni dei tratti o metadati appropriati10.
La fenotipizzazione "ad alto rendimento" può aumentare la velocità e ridurre il lavoro umano durante la raccolta dei dati e/o durante l'analisi dei dati 4,5,6,7. Gli impianti di fenotipizzazione ad alto rendimento hanno consentito l'automazione della raccolta di immagini in ambienti controllati, in cui le piante si spostano su nastri trasportatori o tramite robot verso una stazione di imaging, e vengono automaticamente etichettate con il nome (cioè l'etichetta con i metadati) associato alla pianta 6,11,12,13,14 . Sebbene questi nastri trasportatori o strutture abilitate ai robot consentano l'acquisizione e l'etichettatura di grandi quantità di immagini senza l'intervento umano, ce ne sono pochi al mondo e possono costare milioni di dollari per la loro costruzione, rendendoli inaccessibili a molti ricercatori. Per applicare questi concetti di imaging automatizzato a un costo inferiore, i Raspberry Pi (qui indicati come computer a scheda singola, SBC) sono computer a basso costo (meno di $ 35 ciascuno) che sono stati utilizzati per acquisire, denominare, archiviare e trasferire immagini di piante coltivate in ambienti controllati. Queste configurazioni acquisiscono immagini a intervalli automatici e temporizzati utilizzando un array fino a 72 Raspberry Pi 12,15,16. Sebbene più convenienti dei sistemi a nastro trasportatore che possono costare milioni di dollari, le camere di crescita SBC descritte in precedenza sono più applicabili a una visione dall'alto verso il basso della coltivazione di piccole piante e richiedono ancora numerosi SBC15. In ambienti sul campo, portali, UAS (sistemi aerei senza equipaggio) come droni e satelliti catturano immagini per l'analisi dei tratti delle piante17,18. Sia i sistemi sul campo che le strutture in ambienti controllati ad alta produttività possono essere proibitivi in termini di costi iniziali, manutenzione e conoscenze specialistiche necessarie per costruire e mantenere i sistemi, nonché per analizzare le immagini17,18. I telefoni cellulari e le fotocamere digitali utilizzati da soli per la fotografia di piante presentano sfide dispendiose in termini di tempo per il trasferimento e la denominazione delle immagini con i metadati, che è essenziale per l'analisi delle immagini a valle.
I metodi qui descritti rappresentano una soluzione a basso costo per l'imaging delle piante per le sfide non affrontate da altri sistemi. Una configurazione fotografica, in cui le piante vengono trasportate singolarmente in studio, consente la misurazione efficiente di piante di tutte le dimensioni in vaso. Utilizzando un SBC collegato a una fotocamera digitale, questo metodo fornisce innanzitutto l'acquisizione, l'etichettatura e il trasferimento standardizzati delle immagini. L'utilizzo di una tastiera o di uno scanner di codici QR/codici a barre per denominare le immagini durante l'acquisizione consente di aggirare l'attività dispendiosa in termini di tempo e soggetta a errori di ridenominazione delle immagini con i metadati dopo che sono state acquisite e archiviate. La composizione dell'immagine e le impostazioni consigliate per la fotocamera sono importanti per rendere possibile l'analisi delle immagini a valle e le linee guida sono descritte in dettaglio qui. Infine, viene fornita una pipeline di analisi delle immagini in PlantCV per estrarre tratti quantitativi dalle immagini acquisite, come l'area fogliare, l'altezza, la larghezza e il colore della pianta.
Rispetto ai sistemi a nastro trasportatore, questo metodo è a basso costo e richiede un singolo SBC. La configurazione di fenotipizzazione qui descritta è stata costruita per eliminare i vincoli e i costi di un nastro trasportatore o di sistemi robotici: è possibile misurare piante di qualsiasi dimensione perché la configurazione può essere separata dallo spazio di crescita, è necessario un solo SBC e funziona con numerosi tipi di telecamere per la massima flessibilità e convenienza. Approcci simili a quello presentato qui includono PhenoBox o PhenoRig12,19. Rispetto alle misurazioni manuali, questo metodo consente di risparmiare tempo, ridurre gli errori e non distrugge le piante 4,12,15,19.
Protocollo
I dettagli sui reagenti e sulle attrezzature utilizzate in questo protocollo sono elencati nella Tabella dei Materiali.
1. Allestimento dello studio fotografico
- Appendi un tessuto nero per la fotografia a una parete o al soffitto per l'imaging laterale. Stendere il tessuto per almeno 0,5 m sul pavimento in modo che la pianta sia posizionata sopra il tessuto (Figura 1).
NOTA: Il tessuto deve essere almeno 100 cm più alto e più largo della pianta da fotografare. - Posiziona una carta colorata sullo sfondo, appoggiata a un oggetto come una scatola in modo che sia visibile nell'inquadratura (Figura 1). Per piante più grandi di 30 cm di altezza o larghezza, usa una cartella colori larga almeno 20 cm. Per piante più piccole di 30 cm di altezza o larghezza, usa una cartella colori larga tra i 4 e gli 8 cm.
- Collega una scheda SD a un computer portatile o desktop. Scarica Raspberry Pi Imager dalla sezione software del sito Web (https://www.raspberrypi.com/software/) e scarica il file raspberrypi-image-capture.img.gz fornito (https://github.com/danforthcenter/photo-maize-paper) sul laptop o sul computer desktop.
- Avviare la termocamera aprendo il file della termocamera (.exe) scaricato. Fare clic sul pulsante Scegli sistema operativo e selezionare il pulsante Usa personalizzato in basso. Seleziona il file image-capture.img.gz.
- Quindi, fai clic sul pulsante Scegli archiviazione e seleziona la scheda SD collegata al computer. Fare clic sul pulsante Scrivi .
NOTA: Assicurarsi che sia stata selezionata la scheda SD corretta, poiché questo processo eliminerà tutti i dati memorizzati sulla scheda.
- Quindi, fai clic sul pulsante Scegli archiviazione e seleziona la scheda SD collegata al computer. Fare clic sul pulsante Scrivi .
- Collega l'ultima versione di SBC a una tastiera utilizzando un cavo USB, a un monitor esterno utilizzando un cavo HDMI e a una ciabatta utilizzando un cavo di alimentazione con un interruttore. Collegare il monitor alla ciabatta utilizzando il cavo di alimentazione fornito con il monitor. Inserire la scheda SD nell'SBC. Vedere la Figura 1 per uno schema etichettato della configurazione.
- Accendere la ciabatta premendo l'interruttore sulla striscia, accendere il monitor utilizzando il pulsante sul monitor, quindi accendere l'SBC spostando l'interruttore sul cavo di alimentazione su "on".
- Collega l'SBC alla rete Internet locale tramite WIFI o un cavo Ethernet.
- Se le foto devono essere trasferite direttamente su un server o su un archivio cloud:
- Modificare il file "CAPTURE-PHOTO.py" sull'SBC in modo che contenga il percorso del file per completare il trasferimento.
- Modifica il file win-credentials con il percorso e la password per il server o l'archiviazione cloud utilizzando il seguente comando nel terminale: sudo nano /etc/win-credentials.
NOTA: Contattare l'amministratore del sistema Internet per assicurarsi che la cartella disponga dell'autorizzazione per trasferire i file.
- Collegare uno scanner di codici a barre wireless all'SBC utilizzando le istruzioni del produttore fornite con lo scanner di codici a barre, utilizzando USB o Bluetooth.
- Disattiva l'SBC facendo clic sul pulsante Raspberry Pi in alto a sinistra dello schermo, facendo clic su Disconnetti, quindi facendo clic su Spegni per spegnere in sicurezza l'SBC. Dopo lo spegnimento, accendere l'interruttore di alimentazione SBC, portare il cavo di alimentazione su "off" e la ciabatta su "off".
2. Crescita delle piante e trattamento dello stress
NOTA: Qualsiasi pianta è adatta a questo metodo di fenotipizzazione; Questi metodi di crescita delle piante sono applicabili ai risultati rappresentativi qui presentati.
- Procuratevi i semi di B73 o di un'altra varietà di mais da un centro di stoccaggio.
- Preparare i terreni e le condizioni di crescita:
- Riempi i vasi da 3,5 pollici (0,7 L) con una miscela 1:1 in volume di Turface MVP e Berger BM7-35 con 3 libbre/iarda cubica di fertilizzante a lenta cessione 14-14-14 (NPK). Metti le pentole in un vassoio con fori e annaffia accuratamente con acqua ad osmosi inversa (RO).
- Impostare una camera di crescita sulle seguenti condizioni ambientali: 30 °C di giorno/20 °C di notte, 16 ore di durata del giorno, 60% di umidità e 500 mmol.m-2 s-1 di luce a livello del vaso durante il giorno. Impostare una camera di crescita separata con le stesse condizioni, ad eccezione di 38 °C di giorno/28 °C di notte per stress da calore.
- Pianta 20 semi di mais, ciascuno in un vaso da 3,5 pollici, posizionandoli a ~2 cm di profondità nel terreno e coprendolo con il terreno spostato. Innaffia abbondantemente con acqua RO e posiziona nella camera di crescita di controllo. Innaffia quotidianamente con fertilizzante liquido (miscelato secondo le istruzioni del produttore; vedi Tabella dei materiali).
NOTA: Piantare piante extra necessarie per l'esperimento rappresenterà la scarsa germinazione e la mancanza di uniformità prima dell'inizio di un'applicazione di stress. Il tasso di germinazione varia in base alla varietà. - Dopo 21 giorni di crescita, trapiantare 16 delle piante più uniformi (n = 4 piante per trattamento) in vasi da 3,79 L riempiti con la stessa miscela di terriccio, pre-bagnati con acqua RO. Riportare le piante nella camera di crescita di controllo.
- Il giorno 25 (4 giorni dopo il trapianto) spostare metà delle piante (n = 8) nella camera dello stress termico nel momento in cui si accendono le luci nella camera. Smetti di annaffiare le piante stressate dalla siccità in entrambe le camere (n = 4 per camera).
NOTA: Potrebbe essere necessario annaffiare le piante da stress da siccità in modo che le piante sopravvivano fino alla fine dell'esperimento; Qui, le piante colpite dalla siccità sono state annaffiate il giorno 31 e il giorno 37 con acqua fertilizzante. - Il giorno 38 (13 giorni dopo l'inizio dello stress), rimuovi le piante dalle camere di crescita per l'imaging.
3. Imaging delle piante
- Avviare l'SBC (vedi Figura 2):
- Accendere la ciabatta utilizzando l'interruttore sulla ciabatta.
- Accendere il monitor utilizzando il pulsante sul monitor.
NOTA: Se l'SBC viene acceso prima dell'accensione del monitor, non si avvierà correttamente. - Portare l'interruttore del cavo di alimentazione SBC su "on".
NOTA: Questo accenderà l'SBC e si avvierà sul monitor; non c'è un interruttore "on" sull'SBC stesso, solo sul cavo di alimentazione.
- Posizionare la fotocamera su un treppiede o su un tavolo stabile.
NOTA: L'intera pianta e la scheda colore dovrebbero essere visibili sullo schermo della fotocamera. La telecamera dovrebbe essere perpendicolare al suolo e avere una visione diretta (senza angolo) del centro della pianta. La fotocamera deve essere compatibile con il pacchetto gphoto2 e un elenco può essere trovato qui: http://www.gphoto.org/proj/libgphoto2/support.php. - Fornire alimentazione alla fotocamera tramite un cavo di alimentazione con la fotocamera o le batterie. Accendere la fotocamera utilizzando il pulsante sulla fotocamera.
- Collegare la fotocamera alla porta USB dell'SBC utilizzando il cavo di trasferimento dati fornito con la fotocamera.
- Organizzare l'impianto nella configurazione fotografica:
- Posiziona un pezzo di nastro adesivo di colore scuro sul tessuto fotografico dove verrà posizionato il vaso per assicurarti che il posizionamento sia lo stesso per ogni pianta. Assicurati che la cartella colori sia in linea con il pezzo di nastro.
NOTA: Posiziona la carta dei colori ad almeno 10 cm dal bordo della pianta. Durante il controllo di qualità nel flusso di lavoro di PlantCV (sotto), l'immagine dovrebbe poter essere ritagliata per includere solo la pianta e il vaso, ma non la cartella colori, per garantire che i chip di colore non siano inclusi nell'estrazione finale del tratto. Vedere la Figura 2 per esempi di immagini di alta e bassa qualità. - Posiziona la pentola sul pezzo di nastro adesivo. Per le piante che crescono in piano, come il mais (Zea mays) e il sorgo (Sorghum bicolor), rivolgere l'angolo più ampio della pianta verso la fotocamera. Per le piante che non crescono in piano, catturare gli angoli di 0 e 90 gradi della pianta e calcolare la media delle misurazioni quantitative risultanti al termine dell'analisi delle immagini.
- Posiziona un pezzo di nastro adesivo di colore scuro sul tessuto fotografico dove verrà posizionato il vaso per assicurarti che il posizionamento sia lo stesso per ogni pianta. Assicurati che la cartella colori sia in linea con il pezzo di nastro.
- Nella schermata del desktop SBC visualizzata sul monitor, aprire il terminale facendo clic sul pulsante del terminale nella barra dei menu in alto a sinistra.
- Se si salva direttamente su un server o su un cloud storage, montare lo storage utilizzando il seguente comando modificando il percorso di cloud storage necessario (cloud-storage-path nell'esempio seguente) e la password, digitando la riga di codice nella finestra del terminale aperta e premendo invio sulla tastiera. Se non viene modificato rispetto alle impostazioni di fabbrica, l'ID utente (UID) sarà "pi".
NOTA:
sudo mount -t cifs -o credentials=/etc/win-credentials,uid=pi //percorso-archiviazione-cloud /mnt/archiviazione-cloud - Digita il seguente codice nel terminale e premi invio sulla tastiera. Apparirà una nuova finestra.
./RUN-PHOTOSTUDIO-SHELL.sh
NOTA: Il file RUN-PHOTOSTUDIO-SHELL.sh chiama il file CAPTURE-PHOTO.py, che viene precaricato sul pi se è stato caricato utilizzando il metodo sopra. In alternativa, questi script sono stati forniti su GitHub per l'inserimento su una scheda SD Raspberry Pi esistente. - Si aprirà una finestra dell'interfaccia utente grafica. Digita il nome dell'immagine o scansiona il codice a barre o il codice QR dell'impianto utilizzando uno scanner di codici a barre.
- Selezionare un'opzione per salvare l'immagine localmente o su un server/archivio cloud montato.
- Clicca Clicca qui per scattare una foto! bottone.
- Se è stata scelta l'archiviazione locale, aprire la cartella FOTO sul desktop per esaminare l'immagine. Se è stato scelto un server o un archivio cloud, aprire l'immagine in tale posizione.
- Se necessario, regolare le impostazioni della fotocamera scollegandola dall'SBC, regolandola seguendo le istruzioni del produttore e ricollegandola all'SBC.
NOTA: Potrebbe essere necessario regolare impostazioni come esposizione, luminosità, contrasto, ecc. a seconda dell'illuminazione della stanza e dell'oggetto ripreso. Un errore comune è quello di sovraesporre le immagini. Per verificare l'esposizione di un'immagine di prova, procedere con l'immagine di prova attraverso il flusso di lavoro di analisi delle immagini PlantCV, che include un controllo di qualità per l'esposizione. Regolare le impostazioni della fotocamera in base alle esigenze per ridurre o aumentare l'esposizione fino a quando gli istogrammi non sono distribuiti correttamente. - Dopo che tutte le immagini sono state acquisite, trasferirle dall'SBC a un computer locale o a un archivio cloud utilizzando il metodo preferito, ad esempio l'archiviazione USB, il browser Internet o il trasferimento SSH.
4. Estrazione dei tratti dalle immagini
- Scaricare i seguenti file su un computer o server locale: "photo-studio-SV-notebook.ipynb", "workflow.py" e "photo-studio-SV-config.json". Tutti i file sono disponibili su GitHub (https://github.com/danforthcenter/photo-maize-paper).
- Installare PlantCV sul computer o sul server locale utilizzando i passaggi descritti (https://plantcv.readthedocs.io/en/latest/installation/).
- Apri il file photo-studio-SV-notebook.ipynb utilizzando l'editor di codice preferito, ad esempio JupyterLab o Visual Studio Code.
- Eseguire ogni blocco di codice, modificandolo se necessario in base ai parametri delineati nel blocco di codice per ottenere una maschera pulita solo della pianta nell'immagine.
- Quando si è soddisfatti dell'analisi dell'immagine di esempio, aprire il file workflow.py nell'editor di codice preferito. Modifica questo file con tutti i parametri modificati in "photo-studio-SV-notebook.ipynb" e salvalo.
- Aprire il photo-studio-SV-config.json file e modificare i percorsi dei file per indirizzarli a una cartella di immagini di input, a una cartella di output desiderata, ecc., come indicato nel file.
- Apri un terminale ed esegui il seguente codice per attivare PlantCV. Per fare ciò, digita il codice conda activate plantcv nel terminale e premi invio sulla tastiera.
- Modificare la riga di codice seguente con il percorso del file "photo-studio-SV-config.json" (questa è l'unica modifica da apportare nel codice seguente).
- Nel terminale, digita il codice modificato e premi invio sulla tastiera. In questo modo il flusso di lavoro verrà eseguito su tutte le immagini acquisite nell'esperimento.
NOTA:
plantcv-run-workflow --config /Utenti/NOME UTENTE/photo-studio-SV-config.json
- Nel terminale, digita il codice modificato e premi invio sulla tastiera. In questo modo il flusso di lavoro verrà eseguito su tutte le immagini acquisite nell'esperimento.
- Esaminare le immagini di output per determinare se il codice è appropriato per tutte le immagini.
NOTA: Il contorno blu e rosa della pianta dovrebbe essere attorno ai bordi della pianta e non dovrebbero esserci altri oggetti nell'immagine. Se necessario, torna su "photo-studio-SV-analysis.ipynb" e testa le immagini problematiche e modifica il codice, in particolare i passaggi di mascheratura e la riduzione del rumore, per ottenere i risultati desiderati. Vedere la Figura 3 per esempi di segmentazione delle immagini di alta, accettabile e bassa qualità. - Il file di output sarà sotto forma di file .json. Torna al terminale (assicurati che conda activate plantcv sia già stato eseguito), digita il seguente codice e premi invio sulla tastiera per convertire il file in .csv per l'analisi statistica a valle, modificando il percorso del file in quello del file "results-photo-studio.json".
plantcv-utils json2csv -j /Utenti/NOME UTENTE/results-photo-studio.json -c /Utenti/NOME UTENTE/results-photo-studio.csv
NOTA: Per convertire le misure da pixel a un'unità di misura standard, ad esempio cm, utilizzare la conversione fornita dalla funzione find_color_card nel flusso di lavoro di PlantCV. - Eseguire analisi statistiche dei tratti quantitativi di output per determinare le differenze nei fenotipi delle piante dovute a diverse specie vegetali, varietà, trattamenti e punti temporali.
NOTA: Il pacchetto pcvr in R Studio è stato formulato per analizzare gli output di PlantCV per la significatività statistica e la rappresentazione grafica dei risultati20. Si veda un riassunto e una rappresentazione visiva dei tratti raccolti nella Figura 4.
Risultati
Per sviluppare questo metodo, le piante di mais B73 sono state valutate per fenotipi quantitativi a temperature controllate, temperature di calore, condizioni di acqua potabile e siccità, nonché per le loro combinazioni, per un totale di quattro trattamenti. B73 è una nota varietà di mais consanguineo con una ricchezza di dati genetici21.
L'analisi delle immagini in PlantCV ha corretto con successo il colore delle immagini, etichettato i risultati quantitativi con i metadati della pianta e segmentato (in altre parole, separato) la pianta dallo sfondo, come dimostrato nella Figura 3. Un'analisi non riuscita fornirebbe immagini di output che non mostrano il contorno blu o la forma rosa intorno all'esterno dell'impianto, ma piuttosto includono rumore aggiuntivo o parti mancanti dell'impianto (Figura 3, esempio di bassa qualità). Era necessaria una soglia a doppio canale per includere la maggior parte possibile della pianta nella maschera (vedi strategie alternative a canale singolo in "photo-studio-SV-notebook.ipynb"). Tuttavia, alcune piante, in particolare quelle con steli di colore violaceo, non avevano una segmentazione perfetta, ma erano sufficienti per l'analisi a valle (Figura 3).
L'analisi di PlantCV ha fornito 16 caratteri quantitativi a valore singolo, riassunti nella Figura 4. Un singolo valore fornisce un valore per pianta, ad esempio l'area fogliare, l'altezza, la larghezza e la tonalità circolare media. L'area fogliare è stata considerata utilizzando la funzione "analyze.shape" (Figura 4) e ha considerato l'intera pianta. L'altezza e la larghezza, tuttavia, sono state considerate utilizzando la "funzione analyze.bound_horizontal", con un limite tracciato sulla linea del suolo della pianta. Questo perché le piante che si abbassano sul lato del vaso avranno un'altezza maggiore considerando la pianta come un oggetto intero, e invece dovrebbero misurare solo dalla base della pianta dove incontra il terreno. Oltre ai tratti a valore singolo, PlantCV produce tratti multivalore, che sono istogrammi, in particolare dei valori di colore, che rappresentano ogni pixel della pianta. La media circolare della tonalità è un singolo valore che è il valore medio della tonalità per l'intera pianta.
Per determinare quali tratti sono i più interessanti, è stata calcolata la varianza spiegata dal trattamento per ciascun tratto, nonché la correlazione tra i diversi tratti utilizzando la funzione "frem" nel pacchetto pcvr R Studio20 (Figura 5A). L'area fogliare, l'altezza, la larghezza e la media circolare della tonalità sono state selezionate per l'analisi a valle perché spiegavano più del 50% della varianza dovuta al trattamento e sono misure rilevanti della crescita delle piante (area, altezza e larghezza) e della salute (tonalità). Immagini rappresentative della pianta con l'area fogliare più grande e più piccola dimostrano la variazione dei fenotipi raccolti in questo esperimento (Figura 5B, C, rispettivamente). La pianta con l'area fogliare più grande aveva anche la tonalità media più alta ed era una pianta B73, ben irrigata, stressata dal calore (Figura 5B). L'area fogliare più piccola aveva la tonalità media più piccola (Figura 5C) ed era una pianta B73, stressata dalla siccità e dal calore.
Il trattamento delle acque ha avuto un effetto significativo nella determinazione dell'area fogliare (F(1,13) = 226,5, p = 1,32 x 10-9), dell'altezza (F(1,13) = 21,1, p = 0,0005), della larghezza (F(1,13) = 75,5, p = 8,92 x 10-7) e della tonalità media circolare (F(1,13) = 27,8, p = 0,0002), determinata utilizzando un modello di regressione lineare per l'effetto del trattamento e un'ANOVA a due vie in R Studio (risultati completi nella Tabella 1). Il trattamento della temperatura ha avuto un effetto significativo sull'altezza (F(1,13) = 5,94, p = 0,03), ma non sull'area fogliare, sulla larghezza o sulla tonalità circolare media (p > 0,05, Tabella 1). L'interazione tra temperatura e stato dell'acqua non è stata un fattore significativo in nessuno dei caratteri misurati (p > 0,05, Tabella 1).
Successivamente, i singoli trattamenti sono stati confrontati con il controllo e tra loro utilizzando la regressione lineare e il test post-hoc (medie marginali stimate) con una correzione di Sidak per confronti multipli (Figura 6). L'area fogliare, l'altezza della pianta, la larghezza della pianta e la media circolare della tonalità sono risultate significativamente ridotte nelle piante stressate dalla siccità rispetto a quelle ben irrigate in entrambe le condizioni di temperatura (p < 0,05, Figura 6). Lo stress da calore quando ben irrigato ha solo ridotto l'altezza della pianta (p < 0,05, Figura 6), ma non ha ridotto significativamente l'area fogliare, la larghezza della pianta o la tonalità circolare. Questa differenza di tonalità può essere ulteriormente studiata valutando la tonalità del tratto di colore multivalore, tracciata come un istogramma nella Figura 7, piuttosto che come una semplice media. La riduzione della tonalità media dovuta alla siccità è dovuta a un passaggio dai pixel verdi a quelli gialli, noto come clorosi (Figura 7). Sebbene il calore non abbia mostrato una differenza significativa nella tonalità media, ha mostrato una riduzione e un aumento dovuti sia all'ingiallimento (clorosi) che all'oscuramento del colore verde (Figura 7).
Dopo l'imaging, le piante sono state tagliate alla giunzione radice-germoglio ed è stato registrato il peso del tessuto fuori terra. Le radici sono state lavate delicatamente e asciugate con un asciugamano, quindi pesate, per calcolare la biomassa vegetale totale (nota anche come peso) per il confronto con il metodo di fenotipizzazione basato su immagini descritto. L'area fogliare misurata tramite analisi di immagine è fortemente correlata alla biomassa vegetale (R2 = 0,84, Figura 8).

Figura 1: Immagine dell'allestimento dello studio fotografico con componenti etichettati per la raccolta delle immagini delle piante. (A) Tessuto per la fotografia, (B) cartella colori, (C) linea di nastro adesivo per posizionare i vasi in modo uniforme, (D) fotocamera digitale, (E) computer a scheda singola, (F) monitor che visualizza il processo di acquisizione delle immagini. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Panoramica del flusso di lavoro di imaging. Viene fornita un'immagine rappresentativa di "alta qualità" e "bassa qualità". Clicca qui per visualizzare una versione più grande di questa figura.
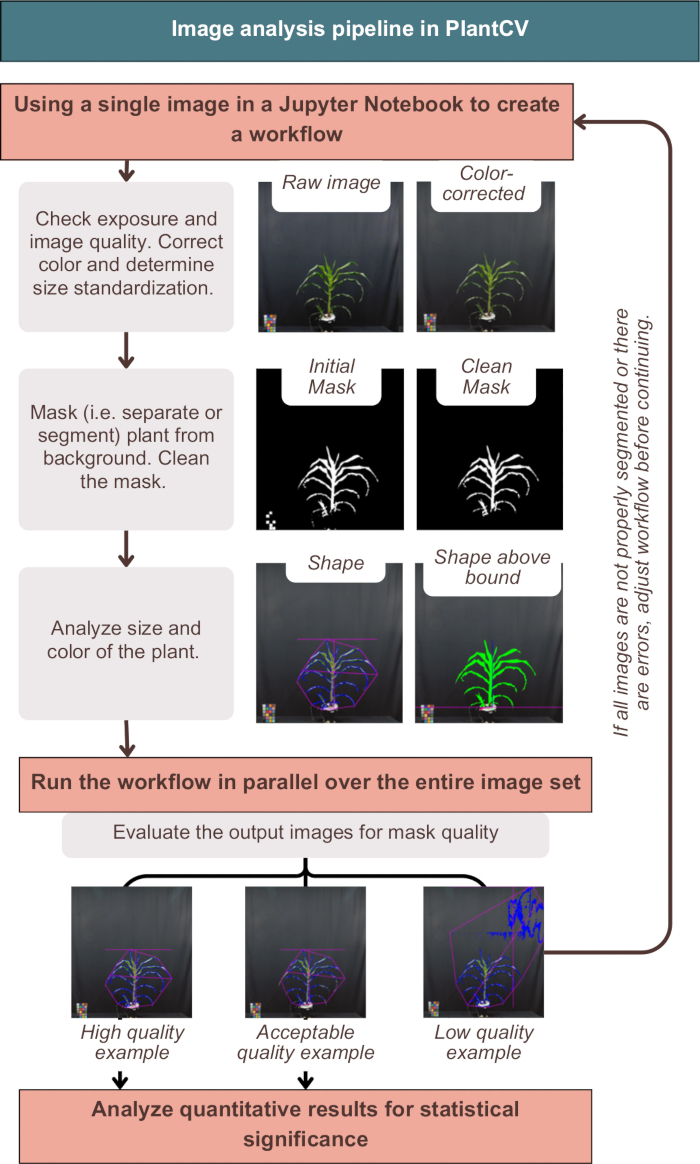
Figura 3: Panoramica del flusso di lavoro dell'analisi delle immagini in PlantCV. Vengono presentate immagini rappresentative di segmentazione di qualità "alta", "accettabile" e "bassa"; I tratti PlantCV sono rappresentati utilizzando un contorno rosa della forma dell'intera pianta e un contorno blu dell'area della pianta. Clicca qui per visualizzare una versione più grande di questa figura.
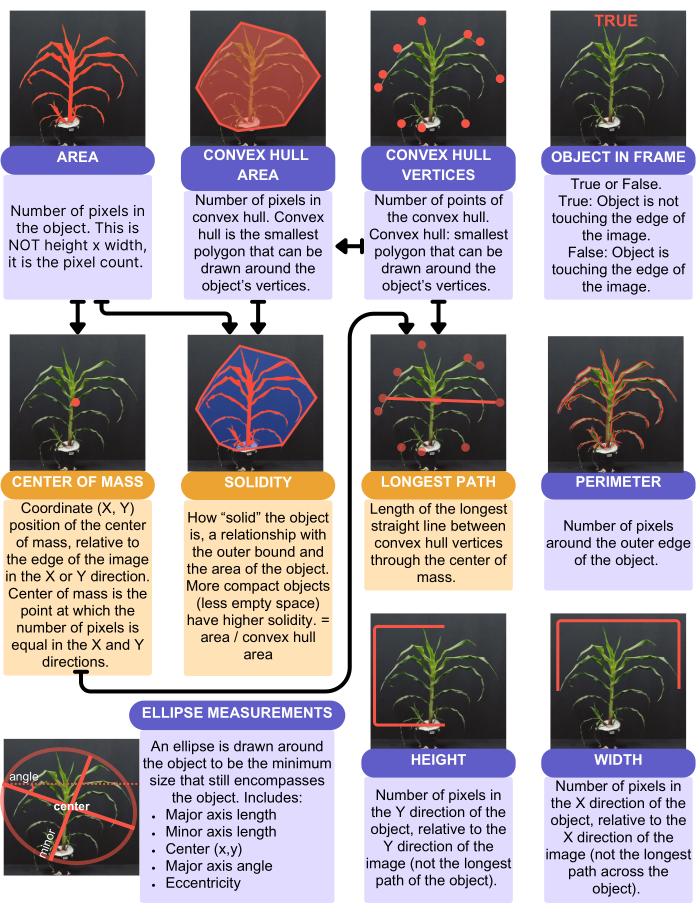
Figura 4: Sintesi e rappresentazione visiva dei tratti di forma acquisiti dall'analisi. Funzione di forma in PlantCV. Le caselle viola rappresentano i tratti che non utilizzano un altro tratto nel loro calcolo. Le caselle arancioni rappresentano i tratti che utilizzano altri tratti nel loro calcolo; Le frecce indicano quali tratti vengono utilizzati in questi calcoli. Clicca qui per visualizzare una versione più grande di questa figura.
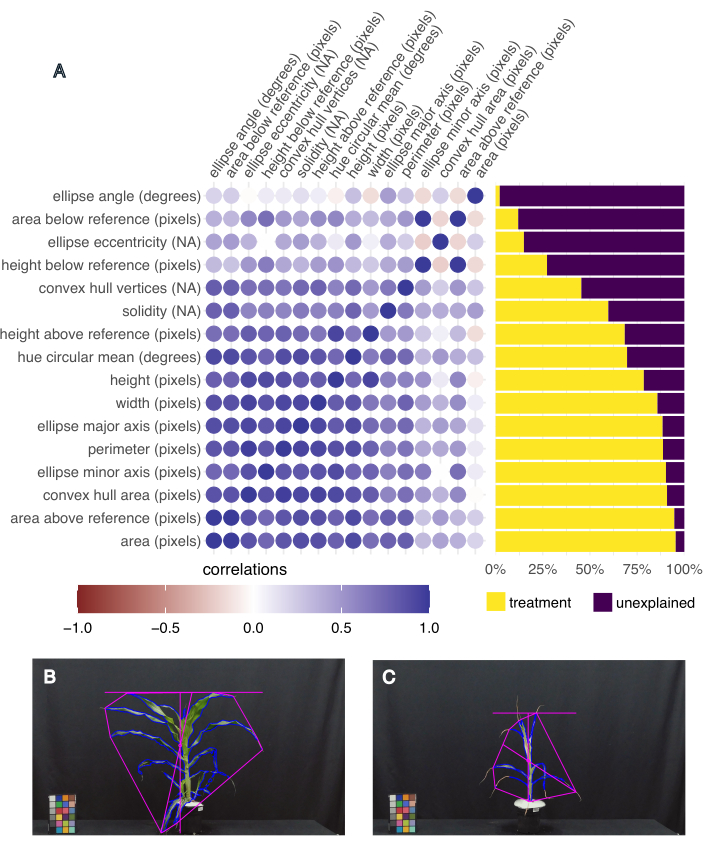
Figura 5: La fenotipizzazione basata su immagini cattura la varianza fenotipica e i tratti correlati. (A) Varianza dei caratteri spiegata dal trattamento (a destra) e correlazione dei caratteri tra loro (a sinistra). Ogni tratto è etichettato, seguito dalla sua unità di misura; "NA" significa che si tratta di una misura senza unità. (B) Immagine della pianta che aveva la più alta area fogliare e la più alta tonalità, media circolare (gradi). (C) Immagine della pianta che aveva l'area fogliare più piccola e la tonalità circolare più bassa media (gradi). Clicca qui per visualizzare una versione più grande di questa figura.
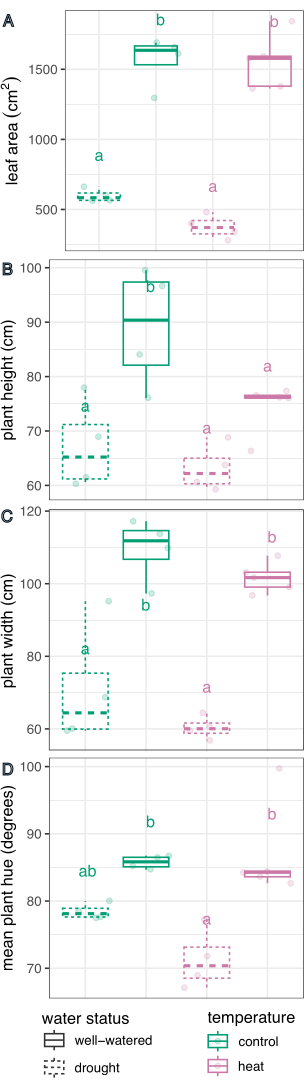
Figura 6: Confronto boxplot degli effetti del trattamento sui fenotipi delle piante. L'area fogliare (A), l'altezza (B), la larghezza (C) e la tonalità circolare media (D) vengono confrontate per l'effetto della siccità, del calore e degli stress combinati. n = 4. Le lettere rappresentano differenze statisticamente significative tra i trattamenti (p < 0,05); i trattamenti sono stati confrontati utilizzando la regressione lineare e il test post-hoc (medie marginali stimate) con una correzione di Sidak per confronti multipli. Clicca qui per visualizzare una versione più grande di questa figura.
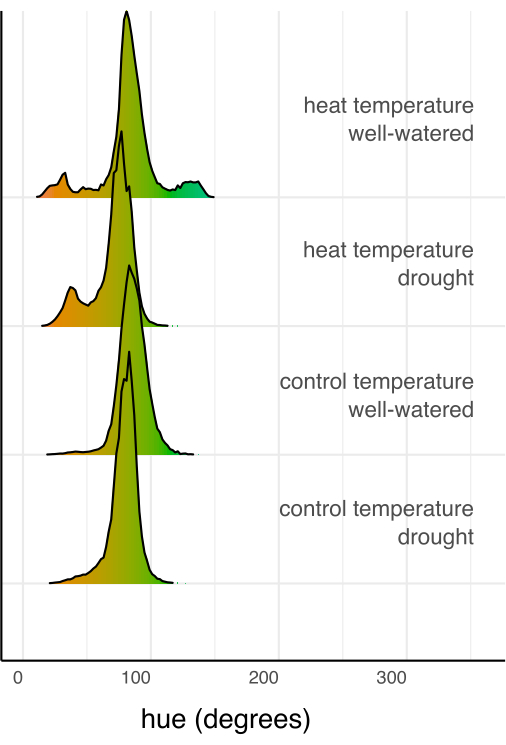
Figura 7: Istogrammi di colore (tonalità) sommati di tutti i pixel di ciascuna combinazione di trattamento, normalizzati dal numero totale di pixel nella combinazione di trattamento. Gli istogrammi sono colorati in base al valore del colore per il grado di tonalità. Clicca qui per visualizzare una versione più grande di questa figura.
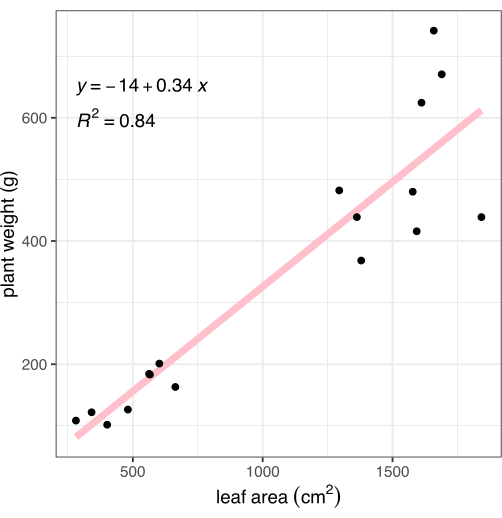
Figura 8: Correlazione dell'area fogliare misurata dalle immagini alla biomassa dell'intera pianta. Ogni punto nero rappresenta una pianta. La linea rosa rappresenta la correlazione lineare tra l'area fogliare (X) e la biomassa (Y). Clicca qui per visualizzare una versione più grande di questa figura.
| Tratto | Fattore | gradi di libertà | Valore F | Valore p |
| area fogliare (cm^2) | Temperatura | 1 | 0.5372 | 0.4766 |
| area fogliare (cm^2) | Acqua | 1 | 226.5318 | 1.32E-09 |
| area fogliare (cm^2) | Temperatura:Acqua | 1 | 2.1526 | 0.1661 |
| area fogliare (cm^2) | Residui | 13 | ||
| Altezza (cm) | Temperatura | 1 | 5.9426 | 0.0298935 |
| Altezza (cm) | Acqua | 1 | 21.107 | 0.0005029 |
| Altezza (cm) | Temperatura:Acqua | 1 | 2.1752 | 0.1640545 |
| Altezza (cm) | Residui | 13 | ||
| larghezza (cm) | Temperatura | 1 | 2.2495 | 0.1575 |
| larghezza (cm) | Acqua | 1 | 75.5474 | 8.92E-07 |
| larghezza (cm) | Temperatura:Acqua | 1 | 0.0839 | 0.7766 |
| larghezza (cm) | Residui | 13 | ||
| Tonalità media circolare (gradi) | Temperatura | 1 | 0.9154 | 0.3561432 |
| Tonalità media circolare (gradi) | Acqua | 1 | 27.7988 | 0.0001509 |
| Tonalità media circolare (gradi) | Temperatura:Acqua | 1 | 3.4792 | 0.0848724 |
| Tonalità media circolare (gradi) | Residui | 13 |
Tabella 1: Risultati del modello di regressione lineare e delle analisi ANOVA a due vie che esaminano gli effetti del trattamento dell'acqua, del trattamento della temperatura e della loro interazione sui tratti delle piante.
Discussione
La fenotipizzazione basata su immagini fornisce misurazioni accurate, rapide e quantitative dei fenotipi delle piante 4,5,6,7. Questo metodo è significativamente più veloce, più preciso, più accurato e fornisce un numero maggiore di misurazioni rispetto all'utilizzo manuale di un righello per registrare l'altezza della pianta o misure qualitative delle caratteristiche del colore della pianta che possono riguardare la salute 4,5,6,7. L'area fogliare è stata osservata qui, e in una precedente ricerca10, fortemente correlata alla biomassa vegetale (Figura 8), suggerendo che questa misura potrebbe sostituire in modo affidabile il processo dispendioso in termini di tempo, distruttivo e soggetto a errori di pesatura manuale della biomassa dell'intera pianta. Mentre una sola telecamera poteva essere utilizzata per acquisire le immagini, l'aggiunta dell'SBC a basso costo ha aumentato la produttività attraverso l'automazione della raccolta delle immagini e dell'etichettatura delle immagini con i metadati necessari15. Questo metodo è adatto da una a ~300 piante che vengono visualizzate una volta al giorno (o meno frequentemente). Per un numero maggiore di piante in un esperimento, un sistema di imaging basato su nastro trasportatore, a portale aereo o abilitato a robot è più adatto poiché il tempo per spostare le piante singolarmente per l'imaging può essere più lungo di un giorno lavorativo11,13. Per i punti temporali più frequenti (ad esempio, un'immagine ogni ora piuttosto che una volta al giorno o meno frequentemente), le configurazioni della fotocamera all'interno di una camera di crescita sono più adatte perché catturano automaticamente le immagini, ma sono limitate dalle dimensioni delle piante e necessitano di SBC15 aggiuntivo.
Mentre PlantCV è presentato come l'opzione di analisi delle immagini qui, esistono altri software. ImageJ (Fiji) è un pacchetto software gratuito per l'analisi delle immagini che può essere utilizzato anche per le immagini delle piante attraverso un'interfaccia utente grafica (GUI)8; tuttavia, non è particolarmente sviluppato per l'analisi delle immagini dell'intero impianto, è meno adattabile al calcolo ad alto rendimento ed è meno facilmente ripetibile rispetto alle pipeline già funzionanti come quelle di PlantCV che possono essere facilmente applicate a nuove immagini9. I metodi di Deep Learning (come BioDock, vedi Tabella dei materiali) e Segment Anything22) sono interessanti per la loro capacità di segmentare facilmente le piante dallo sfondo e potrebbero essere utilizzati per le attività di segmentazione descritte qui. Tuttavia, questi modelli di Deep Learning non forniscono le misurazioni del fenotipo vegetale descritte qui10 e per effettuare queste misurazioni più complesse dei fenotipi quantitativi può essere necessaria una grande quantità di immagini di addestramento (spesso superiori a 10.000) e una potenza di calcolo superiore a quella di un laptop o di un computer desktopdi base 10. Questi modelli richiedono anche una notevole memoria del computer e potenza di elaborazione, che non sono disponibili per molti ricercatori10.
La fase più critica del protocollo consiste nell'organizzare la configurazione dell'imaging della pianta e garantire che non vi siano oggetti (come altre piante, una cartella colori o detriti vegetali) vicino alla pianta di interesse14. Se le immagini non sono impostate in modo appropriato o non viene utilizzata una scheda colore, sarà impossibile analizzarle in un secondo momento perché non esiste un meccanismo per la standardizzazione. Sebbene in questo protocollo non siano necessarie fasi di calibrazione, la correzione del colore e la standardizzazione delle dimensioni durante l'elaborazione sono essenziali. Assicurarsi sempre che l'intera cartella colori sia nell'inquadratura, ma non sovrapposta alla pianta (Figura 2). Un'altra insidia comune è l'illuminazione non uniforme, che può causare ombre o scatti luminosi; si consiglia di utilizzare una luce dall'alto intensa e diffusa e di coprire le finestre della stanza per evitare un'illuminazione irregolare (Figura 2). È possibile aggiungere ulteriori lampade con un angolo rivolto verso l'alto rispetto alla pianta se le luci dall'alto causano ombre. Il tipo di lampadina non è un problema, dato che il colore verrà corretto nell'immagine utilizzando una cartella colori. Le immagini sfocate non sono accettabili per l'analisi: utilizzare la messa a fuoco automatica sulla fotocamera o le istruzioni del produttore per la messa a fuoco manuale per garantire che le immagini siano chiare.
Allo stesso modo, assicurarsi che le immagini non siano sovraesposte (un errore comune) consente un'analisi appropriata (Figura 2); Le immagini sovraesposte non possono essere corrette in una correzione del colore durante l'analisi dell'immagine perché i valori dei pixel sono oltre l'intervallo di rilevamento della fotocamera con le impostazioni correnti14. Per evitare la sovraesposizione, valutare gli istogrammi del colore o i valori RGB per un'immagine di prova utilizzando il flusso di lavoro PlantCV descritto qui (Figura 3) e ridurre l'impostazione della luminosità sulla fotocamera se il test di esposizione determina che l'immagine è sovraesposta. I valori RGB (e quindi gli istogrammi) non dovrebbero avere valori a 0 o a 255, che è l'intervallo di valori perché è possibile che tali valori siano superiori o inferiori all'intervallo di rilevamento e possano registrare solo il valore minimo o massimo piuttosto che il valore reale. Un'immagine di prova dovrebbe sempre essere valutata utilizzando questi metodi per la sovraesposizione (Figura 2 e Figura 3); suggeriamo di utilizzare il controllo di qualità dell'esposizione PlantCV fornito in questa pubblicazione come controllo, che determina che un'immagine di qualità abbia meno del 5% di pixel a 0 o 255 per ogni colore. Come regola generale, le telecamere e il software di analisi forniscono istogrammi, che dovrebbero avere code di istogramma uguali e complete; I valori tagliati al di sotto di 0 (sottoesposto) o al di sopra di 255 (sovraesposto) devono essere ripresi con le impostazioni della fotocamera regolate. Ti consigliamo di scegliere un luogo con un'illuminazione artificiale intensa, uniforme e senza finestre per l'illuminazione, poiché le finestre possono essere troppo luminose, irregolari e cambiare frequentemente l'esposizione alla luce nello spazio. Le immagini devono apparire illuminate in modo uniforme e i quadrati grigi di una scheda di colori non devono apparire bianchi, il che suggerisce una sovraesposizione.
Potrebbe essere necessaria la risoluzione dei problemi per il codice di acquisizione dell'immagine a seconda dell'IP e delle impostazioni di sicurezza dell'istituto dell'utente. Per risolvere il problema, contattare il reparto IT per problemi di connettività Internet e trasferimento file. Un errore comune è l'utilizzo di una scheda SD di dimensioni errate per l'SBC o l'accensione dell'SBC prima dell'accensione del monitor collegato. Per risolvere il problema, seguire esattamente il protocollo sopra e ricaricare il software SBC. I forum online sono un'ottima risorsa per la risoluzione dei problemi e la ricerca di soluzioni per la fotocamera specifica scelta quando si utilizza gphoto2. Per risolvere i problemi relativi all'analisi delle immagini PlantCV e utilizzare il controllo delle versioni più aggiornato, utilizzare la pagina della documentazione (https://plantcv.readthedocs.io/en/latest/) o pubblicare un problema su GitHub (https://github.com/danforthcenter/plantcv/issues). In particolare, se il metodo di soglia in questo protocollo non separa sufficientemente la pianta dallo sfondo, utilizzare metodi di soglia alternativi forniti in "photo-studio-SV-notebook.ipynb" o nella pagina della documentazione.
Questo metodo può essere applicato a qualsiasi specie vegetale e trattamento che può essere trasportato alla configurazione fototografica assemblata descritta. Ad esempio, non è applicabile alle piante che crescono in un campo, poiché non possono essere trasportate alla configurazione fotografica senza scavare la pianta. In questa situazione, le piante potrebbero essere abbattute e successivamente visualizzate utilizzando il protocollo, anche se la misurazione sarebbe distruttiva. È possibile acquisire più piante in un'unica immagine e analizzarle, ma l'utente deve fare attenzione a evitare che le piante si tocchino (Figura 2) e le piante vengono posizionate ogni volta nella stessa posizione sullo sfondo per garantire che il flusso di lavoro possa essere eseguito automaticamente sull'intero set di immagini. Se è necessaria una vista dall'alto verso il basso per piante come l'Arabidopsis thaliana, utilizzare un treppiede per spostare la fotocamera sulla parte superiore delle piante posizionate sul tessuto fotografico; I restanti metodi per l'acquisizione e l'analisi delle immagini rimangono gli stessi.
Come dimostrato qui con il mais, questo metodo è importante sia per la selezione delle piante che per le intuizioni fondamentali su come le piante tollerano gli stress abiotici, come il caldo e la siccità. Il caldo e lo stress da siccità contribuiscono a una perdita di resa del 15%-20% nel mais ogni anno in tutto il mondo23. Si prevede che le perdite di resa aumenteranno con il cambiamento climatico globale, poiché si prevede che le temperature aumenteranno di 1,4-4,4°C entro il 210024. La valutazione della diversità del mais per la tolleranza allo stress, così come la determinazione dei meccanismi fenotipici, fisiologici e molecolari della tolleranza allo stress abiotico, sono obiettivi di ricerca fondamentali per la sicurezza alimentare globale25. Per rispondere a queste domande, i ricercatori di tutti i campi adiacenti - fisiologi, genetisti, selezionatori, biologi molecolari, ecc. - devono essere in grado di valutare in modo rapido, efficace e spesso non distruttivo i fenotipi delle piante di mais in condizioni di stress abiotico25. Il metodo e i risultati dell'esempio mostrati qui rispondono a questa esigenza e sono uno strumento importante per i ricercatori per affrontare le sfide agricole del mondo reale.
I fenotipi osservati qui sono stati ipotizzati, data una ricchezza di ricerche precedenti: lo stress da siccità riduce le dimensioni e il verde delle piante (misurato dalla media circolare della tonalità)26 (Figura 6, Figura 7 e Tabella 1). Il fenotipo dello stress da calore con l'irrigazione inaspettata del pozzo non ha ridotto significativamente l'area fogliare, anche se con più repliche, questa relazione potrebbe essere valutata meglio statisticamente e l'altezza è stata ridotta come previsto (Figura 6). Mentre la media circolare della tonalità non è stata modificata a causa del calore, il profilo cromatico è stato effettivamente modificato per includere sia pixel più gialli che verdi più scuri, dimostrando che la valutazione della sola tonalità media è insufficiente e che dovrebbe essere preso in considerazione l'istogramma del colore con tratti multivalore (Figura 7). Questi cambiamenti fenotipici descrivono la risposta di una pianta all'ambiente e tecniche aggiuntive come le misurazioni dello scambio di gas, l'imaging al microscopio e altro ancora potrebbero essere impiegate per valutare i cambiamenti fisiologici che si verificano in risposta allo stress. Questo risultato dimostra la potenza di misurazioni precise e quantitative che altrimenti andrebbero perse utilizzando misure manuali o qualitative della salute delle piante. Queste informazioni sono fondamentali per l'allevamento e l'ingegnerizzazione di colture resistenti ai cambiamenti climatici, che riducono significativamente i raccolti globali e si prevede che peggioreranno negli anni futuri.
Divulgazioni
Nessuno.
Riconoscimenti
Questo lavoro è stato condotto presso la Phenotyping Core Facility, Donald Danforth Plant Science Center (RRID: SCR_019049), che ha fornito supporto finanziario a K.M.M. e J.G.D. Questo lavoro è stato sostenuto dal Donald Danforth Plant Science Center Enterprise Rent-A-Car Research Institute Grant. D.S. è stato finanziato dal Taylor Geospatial Institute Block Grant e da una sovvenzione (ED21HDQ0240072) del Centro per l'amministrazione dello sviluppo economico degli Stati Uniti per l'AgTech e la scienza e la tecnologia della localizzazione applicata (CATALST). Ringraziamo Kevin Reilly e Kris Haines (Integrated Plant Growth Facility, Donald Danforth Plant Science Center, RRID: SCR_024902) per la loro competenza e assistenza nella crescita delle piante. Ringraziamo Noah Fahlgren, Malia Gehan e Haley Schuhl per i loro consigli su PlantCV.
Materiali
| Name | Company | Catalog Number | Comments |
| 15W USB-C Power Supply | CanaKit | DCAR-RSP-3A5-C | |
| 25604 UC-E6 USB Cable | Nikon | 18208256044 | |
| 3.5 inch pots | Hummert International | 13006000 | |
| Berger BM7 35% Bark HP | Hummert International | 10121500 | |
| Biodock 2024 | AI Software Platform | www.biodock.ai. | |
| Code editing software | Jupyter | https://jupyter.org/install | |
| Code editing software | Visual Studio Code | https://code.visualstudio.com/download | |
| Color Card ColorChecker Classic | Calibrite | CCC | |
| Color Card ColorChecker Passport | Calibrite | CCPP2 | |
| Conviron Growth Chamber | Conviron | BDW80 model | |
| Dell 22 Monitor | Dell | SE2222H | |
| EH-67 AC Adapter | Nikon | AZ185 | |
| Image analysis software | PlantCV | https://plantcv.readthedocs.io/en/latest/installation/ | |
| Jack's Water Soluble Fertilizer 15-5-15 | Hummert International | 7590200 | liquid fertilizer |
| Keyboard and Mouse | Logitech | 1440638 | MK270 wireless keyboard and mouse |
| Maize seed | USDA-ARS Germplasm Resources Information Network (GRIN) | B73 | https://www.ars-grin.gov/ |
| Micro HDMI to HDMI cable | Amazon | B07KSDB25X | |
| Nikon Coolpix B500 camera | Nikon | ||
| Nikon D7200 camera | Nikon | ||
| Osmocote 14-14-14 | Hummert International | 7630000 | |
| Photography-grade fabric | Yayoya | Polyester Fabric Black Photo Backdrop for Photo Video Studio Televison | |
| Raspberry Pi 4 Case with heatsink | iUniker | B07ZVKN262 | |
| Raspberry Pi 4 Model B Rev 1.1 | Raspberry Pi Foundation | RP4B4GB_BP | |
| Raspberry Pi 4 PiSwitch (USB-C) | CanaKit | RSP-PISWITCH-USBC | |
| Raspberry Pi software | Raspberry Pi Imager | https://www.raspberrypi.com/software/ | |
| Statistical analysis software | Rstudio | https://posit.co/downloads/ | |
| Tripod | K&F Concept | T255A3+BH-28L | |
| Turface MVP | Hummert International | 10240000 | |
| Wireless Barcode Scanner | Netumscan | B08X4NMX2M | 1D and 2D barcode scanner |
Riferimenti
- Xiao, Q., Bai, X., Zhang, C., He, Y. Advanced high-throughput plant phenotyping techniques for genome-wide association studies: A review. J Adv Res. 35, 215-230 (2022).
- Mutka, A. M., et al. image-based phenotyping methods provide insight into spatial and temporal dimensions of plant disease. Plant Physiol. 172 (2), 650-660 (2016).
- Mutka, A. M., Bart, R. S. Image-based phenotyping of plant disease symptoms. Front Plant Sci. 5, 734 (2014).
- Fahlgren, N., Gehan, M. A., Baxter, I. Lights, camera, action: High-throughput plant phenotyping is ready for a close-up. Curr Opin Plant Biol. 24 (2), 93-99 (2015).
- Das Choudhury, S., Samal, A., Awada, T. Leveraging image analysis for high-throughput plant phenotyping. Front Plant Sci. 10, 508 (2019).
- Yang, W., et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives. Mol Plant. 13 (2), 187-214 (2020).
- Omari, M. K., et al. Digital image-based plant phenotyping: A review. Korean J Agric Sci. 47 (1), 119-130 (2020).
- Schindelin, J., et al. Fiji: An open-source platform for biolFiogical-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gehan, M. A., et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping. PeerJ. 5, e4088 (2017).
- Murphy, K. M., Ludwig, E., Gutierrez, J., Gehan, M. A. Deep learning in image-based plant phenotyping. Annu Rev Plant Biol. 75 (1), 771-795 (2024).
- Fahlgren, N., et al. A versatile phenotyping system and analytics platform reveals diverse temporal responses to water availability in Setaria. Mol Plant. 8 (3), 1520-1535 (2015).
- Yu, L., et al. Development of a mobile, high-throughput, and low-cost image-based plant growth phenotyping system. bioRxiv. , (2023).
- Atefi, A., Ge, Y., Pitla, S., Schnable, J. Robotic technologies for high-throughput plant phenotyping: Contemporary reviews and future perspectives. Front Plant Sci. 12, 611940 (2021).
- Berry, J. C., Fahlgren, N., Pokorny, A. A., Bart, R. S., Veley, K. M. An automated, high-throughput method for standardizing image color profiles to improve image-based plant phenotyping. PeerJ. 6, e5727 (2018).
- Tovar, J. C., et al. Raspberry Pi-powered imaging for plant phenotyping. Appl Plant Sci. 6, e1031 (2018).
- Jolles, J. W. Broad-scale applications of the Raspberry Pi: A review and guide for biologists. Methods Ecol Evol. 12 (5), 1562-1579 (2021).
- Watt, M., et al. Phenotyping: New windows into the plant for breeders. Annu Rev Plant Biol. 71 (1), 689-712 (2020).
- Xie, C., Yang, C. A review on plant high-throughput phenotyping traits using UAV-based sensors. Comput Electron Agric. 178, 105731 (2020).
- Czedik-Eysenberg, A., et al. The "PhenoBox", a flexible, automated, open-source plant phenotyping solution. New Phytol. 219 (4), 808-823 (2018).
- Sumner, J., Fahlgren, N., Murphy, K. M. PCVR: An R package and tutorials for guided statistical analysis of plant phenotyping data. Authorea Preprints. , (2023).
- Schnable, P. S., et al. The B73 maize genome: complexity, diversity, and dynamics. Science. 326 (5956), 1112-1115 (2009).
- Kirillov, A., et al. Segment Anything. , 3992-4003 (2023).
- Gabaldón-Leal, C., et al. Modelling the impact of heat stress on maize yield formation. Field Crops Res. 198 (3), 226-237 (2016).
- Intergovernmental Panel on Climate Change. . SYNTHESIS REPORT OF THE IPCC SIXTH ASSESSMENT REPORT (AR6). , (2023).
- Quiñones, A., et al. Optimized methods for applying and assessing heat, drought, and nutrient stress of maize seedlings in controlled environment experiments. Cold Spring Harb Protoc. , (2024).
- Murphy, K. M., et al. Maize abiotic stress treatments in controlled environments. Cold Spring Harb Protoc. , (2024).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon