Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identification of Growth Inhibition Phänotypen durch Expression von bakteriellen Typ III Effektoren in Hefe Induced
In diesem Artikel
Zusammenfassung
In diesem Video, beschreiben wir ein Verfahren zur Expression von bakteriellen Typ III Effektoren in Hefe und die Identifizierung der Effektor-induzierte Wachstumshemmung Phänotypen. Solche Phänotypen können anschließend genutzt werden, um Effektor-Funktionen und Ziele zu erläutern.
Zusammenfassung
Viele Gram-negative pathogene Bakterien mit einem Typ III Sekretionssystem für eine Suite von Effektor-Proteine in das Cytosol der Wirtszellen zu translozieren. Innerhalb der Zelle unterwandern Typ III Effektoren Host zelluläre Prozesse, Immunantworten zu unterdrücken und zu fördern Wachstum des Pathogens. Zahlreiche Typ III Effektoren der pflanzlichen und tierischen bakterielle Erreger wurden bisher identifiziert, doch nur wenige von ihnen sind gut charakterisiert. Das Verständnis der Funktion dieser Effektoren wurde durch eine Kombination von funktionellen Redundanz wurde in den Effektor Repertoire eines bestimmten Bakterienstamm untergraben, die subtilen Wirkungen, die sie ausüben, um Virulenz, Rollen, die möglicherweise spezifisch für bestimmte Infektionen Stufen sind, und Schwierigkeiten bei der genetisch erhöhen können Manipulation bestimmter Krankheitserreger. Expression von Typ III Effektoren in der Hefe
Protokoll
I. Entwurf eines Yeast Expression System für Typ III Effektoren
Kalibrieren eines Hefe-System geeignet für die Expression des Typ III Effektor (s) von Interesse ist eine wichtige Aufgabe und kann einige trial and error erfordern. Faktoren von großer Bedeutung, dass betrachtet und optimiert werden sollte bei der Gestaltung eines solchen Systems sind: 1) den Promotor die Expression des Effektors (s), 2) die Kopienzahl der Effektor-Gen, 3) die Epitop-Tag verwendet, um Protein-Expression zu überprüfen und 4) den Hefestamm.
1) Promoter
Da bakterielle Typ III Effektoren können giftig sein, um Hefezellen, sollte ein induzierbarer Promotor verwendet werden, um ihren Ausdruck zu kontrollieren. Die GAL1 Promotor ist üblicherweise für diesen Zweck verwendet und seine Aktivität wird durch die C-Quelle im Nährmedium geregelt: Es wird von Glucose reprimiert und induziert durch Galaktose. Allerdings wurde dieser Promotor als leicht undicht unter Unterdrückung Bedingungen, die sich in unerwünschten Toxizität berichtet. Wenn ein ähnliches Problem aufgetreten ist, ist es ratsam, die Kopienzahl der Effektor-Gen zu minimieren, wie unten beschrieben, oder alternative Promotoren, wie die MET3 oder CUP1 Promotoren 1 zu verwenden. Hier beschreiben wir Verfahren zur Expression von Effektor-Proteine aus einer Galaktose-induzierbarer Promotor.
2) Gene copy-number
Ein weiterer Parameter auf die Höhe der Proteinexpression ist die Anzahl der Kopien der Effektor-Gen in der Zelle. Hohe Expression erreicht werden, wenn der Effektor-Gen durch durchgeführt ist ein 2-Mikron-Plasmid (40-60 Kopien pro Zelle). Intermediate Ausdruck wird mit einem Zentromer-haltigen Plasmid (1-3 Kopien pro Zelle), während niedrige Ausdruck ist erreicht, wenn der Effektor-Gen in die Hefe-Genoms durch homologe Rekombination integriert erhalten. Durch die Kombination einer Zentromer-haltigen Vektor und die GAL1 Promotor haben wir erfolgreich ca. 50 Effektoren des Pflanzenpathogene Xanthomonas campestris pv. Vesicatoria und Pseudomonas syringae pv. ausgedrückt. Tomaten auf einem Niveau nachweisbar durch Immunoblot-Analyse und mit vernachlässigbarer Leckstrom bei unterdrücken Bedingungen (Salomon und Sessa , unveröffentlicht).
3) Epitop-Tag
Wenn Antikörper gegen den Effektor von Interesse nicht verfügbar sind, ist ein Epitop auf die Effektor-Protein fusioniert, um seinen Ausdruck durch Immunoblot überwachen. Häufig verwendete Tags sind Myc, Hämagglutinin (HA) oder Flag, dem zuverlässigen kommerziellen Antikörper verfügbar sind. Wir empfehlen die Verwendung nur einer Kopie der Tag, um unerwünschte schädliche Wirkungen auf die Struktur und Aktivität der Effektor-Proteine zu minimieren.
4) Der Hefestamm
Eine grundlegende Anforderung für den Hefestamm, um die Expression verwendet werden soll Auxotrophie der Selektionsmarker des Expressionsvektors. Es ist wichtig zu beachten, dass verschiedene Hefestämme können unterschiedliche Empfindlichkeiten, um die Expression bestimmter Effektoren anzuzeigen. Zum Beispiel beobachten wir, dass die BY4741 und W303-Stämme unterschiedliche Empfindlichkeiten zu mehreren Xanthomonas campestris pv. Vesicatoria Effektoren (Salomon und Sessa, unveröffentlicht) haben. Daher ist es vorzuziehen, den Effektor (s) von Interesse in verschiedenen Hefestämme zu testen.
II. Vorbereitung der Hefe Medien
Für das Wachstum von Hefe-Stämme, die keine beliebigen Vektors, verwenden YPD (10 g / L Hefeextrakt, 20 g / L Pepton und 2% [w / v] Glukose) als Wachstumsmedium. Für feste Medien, fügen Sie 2% [w / v] Agar, um die Lösung (wenn das feste Medium ist sehr weich, fügen 0,05% [v / v] aus einer 5 N NaOH-Stammlösung auf das Medium vor dem Autoklavieren). Wir empfehlen der Herstellung des Mediums ohne Glukose in 90% der endgültigen Volumen und Autoklavieren. Kurz vor der Verwendung, füllen Sie die Lautstärke auf 100% mit einem Filter-sterilisiert 20% [w / v] Glucose-Lösung (halten 20% Glucose-Lösung bei 4 ° C zur Vermeidung von Kontaminationen).
Für das Wachstum von Hefe-Stämme, die einen Vektor, der Prototrophie bietet einen Selektionsmarker (Leucin, Uracil, Histidin oder Tryptophan), synthetische Drop-out-Medium ohne Leucin, Uracil, Histidin und Tryptophan (6,7 g / L Yeast Nitrogen Base ohne Aminosäuren , 1,4 g / L Hefe synthetischen Drop-out-Medium zu ergänzen) und mit 2% [w / v] Glucose oder 2% [w / v] Galaktose und 1% [w / v] Raffinose. Für feste Medien, fügen Sie 2% [w / v] Agar, um die Lösung. Wir empfehlen der Herstellung des Mediums in 90% der endgültigen Volumen und Autoklavieren. Kurz vor der Verwendung, füllen Sie die Lautstärke auf 100% mit einem Filter-sterilisiert 20% Glukose-Lösung oder ein Filter-sterilisiert 20% Galaktose + 10% Raffinose-Lösung, je nach der gewünschten Kohlenstoffquelle (halten 20% Glucose und 20% Galaktose + 10% Raffinose Lösungen bei 4 ° C zu verhindern Kontamination). Abhängig von der Selektionsmarker des Ausdrucks vector, fügen Sie die anderen Aminosäuren oder Uracil vor Gebrauch (10 ml / L von einem 1 g/100 ml Leucin Lager, 10 ml / L von einem 200 mg/100 ml Uracil Lager, 2 ml / L von einem 1 g/100 ml Histidin Lager oder 2 ml / L von einem 1 g/100 ml Tryptophan Lager). Bei der Vorbereitung Leucin und Uracil Aktien, sterilisieren durch Autoklavieren und halten bei Raumtemperatur. Bei der Vorbereitung Histidin und Tryptophan Aktien, sterilisieren durch Filterung, wickeln Sie die Flasche mit Aluminiumfolie vor Licht zu schützen, und halten bei 4 ° C. In den unten beschriebenen Verfahren, synthetische Drop-out-Medium mit Leucin ergänzt wird Uracil, Histidin und Tryptophan als synthetische Vollmedium bezeichnet.
Solid-Medien Platten können gespeichert werden bis zu zwei Monate bei 4 ° C. Vor dem Gebrauch trocknen die Platten für 20 min in einem sterilen Laminarströmungshaube bei Raumtemperatur.
III. Hefe-Transformation
- Vor Beginn der Hefetransformation Verfahren, bereiten Sie die folgenden drei Lösungen: eine 50% [w / v] Polyethylenglykol (PEG) 3350-Lösung, eine 1 M LiAc pH = 8,0, und ein TE-Lösung (100 mM Tris pH = 6.85 und 10 mM EDTA pH = 8,0). Filter-Sterilisation der Lösungen und halten bei 4 ° C.
- Mit einem langen hölzernen Schaft oder einer sterilen Impföse, wählen Sie eine Hefekolonie (1-2 mm Durchmesser) aus einer frischen Platte und beimpfen 3 ml YPD in ein 15 ml Polypropylen-Röhrchen. Vortex kurz. Legen Sie die Kultur Rohr in einer Rolle und Inkubieren bei 30 ° C mit konstanter Rotation über Nacht.
- Am Morgen, entfernen Sie die Kultur von der Walze und Zentrifuge bei 800 g für 5 min bei Raumtemperatur. Nach der Zentrifugation vollständig zu entfernen Überstand.
- Resuspendieren der Zellen in 1 ml Resuspensionspuffer (10% [v / v] TE-Lösung, 10% [v / v] 1 M LiAc Lösung) und vortexen.
- Für jedes Plasmid transformiert werden und eine zusätzliche Kontrolle Transformation, Übertragung 100 ul der Zellsuspension auf eine Mikroröhrchen.
- Zu jedem Röhrchen werden 5 ul der Einzelstrang Lachssperma-DNA (10 mg / ml) und 250-500 ng Plasmid-DNA transformiert (außer für das Steuerrohr) werden.
- Sorgfältig in jedes Röhrchen 650 ul der Transformation-Lösung (10% [v / v] TE-Lösung, 10% [v / v] 1 M LiAc Lösung, 80% [v / v] 50% [w / v] PEG-Lösung add ) und Wirbel.
- Inkubation bei 30 ° C für 30 min auf einer Walze.
- Entfernen Sie die Schläuche aus dem Shaker, fügen Sie 70 ul DMSO und mischen durch Invertieren der Röhrchen 10-15 mal (nicht vortexen, um die Störung der Zellen).
- Legen Sie die Röhrchen in einem 42 ° C vorgewärmten Wasserbad inkubieren 15 min.
- Entfernen Sie das Röhrchen aus dem Wasserbad und sofort legen sie auf Eis für 2 min.
- Nachdem die Rohre abgekühlt sind, Zentrifuge sie bei 800 g für 5 min bei Raumtemperatur.
- Nehmen Sie die Rohre aus dem Mikrozentrifuge und entfernen Sie vorsichtig die viskose Überstand mit einer Pipette.
- Zellpellet jeder Röhre in 150 ul sterilem doppelt destilliertem Wasser (DDW), und die Ausbreitung der Zellsuspension auf einem synthetischen Vollmedium Platte mit 2% Glucose als Kohlenstoffquelle und ohne die Aminosäure-oder Nukleotid an, die das Plasmid bietet Prototrophie.
- Verlassen Sie die Platten offen für 2 min und es ihnen ermöglichen, in einer Sterilbank trocknen. Legen Sie die Platten in einem 30 ° C Inkubator für 2-3 Tage.
- Sobald Kolonien (1-2 mm Durchmesser) auf den Platten erschienen, nehmen rund 10 einzelne Kolonien von jeder Transformation und Übertragung auf einen frischen Teller. Beim Übertragen der Kolonien, um 2 cm x 2 cm Patches für das Wachstum einer großen Menge von Hefezellen zu ermöglichen. Die Inkubation in einem Brutschrank bei 30 ° C für zwei Tage.
- Wenn die Hefe Patches sind gewachsen, entfernen Sie die Platten aus dem Inkubator und versiegelt sie mit Parafilm. Die Platten können bei 4 ° C gelagert werden Transfer-Kolonien zu einem neuen Platte alle 10-14 Tage.
IV. Vorbereiten einer Hefe-Proteinextrakt zu Effektor-Expression durch Immunoblot Überprüfen
- Pick a Hefekolonie (1-2 mm Durchmesser) aus einer frischen Platte in ein 15 ml Polypropylen-Röhrchen mit 3 ml des entsprechenden synthetischen Vollmedium mit 2% Glucose als Kohlenstoffquelle ergänzt und ohne die Aminosäure-oder Nukleotid an, die das Plasmid der Wahl bietet Prototrophie. Legen Sie die Kultur Rohr in einer Rolle beimpft und über Nacht bei 30 ° C.
- In den folgenden Tag, entfernen Sie die Kultur von der Walze und Zentrifuge bei 800 g für 5 min bei Raumtemperatur. Entfernen Sie das Röhrchen aus der Zentrifuge und den Überstand verwerfen.
- Zellpellet in 3 ml sterile DDW und vortexen.
- Centrifuge wieder Überstand verwerfen und die Zellen in 3 ml sterile DDW.
- Transfer-200 ul der Zellsuspension auf ein neues 15 ml Röhrchen mit 3 ml synthetischen Vollmedium mit 2% Galactose und 1% Raffinose als Kohlenstoffquelle ergänzt, und ohne die Aminosäure-oder Nukleotid an, die das Plasmid der Wahl bietet Prototrophie.Durch Vortexen mischen, in eine Rolle, beimpft und über Nacht bei 30 ° C.
- In den folgenden Morgen, je 1 ml der Kultur (oder mehr für low density Kulturen), um eine Mikroröhrchen. Ernten Sie die Zellen durch Zentrifugation bei 800 g für 5 min bei Raumtemperatur.
- Von hier an, in einem Abzug arbeiten, um zu vermeiden Atmen β-Mercaptoethanol giftige Dämpfe. Entfernen Sie vorsichtig den Überstand mit einer Pipette und Zellpellet in 100 ul eiskaltem Lyse-Lösung (4% [v / v] 5 N NaOH, 0,5% [v / v] β-Mercaptoethanol). Gründlich mischen durch Vortexen und inkubieren auf Eis für 30 min.
- Während der Inkubation bestimmt das Volumen der HCl (6 N) für Titrierung 100 ul Lysepuffer auf pH 9-10 erforderlich. Zu diesem Zweck Platz 10 Mikroröhrchen in einem Rack und Transfer 100 ul der Lysepuffer in jedem von ihnen. Add verschiedenen Volumina (1-10 ul) HCl (6 N) in jedes Röhrchen und vortexen. Nehmen Sie eine Probe und den pH-Wert der Lösung mit einem pH-Indikator Streifen.
- Am Ende der Inkubationszeit, fügen Sie die HCl Volumen in den vorherigen Schritt, um das Lysat und mischen durch Vortexen (für eine bessere Genauigkeit, legen Sie die HCl-Lösung auf der Seite des Rohres, ohne Einlegen der Spitze in das Lysat) bestimmt.
- Add 50 ul 3x Probenpuffer (30% [v / v] Glycerin, 15% [v / v] β-Mercaptoethanol, 37,5% [v / v] 500 mM Tris-HCl pH = 6,8, 0,15% [w / v ] Natriumdodecylsulfat [SDS] und einige Körner Bromphenolblau) zum Lysat und mischen durch Vortexen (wenn die Lösung färbt sich gelb, bedeutet dies, dass der pH-Wert zu niedrig ist, und ein paar Mikroliter Lyse-Lösung sollte, bis die Lösung hinzugefügt werden wird blau).
- Kochen Sie die Lysat-Lösung für 5 min auf einem Heizblock nach Punktion ein Loch in den Deckel des Mikroröhrchen mit einer Nadel.
- Entfernen Sie die Mikroröhrchen aus dem Heizblock und lassen Sie die Lösung für 2 min bei Raumtemperatur abkühlen lassen.
- Vortex Lysat-Lösung und dann drehen Sie es nach unten für 10 sec. Last 30 ul auf eine SDS-PAGE-Gel für Immunoblot-Analyse.
V. Spotting Assay zur Erkennung Effektor-induzierte Wachstumshemmung Phenotypes
- Von einem frischen Teller holen einen Hefe-Kolonie (1-2 mm Durchmesser), welches das Expressionsplasmid von Interesse und impfen, in ein 15 ml Polypropylen-Röhrchen, 3 ml synthetischen Vollmedium mit 2% Glucose als Kohlenstoffquelle ergänzt und ohne die Aminosäure -oder Nukleotid, auf die das Expressionsplasmid bietet Prototrophie. Wiederholen Sie diesen Vorgang für ein Steuerelement Hefe-Stamm, der ein leeres Plasmid. Legen Sie die Kulturröhrchen in einer Rolle beimpft und über Nacht bei 30 ° C mit konstanter Rotation.
- In den folgenden Tag, entfernen Sie die Kulturen von der Walze und Zentrifuge bei 800 g für 5 min bei Raumtemperatur. Entfernen Sie das Röhrchen aus der Zentrifuge und den Überstand verwerfen.
- Zellpellet in 3 ml sterile DDW und vortexen.
- Wiederholen Sie den Zentrifugationsschritt und die Zellen in 3 ml sterile DDW.
- Zur Bestimmung der optischen Dichte (OD), Transfer 100 ul jeder Kultur eine Mikroröhrchen mit 900 ul DDW und vortexen. Gießen Sie den Inhalt der Rohre in einem Kunststoff-Küvette und Messung der Absorption bei einer Wellenlänge von 600 nm (Verwendung als Referenz eine Küvette mit 1 ml DDW gefüllt). Berechnen Sie die OD 600 der ursprünglichen Kulturen durch Multiplikation des OD 600 der Kulturen in der Küvette durch einen Verdünnungsfaktor von 10.
- Als nächstes auf der Grundlage der OD 600 der ursprünglichen Kulturen vorzubereiten Zellsuspensionen (1 ml) mit einer OD 600 = 1,0 in sterile Röhrchen.
- Durch die Verwendung von 3 sterile Röhrchen mit 900 ul sterilem DDW gefüllt, bereiten drei 10-fache serielle Verdünnungen (OD 600 = 0,1, 0,01 und 0,001) von jeder Zellsuspension (OD 600 = 1,0) in den vorherigen Schritt vorbereitet.
- Bereiten Sie zwei Platten mit synthetischen Vollmedium ohne die Aminosäure-oder Nukleotid an, die das Plasmid der Wahl bietet Prototrophie. Das Medium einer Platte sollte mit 2% Glucose und die der anderen Platte mit 2% Galaktose und 1% Raffinose ergänzt werden. Trocknen Sie die Platten in einem sterilen Laminarströmungshaube bei Raumtemperatur für 20 min und legen Sie sie auf einem Raster.
- Für jede Kultur, spot 10 ul aus den vier Verdünnungen in einer Reihe auf beiden unterdrücken und induzieren Medien.
- Nach Spotting, lassen Sie die Platten offen in der Motorhaube für einige Minuten. Wenn die Flecken trocken sind, decken sie die Teller und legen Sie sie in einem 30 ° C Inkubator für 2-3 Tage.
- Nach der Inkubation, nehmen Sie die Platten und analysiert das Wachstum der Hefe. Zunächst bestätigen, dass die Zelldichte ähnlich auf der Platte enthalten, unterdrücken Medium. Dann vergleichen Sie das Wachstum der Kultur zum Ausdruck der Effektor-Protein von Interesse mit der Kultur tragen einen leeren Vektor auf der Platte mit Induktion Medium.
Hinweis: Effektoren können zelluläre Prozesse, die nicht geschwindigkeitsbestimmend zu Hefewachstum unter Standard-Labor Wachstumsbedingungen Ziel.Um die Identifizierung von Wachstumshemmung Phänotypen für solche Effektoren können induzierenden Medien mit Verbindungen, die Hefe Zellfunktionen verändert ergänzt werden, wodurch ihre Empfindlichkeit gegenüber Effektoren. Eine Liste der Chemikalien, die möglicherweise in diesem Verfahren integriert werden können, um Parsons et al beziehen. (2004) 2.
VI. Repräsentative Ergebnisse
Ein Vertreter Hefe Spotting-Assay und den Nachweis von Wachstumshemmung Phänotypen durch die Expression von Typ III Effektoren induziert sind in Abb.. 1. In diesem Experiment wurden die Typ-III-Effektoren AvrPto, HopAA1-1 und AvrE1 der Gram-negativen phytopathogenen Bakterium Pseudomonas syringae pv.. Tomate (Pst) aus dem Zentromer-haltigen Plasmid pGML10 in den Hefestamm BY4741 ausgedrückt und auf ihre Fähigkeit zu hemmen das Wachstum der Hefe. Individuelle Hefekulturen Ausdruck Pst Typ III Effektoren unter der Kontrolle des Galaktose induzierbare GAL1 Promoter oder mit einem leeren Vektor wurden seriell verdünnt und ausplattiert auf unterdrücken (Glukose) oder induzieren (Galaktose) Medien (Abb. 1). Auf unterdrücken Medium zeigte Hefestämme tragen Plasmide für die Expression von AvrPto und HopAA1-1 ein ähnliches Wachstum wie die Kontrollstamm mit einer leeren Vektor. Doch auf dem gleichen Medium, Hefe Durchführung AvrE1 ein leicht geringeres Wachstum angezeigt wird, wahrscheinlich zu einem gewissen Grad der Leckage des GAL1 Promoter und die hohe zytotoxische Wirkung von AvrE1 verwandt. In induzierenden Bedingungen, verursacht Ausdruck der AvrE1 Effektorzellen eine drastische Wachstumshemmung Phänotyp durch das Fehlen von Kolonien in einer Verdünnung wider. Wie bereits von Munkvold et al. 3, Ausdruck HopAA1-1 auch in schweren Hemmung des Wachstums führte, beobachtet, während AvrPto zeigten keine Wirkung.
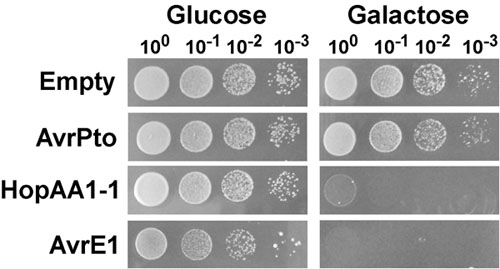
Abbildung 1. Hefe Wachstumshemmung durch Expression des Pseudomonas syringae pv.. Tomate verursacht (Pst) Typ III Effektoren AvrE1 und HopAA1-1. Hefestämme (BY4741), die das Plasmid pGML10, entweder leer oder tragen GAL1-driven-Kassetten für Galaktose-induzierbare Expression von AvrPto , HopAA1-1 oder AvrE1, wurden über Nacht in synthetischen Vollmedium mit Glucose (2%) als Kohlenstoffquelle ergänzt gewachsen, und ohne Leucin. Die Kulturen wurden gewaschen, normiert auf OD 600 = 1,0 und seriellen 10-fach Verdünnungen auf synthetische komplette festen Medien fehlt Leucin und Glucose (2%) oder Galaktose (2%) und Raffinose (1%) wurden gesichtet. Die Fotografien wurden nach 2 und 3 Tagen Wachstum bei 30 ° C für die Hefe wächst in Glukose und Galaktose Medien, bzw. übernommen.
Diskussion
In diesem Vortrag dargestellt wir, wie die Bäckerhefe Saccharomyces cerevisiae als einem heterologen System für die Expression des Typ III bakteriellen Effektor-Proteine und wie Effektor-induzierte Wachstumshemmung Phänotypen identifizieren können. Wichtig ist, dass diese Phänotypen in genetischen Screens genutzt werden, um Unterdrücker der negativen Auswirkungen von Effektoren auf das Wachstum der Hefe zu identifizieren. Suppressors darstellen können entweder direkte Ziele des Effektors studiert o...
Danksagungen
Diese Arbeit wurde von der Israel Science Foundation unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Yeast extract | Difco Laboratories | 212750 | |
| Peptone | Difco Laboratories | 211677 | |
| D-glucose | Sigma-Aldrich | G5767 | |
| Agar | Difco Laboratories | 214010 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Yeast nitrogen base w/o amino acids | Difco Laboratories | 291940 | |
| Yeast synthetic drop-out medium supplement | Sigma-Aldrich | Y2001 | |
| D-galactose | Sigma-Aldrich | G0750 | >99%; <0.1% glucose |
| D-raffinose | Sigma-Aldrich | R0250 | >98% |
| L-leucine | Sigma-Aldrich | L8000 | |
| Uracil | Sigma-Aldrich | U0750 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-histidine | Sigma-Aldrich | H6034 | |
| DNA, single stranded, from salmon testes | Sigma-Aldrich | D7656 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D5879 | Desiccate |
| Hydrochloric acid (HCl) | Sigma-Aldrich | H1758 | |
| Polyethylene glycol (PEG) 3350 | Sigma-Aldrich | P4338 | |
| Lithium acetate (LiAc) | Sigma-Aldrich | L4958 | |
| Tris (base) | JT Baker | 4109-02 | |
| Ethylenediamine-tetraacetic acid (EDTA) | Sigma-Aldrich | E5134 | |
| β-mercapt–thanol | Sigma-Aldrich | M6250 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Bromophenol blue | Sigma-Aldrich | B6131 | |
| Dodecyl sulfate sodium salt (SDS) | Merck & Co., Inc. | 8.22050.1000 | |
| Centrifuge tubes (15 ml) | Corning | 430052 | Sterile |
| Spectrophotometer cuvette (10x4x45 mm) | Sarstedt Ltd | 67.742 | |
| Inoculation loop | Sigma-Aldrich | Z643009 | Sterile |
| Parafilm | Sigma-Aldrich | P7543 | |
| pH indicator strip, pH 6.5-10.0 | Merck & Co., Inc. | 1.09543.0001 |
Referenzen
- Siggers, K. A., Lesser, C. F. The yeast Saccharomyces cerevisiae: a versatile model system for the identification and characterization of bacterial virulence proteins. Cell Host Microbe. 4, 8-15 (2008).
- Parsons, A. B. Integration of chemical-genetic and genetic interaction data links bioactive compounds to cellular target pathways. Nat. Biotechnol. 22, 62-69 (2004).
- Munkvold, K. R., Martin, M. E., Bronstein, P. A., Collmer, A. A survey of the Pseudomonas syringae pv. tomato DC3000 type III secretion system effector repertoire reveals several effectors that are deleterious when expressed in Saccharomyces cerevisiae. Mol. Plant-Microbe Interact. 21, 490-502 (2008).
- Curak, J., Rohde, J., Stagljar, I. Yeast as a tool to study bacterial effectors. Curr. Opin. Microbiol. 12, 18-23 (2009).
- Slagowski, N. L., Kramer, R. W., Morrison, M. F., LaBaer, J., Lesser, C. F. A functional genomic yeast screen to identify pathogenic bacterial proteins. PLoS Pathog. 4, e9-e9 (2008).
- Huang, J., Lesser, C. F., Lory, S. The essential role of the CopN protein in Chlamydia pneumoniae intracellular growth. Nature. 456, 112-115 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten