Method Article
Quantifizierung von Proteinen über ein Peptid Immunoaffinity Enrichment gekoppelt mit der Massenspektrometrie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Stabile Isotope Standards und Capture von Anti-Peptid-Antikörper (SISCAPA) Paare Affinitätsanreicherung von Peptiden mit Isotopenverdünnungsanalyse Massenspektrometrie (MRM-MS) zur quantitativen Messung von Peptiden als Ersatz für ihre jeweiligen Proteine liefern. Hier beschreiben wir das Protokoll mit magnetischen Partikeln in einer teilautomatisierten Format.
Zusammenfassung
Es gibt einen großen Bedarf für quantitative Assays in Messung von Proteinen. Traditionelle Sandwich-Immunoassays, vor allem als der Goldstandard in Quantifizierung sind mit hohen Kosten, langen Lieferzeiten verbunden sind, und sind voller Nachteile (zB heterophile Antikörper, Autoantikörper Störungen "hook-Effekt"). 1 Eine alternative Technik ist Affinitätsanreicherung von Peptiden mit quantitativen Massenspektrometrie gekoppelt, die gemeinhin als SISCAPA (Stable Isotope Standards und Capture von Anti-Peptid-Antikörper) bezeichnet. 2 In dieser Technik Affinitätsanreicherung von Peptiden mit Isotopenverdünnungsanalyse und Detektion von ausgewählten / Multiple Reaction Monitoring-Massenspektrometrie ( SRM / MRM-MS) liefert quantitative Messung von Peptiden als Ersatz für ihre jeweiligen Proteine. SRM / MRM-MS gut ist für eine genaue Quantifizierung kleiner Moleküle 3, 4 eingerichtet und in jüngerer Zeit wurde angepasst, um die Konzentrationen von Proteinen in Plasma-und Zell-Lysaten zu messen. 5-7 Zur Quantifizierung von Proteinen zu erreichen, werden diese größeren Moleküle verdaut Komponente Peptide mit Hilfe eines Enzyms wie Trypsin. Ein oder mehrere ausgewählte Peptide, deren Sequenz ist einzigartig an das Zielprotein in dieser Art (zB "proteotypic" Peptide) werden dann aus der Probe mit anti-Peptid-Antikörper angereichert und gemessen als quantitative stöchiometrische Surrogate für Protein-Konzentration in der Probe. Daher, um Isotopenverdünnungsanalyse (SID) Methoden (dh einer Stachelwalze-in stabilen Isotopen markiertes Peptid-Standard), SRM / MRM kann verwendet werden, um Konzentrationen von proteotypic Peptide als Ersatz für die Quantifizierung von Proteinen in komplexen biologischen Matrices messen. Gekoppelte Die Assays haben mehrere Vorteile im Vergleich zu herkömmlichen Immunoassays. Die Reagenzien sind relativ billiger zu erzeugen, die Spezifität für den Analyten ist ausgezeichnet, die Tests sehr gemultiplext werden können, kann eine Anreicherung von ordentlichen Plasma (kein Abbau erforderlich) durchgeführt werden, und die Technik ist offen für ein breites Spektrum von Proteinen oder Modifikationen von Interesse. 8-13 In diesem Video zeigen wir die grundlegende Protokoll als eine Magnetic-Bead-Plattform angepasst.
Protokoll
Versuchsdurchführung:
Der Test erfordert synthetische Peptide und anti-Peptid-Antikörper. Ausgewählte Peptide sollte eindeutig sein, um das Protein von Interesse, enthalten zwischen 8 und 22 Aminosäuren, und haben keine bekannten post-translationale Modifikationen. Methionin-Reste sind in der Regel vermieden und Peptide, die zweibasische Aminosäuren (zB KK, KR, RR) sind unerwünscht. Bei dieser Technik ist es üblich, stabiles Isotop markierten Peptiden als interne Standards zu verwenden, vereinigt Hochleistungs (13 C und 15 N) markierten Aminosäuren in der C-Terminus des Peptids (dh K oder R gekennzeichnet).
Das folgende Protokoll beschreibt ein Assay entwickelt, um das Peptid GDSLAYGLR von der Maus-Protein Osteopontin messen, mit anti-Peptid-Antikörper aus Epitomics Inc. (Burlingame, CA) und synthetischer Peptide, die aus New England Peptide (Gardner, MA) erhalten. Das Protokoll besteht aus drei Schritten (Abbildung 1): 1) Trypsin Verdauung der komplexen Proteingemisch, 2) Anreicherung von Peptiden 3) Analyse mittels Massenspektrometrie. Es wird auf einer menschlichen Plasmaprobe mit der Maus Osteopontin Protein versetzt nachgewiesen werden.
1. Trypsin enzymatische Verdauung und Bereinigung
- Tauwetter 10 ul ordentlich Plasma Aliquot auf nassem Eis.
- Bestimmen Sie die Gesamt-Protein-Konzentration von BCA-Assay und Zentrifuge die Probe auf eine Entfernung von Schwebstoffen.
- Pipettieren 10 l Aliquot von seiner Lagerung Rohr zu 1000 ul Deepwell Platte und Cover mit Pierce-fähig Film.
- Fügen Sie 20 ml frisches 9M Harnstoff / 30mm Dithiothreitol (DTT) (Endkonzentration 6M Harnstoff / 20mm DTT) zu jeder Probe.
- Inkubieren für 30 Minuten bei 37 ° C.
- Add 3 ml frisches 500 mM iodoacetamide (final IAM 50 mM) zu jeder Probe.
- Inkubieren für 30 Minuten im Dunkeln bei Raumtemperatur.
- Add 257 ul 100 mM Tris (pH 8) (verwässert Harnstoff auf ~ 0,6 Mio.).
- Add 10 l Trypsin-Stammlösung (1 ug / ul; für 01.50 Enzym: Substrat-Verhältnis).
- Inkubieren bei 37 ° C über Nacht (12-16 Stunden).
- Add 3 ul von tausenden von Ameisensäure (Endkonzentration 1%).
- Add stabilen Isotopen-Standard (multiple Standards hinzugefügt werden, wenn der Durchführung einer Multiplex-Assays, in der Regel ist dies etwa 10 ul mit 50-100 fmol von Standard-markiertem Peptid).
- Waschen Sie die Patrone Oasis Platte auch mit 500 ul 0,1% Ameisensäure in 80% Acetonitril, das Verwerfen der Durchströmung. Wiederholen Sie diese 3-fache.
- Äquilibrieren Sie die Patrone Platte durch Zugabe von 500 ul von 0,1% Ameisensäure in Wasser, und entsorgen Sie die Flow-Through. Wiederholen Sie diese 4-fache.
- Legen Sie verdauen Proben, um die Patrone Platte und stellen Sie das Vakuum, so dass die Strömung ist sehr langsam.
- Wash mit 500 ul 0,1% Ameisensäure in Wasser, und entsorgen Sie Flow-Through. Wiederholen Sie diese 3-fache.
- Eluieren Peptide durch Zugabe von 2 x 500 ul von 0,1% Ameisensäure in 80% Acetonitril in 1000 ul Deep-Well-Platte (nicht verwerfen flow-through).
- Lyophilisieren (oder Speedvac) das Eluat. (Gefriertrocknung ist die bevorzugte Methode)
- Rekonstituieren getrockneten Peptide durch Zugabe von 50 ul PBS + 0,03% CHAPS.
2. Peptide Immunaffinitäts Bereicherung
- Übertragen Sie die Probe auf Standard Kingfisher 96 Well-Platten.
- Add 1 pg Antikörper und 1,5 ul Protein-G-beschichteten magnetischen Kügelchen pro Ziel (es ist optional an den Antikörper an die Perlen vor der Zugabe zu vernetzen). Stellen Sie sicher, Perlen sind auch durch Schütteln oder Vortexen suspendiert.
- Die Platte mit Folie.
- Inkubieren über Nacht (12-16 Stunden) mit sanften Taumeln, um sicherzustellen, Perlen aufgehängt sind.
- Centrifuge Platte bei 32 xg für 5 Sekunden, um eine Flüssigkeit aus der Folie Oberfläche zu entfernen.
- Entfernen Sie die Folie abdecken.
- Waschen und Elution der Perlen kann manuell oder in einer automatisierten Weise durchgeführt werden. Dieses Verfahren beschreibt die Schritte auf einem Kingfisher-Plattform automatisiert.
- Waschen Sie die Kugeln 2 mal mit 200 ul PBS + 0,03% CHAPS (1 Minute pro Waschgang).
- Waschen Sie die Kugeln 1 mal mit 200 ul 1:10 Verdünnung des PBS + 0,03% CHAPS (1 Minute).
- Die Peptide werden in 25 uL von 5% Essigsäure + 0,03% CHAPS eluiert.
- Setzen Sie den Elutions-Platte auf einem Magneten und Transfer das Eluat einer 96-Well-Platte, die Pflege nicht auf die übrig gebliebenen Perlen zu übertragen.
- Die Platte mit einem Dichtmatte.
- Die Platte enthält das Eluat ist auf den dreifachen quadrapole Massenspektrometer für die Analyse geliefert.
3. Die Analyse durch multiple reaction monitoring - Massenspektrometrie
- Transitions für SRM / MRM Analyse ausgewählt und durch Infusion einer 1 Picogramm pro Mikroliter Lösung von Peptid-Standard in 30% acetonitrile/0.1% Ameisensäure in das Massenspektrometer bei 0,5 ul / min optimiert werden. Sobald stabile Spray erhalten wird, sammelt ein MS / MS-Spektrum.
- Übergänge sind von der MS / MS-Spektrum durch die Identifizierung thre ausgewählte reichlich Fragment-Ionen in einem Bereich des Spektrums mit wenig Lärm. Für mehrfach geladenen Peptid-Ionen, die y-Ionen-Fragmente bei m / z erkannt> Vorläufer m / z sind in der Regel das Beste für SRM / MRM Assay-Entwicklung. Abbildung 2 zeigt die MS / MS-Spektrum und ausgewählte Übergang Ionen 476,3> 508,3, 579,3 , 692,4 (Übergänge für die schweren stabilen Isotopen Standard 481,3> 518,3, 589,3, 702,4 nicht dargestellt) für das Peptid GDSLAYGLR.
- Je nach Marke und Modell des Massenspektrometers verwendet, gibt es mehrere Parameter, die für jeden Übergang optimiert werden kann. Hier optimierten wir die Aufprallenergie der jeweils ausgewählten Übergang von Rampen die Aufprallenergie und Überwachung der Pegel des Signals. Eine Kollision Energie von 25 wurde für jeden Übergang verwendet.
- Kurz gesagt, ist eine typische Konfiguration für SRM / MRM Analyse wie folgt: Mobile Phase (A) 0,1% Ameisensäure, (B) 90% Acetonitril / 0,1% Ameisensäure, 0,3 x 5 mm C18 Trap-Säule, 75 um ID x 15 cm C18 Trennsäule (Reprosil-Pur C18 AQ, 120 A ° Pore). Das Injektionsvolumen beträgt 10 ul und die Probe für 5 min bei 3% B bei einer Gesamt-Volumenstrom geladen 3 ul / min und eluiert mit einem linearen Gradienten von 3 bis 45% B bei 300 bis 400 nL / min in 10 min. Bedingungen auf einem 4000 QTRAP (ABSCIEX, Foster City, CA) wurden spray Spannung 2.3kV, Ionenquelle 150 ° C, GS1 von 12 und Vorhang Gas 15.
- Die Probe wird durch SRM / MRM durch die Injektion von 10 ul der Probe analysiert. Die Peakflächen sind für leichte und schwere Peptide mit Hilfe des Programms Skyline integriert. 14 Berechnen Sie die Peakflächenverhältnis (PAR) von unmarkiertem / markiertes Peptid in jeder Probe unter Verwendung der am häufigsten vorkommenden Übergang, der frei von Nebengeräuschen ist, in diesem Fall die y5 Übergang (Licht-Peptid 476,3> 579,3, schwere Standard-Peptid 481,3> 589,3).
4. Repräsentative Ergebnisse:
Gemessen Peakflächenverhältnisse (light endogenen Peptids gegenüber versetzt schweren Isotopen-markiertes Peptid) liefern ein quantitatives Maß für die Ziel-Peptid. Abbildung 3 zeigt ein Beispiel-Chromatogramm von leichten und schweren Peptide in einem SISCAPA angereicherten Probe. Beachten Sie, dass leichte und schwere Peptide eluieren gleichzeitig und mehrere Übergänge für jedes Peptid, um die Identität bestätigen können überwacht werden.
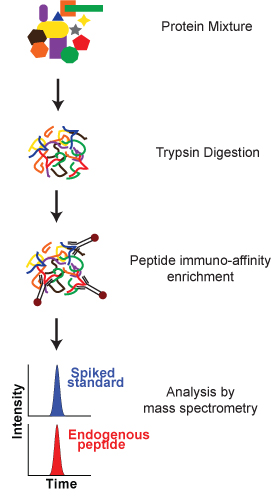
Abbildung 1. Eine schematische Übersicht über die SISCAPA Prozess. Eine komplexe Proteingemisch wird in Peptide verdaut. Gezielte Peptid Analyten (endogenen Analyten und einer Stachelwalze stabile Isotop-markierten internen Standard) angereichert mit anti-Peptid-Antikörper auf Protein G-beschichteten magnetischen Partikeln immobilisiert. Nach der Isolierung sind die gezielte Peptide, die aus den magnetischen Partikeln eluiert und massenspektrometrisch analysiert zur Quantifizierung in Bezug auf den internen Standard.
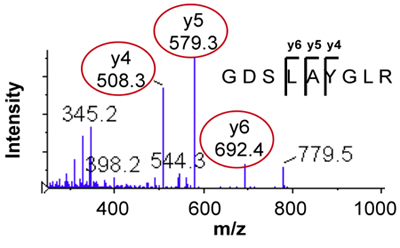
Abbildung 2. MS / MS-Spektrum des Peptids GDSLAYGLR zeigt Auswahl von drei Fragmente für SRM / MRM-Übergänge.
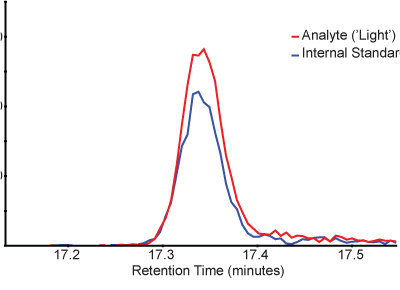
Abbildung 3. Beispiel Chromatogramme zeigen die Peak-Profil eines Licht-Peptid Analyten (rot) und der schweren stabilen Isotopen-markierten internen Standard (blau). Chromatogramme für die y5 Übergang von der Licht-Peptid (476,3> 579,3) und schwere stabile Isotop markiert Standard (481,3> 589,3) sind über der Zeit aufgetragen, wie sie aus der Chromatographie-System zu eluieren.
Diskussion
Der wichtigste Schritt in das Protokoll wie oben beschrieben ist die Gewährleistung der Perlen bleiben gut gemischten während der Inkubationszeit. Erlauben die Perlen auf den Grund des Brunnens / Fläschchen niederlassen wird in erhöhte Variabilität führen. Es ist auch wichtig, Spin-down die Flüssigkeit ab, auf der Spitze des Brunnens / Fläschchen nach der Inkubationszeit bleiben kann. Reproduzierbare Trypsinverdauung ist ebenfalls entscheidend. Das Verfahren für die Verdauung hier beschriebenen wurde ausgiebig in unserem Labor in Verbindung mit SISCAPA eingesetzt, jedoch ist es wahrscheinlich, alternative Methoden der Verdauung für eine gegebene Menge von Target-Proteinen konnte optimiert werden 15 Elution der freien Antikörper nicht mit dem Nachweis der stört. Peptid, wahrscheinlich, weil die Antikörper aus der Säule eluiert oder später als die Peptide ist ausgeschlossen, aber die Antikörper gegen das Protein G Kügelchen vernetzt vor Affinitätsanreicherung in großen Experimente oder wenn freie Antikörper werden zum Problem. Wir haben auch festgestellt, es sinnvoll, einen Magneten unterhalb der Probe Platte auf dem Autosampler sowie das Waschen der Trap-Säule in die umgekehrte Richtung des Flusses (im Vergleich zu Be-) Platz, um restliche Perlen oder Partikel zu entfernen. Zusätzlich zu den Magnetic-Bead-Konzept beschrieben, kann die Technik auch, um eine Spalte Format (dh Affinitätschromatographie) angepasst werden. 12, 16
Sobald der Benutzer bequem mit dem Gesamt-Protokoll, ist die Technik auch zugänglich für mehrere Modifikationen, die die gesamte Assay zu verbessern. 11 Zunächst ist es möglich, mehrere Analyten in einem einzigen Test zu analysieren durch die Kombination von Antikörpern in der Anreicherung (dh Multiplexing). Das Massenspektrometer kann die gleichzeitige Analyse einer großen Anzahl von Analyten. Zweitens erhöht das Volumen der ursprünglichen Probe verbessert die Sensitivität des Tests.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von der NCI Clinical Proteomics Technology Assessment Center (CPTAC) zu gewähren (# U24 CA126476) sowie einen Zuschuss von der Entertainment Industry Foundation (EIF) und der EIF Frauen Cancer Research Fund der Breast Cancer Biomarker Discovery Consortium finanziert und großzügige Geschenke von der Keck Foundation, die Kanarischen Stiftung und der Paul G. Allen Family Foundation.
Materialien
| Name | Company | Catalog Number | Comments |
| Bezeichnung des Stoffes | Firma | Katalog-Nummer | |
|---|---|---|---|
| 1000ul Tiefbrunnen 96-Well-Platten | Eppendorf | 951032786 | |
| Oasis HLB 1 cc Kartusche Platte, Sep-pak, 40 mg 96-Well-Platte | Waters | 186003966 | |
| Kingfisher 96-Well-Platte | Thermo Fisher Scientific | 97002540 | |
| Klare 96-Well, weiß Wandplatte | Bio-Rad | HSP9601 | |
| Folienabdeckung für 96-Well-Platte | Excelscientific | 12-169 | |
| Axymat Dichtmatte für 96-Well-Platte | Axygen | 521-01-151 | |
| X Pierce Film | Sigma-Aldrich | Z722502 | |
| Kingfisher Magnetic-Bead-Prozessor mit PCR Magnetkopf | Thermo Fisher Scientific | HSP 9601 |
Tabelle 1: Werkstoffe
| Bezeichnung des Stoffes | Firma | Katalog-Nummer | Kommentare (optional) |
|---|---|---|---|
| Harnstoff | Sigma Aldrich | U6031 | |
| Trizma Basis | Sigma Aldrich | T1503 | |
| Dithiothreitol (DTT) | Durchstechen | 20291 | "No-wiegen Dithiothreitol" |
| Iodacetamid (IAM) | Sigma Aldrich | I1149 | |
| Trypsin Gold- | Promega | V5280 | |
| Protein G magnetische Kügelchen, 2.8um | Invitrogen | 10004D | |
| Phosphat-gepufferte Salzlösung (PBS) | Thermo Fisher Scientific | L5401 | |
| CHAPS | Thermo Fisher Scientific | 28300 | |
| Ameisensäure | EMD | 11670 | |
| Acetonitril | Thermo Fisher Scientific | A998-1 | |
| Essigsäure | Sigma Aldrich | 242853 |
Tabelle 2: Reagenzien
| Lösung | Kommentare (optional) |
|---|---|
| 100 mM Tris pH 8 | |
| 9 M Harnstoff / 30 mM DTT in 100 mM Tris (pH 8) | muss jedes Mal frisch zubereitet werden |
| 500 mM IAM in 100 mM Tris (pH 8) | muss jedes Mal frisch zubereitet werden |
| Trypsin 1mg/ml in 100 mM Tris pH 8 | |
| Trypsin-Inhibitor 1mg/ml in 100 mM Tris pH 8 | |
| Stabile Isotope Standard Mastermix | |
| Anti-Peptid-Antikörper-Mastermix |
Tabelle 3: Lösungen vorbereitet werden
Referenzen
- Hoofnagle, A. N., Wener, M. H. The fundamental flaws of immunoassays and potential solutions using tandem mass spectrometry. J. Immunol. Methods. 347, 3-11 (2009).
- Anderson, N. L. Mass spectrometric quantitation of peptides and proteins using Stable Isotope Standards and Capture by Anti-Peptide Antibodies (SISCAPA). J Proteome Res. 3, 235-244 (2004).
- Chace, D. H., Kalas, T. A. A biochemical perspective on the use of tandem mass spectrometry for newborn screening and clinical testing. Clin Biochem. 38, 296-309 (2005).
- Want, E. J., Cravatt, B. F., Siuzdak, G. The expanding role of mass spectrometry in metabolite profiling and characterization. Chembiochem. 6, 1941-1951 (2005).
- Barr, J. R. Isotope dilution--mass spectrometric quantification of specific proteins: model application with apolipoprotein A-I. Clin Chem. 42, 1676-1682 (1996).
- Gerber, S. A., Rush, J., Stemman, O., Kirschner, M. W., Gygi, S. P. Absolute quantification of proteins and phosphoproteins from cell lysates by tandem MS. Proc Natl Acad Sci U S A. 100, 6940-6945 (2003).
- Kuhn, E. Quantification of C-reactive protein in the serum of patients with rheumatoid arthritis using multiple reaction monitoring mass spectrometry and 13C-labeled peptide standards. Proteomics. 4, 1175-1186 (2004).
- Whiteaker, J. R. Antibody-based enrichment of peptides on magnetic beads for mass-spectrometry-based quantification of serum biomarkers. Anal Biochem. 362, 44-54 (2007).
- Hoofnagle, A. N., Becker, J. O., Wener, M. H., Heinecke, J. W. Quantification of thyroglobulin, a low-abundance serum protein, by immunoaffinity peptide enrichment and tandem mass spectrometry. Clin Chem. 54, 1796-1804 (2008).
- Kuhn, E. Developing Multiplexed Assays for Troponin I and Interleukin-33 in Plasma by Peptide Immunoaffinity Enrichment and Targeted Mass Spectrometry. Clin Chem. 55, 1108-1117 (2009).
- Whiteaker, J. R., Zhao, L., Anderson, L., Paulovich, A. G. An automated and multiplexed method for high throughput peptide immunoaffinity enrichment and multiple reaction monitoring mass spectrometry-based quantification of protein biomarkers. Mol. Cell. Proteomics. 9, 184-196 (2010).
- Neubert, H., Gale, J., Muirhead, D. Online high-flow peptide immunoaffinity enrichment and nanoflow LC-MS/MS: assay development for total salivary pepsin/pepsinogen. Clin. Chem. 56, 1413-1423 (2010).
- Ackermann, B. L., Berna, M. J. Coupling immunoaffinity techniques with MS for quantitative analysis of low-abundance protein biomarkers. Expert Rev. Proteomics. 4, 175-186 (2007).
- MacLean, B. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Proc, J. L. A quantitative study of the effects of chaotropic agents, surfactants, and solvents on the digestion efficiency of human plasma proteins by trypsin. J. Proteome Res. 9, 5422-5437 (2010).
- Berna, M. Online immunoaffinity liquid chromatography/tandem mass spectrometry determination of a type II collagen peptide biomarker in rat urine: Investigation of the impact of collision-induced dissociation fluctuation on peptide quantitation. Anal. Biochem. 356, 235-243 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten