Method Article
La quantification des protéines à l'aide d'enrichissement d'immunoaffinité peptide couplé à la spectrométrie de masse
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Normes des isotopes stables et capture par anticorps anti-peptide (SISCAPA) l'enrichissement affinités couples de peptides avec des isotopes stables de dilution spectrométrie de masse (MRM-MS) pour fournir une mesure quantitative des peptides comme substituts pour leurs protéines respectives. Nous décrivons ici le protocole en utilisant des particules magnétiques dans un format partiellement automatisés.
Résumé
Il ya un grand besoin pour des analyses quantitatives pour mesurer les protéines. Immunoessais traditionnels sandwichs, largement considéré comme l'étalon-or dans la quantification, sont associées à un coût élevé, de longs délais, et sont pleines de défauts (par exemple, les anticorps hétérophiles, l'interférence des autoanticorps, «effet crochet»). 1 Une technique alternative est l'enrichissement affinités de peptides couplée à la spectrométrie de masse quantitative, communément appelé SISCAPA (Normes des isotopes stables et capture par anticorps anti-peptide). 2 Dans cette technique, l'enrichissement d'affinité de peptides avec une dilution d'isotopes stables et la détection par sélectionnée / multiples réactions de surveillance spectrométrie de masse ( MRS / MRM-MS) fournit une mesure quantitative de peptides en tant que substituts pour leurs protéines respectives. MRS / MRM-MS est bien établie pour la quantification précise de petites molécules 3, 4 et, plus récemment, a été adapté pour mesurer les concentrations de protéines dans le plasma et les lysats cellulaires. 5-7 Pour réaliser la quantification des protéines, ces molécules plus grosses sont digérés peptides composant en utilisant une enzyme comme la trypsine. Un ou plusieurs peptides sélectionnés dont la séquence est unique à la protéine cible de cette espèce (ie "protéotypiques" peptides) sont ensuite enrichies de l'échantillon en utilisant des anticorps anti-peptide et mesurés comme substituts stoechiométrique quantitatifs pour la concentration de protéines dans l'échantillon. Ainsi, couplée à une dilution des isotopes stables (SID) méthodes (c'est à dire un dopés en isotopes stables peptide marqué standard), le SRM / MRM peuvent être utilisés pour mesurer les concentrations de peptides protéotypiques comme substituts pour la quantification des protéines dans des matrices biologiques complexes. Les tests ont plusieurs avantages par rapport aux immunoessais traditionnels. Les réactifs sont relativement moins chers à produire, la spécificité de l'analyte est excellent, les dosages peuvent être fortement multiplexé, l'enrichissement peut être réalisé à partir du plasma soigné (pas de déplétion requis), et la technique se prête à un large éventail de protéines ou des modifications d'intérêt. 8-13 Dans cette vidéo, nous démontrons que le protocole de base adaptée à une plate-forme bille magnétique.
Protocole
Procédure expérimentale:
Le dosage nécessite peptides synthétiques et anticorps anti-peptide. Peptides sélectionnés doivent être uniques à la protéine d'intérêt, contenir entre 8 et 22 acides aminés, et n'ont pas connu modifications post-traductionnelles. Résidus méthionine sont généralement évitées et les peptides contenant des acides aminés dibasiques (par exemple, KK, KR, RR) sont indésirables. Pour cette technique, il est courant d'utiliser des peptides isotopes stables étiquetés comme des normes internes, incorporant lourds (13 C et 15 N) des acides aminés marqués au C-terminale du peptide (ie K ou R étiqueté).
Le protocole suivant décrit un test conçu pour mesurer l'GDSLAYGLR peptide de la protéine de souris ostéopontine, en utilisant des anticorps anti-peptide obtenu à partir de Epitomics Inc (Burlingame, CA) et des peptides synthétiques de la Nouvelle Angleterre Peptide (Gardner, MA). Le protocole comprend trois étapes principales (figure 1): 1) digestion par la trypsine du mélange de protéines complexes, 2) l'enrichissement des peptides 3) Analyse par spectrométrie de masse. Il sera démontré sur un échantillon de plasma humain dopés avec la protéine ostéopontine souris.
1. Trypsine digestion enzymatique et le nettoyage
- Dégeler 10 uL de plasma aliquote pur sur glace mouillée.
- Déterminer la concentration en protéines totales par un dosage BCA et centrifuger l'échantillon pour enlever toute matière en suspension.
- Pipeter 10 uL aliquote de son tube de stockage à un mille Deepwell plaque uL et couvrir avec Pierce-mesure film.
- Ajouter 20 ul de l'urée 9M frais / 30mm dithiothréitol (DTT) (dernière concentration en urée 6M / 20mm TNT) à chaque échantillon.
- Incuber pendant 30 minutes à 37 ° C.
- Ajouter 3 pi de produits frais 500 iodoacétamide mM (finale IAM 50mm) à chaque échantillon.
- Incuber pendant 30 minutes dans le noir à température ambiante.
- Ajouter 257 ul de Tris 100 mM (pH 8) (urée dilue à ~ 0,6 M).
- Ajouter 10 uL de solution mère de trypsine (1 ug / uL, car l'enzyme 01h50: ratio de substrat).
- Incuber à 37 ° C pendant une nuit (12-16 heures).
- Ajouter 3 ul d'acide formique pur (concentration finale de 1%).
- Ajouter la norme des isotopes stables (normes multiples sont ajoutés si vous effectuez un test multiplex, ce n'est généralement environ 10 uL contenant 50-100 fmol de peptide isotopiquement marqués standard).
- Laver la plaque cartouche Oasis bien avec 500 ul d'acide formique 0,1% en 80% d'acétonitrile, en rejetant les actions accréditives. Répétez cela 3 fois.
- Equilibrer la plaque de la cartouche en ajoutant 500 ul d'acide formique 0,1% dans l'eau, et jetez le accréditives. Répétez ce 4 fois.
- Charger les échantillons digérer à la plaque de cartouches et d'ajuster le vide de sorte que le débit est très lent.
- Laver avec 500 ul d'acide formique 0,1% dans l'eau, et jetez accréditives. Répétez cela 3 fois.
- Peptides Eluer en ajoutant 2 x 500 ul d'acide formique 0,1% en 80% d'acétonitrile en 1000 ul puits profond plaque (ne jetez pas l'écoulement continu).
- Lyophiliser (ou speedvac) l'éluat à sec. (La lyophilisation est la méthode préférée)
- Reconstituer séchées peptides en ajoutant 50 pl de PBS + 0,03% de CHAPS.
2. L'enrichissement d'immunoaffinité Peptide
- Transférer l'échantillon à la norme Kingfisher plaques de 96 puits.
- Ajouter 1 antibody ug et 1,5 uL protéine G billes magnétiques recouvertes par cible (il est facultatif pour réticuler l'anticorps sur les billes avant l'addition). Assurez perles sont bien suspendues en secouant ou en vortex.
- Recouvrir la plaque avec du papier.
- Incuber une nuit (12-16 heures) avec douceur tumbling pour assurer perles sont suspendus.
- Centrifuger la plaque à 32 g pendant 5 secondes pour enlever tout liquide de la surface de la feuille.
- Retirez le couvercle en aluminium.
- Lavage et d'élution des perles peut être effectuée manuellement ou de façon automatisée. Cette procédure décrit les étapes automatisées sur une plate-forme Kingfisher.
- Laver les billes deux fois avec 200 ul de PBS + 0,03% de CHAPS (1 minute par lavage).
- Laver les billes une fois avec 200 dilution 1:10 de PBS + 0,03% de CHAPS (1 minute).
- Les peptides sont élués dans 25 ul d'acide acétique à 5% + 0,03% de CHAPS.
- Placer la plaque d'élution sur un aimant et le transfert de l'éluat à une plaque de 96 puits, en prenant soin de ne pas transférer les restes de perles.
- Recouvrir la plaque avec un tapis d'étanchéité.
- La plaque contenant l'éluat est livré à l'spectromètre de masse triple quadrapole pour analyse.
3. L'analyse par suivi de réactions multiples - spectrométrie de masse
- Transitions pour les MRS / MRM analyse peuvent être sélectionnés et optimisés en infusant une picomole 1 par microlitre de solution de peptide standard dans 30% d'acide formique acetonitrile/0.1% dans le spectromètre de masse à 0,5 ul / min. Une fois de pulvérisation stable est obtenu, de recueillir un spectre MS / MS.
- Les transitions sont sélectionnés à partir du spectre MS / MS en identifiant three ions fragments abondants dans une région du spectre avec peu de bruit. Pour multiplier les-ions chargés des peptides, des fragments Y-ion détecté à m / z> précurseur de m / z sont généralement le meilleur pour le développement de tests SRM / GRM. La figure 2 montre le spectre MS / MS et des ions de transition sélectionné 476,3> 508,3, 579,3 , 692.4 (transitions pour les standards isotopes stables lourds 481,3> 518,3, 589,3, 702,4 non représenté) pour l'GDSLAYGLR peptide.
- Selon la marque et le modèle du spectromètre de masse utilisé, il ya plusieurs paramètres qui peuvent être optimisés pour chaque transition. Ici, nous avons optimisé l'énergie de collision de chaque transition choisie par ramping l'énergie de collision et la surveillance du niveau de signal. Une énergie de collision de 25 a été utilisé pour chaque transition.
- En bref, une configuration typique pour SRM / MRM analyse est la suivante: la phase mobile (A) 0,1% d'acide formique; (B) acétonitrile 90% / 0,1% d'acide formique, 0,3 x 5 mm de colonne piège C18, 75 ID um x 15 cm C18 analytique (Reprosil-Pur C18 AQ, 120 A pores °). Le volume d'injection est de 10 uL et l'échantillon est chargé pendant 5 min à B 3% à un débit total de 3 pi / min et éluée par un gradient linéaire de 3 à 45% B à 300 - 400 Nl / min à 10 min. Conditions sur un QTRAP 4000 (ABSCIEX, Foster City, CA) ont été pulvérisée de tension 2.3kV, température de la source d'ions à 150 ° C, GS1 de 12 ans, et 15 de gaz rideau.
- L'échantillon est analysé par MRS / MRM en injectant 10 uL de l'échantillon. Domaines de pointe sont intégrées pour les peptides légers et lourds utilisant le Skyline programme. 14 Calculer le ratio de l'aire du pic (PAR) de peptide non étiquetés / étiquetés dans chaque échantillon en utilisant la transition la plus abondante qui est exempt de bruit de fond, dans ce cas, la transition y5 (peptide légers 476,3> 579,3, 481,3 lourde standard de peptides> 589.3).
4. Les résultats représentatifs:
Mesuré ratios surface du pic (lumière relative aux pointes peptide endogène peptide isotopiquement marqués lourds) fournir une mesure quantitative du peptide cible. La figure 3 montre un chromatogramme par exemple des peptides légers et lourds dans un échantillon SISCAPA enrichi. Notez que les peptides légers et lourds éluer au même moment et de multiples transitions peuvent être surveillés pour chaque peptide pour confirmer l'identité.
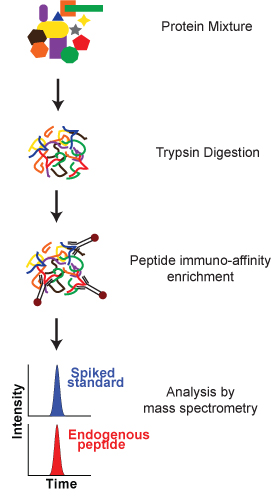
Figure 1. Un aperçu schématique du processus de SISCAPA. Un mélange complexe de protéines sont digérées en peptides. Analytes peptide ciblé (analyte endogène et une pointes marquée par un isotope stable étalon interne) sont enrichis en utilisant des anticorps anti-peptide immobilisé sur la protéine G-particules magnétiques revêtues. Suite à l'isolation, les peptides sont élués ciblés par les particules magnétiques et analysés par spectrométrie de masse pour la quantification par rapport à l'étalon interne.
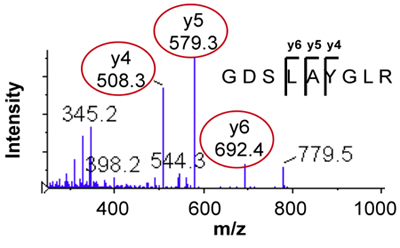
Figure 2. Spectre MS / MS de l'GDSLAYGLR peptide montrant la sélection de trois fragments de MRS / MRM transitions.
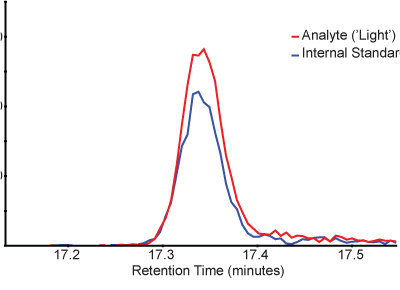
Figure 3. Chromatogrammes Exemple montrant le profil de crête d'un analyte peptide lumière (rouge) et la lourde isotopes stables étiquetés standard interne (en bleu). Chromatogrammes de la transition à partir du peptide y5 léger (476,3> 579,3) et lourd des isotopes stables étiquetés standard (481,3> 589,3) sont tracées au fil du temps comme ils éluer à partir du système de chromatographie.
Discussion
L'étape la plus critique dans le protocole tel que décrit est d'assurer les perles restent bien mélangé pendant la période d'incubation. Permettre aux billes de se déposer au fond du puits / flacon se traduira par une variabilité accrue. Il est également important de spin-down tout liquide qui peut rester sur le haut du puits / flacon après la période d'incubation. Digestion par la trypsine reproductible est également critique. La procédure pour la digestion décrit ici a été largement utilisée dans notre laboratoire en collaboration avec SISCAPA; cependant, il est susceptible de méthodes alternatives de la digestion pourrait être optimisée pour un ensemble donné de protéines cibles 15 élution de l'anticorps libre ne pas interférer avec la détection de l'. peptide, probablement parce que l'anticorps est exclu de la colonne ou élue au plus tard les peptides, mais les anticorps peuvent être réticulés aux billes protéines G avant l'enrichissement d'affinité dans des expériences importantes ou si anticorps libre devient un problème. Nous avons également jugé utile de placer un aimant en dessous de la plaque d'échantillon sur le passeur d'échantillons ainsi que le lavage de la colonne piège dans le sens inverse du flux (par rapport au chargement) pour éliminer toutes les billes ou des particules résiduelles. En plus de l'approche décrite par billes magnétiques, la technique peut également être adapté à un format de colonne (ie la chromatographie d'affinité). 12, 16
Une fois que l'utilisateur est à l'aise avec le protocole général, la technique est également prête à plusieurs modifications qui améliorent le dosage global. 11 Premièrement, il est possible d'analyser des analytes multiples dans un seul test en combinant les anticorps dans l'étape d'enrichissement (multiplexage par exemple). Le spectromètre de masse est capable de analysant simultanément un grand nombre d'analytes. Deuxièmement, l'augmentation du volume de l'échantillon original améliore la sensibilité du dosage.
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été financé par le NCI clinique protéomique Technology Assessment Center (CPTAC) subvention (# U24 CA126476) ainsi que d'une subvention de l'industrie du divertissement de la Fondation (FEI) et le Fonds des femmes FEI recherche sur le cancer au Consortium du cancer du sein découverte de biomarqueurs, et dons généreux de la Fondation Keck, la Fondation des Canaries, et le Paul G. Allen Family Foundation.
matériels
| Name | Company | Catalog Number | Comments |
| Nom de la matière | Société | Numéro de catalogue | |
|---|---|---|---|
| 1000ul assiettes creuses et de 96 puits | Eppendorf | 951032786 | |
| Oasis HLB 1 cc cartouches plaque, Sep-pak, 40 mg plaque de 96 puits | Eaux | 186003966 | |
| Kingfisher plaque de 96 puits | Thermo Fisher Scientific | 97002540 | |
| Effacer 96 puits, la plaque murale blanche | Bio-Rad | HSP9601 | |
| Fleuret couverture pour 96 puits | Excelscientific | 12-169 | |
| Tapis d'étanchéité Axymat pour 96 puits | Axygen | 521-01-151 | |
| X Pierce cinéma | Sigma-Aldrich | Z722502 | |
| Kingfisher processeur bille magnétique avec tête magnétique PCR | Thermo Fisher Scientific | HSP 9601 |
Tableau 1: Matériaux
| Nom de la matière | Société | Numéro de catalogue | Commentaires (optionnel) |
|---|---|---|---|
| L'urée | Sigma-Aldrich | U6031 | |
| Trizma de base | Sigma-Aldrich | T1503 | |
| Dithiothréitol (DTT) | Pierce | 20291 | «Non-pèsent dithiothréitol" |
| Iodoacétamide (IAM) | Sigma-Aldrich | I1149 | |
| Or la trypsine | Promega | V5280 | |
| Protéines G perles magnétiques, 2.8um | Invitrogen | 10004D | |
| Tampon phosphate salin (PBS) | Thermo Fisher Scientific | L5401 | |
| CHAPS | Thermo Fisher Scientific | 28300 | |
| L'acide formique | EMD | 11670 | |
| Acétonitrile | Thermo Fisher Scientific | A998-1 | |
| Acide acétique | Sigma-Aldrich | 242853 |
Tableau 2: Réactifs
| Solution | Commentaires (optionnel) |
|---|---|
| Tris 100 mM pH 8 | |
| 9 M d'urée / 30 mM de DTT dans 100 mM de Tris (pH 8) | doivent être préparés frais chaque fois que |
| 500 mM IAM dans 100 mM de Tris (pH 8) | doivent être préparés frais chaque fois que |
| Trypsine 1mg/mL dans 100 mM Tris pH 8 | |
| Inhibiteur de la trypsine 1mg/mL dans 100 mM Tris pH 8 | |
| Isotopes stables standards mastermix | |
| Mastermix anticorps anti-peptide |
Tableau 3: Des solutions pour se préparer
Références
- Hoofnagle, A. N., Wener, M. H. The fundamental flaws of immunoassays and potential solutions using tandem mass spectrometry. J. Immunol. Methods. 347, 3-11 (2009).
- Anderson, N. L. Mass spectrometric quantitation of peptides and proteins using Stable Isotope Standards and Capture by Anti-Peptide Antibodies (SISCAPA). J Proteome Res. 3, 235-244 (2004).
- Chace, D. H., Kalas, T. A. A biochemical perspective on the use of tandem mass spectrometry for newborn screening and clinical testing. Clin Biochem. 38, 296-309 (2005).
- Want, E. J., Cravatt, B. F., Siuzdak, G. The expanding role of mass spectrometry in metabolite profiling and characterization. Chembiochem. 6, 1941-1951 (2005).
- Barr, J. R. Isotope dilution--mass spectrometric quantification of specific proteins: model application with apolipoprotein A-I. Clin Chem. 42, 1676-1682 (1996).
- Gerber, S. A., Rush, J., Stemman, O., Kirschner, M. W., Gygi, S. P. Absolute quantification of proteins and phosphoproteins from cell lysates by tandem MS. Proc Natl Acad Sci U S A. 100, 6940-6945 (2003).
- Kuhn, E. Quantification of C-reactive protein in the serum of patients with rheumatoid arthritis using multiple reaction monitoring mass spectrometry and 13C-labeled peptide standards. Proteomics. 4, 1175-1186 (2004).
- Whiteaker, J. R. Antibody-based enrichment of peptides on magnetic beads for mass-spectrometry-based quantification of serum biomarkers. Anal Biochem. 362, 44-54 (2007).
- Hoofnagle, A. N., Becker, J. O., Wener, M. H., Heinecke, J. W. Quantification of thyroglobulin, a low-abundance serum protein, by immunoaffinity peptide enrichment and tandem mass spectrometry. Clin Chem. 54, 1796-1804 (2008).
- Kuhn, E. Developing Multiplexed Assays for Troponin I and Interleukin-33 in Plasma by Peptide Immunoaffinity Enrichment and Targeted Mass Spectrometry. Clin Chem. 55, 1108-1117 (2009).
- Whiteaker, J. R., Zhao, L., Anderson, L., Paulovich, A. G. An automated and multiplexed method for high throughput peptide immunoaffinity enrichment and multiple reaction monitoring mass spectrometry-based quantification of protein biomarkers. Mol. Cell. Proteomics. 9, 184-196 (2010).
- Neubert, H., Gale, J., Muirhead, D. Online high-flow peptide immunoaffinity enrichment and nanoflow LC-MS/MS: assay development for total salivary pepsin/pepsinogen. Clin. Chem. 56, 1413-1423 (2010).
- Ackermann, B. L., Berna, M. J. Coupling immunoaffinity techniques with MS for quantitative analysis of low-abundance protein biomarkers. Expert Rev. Proteomics. 4, 175-186 (2007).
- MacLean, B. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Proc, J. L. A quantitative study of the effects of chaotropic agents, surfactants, and solvents on the digestion efficiency of human plasma proteins by trypsin. J. Proteome Res. 9, 5422-5437 (2010).
- Berna, M. Online immunoaffinity liquid chromatography/tandem mass spectrometry determination of a type II collagen peptide biomarker in rat urine: Investigation of the impact of collision-induced dissociation fluctuation on peptide quantitation. Anal. Biochem. 356, 235-243 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon