Method Article
La cuantificación de proteínas mediante el enriquecimiento de inmunoafinidad péptido acoplado a espectrometría de masas
* Estos autores han contribuido por igual
En este artículo
Resumen
Normas de isótopos estables y de captura de anticuerpos anti-péptido (SISCAPA) enriquecimiento de las parejas de afinidad de los péptidos con una dilución de isótopos estables de espectrometría de masas (MRM-MS) para proporcionar una medición cuantitativa de los péptidos como sustitutos de sus respectivas proteínas. A continuación se describe el protocolo de uso de partículas magnéticas en un formato parcialmente automatizado.
Resumen
Hay una gran necesidad de ensayos cuantitativos en la medición de las proteínas. Inmunoensayos tradicional bocadillo, en gran medida considera el estándar de oro en la cuantificación, se asocian con un alto costo, larga duración, y están llenos de inconvenientes (por ejemplo, anticuerpos heterófilos, la interferencia de autoanticuerpos, 'gancho-efecto "). 1 Una técnica alternativa es el enriquecimiento de afinidad de péptidos acoplada a espectrometría de masas cuantitativa, comúnmente conocida como SISCAPA (Normas de isótopos estables y captura de anticuerpos anti-péptido). 2 En esta técnica, el enriquecimiento de afinidad de los péptidos con una dilución de isótopos estables y de detección por determinados / espectrometría de masa de reacción múltiple de control ( SRM / MRM-MS) proporciona una medición cuantitativa de los péptidos como sustitutos de sus respectivas proteínas. SRM / MRM-MS está bien establecida para la cuantificación exacta de las moléculas pequeñas 3, 4 y más recientemente ha sido adaptado para medir las concentraciones de proteínas en el plasma y lisados celulares. 5.7 Para lograr la cuantificación de las proteínas, las moléculas de mayor tamaño son digeridos a péptidos componente usando una enzima como la tripsina. Uno o más péptidos seleccionados cuya secuencia es única a la proteína diana en la especie (es decir, "proteotypic" péptidos) son continuación de la muestra enriquecida con anticuerpos anti-péptido y se mide como sustitutos estequiométrica cuantitativa de la concentración de proteínas en la muestra. Por lo tanto, junto a la dilución de isótopos estables (SID) métodos (es decir, una enriquecida en isótopos estables etiquetados péptido estándar), SRM / MRM se puede utilizar para medir las concentraciones de péptidos proteotypic como sustitutos para la cuantificación de proteínas en matrices biológicas complejas. Los ensayos tienen varias ventajas en comparación con los inmunoensayos tradicionales. Los reactivos son relativamente menos caros para generar, la especificidad para el analito es excelente, los ensayos pueden ser altamente multiplexado, el enriquecimiento se puede realizar a partir de plasma puro (no requiere el agotamiento), y la técnica se presta a una amplia variedad de proteínas o modificaciones de interés. 8-13 En este video se demuestra el protocolo básico adaptado a una plataforma de cuentas magnéticas.
Protocolo
Procedimiento experimental:
El ensayo requiere péptidos sintéticos y anticuerpos anti-péptido. Péptidos seleccionados deben ser exclusivos de la proteína de interés, contienen entre 8 y 22 aminoácidos, y no se conocen modificaciones post-traduccionales. Los residuos de metionina generalmente se evitan y los péptidos que contienen aminoácidos dibásicos (por ejemplo, KK, KR, RR) no son deseables. Para esta técnica, es común el uso de péptidos de isótopos estables etiquetados como estándares internos, la incorporación de pesados (13 C y N 15) aminoácidos marcados en el C-terminal del péptido (es decir, K o R etiqueta).
El siguiente protocolo describe un ensayo para medir el GDSLAYGLR péptido de la osteopontina proteína de ratón, utilizando anticuerpos anti-péptido obtenido de Epitomics Inc. (Burlingame, CA) y péptidos sintéticos de Nueva Inglaterra Péptidos (Gardner, MA). El protocolo consta de tres pasos principales (Figura 1): 1) la digestión de tripsina de la mezcla compleja de proteínas, 2) Enriquecimiento de péptidos 3) El análisis por espectrometría de masas. Se demostrará en una muestra de plasma humano enriquece con la proteína osteopontina ratón.
1. Tripsina digestión enzimática y la limpieza
- Descongele 10 l alícuota de plasma limpia en hielo.
- Determinar la concentración de proteínas totales mediante el ensayo de BCA y centrifugar la muestra para eliminar los sólidos en suspensión.
- Pipetear 10 l alícuota de su tubo de almacenamiento a una placa de 1000 l pozos profundos y cubrir con Pierce-capaz de película.
- Añadir 20 l de 9 millones de nuevos urea / 30 mM ditiotreitol (DTT) (6M final de la concentración de urea / 20 mM DTT) a cada muestra.
- Incubar durante 30 minutos a 37 ° C.
- Añadir 3 l de 500 mM yodoacetamida fresca (final IAM 50 mm) a cada muestra.
- Incubar durante 30 minutos en la oscuridad a temperatura ambiente.
- Añadir 257 l de 100 mM Tris (pH 8) (urea diluye a ~ 0,6 M).
- Añadir 10 l de solución madre de tripsina (1 mg / l; de 1:50 enzima: sustrato de relación).
- Incubar 37 ° C durante la noche (12-16 horas).
- Añadir 3 l de ácido fórmico puro (concentración final de 1%).
- Añadir estándar de isótopos estables (normas se agregan varias si se realiza un ensayo multiplex, por lo general se trata de 10 l que contiene 50-100 fmol de péptido estándar marcados con isótopos).
- Lavar la placa cartucho de Oasis y con 500 l de 0,1% de ácido fórmico en acetonitrilo 80%, descartando el flujo continuo. Repita esto 3 veces.
- Equilibre la placa del cartucho mediante la adición de 500 l de 0,1% de ácido fórmico en agua, y descartar el filtrado. Repita este 4 veces.
- Cargar las muestras de digerir a la placa del cartucho y ajustar el vacío para que el flujo es muy lento.
- Lavar con 500 l de 0,1% de ácido fórmico en agua, y descartar el filtrado. Repita esto 3 veces.
- Péptidos eluir mediante la adición de 2 x 500 l de 0,1% de ácido fórmico en acetonitrilo al 80% en 1000 l profunda y placa (no se descarta el flujo a través).
- Liofilizar (o speedvac) el eluyente hasta desecación. (La liofilización es el método preferido)
- Reconstituir seca péptidos mediante la adición de 50 l de PBS + 0,03% CHAPS.
2. Péptido de enriquecimiento de inmunoafinidad
- Transferir la muestra a nivel Kingfisher placas de 96 pocillos.
- Añadir un anticuerpo mg y 1,5 l proteína G-magnéticas recubiertas por objetivo (que es opcional para entrecruzar los anticuerpos a las bolas antes de la adición). Asegúrese de cuentas son también suspendido por temblor o agitación.
- Cubrir la placa con papel de aluminio.
- Incubar toda la noche (16.12 h) con suave volteretas para asegurarse de cuentas son suspendidas.
- Centrifugar la placa a 32 xg durante 5 segundos para eliminar el líquido de la superficie de la lámina.
- Quite la cubierta de aluminio.
- Lavado y la elución de las cuentas se puede realizar manualmente o de forma automatizada. Este procedimiento describe los pasos automatizados en una plataforma de Kingfisher.
- Lavar las perlas de dos veces con 200 l de PBS + 0,03% CHAPS (1 minuto por lavado).
- Lavar las perlas de una vez con 200 l de dilución de 1:10 de PBS + 0,03% CHAPS (1 minuto).
- Los péptidos se eluyeron en 25 l de ácido acético al 5% + 0,03% CHAPS.
- Coloque la placa de elución de un imán y la transferencia que el efluente de una placa y 96, teniendo cuidado de no transferencia de las cuentas de sobra.
- Cubrir la placa con una estera de sellado.
- La placa que contiene el efluente se entrega el espectrómetro de masas de triple cuadrupolo para el análisis.
3. Análisis mediante el control de reacción múltiple - espectrometría de masas
- Transiciones de SRM / MRM análisis pueden ser seleccionados y optimizados mediante la infusión de una solución picomole 1 por microlitro de péptido estándar en acetonitrile/0.1% al 30% de ácido fórmico en el espectrómetro de masas de 0,5 l / min. Una vez rociado estable se obtiene, recoge un espectro MS / MS.
- Las transiciones son seleccionados de todo el espectro MS / MS mediante la identificación de three iones abundantes fragmentos en una región del espectro, con poco ruido. Para multiplicar cargado de iones de péptidos, los fragmentos y de iones detectados en m / z> precursor m / z son generalmente las mejores para el desarrollo de ensayos de SRM / MRM. La figura 2 muestra el espectro MS / MS y otros iones de transición 476,3> 508,3, 579,3 , 692.4 (transiciones para el pesado de isótopos estables estándar 481.3 no> 518,3, 589,3, 702,4 se muestra) para la GDSLAYGLR péptido.
- Dependiendo de la marca y modelo del espectrómetro de masa utilizado, hay varios parámetros que pueden ser optimizados para cada transición. Aquí, hemos optimizado la energía de colisión de cada transición seleccionada por la rampa de la energía de la colisión y el control del nivel de señal. Una energía de colisión de 25 se utilizó para cada transición.
- En pocas palabras, una configuración típica de SRM / MRM análisis es el siguiente: Fase móvil (A) de ácido fórmico al 0,1%, (B) acetonitrilo 90% / 0,1% de ácido fórmico, 0,3 x 5 mm de columna trampa de C18, 75 ID m x 15 cm C18 analítica (Reprosil-Pur C18 AQ, 120 A poro °). El volumen de inyección es de 10 l, y la muestra se carga durante 5 minutos a 3% B con un caudal total de 3 l / min y se eluyó con un gradiente lineal de 3-45% B en 300 a 400 nL / min en 10 min. Condiciones en un QTRAP 4000 (ABSCIEX, Foster City, CA) fueron spray 2.3kV tensión, temperatura de la fuente de iones de 150 ° C, GS1 de 12 años, y el gas cortina 15.
- La muestra es analizada por SRM / MRM mediante la inyección de 10 l de la muestra. Áreas de los picos están integrados por péptidos ligeros y pesados, utilizando el programa de Skyline. 14 Calcular la relación del área de pico (PAR) del péptido no marcado / etiquetado de cada muestra con la transición más abundante que está libre de ruido de fondo, en este caso, la transición y5 (péptido luz 476,3> 579,3, pesado estándar péptido 481,3> 589,3).
4. Los resultados representativos:
Medida ratios área del pico (en relación con la luz péptido endógeno de pinchos de péptidos marcados con isótopos pesados) proporcionan una medida cuantitativa del péptido objetivo. La figura 3 muestra un cromatograma de ejemplo de péptidos ligeros y pesados en una muestra SISCAPA enriquecido. Tenga en cuenta que los péptidos ligeros y pesados eluyen al mismo tiempo y múltiples transiciones pueden ser monitoreados para cada péptido para confirmar la identidad.
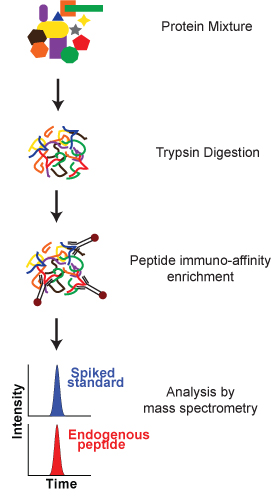
Figura 1. Un esquema resumen del proceso de SISCAPA. Una mezcla compleja de proteínas se digieren en péptidos. Objetivo analitos péptido (analito endógeno y pinchos de isótopos estables marcado patrón interno) se enriquecen con anticuerpos anti-péptido inmovilizado en la proteína G-revestido partículas magnéticas. Tras el aislamiento, los péptidos específicos son eluidos de las partículas magnéticas y analizados por espectrometría de masas para la cuantificación en relación con el patrón interno.
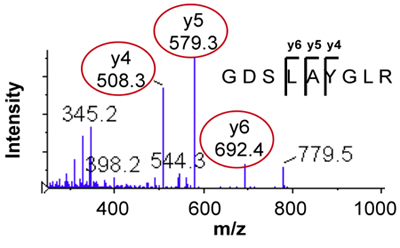
Figura 2. MS / MS espectro de la GDSLAYGLR péptido que muestra la selección de tres fragmentos de SRM / MRM transiciones.
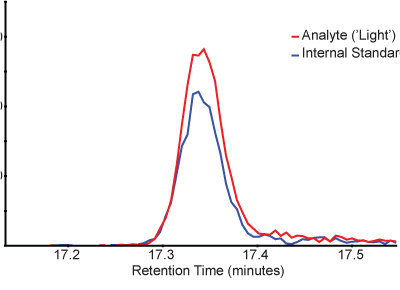
Figura 3. Cromatogramas ejemplo que muestra el perfil de un pico de analito péptido (rojo) y el pesado de isótopos estables marcado patrón interno (azul). Cromatogramas de la transición y5 del péptido de luz (476,3> 579,3) y el isótopo pesado estable etiqueta estándar (481,3> 589,3) están representados a través del tiempo, ya que eluyen de la sistema de cromatografía.
Discusión
El paso más crítico en el protocolo como se describe es asegurar las cuentas siguen siendo bien mezclada durante el período de incubación. Permitiendo que los gránulos se depositan en el fondo del pozo / vial se traducirá en una mayor variabilidad. También es importante para hacer girar hacia abajo el líquido que puede permanecer en la parte superior del pozo / vial tras el período de incubación. Digestión de la tripsina reproducibles también es fundamental. El procedimiento para la digestión descrito aquí se ha utilizado ampliamente en nuestro laboratorio en colaboración con SISCAPA, sin embargo, es probable que los métodos alternos de la digestión puede ser optimizado para un determinado conjunto de proteínas de la meta 15 de elución de los anticuerpos libres no interferir con la detección de la. péptido, probablemente debido a que el anticuerpo se excluye de la columna o eluye más tarde de los péptidos, pero el anticuerpo puede ser reticulado a las perlas de proteína G antes de enriquecimiento de afinidad en los experimentos de gran tamaño o si anticuerpo libre se convierte en un problema. También hemos encontrado que es útil para colocar un imán debajo de la placa de muestra en el muestreador automático, así como el lavado de la columna de la trampa en la dirección opuesta del flujo (en comparación con la carga) para eliminar cualquier residuo granos o partículas. Además de las bolas magnéticas enfoque descrito, la técnica también puede ser adaptado a un formato de columna (es decir, la cromatografía de afinidad). 12, 16
Una vez que el usuario se sienta cómodo con el protocolo general, la técnica también es susceptible de varias modificaciones que mejoran el ensayo general. 11 En primer lugar, es posible analizar múltiples analitos en un único ensayo por la combinación de anticuerpos en la etapa de enriquecimiento (multiplexación por ejemplo). El espectrómetro de masas es capaz de analizar simultáneamente un gran número de analitos. En segundo lugar, aumentar el volumen de la muestra original mejora la sensibilidad de la prueba.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue financiado por el Instituto Nacional del Cáncer Clínica Proteómica del Centro de Tecnología de Evaluación (CPTAC) beca (# U24 CA126476), así como una beca de la Industria del Entretenimiento Foundation (EIF) y el Fondo de las Mujeres del FEI de Investigación del Cáncer en el Consorcio el cáncer de mama el descubrimiento de biomarcadores y las generosas donaciones de la Fundación Keck, de la Fundación Canarias, y la Paul G. Allen Family Foundation.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del material | Empresa | Número de catálogo | |
|---|---|---|---|
| 1000ul placas de pozos profundos de 96 pozos | Eppendorf | 951032786 | |
| Oasis HLB 1 cc cartucho de la placa, Sep-pak, 40 mg de placa de 96 pocillos | Aguas | 186003966 | |
| Kingfisher placa de 96 pocillos | Thermo Fisher Scientific | 97002540 | |
| Claro de 96, blanco placa de pared | Bio-Rad | HSP9601 | |
| Papel de la cubierta de placa de 96 pocillos | Excelscientific | 12-169 | |
| Axymat alfombra de sellado de placa de 96 pocillos | Axygen | 521-01-151 | |
| X perforar la película | Sigma-Aldrich | Z722502 | |
| Kingfisher magnética procesador de grano con la cabeza imán PCR | Thermo Fisher Scientific | HSP 9601 |
Tabla 1: Materiales
| Nombre del material | Empresa | Número de catálogo | Comentarios (opcional) |
|---|---|---|---|
| Urea | Sigma Aldrich | U6031 | |
| Trizma base de | Sigma Aldrich | T1503 | |
| Ditiotreitol (DTT) | Atravesar | 20291 | "No pesan ditiotreitol" |
| Yodoacetamida (IAM) | Sigma Aldrich | I1149 | |
| Tripsina Oro | Promega | V5280 | |
| Proteína G magnética cuentas, 2.8um | Invitrogen | 10004D | |
| Tampón fosfato salino (PBS) | Thermo Fisher Scientific | L5401 | |
| CHAPS | Thermo Fisher Scientific | 28300 | |
| Ácido fórmico | EMD | 11670 | |
| Acetonitrilo | Thermo Fisher Scientific | A998-1 | |
| El ácido acético | Sigma Aldrich | 242853 |
Tabla 2: Reactivos
| Solución | Comentarios (opcional) |
|---|---|
| 100 mM Tris pH 8 | |
| 9 M urea / 30 mM de TDT en 100 mM Tris (pH 8) | debe prepararse cada vez |
| 500 mM IAM en 100 mM Tris (pH 8) | debe prepararse cada vez |
| Tripsina 1mg/ml en 100 mM Tris pH 8 | |
| Inhibidor de tripsina de 1mg/ml en 100 mM Tris pH 8 | |
| De isótopos estables estándar mastermix | |
| Anticuerpos anti-péptido mastermix |
Tabla 3: Soluciones de estar preparados
Referencias
- Hoofnagle, A. N., Wener, M. H. The fundamental flaws of immunoassays and potential solutions using tandem mass spectrometry. J. Immunol. Methods. 347, 3-11 (2009).
- Anderson, N. L. Mass spectrometric quantitation of peptides and proteins using Stable Isotope Standards and Capture by Anti-Peptide Antibodies (SISCAPA). J Proteome Res. 3, 235-244 (2004).
- Chace, D. H., Kalas, T. A. A biochemical perspective on the use of tandem mass spectrometry for newborn screening and clinical testing. Clin Biochem. 38, 296-309 (2005).
- Want, E. J., Cravatt, B. F., Siuzdak, G. The expanding role of mass spectrometry in metabolite profiling and characterization. Chembiochem. 6, 1941-1951 (2005).
- Barr, J. R. Isotope dilution--mass spectrometric quantification of specific proteins: model application with apolipoprotein A-I. Clin Chem. 42, 1676-1682 (1996).
- Gerber, S. A., Rush, J., Stemman, O., Kirschner, M. W., Gygi, S. P. Absolute quantification of proteins and phosphoproteins from cell lysates by tandem MS. Proc Natl Acad Sci U S A. 100, 6940-6945 (2003).
- Kuhn, E. Quantification of C-reactive protein in the serum of patients with rheumatoid arthritis using multiple reaction monitoring mass spectrometry and 13C-labeled peptide standards. Proteomics. 4, 1175-1186 (2004).
- Whiteaker, J. R. Antibody-based enrichment of peptides on magnetic beads for mass-spectrometry-based quantification of serum biomarkers. Anal Biochem. 362, 44-54 (2007).
- Hoofnagle, A. N., Becker, J. O., Wener, M. H., Heinecke, J. W. Quantification of thyroglobulin, a low-abundance serum protein, by immunoaffinity peptide enrichment and tandem mass spectrometry. Clin Chem. 54, 1796-1804 (2008).
- Kuhn, E. Developing Multiplexed Assays for Troponin I and Interleukin-33 in Plasma by Peptide Immunoaffinity Enrichment and Targeted Mass Spectrometry. Clin Chem. 55, 1108-1117 (2009).
- Whiteaker, J. R., Zhao, L., Anderson, L., Paulovich, A. G. An automated and multiplexed method for high throughput peptide immunoaffinity enrichment and multiple reaction monitoring mass spectrometry-based quantification of protein biomarkers. Mol. Cell. Proteomics. 9, 184-196 (2010).
- Neubert, H., Gale, J., Muirhead, D. Online high-flow peptide immunoaffinity enrichment and nanoflow LC-MS/MS: assay development for total salivary pepsin/pepsinogen. Clin. Chem. 56, 1413-1423 (2010).
- Ackermann, B. L., Berna, M. J. Coupling immunoaffinity techniques with MS for quantitative analysis of low-abundance protein biomarkers. Expert Rev. Proteomics. 4, 175-186 (2007).
- MacLean, B. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Proc, J. L. A quantitative study of the effects of chaotropic agents, surfactants, and solvents on the digestion efficiency of human plasma proteins by trypsin. J. Proteome Res. 9, 5422-5437 (2010).
- Berna, M. Online immunoaffinity liquid chromatography/tandem mass spectrometry determination of a type II collagen peptide biomarker in rat urine: Investigation of the impact of collision-induced dissociation fluctuation on peptide quantitation. Anal. Biochem. 356, 235-243 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados