Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Reinigung von Hsp104, ein Protein Disaggregase
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll für die Reinigung von hochaktiven Hsp104, ein hexameren AAA +-Protein aus Hefe, die Paare ATP-Hydrolyse, um Protein Disaggregation. Dieses Schema nutzt eine His6-markierten Konstruktes zur Affinitätsreinigung von E. coli Durch Anionenaustausch-Chromatographie, His6-tag-Entfernung mit TEV-Protease und Größenausschlusschromatographie gefolgt.
Zusammenfassung
Hsp104 ist ein hexameren AAA + protein 1 aus Hefe, die Paare ATP-Hydrolyse, um Protein Aufschlüsselung 2-10 (Abb. 1). Diese Aktivität vermittelt zwei wichtige selektive Vorteile. Erstens ermöglicht Renaturierung ungeordnete Aggregate von Hsp104 Hefe Überleben nach verschiedenen Protein-Fehlfaltung betont, einschließlich Hitzeschock 3,5,11,12. Zweitens ermöglicht die Umgestaltung des Cross-Beta-Amyloid-Fibrillen durch Hsp104 Hefe unzähligen Prionen (infektiöse Amyloide) als ein Reservoir von Vorteil zu nutzen und vererbbare phänotypische Variation 13-22. Bemerkenswert ist, dass Hsp104 direkt umbaut preamyloid Oligomere und Amyloidfibrillen, einschließlich derjenigen der Hefe Prion-Proteine Sup35 und Ure2 23-30 zusammen. Das Amyloid-Umbau-Funktionalität ist ein spezialisierter Facette der Hefe Hsp104. Die E. coli Ortholog, ClpB, nicht umgestalten preamyloid Oligomere oder Amyloid-Fibrillen 26,31,32.
Hsp104 Orthologe sind in allen Reichen des Lebens außer, verblüffend, Tiere gefunden. In der Tat, ob tierische Zellen keine enzymatische System besitzen, dass Paare Protein Aufschlüsselung nach Renaturierung (statt Abbau) bleibt unbekannt 33-35. So haben wir und andere vorgeschlagen, dass Hsp104 als Therapeutikum für verschiedene neurodegenerative Erkrankungen, die mit der Fehlfaltung von spezifischen Proteinen in toxische preamyloid Oligomere und Amyloidfibrillen 4,7,23,36-38 verbunden entwickelt werden könnten. Es gibt keine Behandlungen, die direkt auf den aggregierten Spezies mit diesen Erkrankungen assoziiert. Doch löst sich Hsp104 toxischen Oligomere und Amyloidfibrillen von Alpha-Synuclein, die mit Morbus Parkinson 23 sowie Amyloid Formen von PrP 39 verbunden zusammengesetzt sind. Wichtig ist, reduziert Hsp104 Proteinaggregation und verbessert Neurodegeneration in Nagermodellen der Parkinson-Krankheit 23 und Huntington-Krankheit 38. Idealerweise auf die Therapie zu optimieren und die Nebenwirkungen minimieren, wäre Hsp104 entwickelt und werden verstärkt gezielt umzugestalten spezifische Aggregate von zentraler Bedeutung für die betreffende Krankheit 4,7. Doch die begrenzte strukturelle und mechanistische Verständnis davon, wie Hsp104 disaggregiert ein so vielfältiges Repertoire von aggregierten Strukturen und verwandte Proteine frustriert diese Bemühungen 30,40-42.
Um zu verstehen, die Struktur und den Mechanismus der Hsp104, ist es wichtig, die reine Protein-und rekonstruieren ihre disaggregase Aktivität mit minimalen Komponenten zu untersuchen. Hsp104 ist ein 102kDa Protein mit einem pI von ca. 5,3, die in Gegenwart von ADP oder ATP oder bei hohen Proteinkonzentrationen in Abwesenheit von Nukleotid 43-46 hexamerizes. Hier beschreiben wir ein optimiertes Protokoll für die Reinigung von hoch aktiven, stabilen Hsp104 von E. coli. Die Verwendung von E. coli ermöglicht eine vereinfachte Großproduktion und unsere Methode kann schnell und zuverlässig für zahlreiche Hsp104 Varianten durchgeführt werden. Unser Protokoll erhöht Hsp104 Reinheit und vereinfacht His 6-tag Entfernung im Vergleich zu einer früheren Reinigungsverfahren aus E. coli 47. Darüber hinaus ist unser Protokoll mehr einfache und bequemer als zwei neuere Protokolle 26,48.
Protokoll
1. Expression von Hsp104
- Das Plasmid für die Reinigung in E. beschäftigt coli, pPROEX-HTB-Hsp104 enthält die Hsp104 offene Leserahmen unter der induzierbaren Kontrolle des trc-Promotors 26. Das Plasmid produziert Hsp104 mit einem N-terminalen His 6-Tag, das durch TEV-Protease-Spaltung entfernt werden können. Transform pPROEX-HTB-Hsp104 in Codon-optimierten E. coli BL21-CodonPlus-RIL Zellen (Stratagene, Agilent Technologies) mit einer typischen bakteriellen Transformation Verfahren (zB nach den Anweisungen des Herstellers). Es ist wichtig, um eine Codon-optimierten E. Benutzung coli-Stamm, weil Hsp104 hat eine ungewöhnliche Codon Bias.
- Impfen einen 100 2xYT Kultur (USB. Rezept: 16g / L Caseinpepton, 10 g / L Hefeextrakt, 5 g / L NaCl, pH 7,0) mit 100μg/mL Ampicillin und Chloramphenicol 34μg/mL mit frischen Transformanten ergänzt und wachsen Nacht bei 37 ° C unter Schütteln bei 200rpm.
- Impfen 10mL der Übernacht-Kultur in 6 X 1L 2xYT mit 100μg/mL Ampicillin und Chloramphenicol 34μg/mL in 2L Flaschen. Schütteln bei 250rpm bei 37 ° C bis OD 600 = 0,4-0,6. Aufhören zu zittern und Absenken der Temperaturen auf 15 ° C. Lassen Zellen äquilibrieren unaufgeregt für 30min in den Brutkasten, bis es 15 ° C erreicht Sobald 15 ° C erreicht ist induzieren Proteinexpression durch Zugabe von IPTG zu einer Endkonzentration von 1 mM. Fortsetzen Schütteln bei 250rpm über Nacht (12-16 Stunden).
2. Zellernte und Lyse
- Ernten Sie die Zellen durch Zentrifugation (in einem vorgekühlten Rotor) bei 4.000 min für 20 Minuten bei 4 ° C (wir verwenden einen Sorvall RC 3BP + Zentrifuge). Nachfolgende Schritte müssen sofort ausgeführt werden, da Hsp104 Aktivität ist vermindert, wenn E. coli-Zellen werden eingefroren. Darüber hinaus sollten alle nachfolgenden Schritte auf Eis oder bei 4 ° C durchgeführt werden
- Resuspendieren Zellpellets in vorgekühlte (auf Eis) 10 ml Lysepuffer (40 mM HEPES-KOH pH 7,4, 500 mM KCl, 20 mM MgCl 2, 2,5% (w / v) Glycerin, 20 mM Imidazol, 5 &mgr; M Pepstatin A, komplette Protease-Inhibitor-Cocktail (1 EDTA -freie tablet/50mL) und 2mM β-Mercaptoethanol).
- Lyse-Zellen mit einem Französisch drücken (Emulsiflex) Homogenisator, dass ein Wärmetauscher in Eiswasser getaucht verwendet. Vor dem Gebrauch sicherstellen, dass alle Zellklumpen wurden gelöst. Nach Gleichgewichtseinstellung Homogenisator mit Lysepuffer, ist zwei Durchgängen durch den Zylinder mit einem Druck von 15.000-18.000 psi ausreichend für die vollständige Lyse. Wenn ein Französisch Presse ist nicht vorhanden, kann der Inkubation mit Lysozym mit anschließender Ultraschallbehandlung verwendet werden (siehe Diskussion) werden. Speichern Sie eine kleine Auswahl von lysierten Zellen für die SDS-PAGE-Analyse (1μL) (Lysate in Abb. 2)..
- Entfernen Zelltrümmer durch Zentrifugation bei 16.000 rpm für 20 Minuten bei 4 ° C (wir verwenden einen Sorvall RC5C + centifuge). Bewahren Überstand für den nächsten Schritt, und speichern Sie eine kleine Probe (1μL) der Überstand für die SDS-PAGE-Analyse (Ni Laden in Abb. 2)..
3. Hsp104 Purification
- Mix Überstand aus Schritt 2.4 mit 12ml (2 ml Ni-Sepharose Kügelchen pro 1L von Zellen) von 50% Gülle Lysepuffer äquilibrierte Ni-Sepharose-Beads (GE).
- Drehen Probe langsam für 3 Stunden bei 4 ° C, so dass Ni-Sepharose gleichmäßig suspendiert und Schaumbildung auf ein Minimum reduziert bleibt. Kürzere Inkubationszeiten sind möglich, aber Ausbeute verringern. Bei 4 ° C in Gegenwart von Protease-Inhibitoren, kommt wenig Abbau und eine 3-stündige Ni-Sepharose Inkubationszeit nicht verringert Aktivität des Endprodukts. Sammeln Ni-Sepharose durch Zentrifugation für 2 min bei 4 ° C bei 2.000 rpm (Eppendorf 5810R-Zentrifuge). Speichern Sie eine kleine Auswahl der Überstand für die SDS-PAGE-Analyse (1μL) und entsorgen Sie den Rest (Ni Flow-Through (FT) in Abb. 2)..
- Waschen Sie die abgerufenen Ni-Sepharose mit 25 Säulenvolumen Waschpuffer (40 mM HEPES-KOH pH 7,4, 150 mM KCl, 20 mM MgCl 2, 2,5% (w / v) Glycerin, 20 mM Imidazol, 2 mM β-Mercaptoethanol), 5 Säulenvolumen Waschpuffer mit 1 M KCl, um Verunreinigungen elektrostatisch auf Hsp104 gebunden, und 25 Säulenvolumen Waschpuffer zu entfernen. Sammeln Ni-Sepharose nach jedem Waschen durch Zentrifugation für 2 min bei 4 ° C bei 2.000 rpm (Eppendorf 5810R-Zentrifuge). Nach jedem Waschen und Zentrifugation Zyklus, zu entfernen Puffer durch Aspiration.
- Um Hsp104, mischen Ni-Sepharose mit 1 ml Elutionspuffer (40 mM HEPES-KOH pH 7,4, 150 mM KCl, 20 mM MgCl 2, 2,5% (w / v) Glycerin, 350mm Imidazol, 2 mM β-Mercaptoethanol) pro 1 ml Ni-Sepharose und eluieren Drehen bei 4 ° C für 20 Minuten. Die hohe Imidazol-Konzentration stört das Ni bead-His 6-Interaktion. Nehmen Sie Ni-Sepharose mit leeren 1,2 ml Spin-Säulen (Bio-Rad) durch Zentrifugation für 2 min bei 4 ° C bei 2.000 rpm (Eppendorf 5810R-Zentrifuge). Speichern 1μL Eluat für SDS-PAGE-Analyse (Ni-Eluat in Abb. 2).. Für jeden Liter-Zellen, ist ~ 15 mg Hsp104 bei diesem Schritt erhalten.
- Buffer Austausch Eluat in Buffer Q (20 mM Tris-HCl pH 8, 0,5 mM EDTA, 5 mM MgCl 2, 50mM NaCl) mit MWCO 30.000 Amicon Ultra (Millipore) 15mL Zentrifugenkonzentrator Einheiten. Zunächst konzentrieren Protein ~ 1.5ml fügen Sie dann 14.5mL von Buffer Q-Protein 10-fach verdünnt. Wiederholen Sie die 3-mal für Puffer auszutauschen. Der erhöhte pH-Wert und salzarme sorgt dafür, dass Hsp104 eine hohe negative Ladung hat.
- Purify Hsp104 über Anionenaustauschchromatographie mit einem Buffer Q äquilibriert Resource Q 6mL Spalte (GE). Vor der Injektion wird das Protein durch eine geringe Proteinbindung Millex GP PES Membrane 0,22 &mgr; m Spritzenfilter (Millipore) filtriert. Wir beschäftigen eine Durchflussrate von 1ml/min. Wegspülen schwach bindende Proteine mit 1 Säulenvolumen 20% Puffer Q + (20 mM Tris-HCl pH 8, 0,5 mM EDTA, 5 mM MgCl 2, 1 M NaCl). Eluieren Hsp104 mit linearen Gradienten (20% -50% Puffer Q +) über 5 Säulenvolumen (Abb. 3). Sammeln Sie 1 ml Fraktionen. Hsp104 und die meisten Varianten in der Regel bei ~ 400mm NaCl (31mS/cm) eluieren. Speichern Proben von Peak-Fraktionen (1μL) für die SDS-PAGE-Analyse (Abb. 3, Einschub).
- Um His 6-Tag zu entfernen, Austausch-Protein mit dem Amicon Zentrifugenkonzentrator wie oben beschrieben (siehe Schritt 3.5) in Spaltpuffer (20 mM HEPES-KOH pH 7,4, 140mm KCl und 10 mM MgCl 2). Verwenden Sie Protev Protease (Promega) oder AcTEV Protease (Invitrogen) nach Herstellerangaben. Für Hsp104, haben wir festgestellt, dass höhere Verhältnisse von TEV-Protease: Hsp104 für die vollständige Spaltung erforderlich sind. Wir verwenden ein Verhältnis von 1-Protease-Einheit pro 12μg Hsp104. Die Spaltung sollte bei 30 ° C für 2-4 Stunden durchgeführt werden, gefolgt von einer 16-stündigen Inkubation bei 4 ° C. Finale Hsp104 Konzentration sollte zwischen 20-75 um Monomer sein. Führen die verbleibenden His6-markierten Hsp104 und TEV-Protease mit Ni-Sepharose (GE), indem Ni-Sepharose über die Höhe der Hsp104 in der Spaltungsreaktion (unter der Annahme Perlen binden können 15mg Protein pro ml verpackt Harz). Entfernen Perlen mit leeren Spin-Säulen (Bio-Rad) durch Zentrifugation für 2 min bei 2.000 rpm bei 4 ° C. Proben werden vor und nach der Spaltung (1μL) für die SDS-PAGE-Analyse zur Spaltung, und die Beseitigung von ungespaltenen Protein (Abb. 4) zu gewährleisten.
- Exchange in Size Exclusion-Puffer (20 mM HEPES-KOH pH 7,4, 140mm KCl, 10 mM MgCl 2, 0,1 mM EDTA, 1 mM DTT) mit MWCO 30.000 Amicon Ultra (Millipore) wie in Schritt 3.5 beschrieben.
- Weiter zu reinigen Hsp104 über Größenausschlusschromatographie. Verwenden Sie Größenausschlusschromatographie Puffer äquilibriert Superose 6 oder Superdex 200-Säule (beide GE) (Abb. 5). Vor der Injektion wird das Protein durch eine geringe Proteinbindung Millex GP PES Membrane 0,22 &mgr; m Spritzenfilter (Millipore) filtriert. Für weniger als 10 mg Protein, kann die Größe 10/300 Spalten (24ml) verwendet werden und 0,5 ml Fraktionen gesammelt. Für Proben mit mehr als 10mg, sollte die Größe 12/60 Säule (120 ml) verwendet werden und 1 ml Fraktionen gesammelt. Siehe Anweisungen des Herstellers für Flussraten und Probenmengen geladen werden. Nach der Reinigung werden Hsp104 Fraktionen gepoolt, wie in Abb. gezeigt. 5 und in Amicon Zentrifugenkonzentrator Units (Millipore) konzentriert. Hsp104 gespeichert ist, wie unten beschrieben. Durch den Verlust von Material zu trennen Hsp104 Abbauprodukten und Verunreinigungen die endgültige Ausbeute an gereinigtem full-length Hsp104 ist ~ 1-3mg pro Liter ab Kultur.
4. Hsp104 Disaggregase Activity
- Nach der Reinigung ist es gute Praxis, Hsp104 Aktivität in einer Disaggregation Assay zu beurteilen. Normalerweise beschäftigen wir eine Luciferase Assay Reaktivierung 2 (Abb. 6). In diesem Test Hsp104 in Verbindung mit dem Hsp70-Chaperon-System (Hsp70 und Hsp40) disaggregiert Harnstoff-denaturierter Glühwürmchenluciferase Aggregate 2. Löslich Luciferase katalysiert die Oxidation von Luciferin zu Oxyluciferin, eine Reaktion, die Licht-Releases. Durch die Überwachung der Lumineszenz, die relative Reaktivierung und damit Disaggregation kann der Luziferase bestimmt werden. Hsp104 Lage sein muss, synergistisch mit dem Hsp70 Co-Chaperon-Systems zusammenarbeiten. Die Menge der verwerteten Lumineszenz hängt von der genauen Hsp70: Hsp40-Paar verwendet. Wir setzen routinemäßig Hsp72 und Hsp40 (Assay Designs). Zumindest sollten aktive Hsp104 produzieren eine 5-fache Steigerung der Reaktivierung von Luciferase, wenn die Reaktivierung von Hsp72 Vergleich: Hsp40 allein (Abb. 6). Hsp104 Zubereitungen, die dieses Maß an Aktivität nicht erreichen, werden verworfen.
5. Hsp104 Lagerung
- Für kurzfristige Lagerung kann Hsp104 bei 4 ° C in der Ausschluss-Puffer gehalten werden. Allerdings wird Aktivität nach 2-3 Tagen bei 4 ° C sinken und scharf nach 1 Woche bei 4 ° C. Für Disaggregation Assays empfehlen wir, dass Hsp104 so bald wie möglich sollte nach der Reinigung verwendet werden. Idealerweise sollte Hsp104 sofort für Amyloid Disaggregation Assays verwendet werden.
- Wenn Eiweiß muss langfristig aufbewahrt werden, ist Hsp104 Einlagerung Puffer (20 mM HEPES-KOH pH 7,4, 140mm KCl, 10 mM MgCl 2, 0,1 mM EDTA, 1 mM DTT, 10% Glycerin (w / v)) ausgetauscht. 100 &mgr; l Aliquots in flüssigem Stickstoff eingefroren und bei -80 ° C. Frost-Tau-Zyklen drastisch reduzieren Hsp104 Aktivitätund sollte vermieden werden. Wir empfehlen langsam auftauen Hsp104 auf Eis. Amyloid-disaggregase Aktivität wird stark zurückgehen nach 1 Monat bei -80 ° C.
6. Repräsentative Ergebnisse und Fakten:
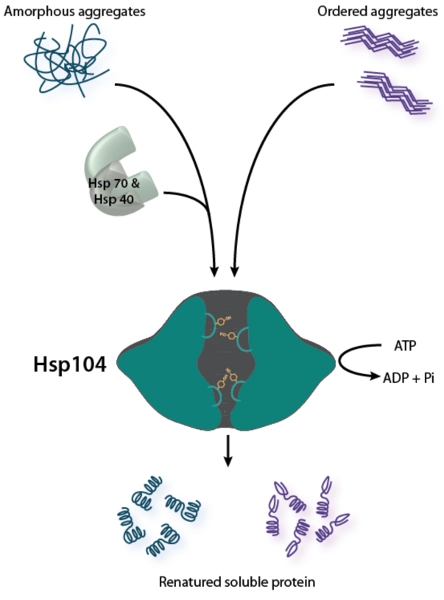
Abbildung 1. Hsp104 ist ein bifunktionelles disaggregase. Disaggregation der ungeordnete Aggregate (siehe links) erfordert die Zusammenarbeit der Hsp70-Chaperon-System (Hsp70 und Hsp40) 2. Hsp104 umbaut bestellt Amyloidaggregaten (siehe rechts) ohne die Hilfe von Hsp70 und Hsp40 in vitro, sondern Hsp70 und Hsp40 kann Hsp104 Aktivität gegen Amyloid 26,28 zu verbessern. Für beide Arten der aggregierten Strukturen, Hsp104 Paare ATP-Hydrolyse, um Substrat Translokation durch seine zentrale Kanal Disaggregation zu fördern. Tyrosin-tragenden Pore Schleifen engagieren und Shuttle-Substrat durch den zentralen Kanal 49-52.

Abbildung 2. SDS-PAGE-Analyse von Ni-Sepharose Affinitätsreinigung Schritt. Lysate, Ni Load, Ni FT und Ni-Eluat-Proben wurden durch SDS-PAGE mit einem 4-20% Tris-HCl 1,0 Criterion-Gel (Bio-Rad) und Coomassie gefärbt . Beachten Sie, dass nicht alle Hsp104 kann in die Ni-Sepharose binden. Broad Range Molekulargewichtsmarker (Bio-Rad) angezeigt werden (linke Spur).
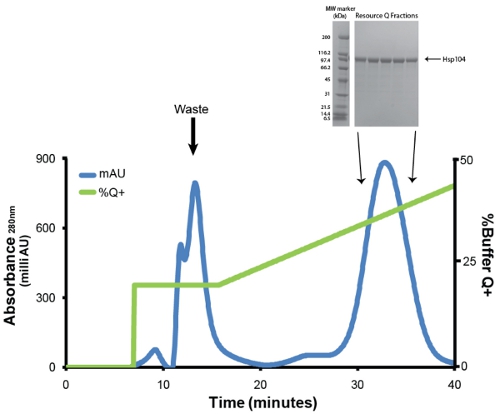
Abbildung 3. Resource Q Reinigung von Ni-Sepharose-Eluat. Blaue Kurve stellt die Absorption bei 280nm und grüne Kurve stellt% Puffer Q + (Maximum bei 50%). Der erste Peak, der bei 20% eluiert Q + enthält Verunreinigungen, Abbauprodukte und falsch gefalteten Hsp104. Die zweite und Hauptpeak enthält korrekt gefaltet und aktiv Hsp104. Die Fließgeschwindigkeit betrug 1ml/min. Gradient von 20 bis 50% Q + 30 Minuten oder 5 Säulenvolumen. Kleines Bild: Peak-Fraktionen werden durch SDS-PAGE-Analyse mit einem 4-20% Tris-HCl 1,0 Criterion-Gel (Bio-Rad) und Coomassie gefärbt aufgelöst. Broad Range Molekulargewichtsmarker (Bio-Rad) angezeigt (links).

Abbildung 4. SDS-PAGE-Analyse von Protev Proteaseschnittstelle Schritt. His 6-Hsp104 von Resource Q Reinigung wurde mit Protev Protease 4 Stunden bei 30 ° C und dann 16 Stunden bei 4 ° C behandelt Die Proben wurden die durch SDS-PAGE mit einem 4-20% Tris-HCl 1,0 Criterion-Gel (Bio-Rad) und Coomassie gefärbt. Beachten Sie, dass Protev gespalten Hsp104 schneller wandert. TEV-Protease und ungespaltenen Hsp104 haben mit Ni-Sepharose erschöpft wie in Schritt 3.7 beschrieben. Broad Range Molekulargewichtsmarker (Bio-Rad) angezeigt (links).
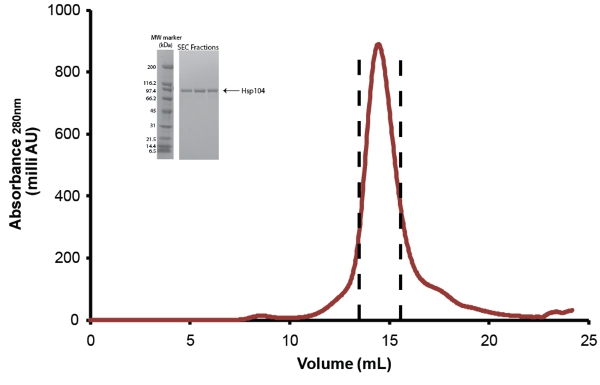
Abbildung 5. Ausschluss-Reinigung von Hsp104. Cleaved Hsp104 weiteren wurde über eine Superose 6 Gelfiltrationssäule (10/300, GE) gereinigt. Durchfluss = 0.4ml/min. Der Gipfel zwischen den gestrichelten Linien stellt gepoolten Fraktionen. Kleines Bild: Peak-Fraktionen werden durch SDS-PAGE-Analyse mit einem 4-20% Tris-HCl 1,0 Criterion-Gel (Bio-Rad) und Coomassie gefärbt aufgelöst. Broad Range Molekulargewichtsmarker (Bio-Rad) angezeigt (links).
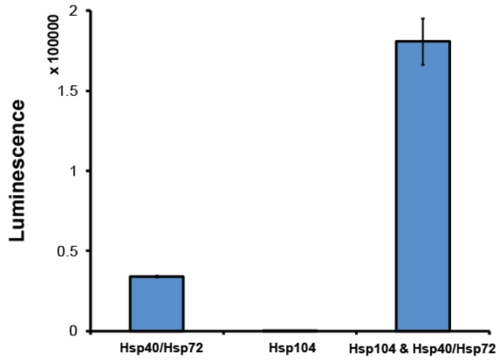
Abbildung 6. Luciferase-Assay Reaktivierung. Vergällt Glühwürmchenluciferase Aggregate (50nm) wurden entweder mit Hsp72 und Hsp40 (1 &mgr; M) (beide von Assay Designs), Hsp104 (6μM Monomer) oder Hsp104, Hsp72 und Hsp40 für 90min bei 25 ° C inkubiert Denaturierten Luciferase ist nur dann voll in der Anwesenheit beider Hsp104 und Hsp40/Hsp72 reaktiviert. Recovered Lumineszenz basiert auf einem Infinite M1000-Reader (Tecan) gemessen. Die Werte stellen Mittelwerte ± SEM (n = 3).
Access restricted. Please log in or start a trial to view this content.
Diskussion
Timeline: Für maximal Hsp104 Aktivität empfehlen wir, dass die gesamte Reinigungsschema so schnell wie möglich abgeschlossen werden. Allerdings macht die Zahl der Reinigungsschritte ein anspruchsvoller Zeitplan, der vielleicht nicht immer praktisch. Wenn die Reinigung Schritte werden so schnell wie möglich durchgeführt werden, die Zeit zwischen dem Ende der Nacht Ausdruck durch die 2-4 Stunden Inkubation bei 30 ° C mit TEV-Protease beträgt ca. 9-11 Stunden. Ein möglicher Ort zum Verweilen ist nach der TEV...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch ein Stipendium des NIH (5T32GM008275-22) und ein American Heart Association Promotionsstipendium (EAS) unterstützt, ein Chemie-Biologie-Schnittstelle Stipendium der NIH (2T32GM071339-06A1) (zu MED) und Zuschüsse aus dem NIH (1DP2OD002177-01 und NS067354-0110), Die Ellison Medical Foundation und The Bill and Melinda Gates Foundation (JS).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
|---|---|---|---|
| BL21-CodonPlus-RIL kompetenter Zellen | Stratagene, Agilent Technologies | 230255 | |
| 2xYT Brühe | USB | 75864 | |
| Complete, Mini, EDTA-free Protease-Inhibitor Tabletten | Roche | 1836170 | |
| Pepstatin A | Sigma | P4265 | |
| Ni-Sepharose 6 Fast Flow | GE Healthcare | 17-5318-02 | |
| Amicon Ultra-15 Zentrifugalfilter Einheiten (MWCO 30.000) | Millipore | UFC903008 | |
| Resource Q - 6ml Spalte | GE Healthcare | 17-1179-01 | |
| Protev Protease | Promega | V6052 | |
| AcTEV Protease | Invitrogen | 12575015 | |
| Superose 6 10 / 300 GL | GE Healthcare | 17-5172-01 | |
| Hsp40 | Assay Designs | SPP-400 | |
| Hsp72 | Assay Designs | ADI-NSP-555 |
Referenzen
- Erzberger, J. P., Berger, J. M. Evolutionary relationships and structural mechanisms of AAA+ proteins. Annu Rev Biophys Biomol Struct. 35, 93-114 (2006).
- Glover, J. R., Lindquist, S. Hsp104, Hsp70, and Hsp40: A Novel Chaperone System that Rescues Previously Aggregated Proteins. Cell. 94, 73-82 (1998).
- Parsell, D. A., Kowal, A. S., Singer, M. A., Lindquist, S. Protein disaggregation mediated by heat-shock protein Hsp104. Nature. 372, 475-478 (1994).
- Vashist, S., Cushman, M., Shorter, J. Applying Hsp104 to protein-misfolding disorders. Biochem Cell Biol. 88, 1-13 (2010).
- Parsell, D. A., Sanchez, Y., Stitzel, J. D., Lindquist, S. Hsp104 is a highly conserved protein with two essential nucleotide-binding sites. Nature. 353, 270-273 (1991).
- Doyle, S. M., Wickner, S. Hsp104 and ClpB: protein disaggregating machines. Trends Biochem Sci. 34, 40-48 (2009).
- Shorter, J. Hsp104: a weapon to combat diverse neurodegenerative disorders. Neurosignals. 16, 63-74 (2008).
- Glover, J. R., Lum, R. Remodeling of protein aggregates by Hsp104. Protein Pept Lett. 16, 587-597 (2009).
- Mogk, A., Haslberger, T., Tessarz, P., Bukau, B. Common and specific mechanisms of AAA+ proteins involved in protein quality control. Biochem Soc Trans. 36, 120-125 (2008).
- Grimminger-Marquardt, V., Lashuel, H. A. Structure and function of the molecular chaperone Hsp104 from yeast. Biopolymers. 93, 252-276 (2010).
- Sanchez, Y., Lindquist, S. L. HSP104 required for induced thermotolerance. Science. 248, 1112-1115 (1990).
- Sanchez, Y., Taulien, J., Borkovich, K. A., Lindquist, S. Hsp104 is required for tolerance to many forms of stress. Embo J. 11, 2357-2364 (1992).
- Alberti, S., Halfmann, R., King, O., Kapila, A., Lindquist, S. A systematic survey identifies prions and illuminates sequence features of prionogenic proteins. Cell. 137, 146-158 (2009).
- Halfmann, R., Alberti, S., Lindquist, S. Prions, protein homeostasis, and phenotypic diversity. Trends Cell Biol. 20, 125-1233 (2010).
- Shorter, J., Lindquist, S. Prions as adaptive conduits of memory and inheritance. Nat Rev Genet. 6, 435-450 (2005).
- True, H. L., Berlin, I., Lindquist, S. L. Epigenetic regulation of translation reveals hidden genetic variation to produce complex traits. Nature. 431, 184-187 (2004).
- True, H. L., Lindquist, S. L. A yeast prion provides a mechanism for genetic variation and phenotypic diversity. Nature. 407, 477-483 (2000).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6, e294-e294 (2008).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi+]. Science. 268, 880-884 (1995).
- Halfmann, R., Lindquist, S. Epigenetics in the extreme: prions and the inheritance of environmentally acquired traits. Science. 330, 629-632 (2010).
- Satpute-Krishnan, P., Langseth, S. X., Serio, T. R. Hsp104-dependent remodeling of prion complexes mediates protein-only inheritance. PLoS Biol. 5, e24-e24 (2007).
- Sweeny, E. A., Shorter, J. Prion proteostasis: Hsp104 meets its supporting cast. Prion. 2, 135-140 (2008).
- Lo Bianco, C. Hsp104 antagonizes alpha-synuclein aggregation and reduces dopaminergic degeneration in a rat model of Parkinson disease. J Clin Invest. 118, 3087-3097 (2008).
- Narayanan, S., Walter, S., Reif, B. Yeast prion-protein, sup35, fibril formation proceeds by addition and substraction of oligomers. Chembiochem. 7, 757-765 (2006).
- Savistchenko, J., Krzewska, J., Fay, N., Melki, R. Molecular chaperones and the assembly of the prion Ure2p in vitro. J Biol Chem. 283, 15732-15739 (2008).
- Shorter, J., Lindquist, S. Hsp104 catalyzes formation and elimination of self-replicating Sup35 prion conformers. Science. 304, 1793-1797 (2004).
- Shorter, J., Lindquist, S. Destruction or potentiation of different prions catalyzed by similar Hsp104 remodeling activities. Mol Cell. 23, 425-438 (2006).
- Shorter, J., Lindquist, S. Hsp104, Hsp70 and Hsp40 interplay regulates formation, growth and elimination of Sup35 prions. Embo J. 27, 2712-2724 (2008).
- Doyle, S. M. Asymmetric deceleration of ClpB or Hsp104 ATPase activity unleashes protein-remodeling activity. Nat Struct Mol Biol. 14, 114-122 (2007).
- Wendler, P. Atypical AAA+ subunit packing creates an expanded cavity for disaggregation by the protein-remodeling factor Hsp104. Cell. 131, 1366-1377 (2007).
- Hinault, M. P. Stable alpha-synuclein oligomers strongly inhibit chaperone activity of the Hsp70 system by weak interactions with J-domain co-chaperones. J Biol Chem. 285, 38173-38182 (2010).
- Tipton, K. A., Verges, K. J., Weissman, J. S. In vivo monitoring of the prion replication cycle reveals a critical role for Sis1 in delivering substrates to Hsp104. Mol Cell. 32, 584-591 (2008).
- Bieschke, J., Cohen, E., Murray, A., Dillin, A., Kelly, J. W. A kinetic assessment of the C. elegans amyloid disaggregation activity enables uncoupling of disassembly and proteolysis. Protein Sci1. 8, 2231-2241 (2009).
- Cohen, E., Bieschke, J., Perciavalle, R. M., Kelly, J. W., Dillin, A. Opposing activities protect against age-onset proteotoxicity. Science. 313, 1604-1610 (2006).
- Cohen, E. Reduced IGF-1 signaling delays age-associated proteotoxicity in mice. Cell. 139, 1157-1169 (2009).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 23, 1191-11201 (2010).
- Carmichael, J. Bacterial and yeast chaperones reduce both aggregate formation and cell death in mammalian cell models of Huntington's disease. Proc Natl Acad Sci. 97, 9701-9705 (2000).
- Vacher, C., Garcia-Oroz, L., Rubinsztein, D. C. Overexpression of yeast hsp104 reduces polyglutamine aggregation and prolongs survival of a transgenic mouse model of Huntington's disease. Hum Mol Genet. 14, 3425-3433 (2005).
- Liu, Y. H. Heat Shock Protein 104 Inhibited the Fibrillization of Prion Peptide 106-126 and Disassembled Prion peptide 106-126 Fibrils in vitro. Int J Biochem Cell Biol. , (2011).
- Wendler, P., Saibil, H. R. Cryo electron microscopy structures of Hsp100 proteins: crowbars in or out. Biochem Cell Biol. 88, 89-96 (2010).
- Wendler, P. Motor mechanism for protein threading through Hsp104. Mol Cell. 34, 81-92 (2009).
- Lee, S., Sielaff, B., Lee, J., Tsai, F. T. CryoEM structure of Hsp104 and its mechanistic implication for protein disaggregation. Proc Natl Acad Sci. 107, 8135-8140 (2010).
- Parsell, D. A., Kowal, A. S., Lindquist, S. Saccharomyces cerevisiae Hsp104 protein. Purification and characterization of ATP-induced structural changes. J Biol Chem. 269, 4480-4487 (1994).
- Schirmer, E. C., Queitsch, C., Kowal, A. S., Parsell, D. A., Lindquist, S. The ATPase activity of Hsp104, effects of environmental conditions and mutations. J Biol Chem. 273, 15546-15552 (1998).
- Schirmer, E. C., Ware, D. M., Queitsch, C., Kowal, A. S., Lindquist, S. L. Subunit interactions influence the biochemical and biological properties of Hsp104. Proc Natl Acad Sci. 98, 914-919 (2001).
- Hattendorf, D. A., Lindquist, S. L. Cooperative kinetics of both Hsp104 ATPase domains and interdomain communication revealed by AAA sensor-1 mutants. EMBO J. 21, 12-21 (2002).
- Schirmer, E. C., Lindquist, S. Purification and properties of Hsp104 from yeast. Methods Enzymol. 290, 430-444 (1998).
- Hattendorf, D. A., Lindquist, S. L. Analysis of the AAA sensor-2 motif in the C-terminal ATPase domain of Hsp104 with a site-specific fluorescent probe of nucleotide binding. Proc Natl Acad Sci. 99, 2732-2737 (2002).
- Lum, R., Niggemann, M., Glover, J. R. Peptide and protein binding in the axial channel of Hsp104. Insights into the mechanism of protein unfolding. J Biol Chem. 283, 30139-30150 (2008).
- Lum, R., Tkach, J. M., Vierling, E., Glover, J. R. Evidence for an unfolding/threading mechanism for protein disaggregation by Saccharomyces cerevisiae Hsp104. J Biol Chem. 279, 29139-29146 (2004).
- Tessarz, P., Mogk, A., Bukau, B. Substrate threading through the central pore of the Hsp104 chaperone as a common mechanism for protein disaggregation and prion propagation. Mol Microbiol. 68, 87-97 (2008).
- Weibezahn, J. Thermotolerance requires refolding of aggregated proteins by substrate translocation through the central pore of ClpB. Cell. 119, 653-665 (2004).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten