このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
Hsp104の精製、タンパク質Disaggregase
要約
ここで、我々は、高活性Hsp104、六量体AAA +タンパク質酵母、タンパク質の分解に結合ATPの加水分解からの精製のためのプロトコルについて説明します。このスキームでは、からアフィニティー精製のためのしたHis6 -タグ付き構造を活用して E.大腸菌 TEVプロテアーゼとアニオン交換クロマトグラフィー、His 6 - タグの削除、およびサイズ排除クロマトグラフィー。
要約
Hsp104は酵母から六量AAA +タンパク質1、タンパク質分解2-10の結合ATPの加水分解(図1)です。この活動は2つの重要な選択的な利点を与える。最初に、Hsp104による無秩序な集合体の再生は、熱ショック3,5,11,12を含む、様々なタンパク質折り畳みのストレスの後に酵母の生存を支援します。第二に、Hsp104によるクロスβアミロイド線維の改造は、有益なの貯水池として無数のプリオン(感染性アミロイド)を悪用し、13から22まで表現型変異を遺伝する酵母を可能にします。驚くべきことに、Hsp104は直接酵母のプリオンタンパク質Sup35由来とUre2 23から30で構成されるものを含むpreamyloidのオリゴマーおよびアミロイド線維を、remodels。このアミロイド-リモデリング機能は、酵母Hsp104の特殊な一面です。 E.大腸菌オルソログ、ClpBは、改造のpreamyloidのオリゴマーまたはアミロイド線維26,31,32に失敗します。
Hsp104のオルソログは、動物、perplexingly、除く生命のすべての国で発見されています。確かに、動物細胞は、任意の酵素システムを持っているかどうかを再生(むしろ劣化より)に結合する蛋白質の分解は、未知の33から35のままという。従って、我々と他のHsp104は毒性preamyloidのオリゴマーおよびアミロイド線維4,7,23,36-38に特定のタンパク質の折り畳みと接続されているさまざまな神経変性疾患の治療薬として開発される可能性がありますことを提案した。直接これらの疾患に関連する集約された種を対象にない治療法はありません。まだ、Hsp104はパーキンソン病23と同様にPrPの39のアミロイドの形態で接続されているα-シヌクレイン、から構成される毒性オリゴマーとアミロイド線維を溶解する。重要なのは、Hsp104は、タンパク質の凝集を低減し、パーキンソン病23ハンチントン病38の齧歯類モデルでの神経変性を改善する。理想的には、治療を最適化し、副作用を最小限に抑えるために、Hsp104は選択問題4,7の疾患に中心的な特定の集合体を改造するように設計し、増強される。しかし、どのようにHsp104 disaggregatesの限られた構造とメカニズムの理解が集約された構造と無関係な蛋白質のそのような多様なレパートリーは、これらの努力の30,40-42をいらだた。
Hsp104の構造とメカニズムを理解するには、最小限のコンポーネントを使用して、純粋な蛋白質と再構成するそのdisaggregaseの活動を研究することが不可欠である。 Hsp104は、ADPまたはATPの存在下で、またはヌクレオチド43から46の非存在下での高タンパク質濃度でhexamerizes〜5.3のpIを持つ102kDa蛋白質である。ここでは、Eからの高活性、安定Hsp104の精製に最適化されたプロトコルを記述する大腸菌 。 E.の使用大腸菌は、簡略化された大規模な生産を可能にし、私たちの手法は、多数のHsp104バリアントのために迅速かつ確実に行うことができます。我々のプロトコルは、Hsp104純度を向上させ、Eからの以前の精製法に比べて彼の6タグの除去を簡素化大腸菌 47。また、我々のプロトコルは2つのより最近のプロトコル26,48よりも容易な、便利です。
プロトコル
1。 Hsp104の発現
- プラスミドは、E.の精製に用いる大腸菌 、pPROEX - HTB - Hsp104は、trcプロモーター26の誘導制御下Hsp104オープンリーディングフレームが含まれています。プラスミドは、TEVプロテアーゼ切断により除去することができるN -末端His 6 - tagをHsp104を生成します。コドン最適化E.にpPROEX - HTB - Hsp104を変換典型的な細菌の形質転換法を用いて大腸菌は BL21 - CodonPlus - RIL細胞(Stratagene、アジレントテクノロジー)(例:製造業者の指示に従って)。コドン最適化E.を使用することが重要です。 Hsp104は珍しいコドンバイアスを持っているので、 大腸菌の菌株。
- 100mLの2XYT文化(USB.レシピさ:16g / Lカゼインペプトン、10gの/ L酵母エキス、5グラム/ LのNaCl、pH 7.0)に植菌、新鮮な形質で100μg/mLアンピシリン及び34μg/mLクロラムフェニコールを添加したが、37で一晩成長° 200rpmで振盪しながらC。
- 2Lフラスコに100μg/mLアンピシリン及び34μg/mLクロラムフェニコールで6 × 1L 2XYTに一晩培養液10mLを接種する。 37 ° CまでのOD 600 = 0.4〜0.6で、250rpmで振とうする。揺れを停止し、15に温度を下げる℃にそれは15℃に達するまで細胞がインキュベーターに30分間落ち着きはらった平衡させる一度15 ° Cは1mmの最終濃度になるようにIPTGを添加し、タンパク質発現を誘導達している。 (12〜16時間)一晩250rpmで振盪しながら再開する。
2。細胞の収穫と溶解
- 4℃20分のための4,000 rpmで遠心分離により細胞を回収(予備冷却したローターで)° C(我々はソーバルRC 3BP +遠心機を使用)。 Hsp104の活性が減少しているため、以降のステップは、すぐに実行する必要があるときにE.大腸菌の細胞が凍結されています。さらに、後続のすべてのステップは氷上または4℃で行ってください
- 予冷した(氷上で)10mLの溶解バッファー(40mmのHEPES - KOH pH7.4の、500mmの塩化カリウム、20mMのMgCl 2を 、2.5%(w / v)のグリセロール、20mMのイミダゾール、5μMのペプスタチン、完全なプロテアーゼ阻害剤カクテル(1 EDTAに再懸濁し、細胞ペレットフリーtablet/50mL)、および2mMβ-メルカプトエタノール)。
- 氷水に浸漬熱交換器を使用するフレンチプレス(Emulsiflex)ホモジナイザーで細胞を溶解する。使用前に、すべての細胞塊が可溶化されていることを確認してください。溶解緩衝液でホモジナイザーを平衡化した後、15,000-18,000 psiの圧力でシリンダーを介して2つのパスが完全に溶解するのに十分です。フレンチプレスを使用できない場合は、超音波処理に続いてリゾチームとのインキュベーションは、(議論を参照)を使用することができる。 SDS - PAGE分析(1μL)(図中のライセート。2)に溶解した細胞の小さなサンプルを保存します。
- 4時20分、16,000 rpmで遠心分離により細胞破片を除去° C(我々は+ centifugeソーバルRC5Cを使用)。次のステップの上清を保持し、SDS - PAGE解析のために上澄みの少量のサンプル(1μL)を保存する(図中のNiのロード。2)。
3。 Hsp104の精製
- 溶解バッファー平衡化したNi -セファロースビーズ(GE)の50%スラリー12mL(細胞の1L当たり2mLのニッケル - セファロースビーズ)で、ステップ2.4から上清を混ぜる。
- 4℃で3時間ゆっくりとサンプルを回転℃でのNi -セファロースが均等に中断し、泡が最小化されているままであるように。短いインキュベーション時間は可能ですが、収量が低下します。 4℃でプロテアーゼ阻害剤の存在下でC、少し劣化が発生し、3時間のNi -セファロースのインキュベーション時間は、最終産物の活性を減少させない。 4℃で2分2000回転(エッペンドルフ5810R遠心)でCのための遠心分離によるNi -セファロースを収集する。 SDS - PAGE解析のために上清の小さなサンプルを保存(1μL)と(図を介してNIの流れ(FT)2)、残りを捨てる。
- 洗浄バッファー(40mmのHEPES - KOH pH7.4の、150MMのKCl、20mMのMgCl 2を 、2.5%(w / v)のグリセロール、20mMのイミダゾール、2mMのβ-メルカプトエタノール)、の5カラム容量の25カラム容積で取得したのNi -セファロースを洗浄してくださいHsp104に静電的に結合した汚染物質、および洗浄緩衝液の25カラムボリュームを削除するには、1M KClでバッファを洗う。 4℃で2分2000回転(エッペンドルフ5810R遠心)でCのための遠心分離により各洗浄後のNi -セファロースを収集する。各洗浄と遠心分離サイクルの後、吸引してバッファーを除去する。
- 1mLのニッケル-セファロースあたりHsp104を溶出するために、1mLの溶出バッファーでミックスのNi -セファロース(40mmのHEPES - KOH pH7.4の、150MMのKCl、20mMのMgCl 2を 、2.5%(w / v)のグリセロール、350mmのイミダゾール、2mMのβ-メルカプトエタノール)と4℃で20分間回転させます。高いイミダゾール濃度は、Niビーズ-彼の6の相互作用を破壊する。 4℃、2分間の遠心により空の1.2 mlをスピンカラム(Bio - Rad社)°、2,000 rpmでC(エッペンドルフ5810R遠心)でNI -セファロースを削除します。 (図中のNiの溶出液2)SDS - PAGE解析のために溶出液の1μLを保存します。細胞の1リットルの場合は、Hsp104の〜15mgは、このステップで得られる。
- MWCO3万アミコンウルトラ(ミルを使用してバッファQ(20mMのトリス- HCl pH8の、0.5mmのEDTA、5mMのMgCl 2を 、50mmの塩化ナトリウム)にバッファー交換の溶出液lipore)15mlの遠心濃縮装置。最初に、10倍のタンパク質を希釈するバッファーQの14.5mLを追加して〜1.5mlのためにタンパク質を集中。緩衝液交換を3回繰り返します。増加したpHと低塩濃度は、Hsp104は高い負の電荷を持つことが保証されます。
- バッファQ平衡化したリソースQ 6mLカラム(GE)を用いて陰イオン交換クロマトグラフィーを経由してHsp104を浄化する。注射の前に、蛋白質は低タンパク結合マイレクスGP PESメンブレン0.22μMシリンジフィルター(Millipore)で濾過する。我々は、1mL/minの流量を採用。 20%のバッファの1列のボリュームと弱く結合タンパク質洗い流すQ +(20mMのトリス- HCl pH8の、0.5mmのEDTA、5mMのMgCl 2を 、1MのNaCl)。 5カラム体積以上の直線勾配で溶出Hsp104(20%〜50%バッファQ +)(図3)。 1ミリリットルフラクションを集める。 Hsp104とほとんどの亜種は、一般的に〜400mmの塩化ナトリウム(31mS/cm)で溶出。 SDS - PAGE解析のためのピーク分画(1μL)(図3、挿入図)のサンプルを保存します。
- アミコン遠心濃縮器を使用して、彼の6タグを削除するには、交換のタンパク質は、切断バッファー(20mMのHEPES - KOH pH7.4の、140mmの塩化カリウム、及び10mMのMgCl 2の )に(ステップ3.5を参照)上記の説明。製造元の指示に従ってproTEVプロテアーゼ(Promega社製)またはAcTEVプロテアーゼ(Invitrogen)を使用してください。 Hsp104完全な切断のために必要とされています:Hsp104のために、我々は、TEVプロテアーゼの高い比率がことを発見した。我々は、12μgHsp104あたり1プロテアーゼのユニットの比率を使用してください。開裂は、4で16時間のインキュベーション℃に続いて、2〜4時間、30℃で実施されるべき最後のHsp104の濃度は20 -75μmの単量体の間でなければなりません。開裂反応(ビーズがパックされた樹脂のmL当たりのタンパク質15mg結合することができると仮定)におけるHsp104の量に対して過剰のNi -セファロースを追加することにより、ニッケル - セファロース(GE)と残りのしたHis6 -タグ付きHsp104とTEVプロテアーゼを使い果たす。 4℃で2,000 rpmで2分間遠心分離して、空のスピンカラム(Bio - Rad社)でビーズを削除℃に開裂、および未切断タンパク質の除去(図4)を確保するためにSDS - PAGE解析のための開裂(1μL)の前と後のサンプルを収集する。
- ステップ3.5で説明したようにMWCO3万アミコンウルトラ(Millipore)を用いてサイズ排除バッファー(20mMのHEPES - KOH pH7.4の、140mmの塩化カリウム、10mMのMgCl 2を 、0.1mmのEDTA、1mMのDTT)に交換。
- さらにサイズ排除クロマトグラフィーを経由してHsp104を浄化する。サイズ排除バッファーで平衡化スーパーローズ6またはSuperdex 200カラム(GEからの両方)(図5)を使用してください。注射の前に、蛋白質は低タンパク結合マイレクスGP PESメンブレン0.22μMシリンジフィルター(Millipore)で濾過する。タンパク質未満の10mgのために、300分の10サイズのカラム(24mL)を使用することができますし、0.5ミリリットル分画を集めています。 10mgのより大きいサンプルについては、60分の12サイズの列は(120mL)を使用する必要がありますし、1mlの画分を収集している。フローレートおよびロードするサンプル量については、製造元の説明書を参照してください。精製後、Hsp104の画分を、図2に示すようにプールされます。 5、アミコン遠心濃縮ユニット(ミリポア)に集中。後述するようにHsp104が格納されています。材料の損失によるHsp104分解生成物と汚染物質を分離するにはフルレングスHsp104を精製の最終収率は出発文化のリットルあたり〜1ミリグラムです。
4。 Hsp104 Disaggregase活動
- 精製後、それは分解アッセイでHsp104活性を評価することをお勧めします。一般的に、我々はルシフェラーゼ再活性化アッセイ2(図6)を採用。このアッセイにおいて、Hsp70のシャペロンシステム(Hsp70のとHSP40)disaggregates尿素変性ホタルルシフェラーゼの凝集体2と共にHsp104。水溶性のルシフェラーゼは、ルシフェリン、光を放出反応にルシフェリンの酸化を触媒する。ルシフェラーゼの監視発光、相対的な再活性化、そのため分解によって決定することができる。 Hsp104は相乗的にHsp70のコシャペロンシステムと協力できなければなりません。利用HSP40ペア:回収された発光量は、正確なHsp70のに依存します。我々は日常的にHsp72とHSP40を(アッセイデザイン)採用。単独でHSP40(図6):最低でも、アクティブHsp104はHsp72によって再活性化と比較してルシフェラーゼの再活性化の5倍の増加を生成する必要があります。活動のこのレベルに達するために失敗したHsp104の準備は破棄されます。
5。 Hsp104ストレージ
- 短期記憶の場合は、Hsp104は° Cサイズ排除バッファ4に保つことができる。しかし、活動は4℃と急激に1週間後に4で2〜3日後に低下する℃に分解アッセイのために我々は、Hsp104は精製後できるだけ早く使用することをお勧めします。理想的には、Hsp104はアミロイド分解アッセイのために直ちに使用する必要があります。
- タンパク質は、長期保存しなければならない場合、Hsp104は保存バッファー(20mMのHEPES - KOH pH7.4の、140mmの塩化カリウム、10mMのMgCl 2を 、0.1mmのEDTA、1mMのDTT、10%グリセロール(W / V))に交換されます。 100μlのアリコートを液体窒素で凍結し、-80℃で保存されているスナップです。凍結融解サイクルが大幅にHsp104活性を低下させるとは避けるべきである。我々は徐々に氷の上Hsp104を解凍することをお勧めします。アミロイド- disaggregase活性が-80 1ヶ月後に急激に減少する℃に
6。代表的な結果と図:
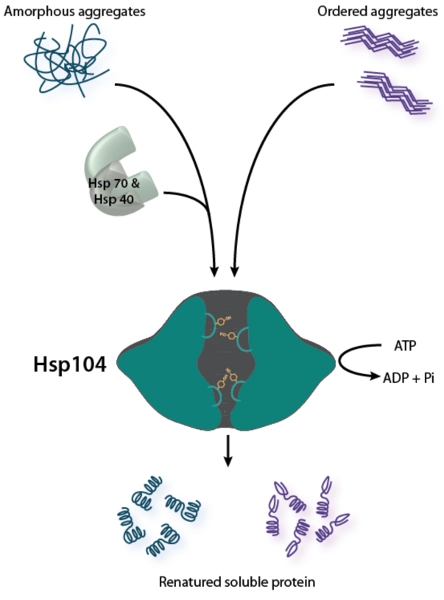
図1。 Hsp104は、二官能性disaggregaseです。不規則な凝集体の分解は、(左図参照)Hsp70のシャペロンシステム(Hsp70のとHSP40)2の協力が必要です。 Hsp104のremodels は 、in vitro での Hsp70のとHSP40の援助なしにアミロイドの凝集体を(右図)注文が、Hsp70のとHSP40は、アミロイド26,28に対してHsp104の活性を向上させることができます。集約された構造、分解を促進するため、その中心的なチャネルを介して基板の転座にHsp104結合ATPの加水分解の両方のタイプの。チロシン-ベアリング細孔ループは中央のチャネル49から52を介して関与し、シャトルの基板。

図2。のNi -セファロースアフィニティー精製ステップのSDS - PAGE分析。ライセート、ニッケルの負荷、ニッケルFTとNiの溶出液のサンプルは4-20%トリス-塩酸1.0ミリメートル基準ゲル(Bio - Rad社)とクマシー染色したを使用してSDS - PAGEで分画し、 。すべてのHsp104は、Ni -セファロースに結合することができることに注意してください。広い範囲の分子量マーカー(Bio - Rad社)(レーン左)が表示されます。
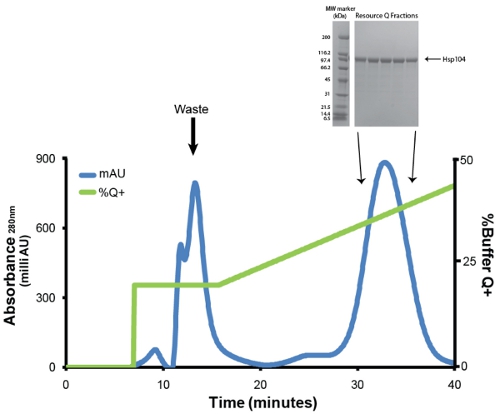
図3。のNi -セファロース溶出液のリソースのQ精製。青のトレースは、280と緑のトレースでの吸光度を表すには%バッファQ +を(50%最大値)を表します。 20%で溶出最初のピーク、Q +不純物、分解生成物と不適切に折り畳まHsp104が含まれています。番目と主要なピークが正しく折り畳まれたとアクティブHsp104が含まれています。流速は1ml/minであった。 20から50パーセントQ +からの勾配は30分または5カラム体積のです。挿入図:ピーク画分を4-20%トリス - 塩酸1.0ミリメートル基準ゲル(Bio - Rad社)とクマシー染色したを使用してSDS - PAGE分析によって解決されます。広い範囲の分子量マーカー(バイオラッド)は、(左)が表示されます。

図4。 proTEVプロテアーゼ切断ステップのSDS - PAGE分析。彼の6 Hsp104リソースQの精製から、30で4次に℃、16時間℃で4時間proTEVのプロテアーゼで処理し、サンプルでは4-20%トリス - 塩酸1.0ミリメートル基準ゲル(Bio - Rad社)とクマシー染色したを使用してSDS - PAGEによって分画した。 proTEV劈開Hsp104がより迅速に移行することに注意してください。ステップ3.7で説明したようにTEVプロテアーゼと未開Hsp104をNi -セファロースで枯渇している。広い範囲の分子量マーカー(バイオラッド)は、(左)が表示されます。
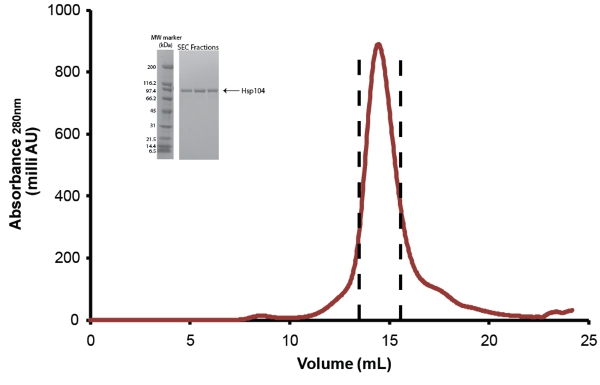
図5。 Hsp104。劈開Hsp104 のサイズ排除精製は、さらにスーパーローズ6ゲル濾過カラム(300分の10、GE)を経由して精製した。流速= 0.4ml/min。破線の間にピークがプールされた画分を表します。挿入図:ピーク画分を4-20%トリス - 塩酸1.0ミリメートル基準ゲル(Bio - Rad社)とクマシー染色したを使用してSDS - PAGE分析によって解決されます。広い範囲の分子量マーカー(バイオラッド)は、(左)が表示されます。
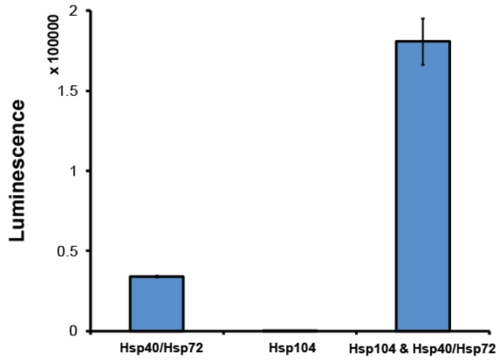
図6。ルシフェラーゼ再活性化アッセイ。変性ホタルルシフェラーゼ凝集体(50nmのは)25℃でHsp72及びHSP40(1μM)(アッセイデザインから両方)、Hsp104(量6μm単量体)またはHsp104、Hsp72と90分のためのHSP40のどちらかとインキュベートした変性ルシフェラーゼは、完全にHsp104とHsp40/Hsp72両方の存在下で再アクティブ化されます。回収された発光は、無限M1000プレートリーダー(テカン)で測定されます。値は平均± SEM(n = 3の)を表す。
ディスカッション
タイムライン:最大Hsp104の活動は私達が全体の精製スキームが可能な限り迅速に完了することをお勧めします。しかし、精製ステップの数は、必ずしも実用的ではないかもしれない厳しいスケジュールになります。精製工程は30℃インキュベーションの2-4時間まで、できるだけ早く一晩式の終わりから時間を実施している場合° TEVプロテアーゼとCは、約9〜11時間です。一時停止のひと?...
開示事項
利害の衝突は宣言されません。
謝辞
この作品は、NIH(5T32GM008275 - 22)とアメリカ心臓協会博士号を取得する前のフェローシップ(EASまで)からの助成金によって支えられて、化学、生物学のインターフェイスのNIH(2T32GM071339 - 06A1)(MEDまで)からフェローシップ、およびからの補助金NIH(1DP2OD002177 - 01とNS067354 - 0110)、エリソン医学財団、ビル&メリンダゲイツ財団(JSまで)。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
|---|---|---|---|
| BL21 - CodonPlus - RILコンピテントセル | ストラタジーン社、アジレントテクノロジー | 230255 | |
| 2XYTスープ | USB | 75864 | |
| 完全な、ミニ、EDTAフリープロテアーゼ阻害剤の錠剤 | ロッシュ | 1836170 | |
| ペプスタチン | シグマ | P4265 | |
| NI -セファロース6 Fast Flowは | GEヘルスケア | 17-5318-02 | |
| アミコンウルトラ- 15遠心フィルターユニット(分画分子量30,000) | ミリポア | UFC903008 | |
| 資源Q - 6ミリリットル列 | GEヘルスケア | 17-1179-01 | |
| proTEVプロテアーゼ | プロメガ | V6052 | |
| AcTEVプロテアーゼ | インビトロジェン | 12575015 | |
| スーパーローズ6 300分の10 GL | GEヘルスケア | 17-5172-01 | |
| HSP40 | アッセイデザイン | SPP - 400 | |
| Hsp72 | アッセイデザイン | ADI - NSP - 555 |
参考文献
- Erzberger, J. P., Berger, J. M. Evolutionary relationships and structural mechanisms of AAA+ proteins. Annu Rev Biophys Biomol Struct. 35, 93-114 (2006).
- Glover, J. R., Lindquist, S. Hsp104, Hsp70, and Hsp40: A Novel Chaperone System that Rescues Previously Aggregated Proteins. Cell. 94, 73-82 (1998).
- Parsell, D. A., Kowal, A. S., Singer, M. A., Lindquist, S. Protein disaggregation mediated by heat-shock protein Hsp104. Nature. 372, 475-478 (1994).
- Vashist, S., Cushman, M., Shorter, J. Applying Hsp104 to protein-misfolding disorders. Biochem Cell Biol. 88, 1-13 (2010).
- Parsell, D. A., Sanchez, Y., Stitzel, J. D., Lindquist, S. Hsp104 is a highly conserved protein with two essential nucleotide-binding sites. Nature. 353, 270-273 (1991).
- Doyle, S. M., Wickner, S. Hsp104 and ClpB: protein disaggregating machines. Trends Biochem Sci. 34, 40-48 (2009).
- Shorter, J. Hsp104: a weapon to combat diverse neurodegenerative disorders. Neurosignals. 16, 63-74 (2008).
- Glover, J. R., Lum, R. Remodeling of protein aggregates by Hsp104. Protein Pept Lett. 16, 587-597 (2009).
- Mogk, A., Haslberger, T., Tessarz, P., Bukau, B. Common and specific mechanisms of AAA+ proteins involved in protein quality control. Biochem Soc Trans. 36, 120-125 (2008).
- Grimminger-Marquardt, V., Lashuel, H. A. Structure and function of the molecular chaperone Hsp104 from yeast. Biopolymers. 93, 252-276 (2010).
- Sanchez, Y., Lindquist, S. L. HSP104 required for induced thermotolerance. Science. 248, 1112-1115 (1990).
- Sanchez, Y., Taulien, J., Borkovich, K. A., Lindquist, S. Hsp104 is required for tolerance to many forms of stress. Embo J. 11, 2357-2364 (1992).
- Alberti, S., Halfmann, R., King, O., Kapila, A., Lindquist, S. A systematic survey identifies prions and illuminates sequence features of prionogenic proteins. Cell. 137, 146-158 (2009).
- Halfmann, R., Alberti, S., Lindquist, S. Prions, protein homeostasis, and phenotypic diversity. Trends Cell Biol. 20, 125-1233 (2010).
- Shorter, J., Lindquist, S. Prions as adaptive conduits of memory and inheritance. Nat Rev Genet. 6, 435-450 (2005).
- True, H. L., Berlin, I., Lindquist, S. L. Epigenetic regulation of translation reveals hidden genetic variation to produce complex traits. Nature. 431, 184-187 (2004).
- True, H. L., Lindquist, S. L. A yeast prion provides a mechanism for genetic variation and phenotypic diversity. Nature. 407, 477-483 (2000).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6, e294-e294 (2008).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi+]. Science. 268, 880-884 (1995).
- Halfmann, R., Lindquist, S. Epigenetics in the extreme: prions and the inheritance of environmentally acquired traits. Science. 330, 629-632 (2010).
- Satpute-Krishnan, P., Langseth, S. X., Serio, T. R. Hsp104-dependent remodeling of prion complexes mediates protein-only inheritance. PLoS Biol. 5, e24-e24 (2007).
- Sweeny, E. A., Shorter, J. Prion proteostasis: Hsp104 meets its supporting cast. Prion. 2, 135-140 (2008).
- Lo Bianco, C. Hsp104 antagonizes alpha-synuclein aggregation and reduces dopaminergic degeneration in a rat model of Parkinson disease. J Clin Invest. 118, 3087-3097 (2008).
- Narayanan, S., Walter, S., Reif, B. Yeast prion-protein, sup35, fibril formation proceeds by addition and substraction of oligomers. Chembiochem. 7, 757-765 (2006).
- Savistchenko, J., Krzewska, J., Fay, N., Melki, R. Molecular chaperones and the assembly of the prion Ure2p in vitro. J Biol Chem. 283, 15732-15739 (2008).
- Shorter, J., Lindquist, S. Hsp104 catalyzes formation and elimination of self-replicating Sup35 prion conformers. Science. 304, 1793-1797 (2004).
- Shorter, J., Lindquist, S. Destruction or potentiation of different prions catalyzed by similar Hsp104 remodeling activities. Mol Cell. 23, 425-438 (2006).
- Shorter, J., Lindquist, S. Hsp104, Hsp70 and Hsp40 interplay regulates formation, growth and elimination of Sup35 prions. Embo J. 27, 2712-2724 (2008).
- Doyle, S. M. Asymmetric deceleration of ClpB or Hsp104 ATPase activity unleashes protein-remodeling activity. Nat Struct Mol Biol. 14, 114-122 (2007).
- Wendler, P. Atypical AAA+ subunit packing creates an expanded cavity for disaggregation by the protein-remodeling factor Hsp104. Cell. 131, 1366-1377 (2007).
- Hinault, M. P. Stable alpha-synuclein oligomers strongly inhibit chaperone activity of the Hsp70 system by weak interactions with J-domain co-chaperones. J Biol Chem. 285, 38173-38182 (2010).
- Tipton, K. A., Verges, K. J., Weissman, J. S. In vivo monitoring of the prion replication cycle reveals a critical role for Sis1 in delivering substrates to Hsp104. Mol Cell. 32, 584-591 (2008).
- Bieschke, J., Cohen, E., Murray, A., Dillin, A., Kelly, J. W. A kinetic assessment of the C. elegans amyloid disaggregation activity enables uncoupling of disassembly and proteolysis. Protein Sci1. 8, 2231-2241 (2009).
- Cohen, E., Bieschke, J., Perciavalle, R. M., Kelly, J. W., Dillin, A. Opposing activities protect against age-onset proteotoxicity. Science. 313, 1604-1610 (2006).
- Cohen, E. Reduced IGF-1 signaling delays age-associated proteotoxicity in mice. Cell. 139, 1157-1169 (2009).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 23, 1191-11201 (2010).
- Carmichael, J. Bacterial and yeast chaperones reduce both aggregate formation and cell death in mammalian cell models of Huntington's disease. Proc Natl Acad Sci. 97, 9701-9705 (2000).
- Vacher, C., Garcia-Oroz, L., Rubinsztein, D. C. Overexpression of yeast hsp104 reduces polyglutamine aggregation and prolongs survival of a transgenic mouse model of Huntington's disease. Hum Mol Genet. 14, 3425-3433 (2005).
- Liu, Y. H. Heat Shock Protein 104 Inhibited the Fibrillization of Prion Peptide 106-126 and Disassembled Prion peptide 106-126 Fibrils in vitro. Int J Biochem Cell Biol. , (2011).
- Wendler, P., Saibil, H. R. Cryo electron microscopy structures of Hsp100 proteins: crowbars in or out. Biochem Cell Biol. 88, 89-96 (2010).
- Wendler, P. Motor mechanism for protein threading through Hsp104. Mol Cell. 34, 81-92 (2009).
- Lee, S., Sielaff, B., Lee, J., Tsai, F. T. CryoEM structure of Hsp104 and its mechanistic implication for protein disaggregation. Proc Natl Acad Sci. 107, 8135-8140 (2010).
- Parsell, D. A., Kowal, A. S., Lindquist, S. Saccharomyces cerevisiae Hsp104 protein. Purification and characterization of ATP-induced structural changes. J Biol Chem. 269, 4480-4487 (1994).
- Schirmer, E. C., Queitsch, C., Kowal, A. S., Parsell, D. A., Lindquist, S. The ATPase activity of Hsp104, effects of environmental conditions and mutations. J Biol Chem. 273, 15546-15552 (1998).
- Schirmer, E. C., Ware, D. M., Queitsch, C., Kowal, A. S., Lindquist, S. L. Subunit interactions influence the biochemical and biological properties of Hsp104. Proc Natl Acad Sci. 98, 914-919 (2001).
- Hattendorf, D. A., Lindquist, S. L. Cooperative kinetics of both Hsp104 ATPase domains and interdomain communication revealed by AAA sensor-1 mutants. EMBO J. 21, 12-21 (2002).
- Schirmer, E. C., Lindquist, S. Purification and properties of Hsp104 from yeast. Methods Enzymol. 290, 430-444 (1998).
- Hattendorf, D. A., Lindquist, S. L. Analysis of the AAA sensor-2 motif in the C-terminal ATPase domain of Hsp104 with a site-specific fluorescent probe of nucleotide binding. Proc Natl Acad Sci. 99, 2732-2737 (2002).
- Lum, R., Niggemann, M., Glover, J. R. Peptide and protein binding in the axial channel of Hsp104. Insights into the mechanism of protein unfolding. J Biol Chem. 283, 30139-30150 (2008).
- Lum, R., Tkach, J. M., Vierling, E., Glover, J. R. Evidence for an unfolding/threading mechanism for protein disaggregation by Saccharomyces cerevisiae Hsp104. J Biol Chem. 279, 29139-29146 (2004).
- Tessarz, P., Mogk, A., Bukau, B. Substrate threading through the central pore of the Hsp104 chaperone as a common mechanism for protein disaggregation and prion propagation. Mol Microbiol. 68, 87-97 (2008).
- Weibezahn, J. Thermotolerance requires refolding of aggregated proteins by substrate translocation through the central pore of ClpB. Cell. 119, 653-665 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved