Method Article
Quantitative und automatisierte Hochdurchsatz-genomweiten RNAi-Screens in C elegans
In diesem Artikel
Zusammenfassung
Wir beschreiben ein Protokoll mit C elegans Und RNAi Fütterung Bibliotheken, die eine automatisierte Messung mehrerer Parameter, wie Fluoreszenz, Größe und die Trübung der einzelnen Würmern in einer Population erlaubt. Wir geben ein Beispiel eines Bildschirms, um Gene in Anti-Pilz-angeborenen Immunität beteiligt sind C elegans.
Zusammenfassung
RNA-Interferenz ist eine leistungsfähige Methode, um Genfunktionen zu verstehen, besonders wenn bei einer gesamten Genoms Maßstab und in einem quantitativen Rahmen durchgeführt. In C. elegans kann Genfunktion sich einfach und effizient durch Zuführen Würmer mit Bakterien Expression eines dsRNA entsprechend einer spezifischen Gen-1 geklopft. Während die Erstellung von Bibliotheken von RNAi-Klone für die meisten der C. elegans Genom 2,3 öffnete den Weg für echte funktionelle Genom-Studien (siehe z. B. 4-7), sind die meisten etablierten Methoden mühsam. Moy und Kollegen haben halbautomatische Protokolle, die Genom-weiten Screens erleichtern 8 entwickelt. Der Ansatz stützt sich auf mikroskopische Bildgebung und Bildanalyse.
Hier beschreiben wir ein alternatives Protokoll für eine Hochdurchsatz-Genom-weite Bildschirm auf Roboter-Handhabung von bakteriellen RNAi Klone, die quantitative Analyse unter Verwendung COPAS Biosort (Union Biometrica (UBI)) auf, und einintegrierte Software: Die MBioLIMS (Laboratory Information Management System von Modul-Bio) eine Technologie, die einen erhöhten Durchsatz für Datenmanagement und Sample Tracking bietet. Das Verfahren ermöglicht Bildschirme auf festem Medium-Platten durchgeführt werden. Dies ist besonders wichtig für einige Studien, wie jene Adressierung Wirt-Pathogen-Interaktionen in C. elegans, da bestimmte Mikroben nicht effizient infizieren sich Würmer in flüssiger Kultur.
Wir zeigen, wie das Verfahren verwendet werden, um die Bedeutung von Genen in Anti-Pilz-angeborenen Immunität bei C zu quantifizieren elegans. In diesem Fall setzt der Ansatz auf der Verwendung eines transgenen Stamm, der eine epidermale Infektion-induzierbaren fluoreszierenden Reporter-Gen mit dem GFP unter der Kontrolle des Promotors des antimikrobiellen Peptid-Gen nlp 29 und einem roten fluoreszierenden Reporter, die konstitutiv in das exprimierte Epidermis. Letzteres bietet ein internes Kontrollsystem für die funktionelle Integrität der Epidermisund unspezifische Transgen-Silencing 9. Als Kontrolle Würmer durch den Pilz infiziert sind sie fluoreszieren grün. Der Zuschlag von RNAi ein Gen für NLP-29 führt zu einer verminderten Expression Fluoreszenz nach der Infektion erforderlich. Derzeit können dieses Protokoll mehr als 3.000 RNAi Klone getestet und pro Woche analysiert werden, die Möglichkeit eröffnet, zum Screenen das gesamte Genom in weniger als 2 Monate.
Protokoll
Das Protokoll, wie unten beschrieben, wird in Schritten an fünf aufeinanderfolgenden Tagen (Abb. 1) unterteilt. Wie bereits erwähnt, können bestimmte Schritte an unterschiedlichen Tagen durchgeführt werden. Die Dauer der einzelnen Schritte, sowie die Menge an Material erforderlich (zB Würmer, Bakterien, Bilder) werden von der Anzahl der Platten mit 96 Vertiefungen pro Versuch der behandelt wird.
Tag 1
1. Herstellung der 96-Well-Platten NGM RNAi
Das folgende Protokoll wird aus dem Standard-Verfahren zur Herstellung Wurm Kultur Platten 10, die Details der Sterilisation Techniken für die verschiedenen Lösungen umfasst angepasst.
- Für 10-12 96-well Platten, Herstellung von 100 ml Nematodenwachstum Media (NGM) in deionisiertem H 2 O: 1,7 g Bactoagar, 0,29 g NaCl, 0,25 g Pepton, 100 ul Cholesterin (5 mg / ml in EtOH).
- Autoclave NGM (5 min bei 121 ° C für 100 ml; 30 min bei 121 ° C für 4 L) und abkühlen lassen, bis sie bei etwa 50 ° C (nur cool eno istugh zu halten). Es ist wichtig, dass die NGM warm genug, so dass es sich nicht verfestigt bleibt.
- Fügen Sie für 100 ml: 2,5 ml Phosphat-Puffer pH 6 (1M), 100 &mgr; l MgSO 4 (1M), 100 &mgr; l CaCl 2 (1M), 400 ul IPTG (1M), 100 ul Ampicillin (100 mg / ml), 100 &mgr; l Tetracyclin (12,5 mg / ml in EtOH).
- Verteilen Sie 75 &mgr; l der NGM in jede Vertiefung einer 96-Well Platte mit flachem Boden. Um Verfestigung des Mediums zu vermeiden, muss sie sich schnell abgegeben werden, es ist zweckmäßig, eine sich wiederholende Spender (zB Repeatman, Eppendorf) zu verwenden. Achten Sie darauf, dass sich keine Luftblasen in den Vertiefungen.
- Lagern Sie die Platten sofort in einer feuchten Kammer (z. B. eine Tupperware-Box mit feuchten Papiertüchern an der Unterseite) bei 4 ° C
Hinweis: Platten bis zu einer Woche im Voraus hergestellt und bei 4 ° C geeignet, kann die Organisation der nachfolgenden Schritte zu erleichtern.
2. Automatische Herstellung der 96-LB-Platten DeepWell
- Vorbereitung und Verteilung von 1,5 ml LB mit Ampicillin und Tetracyclin 100μg/mL 12,5 ug / ml in jede Vertiefung einer 96-Deepwell-Platte, die eine maximale Kapazität von 2 ml / Vertiefung hat. Der Vertrieb wird optimiert mit Hilfe eines Roboter-Liquid-Handling-System (zB TECAN) kann aber auch manuell bei Bedarf ausgeführt werden.
- Fügen Sie einen Deckel auf jeder Platte und lagern bei 4 ° C. Um jede Platte mit 96 Vertiefungen zu verfolgen, sollte eine eindeutige Markierung oder Barcode hinzugefügt, zu diesem Zeitpunkt oder danach werden.
Hinweis: Die Platten können unter 2 bis 3 Tage im Voraus erfolgen und bei 4 ° C; Das kann die Organisation der nachfolgenden Schritte leichter.
3. Wachsen die RNAi bakterielle Klone in den 96-DeepWell LB-Platten (Nacht-Kultur)
- Für die anschließende einfache Handhabung, wenn die ursprünglichen RNAi-Bibliothek Klone in einem 384-Well-Format, erste verteilen die Klone werden in etikettierte oder Standard-Barcodes versehen 96-Well-Platten mit LB mit 100 ug / ml Amp und 12,5 pg / mL Tet ergänzt mit Glycerin (10% Endkonzentration). Wenn der ursprüngliche 384-Well-Platten nicht Klone in alle Wells, wie es der Fall für die am häufigsten verwendeten "Ahringer" Bibliothek, gibt es mehrere Optionen für die Organisation der Tochterplatten. Klone werden kann, so dass es keine leeren Brunnen in den Tochter-Platten neu verteilt. Dies minimiert die Anzahl der Platten bis zum Bildschirm. Im Idealfall jedoch während der Replikation, sollte ein paar Brunnen auf jeder Platte Tochter leer gelassen werden, so dass diese anschließend kann mit Standard-Steuerung Klone (positiv und negativ), die über-Platte Vergleiche zu erleichtern gefüllt werden. Diese 96-Loch-Tochter Platten werden dann verwendet, um die Samen 96-DeepWell LB-Platten für eine Übernachtung (ON) Kultur.
Alle Liquid-Handling-Schritte werden am besten mit einem Roboter-Liquid-Handling-System (zB Tecan) geschehen, sondern kann manuell durchgeführt werden unter Verwendung von zum Beispiel eine 96-polige Replikator. Wenn die RNAi-Bibliothek Klone bereits in einer 96-Well-Format gehen auf 3,5 fort.
- Inkubieren der 96-Well-RNAi Tochter-Platten bei 37 ° C unter Rühren (200 UpM) für 14-15 Stunden.
- Stellen Sie sicher, dass die Bakterien richtig gewachsen und Klone zu identifizieren, die nicht wachsen wollte. Diese werden von späteren Analysen ausgeschlossen werden. Idealerweise wird die OD 600 in jeder Vertiefung gemessen, dieses kann durch einfache Inspektion durchgeführt werden.
- Lagern Sie die 96-Well-Platten RNAi-Tochter bei -80 ° C vor dem Gebrauch.
- Tauen Sie die 96-Well-Platten, die das RNAi-Klone bei Raumtemperatur. Die Platten können dann bei 4 ° C bis zur Verwendung aufbewahrt werden.
- Verteilen Sie 3 ul jeder Bakterien-Klon aus einer 96-Well-Platte zu replizieren, um die 96 Löcher des entsprechend beschrifteten / Strichcode 96-DeepWell LB-Platte mit 1,5 ml LB mit 100 ug / ml Amp und 12,5 pg / mL Tet. Bakterienklone RNAi Effizienz, wie gfp (RNAi) Klon in dem vorliegenden Beispiel, und negative Kontrollen, kann manuell in jedem verfügbaren leeren Vertiefung aufzunehmen.
- Decken Sie die 96-DeepWell pkorreliert mit einer Klebefolie (zB AeraSeal Zellkultur Film aus Dutscher). Ersetzen Sie die verbrauchte 96-Well-Platte replizieren bei -80 ° C
- Die 96-DeepWell markierten Platten werden dann ON bei 37 ° C unter Rühren (200 UpM für 14-15 Stunden).
Tag 2
4. Seed die RNAi Bakterienklonen auf die 96-Well-Platten NGM RNAi
Dieser Schritt sollte in der Früh nach dem auf die Kultur getan werden.
- Nehmen Sie die 96-Well-Platten NGM von 4 ° C und lassen Sie sie aufzuwärmen und für 5-15 min unter einer sterilen Laminar Flow Kabinett trocken. Lassen Sie die Platten zu lange unter der Haube (max. 30 min), da sonst der NGM kann in der Folge zu knacken. Fügen Sie den entsprechenden Etiketten / Barcode auf jeder Platte.
- Rufen Sie die 96-DeepWell auf Kulturplatten von 37 ° C. Zeichnen Sie die Positionen von allen leeren Brunnen (ein leerer Brunnen ist komplett durchsichtig), diese werden von weiteren Analysen ausgeschlossen werden.
- Zentrifugieren 96DeepWell-Platten 5 min bei 4000 Umdrehungen pro Minute.
- Tipp aus dem Überstand durch Drehen der Platte scharf auf den Kopf und trocknen Sie die Kanten der Platte schnell auf ein Papiertuch. Da die Kulturen von rekombinanten Bakterien sind, muss der Überstand, in Übereinstimmung mit den örtlichen Vorschriften (zB autoklaviert) behandelt werden.
- Resuspendieren des bakteriellen Pellets in der Restflüssigkeit (etwa 50 ml) durch Vortexen, idealerweise unter Verwendung eines dedizierten Rührwerk (zB Tecan), so daß jede Platte genau die gleiche Behandlung erhält. Es ist möglich, die OD 600 von jeder Vertiefung genau die bakteriellen Inokulum für den nachfolgenden Schritt zu standardisieren zu messen, aber dies ist nicht unbedingt erforderlich, und wir nicht, eine solche Prüfung durchzuführen.
- Übertragen Sie 5 ul der resuspendierten Bakterien auf den 96-Well-Platten NGM mit einem 8 oder 12-Kanal-Multi-Pipette. Seien Sie vorsichtig, nicht zu berühren oder durchdringen die NGM. Dieser Schritt wird manuell durchgeführt, kann aber unter Verwendung eines optimierten Roboter wie zum Beispiel der Liquidator96 (Rainin) werden, die ermöglicht dieVerteilung von 96 RNAi Klone in einem Schritt.
- Lassen Sie sich die Bakterien trocken unter einer sterilen Laminar Flow Kabinett, regelmäßig zu kontrollieren, um zu vermeiden das NGM zu trocken. Dieser Schritt dauert etwa 2 Stunden.
- Inkubieren Sie die 96-Loch-RNAi-NGM-Platten bei 37 ° C auf in einer feuchten Kammer.
5. Bereiten Sie einen synchronisierten Population von Würmern
Für diesen Schritt verwenden wir transgene Würmer Tragen des Reporter-Gen (e) von Interesse (zB eine Infektion induzierbare p NLP-29 :: GFP-Konstrukt und als interne Kontrolle, konstruieren ein konstitutives p col-12 :: dsRed Transgen) . Aufgrund der beobachteten Abnahme der Transgenexpression mit der Zeit, tauen wir frische Chargen von Würmern alle 6 Wochen. Wir Kultur Würmer für mindestens 2 Generationen vor dem Gebrauch, und sicherzustellen, dass die Würmer noch nie vor dem Test verhungert. Wenn Tag 2 des Protokolls ist Dienstag, bereiten wir am Freitag, Würmer, indem man 30 junge erwachsene Würmer auf einem 9 cm NGM Platte mit OP50 Bakterien verbreitenbei 20 ° C, sie werden bereit sein, Bleiche am Dienstag (siehe Tabelle 1).
- Bleach Würmer nach dem Standardprotokoll 10.
- Lassen Sie die Eier brüten in M9 bei 25 ° C auf unter Rühren zu einer synchronisierten Population von L1-Larven zu bekommen.
Tag 3
6. Verteilen Sie die Würmer auf den 96-Loch-RNAi-NGM Platten für die Fütterung und RNAi
Dieser Schritt sollte morgens erfolgen. L1-Phase synchronisiert Würmer werden auf jeden 96-Loch-Bakterien-Klon für die Fütterung und RNAi verteilt.
- Rufen Sie die 96-Loch-RNAi-NGM-Platten von 37 ° C und lassen sie bei Raumtemperatur abkühlen lassen.
- Rufen Sie die gebleichten Würmer von 25 ° C. Schätzen Sie die Anzahl der Würmer pro 2 ul und stellen Sie mit M9 auf rund 100 bis 120 Würmer pro 2 ul (ungefähr ein einziger Tropfen) haben.
- Verteilen Sie manuell 2 ul der L1-Phase Würmer in jede Vertiefung mit einem sich wiederholenden Spender. Überprüfen Sie jede gut für Würmer (Würmer, sollten Sie schwimmen in Flüssigkeit zu sehen).
- Lassen Sie die Platten trocken unter einer sterilen Laminar-Flow-Schrank (max. 1 Stunde). Überprüfen Sie die Platten regelmäßig; die Würmer kriechen muss und nicht schwimmen. Achten Sie darauf, die NGM darf nicht austrocknen zu viel, sonst wird es zu knacken.
- Legen Sie die Platten bei 25 ° C in einer feuchten Kammer.
7. Testen Sie die Drechmeria coniospora Sporen für eine effektive Infektion
Dieser Schritt ist spezifisch für das vorliegende Bild. Es besteht in der Prüfung verschiedener Chargen von Sporen zu hoch infektiöse diejenigen auszuwählen. Daher sollte so nah wie möglich vor dem Tag 4, Tag der Infektion durchgeführt werden. Es ist notwendig, um eine ausreichende L3-L4 Würmer im Voraus vorbereiten für diese Tests (Tabelle 1). Die Einzelheiten der Verfahren für D. erforderlich coniospora Kultur wurden an anderer Stelle 11 beschrieben.
- Sammeln von Sporen aus einer Probe jeder Pilzkultur in einem kleinen Volumen von M9.
- Geben Sie einen Tropfen jeder Sporensuspension auf einer 35 mm Platte mit NGM OP50 ausgesät, und trocknen lassen.
- In 30-40 L3-L4 Würmer Tragen des Reporter-Gen von Interesse (z. B. p NLP-29 :: GFP).
- Legen Sie die infizierten Platten bei 25 ° C auf.
- Am nächsten Tag, überprüfen Sie die Würmer für GFP Induktion und wählen Sie die Partie von Pilz, der die hellste, breiteste und homogenen Induktion der grünen Fluoreszenz gibt.
Tag 4
8. Infizieren die RNAi-exponierten Würmer mit D. coniospora Sporen
Dieser Schritt wird 30 Stunden nach RNAi-Zuführung, wenn Würmer haben die L3-L4 Stadium erreicht durchgeführt.
- Bestimmen Sie das Volumen der Sporen zu verwenden, um 4 ul pro Well zu verteilen.
- Sammeln Sie frische D. coniospora Sporen aus dem gewählten Ansatz mit M9-Puffer. Verwenden Sie 8-10 ml M9 für ein 9 cm Platte von Sporen.
- Verteilen 4 ul Sporen in jede Vertiefung mit einer sich wiederholenden Spender. Überprüfen Sie jede gut für Sporen (Sie können die Würmer schwimmen in Flüssigkeit zu sehen).
- Lassen Sie die Platten trocken unter einer sterilen Laminar-Flow-Schrank (~ 1 Stunde). Überprüfen Sie regelmäßig die Platten, bis die Würmer krabbeln beginnen. Achten Sie darauf, die NGM darf nicht austrocknen zu viel, sonst wird es zu knacken.
- Legen Sie die infizierten Platten bei 25 ° C in einer feuchten Kammer.
Tag 5
9. Beobachtung und Speicherung der Platten vor der maschinellen quantitative Analyse
Dieser Schritt beginnt 18 Stunden nach der Infektion und wird während Tag 5 durchgeführt. Worms wird erwartet, dass junge Erwachsene beginnen, Eier zu legen sein. Während dieses Schritts wird der Phänotyp visuell bewertet: entweder Würmern fluoreszieren Grün, das mit einer normalen Situation nach der Infektion entspricht, oder die Induktion der GFP in einem gegebenen auch geändert, was bedeutet, dass die stillgelegte Gen verändert die Reaktion auf eine Infektion.
- Schnell beachten Sie die Platten unter dem Fluoreszenz-Mikroskop seziert. Wenn die Platten eine gute allgemeine GFP-Induktion zeigen dann mit dem nächsten Schritt fortfahren, sonst bis zum Nachmittag warten, für eine bessere Induktion, solange es noch Futter für die Würmer verlassen.
- Optisch punkten jede Platte und achten Sie auch mit einer deutlichen Phänotyp (z. B. keine Expression von GFP). Durch die Überprüfung der Ergebnisse mit allen Steuerung RNAi-Klone, die zugesetzt wurden, darf erhalten haben, gibt dieser erste vorläufige Analyse einen Hinweis, ob das Experiment erfolgreich war.
- Mit einem 8 oder 12-Kanal Pipette Multi Transfer die Würmer mit 100 ul 50 mM NaCl-Triton 0,05%, um ein neues entsprechend beschrifteten / Strichcode 96-Well-Rundboden-Platte. Frieren Sie die Platten bei -80 ° C bis zur Analyse.
- Am Tag der Analyse, tauen die Platten bei Raumtemperatur und fahren Sie mit der automatisierten Analyse unter Verwendung des COPAS Biosort. Der Sorter analysiert eine einzelne Platte in 22 Minuten. Die Platten sollten nichtlinks bei Raumtemperatur für mehr als eine Stunde vor der Analyse, und sie können nicht wieder eingefroren werden.
- Die Informationen aus dem COPAS Biosort erhalten werden, in eine Excel-Datei für oberflächliche Überprüfung der Datenqualität transkribiert. Die Rohdaten mit den verschiedenen Parametern, die der COPAS Biosort (optische Dichte (Extinktion), axiale Länge (time of flight), Fluoreszenz-Emissionen, gleichzeitig drei verschiedene Detektoren) gemessen werden dann in einem eigenen Paket LIMS (MBio LIMS, gelagert Modul- Bio) für die nachfolgende detaillierte quantitative Analysen.
10. Repräsentative Ergebnisse
Wenn die Mengen an Würmern und Bakterien oben angegebenen, am Ende des Experiments, in den meisten Vertiefungen, sollte die Tiere alle bis zum Erwachsenenalter entwickelt und es noch immer einige Lebensmittel in allen Vertiefungen verbleiben. Unter diesen Bedingungen sollten die meisten Brunnen enthalten auch Eier. Es sollte eine ausreichende Anzahl von Erwachsenen in jeder Vertiefung, so dass Daten Biosort f erhalten werdenoder mindestens 50 einzelnen Würmer. Auf der anderen Seite, wenn der RNAi ist effizient, wird eine große Anzahl von RNAi Klone provozieren sichtbaren Phänotyp. Zum Beispiel können Würmer sein, unkoordiniert, oder mehr offensichtlich, steril, oder verhaftet in ihrer Entwicklung. Dies sollte häufig auftreten, so daß im allgemeinen mindestens eine Vertiefung pro 96-Well-Platte Schnecken mit einem offensichtlichen RNAi Phänotyp enthält. Wenn der Phänotyp von Interesse die Expression eines GFP-Reporter-Gen ist, bereitzustellen Vertiefungen mit einem GFP-(RNAi) Klon eine robuste Steuerung (Abb. 2). Andere Phänotypen, wie eine Änderung der Größe leicht mit dem Biosort (3A) gemessen werden. Wir beobachteten, dass Würmer könnten auf benachbarte Brunnen bei sehr niedrigen Frequenzen (<1%, BS, unveröffentlichte Ergebnisse) migrieren, wenn das Essen in einem gut ausgehen wollte.
Der Zuschlag die Funktion der verschiedenen Klassen von Genen beeinflussen können Transgene Expression in C. elegans. Einige sind nicht spezifisch für Transgene Expression 12 erforderlich. Andere, wieals Gewebe-spezifische Transkriptionsfaktoren, z. B. die epidermale GATA-3-Faktor ELT 13 wird insbesondere auf Teilmengen von Zellen erforderlich ist. Dies ist einer der Gründe für die Aufnahme von einem nicht verwandten und konstitutive epidermale Transgen-Reporter-Konstrukt (p col-12 :: dsRed) in dem Stamm für die aktuelle Protokoll verwendet. Dies ermöglicht solche Gene aus den ausdrücklich in der Gen-regulatorischen Verfahren untersucht (3B und C) beteiligt zu unterscheiden. Das Transgen wird in allen Larvenstadien exprimiert. Wenn ein Test ein Erfassen Genexpression in Embryonen eine andere Kontroll-Reporter-Gen erforderlich wäre.
Eine der Neuerungen dieses Protokolls ist die Verwendung von Gefrierplatten, ihre Analyse zu verschieben. Einfrieren erlaubt Platten gelagert werden bis zu zwei Wochen ohne wesentliche Veränderung der Ergebnisse. Obwohl die absoluten Werte der gemessenen Fluoreszenz kann geändert werden, bleiben ihre relativen Verhältnisse ähnlich (Abb. 4). Auf der OTihre Hand, ist RNAi eine eigensichere variablen experimentellen Verfahren 14. Um robuste Ergebnisse zu erzielen und die Beseitigung der vielen möglichen False Positives, müssen RNAi-Screens, die repliziert, mindestens einmal werden. Wenn die Versuche erfolgreich durchgeführt werden, sollte es eine vernünftige Korrelation zwischen den Ergebnissen von einer Platte auf verschiedenen Tagen getestet werden. Insbesondere sollten die Gene, die eine starke Wirkung haben eindeutig identifizierbar sein, auch wenn ihre genaue quantitative Wirkung nicht identisch ist zwischen doppelten Platten (Abb. 5).
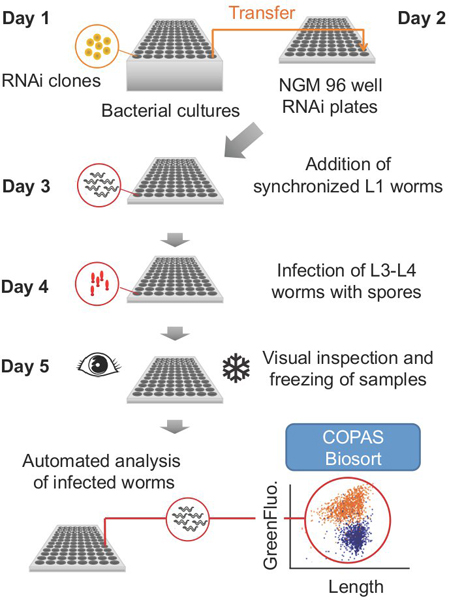
Abbildung 1. Gesamtkonzept für ein typisches Experiment.

Abbildung 2. Control-RNAi-Targeting mit einem Klon GFP-Expression. Transgene Würmer expressing konstitutiv p col-12 :: dsRed Reporter und ein induzierbarer p nlp 29 :: GFP-Reporter auf festem Medium in einer 96-Well-Platte visualisiert unter Verwendung eines GFP Präpariermikroskop mit einem Filterset die gleichzeitige Beobachtung der roten und grünen Fluoreszenz. Die Würmer wurden ausgesetzt steuern (obere Felder) oder ein GFP RNAi-Klon (untere Felder) für 48 h. Die Platten auf der linken Seite sind nicht infizierten Würmer, die auf der rechten Würmer 18 Stunden nach der Infektion mit D. coniospora. Der Maßstab beträgt 1 mm.

Abbildung 3. Die quantitative Analyse einer 96-Well-Platte. Die COPAS Biosort erzeugt numerische Daten für jeden Wurm analysiert. Die Kurven zeigen den Durchschnitt und die Standardabweichung für die Flugzeit (TOF, A), die der Größe der Würmer 15, rote Fluoreszenz (B) und das Verhältnis (X100) der grünen Fluoreszenz der TOF (C) von transgenen r entsprichtrms exprimieren konstitutiv p col-12 :: dsRed Reporter und ein induzierbarer p nlp 29 :: GFP-Reporter, die auf festen Medium in einer 96-Well-Platte kultiviert war mit unterschiedlichen RNAi Klone für 48 h, 18 h nach der Infektion mit D. coniospora. Bestimmte Klone, wie mit dem Pfeil in (A), wirken zu provozieren Wachstumsstörungen und / oder die Expression von P col-12 :: dsRed hervorgehoben. Andere, spezifisch die GFP-Expression, wie durch den Pfeil in (C) markiert. Diese besondere Klon richtet das Gen NSY 1, die zuvor bereits für die Regulierung der NLP 29 13 wichtig. Grüne, rote Fluoreszenz und TOF sind in beliebiger konstanter Einheiten gemessen.
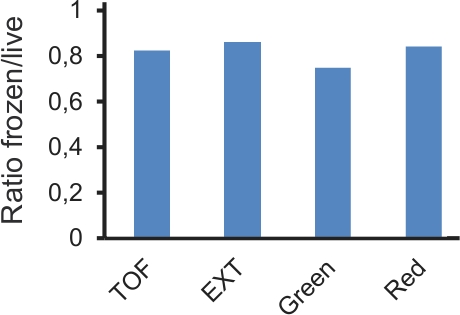
Abbildung 4. CompArison von Daten mit Live-und gefrorenen Proben aus dem gleichen Experiment erhalten. Platten von L4 Würmer Durchführung p NLP-29 :: GFP und p col-12 :: dsRed Transgene wurden entweder mit D. infiziert coniospora oder nicht. Nach 18h, wurde jede Platte in etwa 2 geteilt, die Hälfte wurde sofort analysiert Hälfte bei -80 ° C für 24 Stunden eingefroren, vor dem Auftauen und Analyse. Die Werte für die durchschnittliche TOF (Größe der Würmer), EXT (optische Dichte) und grüne und rote Fluoreszenz wurden für jede Probe berechnet. Die Grafik zeigt das Verhältnis des Durchschnitts infiziert geteilt durch nicht-infizierten Proben, für die Frost-und Lebendfutter Proben (n = 7638 und 5096, 8634 und 3850 und Würmer, jeweils).

Abbildung 5. Vergleichende Analyse von doppelten Platten mit 96 Vertiefungen. Die normalisierte Fluoreszenz-Verhältnis (die mittlere Fluoreszenz-Ratio (hier, 100 * grün / TOF) für jeden gut aufgeteiltdie getrimmt (25. bis 75. Perzentil) bedeuten für jede Platte) für jeden gut aus zweifache Untersuchungen von einer repräsentativen 96-Well Platte.
| Material | Experimentelle Timeline |
| Donnerstag
|
Tabelle 1. Beispiel für eine ein-Cycle-Experiment mit 30 Platten.
Hier wird das Material für eine ein-Cycle-Experiment von 30 Platten und einer Zeitleiste für ein Experiment von Stufe 1 bis 12 auf 9 Tage organisiert Schritt benötigt vorgestellt.
Nematodenwachstum Media (NGM), 100 ml:
| 0.3 g | NaCl |
| 0,25 g | Bactopepton |
| 2 g | Bactoagar |
| 100 ul | 5 mg / ml Cholesterin in EtOH |
| In entionisiertem Wasser auf 100 ml | |
| Autoklaven für 5 Minuten bei 121 ° C und abkühlen lassen, dann zufügen: | |
| 100 ul | 1 M MgSO 4 |
| 100 ul | 1 M CaCl 2 |
| 2,5 ml | 1 M KPO 4 pH 6,0 |
NGM für RNAi-Behandlung in 96-Well-Platte wurden 100 ml:
| 0,29 g | NaCl |
| 0,25 g | Bactopepton |
| 1,7 g | Bactoagar |
| 100 ul | 5 mg / ml Cholesterin in EtOH |
| In de ionisiertes Wasser auf 100 ml | |
| Autoklaven für 5 Minuten bei 121 ° C und abkühlen lassen, dann zufügen: | |
| 100 ul | 1 M MgSO 4 |
| 100 ul | 1 M CaCl 2 |
| 2,5 ml | 1 M KPO 4 pH 6,0 |
| 400 ul | 1 M IPTG |
| 100 ul | 100 mg / ml Ampicillin |
| 100 ul | 12,5 mg / ml Tetracyclin |
Bleichmittel-Lösung, 5 ml:
2,5 ml H 2 O
2,3 ml Bleach
0,2 ml NaOH 50%
NaOH 50%, 100 ml:
50 g NaOH
In entionisiertem Wasser auf 100 ml
NaCl 50 mM-Triton 0,05%, 400 ml:
4 ml 5 M NaCl
1 ml 20% Triton
In entionisiertem Wasser auf 400 ml
2 ml Triton X-100
8 ml VE-Wasser
M9-Puffer, 1L
6 g Na 2 HPO 4 (MG: 178)
3 g KH 2 PO 4 (MG: 136)
5 g NaCl
In deionisiertem Wasser auf 1 L
Autoklavieren
1 ml 1 M MgSO 4
Luria Broth (LB), 1L:
10 g Bactotrypton
20 g Hefe-Extrakt
10 g NaCl
In deionisiertem Wasser auf 1 L
Autoklavieren
1 M MgSO 4, 300 ml:
73,95 g MgSO 4
In entionisiertem Wasser auf 300 ml
Autoklavieren und bei Raumtemperatur lagern
5 mg / ml Cholesterin, 200 ml:
1 g Cholesterin
Fügen Sie 100% EtOH und 200 ml
Filtern sterilisieren und bei Raumtemperatur lagern
1 M CaCl 2, 500 ml:
27.75 g CaCl 2
In entionisiertem Wasser auf 500 ml
Filtern sterilisieren und bei Raumtemperatur lagern
1 M KPO 4, pH 6,0, 4 L:
517 g KH 2 PO 4 (MG: 136)
207 g K 2 HPO 4 (MG: 174)
In entionisiertem Wasser zu 4 l
100 mg / ml Ampicillin (Amp), 10 ml:
1 g Ampicillin
In entionisiertem Wasser auf 10 ml
Lagerung bei -20 ° C
1 M Isopropyl β-D-thiogalactopyranosid (IPTG), 10 ml:
2,38 g IPTG
In entionisiertem Wasser auf 10 ml
Lagerung bei -20 ° C
12,5 mg / ml Tetracyclin, 100 ml
1,25 g Tetracyclin
Fügen Sie 100% EtOH und 100 ml
Tabelle 2. Solutions.
Diskussion
Das aktuelle Protokoll ermöglicht großflächige RNAi-Screening in C elegans Auf festen Medien, unter Verwendung des Niveaus der Expression von einem fluoreszierenden Reporter-Gen als ein Auslesen. Es wurde mit automatisierten Tools optimiert worden, um Hochdurchsatz-Assays ermöglichen, was eine einfache und schnelle Handhabung einer großen Zahl von Proben, in einer effizienten, zuverlässigen und reproduzierbaren Art und Weise. Andere Methoden sind beschrieben worden, dass nicht so stark auf automatisierte Handhabung flüssige Probe 8.
Ein wichtiger organisatorischer Aspekt des Protokolls war die Entwicklung einer robusten Methode der Probe-Tracking durch die Anpassung der kommerziellen MBioLIMS Software zur RNAi-Screen Verfahren in Zusammenarbeit mit der Firma ModulBio. In jedem Stadium des Verfahrens werden die Platten nun von Barcodes innerhalb des LIMS generiert identifiziert. Da die LIMS zeichnet auch die Identität der Klone in jeder Vertiefung auf jeder Platte, und zwar von gut putativen Gens ist einfach. Und darüber hinaus, so dass die LIMS-Anbindung an die Datenbank der Gemeinschaft WormBase, direkter Zugang zum molekularen und funktionellen Daten über ein bestimmtes Gen. Das LIMS ermöglicht auch Analysen auf der Platte oder sogar Chromosom und des gesamten Genoms Ebene geführt werden. Für diese Analysen werden die Daten in einer Weise, die vollständig kompatibel mit zuvor beschriebenen Instrumente wie COPAmulti ist gespeicherten 16.
Ein kritischer Punkt in der experimentellen Verfahren ist die Einstellung der Menge des Lebensmittels relativ zu der Anzahl von Würmern. Für viele Tests ist es wichtig, dass Würmer nicht am Ende des Experiments verhungern aber es ist auch wichtig, nicht zu viel zu essen zu Beginn haben. Wir haben beobachtet, dass eine hohe Dichte von Lebensmitteln Fluoreszenzinduktion abnimmt in dem Protokoll mit Schnecken tragenden p NLP-29 :: GFP-Reporter. Dies könnte auf eine verminderte Effizienz Infektion, wenn Würmer in zu viel Bakterien kultiviert werden (BS, unveröffentlichte Ergebnisse). Gleichzeitig ist die Kulturbedingungen der RNAi-Bakterien auch wichtig, da sie bestimmt, ob dsRNA auf einem optimalen Niveau erzeugt wird. Einige Protokolle für eine groß angelegte RNAi erfordern Kultur der RNAi-Klone auf Agarplatten vor Aussaat Flüssigkulturen 8. Wir erhalten robuste Gen-Silencing Weglassen diesen Schritt. Wir entschieden uns für den Anbau eines Überschusses von jeder Kultur, um eine homogene Ergebnisse zu gewährleisten. Das in der Tat beschrieben wird, liefert genügend Material Anspruch 2 oder 3 Bildschirme parallel durchzuführen. Im Interesse der Erlangung der robustesten Ergebnisse bevorzugen wir jedoch, die Durchführung von Bildschirmen in zweifacher Ausfertigung mit unabhängigen Kulturen von Würmern, RNAi-Bakterien und Pilzsporen.
Ein weiterer kritischer Schritt ist die Verwendung von Medium, das weder zu feucht noch zu trocken ist, verwandt. Wenn die Platten nicht trocken genug sind, wird die RNAi-Bakterien nicht richtig trocknen und Würmer werden während des Tests zu schwimmen. Dies kann Probleme verursachen. Zum Beispiel werden Würmer nicht effizient infiziert D. coniospora In Flüssigkeit (C Couillault, unveröffentlichte Ergebnisse). Auf der anderen Seite, auf trockener Platten, die Osmolarität der Kulturmedium erhöht. Dies hat direkte Auswirkungen auf Wurm Physiologie 17, Einschließlich der Induktion von einigen antimikrobiellen Peptid-Gene, wie z. B. NLP 29 18. Das Volumen verwendet, um Würmer und Sporen zu verzichten ist entscheidend für die effektive und schnelle Trocknung zu ermöglichen. Wenn mehr als 10 l Flüssigkeit auf frische Platten hinzugefügt wird, können die Brunnen viele Stunden dauern, um zu trocknen. Dies kann bedeuten, dass es schwierig, Roboter-Methoden verwenden, um Würmer zu den Platten hinzuzufügen ist. Zum Beispiel wird jeder Wurm durch die Biosort in der Umgebung von 1 ul Flüssigkeit abgegeben. Aktuell vertreiben wir die Würmer manuell als dass erlaubt uns auch, die Tendenz von Würmern in der Lösung in das Sediment zu überwinden.
Wir zuvor berichtet eine Anpassung der Biosort das erlaubt eine 96-Well-Platte auf in 36 Minuten analysiert werden 19. Die Maschinenhersteller UBI nun eine Änderung der Biosort zur weiteren Verringerung der Messzeit. Mit dem "FastReFlex", werden die Proben direkt durch die Flußzelle zur Analyse weitergeleitet, unter Umgehung des Bubble-Sperrfilter. Dies führt zu einer erheblichen Verringerung der Zeit für die Datenerfassung, eine gesamte Platte kann in 22 min ermöglicht etwa 20 Platten pro Tag behandelt werden analysiert werden. Wenn die Platten nicht eingefroren wurden, jedoch würde es eine 8-Stunden-Intervall zwischen dem ersten und letzten sein. Angesichts der relativ kurzen Dauer des Experiments, mit der Infektion dauert nur 18 Stunden, wäre dies eine unzulässige Variable über Platten einzuführen. Nicht nur, dass das Einfrieren Platten vor der Analyse dieses Problem lösen, aber es ist auch wichtig, im Rahmen der High-Throughput-Analysen, mit dem aktuellen Protokoll und Standard-Liquid-Handling-Roboter, 2 Personen problemlos verarbeiten kann bis zu 50 Platten pro Woche. Derzeit haben wir routinemäßig Bildschirm 30 Platten der Woche, die einen gesamten Genoms Bildschirm ermöglichtauf einmal in weniger als 2 Monate durchgeführt werden.
Ein interessantes Merkmal dieses Protokolls ist die Tatsache, dass es feste Agar verwendet, so dass Tests, die nicht in flüssiger Kultur durchgeführt werden können. Als Wurm Physiologie unterscheidet sich in Abhängigkeit von den Kulturbedingungen 20,21, Leistung bringen zu können Assays, die auf einem festen Medium kann auch ergänzen üblichen Flüssig-Bildschirme. Für bestimmte Phänotypen (zB Bewegung), kann die Analyse dann direkt mit 96-Well-Platten festen Medium durchgeführt. Für den aktuellen Bildschirm jedoch als Biosort kann nicht von einem festen Medium Platten Probe werden Würmer erste Flüssigkeit vor dem Einfrieren zu übertragen. Auf der anderen Seite ermöglicht die COPAS Biosort eine Multiparameter quantitative Analyse mit der Messung der Vielzahl von Phänotypen. Es ist wahrscheinlich, dass für einen bestimmten Bildschirm nicht alle gemessenen Parameter wird die primäre Auslesen sein. Dennoch können diese Messungen aus verschiedenen Gründen nützlich. Sie ermöglichen, zu bestimmen, ob der Assay richtig ausgeführt. Zum Beispiel kann man auf der Anzahl und Größe von Würmern in jeder Vertiefung zu überprüfen. Nur dort, wo ein RNAi-Klon soll Wachstum oder Fortpflanzung blockieren sollte es eine Abweichung von den erwarteten Werten sein. Wells mit weniger als 20 Würmer oder / und Tiere 30% kleiner (TOF) mit einem Standard verglichendard Kontrolle ohne Auswirkungen auf das Wachstum ( ZB GFP (RNAi)) Kann von weiteren Analysen ausgeschlossen werden. Die Funktion dieser Klone können adressiert mit einer alternativen Protokoll, wobei Würmer zur RNAi-Bakterien ausgesetzt sind, werden nach ihrer Entwicklung ist weit fortgeschritten 15. Die verschiedenen Parameter können auch verwendet werden, um Kandidaten-Gen (HIT) Auswahl, zum Beispiel zu verfeinern, die Begrenzung Treffern nur jenen, die grüne Fluoreszenz, aber nicht rot zu beeinflussen. Die genaue Methode zur Auswahl gewählt wird, gespielt von der Art des Bildschirms und sein genaue Auslesen ab. Angesichts der Komplexität der Daten aus einer quantitativen genomweiten Screen erzeugt, kann die Zusammenarbeit mit einem Statistiker erforderlich sein.
Das Protokoll beschreiben wir können leicht auf andere Arten von Tests angepasst werden. Zum Beispiel, an der Infektionsstelle Schritt D. coniospora Konnte von keinem anderen Erreger in der Lage, auf festen Medien infizieren ersetzt werden. Dies ist möglicherweise sinnvoll, da wir festgestellt, dass viele der Virulenzfaktoren wichtig für Serratia marcescens-Infektion in flüssiger Kultur spielen keine Rolle bei der Infektion auf festem Medium (E. Pradel, persönliche Mitteilung) haben. Das Protokoll ist auch vollständig kompatibel mit Tests von Chemikalien Verbindungen 22-24, Oder um Verbindungen mit Bioaktivität mit verschiedenen Bibliotheken von Bakterien identifizieren 25, So sollten Sie einen allgemeinen Nutzen in der C elegans Forschungs-Community.
Offenlegungen
Das Labor arbeitet mit Ewbank Union Biometrica und Modul-Bio.
Danksagungen
Wir danken D. Braendle, CL Kurz und F. Montañana-Sanchis für ihre Beiträge. Dieses Projekt wurde durch Zuschüsse aus dem ANR und dem Conseil Regional PACA und institutionelle Förderung von INSERM und CNRS unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Bactoagar | BD Diagnostic Systems | 214010 | |
| Ampicillin | Sigma-Aldrich | A9518 | |
| Bactopepton | BD Diagnostic Systems | 211677 | |
| CaCl 2 | Jeder Lieferant | ||
| AeraSeal Klebefolie | Dutscher | 760214 | |
| Isopropyl β-D-thiogalactopyranosid (IPTG) | Thermo Scientific | AB-0481 | |
| K 2 HPO 4 | Jeder Lieferant | ||
| KH 2 PO 4 | Jeder Lieferant | ||
| MgSO 4 | Jeder Lieferant | ||
| NaCl | Jeder Lieferant | ||
| Na 2 HPO 4 | Jeder Lieferant | ||
| NaOH | Jeder Lieferant | ||
| 96-well-Platte | Thermo Scientific | AB-0932 | |
| 96-Well-Platte flach | FALCON | 353072 | |
| 96-Loch-Runde Platte | FALCON | 353077 | |
| Tetracycline | Sigma-Aldrich | T8032 | |
| Triton X-100 | Jeder Lieferant | ||
| TECAN Roboter | TECAN | ||
| Liquidator96 TM | RAININ | ||
| COPAS Biosort | Union Biometrica | ||
| LIMS | Modul-Bio |
Referenzen
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854-854 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Rual, J. F. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14, 2162-2168 (2004).
- Lee, S. S. A systematic RNAi screen identifies a critical role for mitochondria in C. elegans longevity. Nat. Genet. 33, 40-48 (2003).
- Hamilton, B. A systematic RNAi screen for longevity genes in C. elegans. Genes Dev. 19, 1544-1555 (2005).
- Frand, A. R., Russel, S., Ruvkun, G. Functional genomic analysis of C. elegans molting. PLoS Biol. 3, e312-e312 (2005).
- Parry, D. H., Xu, J., Ruvkun, G. A whole-genome RNAi Screen for C. elegans miRNA pathway genes. Curr. Biol. 17, 2013-2022 (2007).
- O'Rourke, E. J., Conery, A. L., Moy, T. I. Whole-animal high-throughput screens: the C elegans model. Methods Mol. Biol. 486, 57-75 (2009).
- Pujol, N. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Curr. Biol. 18, 481-489 (2008).
- Stiernagle, T. The C. elegans Research Community. Maintenance of C. elegans. WormBook. , 1551-8507 (2006).
- Powell, J. R., Ausubel, F. M. Models of Caenorhabditis elegans Infection by Bacterial and Fungal Pathogens. Methods Mol Biol. Ewbank, J., Vivier, E. 415, Humana Press. 403-427 (2008).
- Cardoso, C. XNP-1/ATR-X acts with RB, HP1 and the NuRD complex during larval development in C. elegans. Dev. Biol. 278, 49-59 (2005).
- Pujol, N. Anti-fungal innate immunity in C. elegans is enhanced by evolutionary diversification of antimicrobial peptides. PLoS Pathog. 4, e1000105-e1000105 (2008).
- Simmer, F. Genome-Wide RNAi of C. elegans Using the Hypersensitive rrf-3 Strain Reveals Novel Gene Functions. PLoS Biol. 1, e12-e12 (2003).
- Couillault, C. TLR-independent control of innate immunity in Caenorhabditis elegans by the TIR domain adaptor protein TIR-1, an ortholog of human SARM. Nat Immunol. 5, 488-494 (2004).
- Morton, E., Lamitina, T. A suite of MATLAB-based computational tools for automated analysis of COPAS Biosort data. Biotechniques. 48, xxv-xxx (2010).
- Rohlfing, A. K., Miteva, Y., Hannenhalli, S., Lamitina, T. Genetic and physiological activation of osmosensitive gene expression mimics transcriptional signatures of pathogen infection in C. elegans. PLoS One. 5, e9010-e9010 (2010).
- Lee, K. Z., Kniazeva, M., Han, M., Pujol, N., Ewbank, J. J. The fatty acid synthase fasn-1 acts upstream of WNK and Ste20/GCK-VI kinases to modulate antimicrobial peptide expression in C. elegans epidermis. Virulence. 1, 113-122 (2010).
- Duverger, Y. A semi-automated high-throughput approach to the generation of transposon insertion mutants in the nematode Caenorhabditis elegans. Nucleic Acids Res. 35, e11-e11 (2007).
- Pierce-Shimomura, J. T. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc. Natl. Acad. Sci. U.S.A. 105, 20982-20987 (2008).
- Ruzanov, P., Riddle, D. L. Deep SAGE analysis of the Caenorhabditis elegans transcriptome. Nucleic Acids Res. 38, 3252-3262 (2010).
- Kurz, C. L., Ewbank, J. J. Infection in a dish: high-throughput analyses of bacterial pathogenesis. Curr. Opin. Microbiol. 10, 10-16 (2007).
- Okoli, I. Identification of antifungal compounds active against Candida albicans using an improved high-throughput Caenorhabditis elegans assay. PLoS One. 4, e7025-e7025 (2009).
- Moy, T. I. Identification of novel antimicrobials using a live-animal infection model. Proc. Natl. Acad. Sci. U. S. A. 103, 10414-10419 (2006).
- Ballestriero, F., Thomas, T., Burke, C., Egan, S., Kjelleberg, S. Identification of compounds with bioactivity against the nematode Caenorhabditis elegans by a screen based on the functional genomics of the marine bacterium Pseudoalteromonas tunicata D2. Appl. Environ. Microbiol. 76, 5710-5717 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten