Method Article
Quantitativos e automatizada de alto rendimento de todo o genoma Telas de RNAi em C. elegans
Neste Artigo
Resumo
Nós descrevemos um protocolo com C. elegans E bibliotecas de alimentação RNAi que permite a medição automatizada de vários parâmetros, tais como fluorescência, tamanho e opacidade de vermes individuais de uma população. Nós dar um exemplo de um ecrã para identificar genes envolvidos na imunidade anti-fúngica inato em C. elegans.
Resumo
RNA de interferência é um poderoso método para compreender a função do gene, especialmente quando conduzida a uma escala do genoma inteiro-e num contexto quantitativa. Em C. elegans, a função do gene pode ser batido para baixo de maneira simples e eficiente pela alimentação de vermes com bactérias que expressam um ARNdc que corresponde a um gene específico 1. Embora a criação de bibliotecas de clones de RNAi cobrindo a maior parte do C. elegans genoma 2,3 abriu o caminho para estudos genômicos funcionais verdadeiros (ver, por exemplo 4-7), a maioria dos métodos estabelecidos são difíceis. Moy e colegas desenvolveram semi-automatizados protocolos que facilitem genome-wide telas 8. A abordagem baseia-se na imagem microscópica e análise de imagem.
Descrevemos aqui um protocolo alternativo para um alto rendimento de tela de todo o genoma, com base no manuseamento robótico da bacteriana RNAi clones, a análise quantitativa utilizando o Biosort COPAS (União Biometrica (UBI)), e umasoftware integrado: o MBioLIMS (Laboratório de Sistema de Gestão da Informação da Modul-Bio) uma tecnologia que proporciona maior produtividade para gerenciamento de dados e rastreamento de amostra. O método permite que as telas para ser conduzido em placas de meio sólido. Isto é particularmente importante para alguns estudos, como as que tratam sistema patógeno-hospedeiro interações em C. elegans, uma vez que certos micróbios não infectam eficientemente vermes em meio líquido.
Nós mostramos como o método pode ser utilizado para quantificar a importância de genes em anti-fúngica imunidade inata em C. elegans. Neste caso, a abordagem baseia-se na utilização de uma estirpe transgénicos portadores uma infecção epidérmica induzível-gene repórter fluorescente, com GFP sob o controlo do promotor do gene antimicrobiana péptido nlp 29 e um repórter vermelho fluorescente que é expressa constitutivamente na epiderme. Este último apresenta um controlo interno para a integridade funcional da epidermee transgene não específica silenciamento 9. Quando vermes de controlo são infectadas pelo fungo que fluorescem verde. Derrubar por RNAi um gene necessário para a PNL 29 resultados de expressão em fluorescência reduzida após a infecção. Actualmente, este protocolo permite que mais de 3.000 clones RNAi a ser testado e analisados por semana, abrindo a possibilidade de peneiramento todo o genoma, em menos de 2 meses.
Protocolo
O protocolo como descrito abaixo é dividida em etapas em cinco dias sucessivos (Fig. 1). Como se observa, certos passos pode ser feito em dias diferentes. A duração de cada passo, bem como a quantidade de material requerida (vermes por exemplo, bactérias, meios de comunicação) irá depender do número de placas de 96 poços tratados por experiência.
Dia 1
1. Preparação das placas de 96 poços NGM RNAi
O protocolo seguinte é adaptado a partir do método padrão para a tomada de placas de cultura de vermes 10, que inclui os pormenores das técnicas de esterilização para as diferentes soluções.
- Para 10-12 placas de 96 poços, a preparação de 100 mL de meio de crescimento nematóide (NGM) em desionizada H2O: 1,7 g BactoAgar, 0,29 g de NaCl, 0,25 g de peptona, 100 uL de colesterol (5 mg / mL em EtOH).
- Autoclave NGM (5 min a 121 ° C durante 100 mL; 30 min a 121 ° C durante 4 L) e deixar arrefecer até que seja menos cerca de 50 ° C (eno apenas frescough para segurar). É importante que o NGM permanece quente suficiente para que ele não se solidifica.
- Adicionar a 100 mL: 2,5 mL de tampão fosfato pH 6 (1M), 100 uL MgSO 4 (1M), 100 uL de CaCl2 (1M), 400 uL IPTG (1M), 100 uL Ampicilina (100 mg / mL), 100 uL de tetraciclina (12,5 mg / mL em EtOH).
- Distribuir 75 uL de NGM a cada poço de uma placa de 96 poços de fundo plano. Para evitar a solidificação do meio, que necessita de ser dispensado rapidamente, é conveniente usar um dispensador repetitivo (por exemplo, Repeatman, Eppendorf). Certifique-se que não há bolhas de ar presentes nos poços.
- Armazenar as placas imediatamente numa câmara húmida (por exemplo, uma caixa de Tupperware com toalhas de papel molhado na parte inferior), a 4 ° C.
Nota: as placas podem ser preparados até uma semana antes e armazenadas a 4 ° C, o que pode tornar a organização de etapas subsequentes mais fácil.
2. Preparação automatizada de as placas de 96-DeepWell LB
- Preparar e distribuir 1,5 mL LB contendo ampicilina e tetraciclina 100μg/mL 12,5 mcg / ml a cada poço de uma placa de 96 DeepWell que tem uma capacidade máxima de 2 mL / poço. A distribuição é optimizada utilizando um sistema de manipulação de robótico líquido (por exemplo TECAN), mas também pode ser realizada manualmente, se necessário.
- Adicionar uma tampa para cada placa e armazenar a 4 ° C. A fim de controlar cada placa de 96 poços, um rótulo de código de barras único ou deve ser adicionado, nesta fase ou posteriormente.
Nota: as placas podem ser preparados de 2 a 3 dias de antecedência e armazenado a 4 ° C, o que pode tornar a organização de etapas subsequentes mais fácil.
3. Crescer os clones RNAi bacterianas nas placas de 96-DeepWell LB (cultura durante a noite)
- Para facilidade de manuseamento subsequente, quando os clones originais de RNAi da biblioteca estão num formato de 384 poços, redistribuir os clones em primeiro rotulados ou de código de barras padrão de 96 poços contendo placas LB com 100 Amp ug / mL e 12,5 mcg / mL Tet suplementado com glicerol (10% de concentração final). Quando as placas originais 384-assim não têm clones em todos os poços, como é o caso para o "Ahringer" mais comumente utilizado biblioteca, existem várias opções para organizar as placas filhas. Os clones podem ser re-distribuído de modo que não existem poços vazios nas placas filhas. Isto minimiza o número de placas a tela. Idealmente, contudo, durante a replicação, alguns poços em cada placa filha deve ser deixado vazio, de modo que estes podem ser posteriormente preenchido com clones de controlo padrão (positivo e negativo) que facilitam ao longo da placa-comparações. Estas placas de 96 poços filhas são então utilizados para sementes das placas 96-DeepWell LB para uma cultura (ON) durante a noite.
Todos os passos de manipulação de líquidos são melhor feito com um sistema de manipulação de robótico líquido (por exemplo Tecan), mas pode ser realizada manualmente utilizando, por exemplo, um replicador de 96 pinos. Se os clones RNAi biblioteca já estão em um formato de 96 poços vá para o passo 3.5.
- Incubar as placas de 96 poços filha RNAi a 37 ° C com agitação (200 rpm) durante 14-15 horas.
- Verificar que as bactérias cresceram correctamente e identificar clones que não crescem. Estes serão excluídos das análises posteriores. Idealmente, o OD 600 é medida em cada poço, mas isto pode ser feito por inspecção simples.
- Armazenar as placas de 96 poços filha RNAi a -80 ° C antes da utilização.
- Descongelar as placas de 96 poços contendo os clones RNAi à temperatura ambiente. As placas podem então ser mantida a 4 ° C até à utilização.
- Distribuir 3 uL de cada clone bacteriano a partir de uma placa de 96 poços replicar para os 96 poços da placa de LB correspondentemente rotulados / código de barras 96-DeepWell contendo 1,5 mL de LB com 100 Amp ug / mL e 12,5 mcg / mL Tet. Clones bacterianos para controlar RNAi eficiência, tal como um clone GFP (RNAi) no presente exemplo, e controlos negativos, pode ser incorporado em qualquer manualmente bem disponível vazio.
- Cobrir a 96-p DeepWelllates com uma película adesiva (película cultura por exemplo AeraSeal celular a partir de Dutscher). Substituir a placa de 96 poços utilizado replicar a -80 ° C.
- As placas 96-DeepWell marcados são, em seguida, incubou-se em a 37 ° C com agitação (200 rpm durante 14-15 horas).
Dia 2
4. Semente os clones RNAi bacterianas sobre as placas de 96 poços NGM RNAi
Esta etapa deve ser feito na manhã seguinte à cultura ON.
- Tomar as placas de 96 poços NGM a partir de 4 ° C e deixá-los aquecer e secar para 5-15 min sob um fluxo laminar estéril armário. Não deixe as placas muito tempo sob o capô (máximo de 30 min), caso contrário o NGM pode posteriormente crack. Adicione o rótulo adequado / código de barras para cada prato.
- Recuperar o 96-DeepWell em placas de cultura de 37 ° C. Gravar as posições de quaisquer poços vazios (um poço vazio é completamente translúcida), os quais serão excluídos das análises subseqüentes.
- Centrifugar a 96-DeepWell placas 5 min a 4000 rpm.
- Ponta fora o sobrenadante, girando a placa de cabeça para baixo acentuadamente e secar as bordas da placa rapidamente sobre uma toalha de papel. À medida que as culturas são de bactérias recombinantes, o sobrenadante tem de ser tratada de acordo com a legislação local (por exemplo, autoclavado).
- Ressuspender o sedimento bacteriano no líquido residual (cerca de 50 uL) em vórtice, de preferência usando um agitador dedicado (por exemplo, Tecan) de modo que cada placa recebe precisamente o mesmo tratamento. É possível medir a OD 600 de cada poço para padronizar precisamente inoculo bacteriano para o passo subsequente, mas isto não é indispensável, e não realizar tal controlo.
- Transferir 5 uL das bactérias ressuspensos para as placas de 96 poços utilizando um NGM 8 ou 12-multi-canal da pipeta. Tenha cuidado para não tocar ou penetrar na NGM. Este passo é executado manualmente, mas pode ser optimizada utilizando um robot, tais como o Liquidator96 (Rainin) que permite que odistribuição de 96 clones RNAi em um passo.
- Deixe as bactérias seco ao abrigo de um gabinete de fluxo laminar estéril, verificando regularmente para evitar o NGM tornar-se demasiado seco. Esta etapa deve levar cerca de 2 horas.
- Incubar as 96-poços RNAi-Ngm as placas a 37 ° C em numa câmara húmida.
5. Prepare uma população sincronizada de worms
Para esta etapa, usamos worms transgênicas com o gene repórter (s) de interesse (por exemplo, uma infecção induzida p-nlp-29 :: GFP construção e, como um controle interno, uma p constitutiva col-12 :: transgene dsRed construção) . Por causa da diminuição observada de expressão do transgene com o tempo, nós descongelar lotes frescas de vermes cada 6 semanas. Nós, os worms de cultura para pelo menos 2 gerações antes da utilização, e garantir que os vermes nunca passaram fome antes do ensaio. Se o dia 2 do protocolo é terça-feira, nos preparamos worms na sexta-feira, colocando 30 vermes adultos jovens em uma placa NGM 9 centímetros espalhar com OP50 bactériasa 20 ° C; eles estará pronto para lixívia na terça-feira (ver Tabela 1).
- Worms Bleach seguindo o protocolo padrão de 10.
- Deixe os ovos eclodem em M9 a 25 ° C ON com agitação para obter uma população sincronizado de larvas L1.
Dia 3
6. Distribua os vermes nos 96-RNAi bem-Ngm placas de alimentação e RNAi
Este passo deve ser feito de manhã. Worms L1 estágio sincronizados são distribuídos em cada clone 96-bacteriana bem para alimentação e RNAi.
- Recuperar os 96-poços RNAi-Ngm placas de 37 ° C e deixá-los à temperatura ambiente para arrefecer.
- Recuperar os vermes esbranquiçados de 25 ° C. Estimar o número de vermes por 2 uL e ajuste com M9 ter cerca de 100-120 vermes por 2 microlitro (aproximadamente uma gota).
- Distribuir manualmente 2 l da L1 estágios worms em cada cavidade utilizando um dispensador repetitivo. Verifique cada bem para os vermes (worms você deve ver nadando em líquido).
- Deixe as placas seco sob um armário estéril de fluxo laminar (hora máximo 1). Verifique regularmente as placas, os vermes deve ser engatinhando e não nadar. Tenha cuidado, a NGM não deve secar muito, senão ele vai quebrar.
- Colocar as placas a 25 ° C em câmara húmida.
7. Teste os esporos Drechmeria coniospora para infecção eficaz
Este passo é específico para o ecrã presente. Ele consiste em testar diferentes lotes de esporos para selecionar aqueles altamente infecciosa. Por conseguinte, deve ser feito tão próximo quanto possível antes do dia 4, o dia da infecção. É necessário preparar suficientes L3-L4 vermes em avanço para estes testes (Tabela 1). Os métodos de execução necessários para a D. cultura coniospora têm sido descritas noutro local 11.
- Collect esporos de uma amostra de cada cultura de fungos em um pequeno volume de M9.
- Coloque uma gota de cada suspensão de esporos em uma placa NGM 35 milímetros semeado com OP50, e deixe secar.
- Adicionar 30-40 L3-L4 vermes com o gene repórter de interesse (por exemplo, p nlp-29 :: GFP).
- Colocar as placas infectadas a 25 ° C ON.
- No dia seguinte, confira os vermes para a indução do GFP e selecione o lote de fungo que dá brilho a indução, a mais ampla e mais homogêneo de fluorescência verde.
Dia 4
8. Infectar o RNAi exposto worms com D. esporos coniospora
Este passo é realizado 30 horas após a alimentação quando RNAi vermes atingiram a fase L3-L4.
- Determinar o volume de esporos para usar para distribuir 4 uL por poço.
- Colete D. fresco esporos coniospora a partir do lote seleccionado com tampão M9. Use 8-10 mL de M9 para uma placa de 9 cm de esporos.
- Distribuir 4 uL de esporos em cada poço com um dispensador repetitivo. Verifique cada poço de esporos (você pode ver a natação worms em líquido).
- Deixe as placas seco sob um armário estéril de fluxo laminar (~ 1 hora). Verifique as placas regularmente até os vermes iniciar o rastreamento. Tenha cuidado, a NGM não deve secar muito, senão ele vai quebrar.
- Colocar as placas infectadas a 25 ° C em câmara húmida.
Dia 5
9. Observação e armazenamento das placas, antes da análise quantitativa automatizado
Este passo é iniciado 18 horas após a infecção e é realizada durante 5 dias. Worms são esperados para ser jovens adultos começam a pôr ovos. Durante este passo, o fenótipo é pontuada visualmente: ou vermes fluorescência verde que corresponde a uma infecção situação normal sequência, ou a indução da GFP é alterada de uma dada bem que significa que as alterações genéticas silenciados a resposta à infecção.
- rapidamente observar as placas sob o microscópio de fluorescência de dissecação. Se as placas mostram uma indução GFP bem geral então prosseguir para o passo seguinte, caso contrário, esperar até à tarde para uma melhor indução, enquanto ainda há comida deixada para os vermes.
- Visualmente marcar cada placa e observe qualquer bem com um fenótipo marcado (por exemplo, nenhuma expressão de GFP). Ao verificar pelos resultados obtidos com quaisquer clones de controlo RNAi que possam ter sido adicionados, esta análise preliminar primeiro dá uma indicação de se a experiência tem sido bem sucedida.
- Usando um 8 ou 12 multi-canal de transferência Pipetar os vermes com 100 ul de NaCl 50 mM-Triton 0,05%, para uma nova correspondentemente rotulados / código de barras de 96 poços de fundo redondo de chapa. Congelar as placas à temperatura de -80 ° C até à análise.
- No dia da análise, descongelar as placas à temperatura ambiente e proceder à análise automatizada usando o Biosort COPAS. O classificador analisa um único prato em 22 minutos. As placas não deve serdeixada à temperatura ambiente por mais de uma hora antes da análise, e não podem ser congelados novamente.
- As informações obtidas a partir da Biosort COPAS são transcritos para um arquivo do Excel para verificação rápida da qualidade dos dados. Os dados em bruto com os diferentes parâmetros medidos pelo Biosort COPAS (densidade óptica (extinção), o comprimento axial (tempo de voo), as emissões de fluorescência, simultaneamente por três detectores diferentes) são então armazenados em um pacote LIMS dedicado (MBiO LIMS, Modul- Bio) para posteriores análises quantitativas detalhadas.
10. Os resultados representativos
Usando as quantidades de vermes e bactérias dadas acima, no final da experiência, na maioria dos poços, os animais devem todos têm desenvolvido até à idade adulta e deve ainda haver algum alimento deixado em todas as cavidades. Sob estas condições, a maioria dos poços devem também conter ovos. Deve haver um número suficiente de adultos em cada poço de modo que os dados são obtidos Biosort fou pelo menos 50 vermes individuais. Por outro lado, se o RNAi é eficiente, um grande número de clones RNAi irá provocar um fenótipo visível. Por exemplo, os worms podem ser descoordenada, ou mais, obviamente, estéril, ou preso em seu desenvolvimento. Isto deve ocorrer com frequência, de modo que, geralmente, pelo menos, um poço por placa de 96 poços contém vermes com um fenótipo RNAi evidente. Quando o fenótipo de interesse é a expressão de um gene repórter da GFP, poços com um clone de GFP (RNAi) proporcionar um controlo robusto (Fig. 2). Outros fenótipos, tais como uma alteração de tamanho podem ser prontamente medido com o Biosort (Fig. 3A). Observamos que os vermes podem migrar para poços adjacentes em freqüência muito baixa (<1%, BS, resultados não publicados), quando o alimento em um poço que acabar.
Derrubar a função de diferentes classes de genes podem afetar a expressão de transgenes C. elegans. Alguns são não-especificamente necessária para expressão transgenes 12. Outros, taiscomo factores de transcrição específicos de tecidos, por exemplo, o factor GATA epidérmica ELT-3 13, será necessária em subconjuntos específicos de células. Esta é uma das razões para a inclusão de um não-relacionadas e constitutiva epidérmica célula repórter transgene construto (p col-12 :: dsRed) na estirpe utilizada para o protocolo de corrente. Isto permite que tais genes a serem distinguidos dos especificamente envolvidas no processo de gene regulador em estudo (Fig. 3B e C). Este transgene é expresso durante todas as fases larvares. Se um ensaio envolve a detecção de expressão do gene em embriões de um gene repórter diferente de controlo seria necessária.
Uma das inovações deste protocolo é o uso de placas de congelamento para adiar a sua análise. Congelação permite placas a ser armazenado por até duas semanas sem alterar substancialmente os resultados. Embora os valores absolutos de fluorescência medida poderá ser alterado, suas proporções relativas permanecem semelhante (Fig. 4). No otsua mão, RNAi é um procedimento de 14 intrinsecamente variável experimental. Para obter resultados robustos e eliminar os potenciais muitos falsos positivos, telas de RNAi precisam ser replicados, pelo menos uma vez. Se as experiências são realizadas com êxito, deve haver uma correlação razoável entre os resultados de uma placa de testado em dias diferentes. Particularmente, os genes que possuem um forte efeito deve ser claramente identificáveis, mesmo se o efeito preciso quantitativa não é idêntica entre as placas em duplicado (Fig. 5).
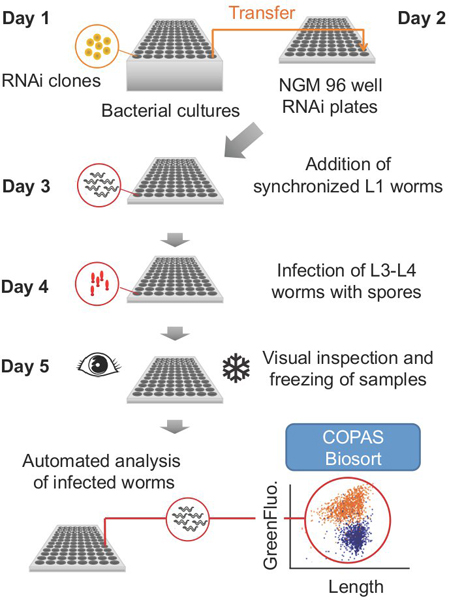
Figura 1. Regime geral de uma experiência típica.

Figura 2. Controlo RNAi utilizando um clone de segmentação expressão da GFP. Transgênicos worms expressividadeng constitutivamente ap col-12 :: repórter dsRed e uma induzível p nlp 29 :: GFP repórter em meio sólido em uma placa de 96 poços visualizada utilizando um microscópio de dissecção GFP com um conjunto de filtro que permite a observação simultânea de fluorescência vermelha e verde. Worms foram expostos a controlar (painéis superiores) ou um GFP RNAi clone (painéis inferiores) durante 48 h. Os painéis à esquerda são vermes não infectados, aqueles sobre os vermes direito 18 horas após a infecção com D. coniospora. A barra de escala é de 1 mm.

Figura 3. A análise quantitativa de uma placa de 96 poços. O Biosort COPAS gera dados numéricos para cada verme analisados. Os gráficos mostram a média e desvio padrão para o tempo de voo (TOF; A), que corresponde ao tamanho dos vermes 15, fluorescência vermelha (B) e da razão (X100) de fluorescência verde para TOF (C) do transgénico WOrms expressando constitutivamente ap col-12 :: repórter dsRed e uma induzível p nlp 29 :: GFP repórter que tinha sido cultivado em meio sólido em uma placa de 96 poços com diferentes clones RNAi durante 48 h, 18 horas após a infecção com D. coniospora. Certos clones, tais como a um destaque com a seta em (A), os defeitos de crescimento e / ou provocam afectar a expressão de p col-12 :: dsRed. Outros, especificamente afetar a expressão da GFP, como destacado pela seta em (C). Este clone particular, tem como alvo o gene nsy 1, previamente demonstrado ser importante para a regulação da PNL 29 13. Verde, vermelho e fluorescência TOF são medidos em unidades arbitrárias, mas constante.
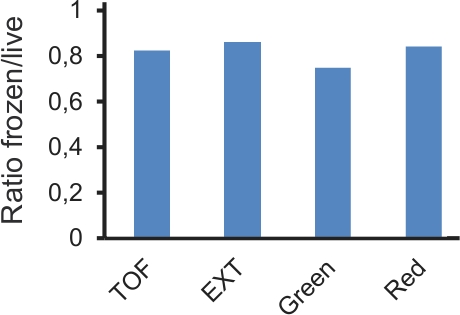
Figura 4. CompArison dos dados obtidos com amostras vivas e congeladas a partir da mesma experiência. Placas de L4 vermes que transportam p nlp-29 :: GFP e col p-12 :: transgenes dsRed ou foram infectados com D. coniospora ou não. Depois de 18h, cada placa foi dividido em 2; meia foi analisado imediatamente e meia congeladas a -80 ° C durante 24 horas, antes de descongelação e análise. Os valores para média TOF (tamanho de vermes), EXT (densidade óptica) e Green e fluorescência vermelha foram calculados para cada amostra. O gráfico mostra a relação entre a média para infectados dividido por não-infectadas amostras, para as amostras congeladas e vivo (n = 7638 e 5096, e 8634 e 3850 vermes, respectivamente).

Figura 5. A análise comparativa dos duplicados placas de 96 poços. A razão de fluorescência normalizada (a razão entre a fluorescência média (aqui, 100 * verde / TOF) para cada poço dividido porrecortada (25 ° ao 75 percentil) significa para cada placa) para cada poço de análises em duplicado de um representante placa de 96 poços.
| Material | Cronograma Experimental |
| Quinta-feira
|
Tabela 1. Exemplo de uma experiência um ciclo com 30 placas.
Aqui é apresentado o material necessário para uma experiência de um ciclo de 30 placas e um cronograma para um experimento a partir do passo 1 a passo 12 organizados em 9 dias.
Nematóides meios de crescimento (NGM), 100 ml:
| 0.3 g | NaCl |
| 0,25 g | BactoPeptone |
| 2 g | BactoAgar |
| 100 L | 5 mg / mL de colesterol em EtOH |
| Adicionar água desionizada para 100 ml | |
| Autoclave durante 5 minutos a 121 ° C e deixar arrefecer, em seguida, adicionar: | |
| 100 L | 1 M MgSO4 |
| 100 L | 1 M de CaCl2 |
| 2,5 mL | 1 M pH 6,0 KPO4 |
NGM para RNAi tratamento em placa de 96 poços, 100 mL:
| 0,29 g | NaCl |
| 0,25 g | BactoPeptone |
| 1,7 g | BactoAgar |
| 100 L | 5 mg / mL de colesterol em EtOH |
| Adicionar de água para ionizado 100 mL | |
| Autoclave durante 5 minutos a 121 ° C e deixar arrefecer, em seguida, adicionar: | |
| 100 L | 1 M MgSO4 |
| 100 L | 1 M de CaCl2 |
| 2,5 mL | 1 M pH 6,0 KPO4 |
| 400 uL | 1 M IPTG |
| 100 L | 100 mg / ml de ampicilina |
| 100 L | 12,5 mg / ml de tetraciclina |
Solução de água sanitária, 5mL:
2,5 mL de H2O
2,3 mL Bleach
0,2 mL de NaOH 50%
NaOH a 50%, 100 mL:
50 g de NaOH
Adicionar água desionizada para 100 ml
NaCl 50 mM-Triton 0,05%, 400 mL:
4 mL de NaCl 5 M
1 mL de Triton 20%
Adicionar água desionizada para 400 ml
2 mL de Triton X-100
8 mL de água deionizada
Tampão M9, 1L
6 g de Na 2 HPO 4 (MW: 178)
3 g KH 2 PO 4 (MW: 136)
5 g de NaCl
Adicionar água desionizada para 1 L
Autoclave
Adicionar 1 mL MgSO4 1 M
Luria Broth (LB), 1L:
10 g BactoTryptone
O extracto de levedura 20 g
10 g de NaCl
Adicionar água desionizada para 1 L
Autoclave
1 M MgSO4, 300 mL:
73,95 g MgSO 4
Adicionar água desionizada para 300 ml
Autoclave e armazenamento em temperatura ambiente
5 mg / Colesterol mL, 200 mL:
1 g Colesterol
Adicionar 100 EtOH% a 200 mL
Filtrar esterilizar e armazenar em temperatura ambiente
1 M de CaCl2, 500 mL:
27.75 g de CaCl2
Adicionar água desionizada para 500 ml
Filtrar esterilizar e armazenar em temperatura ambiente
1 M KPO 4 pH 6,0, 4 L:
517 g KH 2 PO 4 (MW: 136)
207 g de K 2 HPO 4 (MW: 174)
Adicionar água desionizada para 4 L
100 mg / ml de ampicilina (Amp), 10 mL:
1 g de Ampicilina
Adicionar água desionizada a 10 mL
Armazenar a -20 ° C
1 M isopropílico β-d-tiogalactopiranósido (IPTG), 10 mL:
2,38 g IPTG
Adicionar água desionizada a 10 mL
Armazenar a -20 ° C
12,5 mg / ml de tetraciclina, 100 mL
1,25 g de tetraciclina
Adicionar 100% EtOH a 100 mL
Tabela 2. Solutions.
Discussão
O protocolo atual permite grande escala de rastreamento em RNAi C. elegans Em meios sólidos, usando o nível de expressão de um gene repórter fluorescente como uma leitura para fora. Tem sido optimizada com instrumentos automatizados para facilitar alto rendimento ensaios, permitindo a manipulação fácil e rápido de grandes números de amostras, de uma maneira eficiente, fiável e reproduzível. Outros métodos têm sido descritos, que não dependa tanto manipulação automatizada da amostra líquida 8.
Um aspecto chave organizacional para o protocolo tem sido o desenvolvimento de um método robusto de acompanhamento amostra através da adaptação do software MBioLIMS comercial para o procedimento de tela RNAi em colaboração com o ModulBio empresa. Em cada fase do processo, as placas são agora identificados por códigos de barras geradas dentro do LIMS. À medida que o LIMS também regista a identidade dos clones em cada poço em cada placa, indo de bem ao gene putativo é simples. E, além disso, os links LIMS para o banco de dados WormBase comunidade, permitindo o acesso direto aos dados moleculares e funcionais sobre qualquer gene. O LIMS também facilita a análise a ser realizada na placa ou mesmo a nível dos cromossomas e genoma. Para estas análises, os dados são armazenados de uma maneira que é inteiramente compatível com as ferramentas previamente descritos, tais como COPAmulti 16.
Um ponto crítico experimental no método é o ajustamento da quantidade de alimento em relação ao número de vermes. Para muitos ensaios, é essencial que os vermes não fome no final da experiência, mas também é importante não ter muita comida no início. Observou-se que uma densidade elevada de alimentos diminui a indução de fluorescência no protocolo usando vermes que transportam o p Nlp-29 :: Repórter GFP. Isto pode reflectir uma eficiência de infecção diminuída quando os vermes são cultivadas em bactérias demasiado (BS, resultados não publicados). Ao mesmo tempo, as condições de cultura das bactérias RNAi também é importante, uma vez que este determina se ARNdc é produzido a um nível óptimo. Alguns protocolos para grande escala RNAi requerem cultura de RNAi clones em placas de ágar antes de semear culturas líquidas 8. Foram obtidos silenciamento do gene robusto omitindo este passo. Optou-se para o crescimento de um excesso de cada cultura para assegurar resultados homogéneos. O protocolo descrito no facto fornece material suficiente para conduzir 2 ou 3 telas em paralelo. No interesse de obter os resultados mais robustos, nós favorecemos, no entanto, a realização de telas em duplicata, utilizando culturas independentes de vermes, bactérias e esporos de fungos de RNAi.
Outro passo crítico está relacionado ao uso de meio que não é nem muito úmido nem muito seco. Se as placas não são suficientemente seca, a bactéria RNAi não seca de forma adequada e vermes nadará durante o ensaio. Isto pode causar problemas. Por exemplo, os vermes não são eficientemente infectado por D. coniospora Em líquido (C. Couillault, resultados não publicados). Por outro lado, em placas secas, a osmolaridade dos aumentos de meio de cultura. Isso tem efeitos diretos sobre a fisiologia verme 17, Incluindo a indução de alguns genes de péptidos antimicrobianos, tais como Nlp 29 18. O volume utilizado para dispensar vermes e esporos é crítico para permitir a secagem eficaz e rápida. Se mais do que 10 uL de líquido é adicionada às placas frescas, os poços pode levar várias horas para secar. Isto pode significar que é difícil de utilizar métodos robóticos para adicionar vermes para as placas. Por exemplo, cada verme é dispensado pelo Biosort em cerca de 1 uL de líquido. Atualmente, distribuir os vermes manualmente, que também nos permite superar a tendência de vermes em solução de sedimentos.
Nós anteriormente relatado um ajustamento do Biosort que permitiu uma placa de 96 poços a serem analisados em 36 minutos 19. A máquina do fabricante UBI agora oferece uma modificação do Biosort diminuir ainda mais o tempo de análise. Com o "FastReFlex", as amostras são encaminhadas diretamente através da célula de fluxo para análise, ignorando o filtro bolha armadilha. Isto resulta numa redução significativa no tempo de aquisição de dados; uma placa inteira pode ser analisado em 22 min, permitindo a cerca de 20 placas a ser tratado por dia. Se as placas não foram congeladas, no entanto, não haveria um intervalo de 8 horas entre os primeiros e últimos. Dada a duração relativamente curta do experimento, com a infecção dura apenas 18 horas, isso iria introduzir uma variável inaceitável em placas. Não só as placas de congelação antes da análise resolver este problema, mas também é essencial no contexto de alto rendimento como análises, com o protocolo padrão actual e líquido de tratamento de robôs, 2 pessoas podem facilmente processar um máximo de 50 placas de uma semana. Atualmente, nós rotineiramente tela de 30 placas por semana, o que permite uma tela de genoma inteiroa ser conduzida uma vez em menos de 2 meses.
Uma característica interessante deste protocolo é o facto de que ele usa agar sólido, permitindo que os ensaios que não podem ser realizados em cultura líquida. Como fisiologia verme difere dependendo das condições de cultura 20,21, Sendo capaz de realizar ensaios em meio sólido pode também complementar habituais líquido à base de telas. Para certos fenótipos (por exemplo, movimento), a análise pode ser realizada directamente com placas de 96 poços em meio sólido. Para a tela atual, no entanto, como o Biosort não pode provar a partir de placas de meio sólido, worms são primeiro transferidos para líquido antes do congelamento. Por outro lado, o Biosort COPAS permite uma análise quantitativa multiparamétrico compatível com a medição de muitos diferentes fenótipos. É provável que para qualquer determinada tela nem todos os parâmetros medidos será o primário de leitura para fora. No entanto, estas medições podem ser úteis por várias razões. Eles permitem que um para determinar se o ensaio correu correctamente. Por exemplo, pode-se verificar o número e tamanho dos vermes em cada poço. Só quando um clone de RNAi deve bloquear o crescimento ou a reprodução deve haver um desvio dos valores esperados. Wells com menos de 20 vermes e / ou animais 30% menor (TOF) em comparação com um standard controle sem efeito sobre o crescimento ( Por exemplo gfp (RNAi)) Pode ser excluída a partir de análises subsequentes. A função destes clones podem ser tratadas utilizando um protocolo alternativo, em que os vermes são expostos às bactérias RNAi após o seu desenvolvimento é bem avançado 15. Os parâmetros diferentes podem também ser utilizado para refinar gene candidato (batida) de selecção, por exemplo, limitando acessos apenas para os que afectam a fluorescência verde, mas não vermelho. O método preciso escolhido para a selecção acessos dependerá do tipo de tela e sua exacta de leitura para fora. Dada a complexidade dos dados gerados a partir de uma tela de todo o genoma quantitativa, a colaboração com um estatístico pode ser necessária.
O protocolo descrevemos pode ser prontamente adaptado a outros tipos de ensaios. Por exemplo, na fase de infecção D. coniospora Poderia ser substituído por qualquer outro agente patogénico capaz de infectar em meios sólidos. Isto é potencialmente útil como nós descobrimos que muitos dos factores de virulência importantes para Serratia marcescens infecção em cultura líquida não desempenham um papel durante a infecção em meio sólido (E. Pradel, comunicação pessoal). O protocolo é também totalmente compatível com os testes de compostos químicos 22-24, Ou para identificar compostos com bioactividade usando bibliotecas diferentes de bactérias 25, Então deve encontrar uma utilidade geral no C. elegans Comunidade de pesquisa.
Divulgações
O laboratório Ewbank colabora com União Biometrica e Modul-Bio.
Agradecimentos
Agradecemos a D. Braendle, CL Kurz e F. Montañana-Sanchis por suas contribuições. Este projecto foi apoiado por doações da ANR e do Conseil Regional PACA e financiamento institucional de INSERM e do CNRS.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| BactoAgar | BD Diagnostic Systems | 214010 | |
| Ampicilina | Sigma-Aldrich | A9518 | |
| BactoPeptone | BD Diagnostic Systems | 211677 | |
| CaCl 2 | Qualquer fornecedor | ||
| Película adesiva AeraSeal | Dutscher | 760214 | |
| Isopropílico β-d-tiogalactopiranósido (IPTG) | Thermo Scientific | AB-0481 | |
| K 2 HPO 4 | Qualquer fornecedor | ||
| KH 2 PO 4 | Qualquer fornecedor | ||
| MgSO4 | Qualquer fornecedor | ||
| NaCl | Qualquer fornecedor | ||
| Na 2 HPO 4 | Qualquer fornecedor | ||
| NaOH | Qualquer fornecedor | ||
| Placa de 96 poço profundo | Thermo Scientific | AB-0932 | |
| Placa de 96 poços plana | FALCON | 353072 | |
| Placa de 96 poços rodada | FALCON | 353077 | |
| Tetraciclina | Sigma-Aldrich | T8032 | |
| Triton X-100 | Qualquer fornecedor | ||
| TECAN robô | TECAN | ||
| Liquidator96 TM | RAININ | ||
| COPAS Biosort | União Biometrica | ||
| LIMS | Modul-Bio |
Referências
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854-854 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Rual, J. F. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14, 2162-2168 (2004).
- Lee, S. S. A systematic RNAi screen identifies a critical role for mitochondria in C. elegans longevity. Nat. Genet. 33, 40-48 (2003).
- Hamilton, B. A systematic RNAi screen for longevity genes in C. elegans. Genes Dev. 19, 1544-1555 (2005).
- Frand, A. R., Russel, S., Ruvkun, G. Functional genomic analysis of C. elegans molting. PLoS Biol. 3, e312-e312 (2005).
- Parry, D. H., Xu, J., Ruvkun, G. A whole-genome RNAi Screen for C. elegans miRNA pathway genes. Curr. Biol. 17, 2013-2022 (2007).
- O'Rourke, E. J., Conery, A. L., Moy, T. I. Whole-animal high-throughput screens: the C elegans model. Methods Mol. Biol. 486, 57-75 (2009).
- Pujol, N. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Curr. Biol. 18, 481-489 (2008).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1551-8507 (2006).
- Powell, J. R., Ausubel, F. M., Ewbank, J., Vivier, E. Models of Caenorhabditis elegans Infection by Bacterial and Fungal Pathogens. Methods Mol Biol. 415, 403-427 (2008).
- Cardoso, C. XNP-1/ATR-X acts with RB, HP1 and the NuRD complex during larval development in C. elegans. Dev. Biol. 278, 49-59 (2005).
- Pujol, N. Anti-fungal innate immunity in C. elegans is enhanced by evolutionary diversification of antimicrobial peptides. PLoS Pathog. 4, e1000105-e1000105 (2008).
- Simmer, F. Genome-Wide RNAi of C. elegans Using the Hypersensitive rrf-3 Strain Reveals Novel Gene Functions. PLoS Biol. 1, e12-e12 (2003).
- Couillault, C. TLR-independent control of innate immunity in Caenorhabditis elegans by the TIR domain adaptor protein TIR-1, an ortholog of human SARM. Nat Immunol. 5, 488-494 (2004).
- Morton, E., Lamitina, T. A suite of MATLAB-based computational tools for automated analysis of COPAS Biosort data. Biotechniques. 48, xxv-xxx (2010).
- Rohlfing, A. K., Miteva, Y., Hannenhalli, S., Lamitina, T. Genetic and physiological activation of osmosensitive gene expression mimics transcriptional signatures of pathogen infection in C. elegans. PLoS One. 5, e9010-e9010 (2010).
- Lee, K. Z., Kniazeva, M., Han, M., Pujol, N., Ewbank, J. J. The fatty acid synthase fasn-1 acts upstream of WNK and Ste20/GCK-VI kinases to modulate antimicrobial peptide expression in C. elegans epidermis. Virulence. 1, 113-122 (2010).
- Duverger, Y. A semi-automated high-throughput approach to the generation of transposon insertion mutants in the nematode Caenorhabditis elegans. Nucleic Acids Res. 35, e11-e11 (2007).
- Pierce-Shimomura, J. T. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc. Natl. Acad. Sci. U.S.A. 105, 20982-20987 (2008).
- Ruzanov, P., Riddle, D. L. Deep SAGE analysis of the Caenorhabditis elegans transcriptome. Nucleic Acids Res. 38, 3252-3262 (2010).
- Kurz, C. L., Ewbank, J. J. Infection in a dish: high-throughput analyses of bacterial pathogenesis. Curr. Opin. Microbiol. 10, 10-16 (2007).
- Okoli, I. Identification of antifungal compounds active against Candida albicans using an improved high-throughput Caenorhabditis elegans assay. PLoS One. 4, e7025-e7025 (2009).
- Moy, T. I. Identification of novel antimicrobials using a live-animal infection model. Proc. Natl. Acad. Sci. U. S. A. 103, 10414-10419 (2006).
- Ballestriero, F., Thomas, T., Burke, C., Egan, S., Kjelleberg, S. Identification of compounds with bioactivity against the nematode Caenorhabditis elegans by a screen based on the functional genomics of the marine bacterium Pseudoalteromonas tunicata D2. Appl. Environ. Microbiol. 76, 5710-5717 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados