Method Article
Quantitative e automatica High-throughput Genome-wide Schermi RNAi in C. elegans
In questo articolo
Riepilogo
Descriviamo un protocollo usando C. elegans E librerie di alimentazione RNAi che permette la misurazione automatica dei molteplici parametri come la fluorescenza, le dimensioni e l'opacità dei vermi individuali in una popolazione. Diamo un esempio di uno schermo per identificare i geni coinvolti in anti-fungine immunità innata C. elegans.
Abstract
RNA interferenza è un potente metodo per capire la funzione dei geni, soprattutto se condotta in un intero genoma scala e in un contesto quantitativo. In C. elegans, la funzione del gene può essere abbattuto modo semplice ed efficiente da mangiare vermi con i batteri che esprimono un dsRNA corrispondente a un gene specifico 1. Mentre la creazione di librerie di cloni RNAi che copre la maggior parte del C. elegans genoma 2,3 aperto la strada a veri e propri studi di genomica funzionale (vedi per esempio 4-7), la maggior parte dei metodi stabiliti sono laboriosi. Moy e colleghi hanno sviluppato protocolli semi-automatici che facilitano la genome-wide schermi 8. L'approccio si basa su immagini microscopiche e analisi delle immagini.
Qui si descrive un protocollo alternativo per un alto throughput genoma schermo, sulla base del manipolatore batterica cloni RNAi, analisi quantitativa utilizzando la Biosort COPAS (Union Biometrica (UBI)), e unosoftware integrato: il MBioLIMS (Laboratory Information System della Direzione di Modul-Bio), una tecnologia che offre un throughput maggiore per la gestione dei dati e tracciabilità del campione. Il metodo consente di schermi essere realizzato su piastre di terreno solido. Ciò è particolarmente importante per alcuni studi, come quelli riguardanti le interazioni ospite-patogeno in C. elegans, dal momento che alcuni microbi non si infettano in modo efficiente i vermi in coltura liquida.
Si mostrano come il metodo può essere utilizzato per quantificare l'importanza di geni anti-fungini immunità innata in C. elegans. In questo caso, l'approccio si basa sull'utilizzo di un ceppo transgenica che trasporta un epidermica un'infezione-inducibile gene fluorescente reporter, con GFP sotto il controllo del promotore del gene NLP peptide antimicrobico 29 e un reporter fluorescente rossa che si esprime costitutivamente nella epidermide. Quest'ultimo fornisce un controllo interno per l'integrità funzionale dell'epidermidetransgene e aspecifica tacere 9. Quando i vermi di controllo sono infettate dal fungo che fluorescenza verde. Abbattere da RNAi un gene necessario per nlp 29 risultati di espressione a fluorescenza diminuita dopo l'infezione. Attualmente, questo protocollo permette di oltre 3.000 cloni RNAi per essere testato e analizzato a settimana, aprendo la possibilità di screening dell'intero genoma in meno di 2 mesi.
Protocollo
Il protocollo, come descritto di seguito è suddivisa in passi su cinque giorni consecutivi (Fig. 1). Come notato, alcuni passi può essere effettuata in giorni diversi. La durata di ogni fase, così come la quantità di materiale richiesto (vermi ad esempio, batteri, media) dipenderà dal numero di piastre da 96 pozzetti trattati per esperimento.
Giorno 1
1. Preparazione dei 96 e NGM piastre RNAi
Il protocollo seguito è adattato dal metodo standard per la creazione piastre di coltura a vite senza fine 10, che comprende i dettagli delle tecniche di sterilizzazione per le diverse soluzioni.
- Per 10-12 piastre a 96 pozzetti, preparare 100 mL di mezzi di crescita nematodi (NGM) in H 2 O deionizzata: 1,7 g BactoAgar, 0,29 g di NaCl, 0,25 g peptone, colesterolo 100 pl (5 mg / mL in EtOH).
- Autoclave NGM (5 min a 121 ° C per 100 mL; 30 min a 121 ° C per 4 L) e lasciate raffreddare fino a quando non si trova a circa 50 ° C (solo freddo Enough tenere). È importante che la NGM rimane abbastanza caldo in modo che non si solidifichi.
- Aggiungere per 100 mL: 2,5 mL tampone fosfato pH 6 (1M), 100 pl MgSO 4 (1M), 100 pl CaCl 2 (1M), 400 pl IPTG (1M), 100 pl Ampicillina (100 mg / mL), 100 pl Tetraciclina (12,5 mg / mL in EtOH).
- Distribuire 75 microlitri di NGM a ciascun pozzetto di uno a 96 pozzetti a fondo piatto piatto. Per evitare la solidificazione del mezzo, deve essere erogato rapidamente, è conveniente utilizzare un erogatore ripetitivo (ad esempio Repeatman, Eppendorf). Assicurarsi che non vi siano bolle d'aria presenti nei pozzetti.
- Conservare le piastre immediatamente in una camera umida (ad esempio una scatola Tupperware con tovaglioli di carta bagnati in basso) a 4 ° C.
Nota: piastre possono essere preparati fino ad una settimana prima e conservato a 4 ° C, questo può rendere l'organizzazione delle fasi successive più facile.
2. Preparazione automatizzata dei 96 Deepwell piastre LB
- Preparare e distribuire 1,5 mL LB contenente ampicillina e tetraciclina 100μg/mL 12,5 ug / mL in ciascun pozzetto della piastra di 96 Deepwell che ha una capacità massima di 2 mL / pozzetto. La distribuzione è migliorata tramite un sistema robotizzato di manipolazione dei liquidi (per esempio TECAN) ma può anche essere effettuata manualmente, se necessario.
- Aggiungi una copertura per ogni piatto e conservare a 4 ° C. Al fine di tenere traccia di ogni piastra a 96 pozzetti, un'etichetta univoca o codice a barre dovrebbe essere aggiunto in questa fase o successivamente.
Nota: piastre possono essere preparati da 2 a 3 giorni di anticipo e conservati a 4 ° C, questo può rendere l'organizzazione delle fasi successive più facile.
3. Crescere i cloni RNAi batteriche nei 96 Deepwell piastre LB (cultura durante la notte)
- Per facilitare la successiva manipolazione, quando i cloni originali RNAi librerie sono a 384 pozzetti formato, prima ridistribuire i cloni in etichetta o codice a barre standard di piastre da 96 pozzetti contenenti LB con 100 pg / mL Amp e 12,5 ug / ml Tet integrato con glicerolo (10% concentrazione finale). Quando le lastre originali 384-e non hanno cloni in tutti i pozzetti, come avviene per i più comunemente impiegati libreria "Ahringer", ci sono diverse opzioni per organizzare le piastre figlie. I cloni possono essere ridistribuita in modo che non ci sono pozzi vuoti nelle piastre figlie. Ciò minimizza il numero di piastre di schermo. Idealmente, però, durante la replica, alcuni pozzi su ogni piatto figlia deve essere lasciata vuota, in modo che possano essere successivamente riempito con cloni di controllo standard (positivo e negativo) che facilitano in-piastra confronti. Queste piastre a 96 pozzetti figlia vengono quindi utilizzati per le sementi 96-Deepwell piastre LB per una notte (ON) cultura.
Tutte le fasi di manipolazione di liquidi sono fatto meglio con un sistema robotizzato di manipolazione dei liquidi (per esempio Tecan), ma può essere eseguita manualmente usando, per esempio, un 96-pin replicatore. Se i cloni della libreria RNAi sono già in un formato a 96 pozzetti andare al punto 3.5.
- Incubare le piastre a 96 pozzetti figlia RNAi a 37 ° C con agitazione (200 rpm) per 14-15 ore.
- Verificare che i batteri sono cresciuti correttamente e identificare i cloni che non crescono. Questi saranno esclusi dalle analisi successive. Idealmente, la OD 600 viene misurata in ciascun pozzetto, ma questo può essere fatto con la semplice ispezione.
- Conservare le piastre a 96 pozzetti figlia RNAi a -80 ° C prima dell'uso.
- Scongelare le piastre a 96 pozzetti contenenti il cloni RNAi a temperatura ambiente. Le piastre possono essere poi tenuto a 4 ° C fino all'utilizzo.
- Distribuire 3 pl di ciascun clone batterica da una piastra a 96 pozzetti replicare ai pozzetti 96 della corrispondente etichetta / codice a barre 96-Deepwell piastra LB contenente 1,5 ml di LB con 100 pg / mL Amp e 12,5 ug / ml Tet. Cloni batterici per controllare l'efficienza RNAi, ad esempio un (RNAi) clone GFP nel presente esempio, e controlli negativi, possono essere incorporati in qualsiasi manualmente ben disponibile vuoto.
- Coprire il 96-Deepwell ptardive con una pellicola adesiva (es. film AeraSeal coltura cellulare da Dutscher). Sostituire la piastra a 96 pozzetti utilizzati replica a -80 ° C.
- Le piastre 96-Deepwell marcati vengono poi incubate ON a 37 ° C con agitazione (200 rpm per 14-15 ore).
Giorno 2
4. Seed i cloni RNAi batteriche sui 96 e NGM piastre RNAi
Questo passaggio dovrebbe essere fatto al mattino successivo alla cultura.
- Prendere le piastre a 96 pozzetti NGM da 4 ° C e lasciarli riscaldare ed essiccare per 5-15 min sotto un armadietto a flusso laminare sterile. Non lasciare le piastre troppo a lungo sotto il cofano (massimo 30 min), altrimenti il NGM può successivamente incrinarsi. Aggiungere l'etichetta appropriata / codici a barre per ogni piatto.
- Recuperare il 96 Deepwell ON piastre di coltura da 37 ° C. Registrare le posizioni di eventuali pozzetti vuoti (un pozzo vuoto è completamente trasparente), che saranno esclusi dalle analisi successive.
- Centrifugare il 96-Deepwell piastre 5 min a 4000 rpm.
- Tamponare il surnatante ruotando la piastra capovolta bruscamente e asciugare i bordi della piastra rapidamente su un tovagliolo di carta. Come sono le culture di batteri ricombinanti, il surnatante deve essere trattata in conformità alla normativa vigente (ad es autoclavato).
- Risospendere il pellet batterico del liquido residuo (circa 50 mL) nel vortex, preferibilmente utilizzando un agitatore dedicato (per esempio Tecan) in modo che ciascuna piastra riceve esattamente lo stesso trattamento. È possibile misurare la OD 600 di ogni pozzetto per standardizzare precisamente l'inoculo batterico per la fase successiva, ma ciò non è indispensabile, e non eseguire tale controllo.
- Trasferire 5 pl della batteri risospeso sulle piastre a 96 pozzetti utilizzando un NGM 8 o 12-canali multi-pipetta. Fare attenzione a non toccare o penetrare la NGM. Questo passo viene eseguito manualmente, ma possono essere ottimizzati utilizzando un robot come il Liquidator96 (Rainin) che permettela distribuzione di 96 cloni RNAi in un solo passaggio.
- Lasciate che i batteri a secco sotto un armadietto sterile a flusso laminare, controllando regolarmente per evitare la NGM diventare troppo secca. Questo passaggio dovrebbe prendere circa 2 ore.
- Incubare a 96 pozzetti RNAi-NGM piastre a 37 ° C ON in una camera umida.
5. Preparare una popolazione di vermi sincronizzato
Per questo passo, usiamo vermi transgeniche portanti il gene reporter (s) di interesse (ad esempio un'infezione-inducibile p nlp-29 :: GFP costrutto e, come controllo interno, costitutiva p col-12 :: DsRed transgene costrutto) . A causa della diminuzione osservata di espressione del transgene nel tempo, ci scongelare lotti fresche di vermi ogni 6 settimane. Noi vermi cultura per almeno 2 generazioni prima dell'uso, e garantire che i vermi non hanno mai fame prima del dosaggio. Se il giorno 2 del protocollo è il Martedì, ci prepariamo vermi su Venerdì 30 mettendo vermi adulti giovani su un piatto di 9 centimetri NGM diffuso con OP50 batteria 20 ° C, ma sarà pronto per candeggina il Martedì (vedi Tabella 1).
- Vermi Bleach seguito il protocollo standard 10.
- Lasciate che le uova si schiudono in M9 a 25 ° C ON con agitazione per ottenere una popolazione sincronizzata di larve L1.
Giorno 3
6. Distribuire i vermi sui 96 pozzetti RNAi-NGM piastre per l'alimentazione e RNAi
Questo passaggio dovrebbe essere fatto al mattino. Worm L1-stage sincronizzati sono distribuiti su ogni clone a 96 pozzetti batterica per nutrirsi e per RNAi.
- Recuperare i 96 e RNAi-NGM piastre da 37 ° C e lasciarle a temperatura ambiente per raffreddare.
- Recuperare i vermi sbiancati dal 25 ° C. Stimare il numero di vermi per 2 microlitri e regolare con M9 di avere circa 100-120 vermi ogni 2 pl (circa una goccia).
- Distribuire manualmente 2 microlitri di fase L1-vermi in ogni pozzetto usando un dispenser ripetitivo. Controllare ogni bene per i vermi (dovreste vedere i vermi che nuotano in liquido).
- Lasciare le piastre a secco sotto un armadietto a flusso laminare sterile (massimo 1 ora). Controllare regolarmente le piastre, i vermi devono essere strisciare e non nuotare. Attenzione, l'NGM non deve asciugare troppo, altrimenti si crepa.
- Porre le piastre a 25 ° C in una camera umida.
7. Testare le spore coniospora Drechmeria efficace per l'infezione da
Questo passaggio è specifico per lo schermo attuale. Esso consiste nel testare diversi lotti di spore per selezionare quelli altamente infettivi. Pertanto, deve essere fatto il più vicino possibile prima giorno 4, il giorno di infezione. È necessario preparare sufficienti L3-L4 vermi in anticipo per questi test (Tabella 1). I metodi dettagliati, necessari per D. cultura coniospora sono state descritte altrove 11.
- Raccogliere spore da un campione di ciascuna cultura fungina in un piccolo volume di M9.
- Mettere una goccia di ogni sospensione di spore su un piatto NGM 35 millimetri seminato con OP50, e lasciarlo asciugare.
- Aggiungere 30-40 L3-L4 vermi che portano il gene reporter di interesse (ad esempio p nlp-29 :: GFP).
- Posizionare le piastre infetti a 25 ° C ON.
- Il giorno dopo, controllare i vermi per l'induzione GFP e selezionare il lotto di fungo che dà il più brillante, l'induzione più ampia e omogenea di fluorescenza verde.
4 ° giorno
8. Infettare il worm esposto RNAi con D. coniospora spore
Questa operazione viene eseguita 30 ore dopo RNAi alimentazione quando vermi hanno raggiunto la L3-L4 stadio.
- Determinare il volume di spore di utilizzare per la distribuzione 4 pl per pozzetto.
- Raccogliere fresca D. spore coniospora dal batch selezionato con tampone M9. Uso 8-10 mL di M9 per una lastra 9 cm di spore.
- Distribuisci 4 pl di spore ad ogni pozzetto con un dispenser ripetitivo. Controllare ogni bene per spore (si può vedere il nuoto vermi nel liquido).
- Lasciare asciugare le piastre sotto un armadietto sterile a flusso laminare (~ 1 ora). Controllare le piastre regolarmente fino ai vermi iniziare la scansione. Attenzione, l'NGM non deve asciugare troppo, altrimenti si crepa.
- Collocare le piastre infettate a 25 ° C in una camera umida.
5 ° giorno
9. Osservazione e stoccaggio delle piastre prima automatizzata analisi quantitativa
Questo passaggio avvia 18 ore dopo l'infezione e viene eseguita nel giorno 5. Worms dovrebbero essere i giovani adulti iniziano a deporre le uova. Durante questa fase il fenotipo è segnato visivamente: sia vermi fluorescenza verde che corrisponde ad una situazione normale dopo infezione, o l'induzione di GFP è alterata in un ben determinato che significa che le variazioni del gene silenziato la risposta alle infezioni.
- rapidamente osservare i piatti sotto il microscopio a fluorescenza dissezione. Se le piastre a induzione mostrano una buona GFP generale, quindi procedere alla fase successiva, altrimenti attendere fino al pomeriggio per una induzione migliore, finché c'è ancora cibo lasciato per i vermi.
- Visivamente segnare ogni piatto e annotare ogni bene con un fenotipo marcata (ad esempio non espressione della GFP). Controllando per i risultati ottenuti con i cloni RNAi di controllo che possono essere stati aggiunti, questa prima analisi preliminare offre l'indicazione se l'esperimento ha avuto successo.
- Utilizzando un 8 o 12 canali Multi pipetta di trasferimento i vermi con 100 microlitri di NaCl 50 mM, Triton 0,05%, corrispondente a una nuova etichetta / codice a barre a 96 pozzetti a fondo tondo piatto. Congelare le piastre a -80 ° C fino all'analisi.
- Il giorno dell'analisi, scongelare le piastre a temperatura ambiente e procedere all'analisi automatizzata utilizzando il Biosort COPAS. Il selezionatore analizza una singola lastra in 22 minuti. Le piastre non dovrebbe esserelasciato a temperatura ambiente per più di un'ora prima dell'analisi, e non possono essere congelati nuovamente.
- Le informazioni ottenute dal Biosort COPAS sono trascritti in un file di Excel per la verifica sommaria della qualità dei dati. I dati grezzi con i diversi parametri misurati dalla Biosort COPAS (densità ottica (estinzione), la lunghezza assiale (tempo di volo), le emissioni di fluorescenza, contemporaneamente da tre rivelatori diversi) vengono quindi memorizzate in un pacchetto dedicato LIMS (Mbio LIMS, Modul- Bio) per le successive analisi dettagliate quantitative.
10. Risultati rappresentativi
Usando le quantità di vermi e batteri cui sopra, alla fine dell'esperimento, nella maggior parte dei pozzi, gli animali hanno sviluppato dovrebbero adulta e ci dovrebbe essere ancora qualche cibo lasciato in tutti i pozzetti. In queste condizioni, la maggior parte dei pozzetti dovrebbe anche contenere uova. Ci dovrebbe essere un numero sufficiente di adulti in ciascun pozzetto in modo che i dati sono ottenuti Biosort fo almeno 50 singoli vermi. D'altra parte, se la RNAi è efficiente, un gran numero di cloni RNAi provocherà un fenotipo visibile. Ad esempio, i vermi possono essere scoordinato, o più chiaramente, sterile, o arrestati nel loro sviluppo. Questo dovrebbe verificarsi di frequente, in modo che generalmente almeno un pozzetto per piastra da 96 pozzetti contiene vermi con un fenotipo evidente RNAi. Quando il fenotipo di interesse è l'espressione di un gene reporter GFP, pozzetti con un (RNAi) clone GFP fornire un controllo robusto (Fig. 2). Fenotipi Altri, come una modifica della dimensione può essere facilmente misurata con il Biosort (Fig. 3A). Abbiamo osservato che i vermi potrebbero migrare verso pozzetti adiacenti a frequenza molto bassa (<1%, BS, risultati non pubblicati) quando il cibo in un pozzo ha esaurito.
Abbattendo la funzione di differenti classi di geni che possono influenzare l'espressione transgeni in C. elegans. Alcuni sono non specificamente richiesto per l'espressione transgeni 12. Altri, comecome tessuto-specifici fattori di trascrizione, per esempio il fattore GATA epidermico ELT-3 13, sarà richiesto in particolare sottoinsiemi di cellule. Questo è uno dei motivi per l'inserimento di un non-correlata e costitutiva epidermica costrutto reporter di cella transgene (p col-12 :: DsRed) nel ceppo utilizzato per il protocollo corrente. Questo consente di tali geni di essere distinte da quelle specificamente coinvolti nel processo di gene regolatore in studio (Fig. 3B & C). Il transgene è espresso durante tutti gli stadi larvali. Se un test implica l'espressione genica in embrioni rilevare un diverso gene reporter controllo sarebbe necessaria.
Una delle innovazioni di questo protocollo è l'uso di piastre di congelamento di rinviare la loro analisi. Le piastre di congelamento permette di essere conservati per un massimo di due settimane senza alterare sostanzialmente i risultati. Sebbene i valori assoluti di fluorescenza misurata può essere cambiato, le percentuali relative rimangono simili (Fig. 4). Sul otla sua mano, RNAi è un 14 intrinsecamente variabile procedura sperimentale. Per ottenere risultati robusti ed eliminare i molti potenziali falsi positivi, schermi RNAi è necessario replicare, almeno una volta. Se gli esperimenti sono condotti con successo, ci dovrebbe essere una correlazione ragionevole tra i risultati di una piastra testato in giorni diversi. In particolare, i geni che hanno un forte effetto dovrebbe essere chiaramente identificabili, anche se il loro effetto preciso quantitativo non è identica tra le piastre duplicati (Fig. 5).
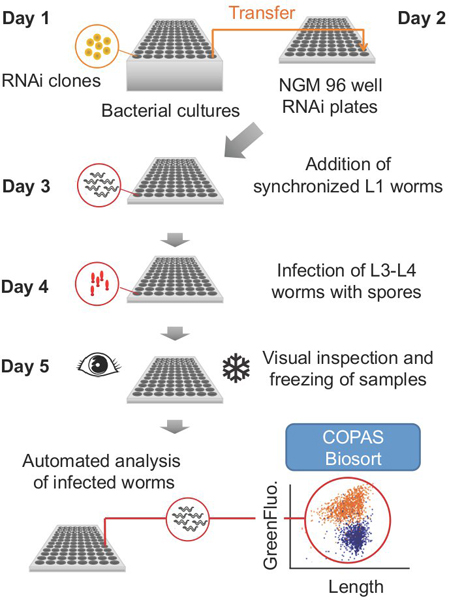
Figura 1. Schema generale per un esperimento tipico.

Figura 2. RNAi di controllo utilizzando un clone di mira l'espressione della GFP. Transgenico vermi espressivong costitutivamente ap col-12 :: DsRed giornalista e un inducibile p nlp 29 :: reporter GFP su terreno solido in una piastra a 96 pozzetti visualizzati utilizzando un microscopio a dissezione GFP con filtro che consente l'osservazione simultanea di fluorescenza rossa e verde. Worms sono stati esposti a controllare (pannelli superiori) o un GFP RNAi clone (pannelli inferiori) per 48 h. I pannelli a sinistra sono vermi infetti, quelli sui vermi giusto 18 ore dopo l'infezione con il D. coniospora. La barra della scala è di 1 mm.

Figura 3. L'analisi quantitativa di una piastra a 96 pozzetti. Il Biosort COPAS genera dati numerici per ogni verme analizzati. I grafici mostrano la deviazione media e standard per il tempo di volo (TOF; A) che corrisponde alla dimensione del vermi 15, fluorescenza rossa (B) e il rapporto (X100) di fluorescenza verde TOF (C) di transgenica worms esprimono costitutivamente col AP-12 :: giornalista DsRed e uno inducibile p nlp 29 :: reporter GFP che erano stati coltivati su mezzo solido in una piastra a 96 pozzetti con differenti cloni RNAi per 48 ore, 18 ore dopo l'infezione con D. coniospora. Alcuni cloni, come quella evidenziata con la freccia in (A), provocano difetti di crescita e / o influenzare l'espressione di p col-12 :: DsRed. Altri, in particolare influenzano l'espressione della GFP, come evidenziato dalla freccia in (C). Questo particolare clone bersaglia il nsy gene 1, in precedenza dimostrato di essere importante per la regolazione della PNL 29 13. Green, fluorescenza rossa e TOF vengono misurate in unità arbitrarie, ma costante.
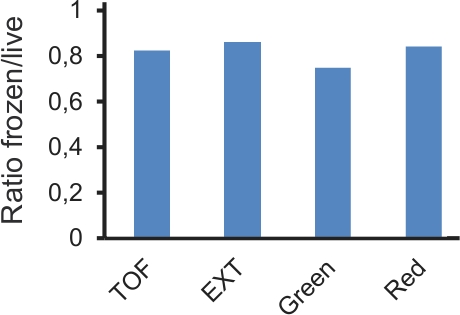
Figura 4. CompArison dei dati ottenuti con campioni dal vivo e congelato lo stesso esperimento. Lastre di L4 vermi che trasportano p nlp-29 :: GFP e col p-12 :: transgeni DsRed sono stati o infettati con D. coniospora o non. Dopo 18h, ogni piastra è stata divisa in 2; metà è stata analizzata immediatamente e mezzo congelato a -80 ° C per 24 ore, prima che lo scongelamento e di analisi. I valori di media TOF (dimensione dei vermi), EXT (densità ottica) e la fluorescenza verde e rosso sono stati calcolati per ogni campione. Il grafico mostra il rapporto della media infettato diviso non infette campioni, per i campioni congelati e vivo (n = 7638 e 5096, e 8634 e 3850 vermi, rispettivamente).

Figura 5. L'analisi comparativa dei duplicati piastre da 96 pozzetti. Il rapporto di fluorescenza normalizzato (il rapporto di fluorescenza media (qui, 100 * verde / TOF) per ciascun pozzetto divisola tagliata (25 ° al 75 ° percentile), si intendono per ogni piastra) per ogni bene da analisi duplicate di un rappresentante della piastra a 96 pozzetti.
| Materiale | Timeline Sperimentale |
| Giovedi
|
Tabella 1. Esempio di un ciclo esperimento con 30 piatti.
Qui viene presentato il materiale necessario per un ciclo unico esperimento di 30 piatti e un calendario per un esperimento dal punto 1 al punto 12 organizzato su 9 giorni.
Nematodi media di crescita (NGM), 100 ml:
| 0.3 g | NaCl |
| 0,25 g | BactoPeptone |
| 2 g | BactoAgar |
| 100 pl | 5 mg / mL di colesterolo in EtOH |
| Aggiungere acqua deionizzata a 100 mL | |
| Autoclave per 5 minuti a 121 ° C e lasciate raffreddare, quindi aggiungere: | |
| 100 pl | 1 M MgSO 4 |
| 100 pl | 1 M CaCl 2 |
| 2,5 mL | 1 M KPO 4 pH 6,0 |
NGM per il trattamento RNAi in piastra a 96 pozzetti, 100 mL:
| 0,29 g | NaCl |
| 0,25 g | BactoPeptone |
| 1,7 g | BactoAgar |
| 100 pl | 5 mg / mL di colesterolo in EtOH |
| Aggiungi de acqua deionizzata a 100 mL | |
| Autoclave per 5 minuti a 121 ° C e lasciate raffreddare, quindi aggiungere: | |
| 100 pl | 1 M MgSO 4 |
| 100 pl | 1 M CaCl 2 |
| 2,5 mL | 1 M KPO 4 pH 6,0 |
| 400 microlitri | 1 M IPTG |
| 100 pl | 100 mg / ml di ampicillina |
| 100 pl | 12,5 mg / mL Tetracycline |
Bleach soluzione, 5 ml:
2,5 mL H 2 O
2,3 mL Bleach
0,2 mL di NaOH 50%
NaOH 50%, 100 mL:
50 g NaOH
Aggiungere acqua deionizzata a 100 mL
NaCl 50mM-Triton 0,05%, 400 ml:
4 mL NaCl 5 M
1 ml di Triton 20%
Aggiungere acqua deionizzata a 400 mL
2 mL Triton X-100
8 mL di acqua deionizzata
M9 tampone, 1L
6 g Na 2 HPO 4 (MW: 178)
3 g di KH 2 PO 4 (PM: 136)
5 g NaCl
Aggiungere acqua deionizzata a 1 L
Autoclavare
Aggiungere 1 mL di MgSO 4 1 M
Luria Broth (LB), 1L:
10 g BactoTryptone
20 g Estratto di lievito
10 g di NaCl
Aggiungere acqua deionizzata a 1 L
Autoclavare
1 M MgSO 4, 300 mL:
73,95 g MgSO 4
Aggiungere acqua deionizzata a 300 mL
Autoclave e conservare a temperatura ambiente
5 mg / Colesterolo mL, 200 mL:
1 g Colesterolo
Aggiungere 100% EtOH a 200 mL
Filtrare sterilizzare e conservare a temperatura ambiente
1 M CaCl 2, 500 mL:
27.75 g di CaCl 2
Aggiungere acqua deionizzata a 500 mL
Filtrare sterilizzare e conservare a temperatura ambiente
1 M KPO 4 pH 6,0, 4 L:
517 g KH 2 PO 4 (PM: 136)
207 g K 2 HPO 4 (MW: 174)
Aggiungere acqua deionizzata a 4 L
100 mg / ml di ampicillina (Amp), 10 mL:
1 g ampicillina
Aggiungere acqua deionizzata a 10 mL
Conservare a -20 ° C
1 M isopropil β-D-tiogalattopiranoside (IPTG), 10 mL:
2,38 g IPTG
Aggiungere acqua deionizzata a 10 mL
Conservare a -20 ° C
12,5 mg / ml di tetraciclina, 100 mL
1,25 g Tetracycline
Aggiungere 100% di EtOH 100 ml
Tabella 2. Solutions.
Discussione
L'attuale protocollo consente su larga scala di screening RNAi in C. elegans Su supporti solidi, utilizzando il livello di espressione di un gene reporter fluorescente come lettura-out. È stato ottimizzato con strumenti automatizzati per facilitare high-throughput saggi, permettendo maneggevolezza e veloce di un gran numero di campioni, in modo efficiente, affidabile e riproducibile. Altri metodi sono stati descritti, che non si basano così pesantemente sulla gestione automatizzata del campione di liquido 8.
Un aspetto fondamentale organizzativo al protocollo è stato lo sviluppo di un metodo affidabile di monitoraggio campione adattando il software commerciale MBioLIMS alla procedura schermo RNAi in collaborazione con la ModulBio azienda. In ogni fase della procedura, le piastre sono ora identificati da codici a barre generati all'interno del LIMS. Come LIMS registra anche l'identità dei cloni in ogni pozzetto su ciascuna piastra, andando da pozzetto a gene putativo è semplice. E di là di questo, i collegamenti LIMS per la WormBase banca dati comunitaria, consentendo l'accesso diretto ai dati molecolari e funzionali in merito a qualsiasi dato gene. Il LIMS facilita anche le analisi da effettuare sul piatto o addirittura a livello dei cromosomi e intero genoma. Per queste analisi, i dati vengono memorizzati in un modo che è perfettamente compatibile con utensili precedentemente descritti come COPAmulti 16.
Un punto critico nel metodo sperimentale è la regolazione della quantità di cibo in relazione al numero di vermi. Per molti saggi, è essenziale che i vermi non fame alla fine dell'esperimento, ma è anche importante non avere troppo cibo all'inizio. Abbiamo osservato che un'alta densità di cibo riduce induzione fluorescenza nel protocollo utilizzando vermi trasportano il p Nlp-29 :: Reporter GFP. Questo può riflettere una diminuita efficienza infezione quando i vermi vengono coltivate nei batteri troppo (BS, risultati non pubblicati). Allo stesso tempo, le condizioni di coltura del batterio RNAi è anche importante, poiché questo determina se dsRNA viene prodotta ad un livello ottimale. Alcuni protocolli per la grande RNAi richiedono cultura di cloni RNAi su piastre di agar prima di semina colture liquide 8. Abbiamo ottenuto il silenziamento genico robusta omissione di questo passaggio. Abbiamo optato per la coltivazione di un eccesso di ogni cultura per garantire risultati omogenei. Il protocollo descritto, infatti, fornisce materiale sufficiente per condurre 2 o 3 schermi in parallelo. Nell'interesse di ottenere i risultati più robusti, siamo favorevoli, però, conducendo schermi in duplicato utilizzando culture indipendenti di vermi, batteri e spore fungine RNAi.
Un altro passo fondamentale è legato all'utilizzo di mezzo che non è né troppo umido né troppo secco. Se le piastre non sono asciutti a sufficienza, i batteri RNAi non asciugare bene e vermi nuotare durante il saggio. Ciò può causare problemi. Ad esempio, i worm non sono efficacemente infettati da D. coniospora In un liquido (C. Couillault, risultati non pubblicati). D'altra parte, in lastre secche, l'osmolarità del aumenti medi di coltura. Ciò ha effetti diretti sulla fisiologia a vite senza fine 17, Compresa l'induzione di alcuni geni peptidi antimicrobici, come ad esempio Nlp 29 18. Il volume utilizzato per erogare i vermi e spore è fondamentale per l'essiccazione efficaci e rapidi. Se più di 10 pl di liquido viene aggiunta alle piastre fresche, i pozzetti può richiedere molte ore per asciugare. Ciò può significare che è difficile da usare metodi robotici per aggiungere vermi alle piastre. Ad esempio, ogni vite senza fine viene erogato dalla Biosort in circa 1 ml di liquido. Al momento distribuire i vermi manualmente che ci permette anche di superare la tendenza di vermi in soluzione a sedimentare.
Abbiamo già segnalato un adeguamento del Biosort che ha permesso uno piastra a 96 pozzetti da analizzare in 36 minuti 19. La macchina fabbricante UBI offre una modifica del Biosort per ridurre ulteriormente il tempo di analisi. Con il "FastReFlex", i campioni vengono indirizzate direttamente attraverso la cella di flusso per l'analisi, bypassando la bolla-trappola filtro. Ciò comporta una riduzione significativa del tempo di acquisizione dati; una piastra intero può essere analizzata in 22 min consentendo circa 20 piastre da trattare al giorno. Se i pannelli non sono state congelate, tuttavia, ci sarebbe un intervallo 8 ore tra i primi e gli ultimi. Data la durata relativamente breve dell'esperimento, con l'infezione dura solo 18 ore, ciò introduce una variabile inaccettabile in piastre. Non solo piastre di congelamento prima dell'analisi risolvere questo problema, ma è anche essenziale nel contesto di high-throughput di analisi che, con l'attuale protocollo e il liquido standard di gestione dei robot, 2 le persone possono facilmente elaborare fino a 50 piastre una settimana. Attualmente, abbiamo regolarmente schermo 30 piastre una settimana, che permette uno schermo intero genomada effettuare una volta in meno di 2 mesi.
Una caratteristica interessante di questo protocollo è il fatto che essa utilizza agar solido, permettendo dosaggi che non possono essere eseguite in coltura liquida. Come fisiologia verme differisce a seconda delle condizioni di coltura 20,21, Essendo in grado di eseguire saggi su supporto solido può anche integrare usuali base liquida schermi. Per alcuni fenotipi movimento (ad esempio), l'analisi può essere effettuata direttamente con 96-well solidi piastre di terreno. Per la schermata corrente, tuttavia, come il Biosort non può campionare da piastre di terreno solido, i vermi vengono prima trasferiti a liquido prima del congelamento. D'altra parte, il Biosort COPAS consente un'analisi multiparametrica quantitativa compatibile con la misurazione di molti diversi fenotipi. È probabile che per ogni dato schermo non tutti i parametri misurati sarà la lettura primaria out. Tuttavia, queste misurazioni possono essere utili per varie ragioni. Essi permettono di determinare se il saggio eseguito correttamente. Ad esempio, si può controllare il numero e la dimensione dei vermi in ciascun pozzetto. Solo quando un clone RNAi dovrebbe bloccare la crescita o la riproduzione dovrebbe esserci uno scostamento dai valori attesi. Wells con meno di 20 vermi o / e degli animali del 30% più piccoli (TOF) rispetto ad una standarddard di controllo con nessun effetto sulla crescita ( Ad esempio, GFP (RNAi)) Può essere escluso dal analisi successive. La funzione di questi cloni possono essere identificati utilizzando un protocollo alternativo in cui i vermi sono esposti ai batteri RNAi dopo il loro sviluppo è in fase avanzata 15. I vari parametri possono anche essere utilizzati per perfezionare gene candidato (hit) selezione, per esempio, limitando riscontro solo a quelle che influiscono fluorescenza verde, ma non rosso. Il metodo preciso prescelte per la selezione riscontro dipenderà dal tipo di schermo e la sua esatta lettura-out. Data la complessità dei dati generati da un punto di genome-wide screen, la collaborazione con un esperto di statistica può essere richiesta.
Il protocollo che descriviamo può essere facilmente adattato ad altri tipi di saggi. Ad esempio, al passo infezione D. coniospora Potrebbe essere sostituita da qualsiasi altro agente patogeno in grado di infettare su supporti solidi. Questo è potenzialmente utile che abbiamo trovato che molti dei fattori di virulenza importanti per l'infezione da Serratia marcescens in coltura liquida non svolgono un ruolo durante l'infezione su terreno solido (E. Pradel, comunicazione personale). Il protocollo è inoltre pienamente compatibile con i test di composti chimici 22-24, O per identificare composti con attività biologica utilizzando diverse librerie di batteri 25, Quindi dovrebbe trovare un programma di utilità generale nel C. elegans Comunità di ricerca.
Divulgazioni
Il laboratorio collabora con l'Unione Ewbank Biometrica e Modul-Bio.
Riconoscimenti
Ringraziamo D. Braendle, CL Kurz e F. Montañana-Sanchis per i loro contributi. Questo progetto è stato sostenuto anche da finanziamenti della ANR e il Conseil Régional PACA e finanziamenti istituzionali da INSERM e il CNRS.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| BactoAgar | BD Diagnostic Systems | 214010 | |
| Ampicillin | Sigma-Aldrich | A9518 | |
| BactoPeptone | BD Diagnostic Systems | 211677 | |
| CaCl 2 | Ogni fornitore | ||
| Pellicola adesiva AeraSeal | Dutscher | 760214 | |
| Isopropil β-D-tiogalattopiranoside (IPTG) | Thermo Scientific | AB-0481 | |
| K 2 HPO 4 | Ogni fornitore | ||
| KH 2 PO 4 | Ogni fornitore | ||
| MgSO 4 | Ogni fornitore | ||
| NaCl | Ogni fornitore | ||
| Na 2 HPO 4 | Ogni fornitore | ||
| NaOH | Ogni fornitore | ||
| Piastra da 96 pozzi profondi | Thermo Scientific | AB-0932 | |
| Piastra a 96 pozzetti piatta | FALCON | 353072 | |
| Piastra a 96 pozzetti rotonda | FALCON | 353077 | |
| Tetracycline | Sigma-Aldrich | T8032 | |
| Triton X-100 | Ogni fornitore | ||
| TECAN robot | TECAN | ||
| Liquidator96 TM | RAININ | ||
| COPAS Biosort | Union Biometrica | ||
| LIMS | Modul-Bio |
Riferimenti
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854-854 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Rual, J. F. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14, 2162-2168 (2004).
- Lee, S. S. A systematic RNAi screen identifies a critical role for mitochondria in C. elegans longevity. Nat. Genet. 33, 40-48 (2003).
- Hamilton, B. A systematic RNAi screen for longevity genes in C. elegans. Genes Dev. 19, 1544-1555 (2005).
- Frand, A. R., Russel, S., Ruvkun, G. Functional genomic analysis of C. elegans molting. PLoS Biol. 3, e312-e312 (2005).
- Parry, D. H., Xu, J., Ruvkun, G. A whole-genome RNAi Screen for C. elegans miRNA pathway genes. Curr. Biol. 17, 2013-2022 (2007).
- O'Rourke, E. J., Conery, A. L., Moy, T. I. Whole-animal high-throughput screens: the C elegans model. Methods Mol. Biol. 486, 57-75 (2009).
- Pujol, N. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Curr. Biol. 18, 481-489 (2008).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1551-8507 (2006).
- Powell, J. R., Ausubel, F. M., Ewbank, J., Vivier, E. Models of Caenorhabditis elegans Infection by Bacterial and Fungal Pathogens. Methods Mol Biol. 415, 403-427 (2008).
- Cardoso, C. XNP-1/ATR-X acts with RB, HP1 and the NuRD complex during larval development in C. elegans. Dev. Biol. 278, 49-59 (2005).
- Pujol, N. Anti-fungal innate immunity in C. elegans is enhanced by evolutionary diversification of antimicrobial peptides. PLoS Pathog. 4, e1000105-e1000105 (2008).
- Simmer, F. Genome-Wide RNAi of C. elegans Using the Hypersensitive rrf-3 Strain Reveals Novel Gene Functions. PLoS Biol. 1, e12-e12 (2003).
- Couillault, C. TLR-independent control of innate immunity in Caenorhabditis elegans by the TIR domain adaptor protein TIR-1, an ortholog of human SARM. Nat Immunol. 5, 488-494 (2004).
- Morton, E., Lamitina, T. A suite of MATLAB-based computational tools for automated analysis of COPAS Biosort data. Biotechniques. 48, xxv-xxx (2010).
- Rohlfing, A. K., Miteva, Y., Hannenhalli, S., Lamitina, T. Genetic and physiological activation of osmosensitive gene expression mimics transcriptional signatures of pathogen infection in C. elegans. PLoS One. 5, e9010-e9010 (2010).
- Lee, K. Z., Kniazeva, M., Han, M., Pujol, N., Ewbank, J. J. The fatty acid synthase fasn-1 acts upstream of WNK and Ste20/GCK-VI kinases to modulate antimicrobial peptide expression in C. elegans epidermis. Virulence. 1, 113-122 (2010).
- Duverger, Y. A semi-automated high-throughput approach to the generation of transposon insertion mutants in the nematode Caenorhabditis elegans. Nucleic Acids Res. 35, e11-e11 (2007).
- Pierce-Shimomura, J. T. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc. Natl. Acad. Sci. U.S.A. 105, 20982-20987 (2008).
- Ruzanov, P., Riddle, D. L. Deep SAGE analysis of the Caenorhabditis elegans transcriptome. Nucleic Acids Res. 38, 3252-3262 (2010).
- Kurz, C. L., Ewbank, J. J. Infection in a dish: high-throughput analyses of bacterial pathogenesis. Curr. Opin. Microbiol. 10, 10-16 (2007).
- Okoli, I. Identification of antifungal compounds active against Candida albicans using an improved high-throughput Caenorhabditis elegans assay. PLoS One. 4, e7025-e7025 (2009).
- Moy, T. I. Identification of novel antimicrobials using a live-animal infection model. Proc. Natl. Acad. Sci. U. S. A. 103, 10414-10419 (2006).
- Ballestriero, F., Thomas, T., Burke, C., Egan, S., Kjelleberg, S. Identification of compounds with bioactivity against the nematode Caenorhabditis elegans by a screen based on the functional genomics of the marine bacterium Pseudoalteromonas tunicata D2. Appl. Environ. Microbiol. 76, 5710-5717 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon