Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Modeling and Imaging 3-Dimensional Collective Zellinvasion
In diesem Artikel
Zusammenfassung
Models of Tumorzellinvasion in dreidimensionale extrazelluläre Matrix besser widerspiegeln In vivo Situation als zweidimensionale Beweglichkeit Assays. Mit Matrix-Invasion Assays mit konfokale Bildgebung von Fluoreszenz-markierten Zellen, detaillierte Informationen über die Invasion Modi und die einzelnen Beiträge zu den führenden im Vergleich zu folgenden Zellen kombiniert erreicht werden kann.
Zusammenfassung
Ein charakteristisches Merkmal von Krebs Malignität Invasion und Metastasierung 1. In einigen Krebsarten (e. g. Gliom 2), lokale Invasion in das umgebende gesunde Gewebe ist die Ursache von Krankheit und Tod. Für andere Krebsarten (e. g. Brust, Lunge, etc.), Wird der Prozess der Metastasierung ist, in die Tumorzellen von einem Primärtumor Masse zu bewegen, zu kolonisieren distalen Seiten und letztendlich zum Organversagen beitragen, dass führt schließlich zu Morbidität und Sterblichkeit 3. Es wurde geschätzt, dass der Invasion und Metastasierung verantwortlich für 90% der Krebstodesfälle 4 sind. Als Ergebnis gab es reges Interesse bei der Identifizierung der molekularen Prozesse und kritische Protein Mediatoren der Invasion und Metastasierung für die Zwecke der Verbesserung von Diagnose und Behandlung 5 gewesen.
Eine Herausforderung für Krebsforscher ist Invasionsassays hinreichend ähneln den in-vivo-Situation zu entwickelngenaue Krankheit Modellierung 6 zu ermöglichen. Zweidimensionale Zellmotilität Assays sind nur informativ über einen Aspekt der Invasion und berücksichtigen nicht die extrazelluläre Matrix (ECM)-Protein Umbau, die auch ein kritisches Element zu nehmen. In jüngster Zeit hat die Forschung unser Verständnis von Tumorzellinvasion verfeinert und gezeigt, dass einzelne Zellen kann durch längliche oder runde Modi 7 zu bewegen. Darüber hinaus hat es eine größere Wertschätzung des Beitrags der kollektiven Invasion in die Zellen eindringen in Stränge, Platten und Clustern, insbesondere in hoch differenzierten Tumoren, die epithelialen Eigenschaften beizubehalten, um die Ausbreitung von Krebs 8 wurden.
Wir präsentieren eine raffinierte Methode 9 für die Prüfung der Beiträge der Kandidaten-Proteine, um kollektive Invasion 10. Insbesondere durch technische separate Pools von Zellen auf verschiedene fluoreszierende Proteine exprimieren, ist es möglich, molekular sezieren die Aktivitäten und Proteins in führenden Zellen im Vergleich zu denen in folgenden Zellen erforderlich erforderlich. Die Verwendung von RNAi stellt die molekulare Werkzeug, um experimentell zu zerlegen die Prozesse in einzelne Zelle Invasion sowie in verschiedenen Positionen der kollektiven Invasion beteiligt. In diesem Verfahren, Mischungen von fluoreszenzmarkierten Zellen auf dem Boden eines Transwell einfügen zuvor mit Matrigel ECM-Protein gefüllt sind vernickelt, dann erlaubt, "nach oben" durch den Filter und in den Matrigel Invasion. Rekonstruktion der z-Serie Bildstapeln durch konfokale Bildgebung erhalten, in dreidimensionale Darstellungen ermöglicht Visualisierung von kollektiv eindringenden Stränge und Analyse der Darstellung von fluoreszenzmarkierten Zellen in führenden gegenüber folgenden Positionen.
Protokoll
1. Retrovirale Markierung von Zellen mit fluoreszierenden Proteinen
- Platte retroviralen Verpackung Zellen (zB Phoenix) bei 0,25 x 10 6 Zellen pro Vertiefung einer 6-well Gericht in 10% fötalem Rinderserum (FBS) / DMEM.
- Transfektion von Zellen mit retroviralen DNA 48 Stunden später Zellen mit Effectene nach den Anweisungen des Herstellers.
- Spülen Sie die Taschen zweimal mit Medium 24 Stunden später, dann fügen Sie 1,5 ml 10% FBS / DMEM pro Loch.
- Sammeln verpackt Virus in Zellkulturmedium 48 Stunden später durch Pipettieren und die Übertragung auf 2 mL Mikrozentrifugenröhrchen.
- Zentrifugation bei 1600 rpm für 5 min auf Pellet alle Zellen.
- Entfernen Sie den Überstand in ein sauberes Röhrchen und lagern bei -80 ° C.
- Cells um retroviral transduziert werden werden mit 1,5 x 10 5 Zellen pro Loch einer 6-Well Schale überzogen und in einem befeuchteten Inkubator bei 37 ° C über Nacht.
- Am nächsten Tag, entfernen Sie die Medien aus den Zellen und add 1 ml Virus-Aktie mit 4 ul Polybren (2 mg / ml) in jede Vertiefung ergänzt. Ersetzen Platten im Brutschrank.
- Nach Inkubation für 5-6 Stunden, 2 ml 10% FBS / DMEM in jede Vertiefung und Platten sind in 37 ° C ersetzt inbubator über Nacht.
- Am nächsten Tag, ersetzen Sie die Medien und damit Zellen 24 Stunden vor dem Zusatz von geeigneten selektiven Medien.
- Wenn konfluent (das hängt von der Wachstumsrate der Zellen, und die Effizienz der retroviralen Transduktion, sondern nimmt in der Regel 4 bis 14 Tage), trypsinize Zellen und sort für die Fluoreszenz unter Verwendung von nicht-transduzierten Zellen als Referenz, zu sammeln und Pool-Zellen mit Fluoreszenz größer als nicht-transduzierten Zellen und frieren Aliquots bei -80 ° C für eine spätere Verwendung.
2. Inverse Matrigel Invasion Assays
- Langsam tauen ein Aliquot von kompletten Matrigel (dh mit Wachstumsfaktoren) auf Eis (Abbildung 1A).
- Nach dem Auftauen, verdünnter Matrigel 1:1 in eiskaltem PBS (zusammen mit allen anderen zusätzlichenBehandlungen mit 2x-Konzentration in der PBS vor der Verdünnung; Abbildung 1B). Um es einfacher zu Matrigel vor der Polymerisation Griff, alle Kunststoffprodukte (zB Tipps, Rohre, etc.) sollte eiskalt sein.
- Legen Sie so viele 8 Mikron Porengröße 6,5 mm Durchmesser unbeschichteten Transwells als in den Vertiefungen einer 24-Well-Gewebekulturplatten erforderlich, dann vorsichtig Pipette 100 ul der verdünnten Matrigel in die Vertiefungen und Urlaub für ~ 30 min bei 37 ° C inkubieren sich zu verfestigen (Abbildung 1C).
- Während dieser Zeit bereiten eine oder mehrere fluoreszenzmarkierten Zellsuspensionen auf zwischen 1 bis 4 x 10 5 Zellen pro ml, je nach Zelllinie, aus jedem vorbehandelten Zustand (e. g. SiRNA, medikamentöse Behandlung) in ihr normales Wachstum Medium (Abbildung 1D).
- Wenn die Matrigel erstarrt ist, kehren die Transwells und Pipette 100 ul der Zellsuspension auf der nach oben weisenden Unterseite des Filters (Abbildung 1E).
- Sorgfältig decken die Transwells mit der Basis eines 24-Well-GewebeKultur Teller, den Kontakt mit jedem Tropfen der Zellsuspension (Abbildung 1F).
- Die Platte in den invertierten Zustand für 4 Stunden für Zelladhäsion (Abbildung 1G) zu ermöglichen.
- Nach dieser Zeit werden die Platten right-side-up und waschen jedes Transwell durch sequentielle Eintauchen in 2 x 1 ml serumfreiem Medium (Abbildung 2A).
- Verlassen Sie die Transwells in einem dritten auch mit Drogen oder Behandlungen erforderlich (Abbildung 2B).
- Gently Pipette 100 ul 10% FBS / DMEM plus Lockstoff (e. g. EGF bei 25 ng / ml) in die Transwell auf der Oberseite des erstarrten Matrigel / PBS-Mischung, ersetzen Sie den Deckel ab und inkubieren für 3-5 Tage bei 37 ° C mit 5% CO 2 (Abbildung 2C).
3. Färbung und Visualisierung
- Zellen, die fluoreszierende Proteine können die folgenden Schritte vor der konfokalen Mikroskopie überspringen
- Um das Bild nicht fluoreszierenden Zellen eindringen Matrigel, Ort Transwells in frischen 24-Well-Gerichte und Pipette 1 ml 4 uMCalcein AM Färbelösung übereinander Matrigel-Stecker, so dass sie übergreifen den Seiten und Flecken von oben und unten. Calcein AM (Acetoxymethylester von Calcein) ist ein Live-Cell-Farbstoff, färbt die ganze Zellen grün und erfordert keine Fixierung.
- Inkubieren 1 Stunde bei 37 ° C in 5% CO 2 befeuchteten Atmosphäre, an welcher Stelle Zellen sind voll gefärbt und bereit, durch konfokale Mikroskopie abgebildet werden.
- Alternativ können nicht-fluoreszierenden Zellen eindringen Matrigel fixiert und gefärbt werden, wie unten in 3,5 bis 3,9 beschrieben.
- Übertragen Sie jede Transwell einen frischen 24-Well-Platte. Overlay 1 ml 4% para-formaldehyde/0.2% Triton-X 100.
- Inkubieren bei Raumtemperatur für 0,5 Stunden.
- Entfernen Fixiermittel und 3 x waschen mit 1 ml PBS.
- Entfernen zytoplasmatische RNA mit 30 min RNase-Behandlung mit 100 pg / ml RNase. Wash 2 mal mit PBS.
- Für die Visualisierung hinzu 0,01 mg / ml Propidiumiodid (PI) in PBS verdünnt und lassen bei Raumtemperatur in den dark für 0,5 Stunden. 3 x waschen mit PBS. In diesem Stadium, gebeizt PI Transwell bei Raumtemperatur im Dunkeln für mindestens 1 Monat aufbewahrt werden.
- Präzise bildgebenden Verfahren durch konfokale Mikroskopie sind abhängig von verfügbaren Ressourcen. Mit Hilfe eines invertierten konfokalen Mikroskop, Ort Transwell mit einer kleinen Menge an Rest-PBS auf große Deckglas (sicherstellen, dass keine Luftblasen vorhanden sind) über nicht-Immersions-20 X objektiv und erfassen optische Schnitte alle 10-15 um von der Unterseite der Matrigel-Plug. Individuelle optische Schnitte können verwendet werden, um das Ausmaß der Invasion (Abbildung 3A) zu quantifizieren oder in 3-dimensionale Objekte mittels geeigneter Software wie Volocity (Abbildung 3B) zu bauen.
4. Repräsentative Ergebnisse
Ein Beispiel für eine Z-Serie von optischen Schnitten ist in Abbildung 3A gezeigt. In diesem Fall wurden die Zellen mit Calcein AM angefärbt und die Anzahl der Zellen eindringen aus dem Filter zu sehen, mit Abstand zu verringern. Die Quantifizierung der invasion kann durch die Analyse des Verhältnisses von Calcein AM positive Pixel, negative Punkte bei jedem Intervall, oder über die Fixierung / Färbemethode oben beschriebene Zählen PI positiven Kerne an jeder Position möglich. Ein Vorteil von Calcein AM Färbung ist, dass 3-dimensionale Rekonstruktion der Zellinvasion zusammengebaut mit einer Software wie Volocity, was eine visuelle Darstellung der Modus der Invasion (Abbildung 3B) werden. Wenn Zellen durch die Expression von fluoreszierenden Proteinen markiert sind, dann die Positionen der einzelnen Farben Zelle kann in 3-dimensionale Rekonstruktion visualisiert, entweder von der Seite (Abbildung 3C) oder durch Schnitte durch die Rekonstruktion (Abbildung 3D) angesehen.
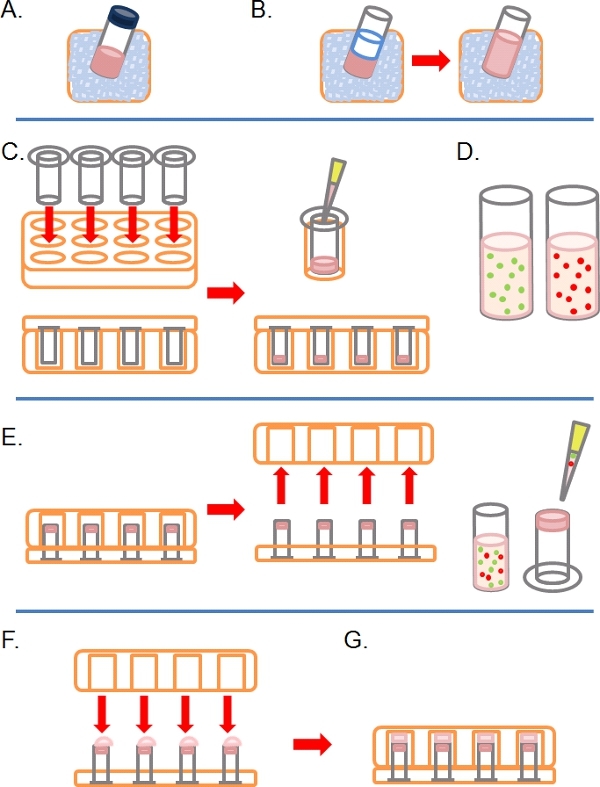
Abbildung 1. Schematische Darstellung der Schritte beim Aufbau einer inversen Invasion Assay beteiligt. A) Matrigel ECM auf Eis aufgetaut. B) Matrigel ist 1:1 mit PBS, das jede medikamentöse Behandlung mit der doppelten Endkonzentration verdünnt. C) Transwell-Einsätze sind in Mikrotiterplatten gegeben und Matrigel in jede pipettiert. D) Zellsuspensionen hergestellt in der gewünschten Konzentration. E) Nach dem Matrigel gesetzt hat, die Platte umgedreht ist und entfernt werden, werden die Zellen auf die Unterseite Filter der Transwell-Einsätze vernickelt. F) In der invertierten Position ist das mehrlagig Platte vorsichtig über Transwell-Einsätze platziert, in Kontakt mit der Zellsuspension. G) Zellen werden dürfen, um den Filter für 4 Stunden einzuhalten.
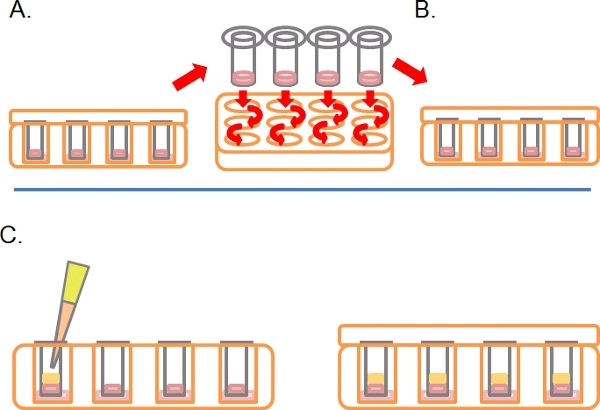
Abbildung 2. Fortführung der Schaltplan für die inverse Invasion Assay. A) Wenn Zellen eingehalten haben, tauchen jede Transwell in serum-freien Medien zweimal zu verlieren Zellen zu entfernen. B) Ort gewaschen Transwell in eine endgültige und mit Medien sowie Behandlungen nach Bedarf. C) Medien mit Lockstoff (zB 10% fötalem Rinderserum) mit Behandlungen nach Bedarf wird vorsichtig auf Matrigel geschichtet.
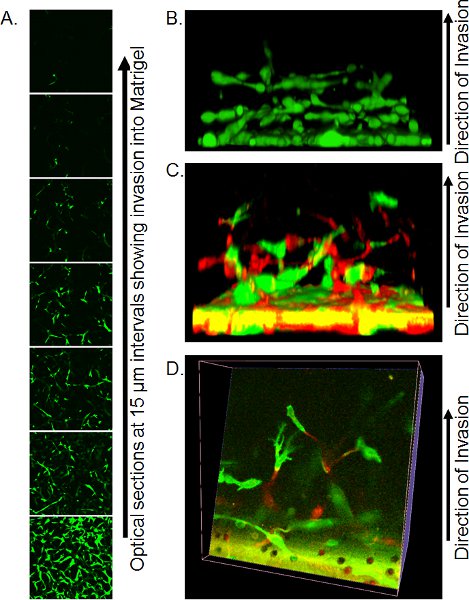
Abbildung 3. Repräsentative Bilder der Ergebnisse der inversen Invasion Assay. A) Optische Schnitte von Zellen mit Calcein gefärbten AM eindringenden Matrigel, aufgenommen bei 15 mu m Abständen durch konfokale Mikroskopie. B) Rekonstruktion eines 3-dimensionalen Rekonstruktion der Zelle Invasion aus einem Stapel von konfokalen Z-Serie Bilder von der Seite angesehen. Wiedergabe aus Lit. 10. C) Dreidimensionale Rekonstruktion des GFP und RFP-markierter Zellen eindringen Matrigel von der Seite betrachtet. D) Optischer Schnitt durch die 3-dimensionale Rekonstruktion der GFP und RFP-markierter Zellen. Wiedergabe aus Lit. 10.
Diskussion
Matrigel Invasion Assays wurden traditionell mit Zellen auf eine Schicht aus extrazellulären Matrix-Proteins mit Lockstoff-induzierten Motilität in Richtung und durch einen Filter am unteren Rand platziert gesetzt. Invasivität wurde als Funktion der, wie viele Zellen könnte auf der Unterseite des Filters gezählt werden gewertet. Obwohl praktisch gibt es kaum einen Unterschied mit dem "inverse" Invasion Assay beschrieben, gibt es wesentlich mehr Informationen über den Prozess der Invasion, die durch die V...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Finanzierung dieser Forschung ist von Cancer Research UK.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
|---|---|---|---|
| DMEM (Dulbecco modifiziertem Eagle-Medium) | GIBCO | 21969 | |
| Fetal Bovine Serum | PAA | A15-101 | |
| Penicillin Streptomycin | GIBCO | 15140 | |
| 200 mM L-Glutamin (100x) | GIBCO | 25050-032 | |
| Puromycin | Sigma-Aldrich | P8833 | |
| 0,05% Trypsin EDTA | GIBCO | 25300 | |
| Polybrene | Sigma-Aldrich | AL-118 | |
| Lipofectamine 2000 Reagent | Invitrogen | 11668019 | |
| 6.5mm Transwells, 8,0 um Porengröße | Corning | 3422 | |
| Komplette Matrigel | BD Biosciences | 354234 | |
| Calcein AM | Invitrogen | C1430 | |
| RNase | Qiagen | 19101 | |
| Propidiumiodid | Sigma-Aldrich | P4864 | |
| Konfokale microcope | Leica | SP2MP |
Referenzen
- Olson, M. F., Sahai, E. The actin cytoskeleton in cancer cell motility. Clin. Exp. Metastasis. 26, 273-287 (2008).
- Hoelzinger, D. B., Demuth, T., Berens, M. E. Autocrine factors that sustain glioma invasion and paracrine biology in the brain microenvironment. J. Natl. Cancer. Inst. 99, 1583-1593 (2007).
- Chaffer, C. L., Weinberg, R. A. A perspective on cancer cell metastasis. Science. 331, 1559-1564 (2011).
- Hanahan, D., Weinberg, R. A. The Hallmarks of Cancer. Cell. 100, 57-70 (2000).
- Francia, G., Cruz-Munoz, W., Man, S., Xu, P., Kerbel, R. S. Mouse models of advanced spontaneous metastasis for experimental therapeutics. Nat. Rev. Cancer. 11, 135-141 (2011).
- Hooper, S., Marshall, J. F., Sahai, E. Tumor cell migration in three dimensions. Methods. Enzymol. 406, 625-643 (2006).
- Croft, D. R., Olson, M. F. Regulating the conversion between rounded and elongated modes of cancer cell movement. Cancer Cell. 14, 349-351 (2008).
- Wolf, K. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell. Biol. 9, 893-904 (2007).
- Hennigan, R. F., Hawker, K. L., Ozanne, B. W. Fos-transformation activates genes associated with invasion. Oncogene. 9, 3591-3600 (1994).
- Scott, R. W. LIM kinases are required for invasive path generation by tumor and tumor-associated stromal cells. J. Cell. Biol. 191, 169-185 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten