Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Einfache Methode zur Fluoreszenz DNA
In diesem Artikel
Zusammenfassung
Here, we present a simple method for performing fluorescence DNA in situ hybridization (DNA ISH) to visualize repetitive heterochromatic sequences on slide-mounted chromosomes. The method requires minimal reagents and it is versatile for use with short or long probes, different tissues, and detection with fluorescence or non-fluorescence-based signals.
Zusammenfassung
DNA in situ Hybridisierung (ISH DNA) ist ein häufig verwendetes Verfahren zum Abbilden von Sequenzen an spezifischen Chromosomenregionen. Dieser Ansatz ist besonders effektiv bei der Kartierung hoch repetitive Sequenzen an heterochromatischen Regionen, in denen Rechenansätze gegen prohibitiv Herausforderungen. Hier beschreiben wir eine optimierte Protokoll für DNA ISH, die Formamid wäscht die Standardschritte in anderen DNA ISH Protokolle umgeht. Unser Protokoll zur Hybridisierung mit kurzen Einzelstrang-DNA-Sonden, die Fluoreszenzfarbstoffe, die effektiv zu markieren repetitive DNA-Sequenzen in hetero chromosomalen Regionen in einer Anzahl verschiedener Insektengewebetypen durch optimiert. Jedoch können Anwendungen erweitert werden, um mit größeren Sonden und Visualisierung von Einzelkopie (nicht-wiederkehrende) DNA-Sequenzen zu verwenden. Wir zeigen, diese Methode durch die Abbildung verschiedene repetitive Sequenzen zu gequetscht Chromosomen von Drosophila melanogaster Nervenzellen und Nasoniavitripennis Spermatozyten. Wir zeigen Hybridisierungsmuster für beide klein, kommerziell synthetisiert Sonden und für eine größere Sonde zum Vergleich. Bei diesem Verfahren wird einfach Laborbedarf und Reagenzien, und ist ideal für Ermittler, die wenig Erfahrung mit der Durchführung DNA ISH haben.
Einleitung
DNA in situ Hybridisierung (ISH DNA) ist ein häufig verwendetes Verfahren zum Abbilden von Sequenzen an spezifischen Chromosomenregionen. Sonden an einzelne Kopie Regionen innerhalb Euchromatin kann durch eine Handvoll von Ansätzen, darunter Nick-Translation oder Endmarkierung langer DNA-Produkte 1,2 und den Einbau von deoxygenin (DIG) -attached Nukleotide und ihre Anerkennung durch eine Vielzahl von erzeugt werden Gruppe-konjugierte Antikörper 1-3. Visualisierung der euchromatischen Sequenzen in wenigen oder einzelnen Kopienzahl erfordert die Verwendung von entweder einzelne große Sonden mit hoher spezifischer Aktivität oder einen Cocktail aus mehreren, kleineren Sonden, die gemeinsam zu verbessern Signal.
Im Gegensatz dazu hoch repetitive Sequenzen in heterochromatin gefunden, wie Satelliten-DNAs, sind leichter Ziele für DNA ISH weil sie existieren normalerweise als zehn bis Tausende von Wiederholungen im einzelnen Chromosomenregionen als Blöcke bekannt geclustert. Transposons können auchbei hohen Kopienzahlen an bestimmten chromosomalen Loci 2 gefunden. In diesen Fällen können einzelne Sonden mit geringer spezifischer Aktivität effektiv beschriften hetero Sequenzen aufgrund ihrer Hybridisierung an mehreren Standorten. Sonden an repetitive Sequenzen können im Handel als kurze Oligonukleotide (30-50 bp) synthetisiert und mit einer der mehreren verschiedenen fluoreszierenden Gruppen chemisch konjugiert. Mapping repetitiven Sequenzen im Heterochromatin durch Verwendung genom Sequenziertechnologien ist schwierig aufgrund der Herausforderungen im Gebäude Gerüste in stark repetitiven Satelliten Blöcke 4-6,7 gestoßen. Derzeit steht ISH als die effektivste Möglichkeit, die Zuordnung dieser Sequenzen am Unterchromosomenebene. Diese Strategie ist wichtig für die Zuordnung einer großen Anzahl von repetitiven Sequenzen, die von laufenden Genom und Transkriptom-Sequenzierung Studien aufgedeckt werden.
Die Effizienz und einfache Zuordnung repetitiven Sequenzen auf Objektträger angebrachten Chromosomen würde gre werdenatly durch ein vereinfachtes Protokoll für DNA ISH verbessert. So können bereits vorhandene Protokolle für DNA ISH beinhalten mehrere Wäschen hybridisiert Gewebe in Formamid-Lösung 2,8, somit im wesentlichen auf die erforderliche Zeit für die Zuordnung von Sequenzen hinzufügen und auch große Mengen chemischen Abfalls für diese teuren Reagenz. Hier beschreiben wir eine geänderte DNA ISH Verfahren, das die Notwendigkeit für Formamid Wäschen umgeht und setzt grundlegende Laborgeräten und Reagenzien. Dieses Verfahren wurde ursprünglich für die rasche Kartierung hoch repetitive DNA-Sequenzen in heterochromatischen Regionen Drosophila Larvenneuroblasten mit handels synthetisierte Oligonukleotide, die mit Fluoreszenzfarbstoffe konjugiert sind entworfen. Allerdings funktioniert diese Methode auch für die Abbildung repetitiven Sequenzen durch den Einsatz größerer Sonden durch andere Mittel 9,10 und über mehrere verschiedene Gewebe und Chromosomentypen synthetisiert. Zusätzlich kann dieses Verfahren verwendet werden, um euchromatic Sequenzen mithilfe länger oder multip abzubildenle, kurze Sonden innerhalb der euchromatischen Sequenz von Interesse.
Protokoll
1. Die Gewebe Dissection und Fixation (60 min)
- Für Drosophila Gehirn legen 3. Larvenstadium in einem Tropfen 1x PBS (phosphatgepufferte Salzlösung). Wählen Sie große 3. Larvenstadium, die aktiv kriechen aus Ampullen oder Flaschen, die nicht überfüllt sind.
- Verwenden Sie einen ultrafeinen Pinzettenpaar zu halten den Mund Haken und andere Pinzettenpaar zu greifen zwei Drittel über die gesamte Länge des Körpers (1A, B) zu greifen. Ziehen Sie vorsichtig an den Mund Haken, um das Gehirn, ventralen Ganglien, Speicheldrüsen und ein Teil der Larvenverdauungstrakt aus. Mit der Pinzette, um das Gehirn und ventralen ganglia trennen (1A, B) von den anderen Geweben und in einem Tröpfchen 1x PBT (phosphatgepufferte Kochsalzlösung mit Tween) auf einer Kunststoff-Petriplatte.
- Für Nasonia Hoden Dissektionen, wählen Männchen 3 Tage alten Puppen (gelber Körper mit roten Augen). Männlich Nasonia haben kleine Flügelblocklängen in Bezug auf females im Puppenstadium (1C).
- Halten Sie die Puppe an der Spitze des Bauches in der Nähe des Brustbereich mit einer Pinzette Paar, und mit dem anderen Paar Pinzette, greifen die sehr distalen Spitze des Bauches, und ziehen Sie die tropfenförmigen Hoden (sie werden von Fett umgeben sein Körper, die leicht geschüttelt werden kann weg; 1D). Trennen Sie alle Karosserieaußenteile aus den Hoden, die richtige Quetschen, und in einem Tropfen 1x PBT auf einer Kunststoffpetrischale verhindert.
- Für jede Folie, sezieren insgesamt vier oder fünf Gewebeproben (dh., Drosophila Larven Gehirn oder Nasonia Hoden).
HINWEIS: Mehr als fünf Proben werden überfüllt Kerne und Chromosomen führen. - Optional, einige Trennung von Schwesterchromatiden in euchromatischen Regionen der mitotischen Chromosomen erreichen, behandeln das Gewebe mit einer hypotonischen Lösung: Transfer Gehirne von 1x PBT einem Rückgang von 0,5% Natriumcitrat 5-10 (nicht mehr als 10) min.
HINWEIS: Colchicin (a Mitoseinhibitor) kann hilfreich für die Erhöhung der Anzahl von Mitosen auf Metaphase 11 sein. Jedoch ist seine Verwendung nicht erforderlich, wenn sehr große gesunde Larven werden verwendet, und sogar unerwünscht, da sie sich negativ auf das Chromosom Auflösung, Ausbreiten und Morphologie. - Platzieren eines Tröpfchens (~ 20 & mgr; l) der Fixierungslösung (2,5% Paraformaldehyd in 45% Essigsäure) auf die Oberfläche einer sauberen Sigmacote behandelten Deckgläschen.
HINWEIS: Stellen Sie Fixierlösung frisch für jeden Tag der Nutzung. Fixative Lösungen, die 1,8 bis 3,7% Paraformaldehyd in 45% Essigsäure die besten Ergebnisse für die Fische. Die Verwendung anderer als Verstand oder Hoden Gewebe oder Anpassung dieses Protokoll für Immuno-FISH kann verlangen, das Experimentieren mit verschiedenen Fixiermitteln (für eine Liste von verschiedenen Fixiermittel, siehe 12). - Jede Gewebeprobe aus dem Seziersaal Puffers (1x PBT) vorsichtig in die Fixierungstropfens über mit ultrafeinen Pinzette, minimizing die Übertragung von Sezieren Puffer in die Fixierlösung. Positionieren der Gewebeproben, so dass sie gleichmäßig voneinander im Fixiermittel Tröpfchen beabstandet. Das Gewebe in Fixativ Inkubieren für 4 min bei Raumtemperatur.
- Vorsichtig ein Poly-Lysin-beschichteten Objektträger nach unten, auf das Gewebe und Deckglas. Nicht nach unten drücken zu diesem Zeitpunkt nicht, sondern ermöglichen die beiden auf die leichte Schulter zu kontaktieren, damit das Deckglas klebt an der Unterseite des Schlittens. Kehren Sie die Rutsche so dass das Deckglas nach oben zeigt.
- Sandwich die Folie / Gewebe / Deckglas in einem gefalteten Stück Filterpapier. Auf eine stabile Oberfläche, mit dem Daumen drücken ganz fest gerade nach unten auf die Position direkt über dem Deckglas. Achten Sie darauf, seitliche Verrutschen des Deckglases (dies führt dazu, dass ein Verschmieren des Gewebes).
- Tauchen Sie den Schiebe / Gewebe / Deckglas in flüssigen Stickstoff (siehe Gerät in 1E, F), und stehen lassen, bis die Stickstoff stoppt Kochen (mehr ist fin). Entfernen Sie die Folie und sofort abknicken das Deckglas mit einer frischen Rasierklinge über einen Schiebe Ecke des Deckglas nach oben (nicht zerkratzt fixiertem Gewebe mit der Rasierklinge).
- Vorkühlung des Schiebe- / Gewebe / Deckglas auf einem Block von Trockeneis, Abdeckung nach oben rutschen, für 1-2 Minuten vor dem Eintauchen in flüssigen Stickstoff wird verhindert, dass die Folien vor dem Knacken.
- Sofort den Objektträger mit Gewebe in eine Coplin-Gefäß mit 100% Ethanol bei Raumtemperatur gefüllt und lassen Sie stehen für mindestens 5 min (diese Zeit kann länger sein, wenn erforderlich).
HINWEIS: kaltem 100% Ethanol ebenfalls verwendet werden könnten. - Entfernen Sie die Folie mit dem Gewebe, Docht überschüssige Ethanol mit einem Kimwipe (ohne Berührung der festen Gewebe), und lassen Sie die Folie an der Luft trocknen für 1 Stunde.
- Gehen Sie direkt zu Schritt 2 oder Folien halten trocken in geringer Luftfeuchtigkeit Luft oder in einer Trocknungskammer für Wochen, sogar Monate vor der Durchführung der Hybridisierung.
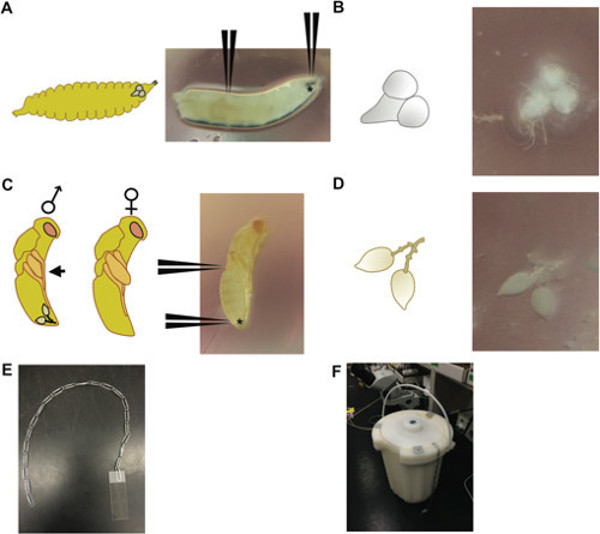
Abbildung 1: (A) A 3. Larvenstadium Drosophila Larve (rechts), mit Positionen, wo die Mundhaken greifen angezeigt (gekennzeichnet mit *) und 2/3 der Weg nach unten die Larven Gehirn sezieren; (Links) eine schematische Darstellung einer Larve eine der gleichen Entwicklungsstufe, der Darstellung der relativen Position des Gehirns im Larvenkopf. (B) Das Gehirn und ventralen Ganglien von einem 3. Larvenstadium Drosophila Larve (rechts) und eine schematische Darstellung dieses seziert Gewebe (links). (C) 3 Tage alten Nasonia Puppe an der Gelbkörper-Rote-Augen-Bühne. (D) Ein Paar Hoden von einem 3 Tage alten Nasonia männlichen Puppe (rechts) seziert mit Positionen anzeigt, wo die Puppe greifen am hinteren Teil des Abdomens (bezeichnet mit einem *) und in der Mitte auf dem Körper; (Links) Schema, männlich und weiblich warp Puppen; männliche Puppen durch Flügel, die nicht an der sagittalen Profil (schwarzer Pfeil) erstrecken, im Gegensatz zu den Weibchen, die Flügel, die hinter dem Profil erstrecken unterschieden werden; die relative Position des Hodens Paar wird im männlichen Puppe (E) Das Gerät-ein String von Büroklammern-und (F) Verfahren verwendet werden, um Folien in einem Behälter mit flüssigem Stickstoff getaucht gezeigt.. Mehrere Büroklammer-Strings können zur gleichzeitigen Eintauchen der mehrere Folien verwendet werden.
2. In situ-Hybridisierung (30 min am Tag 1; 1 h & ndash; 2,5 h bei langen Sonden-am Tag 2)
- 1 ul (100 ng) jeder Sonde zu 20 ul 1,1x Hybridisierungspuffer. Pipettensonde / Hybridisierungspuffer auf die Oberfläche der festen Gewebe (berühren Sie nicht die Gewebe).
- Vorsichtig ein Deckglas direkt auf die Sonde / Hybridisierungspuffer, um sicherzustellen, dass das Deckglas wird direkt über das Gewebe zentriert. Der Puffer sollte die Migration zu thE äußeren Rand des Deckglases, so dass keine Luftblasen.
HINWEIS: Kleine Blasen, die nicht in Verbindung treten Gewebe keine Probleme mit dem Verfahren stellen. Entfernen Sie große Blasen durch vorsichtiges Anheben einer Ecke des Deckglases und sorgfältig fallen sie zurück auf die Folie. - Legen Sie das Dia / Gewebe / Deckglas auf die Oberfläche eines Blocks zu 95 ° C vorgewärmt (Deckglas nach oben). Deckel mit einem großen Stück Alufolie auf Belichtung zu verhindern. Lassen der Schieber Inkubieren bei 95 ° C für 5 min.
HINWEIS: Eine typische Wärmeblock mit Bohrungen für Rohre können umgedreht werden, um eine ebene Fläche, auf der die Folie / Gewebe / Deckglas legen werden. - Entfernen Sie die Folie und lassen ihn etwas abkühlen lassen, bis es warm anfühlen wird. Ein Stück Parafilm gestreckt sorgfältig einzuwickeln um das Deckglas, um die Flüssigkeit unter ihm zu versiegeln.
- Das verschlossene Rutsche in einer feuchten Kammer und legen Sie die Kammer in einem Inkubator auf 30 ° C vorgewärmt. Inkubiere bei 30 ° C für 4 h bis über Nacht.
- Erstellen Sie eine Feuchtigkeitskammer von einer leeren Spitze Box oder Deckeltupperdose mit angefeuchteten Kimwipes oder Papiertücher an der Unterseite angebracht.
HINWEIS: Einzelstrang-DNA-Oligo-Sonden wurden für 28-33 Basen, um eine theoretische Schmelztemperatur (T m) von 45-47 ° C zu erreichen. Diese Länge und T m Bereiche spiegeln die Tatsache wider, dass viele repetitive Sequenzen, die wir studiert haben, sind AT-reichen und damit sehr niedrige GC-Gehalt. Längere Sonden werden wahrscheinlich höhere T m -Werte; Diese können erhöhte Hintergrund-Hybridisierung auf dem Standard-Hybridisierungstemperatur von 30 ° C führen. So können einige Fehlersuche mit Hybridisierungstemperaturen erforderlich, um die besten Ergebnisse zu erzielen. Um die beste Hybridisierungstemperatur, Zunahme (oder Abnahme) die Temperatur um 5 ° C zu finden, inkrementell.
- Erstellen Sie eine Feuchtigkeitskammer von einer leeren Spitze Box oder Deckeltupperdose mit angefeuchteten Kimwipes oder Papiertücher an der Unterseite angebracht.
- Entfernen Sie vorsichtig den Parafilm von der Folie und dann vorsichtig die Deckglas durch langsames Anheben einer Ecke. Wash der Schlitten drei Mal für je 15 min in 0,1 × SSC Waschpuffer. Decken Sie die Coplin-Gefäß mit Aluminiumfolie in den Waschanlagen, um Belichtung zu minimieren.
- Wenn nicht mit einem langen biotinylierte Sonde, fahren Sie mit Schritt 2.8.
- Wenn Sie eine lange biotinylierte Sonde, trocknen Sie den Bereich um das Gewebe mit einer Kimwipe, man aufpassen, das Gewebe selbst nicht zu berühren. Zeigen 100 l Blocking-Lösung über das Gewebe und die Abdeckung vorsichtig mit einem Deckglas, wobei darauf zu Fangblasen zu vermeiden. Wickeln Sie die Folie über das Deckglas mit Parafilm und bei 37 ° C für 30 min.
- Entfernen Sie vorsichtig das Deckglas und tupfen um das Gewebe mit einer Kimwipe. Je 100 & mgr; l von Rhodamin-Avidin, 1: 1000 in SBT über das Gewebe und die Abdeckung vorsichtig mit einem Deckglas, wobei darauf zu achten Fangblasen. Wickeln Sie die Folie über das Deckglas mit Parafilm und bei 37 ° C für 30 min.
- Entfernen Sie vorsichtig das Deckglas und waschen Sie die Folie 3 Mal für jeweils 5 min in 4x SSCT- und dann3-mal für jeweils 5 min in 0,1 x SSC.
HINWEIS: Folien können für längere Zeiträume gewaschen werden.
- Entfernen Sie die Folie und tupfen um das Gewebe mit einem trockenen Kimwipe um überschüssige Puffer zu entfernen (berühren Sie nicht die Gewebe). Stellen Sie den Schiebegewebeseite nach oben an einem dunklen Ort für 10-15 Minuten oder bis die Feuchtigkeit vollständig verflüchtigt.
- Pipette 11 ul Vectashield Befestigungsmediums (4 ', 6-Diamidino-2-phenylindol-DAPI) auf das Gewebe. Vorsichtig ein sauberes Deckglas (nicht mit Sigmacote behandelt) direkt über der Mitte des Montagemedium und Gewebe. Die Einschlussmittel sollte langsam nach außen zu den Kanten des Deckglases zu migrieren.
HINWEIS: Wenn die Montagemedium ausfällt, um die Kante des Deckglases an allen Seiten zu erreichen, wird eine zusätzliche 1-2 ul Befestigungsmedium kann mit einer Position am Rand des Deckglases aufgebracht, um in der benötigten Volumen füllen. In diesem Fall müssen Sie wischen Sie überschüssiges Medium aus der Gleitfläche vorAbdichtung. - Seal die Ränder des Deckglases mit Nagellack. Vermeiden Sie die Malerei der Nagellack über die Gewebeprobe.
- Den Objektträger aufrecht an einem dunklen Ort und lassen Sie den trockenen Nagellack bis zur vollständigen Fest (in der Regel 30 Minuten oder mehr). An dieser Stelle Bild das Gewebe oder bei -20 ° C für bis zu 1 Woche für die spätere Bildgebung.
Buffer / Lösung Rezepte
10x PBS
- 80 g NaCl
- 2,0 g KCl
- 14,4 g Na 2 HPO 4
- 2,4 g KH 2 PO 4
- pH auf 7,4, H 2 O auf 1 l
1x PBT
- 5 ml 10x PBS
- 45 ml H 2 O
- 0,1% Tween 20
20x SSC
- 175,3 g NaCl
- 88,2 g Na-Citrat
- in 800 ml H 2 O
- pH-Wert auf 7, H 2 O auf 1 l
4x SSCT-
- 200 ml 20x SSC
- 799 ml H 2 O
- 0,1% Tween 20
0,1x SSC
- 5 ml 20x SSC
- 995 ml H 2 O
Hybridisierungsmischung (20 & mgr; l; ab 11 modifiziert)
- 10 ul Formamid
- 4 ul 50% Dextransulfat
- 2 ul 20x SSC
- 4 & mgr; l H 2 O
SBT 8 (10 ml)
- 2 ml 20x SSC
- 0,01 g Rinderserumalbumin (BSA)
- 10 ul Tween 20
- 7,9 ml H 2 O
Blockierungslösung 8 (10 ml)
- 0,3 g BSA
- 10 ul Tween 20
- 2 ml 20x SSC
- 8 ml H 2 O
Fixierlösung mit Paraformaldehyd (1 ml)
- 393,75 ul H 2 O (Wasser hinzufügen zuerst)
- 450 ul Eisessig
- 156,25 & mgr; l 16% igem Paraformaldehyd
Ergebnisse
Um dieses Verfahren zu demonstrieren, hybridisierten wir eine Reihe von kleinen kommerziell synthetisiert Oligos, die chemisch mit Fluoreszenz-Konjugate (Abbildung 2) und einem längeren biotinylierte Sonde verändert wurden (durch Nick-Translation eines PCR-Produktes hergestellt; 2B), um die Chromosomen aus verschiedenen Gewebe Typen (siehe Tabelle 1). Die Zielsequenzen enthalten Satelliten-Wiederholungen in pericentromeric (Heterochromatin) Regionen der mitotischen Ch...
Diskussion
DNA ISH wird häufig verwendet, um spezifische Sequenzen auf Chromosomen kartieren. Wir haben ein einfaches Verfahren zur DNA ISH für hohe Kopienzahl, hetero Sequenzen optimiert beschrieben. Anstatt mit Waschungen in einem Formamid-Lösung, die eine Anforderung in anderen vorhandenen DNA ISH Protokolle ist, legen wir gewebe Dias direkt auf einen vorgeheizten Block an DNA zu denaturieren. Dieses Verfahren umgeht die Verwendung von großen Mengen an Formamid. Ein wichtiger Schritt für die Herstellung von klaren Hybridis...
Offenlegungen
The authors declare that they have no competing financial or any other conflict of interest.
Danksagungen
We thank Zhaohua Irene Tang in the W. M. Keck Science Department for the use of her epifluorescence microscope and the Werren lab for donating Nasonia for dissections. This work was supported in part by an NIH-NRSA fellowship (5F32GM105317-02) to AML.
Materialien
| Name | Company | Catalog Number | Comments |
| Poly-L-lysine coated slides (regular slides also can be used) | Sigma Aldrich | ||
| Ultrafine tweezers (5 gauge) | Dumont | ||
| 22 x 22 mm cover slips | Fisher | Sigmacote-treated by immersion for 15 sec, blotting dry, and wiping away all traces of Sigmacote so that cover slip is clear | |
| Sigmacote | Sigma | ||
| Filter paper | 75 - 150 mm | ||
| Paraffin wax paper | |||
| Heat block with thermometer | |||
| Dry incubator | |||
| Razor blades | |||
| Humidity chamber | empty pipette tip box or Tupperware, lined with moistened paper towels or Kimwipes | ||
| Coplin jars | with slide grooves | ||
| Aluminum foil | |||
| Pasteur pipettes | |||
| 1.5 ml microfuge tubes | |||
| Nail polish | clear or colored | ||
| P20 micropipette and plastic tips | |||
| Paperclips | 20 - 25 standard metal paperclips linked to form a chain | ||
| Reagents | |||
| 16% EM grade paraformaldehyde | Electron Microscopy Reagents | ||
| Acetic acid | Sigma | ||
| Liquid nitrogen | |||
| 100% Ethanol, chemical grade | |||
| Commercially synthesized, fluorescently labeled oligos | |||
| Long biotinylated probe | Invitrogen; Alternative steps 2.7.1-2.7.3 | e.g., nick translated and biotinylated with BioNick from Invitrogen | |
| Rhodamine-Avidin | Roche; Alternative steps 2.7.1-2.7.3 | for detection of long biotinylated probe | |
| Hybridization buffer | Recipe above | ||
| 4x SSCT | Recipe above | saline-sodium citrate + Tween | |
| 0.1x SSC | Recipe above | saline-sodium citrate | |
| Blocking solution | Recipe above | ||
| SBT | Recipe above | SSC, bovine serum albumin, Tween | |
| 1x PBT | Recipe above | phosphate-buffered saline + Tween | |
| 1x PBS | phosphate-buffered saline | ||
| Hypotonic solution | 0.5% sodium citrate in H2O | ||
| Formamide | Sigma Aldrich | ||
| Vectashield mounting medium with DAPI | Vector laboratories | ||
Referenzen
- Blattes, R., Kas, E. Fluorescent in situ hybridization (FISH) on diploid nuclei and mitotic chromosomes from Drosophila melanogaster larval tissues. Cold Spring Harbor Protocols. 2009 (9), (2009).
- Dimitri, P. Fluorescent in situ hybridization with transposable element probes to mitotic chromosomal heterochromatin of Drosophila. Methods in Molecular Biology. 260, 29-39 (2004).
- Pardue, M. L. In situ hybridization to polytene chromosomes in Drosophila using digoxigenin-labeled probes. Cold Spring Harbor Protocols. 2011 (8), 1003-1006 (2011).
- Hoskins, R. A., et al. Heterochromatic sequences in a Drosophila whole-genome shotgun assembly. Genome Biology. 3 (12), (2002).
- Hoskins, R. A., et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin. Science. 316 (58331), 1625-1628 (2007).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nature Reviews Genetics. 13 (1), 36-46 (2012).
- He, B., et al. Mapping the pericentric heterochromatin by comparative genomic hybridization analysis and chromosome deletions in Drosophila melanogaster. Genome Research. 22 (12), 2507-2519 (2012).
- Pimpinelli, S., Bonaccorsi, S., Fanti, L., Gatti, M. Fluorescent in situ hybridization (FISH) of mitotic chromosomes from Drosophila larval brain. Cold Spring Harbor Protocols. 2010 (3), (2010).
- Larracuente, A. M., Noor, M. A., Clark, A. G. Translocation of Y-linked genes to the dot chromosome in Drosophila pseudoobscura. Molecular Biology and Evolution. 27 (7), 1612-1620 (2010).
- Ferree, P. M., Barbash, D. A. Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biology. 7 (10), e1000234 (2009).
- Williams, B. C., Karr, T. L., Montgomery, J. M., Goldberg, M. L. The Drosophila l(1)zw10 gene product, required for accurate mitotic chromosome segregation, is redistributed at anaphase onset. The Journal of Cell Biology. 118 (4), 759-773 (1992).
- Gatti, M., Bonaccorsi, S., Pimpinelli, S. Looking at Drosophila Mitotic Chromosomes. Methods in Cell Biology. 44, 371-391 (1994).
- Werren, J. H., Stouthamer, R. PSR (paternal sex ratio) chromosomes: the ultimate selfish genetic elements. Genetica. 117 (1), 85-101 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten