JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
형광 DNA를위한 간단한 방법
요약
Here, we present a simple method for performing fluorescence DNA in situ hybridization (DNA ISH) to visualize repetitive heterochromatic sequences on slide-mounted chromosomes. The method requires minimal reagents and it is versatile for use with short or long probes, different tissues, and detection with fluorescence or non-fluorescence-based signals.
초록
현장 하이브리드 (DNA ISH)에서 DNA는 특정 염색체 영역에 매핑 시퀀스 일반적으로 사용되는 방법이다. 이 방법은 계산 방법이 엄청난 도전에 직면 염색질 영역에 매핑 매우 반복적 인 시퀀스에서 특히 효과적이다. 여기에서 우리는 다른 DNA ISH 프로토콜의 표준 단계입니다 포름 아미드 세척을 우회 DNA ISH에 대한 간소화 된 프로토콜을 설명합니다. 우리 프로토콜은 효과적으로 다른 곤충 조직 유형의 개수에 걸쳐 염색질 염색체 영역 내의 반복적 인 DNA 서열을 표시 형광 염료를 수행 짧은 단일 가닥 DNA 프로브와 혼성화를 위해 최적화된다. 그러나, 애플리케이션은 큰 프로브 및 단일 카피 (비 반복) DNA 서열의 시각화와 함께 사용하기 위해 확장 될 수있다. 우리는 신경 세포와 Nasonia을 melanogaster의 초파리에서 숙청 염색체에 여러 가지 반복적 인 시퀀스를 매핑하여이 방법을 보여vitripennis 정모 세포. 우리는 모두 작은, 상업적으로 합성 프로브 및 비교를위한 더 큰 프로브 하이브리드 패턴을 보여줍니다. 이 절차는 간단한 실험실 소모품 및 시약을 사용하고, DNA ISH을 수행하는 작은 경험이 연구자에 이상적입니다.
서문
현장 하이브리드 (DNA ISH)에서 DNA는 특정 염색체 영역에 매핑 시퀀스 일반적으로 사용되는 방법이다. euchromatin 내에서 단일 복사본 지역으로 프로브의 다양한 통해 닉 번역 또는 긴 DNA 제품 1, 2의 최종 라벨링 및 deoxygenin (DIG)의 통합을 포함하여 접근, -attached 뉴클레오티드과 인식의 소수를 생성 할 수 있습니다 그룹 결합 항체 1-3. 몇 또는 단일 복사본 수의 실 염색질 (euchromatin) 시퀀스의 시각화가 높은 특정 활동 또는 집합 신호를 향상 작은 여러 개의 프로브의 칵테일 하나의 큰 프로브 중 하나를 사용해야합니다.
그들은 일반적으로 수십 블록으로 알려진 단일 염색체 영역에서 클러스터 반복 수천로서 존재하기 때문에 대조적으로, 위성과 같은 이질 염색질의 DNA에서 발견되는 높은 서열이 반복은, DNA ISH 쉬워 표적이다. 전이 될 또한이 될 수 있습니다별개의 염색체 유전자좌 2에서 높은 사본 번호로 발견했다. 이 경우, 낮은 특정 활동과 단일 프로브 효과적으로 인해 여러 사이트에서 자신의 하이브리드에 염색질 시퀀스에 레이블을 지정할 수 있습니다. 반복적 서열 프로브 상업적 짧은 올리고 뉴클레오티드 (30-50 BP)을 화학적으로 합성하여 여러 가지 형광 그룹 중 하나와 결합 될 수있다. 게놈 시퀀싱 기술을 사용하여 이질 염색질 내에서 반복적 인 시퀀스를 매핑 인해 매우 반복적 인 위성 블록 4-6,7 내 건물 비계에서 발생하는 문제로 어렵다. 현재, ISH는 서브 염색체 수준에서 이러한 시퀀스를 맵핑하는 가장 효과적인 방법으로 서있다. 이 전략은 지속적인 유전체 및 전 사체 염기 서열 연구에 의해 발견되는 반복적 인 시퀀스의 큰 숫자를 매핑하는 것이 중요하다.
슬라이드에 장착 된 염색체에 매핑 반복적 인 시퀀스의 효율성과 편의성은 GRE 될 것이다atly DNA ISH에 대한 단순화 된 프로토콜에 의해 강화. 예를 들어, DNA ISH 기존 프로토콜 따라서 매핑 시퀀스에 필요한 시간을 실질적으로 첨가하고이 비싼 시약 화학 폐기물의 대량 생산, 2,8- 포름 아미드 용액에서 하이브리드 화의 다수의 조직 세척을 포함한다. 여기에서 우리는 포름 아미드 세척의 필요성을 우회하고 기본적인 실험실 장비 및 시약을 이용하여 수정 된 DNA ISH 방법을 설명합니다. 이 방법은 원래의 형광 염료와 접합 된 상업적으로 합성 된 올리고를 사용하여 초파리 유충 neuroblasts 염색질의 영역에서 고도로 반복적 인 DNA 서열의 빠른 매핑을 위해 설계되었다. 그러나,이 방법은 또한, 다른 수단을 통해 복수 9,10 상이한 조직 유형 염색체 및 합성 걸쳐 큰 프로브를 사용하여 반복적 인 시퀀스를 매핑하기 위해 작동한다. 또한,이 방법은 더 길거나 multip 사용하여 실 염색질 (euchromatin) 시퀀스를 매핑하는데 사용될 수있다르 관심 실 염색질 (euchromatin) 시퀀스 내 짧은 프로브.
프로토콜
1. 조직의 해부 및 고정 (60 분)
- 초파리의 뇌를 들어, 1X PBS (인산염 완충 식염수)의 드롭 3 차 령 유충을 배치합니다. 적극적으로 혼잡하지 않은 유리 병 또는 병에서 크롤링 큰 3 차 령 유충을 선택합니다.
- 입 후크를 잡고 몸 (그림 1A, B)의 길이 아래로 2/3을 잡기 위해 다른 트위터 쌍을 잡기 위해 하나의 초 미세 트위터 쌍을 사용합니다. 애벌레 소화 기관의 뇌, 복부 신경절, 침샘과 일부를 노출 입 후크를 부드럽게 잡아 당깁니다. 뇌와 복부 신경 분리 핀셋을 사용하여 (그림 1A를, B) 플라스틱 페트리 접시에 1X PBT (트윈 (Tween)와 인산염 완충 식염수)의 드롭 릿에서 다른 조직과 장소에서.
- Nasonia의 고환 해부를 들어, 남성 3 일짜리 번데기 (붉은 눈 노란색 기관)을 선택합니다. 남성 Nasonia은 F에 비해 작은 날개 패드의 길이가번데기 (그림 1C) 동안 emales.
- 한 트위터 쌍, 다른 트위터 쌍을 사용와 흉부 지역 근처 복부의 상단에있는 번데기를 잡고, 복부의 가장 말단 팁을 잡고 눈물 방울 모양의 고환을 잡아 당깁니다 (그들은 지방에 둘러싸여됩니다 부드럽게 떨어져 흔들릴 수 있습니다 몸도 1D). 플라스틱 페트리 접시에 1X PBT의 물방울에 적절한 짓눌러과 장소를 방지 할 수 고환에서 어떤 외부 신체 부위를 분리합니다.
- 각 슬라이드를 들어, 네 다섯 조직 샘플 (예., 초파리 애벌레 두뇌 또는 Nasonia의 고환)의 총을 해부하다.
참고 : 5 개 이상의 샘플 과밀 핵 및 염색체로 이어질 것입니다. - 선택적으로, 유사 분열의 염색체 영역 실 염색질 (euchromatin) 자매 염색 분체의 일부를 분리하기 저장성 용액 조직을 치료하는 (5-10 0.5 % 구연산 나트륨의 하락 1X PBT로부터 뇌를 전송할10 개 이상) 분.하지
주 : 콜히친 (유사 분열 억제제) 중기 (11) 유사 분열의 수를 증가 시키는데 도움이 될 수있다. 부정적인 염색체 해상도에 영향을 줄 수 있기 때문에 매우 큰 건강한 유충 확산 및 형태, 사용하고, 원하지 않는 경우에도 그러나, 그것의 사용은 불필요하다. - 깨끗한 Sigmacote 처리 된 커버 슬립의 표면에 정착 용액 (45 % 초산 2.5 % 파라 포름 알데히드)의 방울 (~ 20 μL)을 놓습니다.
참고 : 사용의 매일 정착 솔루션은 신선한합니다. 45 % 아세트산에 1.8-3.7 % 파라 포름 알데히드에 이르기까지 정착액 솔루션은 물고기에 대한 최상의 결과를 얻을 수 있습니다. 면역 FISH이 프로토콜을 뇌 또는 고환이 아닌 다른 조직을 사용하여, 또는 적응하는 것은 다른 고정 제 실험이 필요할 수 있습니다 (다른 고정 제의 목록, 12 참조). - 조심스럽게 초 미세 핀셋, minimi으로 정착 방울로 해부 버퍼 (1X PBT)에서 각 조직 샘플을 전송정착액 솔루션으로 버퍼를 해부의 전송을 쌩쌩. 골고루 고정액 액적 내에 서로 이격되도록 조직 샘플을 위치. 실온에서 4 분 동안 고정 제의 조직을 배양한다.
- 조심스럽게 아래 조직 및 커버 슬립 위에 폴리 라이신 코팅 슬라이드 얼굴을 배치합니다. 이 시점에서 아래로 눌러 대신 커버 슬립 슬라이드의 아래쪽에 붙어 있도록 두 가볍게 접촉하지 마십시오. 커버 슬립이 상단에 위치하도록 슬라이드 전환.
- 필터 종이의 접힌 부분 내부 슬라이드 / 조직 / 커버 슬립을 샌드위치. 안정된 표면에 매우 단단히 똑바로 직접 커버 슬립 위의 위치 상 엄지 손가락, 프레스를 사용하여. (이 조직의 번짐의 원인이됩니다) 커버 슬립의 슬라이딩 측면 않도록주의해야합니다.
- 액체 질소에 슬라이드 / 조직 / 커버 슬립 잠수함 (그림 1E, F에 장치 참조), 그리고 질소가 끓는 멈출 때까지 세우 (이상은 F입니다오프라인). 슬라이드를 제거하고 즉시 (면도날 고정 조직 긁히지 않도록) 위쪽으로 커버 슬립의 코너를 쓸어 넘겨 신선한 면도날로 커버 슬립을 떼어냅니다.
- 드라이 아이스의 블록에 슬라이드 / 조직 / 커버 슬립을 사전 냉각 전에 커버 균열에서 슬라이드를 예방하는 데 도움이됩니다 액체 질소에 침지에 1-2 분 동안, 최대 슬립.
- 즉시 실온에서 100 % 에탄올로 가득 코 플린 항아리에 조직과 슬라이드를 배치하고 (필요한 경우이 시간이 더 길어질 수 있습니다) 적어도 5 분간 방치 할 수 있습니다.
주 : 냉간 100 % 에탄올이 또한 사용될 수있다. - 조직과 슬라이드를 분리합니다 (고정 조직을 건드리지 않고) 킴과 초과 에탄올을 멀리 심지, 1 시간 동안 공기 건조에 슬라이드를 둡니다.
- 2 단계 또는 몇 주에 하이브리드를 수행하기 전에 몇 달을 위해 습도가 낮은 공기 또는 탈수 챔버 건조 슬라이드를 유지하기 위해 직접 진행합니다.
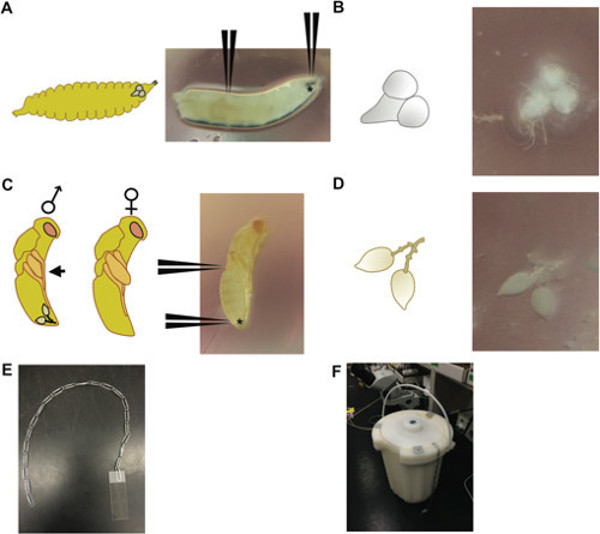
그림 1 : (A) 3 차 령 초파리 유충 (오른쪽), 입 후크를 잡아 수있는 위치에 대한 지시 위치와는 (로 표시 *)과 뇌를 해부 유충을 끝까지 2/3; (왼쪽) 유생 헤드 내의 뇌의 상대 위치를 묘사 한 동일한 발달 단계 유충의 개략도. (B) 뇌와 3 차 령 초파리 유충 (오른쪽)이 개략적 해부 복부 신경절 조직 (왼쪽). 노란색 몸에 붉은 눈 단계에서 (C) 3 일 된 Nasonia 번데기. (D) 번데기를 잡아하는 방법을 나타내는 위치와 (오른쪽) 3 일 이전 Nasonia 남성 번데기 해부 고환 쌍 복부의 뒤쪽에 (* 표시) 몸에 중간에; (왼쪽) 개략적으로 묘사 남성과 여성이었다P는 번데기; 남성 번데기는 프로파일 지나서 연장 날개가 암컷 대조적 져널 프로파일 (흑색 화살표)를지나 연장하지 않는다 날개에 의해 구별 될 수있다; 고환 쌍의 상대적 위치는 남성 번데기에 표시됩니다. 액체 질소 용기에 슬라이드를 담가 사용 클립 및 (F) 방법 (E) 장치 - 문자열. 다중 클립 문자열은 여러 슬라이드를 동시에 침수 사용할 수 있습니다.
현장 하이브리드 2. (1 일 30 분, 오랫동안 1 시간 - 2.5 시간 프로브-에 하루 2)
- 1.1 하이브리드 버퍼의 20 μL 각 프로브 1 μL (100 NG)를 추가합니다. 피펫 프로브 / 고정 조직의 표면에 하이브리드 버퍼 (조직을 건드리지 않도록).
- 조심스럽게 커버 슬립이 조직을 통해 직접 중앙에 있는지 확인하고, 프로브 / 하이브리드 버퍼에 직접 커버 슬립을 배치합니다. 버퍼는 토륨으로 마이그레이션해야합니다기포를 남기지 않고, 커버 슬립의 외연 전자.
참고 : 절차에 대한 문제를 제기하지 않는 조직을 접촉하지 않는 작은 거품을. 조심스럽게 슬라이드에 다시 떨어 조심스럽게 커버 슬립의 모서리를 들어 올려으로 큰 거품을 제거합니다. - 블록 (커버 쪽지를) 95 ° C로 예열의 표면에 슬라이드 / 조직 / 커버 슬립을 놓습니다. 노광을 방지하기 위해 알루미늄 포일의 큰 조각으로 덮을. 슬라이드를 5 분 동안 95 ° C에서 배양한다하자.
참고 : 튜브 구멍 전형적인 열 블록은 슬라이드 / 조직 / 커버 슬립을 배치 할 평면을 제공하기 위해 뒤집어 수 있습니다. - 이 터치에 따뜻한 때까지 약간 냉각시키는, 슬라이드를 제거합니다. 조심스럽게 그 아래에 액체를 밀봉하는 커버 슬립 씌워진 파라 필름 조각을 포장.
- 습도 챔버 내부의 밀봉 된 슬라이드를 삽입하고 30 ° C로 예열 인큐베이터에 실을 배치합니다. 4 시간 하룻밤에 30 ° C에서 품어.
- 맨 아래에 배치 젖은 Kimwipes이나 종이 타월로 빈 팁 상자에서 습도 챔버 또는 리드 형 유리 섬유 복합 재질의 컨테이너를 만듭니다.
주 : DNA 단일 가닥 올리고 프로브는 45-47 ℃의 이론적 용융 온도 (Tm) 28-33 염기 달성 할 수 있도록 설계되었다. 선택된 길이와 T m 범위는 우리가 공부 반복적 인 시퀀스들이 AT 풍부하며, 따라서 매우 낮은 GC 함량을 갖는다는 사실을 반영한다. 긴 프로브 가능성이 높은 T의 m 값을 가질 것이다; 이 30 ° C의 표준 하이브리드 온도에서 높은 배경 하이브리드의 원인이 될 수 있습니다. 따라서, 혼성화 온도가 몇몇 문제가 최상의 결과를 달성하기 위해 요구 될 수있다. 점진적으로, 최고의 하이브리드 온도 증가 (또는 감소) 5 ° C로 온도를 찾을 수 있습니다.
- 맨 아래에 배치 젖은 Kimwipes이나 종이 타월로 빈 팁 상자에서 습도 챔버 또는 리드 형 유리 섬유 복합 재질의 컨테이너를 만듭니다.
- 조심스럽게 슬라이드에서 파라 필름을 제거하고 조심스럽게 천천히 한쪽 모서리를 들어 올려 커버 슬립을 제거합니다. 워싱턴15 분 0.1X SSC 버퍼의 각 세척 3 회 밀어 쉬. 빛의 노출을 최소화하기 위해 세척시 알루미늄 호일로 코 플린 항아리를 커버.
- 긴 비오틴 프로브를 사용하지 않는 경우, 2.8 단계로 진행합니다.
- 긴 바이오틴 프로브를 사용하는 경우, 조직 자체를 만지지 않도록 조심하면서, 킴과 조직 주변을 건조. 조직을 통해 차단 솔루션의 100 μl를 놓고 트래핑 거품을 피하기 위해주의하면서 커버 슬립으로 조심스럽게 다룹니다. 30 분 동안 37 ° C에서 파라 필름과 커버 슬립과 장소를 통해 슬라이드를 감 쌉니다.
- 조심스럽게 커버 슬립을 제거하고 킴과 조직 주위시킨다. 피펫은 로다 민 - 아비딘 1 희석 100 ㎕ : 조직을 통해 SBT 1,000 부드럽게 커버 슬립 커버, 돌보는 거품을 트래핑 방지 할 수 있습니다. 30 분 동안 37 ° C에서 파라 필름과 커버 슬립과 장소를 통해 슬라이드를 감 쌉니다.
- 조심스럽게 커버 슬립을 제거하고 배 SSCT과 5 분 동안 슬라이드를 각각 3 회 세척0.1X의 SSC에서 5 분 각 3 회.
참고 : 슬라이드가 오랜 시간 세척 할 수있다.
- 슬라이드를 제거하고 (조직에 닿지 않도록)를 초과하는 버퍼를 제거하는 건조 킴과 조직 주위시킨다. 10-15 분 동안 또는 수분이 완전히 소산까지 어두운 곳에 슬라이드 조직 쪽을 놓는다.
- Vectashield 장착 매체의 피펫 11 μL ( '4, 6 diamidino -2- 페닐 인돌 - DAPI) 조직 상. 조심스럽게 직접 장착 매체 및 조직의 중심을 통해 (안 Sigmacote 처리) 깨끗한 커버 슬립을 배치합니다. 장착 매체 커버 슬립의 가장자리를 향해 외측으로 천천히 이동한다.
주 : 장착 매체 사방 커버 슬립의 가장자리에 도달하지 못한 경우, 실장 매체의 추가적인 1-2 μL가 필요한 볼륨 채울 커버 슬립의 가장자리에 하나 위치에 적용될 수있다. 이 경우, 이전 슬라이드 표면에서 초과 매체를 닦아주십시오밀봉. - 매니큐어와 커버 슬립의 가장자리를 밀봉합니다. 조직 샘플을 통해 매니큐어를 칠 마십시오.
- 어두운 곳에서 수직 슬라이드를 삽입하고 완전히 하드 (일반적으로 30 분 이상)까지 매니큐어 건조를 할 수 있습니다. 이 시점에서, 이미지 나중에 이미징을위한 최대 1 주일 -20 ° C에서 조직 또는 저장.
버퍼 / 솔루션 조리법
10 배 PBS
- 80g의 NaCl
- 2.0 g의 KCl
- 14.4 g 나 2 HPO 4
- 2.4 g의 KH 2 PO 4
- pH 7.4까지, H 2 O 1 L
1X PBT
- 5 ml의 10 배 PBS
- 45 ML의 H 2 O
- 0.1 % 트윈 20
20 배 SSC
- 175.3 g의 NaCl
- 88.2 g 나 구연산
- 800 ML의 H 2 O에
- pH7로, H 2 O 1 L
배 SSCT
- 200 ml의 20 배 SSC
- 799 ML의 H 2 O
- 0.1 % 트윈 20
0.1X SSC
- 5 ml의 20 배 SSC
- 995 ML의 H 2 O
하이브리드 믹스 (20 μl의 11에서 수정)
- 10 μL 포름 아미드
- 4 ㎕의 50 % 덱스 트란 설페이트
- 2 μL 20 배 SSC
- 4 μL의 H 2 O
SBT 8 (10 mL) 중
- 2 ml의 20 배 SSC
- 0.01 g 소 혈청 알부민 (BSA)
- 10 μL 트윈 20
- 7.9 ML의 H 2 O
블로킹 용액 (8) (10 mL) 중
- 0.3 g의 BSA
- 10 μL 트윈 20
- 2 ml의 20 배 SSC
- 8 ML의 H 2 O
파라 포름 알데히드 고정액 용액 (1 mL) 중
- 2 O (시 물을 추가) 393.75 μL의 H
- 450 μL의 빙초산
- 156.25 μL 16 % 파라 포름 알데히드
결과
여러 조직에서 염색체, (그림 2B PCR 제품의 닉 번역을 통해 만든)이 방법을 설명하기 위해, 우리는 화학적으로 형광 복합체 (그림 2)와 이상 바이오틴 프로브 수정 된 작은 상업적으로 합성 올리고 세트를 교배 타입 (표 1 참조). 대상 시퀀스 D.에서 유사 분열 염색체의 pericentromeric (염색질) 지역에 위치한 위성 반복을 포함 번데기 N.에서 애벌레 neuro...
토론
DNA ISH 자주 염색체 특정 시퀀스를 매핑하는 데 사용된다. 우리는 높은 사본 번호, 염색질 시퀀스에 최적화 된 DNA ISH을위한 간단한 방법을 설명 하였다. 오히려 기존의 DNA의 ISH 프로토콜의 요구 사항 인 포름 아미드 용액에 세척을 사용하는 것보다, 우리는 DNA를 변성 직접 미리 가열 블록에 조직에 장착 된 슬라이드를 배치합니다. 이 방법은 많은 양의 포름 아미드의 사용을 회피. 선명 혼성화 신호?...
공개
The authors declare that they have no competing financial or any other conflict of interest.
감사의 말
We thank Zhaohua Irene Tang in the W. M. Keck Science Department for the use of her epifluorescence microscope and the Werren lab for donating Nasonia for dissections. This work was supported in part by an NIH-NRSA fellowship (5F32GM105317-02) to AML.
자료
| Name | Company | Catalog Number | Comments |
| Poly-L-lysine coated slides (regular slides also can be used) | Sigma Aldrich | ||
| Ultrafine tweezers (5 gauge) | Dumont | ||
| 22 x 22 mm cover slips | Fisher | Sigmacote-treated by immersion for 15 sec, blotting dry, and wiping away all traces of Sigmacote so that cover slip is clear | |
| Sigmacote | Sigma | ||
| Filter paper | 75 - 150 mm | ||
| Paraffin wax paper | |||
| Heat block with thermometer | |||
| Dry incubator | |||
| Razor blades | |||
| Humidity chamber | empty pipette tip box or Tupperware, lined with moistened paper towels or Kimwipes | ||
| Coplin jars | with slide grooves | ||
| Aluminum foil | |||
| Pasteur pipettes | |||
| 1.5 ml microfuge tubes | |||
| Nail polish | clear or colored | ||
| P20 micropipette and plastic tips | |||
| Paperclips | 20 - 25 standard metal paperclips linked to form a chain | ||
| Reagents | |||
| 16% EM grade paraformaldehyde | Electron Microscopy Reagents | ||
| Acetic acid | Sigma | ||
| Liquid nitrogen | |||
| 100% Ethanol, chemical grade | |||
| Commercially synthesized, fluorescently labeled oligos | |||
| Long biotinylated probe | Invitrogen; Alternative steps 2.7.1-2.7.3 | e.g., nick translated and biotinylated with BioNick from Invitrogen | |
| Rhodamine-Avidin | Roche; Alternative steps 2.7.1-2.7.3 | for detection of long biotinylated probe | |
| Hybridization buffer | Recipe above | ||
| 4x SSCT | Recipe above | saline-sodium citrate + Tween | |
| 0.1x SSC | Recipe above | saline-sodium citrate | |
| Blocking solution | Recipe above | ||
| SBT | Recipe above | SSC, bovine serum albumin, Tween | |
| 1x PBT | Recipe above | phosphate-buffered saline + Tween | |
| 1x PBS | phosphate-buffered saline | ||
| Hypotonic solution | 0.5% sodium citrate in H2O | ||
| Formamide | Sigma Aldrich | ||
| Vectashield mounting medium with DAPI | Vector laboratories | ||
참고문헌
- Blattes, R., Kas, E. Fluorescent in situ hybridization (FISH) on diploid nuclei and mitotic chromosomes from Drosophila melanogaster larval tissues. Cold Spring Harbor Protocols. 2009 (9), (2009).
- Dimitri, P. Fluorescent in situ hybridization with transposable element probes to mitotic chromosomal heterochromatin of Drosophila. Methods in Molecular Biology. 260, 29-39 (2004).
- Pardue, M. L. In situ hybridization to polytene chromosomes in Drosophila using digoxigenin-labeled probes. Cold Spring Harbor Protocols. 2011 (8), 1003-1006 (2011).
- Hoskins, R. A., et al. Heterochromatic sequences in a Drosophila whole-genome shotgun assembly. Genome Biology. 3 (12), (2002).
- Hoskins, R. A., et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin. Science. 316 (58331), 1625-1628 (2007).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nature Reviews Genetics. 13 (1), 36-46 (2012).
- He, B., et al. Mapping the pericentric heterochromatin by comparative genomic hybridization analysis and chromosome deletions in Drosophila melanogaster. Genome Research. 22 (12), 2507-2519 (2012).
- Pimpinelli, S., Bonaccorsi, S., Fanti, L., Gatti, M. Fluorescent in situ hybridization (FISH) of mitotic chromosomes from Drosophila larval brain. Cold Spring Harbor Protocols. 2010 (3), (2010).
- Larracuente, A. M., Noor, M. A., Clark, A. G. Translocation of Y-linked genes to the dot chromosome in Drosophila pseudoobscura. Molecular Biology and Evolution. 27 (7), 1612-1620 (2010).
- Ferree, P. M., Barbash, D. A. Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biology. 7 (10), e1000234 (2009).
- Williams, B. C., Karr, T. L., Montgomery, J. M., Goldberg, M. L. The Drosophila l(1)zw10 gene product, required for accurate mitotic chromosome segregation, is redistributed at anaphase onset. The Journal of Cell Biology. 118 (4), 759-773 (1992).
- Gatti, M., Bonaccorsi, S., Pimpinelli, S. Looking at Drosophila Mitotic Chromosomes. Methods in Cell Biology. 44, 371-391 (1994).
- Werren, J. H., Stouthamer, R. PSR (paternal sex ratio) chromosomes: the ultimate selfish genetic elements. Genetica. 117 (1), 85-101 (2003).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유