Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Méthode simple pour l'ADN fluorescence
Dans cet article
Résumé
Here, we present a simple method for performing fluorescence DNA in situ hybridization (DNA ISH) to visualize repetitive heterochromatic sequences on slide-mounted chromosomes. The method requires minimal reagents and it is versatile for use with short or long probes, different tissues, and detection with fluorescence or non-fluorescence-based signals.
Résumé
ADN hybridation in situ (ISH ADN) en est une méthode couramment utilisée pour la cartographie des séquences à des régions spécifiques de chromosomes. Cette approche est particulièrement efficace pour la cartographie des séquences hautement répétitives à des régions hétérochromatiques, où les approches de calcul face à des défis prohibitifs. Ici, nous décrivons un protocole simplifié pour l'ADN ISH qui contourne lavages formamide qui sont des mesures standard dans d'autres protocoles ADN ISH. Notre protocole est optimisé pour l'hybridation avec de courtes sondes d'ADN simple brin qui transportent des colorants fluorescents, qui marquent efficacement des séquences d'ADN répétitives à l'intérieur des régions chromosomiques hétérochromatiques dans un certain nombre de types de tissus de différents insectes. Toutefois, les applications peuvent être étendues à utiliser avec des sondes et la visualisation de séquences d'ADN copie unique (non répétitive) grands. Nous démontrons cette méthode en cartographiant plusieurs différentes séquences répétitives à des chromosomes de Drosophila melanogaster écrasés cellules neurales et Nasoniavitripennis spermatocytes. Nous montrons schémas d'hybridation pour les deux petites sondes, synthétisées dans le commerce et pour une plus grande sonde pour comparaison. Cette procédure utilise des consommables et réactifs de laboratoire simples, et est idéal pour les enquêteurs qui ont peu d'expérience avec l'exécution de l'ADN ISH.
Introduction
ADN hybridation in situ (ISH ADN) en est une méthode couramment utilisée pour la cartographie des séquences à des régions spécifiques de chromosomes. Sondes aux régions à copie unique dans euchromatine peuvent être générés par une poignée d'approches, y compris translation de coupure ou la fin-étiquetage des produits de 1,2 ADN longues et l'incorporation de deoxygenin (DIG) nucléotides -attached et leur reconnaissance à travers une grande variété de conjugué anticorps-groupe 1-3. Visualisation des séquences euchromatiques dans peu ou seul numéro de copie nécessite l'utilisation soit simples, grandes sondes ayant une activité spécifique élevée ou un cocktail de plusieurs petites sondes, qui améliorent collectivement signaux.
En revanche, les séquences hautement répétitives trouvés dans hétérochromatine, comme ADN satellites, sont des cibles plus faciles pour l'ADN ISH parce qu'ils existent normalement que des dizaines de milliers de répétitions groupées dans les régions chromosomiques simples appelés blocs. Les éléments transposables peuvent également êtretrouvé à grand nombre de copies à distincte loci chromosomiques 2. Dans ces cas, les sondes simples avec faible activité spécifique peuvent effectivement étiqueter séquences hétérochromatiques en raison de leur hybridation à plusieurs sites. Sondes à séquences répétitives peuvent être synthétisés oligonucléotides commercialement aussi courtes (30-50 pb) et chimiquement conjugués avec une des multiples différents groupes fluorescents. Cartographie de séquences répétitives au sein hétérochromatine en utilisant des technologies de séquençage des génomes est difficile en raison de difficultés rencontrées dans les échafaudages de construction dans les blocs de satellites hautement répétitives 4-6,7. Actuellement, ISH se présente comme le moyen le plus efficace de la cartographie de ces séquences au niveau des sous-chromosome. Cette stratégie est importante pour cartographier un grand nombre de séquences répétitives qui sont découverts par des études génomiques et de séquençage du transcriptome en cours.
L'efficacité et la facilité de cartographie des séquences répétitives sur les chromosomes de diapositives montées seraient GREatly renforcée par un protocole simplifié pour l'ADN ISH. Par exemple, les protocoles existants pour l'ADN ISH impliquent de multiples lavages de tissus hybrides en solution formamide 2,8, ajoutant ainsi sensiblement au temps requis pour les séquences de cartographie et de produire de grandes quantités de déchets chimiques pour ce réactif coûteux. Nous décrivons ici un procédé ADN ISH révisé qui contourne la nécessité de lavages formamide et utilise le matériel et les réactifs de laboratoire de base. Cette méthode a été initialement conçu pour la cartographie rapide de séquences d'ADN hautement répétitives dans des régions hétérochromatiques neuroblastes larves de Drosophila en utilisant les oligos de synthèse dans le commerce qui sont conjugués avec des colorants fluorescents. Cependant, cette méthode fonctionne également pour mettre en correspondance des séquences répétitives en utilisant de plus grandes sondes synthétisées par d'autres moyens et 9,10 dans de multiples types de tissus et de chromosomes différents. En outre, cette méthode peut être utilisée pour cartographier des séquences euchromatiques en utilisant plus ou Multiple, de courtes sondes dans la séquence euchromatique d'intérêt.
Protocole
1. Tissue Dissection et fixation (60 min)
- Pour cerveaux drosophile, placer 3 ème stade larvaire dans une goutte de 1x PBS (tampon phosphate salin). Choisissez grand stade larvaire 3 ème qui sont activement l'exploration à partir des flacons ou des bouteilles qui ne sont pas surpeuplées.
- Utilisez une pince à épiler ultrafine paire de se emparer de la bouche des crochets et une autre paire de pince pour saisir 2/3 sur la longueur du corps (figure 1A, B). Tirez doucement sur la bouche crochets pour exposer le cerveau, les ganglions ventrale, glandes salivaires et une partie du tube digestif des larves. Utilisez la pince à épiler pour séparer le cerveau et les ganglions ventrale (figure 1A, B) des autres tissus et les placer dans une gouttelette de 1x PBT (tampon phosphate salin avec du Tween) sur une plaque de Petri en plastique.
- Pour Nasonia dissections des testicules, choisissez mâle 3-day-old pupes (corps jaune avec des yeux rouges). Homme Nasonia ont de petites longueurs de rembourrage d'aile par rapport à females pendant le stade de pupe (figure 1C).
- Maintenez la nymphe en haut de l'abdomen près de la région thoracique avec une paire de pince à épiler, et en utilisant l'autre paire de pince à épiler, prenez la pointe distale de l'abdomen et sortir les testicules en forme de larme (ils seront entourés par de la graisse corps qui peut être doucement secoué loin; figure 1D). Détachez les pièces extérieures de carrosserie des testicules, ce qui empêchera l'écrasement bon, et le placer dans une gouttelette de 1x PBT sur une plaque de Petri en plastique.
- Pour chaque diapositive, disséquer un total de quatre ou cinq échantillons de tissus (ce est à dire., Drosophila cerveaux larvaires ou testicules Nasonia).
NOTE: Plus de cinq échantillons mèneront à noyaux et les chromosomes surpeuplées. - Eventuellement, pour atteindre une certaine séparation des chromatides soeurs dans les régions euchromatiques de chromosomes mitotiques, traiter le tissu avec une solution hypotonique: transférer cerveaux de 1x PBT à une baisse de citrate de sodium à 0,5% pour les 5-10 (pas plus de 10) min.
NOTE: La colchicine (un inhibiteur mitotique) peut être utile pour augmenter le nombre de figures de mitose à la métaphase 11. Cependant, son utilisation ne est pas nécessaire si très grand larves saines sont utilisés, et même souhaitable car elle peut nuire à la résolution des chromosomes, la diffusion et la morphologie. - Placer une goutte (~ 20 ul) de solution de fixation (2,5% de paraformaldehyde dans de l'acide acétique à 45%) sur la surface d'une lamelle couvre-Sigmacote traité propre.
NOTE: Préparer une solution de fixateur frais pour chaque jour d'utilisation. Solutions fixateur allant de 1,8 à 3,7% de paraformaldehyde dans de l'acide acétique 45% donnent les meilleurs résultats pour les poissons. En utilisant des tissus autres que le cerveau ou les testicules, ou l'adaptation de ce protocole pour immuno-FISH peut exiger expérimenter avec différents fixateurs (pour une liste des différents fixateurs, voir 12). - Transférer soigneusement chaque échantillon de tissu de la mémoire tampon de dissection (1x PBT) dans la gouttelette fixateur avec des pincettes ultrafines, minimizing le transfert de dissection tampon dans la solution de fixateur. Placer les échantillons de tissu de sorte qu'ils sont uniformément espacés les uns des autres à l'intérieur de la gouttelette fixateur. Incuber les tissus de fixateur pendant 4 min à température ambiante.
- Placez délicatement une surface de glissement poly-lysine vers le bas sur le glissement des tissus et de la couverture. Ne appuyez pas à ce point, mais au lieu de permettre aux deux à contacter légèrement de sorte que la lamelle colle sur la face inférieure de la diapositive. Inversez la diapositive de sorte que la lamelle est sur le dessus.
- Sandwich glissement coulissant / tissus / couvercle intérieur d'un morceau de papier plié de filtre. Sur une surface stable, avec le pouce, appuyez fermement vers le bas sur la position directement au-dessus de la lamelle. Soyez prudent pour éviter un glissement latéral de la lamelle (cela va provoquer des bavures du tissu).
- Immerger la diapositive / tissus / lamelle dans l'azote liquide (voir appareil de la figure 1E, F), et laisser reposer jusqu'à ce que le azote arrête bouillante (plus est fine). Retirer la lame et enclenchez immédiatement la lamelle avec une lame de rasoir propre en appuyant sur un coin de la lamelle dans une direction ascendante (éviter de rayer le tissu fixe avec la lame de rasoir).
- Pré-refroidissement de la lame / tissus / lamelle sur un bloc de glace sèche, lamelle jusqu'à, pendant 1-2 min avant immersion dans l'azote liquide aidera à prévenir les diapositives de la fissuration.
- Placer immédiatement la diapositive avec le tissu dans une jarre Coplin remplie d'éthanol à 100% à la température ambiante et laisser reposer pendant au moins 5 minutes (ce temps peut être plus long si nécessaire).
REMARQUE: éthanol à 100% froid pourrait également être utilisé. - Retirer la lame avec le tissu, évacuer excès d'éthanol avec un Kimwipe (sans toucher le tissu fixe), et de laisser la lame sécher à l'air pendant 1 heure.
- Passez directement à l'étape 2 ou garder diapositives secs dans l'air à basse humidité ou dans une chambre de dessiccation pendant des semaines voire des mois à avant d'effectuer l'hybridation.
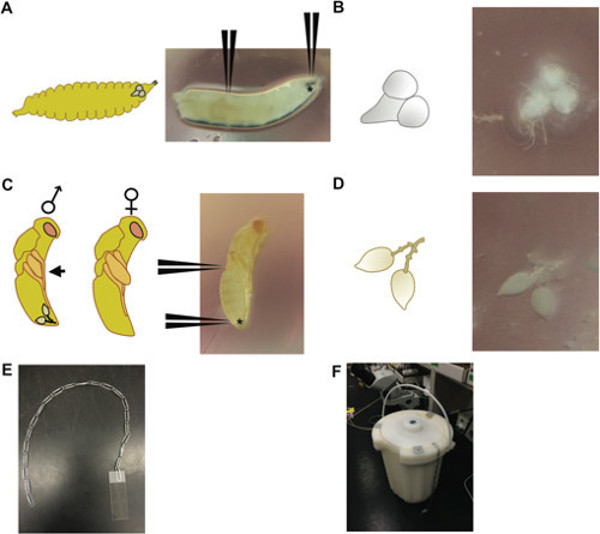
Figure 1: (A) Un 3 ème stade larve de drosophile (à droite), avec des positions indiquées pour savoir où récupérer les crochets buccaux (notée avec *) et 2/3 de la descente de la larves de disséquer le cerveau; (À gauche) un schéma d'une larve d'un même stade de développement, représentant la position relative du cerveau dans la tête des larves. (B) Le cerveau et les ganglions ventrale disséqué d'une 3 ème stade larve de drosophile (à droite) et un schéma de ce tissus (à gauche). (C) 3-day-old nymphe Nasonia au stade de l'oeil du corps rouge jaune. (D) Une paire de testicule disséqué d'un homme âgé de nymphe 3 jours Nasonia (à droite) avec des positions indiquant où saisir la nymphe à la partie postérieure de l'abdomen (désigné par *) et à mi-chemin sur le corps; (À gauche) schématique mâle et femelle était dépeignantp pupes; pupes mâles se distingue par des ailes qui ne se étendent pas au-delà du profil sagittal (flèche noire), contrairement aux femelles, qui ont des ailes qui se étendent au-delà du profil; la position relative de la paire de testicule est montré dans la nymphe mâle. (E) L'appareil-une chaîne de trombones-et méthode (F) utilisés pour immerger les lames dans un récipient d'azote liquide. Plusieurs chaînes de trombone peuvent être utilisés pour l'immersion simultanée de plusieurs diapositives.
2. Hybridation in situ (30 min le jour 1; 1 h-2,5 heures pendant de longues sondes sur deux jours)
- Ajouter 1 pi (100 ng) de chaque sonde de 20 ul de tampon d'hybridation 1,1x. Pipet sonde / tampon d'hybridation sur la surface du tissu fixe (éviter de toucher le tissu).
- Placez délicatement une lamelle directement sur le tampon sonde / hybridation, faire en sorte que la lamelle est centré directement sur le tissu. Le tampon doit migrer vers the bord extérieur de la lamelle, ne laissant aucune bulles d'air.
NOTE: Les petites bulles qui ne contactent pas les tissus ne pose pas de problèmes à la procédure. Retirer les grosses bulles en soulevant délicatement un coin de la lamelle et soigneusement laisser tomber en arrière sur la diapositive. - Placez le glissement coulissant / tissus / couvercle sur la surface d'un bloc pré-chauffé à 95 ° C (couverture glisser vers le haut). Couvrir avec un grand morceau de papier d'aluminium pour éviter exposition à la lumière. Laissez la diapositive incuber à 95 ° C pendant 5 min.
REMARQUE: Un bloc de chaleur typique avec des trous pour les tubes peut être retourné pour fournir une surface plane sur laquelle placer la diapositive / tissus / lamelle. - Retirer la lame, le laisser refroidir légèrement jusqu'à ce qu'il soit chaud au toucher. Envelopper soigneusement un morceau de Parafilm étiré autour de la lamelle pour sceller le liquide en dessous.
- Placer la lame scellé à l'intérieur d'une chambre d'humidité et placer la chambre dans un incubateur préchauffé à 30 ° C. Incuber à 30 ° C pendant 4 heures à une nuit.
- Créer une chambre humide à partir d'une boîte de pointe vide ou un récipient à couvercle de Tupperware avec Kimwipes mouillées ou des serviettes en papier placés au fond.
REMARQUE: Les sondes ADN simples brins oligonucléotidiques ont été conçues pour être de 28 à 33 bases pour atteindre une température de fusion théorique (T m) de 45 à 47 ° C. Ces longueur et T m fourchettes reflètent le fait que beaucoup de séquences répétitives que nous avons étudiés sont AT-riche et donc très faible teneur en GC. Des sondes plus longues auront probablement des valeurs plus élevées de m T; il peut en résulter une hybridation ultérieure de fond à la température d'hybridation standard de 30 ° C. Ainsi, un peu de dépannage avec des températures d'hybridation peut être nécessaire pour obtenir les meilleurs résultats. Pour trouver la meilleure température d'hybridation, augmentation (ou diminution) de la température de 5 ° C, progressivement.
- Créer une chambre humide à partir d'une boîte de pointe vide ou un récipient à couvercle de Tupperware avec Kimwipes mouillées ou des serviettes en papier placés au fond.
- Retirez délicatement le Parafilm de la diapositive, puis retirez soigneusement la lamelle en soulevant lentement un coin. Washingtonsh la diapositive trois fois pour 15 minutes chaque lavage dans un tampon SSC 0,1x. Couvrir la jarre de Coplin avec du papier d'aluminium pendant les lavages à minimiser exposition à la lumière.
- Si vous ne utilisez pas une longue sonde biotinylée, passez à l'étape 2.8.
- Si vous utilisez une sonde à long biotinylé, sécher la zone autour du tissu avec un Kimwipe, en faisant attention de ne pas toucher le tissu lui-même. Placez 100 pi de solution de blocage sur le tissu et recouvrir délicatement avec une lamelle, en prenant soin d'éviter les bulles de piégeage. Enveloppez la lame sur la lamelle avec du Parafilm et lieu à 37 ° C pendant 30 min.
- Retirez délicatement la lamelle et épongez autour du tissu avec un Kimwipe. Distribuer 100 ul de la rhodamine-avidine diluée 1: 1000 dans les soins SBT sur le tissu et recouvrir délicatement avec une lamelle, en prenant pour éviter la formation de bulles. Enveloppez la lame sur la lamelle avec du Parafilm et lieu à 37 ° C pendant 30 min.
- Retirez délicatement la lamelle et laver la lame 3 fois pendant 5 minutes chacun dans 4x SSCT puis3 fois pendant 5 min chacun dans 0,1x SSC.
REMARQUE: Les diapositives peuvent être lavé pendant des périodes de temps plus longues.
- Retirer la lame et épongez autour du tissu avec un Kimwipe sec pour enlever l'excès de tampon (éviter de toucher le tissu). Placez le côté tissu de glisser vers le haut dans un endroit sombre pendant 10-15 min ou jusqu'à ce que l'humidité se dissipe complètement.
- Pipeter 11 ul de milieu de montage Vectashield (avec 4 ', 6-diamidino-2-phénylindole-DAPI) sur le tissu. Placez délicatement une lamelle propre (pas traité avec Sigmacote) directement sur le centre du milieu de montage et de tissus. Le milieu de montage devrait migrer lentement vers l'extérieur vers les bords de la lamelle.
NOTE: Si le milieu de montage ne parvient pas à atteindre le bord de la lamelle sur tous les côtés, puis un 1-2 ul supplémentaire de milieu de montage peut être appliquée à une position au bord de la lamelle à remplir le volume nécessaire. Dans ce cas, veillez à essuyer tout excès du milieu de la surface de glissement avantétanchéité. - Scellez les bords de la lamelle avec du vernis à ongles. Évitez de peindre le vernis à ongles sur l'échantillon de tissu.
- Placer la lame en position verticale dans un endroit sombre et laisser le vernis à ongles à sec jusqu'à ce que complètement dur (généralement 30 minutes ou plus). À ce stade, l'image du tissu ou de conserver à -20 ° C pendant jusqu'à 1 semaine pour l'imagerie tard.
Buffer / Recettes Solution
10x PBS
- 80 g de NaCl
- 2,0 g de KCl
- 14,4 g Na 2 HPO 4
- 2,4 g de KH 2 PO 4
- pH à 7,4, H 2 O de 1 L
1x PBT
- 5 ml de PBS 10x
- 45 ml H 2 O
- 0,1% de Tween 20
20x SSC
- 175,3 g de NaCl
- 88,2 g de citrate de Na
- dans 800 ml de H 2 O
- pH à 7, H 2 O à 1 L
4x SSCT
- 200 ml 20x SSC
- 799 ml H 2 O
- 0,1% de Tween 20
0,1x SSC
- 5 ml 20x SSC
- 995 ml H 2 O
Mélange d'hybridation (20 pi; modifié à partir 11)
- 10 ul de formamide
- 4 ul de 50% de sulfate de dextrane
- 2 ul 20x SSC
- 4 ul de H 2 O
SBT 8 (10 ml)
- 2 ml 20x SSC
- 0,01 g de sérumalbumine bovine (BSA)
- 10 pi de Tween 20
- 7,9 ml H 2 O
8 La solution de blocage (10 ml)
- 0,3 g de BSA
- 10 pi de Tween 20
- 2 ml 20x SSC
- 8 ml de H 2 O
Solution de fixation au paraformaldéhyde (1 ml)
- 393,75 pi H 2 O (ajouter de l'eau en premier)
- 450 ul d'acide acétique glacial
- 156,25 ul 16% de paraformaldehyde
Résultats
Pour illustrer cette méthode, nous avons hybridé un ensemble de petits oligos synthétisés dans le commerce qui ont été modifiés chimiquement avec des conjugués fluorescents (figure 2) et une sonde plus biotinylé (fait par translation de coupure d'un produit PCR; Figure 2B), les chromosomes de plusieurs tissus différents types (voir le tableau 1). Les séquences cibles incluses répétitions satellites situés dans les régions péricentromériques (hétér...
Discussion
ADN ISH est fréquemment utilisé pour cartographier des séquences spécifiques à chromosomes. Nous avons décrit une méthode simple pour l'ADN ISH optimisé pour nombre élevé de copies, séquences hétérochromatiques. Plutôt que d'utiliser des lavages dans une solution de formamide, ce qui est une exigence dans d'autres protocoles ADN ISH existants, on place les lames de tissus montés directement sur un bloc de pré-chauffé pour dénaturer l'ADN. Ce procédé évite l'utilisation de grandes...
Déclarations de divulgation
The authors declare that they have no competing financial or any other conflict of interest.
Remerciements
We thank Zhaohua Irene Tang in the W. M. Keck Science Department for the use of her epifluorescence microscope and the Werren lab for donating Nasonia for dissections. This work was supported in part by an NIH-NRSA fellowship (5F32GM105317-02) to AML.
matériels
| Name | Company | Catalog Number | Comments |
| Poly-L-lysine coated slides (regular slides also can be used) | Sigma Aldrich | ||
| Ultrafine tweezers (5 gauge) | Dumont | ||
| 22 x 22 mm cover slips | Fisher | Sigmacote-treated by immersion for 15 sec, blotting dry, and wiping away all traces of Sigmacote so that cover slip is clear | |
| Sigmacote | Sigma | ||
| Filter paper | 75 - 150 mm | ||
| Paraffin wax paper | |||
| Heat block with thermometer | |||
| Dry incubator | |||
| Razor blades | |||
| Humidity chamber | empty pipette tip box or Tupperware, lined with moistened paper towels or Kimwipes | ||
| Coplin jars | with slide grooves | ||
| Aluminum foil | |||
| Pasteur pipettes | |||
| 1.5 ml microfuge tubes | |||
| Nail polish | clear or colored | ||
| P20 micropipette and plastic tips | |||
| Paperclips | 20 - 25 standard metal paperclips linked to form a chain | ||
| Reagents | |||
| 16% EM grade paraformaldehyde | Electron Microscopy Reagents | ||
| Acetic acid | Sigma | ||
| Liquid nitrogen | |||
| 100% Ethanol, chemical grade | |||
| Commercially synthesized, fluorescently labeled oligos | |||
| Long biotinylated probe | Invitrogen; Alternative steps 2.7.1-2.7.3 | e.g., nick translated and biotinylated with BioNick from Invitrogen | |
| Rhodamine-Avidin | Roche; Alternative steps 2.7.1-2.7.3 | for detection of long biotinylated probe | |
| Hybridization buffer | Recipe above | ||
| 4x SSCT | Recipe above | saline-sodium citrate + Tween | |
| 0.1x SSC | Recipe above | saline-sodium citrate | |
| Blocking solution | Recipe above | ||
| SBT | Recipe above | SSC, bovine serum albumin, Tween | |
| 1x PBT | Recipe above | phosphate-buffered saline + Tween | |
| 1x PBS | phosphate-buffered saline | ||
| Hypotonic solution | 0.5% sodium citrate in H2O | ||
| Formamide | Sigma Aldrich | ||
| Vectashield mounting medium with DAPI | Vector laboratories | ||
Références
- Blattes, R., Kas, E. Fluorescent in situ hybridization (FISH) on diploid nuclei and mitotic chromosomes from Drosophila melanogaster larval tissues. Cold Spring Harbor Protocols. 2009 (9), (2009).
- Dimitri, P. Fluorescent in situ hybridization with transposable element probes to mitotic chromosomal heterochromatin of Drosophila. Methods in Molecular Biology. 260, 29-39 (2004).
- Pardue, M. L. In situ hybridization to polytene chromosomes in Drosophila using digoxigenin-labeled probes. Cold Spring Harbor Protocols. 2011 (8), 1003-1006 (2011).
- Hoskins, R. A., et al. Heterochromatic sequences in a Drosophila whole-genome shotgun assembly. Genome Biology. 3 (12), (2002).
- Hoskins, R. A., et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin. Science. 316 (58331), 1625-1628 (2007).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nature Reviews Genetics. 13 (1), 36-46 (2012).
- He, B., et al. Mapping the pericentric heterochromatin by comparative genomic hybridization analysis and chromosome deletions in Drosophila melanogaster. Genome Research. 22 (12), 2507-2519 (2012).
- Pimpinelli, S., Bonaccorsi, S., Fanti, L., Gatti, M. Fluorescent in situ hybridization (FISH) of mitotic chromosomes from Drosophila larval brain. Cold Spring Harbor Protocols. 2010 (3), (2010).
- Larracuente, A. M., Noor, M. A., Clark, A. G. Translocation of Y-linked genes to the dot chromosome in Drosophila pseudoobscura. Molecular Biology and Evolution. 27 (7), 1612-1620 (2010).
- Ferree, P. M., Barbash, D. A. Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biology. 7 (10), e1000234 (2009).
- Williams, B. C., Karr, T. L., Montgomery, J. M., Goldberg, M. L. The Drosophila l(1)zw10 gene product, required for accurate mitotic chromosome segregation, is redistributed at anaphase onset. The Journal of Cell Biology. 118 (4), 759-773 (1992).
- Gatti, M., Bonaccorsi, S., Pimpinelli, S. Looking at Drosophila Mitotic Chromosomes. Methods in Cell Biology. 44, 371-391 (1994).
- Werren, J. H., Stouthamer, R. PSR (paternal sex ratio) chromosomes: the ultimate selfish genetic elements. Genetica. 117 (1), 85-101 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon