É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Método simples para DNA de Fluorescência
Neste Artigo
Resumo
Here, we present a simple method for performing fluorescence DNA in situ hybridization (DNA ISH) to visualize repetitive heterochromatic sequences on slide-mounted chromosomes. The method requires minimal reagents and it is versatile for use with short or long probes, different tissues, and detection with fluorescence or non-fluorescence-based signals.
Resumo
ADN de hibridação in situ (ISH ADN) é um método vulgarmente utilizado para mapeamento de sequências para as regiões cromossómicas específicas. Esta abordagem é particularmente eficaz em mapeamento de sequências altamente repetitivas para regiões heterocromáticas, onde abordagens computacionais enfrentam desafios proibitivos. Aqui nós descrevemos um protocolo simplificado para DNA ISH que contorna lavagens formamida que são etapas padrão em outros protocolos de DNA ISH. Nosso protocolo é otimizado para a hibridação com sondas de DNA vertente individuais curtas que levam corantes fluorescentes, o que efetivamente marcam sequências de DNA repetitivas dentro de regiões cromossômicas heterocromáticos através de um número de diferentes tipos de tecidos do inseto. No entanto, os pedidos podem ser estendido para usar com sondas maiores e visualização de sequências de DNA única cópia (não repetitivo). Demonstramos esse método mapeando várias sequências repetitivas diferentes de cromossomos esmagados de Drosophila melanogaster células neurais e Nasoniavitripennis espermatócitos. Mostramos padrões de hibridização, quer para pequenas sondas, sintetizados comercialmente e para uma sonda maior para comparação. Este procedimento utiliza material de laboratório simples e reagentes, e é ideal para os investigadores que têm pouca experiência com a realização de DNA ISH.
Introdução
ADN de hibridação in situ (ISH ADN) é um método vulgarmente utilizado para mapeamento de sequências para as regiões cromossómicas específicas. Sondas para regiões de cópia única dentro euchromatin pode ser gerado através de um punhado de abordagens, incluindo nick-tradução ou de fim-de rotulagem dos produtos de ADN longas 1,2 ea incorporação de deoxygenin (DIG) nucleotídeos -attached e seu reconhecimento através de uma ampla variedade de anticorpos conjugados do grupo 1-3. Visualização das sequências eucromáticos em poucos ou número único de cópia requer a utilização quer de sondas individuais grandes, com uma actividade específica elevada ou um cocktail de sondas múltiplas mais pequenas, que aumentam colectivamente sinal.
Em contraste, as sequências altamente repetitivas encontradas na heterocromatina, tais como DNAs satélite, são alvos mais fáceis para DNA ISH porque normalmente existe como dezenas de milhares de repetições agrupados em regiões cromossômicas únicos conhecidos como blocos. Elementos de transposição também pode serencontrado em altos números de cópia na distinta loci cromossômica 2. Nestes casos, as sondas individuais com baixa actividade específica pode rotular eficazmente sequências de heterocromatina, devido à sua hibridação em múltiplos locais. Sondas para seqüências repetitivas podem ser sintetizados oligonucleotídeos comercialmente como curtos (30-50 pb) e quimicamente conjugado com qualquer um dos vários grupos fluorescentes diferentes. Mapeando seqüências repetitivas dentro heterochromatin por meio de tecnologias de sequenciamento do genoma-se difícil devido aos desafios enfrentados em andaimes de construção dentro de blocos de satélite altamente repetitivas 4-6,7. Atualmente, ISH se destaca como a forma mais eficaz de mapear essas seqüências ao nível sub-cromossomo. Essa estratégia é importante para o mapeamento de um grande número de sequências repetitivas que estão sendo descobertos por estudos de genoma e transcriptoma de sequenciamento em curso.
A eficiência e facilidade de mapeamento de seqüências repetitivas nos cromossomos-montado slides seria greatly reforçada por um protocolo simplificado de DNA ISH. Por exemplo, os protocolos existentes para ADN ISH envolver múltiplas lavagens de tecidos hibridados em solução de formamida a 2,8, adicionando assim substancialmente para o tempo requerido para as sequências de mapeamento e também a produção de grandes quantidades de resíduos químicos para este reagente dispendioso. Aqui nós descrevemos um método DNA ISH revista que contorna a necessidade de lavagens formamida e utiliza equipamentos básicos de laboratório e reagentes. Este método foi originalmente projetado para o rápido mapeamento de seqüências de DNA altamente repetitivos em regiões heterocromáticas de neuroblastos de larvas de Drosophila usando oligos sintetizados no mercado que são conjugados com corantes fluorescentes. No entanto, este método também funciona para o mapeamento de seqüências repetitivas usando sondas maiores sintetizados através de outros meios 9,10 e em vários tipos de tecidos e de cromossomos diferentes. Além disso, este método pode ser utilizado para mapear sequências eucromáticos utilizando mais ou multiple, sondas curtas dentro da sequência euchromatic de interesse.
Protocolo
1. Dissecção de tecido e fixação (60 min)
- Para cérebros de Drosophila, colocar larvas 3rdin uma gota de 1x PBS (solução salina tamponada com fosfato). Escolha grande larvas 3º que estão rastreando ativamente de frascos ou garrafas que não estão superlotadas.
- Use um par ultrafine pinça para agarrar os ganchos na boca e outro par de pinça para agarrar 2/3 para baixo o comprimento do corpo (Figura 1A, B). Puxe delicadamente sobre os ganchos na boca para expor o cérebro, os gânglios ventral, glândulas salivares e parte do trato digestivo das larvas. Use as pinças para separar o cérebro e gânglios ventral (Figura 1A, B) a partir de outros tecidos e introduzi-lo numa gotícula de 1x PBT (salino tamponado com fosfato com Tween) numa placa de Petri de plástico.
- Para Nasonia dissecções testículo, escolha do sexo masculino pupas 3 dias de idade (corpos amarelos com olhos vermelhos). Masculino Nasonia têm comprimentos pequenos almofada asa em relação à females durante a fase de pupa (Figura 1C).
- Segure a pupa, na parte superior do abdômen, perto da região torácica, com um par de pinça, e usando o outro par de pinça, pegue a ponta muito distal do abdômen e retirar os testículos lágrima em forma de gota (que será cercado por gordura corpo que pode ser abalado suavemente; Figura 1D). Retire qualquer parte do corpo exteriores dos testículos, o que evitará esmagamento adequada, e coloque em uma gota de 1x PBT em uma placa de Petri de plástico.
- Para cada slide, dissecar um total de quatro ou cinco amostras de tecido (ie., Drosophila cérebros larval ou testículos Nasonia).
NOTA: Mais de cinco amostras levará a núcleos superlotadas e cromossomos. - Opcionalmente, para conseguir alguma separação de cromátides irmãs em regiões eucromáticos de cromossomos mitóticos, tratar o tecido com uma solução hipotônica: transferir cérebros de 1x PBT para uma gota de citrato de sódio a 0,5% para 5-10 (não mais do que 10) min.
NOTA: A colchicina (um inibidor da mitose) pode ser útil para aumentar o número de figuras de mitose na metáfase 11. No entanto, o seu uso é desnecessário se muito grande larvas saudáveis são usados, e até mesmo indesejável, pois pode afetar negativamente a resolução cromossomo, espalhando e morfologia. - Coloque uma gota (~ 20 ul) de solução fixadora (2,5% de paraformaldeído em ácido acético 45%) sobre a superfície de uma lamela de cobertura tratadas com Sigmacote limpo.
NOTA: Certifique solução fixadora nova para cada dia de uso. Soluções fixadoras que variam 1,8-3,7% de paraformaldeído em ácido acético 45% produzir os melhores resultados para os peixes. Usando diferentes do cérebro ou testículos tecidos, ou adaptar este protocolo de imuno-FISH pode exigir experimentando com diferentes fixadores (para uma lista de diferentes fixadores, ver 12). - Transferir cuidadosamente cada amostra de tecido a partir do tampão de dissecação (1x PBT) na gotícula fixador com pinças ultrafinas, minimizing a transferência de tampão de dissecação para a solução fixadora. Colocar as amostras de tecido de modo a que eles estão uniformemente espaçados uns dos outros no interior da gotícula de fixador. Incubar os tecidos em fixador durante 4 minutos à temperatura ambiente.
- Com cuidado, coloque um slide rosto poli-revestido de lisina para baixo para o deslizamento de tecido e tampa. Não pressione a este ponto, mas sim permitir que os dois entrar em contato com levemente para que a lamínula varas para o lado de baixo do slide. Inverter o slide para que o deslizamento tampa está no topo.
- Sanduíche do deslizamento slide / tecido / cobertura dentro de um pedaço dobrado de papel de filtro. Em uma superfície estável, usando o polegar, pressione muito firmemente em linha reta para baixo para a posição diretamente acima da lamela. Tenha cuidado para evitar deslizamento lateral da lamela (isso irá provocar manchas do tecido).
- Mergulhe o slide / tecido / tampa de deslizamento em nitrogênio líquido (ver aparelho na Figura 1E, F), e deixe descansar até que o nitrogênio pára fervente (não é fINE). Remover o slide e encaixe imediatamente a lamínula com uma lâmina de barbear fresco sacudindo um canto da lamínula em um sentido ascendente (evitar riscar o tecido fixado com a lâmina de barbear).
- O pré-resfriamento do slide / tecido / lamela em um bloco de gelo seco, tampa escorregar, por 1-2 min antes de submergir em nitrogênio líquido vai ajudar a prevenir os slides de rachar.
- Colocar imediatamente o diapositivo com o tecido em um frasco Coplin preenchido com etanol a 100% à temperatura ambiente e deixar em repouso durante pelo menos 5 minutos (este tempo pode ser mais longo, se necessário).
NOTA: etanol a 100% frio também poderia ser utilizado. - Remover o slide com o tecido, pavio fora excesso de etanol com uma Kimwipe (sem tocar no tecido fixo), e deixar o slide ao ar seco por 1 hora.
- Prossiga diretamente para a etapa 2 ou manter lâminas secas em baixa umidade do ar ou em uma câmara de dessecação por semanas a até meses antes de realizar a hibridação.
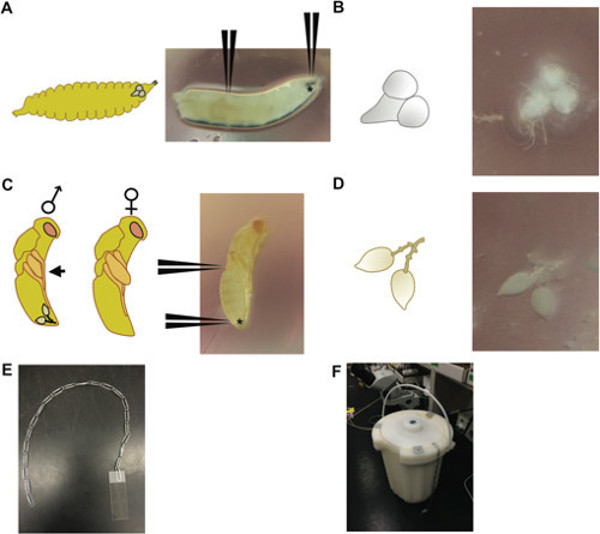
Figura 1: (A) A 3 rd instar Drosophila larva (à direita), com posições indicadas por onde pegar os ganchos na boca (indicado com *) e 2/3 do caminho para baixo as larvas para dissecar cérebros; (À esquerda) uma vista esquemática de uma larva de uma mesma fase de desenvolvimento, que descreve a posição relativa do cérebro no interior da cabeça de larvas. (B) O cérebro e gânglios dissecados a partir de uma ventral 3 Drosophila larva de terceiro instar (direita) e uma representação esquemática do presente tecido (à esquerda). pupa Nasonia (C) 3 dias de idade, em fase de olhos vermelhos corpo-amarelo. (D) Um par testis dissecado a partir de um velho pupa macho Nasonia 3 dia (à direita) com posições indicando onde pegar a pupa no posterior do abdómen (denotado com *) e a meio caminho no corpo; (À esquerda) esquemático masculino que descreve e feminino foip pupas; pupas macho pode ser distinguido pelas asas que não se estendem passado o perfil sagital (seta preta), em contraste com as fêmeas, que têm asas que se estendem para além do perfil; a posição relativa do par de testículo é mostrado na pupa de macho. (E) O aparelho de uma sequência de clipes-e método (F) utilizadas para mergulhar as lâminas em um vaso de azoto líquido. Várias cordas paperclip pode ser utilizado para a imersão simultânea de múltiplas lâminas.
2. A hibridação in situ (30 minutos no dia 1; 1 hr-2,5 h para sondas longas-no dia 2)
- Adicionar 1 ul (100 ng) de cada sonda de 20 ul de tampão de hibridação de 1,1x. Pipetar sonda / tampão de hibridação sobre a superfície do tecido fixado (evitar tocar no tecido).
- Com cuidado, coloque uma lamela diretamente sobre o tampão sonda / hibridação, certificando-se de que a tampa de deslizamento é centrado diretamente sobre o tecido. O tampão deve migrar para poenviar um e borda externa da tampa de deslizamento, sem deixar bolhas de ar.
Nota: Pequenas bolhas que não contactam tecido não colocam quaisquer problemas para o procedimento. Remover grandes bolhas com cuidado levantando um canto da lamela e cuidadosamente soltando-o de volta para o slide. - Coloque o deslizamento slide / tecido / cobertura sobre a superfície de um bloco pré-aquecido a 95 ° C (cover escorregar). Cubra com um pedaço grande de papel alumínio para evitar a exposição à luz. Com o deslizar incubar a 95 ° C durante 5 min.
NOTA: Um bloco de calor típico com furos para tubos podem ser viradas para proporcionar uma superfície plana para colocar o slide / tecido / lamela. - Retire o slide, deixando-a esfriar um pouco até que esteja quente ao toque. Cuidadosamente enrole um pedaço de Parafilm esticada ao redor da lamínula para selar o líquido abaixo dela.
- Colocar a lâmina selada dentro de uma câmara de humidade e colocar a câmara numa incubadora com uma pré-aquecido a 30 ° C. Incubar a 30 ° C durante 4 h até durante a noite.
- Criar uma câmara de umidade a partir de uma caixa de ponta vazio ou recipiente Tupperware pálpebras com Kimwipes umedecidas ou toalhas de papel colocadas na parte inferior.
NOTA: As sondas de oligo de ADN de cadeia simples foram concebidos para ser 28-33 bases para alcançar uma temperatura de fusão teórica (T m) dos 45-47 ° C. Estes m varia de comprimento e T refletir o fato de que muitas seqüências repetitivas que estudamos são AT-ricos e, portanto, têm baixo teor de GC. Sondas maiores, provavelmente, têm valores maiores M T; isto pode resultar em maior fundo de hibridação à temperatura de hibridação padrão de 30 ° C. Assim, resolução de alguns problemas com as temperaturas de hibridação podem ser necessários para obter os melhores resultados. Para encontrar a melhor temperatura de hibridização, aumento (ou redução) a temperatura de 5 ° C, de forma incremental.
- Criar uma câmara de umidade a partir de uma caixa de ponta vazio ou recipiente Tupperware pálpebras com Kimwipes umedecidas ou toalhas de papel colocadas na parte inferior.
- Retire cuidadosamente o Parafilm do slide e, em seguida, retire cuidadosamente a cobertura de vidro, levantando lentamente um canto. Wash o slide três vezes para 15 min cada lavagem em tampão SSC 0,1x. Cubra o recipiente Coplin com folha de alumínio durante as lavagens para minimizar a exposição à luz.
- Se não estiver usando uma sonda biotinilado longa, vá para o passo 2.8.
- Se estiver usando uma sonda longa biotinilado, secar a área em volta do tecido com um Kimwipe, tomando cuidado para não tocar no próprio tecido. Coloque 100 ml de solução sobre o tecido bloqueio e cobrir suavemente com uma lamela, tomando cuidado para evitar bolhas de armadilhagem. Enrole a deslizar sobre a lamela com Parafilm e lugar a 37 ° C durante 30 min.
- Remova cuidadosamente a lamela e secar ao redor do tecido com um Kimwipe. Pipet 100 l de rhodamine-avidina diluído 1: 1.000 em cuidados SBT sobre o tecido e cubra delicadamente com uma lamela, tomando para evitar a captura de bolhas. Enrole a deslizar sobre a lamela com Parafilm e lugar a 37 ° C durante 30 min.
- Remova cuidadosamente a lamela e lavar o slide 3 vezes por 5 minutos cada em 4x SSCT e depois3 vezes por 5 minutos cada em 0,1x SSC.
NOTA: As lâminas podem ser lavadas por longos períodos de tempo.
- Remover o slide e secar ao redor do tecido com um Kimwipe seco para remover o excesso de tampão (evite tocar no tecido). Coloque o lado do tecido slide-up em um local escuro durante 10-15 minutos ou até que a umidade se dissipa completamente.
- Pipetar 11 ul de meio de montagem Vectashield (com 4 ', 6-diamidino-2-fenilindole-DAPI) sobre o tecido. Com cuidado, coloque uma lamela limpa (não tratados com Sigmacote) diretamente sobre o centro do meio de montagem e tecido. O meio de montagem deve migrar lentamente para fora, para as bordas da lamínula.
NOTA: Se o meio de montagem não conseguir atingir a extremidade da lamela em todos os lados, em seguida, um adicional de 1-2 uL de meio de montagem pode ser aplicada a uma posição na borda da lâmina de cobertura para preencher o volume necessário. Neste caso, certifique-se de limpar qualquer excesso médio da superfície do slide antesvedação. - Selar as bordas da lamínula com unha polonês. Evite pintar a unha polonês sobre a amostra de tecido.
- Coloque o slide na posição vertical em um local escuro e deixar a unha seca polonês até que esteja completamente rígido (normalmente, 30 min ou mais). Neste ponto, a imagem do tecido ou armazenar a -20 ° C durante até 1 semana mais tarde para a imagiologia.
Buffer / Receitas Solution
10x PBS
- 80 g de NaCl
- 2,0 g de KCl
- 14,4 g de Na 2 HPO 4
- 2,4 g de KH 2 PO 4
- pH para 7,4, H2O a 1 L
1x PBT
- 5 ml de PBS 10x
- 45 ml de H2O
- 0,1% de Tween 20
20x SSC
- 175,3 g de NaCl
- 88,2 g de Na Citrato
- em 800 ml de H2O
- pH a 7, H2O a 1 L
4x SSCT
- 200 ml de 20x SSC
- 799 ml H 2 O
- 0,1% de Tween 20
0,1x SSC
- 5 ml de 20x SSC
- 995 ml de H2O
Mistura de hibridação (20 ul; modificado a partir do 11)
- 10 ul de formamida
- 4 ul de 50% de sulfato de dextrano
- 2 ul de 20x SSC
- 4 ul de H2O
SBT 8 (10 ml)
- 2 ml de 20x SSC
- 0,01 g de Albumina de Soro Bovino (BSA)
- 10 ul de Tween 20
- 7,9 ml de H2O
A solução de bloqueio 8 (10 ml)
- 0,3 g de BSA
- 10 ul de Tween 20
- 2 ml de 20x SSC
- 8 ml de H2O
Solução fixadora com paraformaldeído (1 ml)
- 393,75 ul H 2 O (adicione água primeiro)
- 450 ácido acético glacial ul
- 156,25 ul 16% de paraformaldeído
Resultados
Para demonstrar este método, que hibridou um conjunto de pequenos oligonucleótidos sintetizados comercialmente que foram quimicamente modificados com conjugados fluorescentes (Figura 2) e uma sonda mais longa biotinilado (feita através de tradução por cortes de um produto de PCR; Figura 2B), a partir de vários tecidos cromossomas diferente tipos (ver Tabela 1). As sequências alvo incluídas repete satélite localizadas em regiões (pericentroméricas heterocromá...
Discussão
DNA ISH é freqüentemente usado para mapear sequências específicas para os cromossomas. Nós descrevemos um método simples para o ADN ISH optimizado para elevado número de cópias, sequências de heterocromatina. Em vez de utilizar lavagens numa solução de formamida, que é um requisito em outros protocolos ISH ADN existentes, colocamos lâminas montadas-tecido directamente sobre um bloco de pré-aquecida para desnaturar o ADN. Este método evita o uso de grandes quantidades de formamida. Um passo fundamental par...
Divulgações
The authors declare that they have no competing financial or any other conflict of interest.
Agradecimentos
We thank Zhaohua Irene Tang in the W. M. Keck Science Department for the use of her epifluorescence microscope and the Werren lab for donating Nasonia for dissections. This work was supported in part by an NIH-NRSA fellowship (5F32GM105317-02) to AML.
Materiais
| Name | Company | Catalog Number | Comments |
| Poly-L-lysine coated slides (regular slides also can be used) | Sigma Aldrich | ||
| Ultrafine tweezers (5 gauge) | Dumont | ||
| 22 x 22 mm cover slips | Fisher | Sigmacote-treated by immersion for 15 sec, blotting dry, and wiping away all traces of Sigmacote so that cover slip is clear | |
| Sigmacote | Sigma | ||
| Filter paper | 75 - 150 mm | ||
| Paraffin wax paper | |||
| Heat block with thermometer | |||
| Dry incubator | |||
| Razor blades | |||
| Humidity chamber | empty pipette tip box or Tupperware, lined with moistened paper towels or Kimwipes | ||
| Coplin jars | with slide grooves | ||
| Aluminum foil | |||
| Pasteur pipettes | |||
| 1.5 ml microfuge tubes | |||
| Nail polish | clear or colored | ||
| P20 micropipette and plastic tips | |||
| Paperclips | 20 - 25 standard metal paperclips linked to form a chain | ||
| Reagents | |||
| 16% EM grade paraformaldehyde | Electron Microscopy Reagents | ||
| Acetic acid | Sigma | ||
| Liquid nitrogen | |||
| 100% Ethanol, chemical grade | |||
| Commercially synthesized, fluorescently labeled oligos | |||
| Long biotinylated probe | Invitrogen; Alternative steps 2.7.1-2.7.3 | e.g., nick translated and biotinylated with BioNick from Invitrogen | |
| Rhodamine-Avidin | Roche; Alternative steps 2.7.1-2.7.3 | for detection of long biotinylated probe | |
| Hybridization buffer | Recipe above | ||
| 4x SSCT | Recipe above | saline-sodium citrate + Tween | |
| 0.1x SSC | Recipe above | saline-sodium citrate | |
| Blocking solution | Recipe above | ||
| SBT | Recipe above | SSC, bovine serum albumin, Tween | |
| 1x PBT | Recipe above | phosphate-buffered saline + Tween | |
| 1x PBS | phosphate-buffered saline | ||
| Hypotonic solution | 0.5% sodium citrate in H2O | ||
| Formamide | Sigma Aldrich | ||
| Vectashield mounting medium with DAPI | Vector laboratories | ||
Referências
- Blattes, R., Kas, E. Fluorescent in situ hybridization (FISH) on diploid nuclei and mitotic chromosomes from Drosophila melanogaster larval tissues. Cold Spring Harbor Protocols. 2009 (9), (2009).
- Dimitri, P. Fluorescent in situ hybridization with transposable element probes to mitotic chromosomal heterochromatin of Drosophila. Methods in Molecular Biology. 260, 29-39 (2004).
- Pardue, M. L. In situ hybridization to polytene chromosomes in Drosophila using digoxigenin-labeled probes. Cold Spring Harbor Protocols. 2011 (8), 1003-1006 (2011).
- Hoskins, R. A., et al. Heterochromatic sequences in a Drosophila whole-genome shotgun assembly. Genome Biology. 3 (12), (2002).
- Hoskins, R. A., et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin. Science. 316 (58331), 1625-1628 (2007).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nature Reviews Genetics. 13 (1), 36-46 (2012).
- He, B., et al. Mapping the pericentric heterochromatin by comparative genomic hybridization analysis and chromosome deletions in Drosophila melanogaster. Genome Research. 22 (12), 2507-2519 (2012).
- Pimpinelli, S., Bonaccorsi, S., Fanti, L., Gatti, M. Fluorescent in situ hybridization (FISH) of mitotic chromosomes from Drosophila larval brain. Cold Spring Harbor Protocols. 2010 (3), (2010).
- Larracuente, A. M., Noor, M. A., Clark, A. G. Translocation of Y-linked genes to the dot chromosome in Drosophila pseudoobscura. Molecular Biology and Evolution. 27 (7), 1612-1620 (2010).
- Ferree, P. M., Barbash, D. A. Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biology. 7 (10), e1000234 (2009).
- Williams, B. C., Karr, T. L., Montgomery, J. M., Goldberg, M. L. The Drosophila l(1)zw10 gene product, required for accurate mitotic chromosome segregation, is redistributed at anaphase onset. The Journal of Cell Biology. 118 (4), 759-773 (1992).
- Gatti, M., Bonaccorsi, S., Pimpinelli, S. Looking at Drosophila Mitotic Chromosomes. Methods in Cell Biology. 44, 371-391 (1994).
- Werren, J. H., Stouthamer, R. PSR (paternal sex ratio) chromosomes: the ultimate selfish genetic elements. Genetica. 117 (1), 85-101 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados