Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine grafische Benutzeroberfläche für die softwaregestützte Verfolgung der Proteinkonzentration in dynamischen zellulären Vorsprüngen
In diesem Artikel
Zusammenfassung
Wir präsentieren eine Softwarelösung zur halbautomatischen Verfolgung der relativen Proteinkonzentration entlang der Länge der dynamischen zelligen Vorsprünge.
Zusammenfassung
Filopodien sind dynamische, fingerähnliche zelluläre Vorsprünge, die mit Migration und Zell-Zell-Kommunikation verbunden sind. Um die komplexen Signalisierungsmechanismen, die der filopodialen Initiation, der Dehnung und der anschließenden Stabilisierung oder dem Retraktion zugrunde liegen, besser zu verstehen, ist es entscheidend, die räumlich-zeitliche Proteinaktivität in diesen dynamischen Strukturen zu bestimmen. Um die Proteinfunktion in Filopodien zu analysieren, haben wir vor kurzem einen halbautomatischen Tracking-Algorithmus entwickelt, der sich an filopodiale Formänderungen anpasst und so eine parallele Analyse der Protrusionsdynamik und der relativen Proteinkonzentration entlang der gesamten filopodialen Länge ermöglicht. Hier stellen wir Ihnen ein detailliertes Schritt-für-Schritt-Protokoll für optimierte Zellhandling, Bilderfassung und Softwareanalyse vor. Für die Verwendung von optionalen Merkmalen während der Bildanalyse und der Datendarstellung stehen wir Ihnen weiter zur Verfügung, sowie Leitfaden zur Fehlerbehebung für alle kritischen Schritte auf dem Weg. Schließlich beinhalten wir auch einen Vergleich der dBeschreibende Bildanalyse-Software mit anderen Programmen zur Filopodien-Quantifizierung. Zusammen stellt das vorgestellte Protokoll einen Rahmen für eine genaue Analyse der Proteindynamik in filopodialen Vorsprüngen mit Bildanalyse-Software dar.
Einleitung
Die räumlich-zeitliche Kontrolle von Aktin-regulatorischen Proteinen ist mit der Filopodium-Dynamik 1 , 2 verbunden . Die Verfolgung der räumlich aufgelösten Proteinkonzentration entlang der gesamten filopodialen Länge durch die Zeit ist daher entscheidend, um unser Verständnis der Mechanismen, die der Einleitung, der Dehnung, der Stabilisierung oder dem Zusammenbruch dieser dynamischen Strukturen 3 , 4 zugrunde liegen, voranzutreiben. Im Gegensatz zur Proteinanalyse im Cytosol, wo viele Zellformveränderungen in einem größeren Maßstab auftreten, sind Filopodien dynamische Mikrostrukturen, die sich kontinuierlich 5 abbiegen und sich biegen, wodurch eine Analyse unter Verwendung eines einfachen Ansatzes wie einer Zeilenabtastung verhindert wird.
Verschiedene Softwarelösungen für die Verfolgung der filopodischen Form sind verfügbar 6 , 7 , 8 , 9 . GleichermaßenIse, Software zur ratiometrischen Verfolgung der Proteindynamik innerhalb des Zellkörpers wurde entwickelt 10 , 11 . Um die automatisierte Verfolgung der filopodischen Form und der räumlich-zeitlichen Proteinanalyse zu kombinieren, haben wir vor kurzem eine Bildanalyse-Software entwickelt, die auf dem Konvex-Rumpf-Algorithmus 12 basiert. Dieses neuartige Analyseverfahren, das über eine grafische Benutzeroberfläche (GUI) betrieben wird, kombiniert erstmals die relative Proteinkonzentration entlang der filopodialen Länge und der Wachstumsgeschwindigkeit und ermöglicht so die genaue Messung der räumlich-zeitlichen Proteinverteilung unabhängig von der Bewegung dieser Dynamische Strukturen 12
Die Idee hinter der Software (Quellcode ist frei verfügbar, siehe unten) ist, dass einer der Ecken des konvexen Rumpfes mit der Spitze des Filopodiums übereinstimmt ( Abbildung 1A ). Durch das Betrachten des nachfolgenden RahmensR der nächste Scheitel des konvexen Rumpfes, die bewegte Spitze kann während des ganzen Films verfolgt werden. Sobald die Spitze in jedem Rahmen erfaßt wird, wird ihre Position verwendet, um eine Achse zu zeichnen, indem sie die Spitze mit einem Bezugspunkt an der Basis des Filopodiums verbindet ( Fig. 1B ). Schließlich werden unter Verwendung äquidistenter Knotenpunkte, deren Positionen durch das mittlere Pixel mit maximaler Intensität entlang der Linie orthogonal zur Achse bestimmt werden, verwendet, um ein Rückgrat zu bestimmen, das der filopodischen Form folgt. Unter Ausnutzung dieses adaptiven Rückgrats wird ein Kymograph erzeugt, um filopodiales Wachstum und Proteinkonzentrationen für bis zu drei Kanäle entlang der filopodialen Länge zu verfolgen ( Abbildung 1C ).
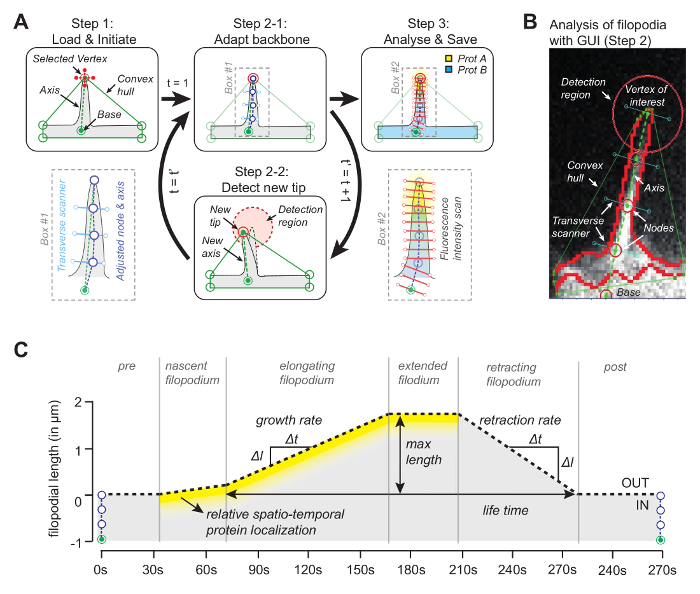
Abbildung 1: Funktionsprinzip der Bildanalyse-Software. ( A ) Der Algorithmus dahinterdie Software. In Schritt 1 spezifiziert der Benutzer die Referenz (Basis) und den Scheitelpunkt (die Spitze) des Filopodiums. In Schritt 2-1 wird das Rückgrat des Filopodiums unter Verwendung des Medianpixels mit maximalem Intensitätswert erhalten. In Schritt 3 wird das Rückgrat für das räumliche Proteinintensitätsprofil verwendet. In Schritt 2-2 verfolgt die Software automatisch die Spitze im nachfolgenden Rahmen. Die ganze Prozedur iteriert. ( B ) Schnappschuss des Algorithmus mit echtem Filopodium, das wichtige Elemente wie den konvexen Rumpf einführt, der für die Verfolgung verwendet wird. ( C ) Übersicht der Parameter, die mit dem Algorithmus gemessen werden können. Diese Zahl wurde aus Referenz 12 geändert. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Die Bildanalyse-Software wird in Matlab (als Programmiersoftware bezeichnet) über einen grafischen Einsatz betriebenR-Schnittstelle Um die Flexibilität und Robustheit für die jeweilige experimentelle Einstellung zu maximieren, kann der Anwender eine Reihe von Tracking-Parametern ( zB zulässiger Biegewinkel und Interframe-Bewegung) anpassen und auch Korrekturen an den Filmen vornehmen ( zB Zuschneiden, Drehen, Entfernen von unerwünschten Objekten) ( Fig. 2A und Tabelle 1) .
| GUI | Nein. | Zwingend notwendig | Beschreibung | Name (in GUI) | ||||
| # 1 | 1a | Y | Laden der gestapelten .tiff-Datei, die den Zellenkörper darstellt (mit dem eingecheckten Feld) oder den überlagerten Zellenkörper aus Kanälen erstellen | CellBody | ||||
| # 1 | 1b | Y | Laden der gestapelten Bilddatei entsprechend Protein 1 | Protein 1 | ||||
| # 1 | 1c | Y | Laden der gestapelten Bilddatei entsprechend Protein 2 | Protein 2 | ||||
| # 1 | 1d | Y | Laden der gestapelten Bilddatei entsprechend Protein 3 | Protein 3 | ||||
| # 1 | 1e | N | Setzt alles auf vorgespeicherte gestapelte Bilddateien zurück | Zurücksetzen | ||||
| # 1 | 2a | Y | Bildlaufleiste zur Ermittlung des Anfangsrahmens für die Analyse im GUI-Fenster # 2 | N / A | ||||
| # 1 | 2b | Y | Bildlaufleiste zur Bestimmung des endgültigen Rahmens für die Analyse im GUI-Fenster # 2 | N / A | ||||
| # 1 | 2c | Y | Bildlaufleiste für KurveNt Rahmen | N / A | ||||
| # 1 | 2d | N | Der Grauwert der Pixel, unterhalb derer alle Pixel auf Null gesetzt werden | N / A | ||||
| # 1 | 2e | N | Der Grauwert der Pixel, über die alle Pixel auf Maximalwerte gesetzt werden | N / A | ||||
| # 1 | 2f | N | Setzen Sie die Intensitätswerte der von <2e> & <2f> angegebenen Pixel | Set | ||||
| # 1 | 2g | N | Spielen Sie den Intensitäts-angepassten Film | Spielen | ||||
| # 1 | 2h | N | Bild zuschneiden | Ernte | ||||
| # 1 | 2i | N | Bild drehen | Drehen | ||||
| # 1 | 2j | N | Regionen im ganzen Stapel löschen | Regionen löschen | ||||
| # 1 | 3a | Y | Klicken Sie auf, um das 'Analysis Window' zu öffnen (GUI-Fenster # 2) | Tracking-Fenster | ||||
| # 1 | 3b | Y | Geben Sie die Größe eines Pixels in Mikron ein | Geben Sie die Pixelgröße ein | ||||
| # 2 | 4 | Y | Klicken Sie hier, um das Rand- / Randbild des überlagerten Zellenkörpers zu erzeugen | Grenze | ||||
| # 2 | 5 | Y | Klicken Sie auf die Basis und die Spitze der Filopodien | Referentum | ||||
| # 2 | 6a | Y | Geben Sie die Anzahl der Segmente oder Knoten ein | Anzahl der Segmente | ||||
| # 2 | 6b | Y | Geben Sie die Scanlänge ein (senkrecht zur Achse) | Scan-Breite | ||||
| # 2 | 6c | Y | Geben Sie die Länge ein, über die die Filopodie beginnt zu verbiegen | Genaue Meas nach | ||||
| # 2 | 6d | Y | Geben Sie den Radius des Spitzenerkennungskreises ein (dh Bereich, in dem der Scheitelpunkt im nächsten Rahmen lokalisiert werden kann) | Radius der Spitzenerkennung | ||||
| # 2 | 6e | Y | Geben Sie den maximalen Winkel ein, den das Filopodium von der vertikalen Achse biegen kann | Winkelschwelle | ||||
| # 2 | 6f | N | Fügen Sie Referenzpunkte für Basis und Spitze für diesen bestimmten Rahmen hinzu | Referenz auswählen | ||||
| # 2 | 6g | N | Geben Sie die Länge ein, über die die Filopodie für diesen bestimmten Rahmen beginnt | Genaue Meas nach | ||||
| # 2 | 6h | N | Geben Sie den Radius des Erkennungskreises für diesen Rahmen ein | Radius der Spitzenerkennung | ||||
| # 2 | 6i | N | Nach Eingabe aller Parameter für den jeweiligen Rahmen klicken Sie, um die Werte in den Speicher und die Datei für weitere Referenz zu speichern | Hinzufügen | ||||
| # 2 | 6j | N | Klicken Sie hier, um die manuellen Parameter für diesen Rahmen zu löschen | Löschen | ||||
| # 2 | 6k | N | Klicken Sie auf, um alle Parameter zu löschen, die manuell über das 'optionale Merkmalsfeld' für alle Frames gespeichert ist | Zurücksetzen | ||||
| # 2 | 6l | N | Check-in vor dem Tracking, um alle Tracking-Ergebnisse im Speicher für zukünftige Referenz zu speichern | Geschichtespur | ||||
| # 2 | 7 | Y | Klicken Sie hier, um das Tracking zu starten | Track & Analysieren | ||||
| # 2 | 8a | N | Klicken Sie hier, um die Intensität des Proteinkanals zu verfolgen | Analysieren Sie Proteinintensitäten | ||||
| # 2 | 8b | N | Check-in, um die Protein-Kanal-Intensität entlang der filopodialen Länge zu verfolgen | Ganze Filopodien | ||||
| # 2 | 8c | N | Check in für die Verfolgung der Referenz-Protein oder Protein A | ProteinA | ||||
| # 2 | 8d | N | Check in für die Verfolgung des Proteins B | ProteinB | ||||
| # 2 | 8e | N | Check in für die Verfolgung des Proteins C | Protein | ||||
| # 2 | 8f | N | Check in, um die durchschnittliche Proteinintensität in th zu verfolgenE Tipp | Führender Tipp | ||||
| # 2 | 8g | N | Geben Sie die Länge der Spitze ein | Tipplänge | ||||
| # 2 | 8h | N | Geben Sie den Mindestabstand von der Basis ein, über der die Spitze beginnt zu bilden | Schwelle | ||||
| # 2 | 8i | N | Klicken Sie hier, um die Ergebnisse der führenden Tip-Analyse zu speichern | Knopf drücken | ||||
| # 2 | 9a | N | Klicken Sie hier, um eine ratiometrische Proteinanalyse einzuleiten | Vergleichen | ||||
| # 2 | 9b | N | Check in, um Protein B in Bezug auf A zu vergleichen | Log10 (B / A) | ||||
| # 2 | 9c | N | Check in, um Protein C in Bezug auf A zu vergleichen | Log10 (C / A) | ||||
| # 2 | 9d | N | Check in, um Protein B in Bezug auf A an der Spitze zu vergleichen | Log10 (B / A) | ||||
| # 2 | 9e | N | Check in, um Protein C in Bezug auf A an der Spitze zu vergleichen | Log10 (C / A) | ||||
| # 2 | 10 a | N | Wähle andere Farbkarte (Voreinstellung: Jetplot) | Farbkarte | ||||
| # 2 | 10b | N | Bearbeiten Sie die Farbkarte | Farbkarte bearbeiten | ||||
Tabelle 1: Übersicht aller Funktionen In der GUI Windows # 1 und # 2 präsentieren.
Sobald dies geschehen ist, erstellt das Programm einen konvexen Rumpf und verfolgt automatisch die Spitze während des Films. Aus dem Film extrahierte Parameter, wie z. B. ein ratiometrisches Kymograph, Wachstumsgeschwindigkeit und filopodiale Länge aWieder angezeigt und auch im Arbeitsordner als Bilder und als Datendateien gespeichert. Andere Parameter wie filopodiale Lebensdauer, Wachstumsrate und Retraktionsrate können dann extrahiert und aus den gespeicherten Datendateien weiter analysiert werden ( Abbildung 2B ).
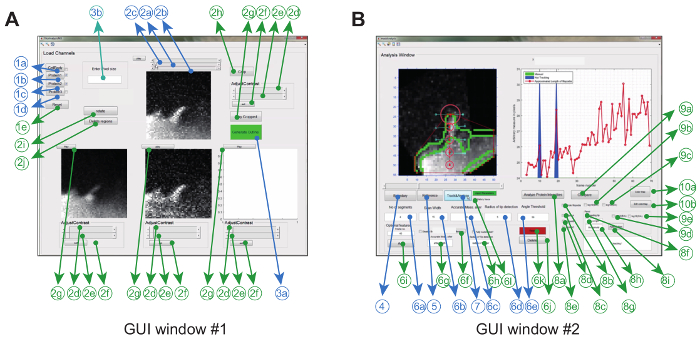
Abbildung 2: Grafische Benutzeroberfläche zur Verwendung der Bildanalyse-Software. ( A ) GUI-Fenster # 1 dient zum Laden und Verarbeiten von Bildern. Das Programm kann bis zu 3 Proteinkanäle laden, wobei 2 Kanäle paarweise verglichen werden. Das Fenster kommt mit obligatorischen (blauen) und optionalen Funktionen (grün) für die Vorverarbeitung der Bilder vor dem Tracking ( B ) GUI Window # 2 wird für die Verfolgung der Filopodium sowie räumlich-zeitliche und Verhältnis-metrische Protein-Analyse verwendet. Auch hier sind die optionalen Funktionen grün markiert. Diese Figur ist modifi gewesenAus Referenz 12 . Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Hier stellen wir Ihnen ein detailliertes Protokoll zur Probenvorbereitung und Softwarehandhabung vor. Wir beginnen mit detaillierten Anweisungen zur Kultivierung von Zellen und dem Erwerb von Filmen, die für die Bildanalyse optimiert sind. In diesem Abschnitt zur Datenerfassung folgt eine detaillierte Beschreibung für den Betrieb der Bildanalyse-Software. Während des gesamten Protokolls führen wir kritische Schritte und optionale Funktionen ein, die beim Sammeln und Verarbeiten von Daten berücksichtigt werden sollten. Schließlich analysieren wir Filopodien aus verschiedenen Modellsystemen mit der Bildanalyse-Software, bevor wir mit einem Vergleich der beschriebenen Bildanalyse-Software mit anderen Programmen zur filopodischen Quantifizierung und einer Diskussion über Einschränkungen und zukünftige Richtung schließen.
Protokoll
1. Zellkultur
- Kultur-HeLa- oder COS-Zellen in Dulbecco's Modified Eagle Medium (DMEM) mit 4,5 g / l D-Glucose, L-Alanin-L-glutamintipeptid, Pyruvat, 10% fötalem Rinderserum und 10 Einheiten / ml Penicillin / Streptomycin. Kultur-Neuronen in Kulturmedien ohne L-Glutamin, Glutaminsäure oder Asparaginsäure, ergänzt mit 0,5 mM L-Alanin-L-glutamin-Dipeptid, serumfreien neuronalen Nahrungsergänzungsmitteln und 10 Einheiten / ml Penicillin / Streptomycin.
- Sobald 40% Konfluenz erreicht ist, transfizieren Zellen mit Konstrukten der Wahl mit einem Transfektionsreagenz nach Herstelleranweisungen. Halten Sie transfizierte Zellen im Inkubator bei 37 ° C und 5% CO 2 für 15-18 h.
- Um die Veränderungen des pH-Werts und der Osmolarität während der Bildaufnahme zu reduzieren, füllen Sie die Zellkulturkammern (vor der Bildgebung) bis zu 90% mit 37 ° C vorgewärmtem Medium, das 20 mM 4- (2-Hydroxyethyl) -1 enthält -piperazethansulfonsäure (HEPES). VersiegelnDen Deckel, eine dünne Schicht Vakuumfett auf der Innenseite des Deckels auftragen und ihn vorsichtig auf die Kulturkammer auftragen, die die transfizierten Zellen enthält.
2. Bildaufnahme
HINWEIS: Die Länge der Filopodien variiert von 2-10 μm 13 . Filopodien wachsen mit einer durchschnittlichen Geschwindigkeit von 0,05-0,1 μm / s 13 , 14 .
- Erfassen von Bildern mit einem Mikroskop mit 60X oder 100X Objektiv und kein Pixel Binning (hier ein Spinndisk konfokales Mikroskop). Verwenden Sie Erfassungsraten größer als 1 Hertz (Hz), um die filopodiale Dynamik zu verfolgen. Zur Minimierung von unscharfen Artefakten, Bildfilopodien nahe der Basalmembran ( dh Substratoberfläche).
- Um eine reibungslose Verfolgung zu gewährleisten, stellen Sie die Belichtungszeit der Kamera und die Laserintensität so ein, dass das Signal-Rausch-Verhältnis (SNR) größer als 4 ist. Vermeiden Sie die Sättigung einzelner Kanäle ( dh Pixelwerte von255 für 8-Bit-Bilder und 65.535 für 16-Bit-Bilder), da dies eine anschließende Bildanalyse ausschließt.
- Um ein Durchbluten zu vermeiden, verwenden Sie nur Fluoreszenzmarkierungen, die mit Laserlinien und Filtern des Mikroskops kompatibel sind (Details siehe Referenz 15 ).
3. Bildvorverarbeitung
HINWEIS: Verwenden Sie ImageJ oder andere verfügbare Software zur Vorverarbeitung von Bildern 16 , 17 .
- Wenn sich die Probe bewegt, korrigieren Sie die seitliche Drift mit der verfügbaren Software ( zB https://github.com/NMSchneider/fixTranslation-Macro_for_ImageJ) vor der Analyse. Filme mit axialer Drift ausschließen (dh Bewegung in z-Richtung).
- Korrekter Hintergrund ( dh Grauwerte von Bereichen außerhalb der Zelle), Bleichen ( dh kontinuierlicher Verlust bei Fluoreszenzintensität durch Beschädigung von Fluoreszenzproteinen) und eventueller Entlüftung ( dh Signal von einer GrippeOrescence-Sonde in den beiden Kanälen) mit der verfügbaren Software ( zB http://imagej.net/Kategorie:Plugins und 16 , 17 ).
Hinweis: Fluoreszenzintensitäten einzelner Kanäle werden durch die Software nicht verändert. - Um eine spätere Bildanalyse sicherzustellen, speichern Sie Filme, die bestimmten Proteinkanälen in Graustufen entsprechen, als '.tiff' gestapeltes Dateiformat im Arbeitsordner.
HINWEIS: Die Abmessungen (Größe, Länge) der Stapel müssen für alle Kanäle gleich sein.
4. Bildanalyse - Schritt 1: Bilder laden
HINWEIS: Die hier beschriebene Software wurde in Matlab (als Programmiersoftware bezeichnet) geschrieben und läuft nur mit diesem Programm.
- Laden Sie den gezippten Ordner mit allen benötigten Dateien für die Bildanalyse von der folgenden Seite herunter: https://campus.uni-muenster.de/en/einrichtungen/impb0/nanoscale-forces-in-cells/softwsind/. Entpacken und kopieren Sie Dateien in den Arbeitsordner.
- Nach der Installation öffnen Sie die Programmiersoftware und führen 'filopodiaAnalysisM3.fig' aus. GUI-Fenster # 1 wird geöffnet, wie in Abbildung 2A gezeigt .
- Laden Sie die gespeicherten gestapelten '.tiff'-Dateien, die einem bestimmten Protein von Interesse im GUI-Fenster # 1 entsprechen, mit den Tasten <1b> für Protein A, <1c> für Protein B, <1d> für Protein C. Siehe Abbildung 2 und Tabelle 1 für Details.
HINWEIS: Das im GUI-Fenster # 1 gezeigte Protein A fungiert als Referenzkanal für die endgültige ratiometrische Analyse. - Erstellen Sie ein überlagertes Bild der Zelle aus den Proteinkanälen, indem Sie auf <1a> klicken.
- Klicken Sie auf <2a>, um den ersten Frame und <2b> für den letzten Frame zuzuweisen, der für die Analyse verwendet wird.
- Optional können Sie den interessierenden Bereich (ROI) mit dem Filopodium von Interesse mit der Taste <2h> zuschneiden, das Bild drehenSingen Sie die Taste <2i> oder löschen Sie unerwünschte Bereiche mit dem Freiland-Werkzeug <2j>.
HINWEIS: Um die Bildanalyse zu optimieren, empfiehlt es sich, den ROI ( zB Zuschneiden, Drehen, Löschen) zusammen mit den anderen Vorverarbeitungsschritten (Hintergrundsubtraktion, Bleichkorrekturen etc.) mit ImageJ zu isolieren. - Bewegen Sie den Schieberegler <2c> für jeden Rahmen für die Qualitätskontrolle und prüfen Sie, ob das Filopodium im ganzen Film deutlich sichtbar bleibt.
5. Bildanalyse - Schritt 2: Trace erzeugen
- Klicken Sie im GUI-Fenster # 1 auf die Schaltfläche <3a>, um das GUI-Fenster # 2 zu öffnen (siehe Abbildung 2B ).
- Klicken Sie im GUI-Fenster # 2 auf die Schaltfläche <4>, um die Maske des überlagerten Zellenkörpers zu erzeugen (im GUI-Fenster # 1 nach dem Klicken auf <1a> generiert). Das Programm erzeugt auch die Grenze der Maske, wo der konvexe Rumpf implementiert wird, um die Scheitelpunkte zu erhalten.
- CliCk-Taste <5>; Ein Cursor erscheint. Verwenden Sie den Cursor, um die Basis zu wählen (von wo aus der Abstand der filopodischen Spitze gemessen wird), gefolgt von der Spitze der Filopodien im Rahmen, wo sie zuerst erscheint. Um dies zu tun, verschiebe den Schieberegler im Fenster # 2).
HINWEIS: Um Fehler bei der Datenausgabe zu minimieren, legen Sie den Basispunkt senkrecht unter die filopodiale Spitze entlang der Achse. Die Positionierung des Basispunktes an anderen Stellen ( z. B. seitlich verschoben) kann eine Richtungsvorspannung in Fluoreszenzwerten innerhalb des Zellkörpers einführen. - Wählen Sie mit <6c> die Schwellenlänge (oberhalb der sich die Filopodien biegen).
Anmerkung: Diese Länge ist definiert als der Abstand zwischen dem Basispunkt (ausgewählt unter <5>) und der Grenze des Zellkörpers ( dh der Region, von wo aus die Filopodien anfangen zu wachsen). Der Abstand wird in Pixeln gemessen. - Geben Sie die Anzahl der Segmente an, die verwendet werden, um die Form der Filopodien im Feld <6a> zu approximieren.
HINWEIS: Die mDie maximale Anzahl der Segmente hängt von der maximalen Länge ab, die durch das Filopodium erreicht wird, aber auch, wie sich das Filopodium biegt. Wählen Sie nicht eine Anzahl von Segmenten, die größer sind als die Anzahl der Pixel zwischen der Basis und der Spitze ( dh die Schwellenlänge). Auswählen mehrerer Segmente als die in Schritt 5.4 definierte Schwellenlänge. ( Dh Anzahl von Pixeln zwischen Basis und Spitze) führt zu einer Überschätzung der Länge des Filopodiums. - Geben Sie die Scan-Breite an, die als horizontaler Scanner dient, um die Knoten in <6b> zu platzieren.
HINWEIS: Diese Knoten werden vom Programm verwendet, um die Linie zu verbinden, die die Basis und die Spitze mit dem Körper des Filopodiums verbindet ( dh das Rückgrat zu schaffen). Als Ausgangspunkt setzen Sie einen Wert in Pixel gleich der maximalen Länge multipliziert mit einem Faktor von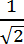 .
. - Geben Sie den Scan-Radius (in Pixeln) im Feld <6d> an. Verwenden Sie einen Wert, der etwa 50% größer ist als der obServiert Inter-Frame-Spitze Verschiebung.
HINWEIS: Bei Verwendung eines sehr großen Scan-Radius können unerwünschte Konvex-Rumpf-Punkte in Rahmen aufgenommen werden, in denen keine echte Filopodialspitze vorhanden ist ( z. B. außerhalb der Flugzeugbewegung oder des niedrigen SNR). - Geben Sie den Biegewinkel im Feld <6e> an.
HINWEIS: Die Winkelschwelle wird durch den maximalen Winkel bestimmt, den das Filopodium während der gesamten Analyse biegt. Die Angabe der Winkelschwelle hilft der Software, die Verfolgung von unerwünschten Strukturen, die von der Seite der Filopodien wachsen, auszuschließen, wenn sich die Filopodien in Richtung des Zellkörpers biegt. Das Programm arbeitet zuverlässig für Filopodien mit Kippwinkeln von weniger als 45 Grad von der vertikalen Achse. - Um das Tracking zu starten, klicken Sie im GUI-Fenster # 2 auf die Schaltfläche . Klicken Sie auf das Feld "History Trace" im GUI-Fenster # 2, um das gesamte Tracking-Protokoll für zukünftige Referenz zu speichern.
HINWEIS: Nach Abschluss des Tracking-Vorgangs wird die Länge des Filopodiums in jedem Frame gespeichertDas Blatt 'length_vel' von 'dynamics.xlsx' für zukünftige Referenzen. Ebenso werden alle anderen Tracking-Parameter in dem Blatt mit dem Namen 'parameters' von 'dynamics.xlsx' gespeichert. - Wenn die Filopodie nicht automatisch in allen Frames erkannt wird, verwenden Sie die folgenden Schritte, um sie manuell zu korrigieren.
- Besuchen Sie den jeweiligen Rahmen mit dem Schieberegler in Fenster # 2 und wählen Sie die Spitze des Filopodiums manuell aus.
Hinweis: Frames, bei denen keine konvexen Rumpfpunkte erkannt werden, werden durch eine blaue Region im Tracking-Fenster des GUI Window # 2 dargestellt. Es ist zwingend erforderlich, die Schaltfläche "History Trace" zu überprüfen, bevor das Tracking-Programm für den Zugriff auf die Knotenkoordinaten in diesem Frame initiiert wurde. - Wählen Sie die Referenzpunkte (Basis gefolgt von Tipp) mit <6f> im GUI-Fenster # 2. Spezifizieren Sie die anderen Parameter wie 'Scanlänge', 'Genaue Messung nach' und 'Maximaler Biegewinkel'; Für diesen Rahmen, wie in den Schritten 5.4-5.8 beschrieben.
- Klicken Sie auf die Schaltfläche <6i>, um die neuen Parameter zu speichern (spezifisch für diesen Rahmen). Die Schritte sind für alle Frames zu wiederholen, die durch den blauen Bereich angezeigt werden.
- Wenn Sie fertig sind, initialisieren Sie den Tracking-Vorgang mit <7> neu.
HINWEIS: Diese Korrektur kann auch verwendet werden, wenn das Programm plötzlich von einem Filopodial zum anderen innerhalb eines Films wechselt. Als Alternative, betrachten Sie Kuchen Filme, die mehrere Filopodien enthalten.
- Besuchen Sie den jeweiligen Rahmen mit dem Schieberegler in Fenster # 2 und wählen Sie die Spitze des Filopodiums manuell aus.
6. Bildanalyse - Schritt 3: räumlich-zeitliche Proteinanalyse
- Zur räumlich-zeitlichen Analyse wählen Sie die durch <8b> dargestellte Box, gefolgt von dem interessierenden Proteinkanal (<8c> und / oder <8d> und / oder <8e>).
Hinweis: Es ist zwingend erforderlich, Protein A vor der ratiometrischen Analyse (<9a>, <9b> und / oder <9c>) zu wählen, da Protein A als Referenz verwendet wird. - Klicken Sie auf <8a>Um die Proteinverfolgung entlang der filopodialen Länge unter Verwendung der in Schritt 5.9 erzeugten Spur zu initiieren.
7. Bildanalyse - Schritt 4: Verhältnis-metrische Proteinanalyse
- Kontrollkästchen <9b> oder <9c> und klicke auf die Schaltfläche <9a>, um die räumlich-zeitliche ratiometrische Darstellung zu erhalten.
HINWEIS: Um eine falsche Darstellung der relativen Proteinkonzentration zu vermeiden, ist das ratiometrische Bild nicht als X / Y, sondern als log (X / Y) aufgetragen. Für die zukünftige Verwendung wird das ratiometrische Diagramm in den Dateien '.png' und '.fig' exportiert und rohe Plotdaten werden in der Datei 'dynamics.xlsx' gespeichert.
8. Bildanalyse - Schritt 5: Filopodiale Tipanalyse
- Kontrollkästchen <8f> und spezifizieren die Spitzenlänge und die Schwellenlänge von der Basis mit <8g> und <8h>. Klicken Sie auf , um Daten in die Datei 'dynamics.xlsx' zu speichern. Klicken Sie auf , um das zu erzeugenSpuren von Proteinintensitäten der filopodialen Spitze und zur rettiometrischen Analyse zu speichern.
HINWEIS: Die Tipplänge bestimmt die Anzahl der Pixel an der Spitze, die für die Analyse verwendet wird. Die Software gibt den durchschnittlichen Intensitätswert der Pixel an der Spitze über jeden Rahmen zurück. Die Schwellenlänge bestimmt den Mindestabstand von der Basis bis zur Spitze, über der das Filopodium (filopodiale Spitze) anfängt zu wachsen (und wenn das Programm beginnt, den Intensitätswert der Spitze zu verfolgen). So muss die Spitzenlänge kleiner sein als die Schwellenlänge. - Klicken Sie mit <9d> oder <9e> auf das gewünschte Verhältnis.
- Klicken Sie auf die Schaltfläche Vergleichen <9a>, um die ratiometrischen Daten zu erzeugen.
Ergebnisse
Unter Verwendung von COS-Zellen, die mit einem Marker für filamentösen Actin (f-Tractin 18 , rot) und einem cytosolischen Referenz (grün) transfiziert wurden, fanden wir aktinreiche filopodiale Vorsprünge ( Abbildung 3A , obere Platte). Zeitreihen zeigten, dass Filopodien sich schnell ausdehnen und zurückziehen ( Abbildung 3A , Mitteltafel). Mit der Bildanalyse-Software haben wir dann einzelne Filopo...
Diskussion
Hier stellen wir ein detailliertes Protokoll zur Verfolgung der filopodischen Wachstumsdynamik und der Analyse der relativen Proteinkonzentrationen in diesen dynamischen Strukturen über den konvexen Rumpfalgorithmus vor. Mit der Software können bis zu 3 Kanäle paarweise in einem einzigen Durchlauf verglichen werden, wobei die relativen Konzentrationen von zwei Kanälen ( dh Proteinen) während des gesamten Extensions- / Retraktionszyklus bestimmt und als Bild- und Datendateien in separaten Ordnern gespeicher...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Autoren bestätigen die Finanzierung von der DFG (EXC-1003 bis MG).
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referenzen
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten