Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Una interfaz gráfica de usuario para el seguimiento asistido por software de la concentración de proteínas en protones dinámicos celulares
En este artículo
Resumen
Presentamos una solución de software para el seguimiento semi-automatizado de la concentración relativa de proteínas a lo largo de la longitud de protrusiones celulares dinámicas.
Resumen
Los filopodios son protrusiones celulares dinámicas parecidas a los dedos asociadas con la migración y la comunicación célula-célula. Con el fin de comprender mejor los complejos mecanismos de señalización subyacentes a la iniciación filopodial, el alargamiento y posterior estabilización o retracción, es crucial determinar la actividad de la proteína espacio-temporal en estas estructuras dinámicas. Para analizar la función de la proteína en los filopodios, recientemente se desarrolló un algoritmo de seguimiento semi-automatizado que se adapta a los cambios de forma filopodial, lo que permite el análisis paralelo de la dinámica de protrusión y la concentración relativa de proteínas a lo largo de toda la longitud filopodial. Aquí presentamos un detallado protocolo paso a paso para el manejo optimizado de células, adquisición de imágenes y análisis de software. Además, proporcionamos instrucciones para el uso de funciones opcionales durante el análisis de imágenes y la representación de datos, así como pautas de solución de problemas para todos los pasos críticos en el camino. Por último, también se incluye una comparación de la dEscribed software de análisis de imágenes con otros programas disponibles para la cuantificación filopodia. En conjunto, el protocolo presentado proporciona un marco para el análisis preciso de la dinámica de proteínas en protrusiones filopodiales utilizando software de análisis de imágenes.
Introducción
El control espacio-temporal de las proteínas reguladoras de la actina se asocia con la dinámica del filopodio 1 , 2 . El seguimiento de la concentración de proteína resuelta espacialmente a lo largo de toda la longitud filopodial a través del tiempo es crucial para avanzar en nuestra comprensión de los mecanismos subyacentes de iniciación, elongación, estabilización o colapso de estas estructuras dinámicas 3 , 4 . A diferencia del análisis de proteínas en el citosol, donde muchos cambios en la forma celular ocurren a mayor escala, los filopodios son micro estructuras dinámicas que constantemente doblan 5 y se doblan, impidiendo así el análisis usando un enfoque simple como un barrido lineal.
Existen diferentes soluciones de software para el seguimiento de la forma filopodial disponibles 6 , 7 , 8 , 9 . ComoIse, se ha desarrollado un software para el seguimiento ratiométrico de la dinámica de proteínas dentro del cuerpo celular 10 , 11 . Para combinar el seguimiento automatizado de la forma filopodial y el análisis de proteínas espacio-temporales, recientemente hemos desarrollado un software de análisis de imágenes basado en el convexo casco algoritmo [ 12] . Este nuevo método de análisis, que funciona a través de una interfaz gráfica de usuario (GUI), combina por primera vez la concentración relativa de proteínas a lo largo de la longitud filopodial y la velocidad de crecimiento, permitiendo así la medición precisa de la distribución espacial-temporal de proteínas independiente del movimiento de estos Estructuras dinámicas 12 .
La idea detrás del software (el código fuente está disponible libremente, véase más abajo) es que uno de los vértices del casco convexo coincidirá con la punta del filopodio ( Figura 1A ). Al mirar en el siguiente marco foR el vértice más cercano del casco convexo, la punta en movimiento puede ser rastreada a lo largo de toda la película. Una vez que se detecta la punta en cada marco, su posición se utiliza para dibujar un eje uniendo la punta con un punto de referencia en la base del filopodio ( Figura 1B ). Finalmente, se utilizan puntos nodales equidistantes, cuyas posiciones son determinadas por el píxel mediano con máxima intensidad a lo largo de la línea ortogonal al eje, para determinar una columna vertebral que sigue a la forma filopodial. Aprovechando esta estructura adaptativa, se genera un quimógrafo para rastrear el crecimiento filopodial y las concentraciones de proteínas de hasta tres canales a lo largo de la longitud filopodial ( Figura 1C ).
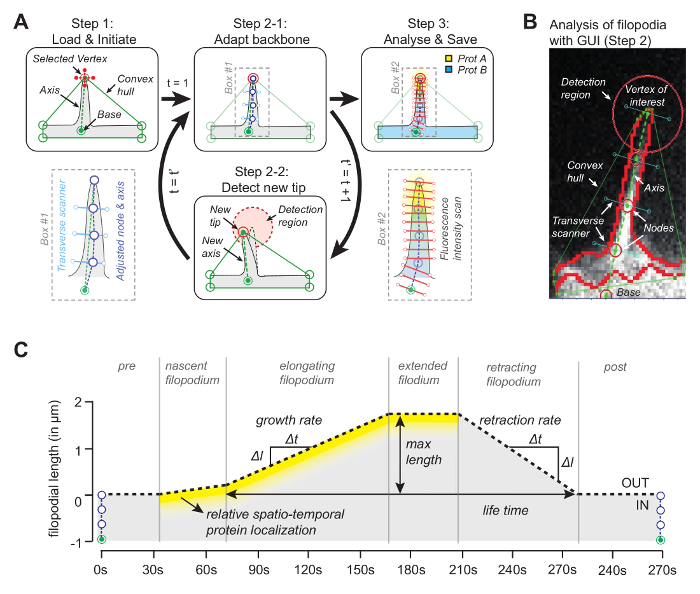
Figura 1: Principio de trabajo del software de análisis de imágenes. ( A ) El algoritmo detrásEl software. En Step1 el usuario especifica la referencia (base) y el vértice (la punta) del filopodio. En el Paso 2-1, la columna vertebral del filopodio se obtiene usando el píxel medio con el valor de intensidad máxima. En el Paso 3 se utiliza la columna vertebral para el perfil de intensidad de proteína espacial. En el paso 2-2, el software rastrea automáticamente la punta en el siguiente cuadro. Todo el procedimiento itera. ( B ) Instantánea del algoritmo con filopodio real introduciendo elementos importantes como el casco convexo que se está utilizando para el seguimiento. ( C ) Visión general de los parámetros que se pueden medir con el algoritmo. Esta cifra ha sido modificada a partir de la referencia 12 . Haga clic aquí para ver una versión más grande de esta figura.
El software de análisis de imágenes se opera en Matlab (denominado software de programación) mediante un uso gráficoR. Para maximizar la flexibilidad y la robustez del entorno experimental particular, el usuario puede ajustar una serie de parámetros de seguimiento ( por ejemplo , ángulo de flexión permitido y movimiento entre fotogramas) y también realizar algunas correcciones en las películas ( por ejemplo , recorte, rotación, eliminación de objetos no deseados) ( Figura 2A y Tabla 1) .
| GUI | No. | Obligatorio | Descripción | Nombre (en GUI) | ||||
| # 1 | 1a | Y | Cargar archivo .tiff apilado que representa el cuerpo de la celda (con la casilla marcada) o crear el cuerpo de celda superpuesto desde los canales | Cuerpo de la célula | ||||
| # 1 | 1b | Y | Cargando el archivo de imágenes apiladas correspondiente a la proteína 1 | Proteína 1 | ||||
| # 1 | 1c | Y | Carga del archivo de imágenes apiladas correspondiente a la proteína 2 | Proteína 2 | ||||
| # 1 | 1d | Y | Cargar archivo de imágenes apiladas correspondiente a la proteína 3 | Proteína 3 | ||||
| # 1 | 1e | norte | Restablece todo a archivos de imagen apilados precargados | Reiniciar | ||||
| # 1 | 2a | Y | Barra de desplazamiento para determinar el marco inicial para el análisis en la ventana GUI # 2 | N / A | ||||
| # 1 | 2b | Y | Barra de desplazamiento para determinar el marco final para el análisis en la ventana GUI # 2 | N / A | ||||
| # 1 | 2c | Y | Barra de desplazamiento que representa curreNt marco | N / A | ||||
| # 1 | 2d | norte | El valor de gris de los píxeles por debajo del cual todos los píxeles se pondrán a cero | N / A | ||||
| # 1 | 2e | norte | El valor de gris de los píxeles por encima del cual todos los píxeles se establecerán en valores máximos | N / A | ||||
| # 1 | 2f | norte | Establezca los valores de intensidad de los píxeles especificados por <2e> y <2f> | Conjunto | ||||
| # 1 | 2g | norte | Reproducir la película de intensidad ajustada | Jugar | ||||
| # 1 | 2h | norte | Delimitar imagen | Cultivo | ||||
| # 1 | 2i | norte | Rotar imagen | Girar | ||||
| # 1 | 2j | norte | Eliminar regiones de toda la pila | Eliminar regiones | ||||
| # 1 | 3a | Y | Haga clic para abrir la "ventana de análisis" (ventana GUI # 2) | Ventana de seguimiento | ||||
| # 1 | 3b | Y | Introduzca el tamaño de un píxel en micras | Introducir tamaño de píxel | ||||
| # 2 | 4 | Y | Haga clic para generar la imagen de límite / borde del cuerpo celular superpuesto | Límite | ||||
| # 2 | 5 | Y | Haga clic para seleccionar la base y la punta de los filopodios | Referenciar | ||||
| # 2 | 6a | Y | Introduzca el número de segmentos o nodos | Número de segmentos | ||||
| # 2 | 6b | Y | Introduzca la longitud de la exploración (perpendicular al eje) | Ancho de la exploración | ||||
| # 2 | 6c | Y | Introduzca la longitud por encima de la cual filopodia comienza a doblarse | Accurate Meas after | ||||
| # 2 | 6d | Y | Introduzca el radio del círculo de detección de la punta (es decir, el área donde el vértice se puede localizar en el siguiente fotograma) | Detección de radio de punta | ||||
| # 2 | 6e | Y | Introduzca el ángulo máximo que el filopodio puede doblar desde el eje vertical | Umbral de ángulo | ||||
| # 2 | 6f | norte | Agregar puntos de referencia para la base y la punta para ese marco específico | Seleccionar referencia | ||||
| # 2 | 6g | norte | Introduzca la longitud por encima de la cual filopodia comienza a doblarse para ese marco específico | Accurate Meas after | ||||
| # 2 | 6h | norte | Introduzca el radio del círculo de detección para ese marco específico | Detección de radio de punta | ||||
| # 2 | 6i | norte | Después de ingresar todos los parámetros para el marco específico, haga clic para almacenar los valores en la memoria y el archivo para referencia adicional | Añadir | ||||
| # 2 | 6j | norte | Haga clic para eliminar el conjunto de parámetros manuales para ese marco | Borrar | ||||
| # 2 | 6k | norte | Haga clic para borrar todos los parámetros almacenados manualmente usando el "panel de características opcionales" para todos los marcos | Reiniciar | ||||
| # 2 | 6l | norte | Registrarse antes de realizar un seguimiento para almacenar todos los resultados de seguimiento en la memoria para referencia futura | Rastro de la historia | ||||
| # 2 | 7 | Y | Haga clic para iniciar el seguimiento | Seguimiento y análisis | ||||
| # 2 | 8a | norte | Haga clic para iniciar el seguimiento de la intensidad del canal de proteínas | Analizar las Intensidades de las Proteínas | ||||
| # 2 | 8b | norte | Registro para rastrear la intensidad del canal proteico a lo largo de la longitud filopodial | Filopodias enteras | ||||
| # 2 | 8c | norte | Registro para el seguimiento de la proteína de referencia o proteína A | Proteina | ||||
| # 2 | 8d | norte | Registro para el seguimiento de la proteína B | Proteína b | ||||
| # 2 | 8e | norte | Check in para el seguimiento de la proteína C | Proteína | ||||
| # 2 | 8f | norte | Compruebe la intensidad media de proteínasSugerencia | Punta líder | ||||
| # 2 | 8g | norte | Introduzca la longitud de la punta | Longitud de la punta | ||||
| # 2 | 8h | norte | Introduzca la distancia mínima desde la base por encima de la cual comienza a formarse la punta | límite | ||||
| # 2 | 8i | norte | Haga clic para guardar los resultados del análisis de puntaje principal en el archivo | Presionar el botón | ||||
| # 2 | 9a | norte | Haga clic para iniciar el análisis de proteínas ratiométricas | Comparar | ||||
| # 2 | 9b | norte | Verifique para comparar la proteína B con respecto a A | Log10 (B / A) | ||||
| # 2 | 9c | norte | Comprobar para comparar la proteína C con respecto a A | Log10 (C / A) | ||||
| # 2 | 9d | norte | Verifique para comparar la proteína B con respecto a A en la punta | Log10 (B / A) | ||||
| # 2 | 9e | norte | Verifique para comparar la proteína C con respecto a A en la punta | Log10 (C / A) | ||||
| # 2 | 10 a | norte | Elija otro mapa de color (predeterminado: Jetplot) | Mapa del color | ||||
| # 2 | 10b | norte | Editar el mapa de colores | Editar mapa de color | ||||
Tabla 1: Resumen de todas las funciones presentes en la GUI Windows # 1 y # 2.
Una vez realizado esto, el programa crea un casco convexo y rastrea automáticamente la punta a lo largo de la película. Parámetros extraídos de la película, como un quimógrafo ratiométrico, velocidad de crecimiento y longitud filopodial aSe visualizan y también se almacenan en la carpeta de trabajo como imágenes y como archivos de datos. Otros parámetros tales como la vida filopodial, la velocidad de crecimiento y la tasa de retracción pueden extraerse y analizarse posteriormente a partir de los archivos de datos almacenados ( Figura 2B ).
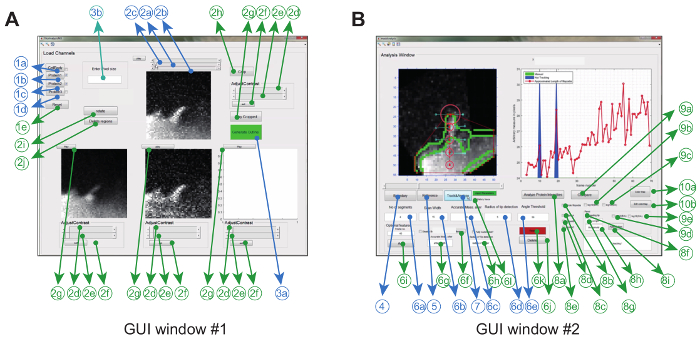
Figura 2: Interfaz gráfica de usuario para el uso del software de análisis de imágenes. ( A ) La Ventana GUI # 1 se utiliza para cargar y procesar imágenes. El programa puede cargar hasta 3 canales de proteína, por lo que 2 canales se comparan en pares. La ventanilla viene con las características obligatorias (azules) y opcionales (verde) para el pre-procesamiento de las imágenes antes del seguimiento ( B ) La ventana GUI # 2 se utiliza para el seguimiento del filopodio así como el análisis espacio-temporal y la relación-métrica de proteínas. Nuevamente, las funciones opcionales están marcadas en verde. Esta cifra ha sido modificadaEd de la referencia 12 . Haga clic aquí para ver una versión más grande de esta figura.
Aquí presentamos un protocolo detallado para la preparación de muestras y manejo de software. Comenzamos con instrucciones detalladas sobre el cultivo de células y la adquisición de películas optimizadas para el análisis de imágenes. Esta sección sobre la adquisición de datos va seguida de una descripción detallada para el funcionamiento del software de análisis de imágenes. A lo largo del protocolo, introducimos pasos críticos y características opcionales que deben tenerse en cuenta al recopilar y procesar datos. Finalmente, analizamos filopodios de diferentes sistemas de modelos con el software de análisis de imágenes, antes de cerrar con una comparación del software de análisis de imágenes descrito con otros programas disponibles para la cuantificación filopodial y una discusión sobre limitaciones y dirección futura.
Protocolo
1. Cultivo celular
- Cultivo de células HeLa o COS en medio de Eagle modificado por Dulbecco (DMEM) que contiene 4,5 g / l de D-glucosa, dipéptido L-alanina-L-glutamina, piruvato, suero fetal bovino al 10% y 10 unidades / ml de penicilina / estreptomicina. Cultivo de neuronas en medio de cultivo sin L-glutamina, ácido glutámico o ácido aspártico, suplementado con 0,5 mM de L-alanina-L-glutamina dipéptido, suero libre de suplementos neuronales y 10 unidades / ml de penicilina / estreptomicina.
- Una vez que se alcanza el 40% de confluencia, transfectar las células con construcciones de elección usando un reactivo de transfección según las instrucciones del fabricante. Mantener las células transfectadas en incubadora a 37 ° C y 5% de CO 2 durante 15-18 h.
- Para reducir los cambios en el pH y la osmolaridad (debido a la evaporación) durante la adquisición de la imagen, llenar las cámaras de cultivo celular (antes de la formación de imágenes) hasta 90% con medio pre-calentado a 37ºC que contiene 20 mM 4- (2-hidroxietil) -1 -piperazinoetanosulfónico (HEPES). Para sellarLa tapa, aplicar una fina capa de grasa al vacío en el interior de la tapa y presionar suavemente sobre la cámara de cultivo que contiene las células transfectadas.
2. Adquisición de imágenes
NOTA: La longitud de los filopodios varía de 2-10 μm 13 . Los filopodios crecen a una velocidad media de 0,05-0,1 μm / s 13 , 14 .
- Adquirir imágenes utilizando un microscopio con objetivo de 60X o 100X y sin binario de píxeles (aquí, un microscopio confocal de disco giratorio). Utilice velocidades de adquisición superiores a 1 Hz (Hz) para realizar un seguimiento de la dinámica filopodial. Para minimizar los artefactos fuera de foco, imagen filopodia cerca de la membrana basal ( es decir , la superficie del sustrato).
- Para asegurar un seguimiento suave, ajuste el tiempo de exposición de la cámara y la intensidad del láser de modo que la relación señal a ruido (SNR) sea mayor que 4. Evite la saturación de canales individuales ( es decir, valores de píxeles de255 para imágenes de 8 bits y 65.535 para imágenes de 16 bits), ya que esto impedirá el análisis posterior de imágenes.
- Para evitar el sangrado, utilice únicamente etiquetas de fluorescencia compatibles con líneas de láser y filtros del microscopio (para detalles, consulte la referencia 15 ).
3. Pre-procesamiento de imágenes
NOTA: Utilice ImageJ u otro software disponible para procesar previamente las imágenes 16 , 17 .
- Si la muestra se está moviendo, corrija la deriva lateral utilizando el software disponible ( por ejemplo, https://github.com/NMSchneider/fixTranslation-Macro_for_ImageJ) antes del análisis. Excluir películas con desplazamiento axial (es decir, movimiento en la dirección z).
- Corregir el fondo ( es decir, los valores de grises de las áreas fuera de la célula), blanqueamiento ( es decir, pérdida continua si la intensidad de la fluorescencia debida a daño a las proteínas de fluorescencia) y posible sangradoSonda de orestesia en los dos canales) utilizando el software disponible ( por ejemplo, http://imagej.net/Category:Plugins y 16 , 17 ).
Nota: Las intensidades de fluorescencia de los canales individuales no son alteradas por el software. - Para asegurar el posterior análisis de imagen, guarde las películas correspondientes a canales de proteínas particulares en escala de grises como formato de archivo apilado ".tiff" en la carpeta de trabajo.
NOTA: Las dimensiones ( es decir , tamaño, longitud) de las pilas deben ser las mismas para todos los canales.
4. Análisis de imagen - Paso 1: cargar imágenes
NOTA: El software descrito aquí se escribió en Matlab (denominado software de programación) y se ejecutará sólo con este programa.
- Descargue la carpeta comprimida que contiene todos los archivos necesarios para el análisis de imágenes desde el siguiente sitio: https://campus.uni-muenster.de/en/einrichtungen/impb0/nanoscale-forces-in-cells/softwson/. Descomprimir y copiar archivos en la carpeta de trabajo.
- Una vez instalado, abra el software de programación y ejecute 'filopodiaAnalysisM3.fig'. GUI Ventana # 1 se abrirá como se muestra en la Figura 2A .
- Cargue los archivos '.tiff' guardados guardados correspondientes a una proteína particular de interés en la ventana GUI # 1 usando los botones <1b> para la proteína A, <1c> para la proteína B, <1d> para la proteína C. Vea la Figura 2 y la Tabla 1 para más detalles.
NOTA: La proteína A mostrada en la ventana GUI # 1 actúa como el canal de referencia para el análisis ratiométrico final. - Crear una imagen superpuesta de la célula de los canales de proteína haciendo clic en <1a>.
- Haga clic en <2a> para asignar el primer fotograma y <2b> para el último fotograma utilizado para el análisis.
- Opcionalmente, recorte la región de interés (ROI) que contiene el filopodio de interés mediante el botón <2h>, gire la imagen uCantar el botón <2i>, o eliminar regiones no deseadas con la herramienta de dibujo a mano <2j>.
NOTA: Para optimizar el análisis de imagen, se recomienda aislar el ROI ( es decir , recortar, rotar, eliminar) junto con los otros pasos de preprocesamiento (substracción de fondo, correcciones de blanqueo, etc.) usando ImageJ. - Mueva el control deslizante <2c> para cada cuadro para el control de calidad y compruebe si el filopodio permanece claramente visible a lo largo de toda la película.
5. Análisis de la imagen - Paso 2: Generar Trace
- Haga clic en el botón <3a> en la ventana GUI # 1 para abrir la ventana GUI # 2 (vea la Figura 2B ).
- Haga clic en el botón <4> en la ventana GUI # 2 para generar la máscara del cuerpo celular superpuesto (generado en la ventana GUI # 1 después de hacer clic en <1a>). El programa también genera el límite de la máscara, donde el casco convexo se implementa para obtener los puntos de vértice.
- CliBotón ck <5>; Aparecerá un cursor. Utilice el cursor para seleccionar la base (desde donde se mide la distancia de la punta filopodial) seguida por la punta de los filopodios en el marco donde aparece por primera vez. Para ello, mueva el control deslizante en la Ventana # 2).
NOTA: Para minimizar los errores en la salida de datos, coloque el punto base verticalmente por debajo de la punta filopodial a lo largo del eje. Posicionar el punto base en otros lugares ( p . Ej. Desplazados lateralmente) puede introducir un sesgo direccional en los valores de fluorescencia dentro del cuerpo celular. - Seleccione la longitud umbral (por encima de la cual se doblará el filopodio) usando <6c>.
Nota: Esta longitud se define como la distancia entre el punto base (seleccionado con <5>) y el límite del cuerpo celular ( es decir, la región desde la cual comienza a crecer el filopodio). La distancia se mide en píxeles. - Especifique el número de segmentos utilizados para aproximar la forma de los filopodios en la casilla <6a>.
NOTA: El mEl número mínimo de segmentos depende de la longitud máxima alcanzada por el filopodio, pero también de cómo se dobla el filopodio. No seleccione un número de segmentos mayor que el número de píxeles entre la base y la punta ( es decir, la longitud umbral). Selección de más segmentos que la longitud umbral definida en el paso 5.4. (Es decir, número de píxeles entre la base y la punta) resultará en una sobreestimación de la longitud del filopodio. - Especifique el ancho de exploración, que actúa como escáner horizontal para colocar los nodos, en <6b>.
NOTA: Estos nodos son utilizados por el programa para ajustar la línea que une la base y la punta con el cuerpo del filopodio ( es decir, para crear la columna vertebral). Como punto de partida, ponga un valor en píxeles igual a la longitud máxima multiplicada por un factor de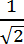 .
. - Especifique el radio de exploración (en píxeles) en el cuadro <6d>. Utilice un valor que sea aproximadamente 50% mayor que el obServido inter-marco desplazamiento de la punta.
NOTA: El uso de un radio de barrido muy grande podría atrapar puntos no deseados del casco convexo en los marcos donde no hay punta filopodial real presente ( por ejemplo, fuera del movimiento del plano o bajo SNR). - Especifique el ángulo de flexión en la casilla <6e>.
NOTA: El umbral angular se determina por el ángulo máximo que el filopodio está doblando durante todo el análisis. Especificar el umbral de ángulo ayuda al software a excluir el seguimiento de las estructuras no deseadas que crecen desde el lado de los filopodios cuando los filopodios se dobla hacia el cuerpo celular. El programa funciona de forma fiable para filopodia con ángulos de inclinación de menos de 45 grados desde el eje vertical. - Para iniciar el seguimiento, haga clic en el botón en la ventana GUI # 2. Haga clic en el cuadro "Historial de seguimiento" en la ventana GUI # 2 para guardar el protocolo de seguimiento completo para referencia futura.
NOTA: Una vez finalizado el procedimiento de seguimiento, la longitud del filopodio en cada fotograma se almacena enLa hoja denominada 'length_vel' de 'dynamics.xlsx' para futuras referencias. Del mismo modo, todos los demás parámetros de seguimiento se almacenan en la hoja llamada 'parameters' de 'dynamics.xlsx'. - Opcionalmente, si los filopodios no se detectan automáticamente en todos los marcos, utilice los siguientes pasos para corregirlos manualmente.
- Visite el marco respectivo utilizando el deslizador de la Ventana # 2 y seleccione manualmente la punta del filopodio.
Nota: Los marcos, donde no se detectan puntos convexos del casco, serán representados por una región azul en la ventana de seguimiento de la Ventana GUI # 2. Es obligatorio comprobar el botón "Historial rastreo" antes de iniciar el programa de seguimiento para acceder a las coordenadas nodales en ese marco. - Seleccione los puntos de referencia (base seguida de punta) usando <6f> en la Ventana GUI # 2. Especifique los otros parámetros como 'longitud de barrido', 'Medición exacta después' y 'ángulo de plegado máximo'; Para ese cuadro como se describe en los pasos 5.4-5.8.
- Haga clic en el botón <6i> para guardar los nuevos parámetros (específicos de ese cuadro). Los pasos se deben repetir para todos los marcos indicados por la región azul.
- Cuando termine, vuelva a inicializar el procedimiento de seguimiento con <7>.
NOTA: Esta corrección también se puede utilizar si el programa cambia de repente de un filopodial a otro dentro de una película. Como alternativa, considere recortar películas que contienen varios filopodios.
- Visite el marco respectivo utilizando el deslizador de la Ventana # 2 y seleccione manualmente la punta del filopodio.
6. Análisis de Imagen - Paso 3: Análisis de Proteínas Espaciales-Temporales
- Para el análisis espacio-temporal, seleccione el cuadro representado por <8b> seguido por el canal de proteína de interés (<8c> y / o <8d> y / o <8e>).
Nota: Es obligatorio seleccionar la Proteína A antes del análisis ratiométrico (<9a>, <9b> y / o <9c>) ya que la Proteína A se utiliza como referencia. - Haga clic en <8a>Para iniciar el seguimiento de proteínas a lo largo de la longitud filopodial utilizando la traza generada en el paso 5.9.
7. Análisis de imagen - Paso 4: Análisis de proteína de relación-métrica
- Marque la casilla <9b> o <9c> y haga clic en el botón <9a> para obtener el diagrama ratiométrico espacio-temporal.
NOTA: Para evitar la representación errónea de la concentración relativa de proteína, la imagen ratiométrica no se representa como X / Y sino como log (X / Y). Para uso futuro, la trama ratiométrica se exporta en archivos de formato '.png' y '.fig', y los datos de trama sin procesar se almacenan dentro del archivo 'dynamics.xlsx'.
8. Análisis de imagen - Paso 5: Análisis de punta filopodial
- Marque la casilla <8f> y especifique la longitud de la punta y la longitud del umbral desde la base usando <8g> y <8h>. Haga clic en para guardar los datos en el archivo 'dynamics.xlsx'. Haga clic en para generar elTraza de intensidad de proteínas de la punta filopodial y guardar para el análisis ratiométrico.
NOTA: La longitud de la punta determina el número de píxeles en la punta utilizada para el análisis. El software devolverá el valor de intensidad promedio de los píxeles en la punta sobre cada trama. La longitud del umbral determina la distancia mínima entre la base y la punta por encima de la cual comienza a crecer el filopodio (punta filopodial) (y cuando el programa comienza a rastrear el valor de intensidad de la punta). Así que la longitud de la punta debe ser menor que la longitud del umbral. - Haga clic en la proporción deseada para analizar con <9d> o <9e>.
- Haga clic en el botón de comparación <9a> para generar los datos ratiométricos.
Resultados
Utilizando células COS transfectadas con un marcador de actina filamentosa (f-tractina 18 , rojo) y una referencia citosólica (verde), encontramos protrusiones filopodiales ricas en actina ( Figura 3A , panel superior). Las series de tiempo mostraron que los filopodios se extienden y retraen rápidamente ( Figura 3A , panel central). Utilizando el software de análisis de imágenes, seguimos los filopod...
Discusión
Aquí presentamos un protocolo detallado para el seguimiento de la dinámica de crecimiento filopodial y el análisis de las concentraciones relativas de proteínas en estas estructuras dinámicas a través del algoritmo de casco convexo. Usando el software, se pueden comparar hasta 3 canales en pares en una sola ejecución, por lo que las concentraciones relativas de dos canales ( es decir, proteínas) se determinan a lo largo del ciclo de extensión / retracción y se almacenan como archivos de imagen y datos...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores reconocen el financiamiento del DFG (EXC-1003 a MG).
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referencias
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados