Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une interface utilisateur graphique pour le suivi assisté par logiciel de la concentration de protéines dans les protrusions cellulaires dynamiques
Dans cet article
Résumé
Nous présentons une solution logicielle pour le suivi semi automatisé de la concentration relative de protéines sur toute la longueur des protrusions cellulaires dynamiques.
Résumé
Filopodia sont des protrusions cellulaires dynamiques, semblables à des doigts associées à la migration et à la communication cellule-cellule. Afin de mieux comprendre les mécanismes de signalisation complexes sous-jacents à l'initiation filopodiale, à l'allongement et à la stabilisation ou à la rétraction ultérieure, il est crucial de déterminer l'activité spatio-temporelle de la protéine dans ces structures dynamiques. Pour analyser la fonction des protéines dans filopodia, nous avons récemment développé un algorithme de suivi semi-automatisé qui s'adapte aux changements de forme filopodique, permettant ainsi une analyse parallèle de la dynamique de protrusion et de la concentration relative de protéines sur toute la longueur de la filopodie. Nous présentons ici un protocole détaillé étape par étape pour la gestion optimisée des cellules, l'acquisition d'images et l'analyse de logiciels. Nous fournissons également des instructions pour l'utilisation de fonctionnalités facultatives lors de l'analyse d'image et de la représentation des données, ainsi que des directives de dépannage pour toutes les étapes critiques en cours de route. Enfin, nous incluons également une comparaison de la dLogiciel d'analyse d'image écrited avec d'autres programmes disponibles pour la quantification de filopodia. Ensemble, le protocole présenté fournit un cadre pour une analyse précise de la dynamique des protéines dans les protubérances filopodiales à l'aide d'un logiciel d'analyse d'image.
Introduction
Le contrôle spatio-temporel des protéines régulatrices d'actine est associé à la dynamique du filopodium 1 , 2 . Le suivi de la concentration de protéines spatialement résolue tout au long de la longueur filopodiale dans le temps est donc crucial pour faire progresser notre compréhension des mécanismes sous-jacents à l'initiation, l'allongement, la stabilisation ou l'effondrement de ces structures dynamiques 3 , 4 . Contrairement à l'analyse des protéines dans le cytosol, où de nombreux changements de forme cellulaire se produisent à plus grande échelle, les filopodes sont des micro structures dynamiques qui bouclent constamment 5 et se courbent, ce qui exclut l'analyse en utilisant une approche simple telle qu'une analyse de ligne.
Différentes solutions logicielles pour le suivi de la forme filopodique sont disponibles 6 , 7 , 8 , 9 . LikewIse, un logiciel pour le suivi ratitimétrique de la dynamique des protéines dans le corps cellulaire a été développé 10 , 11 . Pour combiner le suivi automatique de la forme filopodique et l'analyse spatio-temporelle des protéines, nous avons récemment développé un logiciel d'analyse d'image basé sur l'algorithme de la coque convexe 12 . Cette nouvelle méthode d'analyse, qui fonctionne via une interface utilisateur graphique (GUI), combine pour la première fois, la concentration relative de protéines le long de la longueur et de la vitesse de croissance filopodiales, permettant ainsi une mesure précise de la répartition spatio-temporelle des protéines indépendamment du mouvement de ces Structures dynamiques 12 .
L'idée derrière le logiciel (le code source est disponible gratuitement, voir ci-dessous) est que l'un des sommets de la coque convexe coïncide avec la pointe du filopodium ( Figure 1A ). En regardant dans le cadre ultérieur pourLe sommet le plus proche de la coque convexe, la pointe mobile peut être suivie tout au long du film. Une fois que la pointe est détectée dans chaque cadre, sa position est utilisée pour dessiner un axe en joignant la pointe avec un point de référence à la base du filopodium ( figure 1B ). Enfin, en utilisant des points nodaux équidistants, dont les positions sont déterminées par le pixel médian avec une intensité maximale le long de la ligne orthogonale à l'axe, sont utilisés pour déterminer un squelette qui suit la forme filopodique. Profitant de ce squelette adaptatif, un kymographe est généré pour tracer la croissance filopodiale et les concentrations de protéines pour jusqu'à trois canaux le long de la longueur filopodique ( figure 1C ).
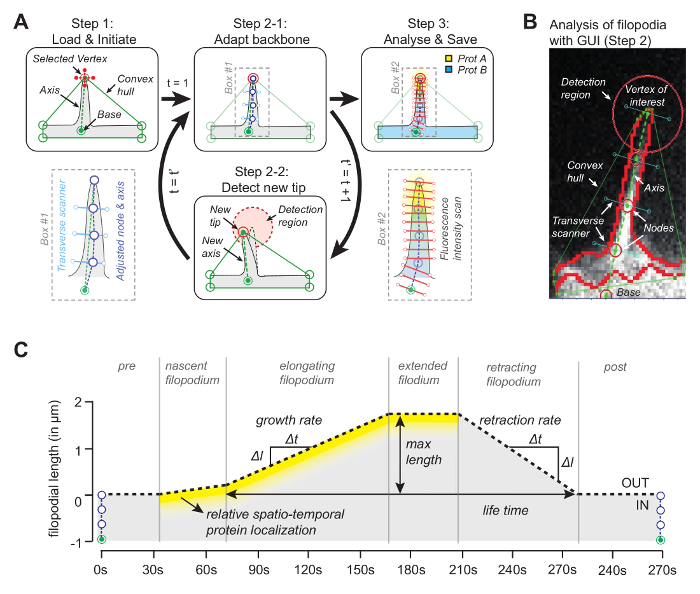
Figure 1: Principe de fonctionnement du logiciel d'analyse d'image. ( A ) L'algorithme derrièreles logiciels. À l'étape 1, l'utilisateur spécifie la référence (base) et le sommet (la pointe) du filopodium. À l'étape 2-1, l'ossature du filopodium est obtenue en utilisant le pixel médian avec une valeur d'intensité maximale. À l'étape 3, l'ossature est utilisée pour le profil d'intensité de protéine spatiale. À l'étape 2-2, le logiciel suit automatiquement la pointe dans la trame suivante. La procédure entière itère. ( B ) Instantané de l'algorithme avec du filopodium réel présentant des éléments importants tels que la coque convexe qui est utilisée pour le suivi. ( C ) Vue d'ensemble des paramètres qui peuvent être mesurés avec l'algorithme. Ce chiffre a été modifié à partir de la référence 12 . Cliquez ici pour voir une version plus grande de ce chiffre.
Le logiciel d'analyse d'image est utilisé dans Matlab (appelé logiciel de programmation) par une utilisation graphiqueR interface. Pour maximiser la flexibilité et la robustesse pour le réglage expérimental particulier, l'utilisateur peut ajuster une série de paramètres de suivi ( p. Ex. Angle de flexion autorisé et mouvement entre images) et effectuer également des corrections aux films ( p. Ex., Recadrage, rotation, élimination des objets indésirables) ( Figure 2A et tableau 1) .
| GUI | Non. | Obligatoire | La description | Nom (en GUI) | ||||
| #1 | 1a | Y | Chargement du fichier .tiff empilé représentant le corps de la cellule (avec la case cochée) ou créez le corps cellulaire superposé à partir des canaux | Corps cellulaire | ||||
| #1 | 1b | Y | Chargement d'un fichier d'image empilé correspondant à la protéine 1 | Protéines 1 | ||||
| #1 | 1c | Y | Chargement d'un fichier d'image empilé correspondant à la protéine 2 | Protéines 2 | ||||
| #1 | 1d | Y | Chargement du fichier d'image empilé correspondant à la protéine 3 | Protéines 3 | ||||
| #1 | 1e | N | Réinitialise tout à précharger les fichiers d'images empilés | Réinitialiser | ||||
| #1 | 2a | Y | Barre de défilement pour déterminer la trame initiale pour l'analyse dans la fenêtre GUI n ° 2 | N / A | ||||
| #1 | 2b | Y | Barre de défilement pour déterminer le cadre final pour l'analyse dans la fenêtre GUI n ° 2 | N / A | ||||
| #1 | 2c | Y | Barre de défilement représentant le curreCadre nt | N / A | ||||
| #1 | 2d | N | La valeur grise des pixels au-dessous de laquelle tous les pixels seront mis à zéro | N / A | ||||
| #1 | 2e | N | La valeur grise des pixels au-dessus de laquelle tous les pixels seront définis sur les valeurs maximales | N / A | ||||
| #1 | 2f | N | Définissez les valeurs d'intensité des pixels spécifiées par <2e> & <2f> | Ensemble | ||||
| #1 | 2 g | N | Jouez le film ajusté en intensité | Jouer | ||||
| #1 | 2h | N | Image de culture | Surgir | ||||
| #1 | 2i | N | Faire pivoter l'image | Tourner | ||||
| #1 | 2j | N | Supprimer les régions dans l'ensemble de la pile | Supprimer les régions | ||||
| #1 | 3a | Y | Cliquez pour ouvrir la «fenêtre d'analyse» (fenêtre GUI n ° 2) | Fenêtre de suivi | ||||
| #1 | 3b | Y | Entrez la taille d'un pixel en microns | Entrer la taille du pixel | ||||
| # 2 | 4 | Y | Cliquez pour générer l'image limite / bord du corps cellulaire superposé | Frontière | ||||
| # 2 | 5 | Y | Cliquez pour sélectionner la base et la pointe du filopodia | Referernce | ||||
| # 2 | 6a | Y | Entrez le nombre de segments ou de nœuds | Nombre de segments | ||||
| # 2 | 6b | Y | Entrez la longueur de balayage (perpendiculaire à l'axe) | Largeur de numérisation | ||||
| # 2 | 6c | Y | Entrez la longueur au-dessus de laquelle filopodia commence à se pencher | Mes exacte après | ||||
| # 2 | 6d | Y | Entrez le rayon du cercle de détection de la pointe (c'est-à-dire la zone où le sommet peut être localisé dans la prochaine image) | Radius de détection de pointe | ||||
| # 2 | 6e | Y | Entrez l'angle maximum que le filopodium peut se pencher de l'axe vertical | Seuil d'angle | ||||
| # 2 | 6f | N | Ajouter des points de référence pour la base et la pointe pour ce cadre spécifique | Sélectionnez la référence | ||||
| # 2 | 6 g | N | Entrez la longueur au-dessus de laquelle filopodia commence à se plier pour ce cadre spécifique | Mes exacte après | ||||
| # 2 | 6h | N | Entrez le rayon du cercle de détection pour ce cadre spécifique | Radius de détection de pointe | ||||
| # 2 | 6i | N | Après avoir entré tous les paramètres pour le cadre spécifique, cliquez pour enregistrer les valeurs dans la mémoire et le fichier pour plus d'informations. | Ajouter | ||||
| # 2 | 6j | N | Cliquez pour supprimer les paramètres manuellement définis pour cette image | Effacer | ||||
| # 2 | 6k | N | Cliquez pour supprimer tous les paramètres stockés manuellement à l'aide du «panneau des fonctions optionnelles» pour tous les cadres | Réinitialiser | ||||
| # 2 | 6l | N | Check in avant le suivi pour stocker tous les résultats de suivi en mémoire pour référence future | Histoire trace | ||||
| # 2 | 7 | Y | Cliquez pour commencer le suivi | Suivre et analyser | ||||
| # 2 | 8a | N | Cliquez pour commencer le suivi de l'intensité du canal protéique | Analyser les intensités de protéines | ||||
| # 2 | 8b | N | Check in pour suivre l'intensité du canal protéique le long de la longueur filopodiale | Filopodes entiers | ||||
| # 2 | 8c | N | Vérifier le suivi de la protéine de référence ou de la protéine A | ProteinA | ||||
| # 2 | 8d | N | Check in pour le suivi de la protéine B | ProteinB | ||||
| # 2 | 8e | N | Check in pour le suivi de la protéine C | Protéines | ||||
| # 2 | 8f | N | Check in pour suivre l'intensité moyenne de la protéine dansE tip | Conseil principal | ||||
| # 2 | 8 g | N | Entrez la longueur de la pointe | Longueur des embouts | ||||
| # 2 | 8h | N | Entrez la distance minimale à partir de la base au-dessus de laquelle la pointe commence à former | seuil | ||||
| # 2 | 8i | N | Cliquez pour enregistrer les résultats de l'analyse de pointe avant de déposer | Bouton poussoir | ||||
| # 2 | 9a | N | Cliquez pour lancer l'analyse de la protéine ratiométrique | Comparer | ||||
| # 2 | 9b | N | Check in pour comparer la protéine B par rapport à A | Log10 (B / A) | ||||
| # 2 | 9c | N | Check in pour comparer la protéine C par rapport à A | Log10 (C / A) | ||||
| # 2 | 9d | N | Check in pour comparer la protéine B par rapport à A à la pointe | Log10 (B / A) | ||||
| # 2 | 9e | N | Check in pour comparer la protéine C par rapport à A à la pointe | Log10 (C / A) | ||||
| # 2 | 10a | N | Choisissez d'autres cartes de couleurs (par défaut: Jetplot) | Carte de couleur | ||||
| # 2 | 10b | N | Modifier la couleur | Modifier la couleur | ||||
Tableau 1: Vue d'ensemble de toutes les fonctions Présentes dans la GUI Windows n ° 1 et n ° 2.
Une fois que cela est accompli, le programme crée une coque convexe et suit automatiquement la pointe tout au long du film. Paramètres extraits du film, tels qu'un kymographe ratiométrique, une vitesse de croissance et une longueur filopodique aSont affichés et enregistrés dans le dossier de travail sous forme d'images et de fichiers de données. D'autres paramètres tels que la durée de vie filopodiale, le taux de croissance et le taux de retrait peuvent ensuite être extraits et analysés plus avant à partir des fichiers de données stockés ( Figure 2B ).
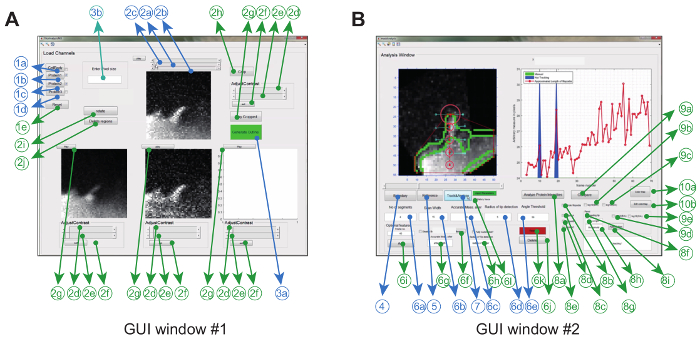
Figure 2: Interface utilisateur graphique pour l'utilisation du logiciel d'analyse d'image. ( A ) La fenêtre GUI # 1 est utilisée pour charger et traiter des images. Le programme peut charger jusqu'à 3 canaux de protéines, par lequel 2 canaux sont comparés par paire. La fenêtre est livrée avec des fonctions obligatoires (bleu) et optionnelles (vert) pour prétraiter les images avant le suivi ( B ). La fenêtre GUI # 2 est utilisée pour suivre l'analyse des protéines filopodium ainsi que spatio-temporelles et proportionnelles. Encore une fois, les fonctionnalités facultatives sont marquées en vert. Ce chiffre a été modifiDe la référence 12 . Cliquez ici pour voir une version plus grande de ce chiffre.
Ici, nous présentons un protocole détaillé pour la préparation des échantillons et la gestion du logiciel. Nous commençons par des instructions détaillées sur la culture des cellules et l'acquisition de films optimisés pour l'analyse d'image. Cette section sur l'acquisition de données est suivie d'une description détaillée pour l'utilisation du logiciel d'analyse d'image. Tout au long du protocole, nous présentons des étapes critiques et des fonctionnalités facultatives qui devraient être prises en compte lors de la collecte et du traitement des données. Enfin, nous analysons filopodia à partir de différents modèles de systèmes avec le logiciel d'analyse d'image, avant de terminer avec une comparaison du logiciel d'analyse d'image décrit avec d'autres programmes disponibles pour la quantification filopodiale et une discussion sur les limites et les orientations futures.
Protocole
1. Culture cellulaire
- Culture HeLa ou cellules COS dans le milieu Eagle Eagle modifié par Dulbecco (DMEM) contenant 4,5 g / L de D-glucose, dipeptide L-alanine-L-glutamine, pyruvate, 10% de sérum bovin fœtal et 10 unités / ml de pénicilline / streptomycine. Les neurones de la culture dans les milieux de culture sans L-glutamine, l'acide glutamique ou l'acide aspartique, additionnés de 0,5 mM de dipeptide L-alanine-L-glutamine, de suppléments neuronaux exempts de sérum et 10 unités / ml de pénicilline / streptomycine.
- Une fois que la concentration de 40% est atteinte, transférez les cellules avec des constructions de choix en utilisant un réactif de transfection selon les instructions du fabricant. Garder les cellules transfectées dans l'incubateur à 37 ° C et 5% de CO 2 pendant 15-18 h.
- Pour réduire les variations de pH et d'osmolarité (due à l'évaporation) pendant l'acquisition de l'image, remplissez les chambres de culture cellulaire (avant l'imagerie) jusqu'à 90% avec un milieu préchauffé à 37 ° C contenant 20 mM de 4- (2-hydroxyéthyl) -1 -pipérazine-éthanesulfonique (HEPES). À scellerLe couvercle, appliquer une fine couche de graisse sous vide à l'intérieur du couvercle et appuyer doucement sur la chambre de culture contenant les cellules transfectées.
2. Acquisition d'image
NOTE: La longueur de filopodia varie de 2 à 10 μm 13 . Filopodia se développe à une vitesse moyenne de 0,05-0,1 μm / s 13 , 14 .
- Acquérir des images à l'aide d'un microscope avec objectif 60X ou 100X et sans binage de pixels (ici, un microscope confocal à disque tournant). Utilisez des taux d'acquisition supérieurs à 1 Hertz (Hz) pour suivre la dynamique des filopodies. Pour minimiser les artefacts hors focus, image filopodia près de la membrane basale ( c'est-à-dire la surface du substrat).
- Pour assurer un suivi en douceur, ajustez le temps d'exposition de l'appareil photo et l'intensité du laser de telle sorte que le rapport signal / bruit (SNR) soit supérieur à 4. Évitez la saturation des canaux individuels ( c.-à-d. Valeurs de pixels de255 pour les images 8 bits et 65 535 pour les images 16 bits), car cela empêchera l'analyse d'image ultérieure.
- Pour éviter la purge, utilisez uniquement des étiquettes de fluorescence compatibles avec les lignes laser et les filtres du microscope (pour plus de détails, voir la référence 15 ).
3. Prétraitement de l'image
REMARQUE: utilisez ImageJ ou autre logiciel disponible pour prétraiter les images 16 , 17 .
- Si l'échantillon se déplace, corrigez la dérive latérale à l'aide du logiciel disponible ( par exemple https://github.com/NMSchneider/fixTranslation-Macro_for_ImageJ) avant l'analyse. Exclure les films avec une dérive axiale (c'est-à-dire un mouvement en direction z).
- L'arrière-plan correct ( c.-à-d. Les valeurs de gris des zones en dehors de la cellule), le blanchiment ( c.- à-d. Une perte continue si l'intensité de fluorescence due aux effets des protéines de fluorescence) et une purge possible ( c'est -à- dire le signal d'une grippeSonde d'orescence dans les deux canaux) en utilisant le logiciel disponible ( p. Ex. Http://imagej.net/Category:Plugins et 16 , 17 ).
Remarque: Les intensités de fluorescence des canaux individuels ne sont pas modifiées par le logiciel. - Pour assurer une analyse d'image ultérieure, enregistrez les films correspondant à des chaînes de protéines particulières en échelle de gris sous forme de fichier empilé .tiff dans le dossier de travail.
REMARQUE: les dimensions ( c'est-à-dire la taille, la longueur) des piles doivent être identiques pour tous les canaux.
4. Analyse d'image - Étape 1: Charger des images
REMARQUE: Le logiciel décrit ici a été écrit dans Matlab (appelé logiciel de programmation) et fonctionnera uniquement avec ce programme.
- Téléchargez le dossier zippé contenant tous les fichiers requis pour l'analyse d'image à partir du site suivant: https://campus.uni-muenster.de/en/einrichtungen/impb0/nanoscale-forces-in-cells/softwsont/. Décompressez et copiez les fichiers dans le dossier de travail.
- Une fois installé, ouvrez le logiciel de programmation et exécutez 'filopodiaAnalysisM3.fig'. La fenêtre GUI n ° 1 s'ouvrira comme indiqué sur la figure 2A .
- Chargez les fichiers ".tiff" empilés sauvegardés correspondant à une protéine particulière d'intérêt dans la fenêtre GUI # 1 en utilisant les boutons <1b> pour la protéine A, <1c> pour la protéine B, <1d> pour la protéine C. Voir la figure 2 et la table 1 pour plus de détails.
REMARQUE: la protéine A présentée dans la fenêtre GUI # 1 sert de chaîne de référence pour l'analyse ratiométrique finale. - Créez une image superposée de la cellule à partir des canaux protéiques en cliquant <1a>.
- Cliquez sur <2a> pour attribuer la première image et <2b> pour la dernière image utilisée pour l'analyse.
- En option, recadrer la région d'intérêt (ROI) contenant le filopodium d'intérêt en utilisant le bouton <2h>, faire pivoter l'image.Chanter le bouton <2i>, ou supprimer des régions indésirables à l'aide de l'outil de dessin gratuit <2j>.
REMARQUE: pour rationaliser l'analyse d'image, il est recommandé d'isoler le ROI ( c.-à-d. Recadrer, faire pivoter, supprimer) avec les autres étapes de prétraitement (soustraction d'arrière-plan, corrections de blanchiment, etc.) en utilisant ImageJ. - Déplacez le curseur <2c> pour chaque image pour le contrôle de la qualité et pour vérifier si le filopodium reste visible sur tout le film.
5. Analyse d'image - Étape 2: générer une trace
- Cliquez sur le bouton <3a> dans la fenêtre GUI # 1 pour ouvrir la fenêtre GUI # 2 (voir la figure 2B ).
- Cliquez sur le bouton <4> dans la fenêtre GUI # 2 pour générer le masque du corps cellulaire superposé (généré dans la fenêtre GUI # 1 après avoir cliqué sur <1a>). Le programme génère également la limite du masque, où la coque convexe est implémentée pour obtenir les points de vertex.
- CliTouche ck <5>; Un curseur apparaîtra. Utilisez le curseur pour sélectionner la base (à partir de laquelle la distance de la pointe du filopodie est mesurée) suivie de la pointe du filopodia dans le cadre où elle apparaît pour la première fois. Pour ce faire, déplacez le curseur dans la fenêtre n ° 2).
REMARQUE: Pour minimiser les erreurs dans la sortie de données, placez le point de base verticalement sous le pointage filopodique le long de l'axe. Le positionnement du point de base sur d'autres endroits ( par exemple déplacé latéralement) peut introduire une polarisation directionnelle dans les valeurs de fluorescence dans le corps de la cellule. - Sélectionnez la longueur de seuil (ci-dessus que le filopodia se pliera) en utilisant <6c>.
Remarque: Cette longueur est définie comme la distance entre le point de base (sélectionné en utilisant <5>) et la limite du corps de la cellule ( c. -à- d. La région d'où le filopodia commence à croître). La distance mesurée est en pixels. - Spécifiez le nombre de segments utilisés pour approximer la forme des filopodes dans la case <6a>.
REMARQUE: le mLe nombre minimum de segments dépend de la longueur maximale atteinte par le filopodium, mais aussi de la façon dont le filopodium se penche. Ne sélectionnez pas un nombre de segments supérieur au nombre de pixels entre la base et la pointe ( c'est -à- dire la longueur de seuil). Sélection de plus de segments que la longueur de seuil définie à l'étape 5.4. (C'est-à- dire le nombre de pixels entre la base et la pointe) entraînera une surestimation de la longueur du filopodium. - Spécifiez la largeur de numérisation, qui agit comme un scanner horizontal pour placer les noeuds, en <6b>.
REMARQUE: ces noeuds sont utilisés par le programme pour s'adapter à la ligne qui relie la base et la pointe avec le corps du filopodium ( c.- à- d. Pour créer l'ossature). Comme point de départ, mettez une valeur en pixels égale à la longueur maximale multipliée par un facteur de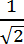 .
. - Spécifiez le rayon de balayage (en pixels) dans la zone <6d>. Utilisez une valeur qui est environ 50% plus grande que l'obA servi de déplacement d'extrémité entre les cadres.
REMARQUE: l'utilisation d'un rayon de balayage très important pourrait prendre des points de coque convexe indésirables dans des cadres où il n'y a pas de pointe filopodique réelle ( p. Ex. Mouvement hors-plan ou faible SNR). - Spécifiez l'angle de flexion dans la zone <6e>.
NOTE: Le seuil d'angle est déterminé par l'angle maximal que le filopodium se penche pendant toute l'analyse. La spécification du seuil d'angle aide le logiciel à exclure le suivi des structures indésirables qui croissent du côté des filopodes lorsque le filopodia se penche vers le corps de la cellule. Le programme fonctionne de manière fiable pour les filopodes avec des angles basculants inférieurs à 45 degrés par rapport à l'axe vertical. - Pour commencer le suivi, cliquez sur le bouton dans la fenêtre GUI # 2. Cliquez sur la case "Historique trace" dans la fenêtre GUI # 2 pour enregistrer tout le protocole de suivi pour une référence ultérieure.
REMARQUE: Une fois la procédure de suivi terminée, la longueur du filopodium dans chaque image est stockée dansLa feuille nommée 'length_vel' de 'dynamics.xlsx' pour les références futures. De même, tous les autres paramètres de suivi sont mémorisés dans la feuille appelée «paramètres» de «dynamics.xlsx». - En option, si le filopodia n'est pas automatiquement détecté dans toutes les images, utilisez les étapes suivantes pour corriger manuellement.
- Visitez le cadre respectif à l'aide du curseur dans la fenêtre n ° 2 et sélectionnez manuellement la pointe du filopodium.
Remarque: Les cadres, où aucun point de coque convexe n'est détecté, seront représentés par une région bleue dans la fenêtre de suivi de la fenêtre GUI # 2. Il est obligatoire de vérifier le bouton 'Historique trace' avant que le programme de suivi ne soit lancé pour accéder aux coordonnées nodales dans cette image. - Sélectionnez les points de référence (base suivie d'un conseil) en utilisant <6f> dans la fenêtre GUI # 2. Spécifiez les autres paramètres tels que «longueur de balayage», «Mesure précise après» et «Angle de flexion maximal»; Pour ce cadre comme décrit aux étapes 5.4-5.8.
- Cliquez sur le bouton <6i> pour enregistrer les nouveaux paramètres (spécifiques à cette image). Les étapes doivent être répétées pour tous les cadres indiqués par la région bleue.
- Une fois terminé, réinitialisez la procédure de suivi en utilisant <7>.
NOTE: Cette correction peut également être utilisée si le programme passe brusquement d'un filopodie à l'autre dans un film. En alternative, envisagez de recadrer des films contenant plusieurs filopodes.
- Visitez le cadre respectif à l'aide du curseur dans la fenêtre n ° 2 et sélectionnez manuellement la pointe du filopodium.
6. Analyse d'image - Étape 3: analyse spatio-temporelle des protéines
- Pour l'analyse spatio-temporelle, sélectionnez la case représentée par <8b> suivie du canal protéique d'intérêt (<8c> et / ou <8d> et / ou <8e>).
Remarque: Il est obligatoire de sélectionner la protéine A avant l'analyse ratiométrique (<9a>, <9b> et / ou <9c>) puisque la protéine A est utilisée comme référence. - Cliquez sur <8a>Pour initier le suivi des protéines le long de la longueur du filopodie en utilisant la trace générée à l'étape 5.9.
7. Analyse d'image - Étape 4: Analyse de protéines métriques
- Cochez la case <9b> ou <9c> et cliquez sur le bouton <9a> pour obtenir la trame ratiométrique spatio-temporelle.
REMARQUE: pour éviter une représentation inexacte de la concentration relative de protéines, l'image ratiométrique n'est pas tracée comme X / Y mais comme log (X / Y). Pour une utilisation future, le tracé ratiométrique est exporté dans les fichiers au format '.png' et '.fig', et les données du tracé brut sont stockées dans le fichier 'dynamics.xlsx'.
8. Analyse d'image - Étape 5: analyse de pointe de filopodie
- Cochez la case <8f> et spécifiez la longueur de la pointe et la longueur de seuil à partir de la base en utilisant <8g> et <8h>. Cliquez sur pour enregistrer les données dans le fichier 'dynamics.xlsx'. Cliquez sur pour générer leTrace des intensités de protéines de la pointe de la filopodie et pour économiser pour l'analyse ratiométrique.
REMARQUE: la longueur de l'embout détermine le nombre de pixels à la pointe utilisée pour l'analyse. Le logiciel renverra la valeur d'intensité moyenne des pixels à la pointe sur chaque image. La longueur de seuil détermine la distance minimale de la base à la pointe au-dessus de laquelle le filopodium (pointe filopodique) commence à croître (et lorsque le programme commence à suivre la valeur d'intensité de la pointe). La longueur de la pointe doit donc être inférieure à la longueur de seuil. - Cliquez sur le ratio souhaité à analyser en utilisant <9d> ou <9e>.
- Cliquez sur le bouton de comparaison <9a> pour générer les données ratiométriques.
Résultats
En utilisant des cellules COS transfectées avec un marqueur pour l'actine filamenteuse (f-tractine 18 , rouge) et une référence cytosolique (vert), nous avons trouvé des protrusions filopodiales riches en actine ( figure 3A , panneau supérieur). Les séries chronologiques ont montré que les filopodes se prolongent et se rétractent rapidement ( figure 3A , panneau central). En utilisant le logic...
Discussion
Nous présentons ici un protocole détaillé pour le suivi de la dynamique de croissance filopodiale et l'analyse des concentrations protéiques relatives dans ces structures dynamiques via l'algorithme de coque convexe. En utilisant le logiciel, jusqu'à 3 canaux peuvent être comparés par paire dans une seule opération, de sorte que les concentrations relatives de deux canaux ( c'est-à-dire les protéines) sont déterminées tout au long du cycle d'extension / rétractation et stockées...
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Les auteurs reconnaissent le financement du DFG (EXC-1003 à MG).
matériels
| Name | Company | Catalog Number | Comments |
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Références
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon