JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
동적 세포 돌출부에서 소프트웨어를 이용한 단백질 농도 추적을위한 그래픽 사용자 인터페이스
요약
우리는 동적 세포 돌출부의 길이에 따른 상대 단백질 농도의 반자동 추적을위한 소프트웨어 솔루션을 제시합니다.
초록
Filopodia는 이동 및 세포 - 세포 통신과 관련된 동적 인 손가락 모양의 세포 돌출부입니다. 섬유 유두의 개시, 연신율 및 후속 안정화 또는 수축의 근본적인 복잡한 신호 메커니즘을 더 잘 이해하기 위해 이러한 동적 구조에서 시공간 단백질 활성을 결정하는 것이 중요합니다. Filopodia에서 단백질 기능을 분석하기 위해 우리는 최근에 filopodial 모양 변화에 적응하는 반자동 추적 알고리즘을 개발하여 전체 filopodial 길이에 따른 돌출부 역학 및 상대 단백질 농도의 병렬 분석을 허용했습니다. 여기서는 최적화 된 셀 처리, 이미지 수집 및 소프트웨어 분석을위한 자세한 단계별 프로토콜을 제시합니다. 또한 이미지 분석 및 데이터 표현 과정에서 옵션 기능을 사용하는 방법에 대한 지침과 함께 중요한 모든 단계에 대한 문제 해결 지침을 제공합니다. 마지막으로, 우리는 또한 dfilopodia 부량을 위해 유효한 다른 프로그램을 가진 escribed 영상 분석 소프트웨어. 함께 제시된 프로토콜은 이미지 분석 소프트웨어를 사용하여 유두 돌출에서 단백질 역학의 정확한 분석을위한 프레임 워크를 제공합니다.
서문
actin 조절 단백질의 시공간 조절은 filopodium dynamics 1 , 2 와 관련이있다. 시간에 따른 전체의 유두 길이에 따른 공간적으로 분석 된 단백질 농도를 추적하는 것은 이러한 동적 구조의 개시, 신장, 안정화 또는 붕괴의 기초가되는 메커니즘에 대한 우리의 이해를 증진 시키는데 중요하다 3 , 4 . 세포 모양의 변화가 더 큰 규모에서 일어나는 세포질에서의 단백질 분석과는 달리, filopodia는 끊임없이 5 번 굴곡하여 선형 주사와 같은 간단한 접근법을 사용하여 분석을 방해하는 동적 인 미세 구조이다.
유두 모양 추적을위한 다양한 소프트웨어 솔루션을 사용할 수 있습니다 6 , 7 , 8 , 9 . 유사성ise, 세포 동체 내에서 단백질 동역학의 비율 측정 용 소프트웨어가 개발되었습니다 10 , 11 . 우리는 철근 형상의 자동 추적과 시공간 단백질 분석을 결합하기 위해 최근에 convex-hull 알고리즘을 기반으로 한 이미지 분석 소프트웨어를 개발했습니다. 그래픽 사용자 인터페이스 (GUI) 를 통해 작동하는이 새로운 분석 방법은 처음으로 유두 길이와 성장 속도에 따른 상대적 단백질 농도를 결합하여 이들의 움직임에 관계없이 시공간 단백질 분포의 정확한 측정을 가능하게합니다 동적 구조 12 .
소프트웨어의 아이디어 (소스 코드는 자유롭게 사용할 수 있습니다. 아래 참조)는 볼록 선체의 꼭지점 중 하나가 필리 포듐의 팁과 일치한다는 것입니다 ( 그림 1A ). 후속 프레임을 보면r은 convex-hull의 가장 가까운 꼭지점이며 움직이는 팁은 전체 영화에서 추적 할 수 있습니다. 일단 각 프레임에서 팁이 검출되면, 그 위치는 필로소움의 기저에있는 기준점과 팁을 결합함으로써 축을 그리는 데 사용됩니다 ( 그림 1B ). 마지막으로, 축에 직각 인 선을 따라 최대 강도를 갖는 중앙 픽셀에 의해 위치가 결정되는 등거리 노드 점을 사용하여 파일 포트 모양을 따르는 백본을 결정합니다. 이 adaptive backbone을 이용하여 kymograph가 생성되어 filopodial 길이 ( 그림 1C )를 따라 최대 3 개의 채널에 대한 filopodial 성장과 단백질 농도를 추적합니다.
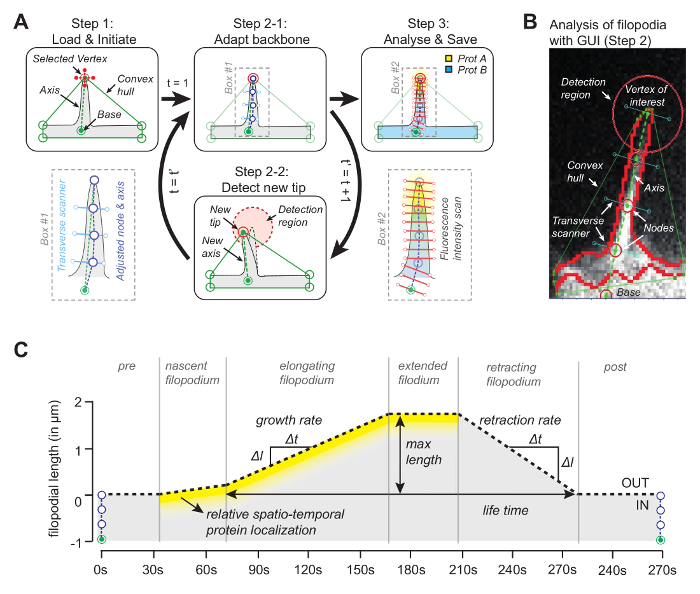
그림 1 : 이미지 분석 소프트웨어의 작동 원리. ( A ) 뒤에있는 알고리즘소프트웨어 1 단계에서 사용자는 필로폰의 참조 (기본)와 꼭지점 (끝)을 지정합니다. 단계 2-1에서, 최대 강도 값을 갖는 중간 값 픽셀을 사용하여 필로폰의 백본을 얻는다. 3 단계에서 백본은 공간 단백질 강도 프로파일에 사용됩니다. 단계 2-2에서 소프트웨어는 자동으로 후속 프레임의 팁을 추적합니다. 전체 절차가 반복됩니다. ( B ) 추적에 사용되는 볼록 선체와 같은 중요한 요소를 소개하는 실제 사상 최고로 알고리즘의 스냅 샷. ( C ) 알고리즘으로 측정 할 수있는 매개 변수 개요. 이 숫자는 참고 문헌 12 에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이미지 분석 소프트웨어는 그래픽 사용을 통해 Matlab (프로그래밍 소프트웨어라고 함)에서 작동합니다.r 인터페이스. 특정 실험 설정에 대한 유연성과 견고성을 극대화하기 위해 사용자는 일련의 추적 매개 변수 ( 예 : 허용 된 굽힘 각도 및 프레임 간 이동)를 조정할 수 있으며 영화 ( 예 : 자르기, 회전, 원하지 않는 객체 제거) ( 도 2a 및 표 1) .
| GUI | 아니. | 필수 | 기술 | 이름 (GUI에서) | ||||
| #1 | 1a | 와이 | 셀 본문을 나타내는 스택 된 .tiff 파일로드 (상자를 체크 인) 또는 채널에서 중첩 된 셀 본문 만들기 | CellBody | ||||
| #1 | 1b | 와이 | 단백질 1에 해당하는 적층 이미지 파일로드 | 단백질 1 | ||||
| #1 | 1c | 와이 | 단백질 2에 상응하는 스택 이미지 파일로드 | 단백질 2 | ||||
| #1 | 1d | 와이 | 단백질 3에 해당하는 적층 이미지 파일로드 | 단백질 3 | ||||
| #1 | 1e | 엔 | 미리로드 된 이미지 파일에 대한 모든 것을 재설정합니다. | 다시 놓기 | ||||
| #1 | 2a | 와이 | GUI 창 # 2에서 분석을위한 초기 프레임을 결정하기 위해 스크롤 막대 | 없음 | ||||
| #1 | 2b | 와이 | GUI 창 # 2에서 분석 할 최종 프레임을 결정하기 위해 스크롤 막대 | 없음 | ||||
| #1 | 2c | 와이 | curre를 나타내는 스크롤 막대NT 프레임 | 없음 | ||||
| #1 | 2d | 엔 | 모든 픽셀이 0으로 설정되는 픽셀의 회색 값 | 없음 | ||||
| #1 | 2e | 엔 | 모든 픽셀이 최대 값으로 설정되는 위의 픽셀의 회색 값 | 없음 | ||||
| #1 | 2f | 엔 | <2e> 및 <2f>에 지정된 픽셀의 강도 값을 설정합니다. | 세트 | ||||
| #1 | 2g | 엔 | 강도 조절 동영상 재생 | 놀이 | ||||
| #1 | 2 시간 | 엔 | 자르기 이미지 | 수확고 | ||||
| #1 | 2i | 엔 | 이미지 회전 | 회전 | ||||
| #1 | 2j | 엔 | 전체 스택에서 영역 삭제 | 영역 삭제 | ||||
| #1 | 3a | 와이 | '분석 창'(GUI 창 # 2)을 열려면 클릭하십시오. | 추적 창 | ||||
| #1 | 3b | 와이 | 픽셀 크기를 미크론 단위로 입력하십시오. | 픽셀 크기 입력 | ||||
| # 2 | 4 | 와이 | 중첩 된 셀 본문의 경계 / 가장자리 이미지를 생성하려면 클릭하십시오. | 경계 | ||||
| # 2 | 5 | 와이 | 유두의 기저부를 클릭하여 선택하십시오. | Referernce | ||||
| # 2 | 6a | 와이 | 세그먼트 또는 노드 수를 입력하십시오. | 세그먼트 수 | ||||
| # 2 | 6b | 와이 | 스캔 길이 (축에 수직)를 입력하십시오. | 스캔 너비 | ||||
| # 2 | 6c | 와이 | 철학이 굴곡을 시작하는 길이를 입력하십시오. | 정확한 측정 후 | ||||
| # 2 | 6d | 와이 | 팁 감지 원의 반지름을 입력하십시오 (즉, 다음 프레임에서 정점이 지역화 될 수있는 영역) | 팁 감지 반경 | ||||
| # 2 | 6e | 와이 | filypodium이 수직 축에서 구부릴 수있는 최대 각도를 입력하십시오. | 각도 임계 값 | ||||
| # 2 | 6f | 엔 | 해당 특정 프레임에 대한 기준점 및 기준점을 추가합니다. | 참조 선택 | ||||
| # 2 | 6g | 엔 | 해당 특정 프레임에 대해 절곡이 시작되는 길이를 입력하십시오. | 정확한 측정 후 | ||||
| # 2 | 6h | 엔 | 특정 프레임에 대한 감지 원의 반지름을 입력하십시오. | 팁 감지 반경 | ||||
| # 2 | 6i | 엔 | 특정 프레임에 대한 모든 매개 변수를 입력 한 후 추가 참조를 위해 값을 메모리 및 파일에 저장하려면 클릭하십시오. | 더하다 | ||||
| # 2 | 6j | 엔 | 해당 프레임의 수동 매개 변수를 삭제하려면 클릭하십시오. | 지우다 | ||||
| # 2 | 6k | 엔 | 모든 프레임에 대해 '선택적 기능 패널'을 사용하여 수동으로 저장된 모든 매개 변수를 삭제하려면 클릭하십시오. | 다시 놓기 | ||||
| # 2 | 6l | 엔 | 추적 전에 모든 추적 결과를 메모리에 저장하여 나중에 참조 할 수 있도록하십시오. | 기록 추적 | ||||
| # 2 | 7 | 와이 | 추적을 시작하려면 클릭하십시오. | 추적 및 분석 | ||||
| # 2 | 8a | 엔 | 단백질 채널 강도 추적 시작하려면 클릭하십시오. | 단백질 농도 분석 | ||||
| # 2 | 8b | 엔 | 유두 길이에 따른 단백질 채널 강도를 추적하려면 체크인하십시오. | 전체 filopodia | ||||
| # 2 | 8c | 엔 | 참조 단백질 또는 단백질 A를 추적하기 위해 체크인하십시오. | ProteinA | ||||
| # 2 | 8d | 엔 | 단백질 B를 추적하기 위해 체크인하십시오. | ProteinB | ||||
| # 2 | 8e | 엔 | 단백질 C를 추적하기 위해 체크인하십시오. | ProteinC | ||||
| # 2 | 8f | 엔 | 체크인시 평균 단백질 강도를 확인하십시오.전자 팁 | 이끄는 팁 | ||||
| # 2 | 8g | 엔 | 팁의 길이를 입력하십시오. | 팁 길이 | ||||
| # 2 | 8 시간 | 엔 | 팁이 형성되기 시작하는 바닥에서 최소 거리를 입력하십시오 | 문지방 | ||||
| # 2 | 8i | 엔 | 선도 팁 분석 결과를 파일로 저장하려면 클릭하십시오. | 누름 단추 | ||||
| # 2 | 9a | 엔 | Ratiometric 단백질 분석을 시작하려면 클릭하십시오 | 비교 | ||||
| # 2 | 9b | 엔 | 단백질 B를 A와 비교하기 위해 체크인하십시오. | log10 (B / A) | ||||
| # 2 | 9c | 엔 | A와 관련하여 단백질 C를 비교하기 위해 체크인하십시오. | log10 (C / A) | ||||
| # 2 | 9d | 엔 | 팁에서 A와 관련하여 단백질 B를 비교하기 위해 체크인하십시오. | log10 (B / A) | ||||
| # 2 | 9e | 엔 | 팁에서 A와 관련하여 단백질 C를 비교하려면 체크인하십시오. | log10 (C / A) | ||||
| # 2 | 10a | 엔 | 다른 색상 맵을 선택하십시오 (기본값 : Jetplot). | 컬러 맵 | ||||
| # 2 | 10b | 엔 | 색상 맵 편집 | 색상 맵 편집 | ||||
표 1 : GUI에있는 모든 기능 개요 Windows # 1 및 # 2.
이 작업이 완료되면 프로그램은 볼록 선체를 만들고 영화 전체에 걸쳐 팁을 자동으로 추적합니다. Ratiometric kymograph, 성장 속도 및 유두 길이와 같은 영화에서 추출한 매개 변수다시 표시되며 이미지 및 데이터 파일로 작업 폴더에 저장됩니다. filopodial lifetime, growth rate 및 retraction rate와 같은 다른 매개 변수를 추출하고 저장된 데이터 파일 ( 그림 2B )에서 추가로 분석 할 수 있습니다.
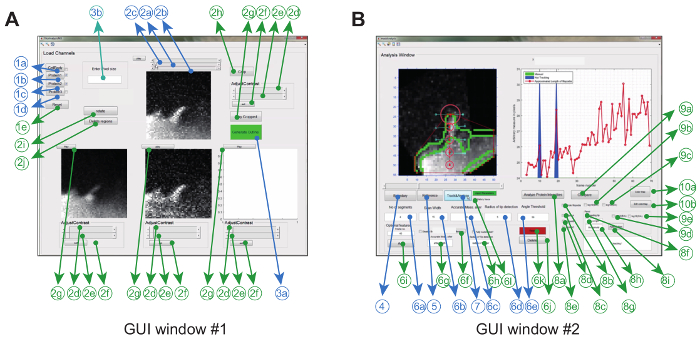
그림 2 : 이미지 분석 소프트웨어를 사용하기위한 그래픽 사용자 인터페이스. ( A ) GUI 창 # 1은 이미지로드 및 처리에 사용됩니다. 이 프로그램은 최대 3 개의 단백질 채널을로드 할 수 있으므로 2 개 채널을 쌍으로 비교합니다. 이 창에는 추적 전에 이미지를 사전 처리하기위한 필수 (파란색) 및 선택적 기능 (녹색)이 있습니다. ( B ) GUI 창 # 2는 시신경 및 비율 메트릭 단백질 분석뿐만 아니라 필로도를 추적하는 데 사용됩니다. 다시 옵션 기능은 녹색으로 표시됩니다. 이 숫자는 수정되었습니다.참고 문헌 12 에서 편집. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
여기에서는 샘플 준비 및 소프트웨어 처리를위한 상세한 프로토콜을 제시합니다. 우리는 세포 배양과 이미지 분석에 최적화 된 영화 획득에 대한 자세한 지침부터 시작합니다. 데이터 수집에 대한이 절 다음에는 이미지 분석 소프트웨어 작동에 대한 자세한 설명이 나와 있습니다. 프로토콜 전반에 걸쳐 데이터를 수집하고 처리 할 때 고려해야 할 중요한 단계와 옵션 기능을 소개합니다. 마지막으로 우리는 이미지 분석 소프트웨어로 다른 모델 시스템의 파일 포리아를 분석 한 후 설명 된 이미지 분석 소프트웨어와 유치원 계량화에 사용할 수있는 다른 프로그램을 비교하고 제한 사항 및 향후 방향에 대한 논의를 마무리합니다.
프로토콜
1. 세포 배양
- 4.5g / L D- 글루코오스, L- 알라닌 -L- 글루타민 디 펩티드, 피루 베이트, 10 % 소 태아 혈청 및 10 유닛 / ml 페니실린 / 스트렙토 마이신을 함유하는 둘 베코 변형 이글 배지 (DMEM) 중의 HeLa 또는 COS 세포 배양. 0.5 mM L- 알라닌 -L- 글루타민 디 펩티드, 무 혈청 신경원 보충제 및 페니실린 / 스트렙토 마이신 10 단위 / ml가 보충 된 L- 글루타민, 글루탐산 또는 아스파르트 산이없는 배지에서의 배양 뉴런.
- 40 %의 confluency가 도달되면 제조 업체의 지침에 따라 transfection 시약을 사용하여 선택의 구조로 세포를 transfect하십시오. 37에서 배양기에 transfected 세포를 유지 ° C와 5 % CO 2 15-18 시간.
- 이미지 수집 중 산도와 삼투압의 변화를 줄이려면 37 ℃에서 미리 90 %까지 세포 배양 챔버를 채우십시오 (예 : 20 mM 4- (2- 하이드 록시 에틸) -1) -1 (HEPES)를 포함한다. 밀봉뚜껑 안쪽에 얇은 진공 그리스를 묻혀 트랜 스펙 션 된 세포가 들어있는 배양기에서 가볍게 누릅니다.
2. 이미지 수집
참고 : filopodia의 길이는 2-10 μm에서 다릅니다. Filopodia는 0.05-0.1 μm / s의 평균 속도로 자랍니다 13 , 14 .
- 60X 또는 100X 대물 렌즈 및 픽셀 비닝 (여기서는 회전 디스크 공 촛점 현미경)이있는 현미경을 사용하여 이미지를 수집하십시오. 골반 동역학을 추적하기 위해 1 헤르츠 (Hz) 이상의 수집 속도를 사용하십시오. 초점이 맞지 않는 아티팩트를 최소화하기 위해 기저막 ( 즉, 기판 표면)에 가까운 영상 필로 디아를 사용합니다.
- 매끄러운 트래킹을 보장하려면 SNR (Signal to Noise Ratio)이 4 이상이되도록 카메라의 노출 시간과 레이저 강도를 조정하십시오. 개별 채널의 채도를 피하십시오 ( 예 :8 비트 이미지의 경우 255, 16 비트 이미지의 경우 65,535), 이후 이미지 분석이 어려워집니다.
- 블리드 스루 (bleed-through)를 피하려면 현미경의 레이저 라인 및 필터와 호환되는 형광 태그 만 사용하십시오 (자세한 내용은 참조 15 참조).
3. 이미지 전처리
참고 : ImageJ 또는 다른 사용 가능한 소프트웨어를 사용하여 이미지 16 , 17 을 사전 처리하십시오.
- 샘플이 움직이는 경우 분석 전에 사용 가능한 소프트웨어 ( 예 : https://github.com/NMSchneider/fixTranslation-Macro_for_ImageJ)를 사용하여 측면 드리프트를 보정하십시오. 축 방향 드리프트가있는 동영상 제외 (예 : z 방향으로의 이동).
- 올바른 배경 ( 즉 , 세포 외부 영역의 회색 값), 표백 (형광 단백질 손상으로 인한 형광 강도의 경우 연속적인 손실) 및 가능한 블리드 물마루 ( 즉 , 한 가지 독감orescence probe) ( 예 : http://imagej.net/Category:Plugins and 16 , 17 ).
참고 : 개별 채널의 형광 강도는 소프트웨어에 의해 변경되지 않습니다. - 후속 이미지 분석을 위해 특정 단백질 채널에 해당하는 영화를 그레이 스케일로 '.tiff'스택 파일 형식으로 작업 폴더에 저장하십시오.
참고 : 스택의 크기 ( 즉, 크기, 길이)는 모든 채널에서 동일해야합니다.
4. 이미지 분석 - 1 단계 : 이미지로드
참고 : 여기에 설명 된 소프트웨어는 Matlab (프로그래밍 소프트웨어라고 함)으로 작성되었으며이 프로그램에서만 실행됩니다.
- 다음 사이트에서 이미지 분석에 필요한 모든 파일이 들어있는 압축 폴더를 다운로드하십시오. https://campus.uni-muenster.de/en/einrichtungen/impb0/nanoscale-forces-in-cells/softw아르/. 압축을 풀고 파일을 작업 폴더에 복사하십시오.
- 일단 설치되면 프로그래밍 소프트웨어를 열고 'filopodiaAnalysisM3.fig'를 실행하십시오. 그림 2A 와 같이 GUI 창 # 1이 열립니다.
- 단백질 A는 <1b>, 단백질 B는 <1c>, 단백질 C는 <1d> GUI 창 # 1에서 관심있는 특정 단백질에 해당하는 저장된 스택 '.tiff'파일을로드하십시오. 그림 2 및 표 참조 자세한 내용은 1 페이지 를 참조하십시오.
참고 : GUI 창 # 1에 표시된 단백질 A는 최종 비율 측정 분석을위한 참조 채널 역할을합니다. - <1a>를 클릭하여 단백질 채널에서 세포의 이미지를 겹쳐서 만듭니다.
- <2a>를 클릭하여 첫 번째 프레임을 지정하고 <2b>를 분석에 사용 된 마지막 프레임으로 지정합니다.
- 선택적으로, <2h> 버튼을 사용하여 원하는 대상자를 포함하는 관심 영역 (ROI)을 자르고, 이미지를노래 버튼 <2i>을 사용하거나 손으로 그리는 도구 <2j>를 사용하여 불필요한 영역을 삭제하십시오.
참고 : 이미지 분석을 간소화하려면 ImageJ를 사용하여 ROI (자르기, 회전, 삭제)를 다른 전처리 단계 (배경 빼기, 표백 수정 등)와 함께 격리하는 것이 좋습니다. - 품질 관리를 위해 각 프레임에 대한 슬라이더 <2c>를 이동하고 전체 영화에서 필로 드가 명확하게 보이는지 확인하십시오.
5. 이미지 분석 - 2 단계 : 추적 생성
- GUI 창 # 1에서 <3a> 버튼을 클릭하여 GUI 창 # 2를 엽니 다 ( 그림 2B 참조).
- GUI 창 # 2에서 <4> 버튼을 클릭하여 겹쳐진 셀 본문의 마스크를 생성하십시오 (<1a>를 클릭 한 후 GUI 창 # 1에서 생성됨). 또한이 프로그램은 마스크의 경계를 생성하며, 볼록 선체가 구현되어 정점을 얻습니다.
- 클리ck 버튼 <5>; 커서가 나타납니다. 커서를 사용하여베이스를 선택하고 (여기에서 필립 드 팁의 거리를 측정 한 후), 처음 나타나는 프레임에서 필로 디아의 팁을 선택하십시오. 이렇게하려면 창 # 2에서 슬라이더를 움직입니다.
참고 : 데이터 출력의 오류를 최소화하려면 축을 따라 원위치를 filopodial 팁 아래에 수직으로 배치하십시오. 기준점을 다른 장소에 배치하면 ( 예 : 옆으로 이동) 세포체 내에서 형광 값에 방향성 편향이 생길 수 있습니다. - <6c>를 사용하여 문턱 길이를 선택하십시오.
참고 :이 길이는 기준점 (<5>를 사용하여 선택)과 세포체의 경계선 ( 즉 , 사상충증이 성장하기 시작하는 영역) 사이의 거리로 정의됩니다. 측정 거리는 픽셀 단위입니다. - <6a> 상자에서 유창 모양을 대략적으로 나타내는 데 사용되는 세그먼트 수를 지정하십시오.
참고 : m가장 많은 수의 세그먼트는 필로폰이 도달하는 최대 길이에 달려 있지만, 또한 필로소움이 어떻게 구부러 지느냐에 달려있다. 기본과 팁 사이의 픽셀 수보다 큰 세그먼트 수 ( 예 : 임계 길이)를 선택하지 마십시오. 5.4 단계에서 정의한 임계 길이보다 많은 세그먼트를 선택합니다. ( 즉, 베이스와 팁 사이의 픽셀 수)는 필로 피이드의 길이를 과대 평가하게됩니다. - <6b>에 노드를 놓을 수평 스캐너 역할을하는 스캔 너비를 지정하십시오.
참고 :이 노드는베이스와 팁을 filopodium의 본문에 연결하는 선에 맞게 프로그램에서 사용됩니다 ( 즉 , 백본을 만드는 데 사용됩니다). 출발점으로 최대 길이에 인수를 곱한 값을 픽셀 단위로 입력하십시오.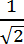 .
. - <6d> 상자에 스캔 반경 (픽셀 단위)을 지정하십시오. ob보다 약 50 % 큰 값을 사용하십시오.프레임 간 팁 이동을 제공했습니다.
참고 : 매우 큰 스캔 반경을 사용하면 실제 filopodial 팁이없는 프레임에서 원하지 않는 convex-hull 점을 얻을 수 있습니다 ( 예 : 평면 이동 또는 낮은 SNR). - <6e> 상자에 굽힘 각도를 지정하십시오.
참고 : 각도 임계 값은 전체 분석 중에 filopodium이 구부려지는 최대 각도로 결정됩니다. 각도 임계 값을 지정하면, 소프트웨어가 filopodia가 세포체쪽으로 구부러져있을 때, filopodia의 측면에서 성장하는 원치 않는 구조의 추적을 제외시키는 데 도움이됩니다. 이 프로그램은 세로 축에서 45도 미만의 기울기 각도를 가진 filopodia에 대해 안정적으로 작동합니다. - 추적을 시작하려면 GUI 창 # 2에서 <트랙 및 분석> 버튼을 클릭하십시오. 나중에 참조 할 수 있도록 전체 추적 프로토콜을 저장하려면 GUI 창 # 2에서 '기록 추적'상자를 클릭하십시오.
참고 : 추적 절차가 완료되면 각 프레임에서 필로폰의 길이가나중에 참조 할 수 있도록 'dynamics.xlsx'의 'length_vel'이라는 시트가 있습니다. 마찬가지로 다른 모든 추적 매개 변수는 'dynamics.xlsx'의 'parameters'시트에 저장됩니다. - 선택적으로, 모든 프레임에서 파일 아이콘이 자동으로 검색되지 않으면 다음 단계를 사용하여 수동으로 수정하십시오.
- 창 # 2의 슬라이더를 사용하여 각각의 프레임을 방문하여 수동으로 필로소움의 팁을 선택하십시오.
참고 : 볼록 선체 점이 발견되지 않은 프레임은 GUI 창 # 2의 추적 창에서 파란색 영역으로 표시됩니다. 해당 프레임의 노드 좌표에 액세스하기 위해 추적 프로그램이 시작되기 전에 '기록 추적'버튼을 선택해야합니다. - GUI 창 # 2에서 <6f>를 사용하여 참조 점 (밑면과 뒤끝)을 선택하십시오. '스캔 길이', '정확한 측정 후'및 '최대 굴곡 각도'와 같은 다른 매개 변수를 지정하십시오.; 그 프레임에 대해서는 5.4-5.8 단계에 설명 된대로.
- <6i> 버튼을 클릭하여 새 매개 변수 (해당 한 프레임에만 해당)를 저장하십시오. 파란색 영역으로 표시된 모든 프레임에 대해 단계가 반복됩니다.
- 완료되면 <7>을 사용하여 추적 절차를 다시 초기화하십시오.
참고 :이 보정은 프로그램 내에서 영화 내에서 돌연변이가 갑자기 바뀌는 경우에도 사용할 수 있습니다. 대안으로, 여러 가지 사체를 포함하는 동영상 자르기를 고려하십시오.
- 창 # 2의 슬라이더를 사용하여 각각의 프레임을 방문하여 수동으로 필로소움의 팁을 선택하십시오.
6. 이미지 분석 - 3 단계 : 시공간 단백질 분석
- 시공간 분석을 위해 <8b>로 표시된 상자를 선택한 다음 관심있는 단백질 채널 (<8c> 및 / 또는 <8d> 및 <8e>)을 선택하십시오.
참고 : Protein A가 참조로 사용되므로 비율 분석 (<9a>, <9b> 및 / 또는 <9c>) 전에 단백질 A를 선택해야합니다. - <8a>를 클릭하십시오.단계 5.9에서 생성 된 추적을 사용하여 유두 길이를 따라 단백질 추적을 시작합니다.
7. 이미지 분석 - 4 단계 : 비율 - 메트릭 단백질 분석
- <9b> 또는 <9c> 확인란을 선택하고 <9a> 버튼을 클릭하여 시공간 비례 곡선을 얻습니다.
참고 : 상대 단백질 농도의 허위 진술을 피하기 위해 비율 계산 이미지는 X / Y로 표시되지 않고 log (X / Y)로 표시됩니다. 나중에 사용하기 위해 비율 그림은 '.png'및 '.fig'형식 파일로 내보내지고 원시 플롯 데이터는 'dynamics.xlsx'파일에 저장됩니다.
8. 이미지 분석 - 5 단계 : 유두 팁 분석
- <8f> 확인란을 선택하고 <8g> 및 <8h>를 사용하여 밑면에서 팁 길이 및 임계 길이를 지정합니다. 데이터를 'dynamics.xlsx'파일에 저장하려면 <누름 버튼>을 클릭하십시오. 을 클릭하여filopodial tip의 단백질 강도를 추적하고 Ratiometric 분석을 위해 저장합니다.
참고 : 팁 길이는 분석에 사용되는 팁의 픽셀 수를 결정합니다. 소프트웨어는 각 프레임의 끝 부분에있는 픽셀의 평균 밝기 값을 반환합니다. 임계 길이는 기지에서 필로움 (filopodial tip)이 자라기 시작하는 팁까지의 최소 거리를 결정합니다 (그리고 프로그램이 팁의 강도 값을 추적하기 시작할 때). 따라서 팁 길이는 임계 길이보다 작아야합니다. - <9d> 또는 <9e>를 사용하여 분석 할 원하는 비율을 클릭하십시오.
- 비교 단추 <9a>를 클릭하여 비율 데이터를 생성하십시오.
결과
섬유질 악틴 (f-tractin 18 , 빨간색)과 세포질 참조 (녹색)에 대한 마커로 형질 감염된 COS 세포를 사용하여 우리는 액틴 풍부 유두 돌출을 발견했습니다 ( 그림 3A , 상단 패널). 시계열은 filopodia가 빠르게 늘어나고 철회하는 것을 보여 주었다 ( 그림 3A , 가운데 패널). 이미지 분석 소프트웨어를 사용하여 우리는 개...
토론
여기에 우리는 철근 - 성장 알고리즘을 통해 이러한 동적 구조에서의 상대 단백질 농도 분석과 생식기 성장 역학을 추적하기위한 상세한 프로토콜을 제시합니다. 이 소프트웨어를 사용하면 최대 3 개의 채널을 한 번에 쌍으로 비교할 수 있으므로 두 채널 ( 즉, 단백질)의 상대적 농도가 확장 / 수축 사이클 전체에서 결정되고 이미지 파일과 데이터 파일로 별도의 폴더에 저장됩니다. 일상적?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
저자는 DFG (EXC-1003에서 MG까지)의 자금 지원을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
참고문헌
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유