Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Membran-Nanoröhrchen ziehen aus riesigen Unilamellar Vesikel
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Viele Proteine in der Zelle spüren und Krümmung der Membran zu induzieren. Wir beschreiben eine Methode zur Membran Nanoröhrchen aus Lipid Vesicles, die Interaktion von Proteinen oder jede Krümmung-aktive Molekül mit gebogenen Membranen in VitroStudie ziehen.
Zusammenfassung
Die Umgestaltung der Zellmembran ist ein integraler Bestandteil der viele zelluläre Phänomene, z. B. Endozytose, Menschenhandel, die Bildung von Filopodien, etc.. Viele verschiedene Proteine assoziieren mit gebogenen Membranen aufgrund ihrer Fähigkeit, Sinn oder Membran Krümmung zu induzieren. Diese Prozesse beinhalten in der Regel eine Vielzahl von Proteinen macht sie zu komplex, um quantitativ in der Zelle zu studieren. Wir beschreiben ein Protokoll, um eine gebogene Membran in Vitro, rekonstruieren imitiert eine gebogene Zellstruktur, wie der endocytic Hals. Ein giant Unilamellar Vesicle (GUV) dient als ein Modell der Zellmembran, dessen Innendruck und Oberflächenspannung mit Mikropipette Aspiration gesteuert werden. Anwendung einer Zugkraft von Punkt auf der optischen Pinzette GUV erstellt ein Nanotube starken Kurven auf eine flache Membran verbunden. Diese Methode ist traditionell verwendet, um die grundlegenden mechanischen Eigenschaften der Lipidmembranen, wie biegen Steifigkeit zu messen. In den letzten Jahren wurde es erweitert um Interaktion von Proteinen mit Membran-Krümmung und die Art und Weise, die Sie die Form und die Mechanik der Membranen beeinflussen, zu studieren. Ein System verbindet Mikromanipulation, Mikroinjektion, optische Pinzette und konfokalen Mikroskopie ermöglicht Messung der Krümmung der Membran Membran Spannung und die Flächendichte von Proteinen, gleichzeitig. Aus diesen Messungen können viele wichtige mechanische und morphologische Eigenschaften von Protein-Membran-System abgeleitet werden. Darüber hinaus legen wir ein Protokoll GUVs im Beisein von physiologischen Salzkonzentration und eine Methode zur Quantifizierung der Flächendichte von Proteinen auf der Membran von Fluoreszenz-Intensität von markierte Proteine und Lipide zu schaffen.
Einleitung
Viele zelluläre Prozesse, wie z. B. Endozytose, Menschenhandel, die Bildung von Filopodien, Infektion, etc., werden durch eine dramatische Veränderung in der Form von Zellmembranen1,2begleitet. In der Zelle beteiligen sich eine Reihe von Proteinen in diesen Prozessen durch Bindung an die Membran und verändern ihre Form. Die bemerkenswertesten Beispiele sind Mitglieder der Bin/gekrümmte/Rvs (BAR)-Proteinfamilie, enthält ein Merkmal intrinsisch geschwungene BAR Domäne3,4,5,6,7. In der Regel interagieren sie mit der Membran durch die Einhaltung der BAR-Domäne an die Oberfläche und in vielen Fällen auch flach amphipathische Helices in der Bilayer einfügen. Form, Größe und Ladung des Geschäftsfeldes BAR zusammen mit der Anzahl der amphipathische Helices bestimmt: (1) die Richtung der Krümmung der Membran (d. h., ob sie Invaginations oder Vorsprünge induziert werden), und (2) das Ausmaß der Membran Krümmung5,8. Der Hinweis hier positive Krümmung der konvexen Seite die gebogene Membran, d.h., die Ausbuchtung in Richtung der wechselwirkenden Teilchen, und negative sonst bezeichnet. Darüber hinaus quantitative Studien der BAR Proteine ergaben, dass ihre Wirkung auf die Membran hängt von einer Reihe von physikalischen Parametern: Dichte von Proteinen, Membran-Spannung und Membran-Form (flach im Vergleich zu röhrenförmigen im Vergleich zu sphärischen Oberfläche Form)7. Abhängig von diesen Parametern BAR Proteine können: (1) dienen als Sensoren der Membran Krümmung, (2) beugen Membranen oder (3) induzieren Membran Spaltung7.
Aufgrund der Vielzahl der Beteiligten bei der Neugestaltung der Membran in die Zelle, studieren die quantitativen Aspekte der Phänomene, wie z. B. Endozytose Komponenten, ist in Vivo extrem schwierig. In-vitro- Rekonstitution der minimalen Komponenten imitiert gekrümmte Membranen in der Zelle ermöglicht ein mechanistischen Verständnis wie Membran-geschwungene Proteine zu betreiben. Dieser Artikel beschreibt ein Protokoll um eine Membran Nanoröhrchen in Vitro mit Mikromanipulation, konfokale Mikroskopie und optische Pinzette wieder zusammenzusetzen. Der Ansatz kann verwendet werden, in einer quantitativen Weise zu untersuchen, wie Proteine, Lipide oder kleine Moleküle mit gebogenen Membranen interagieren. Lipid GUVs dienen als Modelle für eine Zellmembran, deren Krümmung im Vergleich zur Größe der interagierenden Membran-geschwungene Moleküle vernachlässigbar. Sie sind bereit, mit der Electroformation Methode9 in dem die Bläschen gebildet werden, durch ein Lipid-Film feuchtigkeitsspendende und Schwellung es in GUVs unter einem Wechselstrom (AC)10. Am häufigsten verwendeten Substrate auf die GUVs angebaut werden sind entweder halbleitenden Platten beschichtet mit Indium-Zinn-Oxid (ITO) oder Platin (Pt-Drähte)11Drähte. In dieser Arbeit werden GUVs auf Pt-Drähte angebaut, wie diese Methode viel besser als die Alternative bei der Herstellung GUVs in Anwesenheit von Salzen in den Puffer12Arbeiten gezeigt hat. Obwohl das Electroformation-Protokoll ausreichend detailliert, es zu reproduzieren hier beschrieben ist, verweisen wir den Leser auf früheren Artikeln, in denen ähnliche und andere Verfahren zur Herstellung von GUVs im Detail13,14beschrieben wurden. In unseren Händen hat Electroformation auf Pt-Drähte erfolgreich GUVs aus einer Mischung von synthetischen Lipiden oder natürliche Lipid Extrakte in einem Puffer mit ~ 100 mM NaCl erbracht. Darüber hinaus war es auch möglich, Proteine innen GUVs während des Wachstums zu Kapseln. Abbildung 1Azeigt eine Beispiel-Electroformation-Kammer; Es besteht aus zwei ca. 10 cm lange Pt-Drähte eingefügt in eine Halterung hergestellt aus Polytetrafluorethylen (PTFE), die auf beiden Seiten mit Glasdeckgläser versiegelt werden kann ~ 1-2 cm auseinander (Abb. 1A).

Abbildung 1: Versuchsaufbau. (A) die GUV Electroformation Kammer mit elektrischen Anschlüssen an Pt-Drähte befestigt. (B) links: das experimentelle System das Mikroskop zeigt, die experimentelle Kammer über das Ziel und zwei Mikropipetten (links und rechts) die Mikromanipulatoren zugeordnet und eingefügt in die experimentelle Kammer für das Rohr zu ziehen und Protein Injektion. Rechts: eine Nahansicht der experimentellen Kammer über dem Ziel zeigen die Spitzen der Aspiration und die Injektion Mikropipetten eingefügt montiert. (C) eine Spritze ist ausgestattet mit einem dünnen Dispenser in einer Mikropipette an seinem hinteren Ende eingefügt. Der Boden ist eine Nahansicht des Spenders in die Mikropipette mit der blau gepunkteten Linie umreißt die Mikropipette. Dieses System wird verwendet, um die Mikropipette mit Kasein auf die Glasoberfläche passiviert und füllen mit Mineralöl bei Bedarf wieder zu füllen. (D) ein System zur µL Mengen der Proteinlösung Aspirieren. Die Nadel ist verbunden, um eine Spritze und Schläuche, die mit der Injektion Mikropipette verbunden ist. Die Mikropipette Spitze sorgfältig eingetaucht in die Proteinlösung und abgesaugt, also um die Mikropipette Spitze gefüllt. Die Mikropipette wird dann wieder gefüllt mit Mineralöl mit dem System gezeigt im Bedienfeld "C. Klicken Sie bitte hier, um eine größere Version dieser Figur.
Ein Membran-Nanoröhrchen, im Umkreis von 7 nm bis mehrere hundert nm, kann aus einer GUV durch eine externe Kraft gezogen werden. Diese Methode wurde ursprünglich entwickelt, um die elastischen Eigenschaften von Zellmembranen und Vesikel, wie z. B. die Biege Steifigkeit15,16messen. In jüngsten Arbeiten wurde die Methode erweitert, um die Wechselwirkung von Proteinen mit gebogenen Membranen zu studieren, von den Proteinen in der Nähe der gezogenen Nanotube7,17microinjecting. Andere Methoden haben für das Studium geschwungenen Membran Proteine entwickelt worden. In einer Methode sind Proteine mit unterschiedlich großen Liposomen angebunden an eine passivierte Oberfläche inkubiert. Konfokale Mikroskopie wird verwendet, um die Proteinbindung als Funktion der Liposomen Durchmesser messen die Krümmung-induzierte Sortierung18,19angeben können. In einer anderen Methode werden Proteine in der Nähe einer Mikro-aspirierten GUV, Messen Sie ihre Fähigkeit, spontan Tubuli20,21induzieren injiziert. In diesem Protokoll beschriebene Methode eignet sich eindeutig studieren geschwungenen Membran Proteine beteiligt Endozytose, begegnen die meisten Proteine in der Regel vorgeformte Membran Nanoröhren verbinden die Fracht-haltigen Membran Einstülpen mit der zugrunde liegenden flachen Plasmamembran. Darüber hinaus ist bei dieser Methode wird im Gegensatz zu in der Probe mit gefesselte kleine Liposomen Membran Nanotube kontinuierlich an die Membran verbunden; Daher ist es im mechanischen Gleichgewicht mit der GUV, eine Situation erwartet in Vivo. Daher grundlegende Membran Physik gilt, und wir können eine Vielzahl von mechanischen Eigenschaften ableiten, aus unseren Messungen22,23,24.
Für eine vollständige Implementierung dieser Methode beinhaltet die notwendige Ausrüstung ein confocal Mikroskop, optische Pinzette und ein oder zwei Mikropipetten mit einem Wassertank (Abbildung 1 b) verbunden. Durch die Kombination aller drei, ist es möglich, gleichzeitig messen Membran Spannung, Membran Krümmung, Flächendichte von Proteinen, und Kraft25Rohr. Mikropipette Aspiration ist wichtig und es ist einfach aufgebaut, indem man ein Glas Mikropipette in eine Halterung mit einem Wassertank, der über hydrostatischen Druck die Aspiration Druck26 steuertverbunden. Die Mikropipette und der Halter sind mit einem Mikromanipulator und idealerweise in eine Richtung ein Piezo-Aktor für präzise Bewegung gesteuert. Ein Nanoröhrchen zu ziehen, ist Microaspirated GUV kurz auf ein Mikron mittelständische Korn dann zog Weg schaffen ein Nanotube aufgeklebt. In dieser Implementierung ist die Perle von optischen Pinzette gehalten, konstruiert werden kann, indem Sie die folgenden veröffentlichten Protokoll27. Es ist möglich, auf unterschiedliche Weise der optischen Pinzette und Pull-Nanoröhren verzichten zwar auf Kosten der genaue Kraftmessungen. Wenn es zu schwierig, eine optische Falle zu bauen oder Kraftmessungen sind nicht zwingend notwendig, wie will man einfach nur die Vorliebe von Proteinen für gekrümmte Membranen überprüfen kann ein Rohr mit einer Perle an der Spitze einer zweiten Mikropipette28aspiriert gezogen werden. Es ist auch möglich, ziehen Röhrchen mit Gravitationskraft29 oder30,31zu fließen. Darüber hinaus ist auch konfokalen Mikroskopie nicht wesentlich; Es ist jedoch so bevorzugt die Flächendichte von Proteinen zu messen. Es ermöglicht auch die Messung des Nanotube-Radius von Fluoreszenzintensität von Lipiden in der Röhre, also unabhängig von der Membran Kraft und Spannung. Ableitens Rohr-Radius von Fluoreszenz ist besonders wichtig, wenn die Beziehung zwischen diesen Größen von etablierten Gleichungen aufgrund des Vorhandenseins der Membran eingehalten Proteine25abweicht. Wichtig ist, kann nicht einer optischen Falle sowohl der konfokalen Mikroskopie zu verzichten, da es nicht möglich, die Krümmung des Rohres zu messen.
Die Methode wie beschrieben in diesem Protokoll wurde verwendet, um zu studieren, die Krümmung-induzierte Sortierung von verschiedenen peripheren Membranproteinen auf Nanoröhren, vor allem diejenigen aus der BAR Familie25,32,33,34 . Es wurde auch gezeigt, dass die konisch geformten transmembranen Kaliumkanal KvAP auf bereichert ist als BAR-Proteine35Nanoröhren in gleicher Weise gebogen. Durch die Optimierung der Methode um Proteine innen GUVs Kapseln, ist die Interaktion von Proteinen mit negativer Krümmung vor kurzem als gut36untersucht worden. Darüber hinaus wurde diese Methode, die Bildung von Protein Gerüste25,37 aufzuklären und den Mechanismus der Spaltung der Membran durch entweder Linie Spannung38, Protein Dynamin39, oder BAR zu studieren verwendet Proteine-40,-41. Neben Proteinen können kleine Moleküle oder Ionen auch Krümmung auslösen. Mit dieser Methode wurden Calcium-Ionen induzieren positive Krümmung unter salzfreie Bedingungen42gezeigt. Interessanterweise hat auch gezeigt, dass die Lipide Krümmung sortieren, obwohl nur für Kompositionen unterziehen können, die in der Nähe ein demixing Punkt43,44. Zusammenfassend lässt sich sagen die Methode kann verwendet werden, von Forschern interessiert untersucht wie entweder fester Membran-Komponenten (z. B., Lipide oder transmembranen Proteine) oder peripher Moleküle binden (entweder innen oder außen GUVs) interagieren mit zylindrisch gebogenen Membranen aus mechanischen und quantitativen Gesichtspunkten. Es richtet sich auch an Interessenten, die bei der Messung der mechanischen Eigenschaften der Membran selbst22,23,45.
Protokoll
1. Vorbereitung des GUVs von Electroformation auf Pt-Drähte
- Reinigen Sie die Electroformation Kammer (siehe Einführung und Abbildung 1A) und die Pt-Drähte mit einem organischen Lösungsmittel wie Ethanol oder Aceton, die Lipide Weg zu waschen und mit Wasser abzuwaschen die Salze.
Hinweis: Wir empfehlen gründlich abwischen entfernt die Rückstände mit einem Ethanol-getränkten Tuch dann in Aceton, Ethanol, dann Wasser, jeweils für 5 min beschallen. - Bereiten Sie eine Lipid-Mischung aus einer gewünschten Lipidzusammensetzung bei 1 mg/mL in Chloroform. Die Mischung sollte enthalten ~ 0,05 % molare Bruch ein Lipid konjugiert mit Biotin (z.B., 1,2-distearoyl-sn-glycero-3-phosphoethanolamine-N-[biotinyl(polyethyleneglycol)-2000]) und ~ 0,5 % molare Bruch ein Lipid konjugiert mit einem Fluorophore ( z. B.BODIPY-TR-C5-Ceramid).
Hinweis: Nach unserer Erfahrung war es schwierig, GUVs an einen hohen Ertrag mit mehr als 30 % produzieren Lipide in Rechnung gestellt.
Achtung: Chloroform sollte in einem chemischen Kapuze mit entsprechenden Handschuhen gehandhabt werden. - Legen Sie ein Kabelpaar Pt in der Electroformation Kammer. Die Lipid-Mischung auf die Pt-Drähte in Tropfen durch getrennt hinterlegen ~ 2-3 mm (insgesamt ~ 4 µL des Mix).
- Trocknen Sie die Drähte unter Vakuum für 30 – 60 min bei Raumtemperatur.
- Der Boden der Kammer durch die Einhaltung der Deckglas über die Kammer mit Silikonfett zu versiegeln. Füllen Sie die Kammer mit einer gepufferten Lösung mit NaCl, Saccharose und eine Pufferung Agent (z.B.70 mM NaCl, Saccharose 100 mM und 10 mM Tris, pH 7.4). Dieses Medium wird innerhalb der GUVs im Experiment sein. Es ist wichtig, dass die Osmolarität dieses Mediums die Osmolarität des experimentellen Mediums innerhalb entsprechen ~ 10 %. Verwenden Sie Saccharose, um die Osmolarität anzupassen. Fügen Sie Proteine der Lösung bei einer gewünschten Konzentration hinzu, wenn das Ziel ist, ihnen in die GUVs gekapselt.
Achtung: Verschiedene Salze und Zucker im Puffer können die Biegesteifigkeit und die spontane Krümmung der Membranen24,42,46,47auswirken. - Versiegeln Sie die Spitze der Kammer durch die Einhaltung von einem Deckgläschen mit Silikonfett gewährleisten minimale Luft in der Kammer. Anwenden einen Sinus AC-Strom durch die Pt-Drähte bei 500 Hz und 280 mV.
Hinweis: Die Wachstumszeit und die Temperatur müssen optimiert werden, abhängig von der Lipidzusammensetzung und der Empfindlichkeit des Proteins. Bei der Verwendung von natürlichen Lipid-Extrakte, und auch wenn Proteine in GUVs Verkapselung, die beste Ausbeute gelang durch die wachsenden GUVs bei 4 ° C über Nacht. In Ermangelung von Proteinen und für synthetische Lipid Kompositionen, Wachstum bei Raumtemperatur für ~ 2 h war ausreichend.
2. Vorbereitung der experimentellen Kammer und der Mikropipetten
- Um Mikropipetten vorzubereiten, ziehen Sie einer Glas-Kapillare mit einer Pipette Puller. Es wird vorgeschlagen, um eine Glas-Kapillare mit internen und externen Radien, bzw. 0,7 mm und 1 mm zu verwenden. Dann die Spitze der Mikropipette verfeinern, damit seine Innendurchmesser 5–ist 7 µm, mithilfe eines Microforge. Wenn Proteine oder andere Moleküle im Experiment injiziert werden, ziehen Sie ein weiteres Mikropipette und verfeinern Sie ihrer Spitze einen Innendurchmesser von 8–15 µm.
- Eine experimentelle Kammer zu konstruieren, indem man zwei rechteckige Mikroskopie Deckgläsern auf einem metallischen Untergrund, wie in Abbildung 1dargestellt. Die Deckgläsern sollten durch getrennt werden ~ 1 mm. Die Kammer sollte Öffnungen entlang den langen Kanten aufweisen (siehe Abbildung 1). Die offenen Seiten sollte passen die Spitze der Mikropipette, wo die Spitze sollte mindestens die Mitte der Kammer erreichen.
- Bereiten Sie die experimentelle Puffer deren Osmolarität unterscheiden sich nicht von mehr als 10 % aus dem Puffer verwendet, um GUVs wachsen sollte. Passen Sie die Osmolarität mit Glukose. Ein Beispiel für eine experimentelle Puffer, der verwendet wurde, bei der Untersuchung der Wechselwirkung von Endophilin mit Nanoröhren ist 100 mM NaCl, Glukose 40 mM Tris, pH 7,4 gepuffert. Eine Kombination von Saccharose in / Glukose außerhalb sorgt für: (a) ausreichende Phasenkontrast, GUVs mit Hellfeld Mikroskopieren und (b) höhere Dichte innerhalb der GUVs, wodurch sie sich auf den Boden der Kammer zu beobachten. Passen Sie Salzkonzentration basierend auf experimentellen Anforderungen an.
Hinweis: Neben den Auswirkungen auf die mechanischen Eigenschaften der Membran, erfahrungsgemäß beeinträchtigt hohe Glukosekonzentration (> 300 mM) die Einrichtung von Streptavidin-Biotin-Bindungen, erforderlich, um ein Nanoröhrchen zu ziehen. Darüber hinaus zu wenig Salz hemmt Streptavidin-Biotin-Anleihen, während zu hohe Konzentration Bildschirme Membran-Protein Interaktionen. Im Falle von Protein Kapselung, mit sehr hoher Salzkonzentration in der externen Puffer (z.B.> 200 mM NaCl) eignet sich als ein Trick, um die Proteine aus der äußeren Merkblatt36zu lösen. Es ist notwendig, experimentieren Sie mit einer Reihe von Salzkonzentration, die optimale verbindliche Bedingungen für das Molekül des Interesses zu finden. -
30 – 60 min vor der Erhebung GUVs für das Experiment passiviert die Glasflächen durch Ausfüllen der experimentellen Kammer und die Aspiration Mikropipette mit einer 5 mg/mL Lösung ein hochreines β-Casein (z. B.in der experimentellen Puffer aufgelöst). Β-Casein schafft eine Schutzschicht auf den Glasflächen verhindert GUVs zu stark einzuhalten, die diese zerplatzen verursachen würde. Mit β-Casein für 30 – 60 min inkubieren.
Hinweis: Sollte keine Luftblasen im Inneren der Mikropipette, die Membrane Spannung steuern stören würde.- Um einen Spender zu bauen zum Füllen von Mikropipetten, brechen einer Spritzennadel in der Nähe der Kunststoffstecker und kleben hinein eine dünne Silizium-Kapillare (z. B. für die Flüssigchromatographie verwendet) (Abbildung 1).
- Während der Inkubation mit β-Casein montieren Sie die Kammer am Mikroskop und zentrieren Sie ihn über das Ziel. Führen Sie die Spitze der Aspiration Mikropipette durch die Öffnung an der langen Kante und bringen Sie die Spitze über das Mikroskopobjektiv. Einstellen Sie den Wasserstand Tank so, dass der Anspruch Druck nahe Null ist (es sollte keine schwere Strömung in oder aus der Pipette, die unter dem Mikroskop gesehen werden können).
-
Für den Fall, dass die Proteine oder andere Moleküle während des Experiments injiziert werden, füllen die Injektion Mikropipette mit das gewünschte Molekül aufgelöst im experimentellen Puffer in einer Konzentration von Wahl, montieren die Mikropipette in einem Mikromanipulator und durch die Gegenseite der experimentellen Kammer schieben.
- Um eine minimale Menge an Proteinen zu nutzen, füllen Sie nur die Mikropipette Spitze mit dem Protein durch Absaugen. Um dies zu erreichen, wickeln Sie das Ende einer Nadel einer Spritze mit Kunststoffschlauch befestigt. Legen Sie den Schlauch in den hinteren Teil der Injektion Mikropipette.
- Sehr sorgfältig Tauchen Sie die Spitze der Mikropipette in die Proteinlösung und aspirieren Sie es um die Spitze der Mikropipette gefüllt.
Hinweis: Dieses einfache Setup wird angezeigt in Figur 1 und nach unserer Erfahrung ermöglicht Absaugen von so wenigem wie einige µL der Proteinlösung. - Den Rest der Injektion Mikropipette mit Mineralöl Hinterfüllung zu verhindern, mischen die Proteinlösung und das Wasser aus dem Wassertank (über das Setup in Abbildung 1).
- Achten Sie darauf, nicht Luftblasen in die Injektion Pipette einführen wie es instabil Einspritzdruck induziert wird. Alternativ reicht die Menge Protein, füllen Sie die Mikropipette mit den Proteinen genauso wie Füllung mit β-Casein wie unter Punkt 2.4 beschrieben. Der Pegel Wasser Tank zur Minimierung den Aspiration Druck in die Injektion-Pipette.
Hinweis: Die Konzentration der injizierten Molekül oder Ion in der Nähe der Nanotube wird durch Verdünnung niedriger sein und es kann durch die Messung der Fluoreszenz-Intensität-Abnahme als Funktion des Abstandes von der Pipette Ausfahrt42geschätzt werden. Es ist jedoch wichtiger Bilayer-gebundenen Dichte des injizierten Moleküls, wissen die genau gemessen werden kann (siehe Abschnitt 4).
- Entfernen Sie nach der Inkubation mit β-Casein die Lösung aus der Kammer, und spülen Sie mehrere Male mit der experimentellen Puffer. Mit dem experimentellen Puffer zu füllen.
- Electroformation GUVs zu stoppen und direkt aus der Pt-Drähte zu sammeln. Fügen Sie ein paar µL der GUV-Lösung zur experimentellen Kammer. Verwenden Sie nur frisch zubereitete GUVs.
- Die experimentelle Kammer, eine Endkonzentration von Perlen in der Kammer um 0,1 x 10−3 % (w/V) oder weniger ein paar µL Streptavidin beschichteten Perlen hinzufügen. Polystyrol-Kügelchen ~ 3 µm im Durchmesser empfohlen (Polystyrol-Kügelchen im Wasser haben einen nahezu optimale Brechungsindex Kontrast im Hinblick auf die Maximierung der Neigungskraft in Bezug auf die Streuung Kraft in der optischen Falle). Die Perle-Konzentration lässt sich anhand des Experiments: eine sehr niedrige Konzentration macht es schwierig, sie in der Kammer zu finden, und eine zu hohe Konzentration läuft Gefahr, mehrere Perlen in der optischen Pinzette fallen.
3. ziehen ein Membran-Nanoröhrchen aus einer GUV
- Lassen Sie die GUVs und die Perlen auf den Boden der Kammer zu begleichen. Die GUVs zu entlüften, indem man ein wenig des Puffers experimentelle verdunsten, für etwa ~ 15 min. Deflationierung der GUVs unbedingt einige überschüssige Bereich produzieren, die mit der Mikropipette abgesaugt werden kann. GUVs sollte sichtbar Wogen (d.h.erscheinen Diskette) unter dem Lichtmikroskop. Wenn sie angespannt erscheinen, geben Sie mehr Verdunstung Zeit.
Hinweis: Die Osmolarität Änderungsrate hängt die verdunstende Oberfläche, Temperatur usw.und sollte überwacht werden. Im Prinzip der Osmolarität Unterschied könnte durch Anpassen der Saccharose/Glukosekonzentration von Anfang eingestellt werden, jedoch sollte darauf geachtet werden nicht zu einen osmotischen Schock auslösen. - Eine Diskette GUV zu finden und in die Pipette abzusaugen. Die Länge der Aspiration Zunge (der Teil der Membran im Inneren der Pipette) sollte gleich oder größer als der Radius der Pipette für die theoretische Analyse geltenden15,16,22,()23 ( Abbildung 2). Versuchen Sie mehrere GUVs. Wenn keiner die GUVs abgesaugt werden kann, um eine lange Zunge zu produzieren, noch warten Sie ein paar Minuten. Wenn GUVs Disketten genug sind, fahren Sie mit dem nächsten Schritt fort.
- Versiegeln Sie die Kammer mit Mineralöl, um weitere Verdunstung des Puffers zu verhindern. Dazu das Öl an den offenen Rändern der experimentellen Kammer vorsichtig pipettieren.
- Um die Nullstellung der Aspiration Druck einzustellen, erstens eine Perle in der Kammer suchen und streben Pipette Ausgang in der Nähe der Wulst zu positionieren. Die Höheneinstellung des Wassertanks, so dass die Wulst ist weder überwältigt von der Aspiration Pipette angesaugt. Obwohl das Mineralöl Verdunstung und daher weitere Druckänderungen in der Kammer verhindert, null streben Druck vor der Messung jedes GUV.
- Suchen Sie eine GUV und aspirieren Sie es. Verschieben Sie die Mikropipette oben unscharf (zu halten die GUV Weg von der Oberfläche, wo könnte es gezogen werden aus der Pipette durch Scherbeanspruchung zog die Kammer).
- Suchen Sie eine Perle von sorgfältig die Kammer bewegen. Plötzliche Bewegungen können die GUV auswerfen. Fangen sie mit optischen Pinzette entfernt ~ 20 µm von der unteren Kammer entfernt. Sicherstellen Sie, dass der relevante Bereich sauber ohne andere Perlen oder Membranen in Sicht ist. Alles fällt in der optischen Pinzette als die Perle wird die Messung stören.
- Bringen Sie die GUV wieder ins Blickfeld, und Weg von den Wulst mit der Mikropipette ausgerichtet mit der optischen Falle (Abbildung 2).
- Notieren Sie die Bewegung des Wulstes für 1 – 2 min. um die Gleichgewichtslage (erforderlich für Kraftmessungen) zu messen.
- Reduzieren Sie den Druck im Inneren der Mikropipette so weit wie möglich ohne Verlust der GUV so um die Membrane Spannung zu verringern. Bringen Sie sorgfältig die GUV in Kontakt mit der Wulst für ca. eine Sekunde, zur Gründung Streptavidin-Biotin-Anleihen, dann ziehen Sie vorsichtig zurück, ein Nanoröhrchen zu schaffen. Die Bewegung der GUV zu oder Weg von der Perle sollte im Idealfall mit einem Piezo-Aktuator, minimal stören die Perle in der optischen Pinzette erfolgen.
Hinweis: Wenn die Nanoröhre nicht gehört, könnte es durch schlechte Streptavidin-Beschichtung des Wulstes, unzureichende Menge an biotinylierte Lipide in der GUV, unzureichende Konzentration von Salz oder übermäßige Konzentration von Glukose in der experimentellen Puffer oder die Membran sein Spannung in der GUV ist zu hoch48. - Erhöhen Sie den Anspruch Druck um die Aspiration Zunge neu zu erstellen. Richten Sie das Rohr in der Achse der die Aspiration Pipette und maximal auf das Rohr (Abbildung 2) zu liegen.
- Sicherstellen Sie, dass die GUV-Äquator und das Rohr im Mittelpunkt stehen. Die Bewegung des Wulstes mit Hellfeld Mikroskopieren für ein paar Minuten aufzeichnen (Geschwindigkeit der Kamera hier 30 Hz). Zeichnen Sie die Höhe hdes Wasserkastens in Bezug auf die Nullposition. Nehmen Sie ein paar konfokale Bilder des Systems (Abbildung 2).
- Wiederholen Sie den vorherigen Schritt bei verschiedenen Aspiration drücken, implizit Membran Spannungen. Typische Spannung beträgt 0,015 – 0,2 mN/m, mit einer Schrittweite von ca. 0,02 mN/m.
- Wenn Proteine oder Moleküle in der Nähe der Anlage zu injizieren, bringen Sie die Injektion Mikropipette in der Nähe von Nanoröhrchen, um sicherzustellen, dass die Perle in der optischen Falle nicht gestört wird. Sanft zu injizieren, bei einem Druck von rund 1 bis 2 PA.
-
Nachdem die Proteinbindung (der Fluoreszenzintensität des injizierten Proteins auf die Membran bleibt konstant auf die GUV) equilibriert hat, wiederholen Sie die schrittweisen Messungen wie bei der bloßen Membran (Schritte 3.11 und 3.12).
Hinweis: Da die Messungen durchgeführt werden, während der Injektion von Proteinen in der Nähe der GUV bei konstantem Druck, bleibt die größte Konzentration des Proteins etwa gleich in der Nähe der GUV; so sollte während der Messung Protein Desorption vernachlässigbar sein.- Alternativ ist es möglich, die GUVs zusammen mit den Proteinen inkubieren Sie vor der Durchführung von Experimenten Rohr ziehen, um eine konstante Masse Proteinkonzentration zu gewährleisten. Angesichts der Tatsache, dass die relative Membran Bruchteil der Proteine auf der Tube und auf die GUV gemessen wird, die Art und Weise Proteine werden geliefert hat keinen Einfluss auf die Krümmung Sortierung Berechnung (siehe Abschnitt 4).
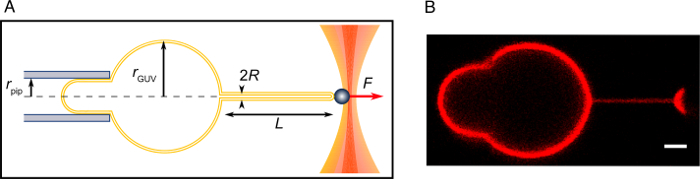
Abbildung 2: Schlauch ziehen Experiment. (A) Schaltpläne des Experiments. (B) ein konfokale Bild einer gezogenen Röhre wie beschrieben in diesem Protokoll. Maßstabsleiste = 2 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
4. Messungen und Datenanalyse
- Membran-Spannung messen
- Berechnen Sie den hydrostatischen Druck für jeden Schritt im ständigen Bestreben Druck:
ΔP = Pgh
wobei p die wasserdichte, Erdbeschleunigung g und h die Höhe des Wassertanks aus Schritt 3.11 ist. - Von konfokalen Bilder Messen der Radius der GUV RGUVund der Aspiration Pipette, RPip (Abbildung 2).
- Berechnen Sie die Membran-Spannung σ, mit Hilfe Laplaces-Gleichung-49:
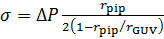
- Berechnen Sie den hydrostatischen Druck für jeden Schritt im ständigen Bestreben Druck:
- Membran-Kraftmessung
- Bestimmen Sie die Steifigkeit der optischen Pinzette, k, mithilfe eines mehrere Kalibrierung Methoden27. Messen Sie in diesem Setup k mit der Viskose Drag Methode27.
- Berechnen Sie die Gleichgewichtslage des Wulstes, eine0als Mittelwert aus einer Messung vor dem Abziehen der Röhre (Schritt 3,8).
- Berechnen Sie für jede konstante Spannung Messung der Gleichgewicht-Membran Kraft, F, aus Hookes Gesetz:
F = k(ein - 0)
wo ist eine durchschnittliche Position der Schweißnaht während dieser Messung.
- Mess-Rohr-radius
- Im Falle einer bloßen Membran (keine zusätzlichen Membran-geschwungene Moleküle) berechnen des Rohr-Radius, R, aus der Kraft als:
R = F/(4πσ)
(Referenzen22,23). - Zur Messung des Rohr-Radius im Beisein von Membran-geschwungene Moleküle und unabhängig von 4.3.1 Schritt zunächst, Aufzeichnen der Lipid-Fluoreszenzintensität entlang der Rohrlänge, ichWanne, und die GUV-Kontur, ichGUV. Messen Sie die durchschnittliche Fluoreszenzintensität einer Fluorophor in oder an die Membran gebunden als durchschnittliche Intensität entlang der hellsten ein Rohr oder eine GUV-Kontur. Wählen Sie einen rechteckigen Kasten mit einem horizontalen Abschnitt des GUV-Kontur oder das Rohr und berechnen Sie die Summe der Fluoreszenz-Intensität von jede horizontale Linie im Feld.
- Teilen Sie jede Summe durch die Anzahl der Pixel der horizontalen Linie (d.h. die Breite der Box). Beachten Sie, dass im Feld ausgewählte keine anderen Membranen vorhanden sein sollte. Die Fluoreszenz Intensität Profil entlang der Länge des ausgewählten Feldes ergibt.
- Nehmen Sie nach Abzug der Hintergrundintensität die durchschnittliche Pixel Fluoreszenzintensität von der hellste Linie. Der Rohr-Radius bezieht sich auf das Verhältnis von Fluoreszenz entlang der Kontur des Rohres und der GUV als linear:
R = KWanneichWanne/ichGUV
wo KWanne eine Kalibrierung Faktor25ist.
Hinweis: Diese Methode kann verwendet werden, zur Messung des Rohr-Radius im Bereich von 10-80 nm (wenn das Rohr schmal genug ist, um in einem konfokalen Voxel Breite liegen) und eine etwas größere Unsicherheit in den 80 nm und Hochtonbereich. Die Empfindlichkeit der Messung hängt von der Einrichtung.
- Bestimmen Sie K-Wanne durch die Durchführung unabhängiger Radius-Messungen in den Schritten 4.3.1 und 4.3.2 an einer einfachen Membran-Komposition mit ungeladenen Lipide. Solche einfachen Membran Zusammensetzung sorgt dafür, dass der Radius und die Kraft, die einfache Beziehung in Schritt 4.3.1 angegeben haben. Wiederholen Sie das Experiment mehrere Male und plot R, abgeleitet aus der Kraft (Schritt 4.3.1) versus ichWanne/ichGUV (Schritt 4.3.2). Berechnen Sie K-Wanne von der Passform. In diesem Setup, K-Wanne = 200 L-α-Phosphatidylcholin (Ei-PC) Membran und eine fluoreszierende Lipid, deren Fluoreszenz minimal Polarisation25 hängt± 50 nm mit Ei.
Hinweis: K-Wanne muss für unterschiedliche Ziele, aufgrund ihrer verschiedenen konfokale Voxel Volumina gemessen werden. Die Lipid-Fluorophor sollten nicht mit den injizierten Proteine/Molekülen interagieren. Die fluoreszierende Lipid empfohlen hier BODIPY TR Ceramid, voraussichtlich keine Interaktionen mit den Proteinen haben. Diese Annahme bestätigte sich mit früheren Studien von BAR und -BAR Domänen25,36,,37, die haben gezeigt, dass bei hohem Proteingehalt Flächendeckung der Rohr-Radius wird behoben, indem die Proteine, die unabhängig von der Spannung in der GUV. Wenn die Lipid-Fluorophor Proteine interagieren, wird eine Erschöpfung oder Anreicherung von der Fluorophor in das Rohr auf abwechslungsreiche hohe Oberfläche Proteinfraktionen beobachtet werden.
- Im Falle einer bloßen Membran (keine zusätzlichen Membran-geschwungene Moleküle) berechnen des Rohr-Radius, R, aus der Kraft als:
- Messung der Oberfläche Dichte von Proteinen
- Vorbereiten der Lipid-Mischungen mit einer einfachen ungeladenen Lipid (z.B. Ei-PC) ergänzt mit etwa fünf verschiedenen Maulwurf Bruchteilen von einem fluoreszierenden Lipid (der gleichen Wellenlänge wie der Farbstoff verwendet, um das Protein, z.B.beschriften BODIPY-FLC5-hexadecanoyl-Phosphatidylcholine (HPC *) für z.B., das Alexa488-Label Protein) im Bereich: XHPC * = 0,01-1 %. Bereiten Sie GUVs in 100 mM Saccharose durch Electroformation auf ITO Platten (folgen Sie Schritt 1 eine vorherige Veröffentlichung13).
- GUVs sammeln und übertragen auf eine experimentelle Kammer mit β-Casein passiviert. Verwenden Sie z. B.100 mM Glukose für die experimentelle Lösung. Warten Sie ein paar Minuten die GUVs zu begleichen.
- Konfokale Fluoreszenzbilder von GUVs nehmen und Aufzeichnen der durchschnittliche Fluoreszenzintensität entlang der GUV Konturen in Abhängigkeit von der Maulwurf Bruchteil der fluoreszierenden Lipid (siehe Punkt 4.3.2). Für jede Komposition berechnen die Flächendichte von fluoreszierenden Lipid, φHPC*. Z. B. durch vorausgesetzt, dass die Fläche pro Lipid 0,7 nm2, φHPC* = 1,43 x 106 XHPC * pro Broschüre. Plot-HPC * Fluoreszenz-Intensität in der GUV, ichHPC *, GUV, im Vergleich zu φHPC*. Passform ermöglicht die Kalibrierung konstant, A, gegeben durch φHPC* = AIHPC *, GUV. A hängt die Mikroskopie-Einstellungen, wie z. B. die Laserleistung und die Detektorempfindlichkeit (d. h.Gewinn), daher erfassen A für verschiedene häufigsten verwendeten Mikroskopie-Einstellungen (siehe Beispiel in Abbildung 3).
- Bereiten Sie mehrere Lösungen von HPC * in Reinigungsmittel, z.B.Natrium-Dodecyl-Sulfat (SDS), aufgelöst in verschiedenen Konzentrationen im Bereich von ~ 1, 10-50 µM. Fluoreszenzintensität der jeweiligen Lösung in großen Mengen erfassen und berechnen Sie die Steigung der Intensität im Vergleich zu Konzentration (Abbildung 3). Wiederholen Sie die Messung mit dem Fluorophor, die an das Protein in einem vergleichbaren Konzentrationsbereich konjugiert werden (siehe das Beispiel mit Alexa488 in Abbildung 3). Diese Messung ist erforderlich, um die Fluoreszenz-Intensitäten der Protein-Label und die Lipid-Label, ichA488, Bulk beziehen / ichHPC *, Bulk, da sie nicht unbedingt auf die gleiche Weise auf die gleiche Masse Konzentration freisetzen können.
- Messen Sie die Anzahl der Fluorophore pro Protein-Molekül mit einer Fluorospectrometer. Beispielsweise kann für den Fall der Alexa488, die Anzahl der Alexa488 Moleküle pro Protein, nA488, berechnet die folgende Beziehung:
n A488 = (A494/εA488) / ((A280 - 0,11A494) / εProt)
wo A494 und A280 sind die Extinktion Werte pro Längeneinheit an 494 nm und 280 nm, bzw., und εA488 und εProt sind die molekulare Absorption Koeffizienten der Alexa488 und Das Protein, beziehungsweise. - Berechnen Sie die Oberflächendichte des Proteins auf die GUV, φProt, GUV, nach folgender Formel:
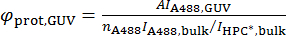
Denken Sie daran, die Polymerisation Zustand des Proteins. Z. B. BAR Proteinen Dimere, daher die berechnete Dichte wird der BAR Monomer pro Fläche wenn das Aussterben Koeffizient der Monomeren Form zu verwenden.

Abbildung 3: ein Beispiel für Protein Oberflächendichte Kalibrierung. Gemessen werden die HPC * Lipid Fluoreszenzintensität in loser Schüttung (links) und GUVs (Mitte). Auch der Großteil Fluoreszenzintensität von Alexa488 (gebunden mit einer BAR-Domäne) sind gemessen (rechts). Fluoreszenzintensität skaliert linear mit der Konzentration. Messungen gezeigt werden für spezifische Erkennung Gewinn und laser-Ausgangsleistung. Grundstücke generierten, basierend auf Daten von Referenz-37. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergebnisse
Das Rohr ziehen Experiment kann wichtige mechanische Informationen über die Membran geben. Bei fehlender Proteine oder andere Moleküle, die paar mit Krümmung der Membran, die Membran zu erzwingen und Rohr-Radius kann mit Membran Spannung bezogen werden, durch die Anwendung der Canham Helfrich Hamiltonian zog Gleichung zu einem Schlauch aus GUV50,51
Diskussion
Die Methode des Ziehens Röhren aus GUVs gibt umfassende Informationen über das Membranprotein System, wie es nicht nur die Mittel ist, um die grundlegenden mechanischen Eigenschaften der Membran zu messen, aber es hilft, die Aufschluss über die Kopplung zwischen Proteinen und Membran Krümmung. Wie in der Einleitung erwähnt, andere Techniken existieren, um die Wirkungen der Membran-geschwungene Proteine messen durch Inkubation der Proteine mit Sub-Mikrometer-Liposomen angebunden an eine passivierte Oberfläche
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren danken Benoit Sorre, Damien Cuvelier, Pierre Nassoy, François Quemeneur und Gil Toombes für ihre wesentliche Beiträge zur Nanotube-Methode in der Gruppe zu etablieren. Die P.B-Gruppe gehört das CNRS-Konsortium CellTiss, Labex CelTisPhyBio (ANR-11-LABX0038) und Paris Sciences et Lettres (ANR-10-IDEX-0001-02). F. C. Tsai wurde von EMBO langfristige Fellowship (ALTF 1527-2014) und Marie Curie Aktionen (H2020-MSCA-IF-2014, Projekt Membran-Ezrin-Aktin) finanziert. M.S. ist Junior Fellow Simons Society of Fellows.
Materialien
| Name | Company | Catalog Number | Comments |
| 1,2-Dioleoyl-sn-glycero-3-phosphatidylcholine (DOPC) | Avanti | 850375 | Example lipid used in data for Figure 3 |
| 1,2-distearoyl-sn-glycero-3-phosphoethanolamine-N-[biotinyl(polyethyleneglycol)-2000] [DSPE-PEG(2000)-biotin] | Avanti | 880129 | biotinylated lipid |
| BODIPY-TR-C5-ceramide | Molecular Probes (ThermoFisher) | D7540 | fluorescent lipid |
| BODIPY- FLC5-hexadecanoyl phosphatidylcholine (HPC*) | Molecular Probes (ThermoFisher) | fluorescent lipid for protein density calibration | |
| egg L-α-phosphatidylcholine (eggPC) | Avanti | 840051 | used for calibrating the tube radius constant |
| β-casein from bovine milk (>99%) | Sigma Aldrich | C6905 | used for passivating the micropipette and the experimental chamber |
| Sucrose | Sigma Aldrich | S7903 | |
| D(+) glucose | Sigma Aldrich | G7021 | |
| NaCl | Sigma Aldrich | S7653 | |
| Tris | Sigma Aldrich | 10708976001 | |
| osmometer | Loser | n/a | |
| Pt-wires, 0.5 mm diameter, 99.99% pure | Goodfellow USA | PT005139 | used for GUV electroformation |
| function generator | n/a | used to create current for GUV electroformation | |
| putty sealant | Vitrex (from CML France) | CRIT 140013 | used to seal the electroformation chamber |
| bath sonicator | n/a | useful to clean the electroformation chamber, but not crucial | |
| Nikon TE2000 inverted microscope, eC1 confocal system (Nikon), with two laser lines (λ = 488 nm and 543 nm); optical tweezers induced by a 5 W ytterbium fiber continuous wave laser (λ > 1070 nm; IPG GmBH Germany) | an example of a confocal microscopy system equipped with optical tweezers | ||
| micromanipulators | n/a | ||
| borosilicate capillaries (with internal and external radii of 0.78 mm and 1 mm, respectively) | Harvard apparatus | 30-0036 | |
| micropipette puller | Sutter Instrument | P-2100 | |
| microforge | Narishige | MF-800 | |
| piezoelectric actuator | Physik Instrumente | n/a | |
| polystyrene streptavidin coated beads (diameter 3.2 µm) | Spherotech | SVP-30-5 |
Referenzen
- McMahon, H. T., Gallop, J. L. Membrane curvature and mechanisms of dynamic cell membrane remodelling. Nature. 438 (7068), 590-596 (2005).
- Johannes, L., Wunder, C., Bassereau, P. Bending "on the rocks"--a cocktail of biophysical modules to build endocytic pathways. Cold Spring Harb Perspect Biol. 6 (1), (2014).
- Dawson, J. C., Legg, J. A., Machesky, L. M. Bar domain proteins: a role in tubulation, scission and actin assembly in clathrin-mediated endocytosis. Trends Cell Biol. 16 (10), 493-498 (2006).
- Mim, C., Unger, V. M. Membrane curvature and its generation by BAR proteins. Trends Biochem Sci. 37 (12), 526-533 (2012).
- Qualmann, B., Koch, D., Kessels, M. M. Let's go bananas: revisiting the endocytic BAR code. EMBO J. 30 (17), 3501-3515 (2011).
- Rao, Y., Haucke, V. Membrane shaping by the Bin/amphiphysin/Rvs (BAR) domain protein superfamily. Cell Mol Life Sci. 68 (24), 3983-3993 (2011).
- Simunovic, M., Voth, G. A., Callan-Jones, A., Bassereau, P. When Physics Takes Over: BAR Proteins and Membrane Curvature. Trends Cell Biol. 25 (12), 780-792 (2015).
- Safari, F., Suetsugu, S. The BAR Domain Superfamily Proteins from Subcellular Structures to Human Diseases. Membranes (Basel). 2 (1), 91-117 (2012).
- Angelova, M. I., Soléau, S., Méléard, P., Faucon, F., Bothorel, P., Helm, C., Lösche, M., Möhwald, H. . Progress in Colloid and Polymer Science Vol. 89: Trends in Colloid and Interface Science VI. 89, 127-131 (1992).
- Meleard, P., Bagatolli, L. A., Pott, T. Giant unilamellar vesicle electroformation from lipid mixtures to native membranes under physiological conditions. Methods Enzymol. 465, 161-176 (2009).
- Montes, L. R., Alonso, A., Goni, F. M., Bagatolli, L. A. Giant unilamellar vesicles electroformed from native membranes and organic lipid mixtures under physiological conditions. Biophys J. 93 (10), 3548-3554 (2007).
- Mathivet, L., Cribier, S., Devaux, P. F. Shape change and physical properties of giant phospholipid vesicles prepared in the presence of an AC electric field. Biophys J. 70 (3), 1112-1121 (1996).
- Collins, M. D., Gordon, S. E. Giant liposome preparation for imaging and patch-clamp electrophysiology. J Vis Exp. (76), (2013).
- Garten, M., Aimon, S., Bassereau, P., Toombes, G. E. Reconstitution of a transmembrane protein, the voltage-gated ion channel, KvAP, into giant unilamellar vesicles for microscopy and patch clamp studies. J Vis Exp. (95), e52281 (2015).
- Hochmuth, R. M., Evans, E. A. Extensional flow of erythrocyte membrane from cell body to elastic tether. I. Analysis. Biophys J. 39 (1), 71-81 (1982).
- Hochmuth, R. M., Wiles, H. C., Evans, E. A., McCown, J. T. Extensional flow of erythrocyte membrane from cell body to elastic tether. II. Experiment. Biophys J. 39 (1), 83-89 (1982).
- Bassereau, P., Sorre, B., Levy, A. Bending lipid membranes: experiments after W. Helfrich's model. Adv Colloid Interface Sci. 208, 47-57 (2014).
- Hatzakis, N. S., et al. How curved membranes recruit amphipathic helices and protein anchoring motifs. Nature chemical biology. 5 (11), 835-841 (2009).
- Bhatia, V. K., et al. Amphipathic motifs in BAR domains are essential for membrane curvature sensing. EMBO J. 28 (21), 3303-3314 (2009).
- Shi, Z., Baumgart, T. Membrane tension and peripheral protein density mediate membrane shape transitions. Nat Commun. 6, 5974 (2015).
- Chen, Z., Shi, Z., Baumgart, T. Regulation of membrane-shape transitions induced by I-BAR domains. Biophys J. 109 (2), 298-307 (2015).
- Cuvelier, D., Derenyi, I., Bassereau, P., Nassoy, P. Coalescence of membrane tethers: experiments, theory, and applications. Biophys J. 88 (4), 2714-2726 (2005).
- Derenyi, I., Julicher, F., Prost, J. Formation and interaction of membrane tubes. Phys Rev Lett. 88 (23), 238101 (2002).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Adv Colloid Interface Sci. 208, 225-234 (2014).
- Sorre, B., et al. Nature of curvature coupling of amphiphysin with membranes depends on its bound density. Proc Natl Acad Sci U S A. 109 (1), 173-178 (2012).
- Evans, E., Rawicz, W. Entropy-driven tension and bending elasticity in condensed-fluid membranes. Phys Rev Lett. 64 (17), 2094-2097 (1990).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nat Methods. 5 (6), 491-505 (2008).
- Capraro, B. R., Yoon, Y., Cho, W., Baumgart, T. Curvature sensing by the epsin N-terminal homology domain measured on cylindrical lipid membrane tethers. J Am Chem Soc. 132 (4), 1200-1201 (2010).
- Bo, L., Waugh, R. E. Determination of bilayer membrane bending stiffness by tether formation from giant, thin-walled vesicles. Biophys J. 55 (3), 509-517 (1989).
- Rossier, O., et al. Giant Vesicles under Flows: Extrusion and Retraction of Tubes. Langmuir. 19 (3), 575-584 (2003).
- Borghi, N., Rossier, O., Brochard-Wyart, F. Hydrodynamic extrusion of tubes from giant vesicles. EPL (Europhysics Letters). 64 (6), 837 (2003).
- Zhu, C., Das, S. L., Baumgart, T. Nonlinear sorting, curvature generation, and crowding of endophilin N-BAR on tubular membranes. Biophys J. 102 (8), 1837-1845 (2012).
- Ramesh, P., et al. FBAR syndapin 1 recognizes and stabilizes highly curved tubular membranes in a concentration dependent manner. Sci Rep. 3, 1565 (2013).
- Knorr, R. L., et al. Membrane morphology is actively transformed by covalent binding of the protein Atg8 to PE-lipids. PLoS One. 9 (12), e115357 (2014).
- Aimon, S., et al. Membrane shape modulates transmembrane protein distribution. Dev Cell. 28 (2), 212-218 (2014).
- Prévost, C., et al. IRSp53 senses negative membrane curvature and phase separates along membrane tubules. Nat Commun. , (2015).
- Simunovic, M., et al. How curvature-generating proteins build scaffolds on membrane nanotubes. Proc Natl Acad Sci U S A. 113 (40), 11226-11231 (2016).
- Allain, J. M., Storm, C., Roux, A., Ben Amar, M., Joanny, J. F. Fission of a multiphase membrane tube. Phys Rev Lett. 93 (15), 158104 (2004).
- Morlot, S., et al. Membrane shape at the edge of the dynamin helix sets location and duration of the fission reaction. Cell. 151 (3), 619-629 (2012).
- Renard, H. F., et al. Endophilin-A2 functions in membrane scission in clathrin-independent endocytosis. Nature. 517 (7535), 493-496 (2015).
- Simunovic, M., et al. Friction Mediates Scission of Tubular Membranes Scaffolded by BAR Proteins. Cell. 170 (1), 172-184 (2017).
- Simunovic, M., Lee, K. Y., Bassereau, P. Celebrating Soft Matter's 10th anniversary: screening of the calcium-induced spontaneous curvature of lipid membranes. Soft Matter. 11 (25), 5030-5036 (2015).
- Sorre, B., et al. Curvature-driven lipid sorting needs proximity to a demixing point and is aided by proteins. Proc Natl Acad Sci U S A. 106 (14), 5622-5626 (2009).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proc Natl Acad Sci U S A. 107 (16), 7208-7213 (2010).
- Dasgupta, R., Dimova, R. Inward and outward membrane tubes pulled from giant vesicles. Journal of Physics D: Applied Physics. 47 (28), 282001 (2014).
- Vitkova, V., Genova, J., Mitov, M. D., Bivas, I. Sugars in the aqueous phase change the mechanical properties of lipid mono- and bilayers. Molecular Crystals and Liquid Crystals. 449, 95-106 (2006).
- Bouvrais, H. . Mechanical properties of giant vesicle membranes investigated by flickering technique. , (2011).
- Koster, G., Cacciuto, A., Derenyi, I., Frenkel, D., Dogterom, M. Force barriers for membrane tube formation. Phys Rev Lett. 94 (6), 068101 (2005).
- Kwok, R., Evans, E. Thermoelasticity of large lecithin bilayer vesicles. Biophys J. 35 (3), 637-652 (1981).
- Helfrich, W. Elastic properties of lipid bilayers: theory and possible experiments. Z Naturforsch C. 28 (11), 693-703 (1973).
- Canham, P. B. The minimum energy of bending as a possible explanation of the biconcave shape of the human red blood cell. J Theor Biol. 26 (1), 61-81 (1970).
- Marcerou, J. P., Prost, J., Gruler, H. Elastic Model of Protein-Protein Interaction. Nuovo Cimento Della Societa Italiana Di Fisica D-Condensed Matter Atomic Molecular and Chemical Physics Fluids Plasmas Biophysics. 3 (1), 204-210 (1984).
- Leibler, S. Curvature Instability in Membranes. J Phys-Paris. 47 (3), 507-516 (1986).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten