Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine Reinigung und In vitro- Aktivität Assay für ein (p) PpGpp Synthestase von Clostridium difficile
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine Methode zur Reinigung von Histidin-markierten Pyrophosphokinase Enzyme und Dünnschichtchromatographie radioaktiv Substrate und Produkte, für die enzymatische Aktivität in Vitroassay nutzen. Die Enzym-Aktivität-Assay ist breit anwendbar auf Kinase, Nukleotid-Cyclase oder Phosphor-Transfer-Reaktion, deren Mechanismus Nukleotid Triphosphat Hydrolyse umfasst.
Zusammenfassung
Kinase und Pyrophosphokinase Enzyme übertragen die Gamma-Phosphat oder Beta-Gamma Pyrophosphat glyko-aus Nukleotid Triphosphat Vorstufen auf Substrate phosphorylierten Produkte zu schaffen. Die Verwendung von γ -32- P bezeichnet NTP Vorstufen ermöglicht die gleichzeitige Überwachung der Substrat-Auslastung und Produkt-Bildung durch Röntgen. Dünnschichtchromatographie (TLC) auf Zellulose Platten ermöglicht schnelle Trennung und sensible Quantifizierung von Substrat und Produkt. Wir präsentieren Ihnen eine Methode zur Nutzung der Dünnschichtchromatographie um die Pyrophosphokinase Aktivität einer gereinigten (p) PpGpp Synthestase assay. Diese Methode wurde zuvor verwendet, um die Aktivität der zyklische Nukleotid und Dinucleotide Synthetasen charakterisieren und eignet sich im großen und ganzen für die Charakterisierung der Aktivität jedes Enzym, das eine Nukleotid Triphosphat Bindung hydrolysiert oder überträgt einen terminal Phosphat von einem Phosphat-Spender zu einem anderen Molekül.
Einleitung
Kinase und Pyrophosphokinase (oder Diphospho-Kinase) Enzyme ins Substrat Moleküle Phosphate aus Nukleotid Triphosphat (NTP) Vorstufen. Die Substrate können andere Nukleotide, Aminosäuren oder Proteine, Kohlenhydrate und Lipide1gehören. Bioinformatische Analysen können manchmal ein Enzym cognate Substrat oder Substrate aufgrund der Ähnlichkeit zu gekennzeichnet Enzyme, aber experimenteller Validierung ist immer noch notwendig. Ebenso müssen die Affinität des Enzyms für ihre Substrate und die Rate, mit der es die Phosphor-Transfer Reaktion katalysiert, und die Auswirkungen der Co-Faktoren, Inhibitoren oder anderen Enzym Effektoren experimentell ermittelt werden. Zur Vermeidung von Erschöpfung der ATP-Vorstufe durch andere Enzyme ATP verbrauchenden in bakteriellen Zytoplasma erfordern quantitative Aktivität Assays gereinigtes Protein.
Proteinreinigung durch Metall Affinitätschromatographie wurde gründlich in die Literatur2,3berichtet. Histidin-Tags bestehend aus sechs aufeinander folgende Histidin Rückstände angehängt, N - oder C-Terminus eines rekombinanten Proteins erlauben schnelle Reinigung von Metall Affinität Chromatographie4,5,6. Diese Sequenzen sind klein im Vergleich zu den Proteinen, die Sie ändern und haben in der Regel einen minimalen Einfluss auf die Proteinfunktion, obwohl sie manchmal Proteinstabilität und/oder Enzym Kinetik7,8zu ändern. Histidin-Tags auf die N - und C-Termini des gleichen Proteins können unterschiedliche Auswirkungen haben schwer vorherzusagen, ohne zu wissen, die Struktur des Proteins in Frage stellen. Histidin-Tags sind in der Regel beim Klonen eines rekombinanten Proteins durch die Gestaltung von Grundierungen, die sechs Histidin Rückstände entweder sofort kodieren 3', die ATG-Start-Codon oder sofort 5' auf das Stopp-Codon der offenen Leserahmen eingearbeitet. Nach Verstärkung ist das Hexahistidine-haltigen gen in einen Vektor unter der Kontrolle eines induzierbaren Promotors ligiert und zum Ausdruck gebracht, in der Regel in einem Laborstamm von E. Coli. Die Rekombination Protein kann dann auf eine Affinität Harz mit immobilisierten zweiwertigen kationen (in der Regel Nickel oder Kobalt)9isoliert werden. Kontaminierende native Metall-bindende Proteine kann durch Titration mit Imidazol, entfernt werden die gebundenen Protein2kompetitiv verdrängt. Zu guter Letzt ist das Zielprotein aus der Spalte mit höheren Konzentrationen von Imidazol eluiert. Gibt es mehrere kommerzielle Quellen immobilisierten Metall kation Harze, und die Hersteller geben Empfehlungen für die Pufferbedingungen und Imidazol-Konzentrationen. Nach der Elution kann Protein von Sodium Dodecyl Sulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) analysiert, dialysiert oder sofort in funktionellen Tests verwendet.
Es gibt mehrere Methoden, um die Kinase-Aktivität durch die Kopplung von ATP-Phosphat-Bindung-Hydrolyse zu einer zweiten Reaktion, die veröffentlicht oder reizt ein Fluorophor oder Chemilumineszenz erzeugt mittelbar zu überwachen, aber diese Reaktionen haben mehrere bewegliche Teile und kann logistisch anspruchsvollen10. Der direkteste Weg, insbesondere Phosphor-Transfer Aktivität zu messen ist, direkt die Übertragung von einem radioaktiven Phosphatgruppe von einem handelsüblichen γ -32-P überwachen NTP Vorstufe zu einem Substrat nicht radioaktiv-11 , 12 , 13. Mischungen von radioaktiven Substrate und Produkte getrennt und durch Dünnschichtchromatographie (TLC) quantifiziert werden können. TLC nutzt die differenzielle Mobilität von gelösten Stoffen in einem bestimmten Lösungsmittel dadurch, dass das Lösungsmittel (flüssige Phase) durch Kapillarwirkung über eine Oberfläche (feste Phase) migrieren, auf denen eine Mischung aus gelösten Stoffen adsorbierten14gewesen ist. Solute, die klein sind und/oder günstige Wechselwirkungen mit der festen Phase fehlt migrieren Entfernungen von ihrem ursprünglichen Speicherort als gelöste Stoffe mit höherer Molekulargewichte oder große Affinitäten für die solide. Für die Prüfung der Phosphor-Transfer erhöhen Phosphat Moieties das Molekulargewicht von Molekülen, die sie hinzugefügt werden, die und fügen Sie negativen Ionischen Ladung bei neutralen oder sauren pH11,12,14. Dies verringert ihre Mobilität auf einer Grundfläche wie PEI-Zellulose. Bei der Entwicklung im sauren Kalium-Phosphat-Puffer können Mischungen von Mono - di-, Tri-, Tetra- und Pentaphosphate Arten leicht auf PEI-Zellulose, so dass die Quantifizierung der einzelnen Arten (Abbildung 2, Abbildung 3) getrennt werden. Solche Tests können mit Zelle Lysates, enthält das Enzym von Interesse durchgeführt werden, aber dies beinhaltet das Potential für die Tätigkeit der anderen Kinasen und Phosphatasen allgemeine ATPasen, Substrat und/oder Produkt zum Abbau. Für eine quantitative in-vitro- Bewertung der Enzym-Aktivität ist es notwendig, das Enzym von Interesse zu reinigen.
Guanosin Tetraphosphate (PpGpp) und Guanosin Pentaphosphate (PppGpp) sind Ribonukleotidreduktase Signalisierung Moleküle gebildet durch die Übertragung von einem Pyrophosphat Gruppe aus einem Vorläufer von Adenosintriphosphat (ATP), bzw. ein Guanosin-diphosphat (BIP) oder Guanosin Tetraphosphate (GTP) Substrat15. Diese einzelnen Ribonukleotidreduktase Signale, zusammen bekannt als (p) PpGpp, vermitteln eine Zelle-weite Reaktion auf Umweltstress, bekannt als die strenge Reaktion in unterschiedlichen Bakterienarten15,16. Konservierte Zweiklassen-Enzyme katalysieren die Bildung von (p) PpGpp15,17 Rel/Spo Homolog (RSH) Enzyme sind "lang" bifunktionelle (p) PpGpp Synthestase/Hydrolasen benannt nach ihrer Ähnlichkeit zu den RelA und SpoT (p) PpGpp metabolische Enzyme aus Escherichia coli enthalten Synthestase, Hydrolase und regulatorischen Domains, während kleine Alarmone Synthestase (SAS) Enzyme kurze monofunktionalen Synthetasen fand ausschließlich in Gram-positiven Bakterien-15, sind 17 , 18. das Spore-bildenden Gram-positiven Bakterium Clostridium Difficile kodiert vermeintliche RSH und SAS Gene19. Hier präsentieren wir Ihnen erste Aktivität-Assays, die bestätigen, dass die C. Difficile RSH Enzym ist eine katalytisch aktiven (p) PpGpp Synthestase.
Protokoll
(1) induzierbaren Überexpression eines Proteins Histidin-markierten
- Rsh von C. Difficile R20291 genomic DNA zu verstärken.
- Verwenden Sie eine High Fidelity Polymerase und folgen Sie den Anweisungen des Herstellers.
- C. Difficile Rsh mit Primer zu verstärken
Rsh_F (CAGGTACCGGTTATATGCATGATAAAGAATTACAAG) und
Rsh_R (CCCTGCAGCTAATGGTGATGGTGATGGTGATTTGTCATTCTATAAATAC), die einen C-terminale Hexahistidine Tag einführt.
Hinweis: Die KpnI und PstI geschnitten Standorte in die Primer-Sequenzen sind fett formatiert. - Den pMMBneo Vektor zu verdauen und das Rsh-his6 PCR-Produkt mit House und KpnI Einschränkung schneiden Websites bei 37 ° C für 45 Minuten.
- Reinigen der linearisierten Vektor und der PCR-Fragmente über Agarose-Gelelektrophorese und anschliessende Reinigung mit den DNA-Gel Extraction Kit.
- Messen Sie die 280 nm Absorption (ein280) des Vektors und die amplifizierten PCR-Produkt. Verwenden Sie die Gleichung
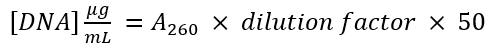 zur Bestimmung der DNA-Konzentration von jedem DNA-Fragment.
zur Bestimmung der DNA-Konzentration von jedem DNA-Fragment.
- Verbinden Sie Rsh in pMMBneo für Ausdruck.
- Kombinieren Sie 25 ng der verdauten PMMBneo Vektor, 125 ng von Rsh-his6 Genprodukt, 2 μl 10-fach Ligase Puffer und 1 μl DNA Ligase. Verwenden Sie die Nuklease freies Wasser um das Gesamtvolumen auf 20 μl anzupassen. Bei 16 ° C 16 h inkubieren.
Hinweis: Die Wirksamkeit der Ligatur Fragmentgröße abhängig und muss je nach Hersteller Protokolle eingestellt werden. - E. Coli DH5-α oder eine andere RecA - Plasmid Wartung Belastung verwandeln Sie der aufgespaltenen Vektor-Produkt. Wählen Sie für die transformierten Zellen auf LB-Platten mit 100 μg/mL Kanamycin und 16 – 24 h bei 37 ° c inkubieren
- Wählen Sie vier Kolonien von der Transformation-Platte (s) und Streifen Sie jeweils auf eine frische Platte mit 100 μg/mL Kanamycin für DNA-Isolierung. Die neuen Platten bei 37 ° C für 16 – 24 h inkubieren und wählen Sie eine für die nachfolgende Proteinexpression isolieren.
- Bestätigen die erfolgreiche Unterbindung der Codierung Region intakt Rsh -Protein, wählen Sie eine Kolonie des gewählten isolieren in 20 μL des Wassers, bei 95 ° C für 10 min erhitzen, und verwenden Sie als Vorlage für eine bestätigende PCR unter Verwendung des gleichen Primers verwendet, um das Gen zu verstärken.
- Kombinieren Sie 25 ng der verdauten PMMBneo Vektor, 125 ng von Rsh-his6 Genprodukt, 2 μl 10-fach Ligase Puffer und 1 μl DNA Ligase. Verwenden Sie die Nuklease freies Wasser um das Gesamtvolumen auf 20 μl anzupassen. Bei 16 ° C 16 h inkubieren.
- Die verifizierte Plasmid gemäß standard Transformation Protokolle für E. Coli Bakterien Plasmid Transformation in E. Coli BL21 System für high-Yield-Protein-Produktion zu verwandeln.
- Ausdrückliche RSH-His6 in E. Coli.
- Wählen Sie eine einzelne Kolonie von E. Coli BL21 verwandelt mit pMMBneo::Rsh und 2 mL LB-Medium mit 50 μg/mL Kanamycin zu impfen. Bei 37 ° C inkubieren 12 – 16 Uhr unter 250 u/min schütteln.
- 500 mL LB-Medium mit 50 μg/mL Kanamycin mit 0,5 mL der Nacht Kultur für Zellwachstum und Protein-Expression zu impfen.
- Inkubieren Sie die Ausdruck Kultur bei 37 ° C und 250 u/min in einem Inkubator-Shaker, bis die Zelldichte eine OD600 0,167 bei 37 ° C erreicht.
- Reduzieren Sie der Inkubator-Temperatur auf 30 ° C und warten Sie 30 min für die Kultur Temperatur sinkt.
- Induzieren Sie RSH Ausdruck durch Zugabe von Isopropyl-β-D-Thiogalactoside (IPTG), eine Endkonzentration von 0,5 mM. Ermöglichen Sie die Induktion bei 30 ° C, bei 250 u/min schütteln über Nacht (für 16 – 18 h) stattfinden.
- Pellet-durch Zentrifugation bei 3.080 x g für 30 min bei 4 ° C.
Hinweis: Das Pellet kann über Nacht bei-20 ° C zur Reinigung Zielprotein am Folgetag ohne spürbaren Verlust von Protein Ertrag oder enzymatische Aktivität gespeichert werden.
(2) Proteinreinigung durch Affinitätschromatographie Nickel
Hinweis: Fahren Sie direkt mit Protein Reinigungsschritte nach Klärung der Zelle lysate unten bereitgestellt. Speichern von geklärt lysate bei 4 ° C über Nacht für nachfolgende Proteinreinigung den Eiweiß-Ertrag reduziert.
- Eiweiß mit 1 mL der Nickel-Nitriloacetic Säure (Ni-NTA) Harz auf eine Schwerkraft-Spalte zu reinigen.
- Am Vortag verwenden, die Spalte über Nacht bei 4 ° C mit 2 mL Gleichgewichtherstellung Puffer (10 mM Tris-HCl pH 7,79, 300 mM NaCl, 50 mM NaH2PO40,5 mg/mL Lysozym, 5 mM MgCl2, 10 mM Imidazol, 0,25 mM DVB-t, 5 mM Phenylmethane Sulfonyl equilibrate Fluorid (PMSF), 10 % Glycerin).
- Am nächsten Tag bringen die Spalte von 4 ° C bis RT vor dem Laden der geklärte lysate und lassen Sie es stehen für ~ 2 – 3 h.
Hinweis: Die Spalte zum thermischen Gleichgewicht zu bringen ist entscheidend für die Luftblasen bilden innerhalb der Spalte zu vermeiden. - Aufzuwirbeln Sie das Pellet in Schritt 1.3.6 in der Lyse-Puffer (10 mM Tris-HCl pH 7,8, 300 mM NaCl, 5 mM MgCl2, 50 mM NaH2PO4, 10 % Glycerin, 0,5 mg/mL Lysozym, 10 mM Imidazol, 0,25 mM DVB-t und 5 mM PMSF) erhaltenen.
- Beschallen Sie Zellen auf Eis für 8 x 10 s Intervalle, Pause 30 s zwischen den Pulsen.
- Durch Zentrifugation bei 3.080 x g für 30 min bei 4 ° C mit einem Microcentrifuge der lysate zu klären.
- Lysate mit gleiche Volumina der Lyse Puffer vorbereiten, dann gelten die vorbereitet lysate auf die Spalte zu klären und die Durchströmung zu sammeln.
- Geklärte lysate Durchflussverfahren in die Spalte anwenden und die sekundäre durchströmten sammeln.
- Waschen Sie die Spalte mit Waschpuffer 1 (10 mM Tris-HCl (pH 7,79), 300 mM NaCl, 5 mM MgCl2, 50 mM NaH2PO4, 30 mM Imidazol, 10 % Glycerin). Die Flow-through zu sammeln.
Hinweis: Aufnahme von 5 mM MgCl2 in den Wasch- und Elution Puffern ist wichtig für die enzymatische Aktivität des gereinigten Proteins. - Waschen Sie die Spalte mit Waschpuffer 2 (10 mM Tris-HCl (pH 7,79), 300 mM NaCl, 5 mM MgCl2, 50 mM NaH2PO4 und 50 mM Imidazol). Die Flow-through zu sammeln.
- 2 mL Elution Puffer (10 mM Tris-HCl (pH 7,79), 300 mM NaCl, 50 mM NaH2PO4, 10 % Glycerin und 75 mM Imidazol) gelten. Sammeln Sie durchströmten in zwei Fraktionen von 1 mL.
Hinweis: Die Spalte nach diesem Schritt kann in Gleichgewichtherstellung Puffer bei 4 ° C gespeichert werden, wenn das gleiche Protein wird in der nächsten Reinigung Test gereinigt werden. Die Spalte kann bis zu 3 mal verwendet werden, bei sachgemäßer Lagerung.
- Bewerten Sie Spalte Brüche für Reinheit durch SDS-PAGE.
- Proteinreinigung qualitativ zu bewerten, laufen Sie 20 μl-Aliquots aller Spalte Fraktionen auf eine 4 % / 10 % Polyacrylamid-Gel für 60 min bei 170 V.
- Fleck das Gel mit 0,1 % Coomassie Blau bei Raumtemperatur für 5 h, Schaukeln sanft auf einem Benchtop-Rocker.
- Das Gel in 40 % Methanol, 10 % Eisessig über Nacht bei Raumtemperatur, Schaukeln auf einem Benchtop-Rocker destain.
Hinweis: Eine repräsentative Gel ist in Abbildung 1dargestellt.
- Dialyse eluieren Bruch 2 über Nacht bei 4 ° C.
- Dialyse gegen Dialyse-Puffer (15,7 mM Tris-HCl (pH 7,6), 471,9 mM NaCl, 15,69 mM MgCl2, 1,57 mM DTT, 1,5 mM PMSF und 15,7 % Glycerin) im Verhältnis 200: 1 mit einer 1 mL-Dialyse-Gerät mit einem 20 kDa Molekulargewicht Cut-Off (MWCO).
- Bestimmen Sie die Konzentration der Probe dialysierten Protein durch Messung der Absorption bei 280 nm und die berechneten Molaren Aussterben Koeffizient 82085 M-1cm-1 20verwenden.
- 5 μl-Aliquots der dialysierten Protein Probe in Aliquote bei-80 ° C bis zur Verwendung aufbewahren.
(3) Protein Activity Assay durch Dünnschichtchromatographie
- Bereiten Sie die Dünnschicht-Chromatographie-Platte.
- Vor der Durchführung der Reaktions, bereiten Polyethyleneimine (PEI)-Zellulose-Platten durch Waschen in entionisiertem Wasser. Legen Sie die Platten in einer Glas-Kammer mit doppelt destilliertem Wasser bis zu einer Tiefe von ~0.5 cm.
- Lassen Sie Wasser auf die Oberseite der Platte zu migrieren.
Hinweis: Die Platten waschen ist nicht unbedingt notwendig, da Platten, ohne dass es verwendet werden jedoch waschen die Übersichtlichkeit der resultierenden Bilder erhöht. Waschen die Platten in zwei senkrechten Richtungen weiter sorgt dafür, dass Verunreinigungen in das Harz in einer Ecke der Platte (Abbildung 2) isoliert werden. - Die Platten aus Glas-Kammer zu bringen und lassen auf ein Benchtop-Rack über Nacht trocknen (12 – 18 h).
- Markieren Sie die Trockenplatten 2,0 cm von einer Kante mit einem weichen Bleistift um anzugeben, wo die Proben für die TLC angewendet wird. Beantragen Sie 2 μL Proben, Proben nicht weniger als 1,0 cm auseinander (Abbildung 2).
Hinweis: Dies ermöglicht klare Trennung zwischen benachbarten Spot, die für Signal-Quantifizierung von entscheidender Bedeutung ist. Solange die Zellulose Harz nicht zerkratzt, kleine ist stören Bleistiftmarkierungen auf der Oberfläche nicht solvent Migration. - Bei der Planung von Experimenten lassen Sie immer eins auf jeder Platte ungenutzt.
Hinweis: Diese liefern eine leere Spur für Probe Quantifizierung (Abbildung 2). Eine 20 cm TLC Platte haben Zimmer für 19 Spots.
- Enzym-Aktivität-assay
- Bereiten Sie eine 5-fach-Puffer-Mischung mit 50 mM Tris-HCl (pH 7,5), 25 mM Ammoniumacetat, 10 mM KCl, 1 mM DTT und 0,6 mM ATP.
Hinweis: Diese Mischung kann in großen Mengen zubereitet und in 10 μl-Aliquots für eine spätere Verwendung eingefroren. Unterliegen Sie Mischung nicht mehrere Gefrier-Tau-Zyklen. - Bereiten Sie individuelle Reaktionen mit 3 μM RSH, 1 X Puffer Mischung, 0,6 mM BIP, 1,2 mM MgCl2 vor. Fügen Sie 1,0 μCi γ -32P-ATP pro 10 μL der Reaktion und verwenden Sie Nuklease freies Wasser zu, um die Reaktion auf eine gewünschte Lautstärke bringen. Die RSH nach Hinzufügen der anderen Komponenten gemischt worden sind, wie die Zugabe von RSH der Nukleotid-haltige Mischung den Enzymaktivität Test initiiert.
Hinweis: Letzte Reaktionsvolumen hängt von der Anzahl der Zeitpunkte abgetastet. Um 2 μL/Timepoint probieren, montieren Sie 10 μL Reaktionsgemisch für jede 4 Zeitpunkte. - Um für die ATP-Hydrolyse von kontaminierenden Nuklease Aktivität zu kontrollieren, montieren Sie 10 μL Reaktion, kein Eiweiß enthalten und inkubieren Sie es parallel. Vor Ort 2 μL Proben bei t = 0 und am Ende des Experiments um sicherzustellen, dass ATP nicht bei fehlender Protein hydrolysiert wurde.
- Sofort bei Zusatz von RSH, entfernen 2 μL und Stelle es auf die beschrifteten PEI-Zellulose-Platte als die t = 0 min Probe.
- Inkubieren Sie die Reaktion bei 37 ° C, 2 μl-Aliquots an gewünschten Zeitpunkten zu entfernen.
Hinweis: Enzymatische Aktivität wird aufhören, wenn die Probe auf die Zellulose-Platte adsorbiert wird. Warten Sie 10-30 min nachdem der letzte Fleck Platte vor Entwicklung damit komplette Adsorption und Probentrocknung hinzugefügt wurde.
- Bereiten Sie eine 5-fach-Puffer-Mischung mit 50 mM Tris-HCl (pH 7,5), 25 mM Ammoniumacetat, 10 mM KCl, 1 mM DTT und 0,6 mM ATP.
- Dünnschichtchromatographie
- Füllen Sie die Chromatographie-Kammer mit 1,5 M 1,5 M KH2PO4 (pH 3,64) bis zu einer Tiefe von 0,5 cm.
Hinweis: Die Lautstärke benötigt die Abmessungen des Panzers Chromatographie abhängen. Glasbehälter mit einem ebenen Boden, der breit genug, um die Einfügung der TLC Platte ohne sich bücken zu ermöglichen ist kann als eine Entwicklerdose mit dem Zusatz von einer Abdeckung verwendet werden. Die TLC Platte kürzbar in schmaler Streifen mit eine saubere Rasierklingen für die Entwicklung in ein Becherglas in Kunststoff-Folie abgedeckt. - Tauchen Sie den unteren Rand der Platte in Lösungsmittel. Lassen Sie das Lösungsmittel, auf der Oberseite der Platte (~ 90 min) zu migrieren.
Hinweis: Während solvent Migration am oberen Rand der Platte angehalten wird und Proben werden nicht verloren gehen oder laufen zusammen während einer längeren eintauchen, sollten Platten nicht in Lösungsmittel über Nacht gelassen werden. Immersionen länger als 4 h können dazu führen, dass das Harz zu trennen von der Platte sichern, Verlust des Signals führen. - Entfernen Sie die Platte aus der Chromatographie-Tank und legen Sie es auf einem Benchtop Wäscheständer.
- Lassen Sie die Platte an der Luft trocknen über Nacht.
Hinweis: Trocknung kann durch den Einsatz von Fön beschleunigt werden. Trockenheit kann beurteilt werden, durch die Farbe der Harz, das dunkler wird, wenn Sie nass und Rückkehr zur Farbe eines ungenutzten Platte, wenn es vollständig getrocknet. - Nachdem die Platte trocken ist, wickeln Sie die Platte in Kunststofffolie zu vermeiden Übertragung von radioaktivem Material auf die Bildgebung Kassette und analysieren durch Autoradiographie (Abbildung 3).
- Füllen Sie die Chromatographie-Kammer mit 1,5 M 1,5 M KH2PO4 (pH 3,64) bis zu einer Tiefe von 0,5 cm.
- Datenanalyse
- Setzen Sie die PEI-Zellulose-Platte mit getrennten Reaktionen auf eine Phosphorimager Kassette für 4 h bei Raumtemperatur.
Hinweis: Dies ist ausreichende Belichtung ein sehr klares Bild über die angegebenen Konzentrationen von frischen γ -32P-ATP liefern. Wenn geringere Mengen an möglicher Substrat verwendet werden, kann die Belichtungszeit, 12-16 h erhöht werden. - Bild der Kassette auf eine Phosphorimager.
- Mit imaging-Software mit einer grafischen Benutzeroberfläche, Regionen of Interest (ROI) durch Auswahl Rechteck zeichnen und mit der Maus, um rechteckige ROIs zu ziehen, um eine ganze Spur und ATP und PpGpp Flecken innerhalb dieser Lane (Abbildung 3 enthaltenen, zeichnen ).
- Verwenden Sie die auswählen, kopieren und einfügen (oder entsprechende Befehle anhand der imaging-Software verwendet wird), identische ROIs in den anderen Spuren um sicherzustellen, dass die ROIs Signal innerhalb identische Bereiche in jeder Bahn messen zu ziehen. Zählen Sie ROIs aus einer ungenutzten Fahrspur als Leerzeichen verwendet werden.
- Mit Hilfe der analysieren | Werkzeuge | ROI-Manager | Fügen Sie Befehle der imaging-Software, wählen Sie alle ROIs auf der PEI-Zellulose-Platte gezogen.
- Mit Hilfe der analysieren | Legen Sie Messungen | Maßnahme Befehle, die Signalintensität innerhalb jedes ROI zu quantifizieren und export der Messwerte als Tabelle (Abbildung 3). Subtrahieren Sie leere ROI-Werte von experimentellen Signale.
- Berechnen Sie, welcher Prozentsatz des ausgeblendeten Signals innerhalb jeder Bahn auf ATP und PpGpp anhand der Formeln zurückzuführen ist

Hinweis: ROIs können erstellt werden und quantitated mit kommerzieller Software kompatibel mit dem Phosphorimager oder frei verfügbare ImageJ-Software (National Institutes of Health).
- Setzen Sie die PEI-Zellulose-Platte mit getrennten Reaktionen auf eine Phosphorimager Kassette für 4 h bei Raumtemperatur.
Ergebnisse
Wir präsentieren eine Methode für die Affinitätsreinigung ein (p) PpGpp Synthestase von Clostridium Difficile und die Beurteilung der seine enzymatische Aktivität. Abbildung 1 veranschaulicht die Proteinreinigung durch Metall Affinitätschromatographie erreicht. Die zweite Elution (E2)-Fraktion aus diese Reinigung war dialysiert und für die enzymatische Aktivität Assay verwendet. Abbildung 2 beschreibt die notwendi...
Diskussion
Hier berichten wir die Reinigung des His-Tags RSH von C. Difficile und stellen eine Methode zur Quantifizierung der Aktivität mit radioaktiven Dünnschichtchromatographie. Diese Methode wurde zuvor verwendet, um die Aktivität der Diguanylate Cyclase Enzyme von C. Difficile, sowie (p) PpGpp Synthestase, Nukleotid-Cyclase, Kinase und Phosphodiesterase Enzyme aus anderen Organismen11,12 bewerten ,13,...
Offenlegungen
Die Autoren erklären keine finanziellen Interessenkonflikte oder andere Interessenkonflikte.
Danksagungen
Diese Arbeit wurde von NIAID 1K22AI118929-01 finanziert. EBP wurde unterstützt durch einen Sommer Research Fellowship Programm Zuschuss von der Office of Research bei Old Dominion University in Norfolk, Virginia, USA.
Materialien
| Name | Company | Catalog Number | Comments |
| Inducible overexpression of a histidine-tagged protein | |||
| Phusion polymerase | New England Biolabs (NEB) | M0530L | |
| QIAEX II DNA Gel Extraction Kit | Qiagen | 20021 | |
| KpnI restriction enzyme | NEB | R0142S | |
| PstI restriction enzyme | NEB | R0140S | |
| T4 DNA ligase | NEB | M0202 | |
| NEB® 5-alpha Competent E. coli (High Efficiency) | NEB | C2987I | |
| BL21 (DE3) Competent E. coli | NEB | C2527I | |
| IPTG | Sigma-Aldrich | 10724815001 | |
| JXN-26 centrifuge with JLA 10.500 rotor | Beckman Coulter Avanti | - | |
| Microcentrifuge with D3024/D3024R rotor | Scilogex | - | |
| MaxQ SHKE6000 Incubator | Thermo Scientific | - | |
| Ultrasonic processor | Sonics | VC-750 | |
| Protein purification by nickel affinity chromatography | |||
| Ni-NTA resin | G Biosciences | 786-940/941 | |
| Pierce Disposable Gravity columns, 10 mL | Thermo Scientific | 29924 | |
| 1 mL Spectra/ Por float-A-lyzer G2 dialysis device (MWCO: 20-kD) | Spectrum | G235033 | |
| Mini-Protean Electrophoresis Cell | BioRad | 1658004 | |
| Protein activity assay by thin layer chromatography | |||
| Thin layer chromatograph (TLC) development tank | General Glass Blowing Company | 80-3 | |
| Polyethylenimine (PEI)-cellulose plates (20 cm x 20 cm, 100 μm thickness) with polyester support | Sigma-Aldrich | Z122882-25EA | |
| ATP, [γ-32P]- 3000 Ci/mmol 10mCi/ml lead, 100 μCi | Perkin Elmer | NEG002A | |
| Adenosine 5’-triphosphate (ATP) 100 mM | Bio Basic Canada | AB0311 | |
| Guanosine-5’-diphosphate disodium salt (GDP) | Alfa Aesar | AAJ61646MC/E | |
| Storage phosphor screen | GE Healthcare Life Sciences | BAS-IP TR 2040 E Tritium Screen | |
| Storm 860 phosphorimager | GE Healthcare Life Sciences | - |
Referenzen
- Cheek, S., Ginalski, K., Zhang, H., Grishin, N. V. A comprehensive update of the sequence and structure classification of kinases. BMC Structural Biology. 5 (1), 6 (2005).
- Porath, J. Immobilized metal ion affinity chromatography. Protein Expression and Purification. 3 (4), 263-281 (1992).
- Arnau, J., Lauritzen, C., Pedersen, J. Cloning strategy, production and purification of proteins with exopeptidase-cleavable His-tags. Nature Protocols. 1 (5), 2326-2333 (2006).
- Porath, J., Carlsson, J., Olsson, I., Belfrage, G. Metal chelate affinity chromatography, a new approach to protein fractionation. Nature. 258 (5536), 598-599 (1975).
- Smith, M. C., Furman, T. C., Ingolia, T. D., Pidgeon, C. Chelating peptide-immobilized metal ion affinity chromatography. A new concept in affinity chromatography for recombinant proteins. Journal of Biological Chemistry. 263 (15), 7211-7215 (1988).
- Graslund, S., et al. Protein production and purification. Nature Methods. 5 (2), 135-146 (2008).
- Booth, W. T., et al. Impact of an N-terminal Polyhistidine Tag on Protein Thermal Stability. ACS Omega. 3 (1), 760-768 (2018).
- Thielges, M. C., Chung, J. K., Axup, J. Y., Fayer, M. D. Influence of histidine tag attachment on picosecond protein dynamics. Biochemistry. 50 (25), 5799-5805 (2011).
- Chaga, G. S. Twenty-five years of immobilized metal ion affinity chromatography: past, present and future. Journal of Biochemical and Biophysical Methods. 49 (1-3), 313-334 (2001).
- Ma, H., Deacon, S., Horiuchi, K. The challenge of selecting protein kinase assays for lead discovery optimization. Expert opinion on drug discovery. 3 (6), 607-621 (2008).
- Tamayo, R., Tischler, A. D., Camilli, A. The EAL domain protein VieA is a cyclic diguanylate phosphodiesterase. Journal of Biological Chemistry. 280 (39), 33324-33330 (2005).
- Purcell, E. B., McKee, R. W., McBride, S. M., Waters, C. M., Tamayo, R. Cyclic Diguanylate Inversely Regulates Motility and Aggregation in Clostridium difficile. Journal of Bacteriology. 194 (13), 3307-3316 (2012).
- Purcell, E. B., Tamayo, R. Identification and characterization of cyclic nucleotide phosphodiesterases. Methods in Molecular Biology. 1016, 235-243 (2013).
- Ross, P., et al. Control of cellulose synthesis Acetobacter xylinum. A unique guanyl oligonucleotide is the immediate activator of the cellulose synthase. Carbohydrate Research. 149 (1), 101-117 (1986).
- Potrykus, K., Cashel, M. (p)ppGpp: still magical?. Annual Review of Microbiology. 62, 35-51 (2008).
- Boutte, C. C., Crosson, S. Bacterial lifestyle shapes stringent response activation. Trends in Microbiology. 21 (4), 174-180 (2013).
- Nanamiya, H., et al. Identification and functional analysis of novel (p)ppGpp synthetase genes in Bacillus subtilis. Molecular Microbiology. 67 (2), 291-304 (2008).
- Gaca, A. O., et al. Basal Levels of (p)ppGpp in Enterococcus faecalis: the Magic beyond the Stringent Response. mBio. 4 (5), (2013).
- Sebaihia, M., et al. The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome. Nature Genetics. 38 (7), 779-786 (2006).
- Gasteiger, E., et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis. Nucleic Acids Research. 31 (13), 3784-3788 (2003).
- Purcell, E. B., et al. A nutrient-regulated cyclic diguanylate phosphodiesterase controls Clostridium difficile biofilm and toxin production during stationary phase. Infection and Immunity. , (2017).
- Mechold, U., Murphy, H., Brown, L., Cashel, M. Intramolecular Regulation of the Opposing (p)ppGpp Catalytic Activities of RelSeq, the Rel/Spo Enzyme from Streptococcus equisimilis. Journal of Bacteriology. 184 (11), 2878-2888 (2002).
- Dalebroux, Z. D., Svensson, S. L., Gaynor, E. C., Swanson, M. S. ppGpp Conjures Bacterial Virulence. Microbiology and Molecular Biology Reviews. 74 (2), 171-199 (2010).
- Bartlett, J. G. Clostridium difficile: progress and challenges. Annals of the New York Academy of Sciences. 1213, 62-69 (2010).
- . . US Department of Health and Human Services. , (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten