Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une Purification et analyse d’activité In Vitro pour un (p) ppGpp synthétase de Clostridium difficile
Dans cet article
Résumé
Nous décrivons ici une méthode pour la purification des enzymes pyrophosphokinase histidine-étiquetées et utilisant la chromatographie sur couche mince des substrats radiomarqués et produits pour le dosage de l' activité enzymatique in vitro. Le dosage de l’activité enzymatique est largement applicable aux kinase, cyclase de nucléotides ou réaction de transfert de phosphore dont le mécanisme comprend l’hydrolyse de nucléotides triphosphate.
Résumé
Enzymes kinases et pyrophosphokinase transférer le phosphate gamma ou la portion de pyrophosphate de bêta-gamma de précurseurs de nucléotides triphosphate de substrats pour créer des produits phosphorylés. L’utilisation de γ -32- P étiqueté précurseurs NTP permet une surveillance simultanée de formation utilisation et produit de substrat par radiographie. Chromatographie sur couche mince (TLC) sur des plaques de cellulose permet une séparation rapide et sensible quantification du substrat et produit. Nous présentons une méthode pour l’utilisation de la chromatographie en couche mince pour doser l’activité pyrophosphokinase d’un (p) ppGpp synthétase purifiée. Cette méthode n’a encore été utilisée pour caractériser l’activité des synthétases cycliques de nucléotide et dinucléotide et est largement adaptée pour caractériser l’activité d’une enzyme qui hydrolyse d’une liaison de triphosphate de nucléotides ou transfère un terminal phosphate d’un donneur de phosphate à une autre molécule.
Introduction
Les enzymes kinases et pyrophosphokinase (ou diphospho-kinase) transfert en phosphates de précurseurs de nucléotides triphosphates (NTP) aux molécules de substrat. Les substrats peuvent inclure les autres nucléotides, des acides aminés ou protéines, glucides et lipides1. Analyses bioinformatiques peuvent parfois prévoir une enzyme substrat apparenté ou substrats fondées sur la similitude aux enzymes caractérisées, mais il faut toujours une validation expérimentale. De même, l’affinité de l’enzyme pour sa fonction et le taux auquel elle catalyse la réaction de transfert de phosphore et les effets des co-facteurs, inhibiteurs, ou autres effecteurs de l’enzyme doivent être déterminées expérimentalement. Afin d’éviter l’appauvrissement du précurseur ATP des autres enzymes ATP consommateurs présents dans le cytoplasme bactérien, tests d’activité quantitative nécessitent de protéine purifiée.
Purification des protéines par chromatographie d’affinité métallique a été couvert abondamment dans la littérature2,3. Les balises de histidine consistant en six résidus histidine consécutives annexées à l’extrémité N - ou C-terminale d’une protéine recombinante permettent rapide purification par affinité métallique chromatographie4,5,6. Ces séquences sont petites par rapport aux protéines ils modifier et ont généralement un effet minime sur la fonction des protéines, bien qu’ils peuvent parfois modifier stabilité des protéines et/ou enzyme cinétique7,8. Histidine balises aux N - et C-terminus de la protéine même peuvent avoir des effets différents, qui sont difficiles à prédire sans connaître la structure de la protéine en question. Tags de l’histidine sont généralement incorporés pendant le clonage d’une protéine recombinante en concevant des amorces qui codent pour six résidus histidine, soit immédiatement 3' pour le codon de début ATG ou juste 5' pour le codon d’arrêt de la trame de lecture ouverte. Après amplification, le gène de la protéine contenant est ligué dans un vecteur sous le contrôle d’un promoteur inductible et exprimé, en général chez une souche de laboratoire d’Escherichia coli. Les protéines de recombinaison peuvent ensuite être isolé sur une résine contenant des cations divalents immobilisé (typiquement de nickel ou de cobalt)9. Contamination des protéines liant le métal natifs peut être enlevé par titrage avec imidazole, qui déplace compétitif lié protein2. Enfin, la protéine-cible est éluée de la colonne avec des concentrations plus élevées de l’imidazole. Il y a plusieurs sources commerciales pour les résines de cations métalliques immobilisés et les constructeurs fournissent des recommandations pour les conditions de la mémoire tampon et les concentrations en imidazol. Après élution, protéine peut être analysé par électrophorèse sur gel sodium dodecyl sulfate polyacrylamide (SDS-PAGE), dialysée ou utilisé immédiatement pour des tests fonctionnels.
Il existe plusieurs méthodes pour contrôler indirectement l’activité kinase en couplant l’hydrolyse des liaisons phosphate de l’ATP à une seconde réaction qui libère ou excite un fluorophore ou génère la chimiluminescence, mais ces réactions ont les pièces mobiles multiples et peuvent être logistiquement difficile10. La façon la plus simple pour mesurer plus précisément l’activité de phosphore-transfert doit directement surveiller le transfert d’un groupement phosphate radiomarqué d’un disponible dans le commerce γ -32P - précurseur NTP à un substrat non radioactif11 , 12 , 13. mélanges de substrats radiomarquées et produits peuvent être séparés et quantifiés par chromatographie sur couche mince (CCM). TLC utilise la mobilité différentielle des solutés dans un solvant donné en permettant le solvant (phase liquide) migrer par capillarité à travers une surface (phase solide) sur lequel un mélange de solutés a été adsorbé14. Les solutés qui sont de petite taille ou les interactions favorables avec la phase solide vont migrer plus longues distances de leur emplacement initial que les solutés avec un poids moléculaire plus élevé ou de grande affinité pour le solide. Pour l’examen du transfert de phosphore, groupements phosphate augmentent le poids moléculaire des molécules, ils sont ajoutés à et ajouter la charge ionique négative à pH neutre ou acide11,12,14. Cela diminue leur mobilité sur une surface de base tels que la PEI-cellulose. Quand développé dans un tampon phosphate acide de potassium, mélanges de mono-, di-, tri-, tétra- et pentaphosphate espèces peuvent être aisément séparées sur PEI-cellulose, ce qui permet la quantification de chaque espèce (Figure 2, Figure 3). Ces tests peuvent être effectuées à l’aide de lysats de cellules contenant l’enzyme d’intérêt, mais cela inclut la possibilité pour l’activité d’autres kinases et phosphatases ATPases générales pour épuiser le substrat ou le produit. Pour une évaluation quantitative in vitro de l’activité enzymatique, il faut purifier l’enzyme d’intérêt.
Guanosine tétraphosphate (ppGpp) et la guanosine pentaphosphate (pppGpp) sont des ribonucléotides signalisation des molécules formées par le transfert d’un pyrophosphate de groupe d’un précurseur de l’adénosine triphosphate (ATP), respectivement, un diphosphate de guanosine (PIB) ou guanosine tétraphosphate (GTP) substrat15. Ces signaux de ribonucléotide unique, collectivement connus comme (p) ppGpp, véhiculent une réponse à l’échelle de la cellule à un stress environnemental connu comme la réponse stricte dans diverses espèces bactériennes15,16. Deux classes conservées des enzymes catalysent la formation de (p) ppGpp15,17 Rel/Spo enzymes homologue (RSH) sont « long » bifonctionnel (p) ppGpp synthétase/hydrolases nommé pour leur ressemblance avec la RelA et SpoT (p) ppGpp métabolique enzymes d’Escherichia coli qui contiennent synthétase, hydrolase et domaines réglementaires, tandis que les petites alarmone synthétase (SAS) enzymes sont courts monofonctionnel synthétases trouvés exclusivement à Gram positif bactéries15, 17 , 18. la bactérie Gram-positive sporulée Clostridium difficile encode putatif RSH et SAS gènes19. Nous présentons ici les tests d’activité initial qui confirment que l’enzyme RSH de c. difficile est un catalytiquement actifs (p) ppGpp synthétase.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. inductible surexpression d’une protéine Histidine-étiquetée
- Amplifier rsh de c. difficile R20291 ADN génomique.
- Utilisez une polymérase de haute fidélité et suivez les instructions du fabricant.
- Amplifier rsh de c. difficile en utilisant des amorces
rsh_F (CAGGTACCGGTTATATGCATGATAAAGAATTACAAG) et
rsh_R (CCCTGCAGCTAATGGTGATGGTGATGGTGATTTGTCATTCTATAAATAC), qui introduit une balise de protéine C-terminal.
NOTE : Le KpnI et PstI couper des sites dans les séquences des amorces sont en caractères gras. - Digérer le vecteur pMMBneo et le produit PCR rsh-his6 avec Angélique et KpnI restriction coupée sites à 37 ° C pendant 45 min.
- Purifier le vecteur linéarisé et les fragments PCR par électrophorèse sur gel d’agarose et purification ultérieure avec un kit d’extraction de gel ADN.
- Mesurer l’absorbance de nm (un280) 280 du vecteur et du produit PCR amplifié. Il faut utiliser l’équation
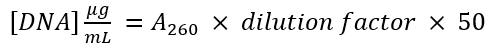 pour déterminer la concentration d’ADN de chaque fragment d’ADN.
pour déterminer la concentration d’ADN de chaque fragment d’ADN.
- Ligaturer rsh dans pMMBneo pour l’expression.
- Mélanger 25 ng de digéré PMMBneo vector, 125 ng de rsh-his6 produit du gène, 2 μL de mémoire tampon ligase 10 x et 1 μL de DNA ligase. Utilisez l’eau libre nucléase pour ajuster le volume total de 20 μL. Incuber à 16 ° C pendant 16 h.
Remarque : L’efficacité de la ligature dépend de la taille du fragment et doit être réglée selon les protocoles du fabricant. - Transformer le produit vectoriel ligaturé en e. coli DH5-α ou d’une autre plasmide recA - entretien-souche. Sélectionner des cellules transformées sur plaques LB avec 100 kanamycine μg/mL et incuber pendant 16 à 24 h à 37 ° C.
- Prélever des quatre colonies de la plaque de transformation (s) et ensemencer chacun sur une assiette fraîche contenant 100 kanamycine μg/mL pour l’isolement de l’ADN. Incuber les plaques à 37 ° C pendant 16 à 24 h de nouveau, puis sélectionnez un isolat pour l’expression de la protéine ultérieures.
- Pour confirmer la ligature réussie de la protéine intacte rsh région codante, pioche une colonie de l’Elu isoler dans 20 μL d’eau, chauffer à 95 ° C pendant 10 min et utiliser comme modèle pour une confirmation PCR en utilisant la même amorce utilisée pour amplifier le gène.
- Mélanger 25 ng de digéré PMMBneo vector, 125 ng de rsh-his6 produit du gène, 2 μL de mémoire tampon ligase 10 x et 1 μL de DNA ligase. Utilisez l’eau libre nucléase pour ajuster le volume total de 20 μL. Incuber à 16 ° C pendant 16 h.
- Transformer le plasmide vérifié conformément aux protocoles standard de transformation transformation, plasmide bactérien Escherichia coli en e. coli BL21 système pour la production de protéines à haut rendement.
- RSH-His6 Express chez e. coli.
- Sélectionnez une seule colonie d’Escherichia coli BL21 transformé avec pMMBneo ::rsh et ensemencer 2 mL de milieu LB avec 50 kanamycine μg/mL. Incuber à 37 ° C 12 – 16 h et en agitant à 250 tr/min.
- Inoculer les 500 mL de milieu LB contenant 50 kanamycine μg/mL avec 0,5 mL de la culture au jour le jour pour la croissance cellulaire et l’expression de la protéine.
- Incuber la culture d’expression à 37 ° C et 250 tr/min dans un shaker incubateur jusqu'à ce que la densité des cellules atteigne une OD600 0,167 à 37 ° C.
- Réduire la température de l’étuve à 30 ° C et attendre 30 min pour la température de la culture de laisser tomber.
- Induire l’expression de RSH en ajoutant l’isopropyl β-D-thiogalactoside (IPTG) à une concentration finale de 0,5 mM. Permettent l’induction se déroulera pendant la nuit (pour les 16-18 h) à 30 ° C, agitation à 250 tr/min.
- Granulés par centrifugation à 3 080 x g pendant 30 min à 4 ° C.
Remarque : Les granulés peuvent être stockés pendant la nuit à-20 ° C pour la purification de protéine cible le lendemain sans perte notable de rendement de protéine ou de l’activité enzymatique.
2. protein Purification par chromatographie d’affinité de Nickel
NOTE : Continuer directement avec les étapes de purification de protéine fournis ci-dessous après clarifiant le lysat cellulaire. Stockage a précisé lysat à 4 ° C durant la nuit pour purification de protéine ultérieure réduit le rendement de protéine.
- Purifier les protéines en utilisant 1 mL de résine acide Nitriloacetic-Nickel (Ni-NTA) sur une colonne de gravité.
- La veille, utiliser, équilibrer la colonne pendant la nuit à 4 ° C avec 2 mL de tampon d’équilibration (10 mM Tris-HCl pH 7.79, 300 mM NaCl, 50 mM NaH2PO4, lysozyme de 0,5 mg/mL, 5 mM MgCl2, imidazole de 10 mM, 0. 25 mM DTT, sulfonyl phenylmethane 5 mM fluorure (PMSF), 10 % de glycérol).
- Le lendemain mettre la colonne de 4 ° C a RT avant le chargement de la clarification lysat et laissez il reposer pendant environ 2 à 3 h.
NOTE : Ce qui porte la colonne à l’équilibre thermique est crucial pour éviter les bulles d’air formant dans la colonne. - Resuspendre le culot obtenu à l’étape 1.3.6 dans le tampon de lyse (10 mM Tris-HCl pH 7.8, 300 mM NaCl, 5 mM MgCl2, 50 mM NaH2PO4, 10 % de glycérol, lysozyme de 0,5 mg/mL, imidazole de 10 mM, 0,25 mM DTT et 5 mM PMSF).
- Ultrasons cellules sur la glace pendant des intervalles de s 8 x 10, faire une pause de 30 s entre les impulsions.
- Clarifier le lysat par centrifugation à 3 080 x g pendant 30 min à 4 ° C à l’aide d’une micro-centrifugeuse.
- Préparer le lysat avec des volumes égaux de tampon de lyse, puis appliquer la préparée a précisé lysate de la colonne et recueillir le cheminement.
- Réappliquez clarifié lysat intermédiaires à la colonne et de recueillir les intermédiaires secondaires.
- Laver la colonne avec le tampon de lavage 1 (10 mM Tris-HCl (pH 7.79), 300 mM NaCl, 5 mM MgCl2, 50 mM NaH2PO4, imidazole de 30 mM, 10 % de glycérol). Recueillir le cheminement.
Remarque : L’Inclusion de 5 mM MgCl2 dans les mémoires tampons de lavage et d’élution est importante à l’activité enzymatique de la protéine purifiée. - Laver la colonne avec le tampon de lavage 2 (10 mM Tris-HCl (pH 7.79), NaCl, 5 mM MgCl2, 50 mM NaH2PO4 et imidazole de 50 mM à 300 mM). Recueillir le cheminement.
- Appliquer le tampon d’élution de 2 mL (10 mM Tris-HCl (pH 7.79), 300 mM NaCl, 50 mM NaH2PO4, 10 % de glycérol et d’imidazole de 75 mM). Recueillir des intermédiaires en deux fractions de 1 mL de chaque.
Remarque : La colonne après cette étape peut être stockée dans le tampon de l’équilibration à 4 ° C si la même protéine sera purifiée dans le prochain test de purification. La colonne peut être utilisée jusqu'à 3 fois si stocké correctement.
- Évaluer les fractions de colonne pour la pureté par SDS-PAGE.
- Evaluer qualitativement purification des protéines, exécutez 20 μL des parties aliquotes de toutes les fractions de colonne sur un gel de polyacrylamide de 4/10 % pendant 60 min à 170 V.
- Coloration du gel par le bleu de Coomassie 0,1 % à température ambiante pendant 5 h, bascule doucement sur une bascule de benchtop.
- Décolorer le gel dans le méthanol 40 %, l’acide acétique 10 % glacial du jour au lendemain à la température ambiante, bascule sur une bascule de benchtop.
Remarque : Un gel représentatif est illustré à la Figure 1.
- Dialyser Éluer la fraction 2 nuit à 4 ° C.
- Dialyser contre tampon de dialyse (15,7 mM Tris-HCl (pH 7,6), 471,9 mM NaCl, 15,69 mM MgCl2, 1,57 mM DTT, PMSF à 1,5 mM et 15,7 % de glycérol) à un ratio de 200 : 1 à l’aide d’un appareil de dialyse de 1 mL avec un seuil de poids moléculaire 20 kDa (MWCO).
- Déterminer la concentration de protéine dialysée échantillon en mesurant l’absorbance à 280 nm et à l’aide de l’extinction molaire calculée coefficient 82085 M-1cm-1 20.
- Conserver 5 aliquotes μl de l’échantillon de protéine dialysée en aliquotes à-80 ° C jusqu'à l’utilisation.
3. protéine activité dosage par chromatographie sur couche mince
- Préparer la plaque de chromatographie sur couche mince.
- Avant d’effectuer la réaction, préparer POLYÉTHYLÈNEIMINE (PEI)-plaques de cellulose par lavage à l’eau désionisée. Placer les plaques dans une enceinte de verre avec l’eau bidistillée jusqu'à une profondeur de ~0.5 cm.
- Permettre à l’eau de migrer vers le haut de la plaque.
Remarque : La vaisselle n’est pas strictement nécessaire, car les plaques peuvent être utilisées sans elle, mais laver augmente la clarté des images qui en résultent. La vaisselle dans deux directions perpendiculaires davantage s’assure que tous les contaminants présents dans la résine soient isolés dans un coin de la plaque (Figure 2). - Sortir les plaques de la chambre de verre et laisser sur un rack de benchtop sécher jusqu’au lendemain (12 – 18 h).
- Marquer les plaques sèches 2,0 cm d’un bord avec un crayon pour indiquer où les échantillons seront appliqués pour TLC. Pour les 2 échantillons μL, appliquer des échantillons pas moins de 1,0 cm de distance (Figure 2).
NOTE : Cela permet une séparation claire entre la place adjacente, ce qui est essentiel pour la quantification du signal. Tant que la résine de cellulose n’est pas rayé, petites marques de crayon sur la surface n’interfèrent pas avec solvant migration. - Lors de la planification d’expériences, toujours laisser une tache sur chaque plaque inutilisés.
Remarque : Ceci fournira une ruelle vide pour la quantification de l’échantillon (Figure 2). Une plaque de 20 cm TLC aura Colocation de 19 points.
- Dosage de l’activité enzymatique
- Préparer un mélange de tampon 5 x contenant 50 mM Tris-HCl (pH 7,5), acétate d’ammonium de 25 mM, 10 mM KCl, 1 TNT et 0,6 mM ATP.
Remarque : Ce mélange peut préparer en grandes quantités et congelé dans 10 μL parties aliquotes pour une utilisation ultérieure. Ne pas soumettre le mélange à plusieurs cycles de gel-dégel. - Préparer les réactions individuelles contenant 3 μM RSH, 0,6 mM PIB, 1 mélange tampon x 1,2 mM MgCl2. Ajouter 1,0 μCi de γ -32P-ATP 10 μL de la réaction et la nucléase gratuit à l’eau apportent la réaction jusqu'à un volume désiré. Ajoutez le RSH après que les autres composantes ont été mitigés, comme l’ajout de RSH au mélange contenant du nucléotide initie le dosage de l’activité enzymatique.
Remarque : Volume réactionnel Final dépendra du nombre de points échantillonnés. Pour effectuer un échantillonnage 2 μL/validant, assembler 10 μl du mélange réactionnel pour chaque 4 h. - Pour contrôler d’hydrolyse de l’ATP d’activité nucléasique contaminante, assembler la réaction 10 μL ne contenant aucune protéine et il incuber en parallèle. Spot 2 échantillons μL à t = 0 et à la fin de l’expérience pour s’assurer que les ATP a été hydrolysé pas en l’absence de la protéine.
- Retirer immédiatement lors de l’addition de RSH, 2 μL et il place sur la plaque de PEI-cellulose marquée par la t = 0 min sample.
- Incubez la réaction à 37 ° C, enlever 2 aliquotes μL à intervalles souhaités.
Remarque : L’activité enzymatique cessera lorsque l’échantillon est adsorbé sur la plaque de cellulose. Attendre 10 à 30 min après que la dernière place est ajoutée à la plaque avant le développement afin d’assurer la complète adsorption et échantillon de séchage.
- Préparer un mélange de tampon 5 x contenant 50 mM Tris-HCl (pH 7,5), acétate d’ammonium de 25 mM, 10 mM KCl, 1 TNT et 0,6 mM ATP.
- Chromatographie sur couche mince
- Remplir la chambre de chromatographie avec 1,5 M 1,5 M KH2PO4 (pH 3,64) jusqu'à une profondeur de 0,5 cm.
Remarque : Le volume nécessaire dépendra des dimensions de la cuve de chromatographie. N’importe quel récipient en verre avec un bas niveau, qui est assez large pour permettre l’insertion de la plaque TLC sans plier peut servir d’une cuve de développement avec l’ajout d’un couvercle. La plaque TLC peut être coupée en lanières étroites avec un rasoir propre à permettre le développement dans un bécher de verre couvert de film plastique. - Immerger le bord inférieur de la plaque dans le solvant. Laisser le solvant migrer vers le haut de la plaque (environ 90 min).
NOTE : Tandis que solvant migration s’arrête en haut de la plaque et les échantillons ne seront pas perdus ou courir ensemble pendant une plus longue immersion, plaques ne doivent pas en dissolvant du jour au lendemain. Immersions en eau plues de 4 h peuvent provoquer la résine à se détacher de la plaque de renfort et causer une perte de signal. - Retirez la plaque de la cuve de chromatographie et placez-le sur une paillasse Etendoir.
- Laissez la plaque à l’air sec du jour au lendemain.
NOTE : Séchage peut être accéléré par l’utilisation d’un séchoir à cheveux. La sécheresse peut être évaluée par la couleur de la résine, qui s’assombrit lorsqu’il est mouillé et retour à la couleur d’une plaque non utilisée lorsqu’il est complètement sec. - Après que la plaque est sèche, enrouler la plaque en plastique pour éviter le transfert de matières radioactives à la cassette d’imagerie et d’analyser par autoradiographie (Figure 3).
- Remplir la chambre de chromatographie avec 1,5 M 1,5 M KH2PO4 (pH 3,64) jusqu'à une profondeur de 0,5 cm.
- Analyse des données
- Exposer la plaque PEI-cellulose contenant des réactions séparées sur une cassette de phosphorimager pendant 4 h à température ambiante.
Remarque : Il s’agit d’une exposition suffisante pour donner une image très claire en utilisant les concentrations indiquées de frais γ -32P-ATP. Si des quantités plus faibles de substrat radiomarqué sont utilisées, temps d’exposition peut être augmentée de 12 à 16 h. - Images de la cassette sur un phosphorimager.
- À l’aide de logiciels d’imagerie avec une interface utilisateur graphique, dessinez des régions d’intérêt (ROIs) en sélectionnant le Rectangle à dessiner à l’aide de la souris pour dessiner des ROIs rectangulaires autour d’un ensemble lane et l’ATP et ppGpp points contenus dans cette voie (Figure 3 ).
- Utiliser les commandes Sélectionner, copier et coller (ou des commandes correspondantes basées sur les logiciels d’imagerie utilisés) pour dessiner des ROIs identiques dans les autres voies pour s’assurer que la ROIs sont mesure de signal au sein des zones identiques dans chaque ruelle. Comprennent les ROIs d’une voie non utilisée, pour être utilisé comme une ébauche.
- À l’aide de la analyser | Outils | Gestionnaire de ROI | Ajouter commandes du logiciel d’imagerie, sélectionnez tous la ROIs sur la plaque de cellulose PEI.
- À l’aide de la analyser | Définir des mesures | Mesure commandes, quantifier l’intensité du signal au sein de chaque ROI et les mesures à l’exportation comme une feuille de calcul (Figure 3). Soustraire des valeurs vides de ROI de signaux expérimentaux.
- Calculer quel pourcentage du signal occulté au sein de chaque voie est attribuable à l’ATP et ppGpp en utilisant les formules

Remarque : Les ROIs peuvent être dessinés et quantifiée à l’aide de logiciels commerciaux compatibles avec le phosphorimager ou les logiciels ImageJ librement disponibles (National Institutes of Health).
- Exposer la plaque PEI-cellulose contenant des réactions séparées sur une cassette de phosphorimager pendant 4 h à température ambiante.
Access restricted. Please log in or start a trial to view this content.
Résultats
Nous présentons une méthode pour la purification d’affinité d’un (p) ppGpp synthétase de Clostridium difficile et l’évaluation de son activité enzymatique. La figure 1 illustre la purification de protéines obtenue par chromatographie d’affinité métallique. La seconde fraction d’élution (E2) de cette purification a été dialysée et utilisée pour le dosage de l’activité enzymatique. La figure 2 d?...
Access restricted. Please log in or start a trial to view this content.
Discussion
Nous rapportons ici la purification de son étiquette RSH de c. difficile et présenter une méthode de quantification de l’activité à l’aide de la chromatographie sur couche mince radiomarqué. Cette méthode a déjà été utilisée pour évaluer l’activité des enzymes cyclase diguanylate de c. difficile, ainsi que (p) ppGpp synthétase, adénylcyclase nucléotide kinase et enzymes de phosphodiestérase d’autres organismes11,12
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs déclarent pas concurrentes d’intérêts financiers ou autres conflits d’intérêts.
Remerciements
Ce travail a été financé par le NIAID 1K22AI118929-01. EBP était soutenu par une subvention du programme Summer recherche Fellowship de l’Office of Research à Old Dominion University, Norfolk, Virginia, USA.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Inducible overexpression of a histidine-tagged protein | |||
| Phusion polymerase | New England Biolabs (NEB) | M0530L | |
| QIAEX II DNA Gel Extraction Kit | Qiagen | 20021 | |
| KpnI restriction enzyme | NEB | R0142S | |
| PstI restriction enzyme | NEB | R0140S | |
| T4 DNA ligase | NEB | M0202 | |
| NEB® 5-alpha Competent E. coli (High Efficiency) | NEB | C2987I | |
| BL21 (DE3) Competent E. coli | NEB | C2527I | |
| IPTG | Sigma-Aldrich | 10724815001 | |
| JXN-26 centrifuge with JLA 10.500 rotor | Beckman Coulter Avanti | - | |
| Microcentrifuge with D3024/D3024R rotor | Scilogex | - | |
| MaxQ SHKE6000 Incubator | Thermo Scientific | - | |
| Ultrasonic processor | Sonics | VC-750 | |
| Protein purification by nickel affinity chromatography | |||
| Ni-NTA resin | G Biosciences | 786-940/941 | |
| Pierce Disposable Gravity columns, 10 mL | Thermo Scientific | 29924 | |
| 1 mL Spectra/ Por float-A-lyzer G2 dialysis device (MWCO: 20-kD) | Spectrum | G235033 | |
| Mini-Protean Electrophoresis Cell | BioRad | 1658004 | |
| Protein activity assay by thin layer chromatography | |||
| Thin layer chromatograph (TLC) development tank | General Glass Blowing Company | 80-3 | |
| Polyethylenimine (PEI)-cellulose plates (20 cm x 20 cm, 100 μm thickness) with polyester support | Sigma-Aldrich | Z122882-25EA | |
| ATP, [γ-32P]- 3000 Ci/mmol 10mCi/ml lead, 100 μCi | Perkin Elmer | NEG002A | |
| Adenosine 5’-triphosphate (ATP) 100 mM | Bio Basic Canada | AB0311 | |
| Guanosine-5’-diphosphate disodium salt (GDP) | Alfa Aesar | AAJ61646MC/E | |
| Storage phosphor screen | GE Healthcare Life Sciences | BAS-IP TR 2040 E Tritium Screen | |
| Storm 860 phosphorimager | GE Healthcare Life Sciences | - |
Références
- Cheek, S., Ginalski, K., Zhang, H., Grishin, N. V. A comprehensive update of the sequence and structure classification of kinases. BMC Structural Biology. 5 (1), 6(2005).
- Porath, J. Immobilized metal ion affinity chromatography. Protein Expression and Purification. 3 (4), 263-281 (1992).
- Arnau, J., Lauritzen, C., Pedersen, J. Cloning strategy, production and purification of proteins with exopeptidase-cleavable His-tags. Nature Protocols. 1 (5), 2326-2333 (2006).
- Porath, J., Carlsson, J., Olsson, I., Belfrage, G. Metal chelate affinity chromatography, a new approach to protein fractionation. Nature. 258 (5536), 598-599 (1975).
- Smith, M. C., Furman, T. C., Ingolia, T. D., Pidgeon, C. Chelating peptide-immobilized metal ion affinity chromatography. A new concept in affinity chromatography for recombinant proteins. Journal of Biological Chemistry. 263 (15), 7211-7215 (1988).
- Graslund, S., et al. Protein production and purification. Nature Methods. 5 (2), 135-146 (2008).
- Booth, W. T., et al. Impact of an N-terminal Polyhistidine Tag on Protein Thermal Stability. ACS Omega. 3 (1), 760-768 (2018).
- Thielges, M. C., Chung, J. K., Axup, J. Y., Fayer, M. D. Influence of histidine tag attachment on picosecond protein dynamics. Biochemistry. 50 (25), 5799-5805 (2011).
- Chaga, G. S. Twenty-five years of immobilized metal ion affinity chromatography: past, present and future. Journal of Biochemical and Biophysical Methods. 49 (1-3), 313-334 (2001).
- Ma, H., Deacon, S., Horiuchi, K. The challenge of selecting protein kinase assays for lead discovery optimization. Expert opinion on drug discovery. 3 (6), 607-621 (2008).
- Tamayo, R., Tischler, A. D., Camilli, A. The EAL domain protein VieA is a cyclic diguanylate phosphodiesterase. Journal of Biological Chemistry. 280 (39), 33324-33330 (2005).
- Purcell, E. B., McKee, R. W., McBride, S. M., Waters, C. M., Tamayo, R. Cyclic Diguanylate Inversely Regulates Motility and Aggregation in Clostridium difficile. Journal of Bacteriology. 194 (13), 3307-3316 (2012).
- Purcell, E. B., Tamayo, R. Identification and characterization of cyclic nucleotide phosphodiesterases. Methods in Molecular Biology. 1016, 235-243 (2013).
- Ross, P., et al. Control of cellulose synthesis Acetobacter xylinum. A unique guanyl oligonucleotide is the immediate activator of the cellulose synthase. Carbohydrate Research. 149 (1), 101-117 (1986).
- Potrykus, K., Cashel, M. (p)ppGpp: still magical? Annual Review of Microbiology. 62, 35-51 (2008).
- Boutte, C. C., Crosson, S. Bacterial lifestyle shapes stringent response activation. Trends in Microbiology. 21 (4), 174-180 (2013).
- Nanamiya, H., et al. Identification and functional analysis of novel (p)ppGpp synthetase genes in Bacillus subtilis. Molecular Microbiology. 67 (2), 291-304 (2008).
- Gaca, A. O., et al. Basal Levels of (p)ppGpp in Enterococcus faecalis: the Magic beyond the Stringent Response. mBio. 4 (5), (2013).
- Sebaihia, M., et al. The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome. Nature Genetics. 38 (7), 779-786 (2006).
- Gasteiger, E., et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis. Nucleic Acids Research. 31 (13), 3784-3788 (2003).
- Purcell, E. B., et al. A nutrient-regulated cyclic diguanylate phosphodiesterase controls Clostridium difficile biofilm and toxin production during stationary phase. Infection and Immunity. , (2017).
- Mechold, U., Murphy, H., Brown, L., Cashel, M. Intramolecular Regulation of the Opposing (p)ppGpp Catalytic Activities of RelSeq, the Rel/Spo Enzyme from Streptococcus equisimilis. Journal of Bacteriology. 184 (11), 2878-2888 (2002).
- Dalebroux, Z. D., Svensson, S. L., Gaynor, E. C., Swanson, M. S. ppGpp Conjures Bacterial Virulence. Microbiology and Molecular Biology Reviews. 74 (2), 171-199 (2010).
- Bartlett, J. G. Clostridium difficile: progress and challenges. Annals of the New York Academy of Sciences. 1213, 62-69 (2010).
- US Department of Health and Human Services. , Centers for Disease Control and Prevention, US Department of Health and Human Services. Atlanta, GA. (2013).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon