È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una purificazione e l'analisi di attività In Vitro per un ppGpp (p) sintetasi da Clostridium difficile
In questo articolo
Riepilogo
Qui, descriviamo un metodo per purificare enzimi pyrophosphokinase istidina-etichettate e utilizzando la cromatografia su strato sottile di radiomarcato substrati e prodotti di test per l' attività enzimatica in vitro. L'analisi di attività dell'enzima è ampiamente applicabile a qualsiasi chinasi, ciclasi del nucleotide o reazione di fosforo-trasferimento cui meccanismo include idrolisi di trifosfato del nucleotide.
Abstract
Gli enzimi della chinasi e pyrophosphokinase trasferire la gamma fosfato o la frazione di pirofosfato di beta-gamma dai precursori di nucleotide trifosfato ai substrati per creare prodotti fosforilati. L'uso di γ -32- P etichettato precursori NTP permette monitoraggio simultaneo della formazione di prodotto e l'utilizzo di substrato dalla radiografia. Cromatografia su strato sottile (TLC) su piastrine di cellulosa consente la rapida separazione e quantificazione sensibile del substrato ed il prodotto. Presentiamo un metodo per utilizzando la cromatografia di strato sottile per analizzare l'attività pyrophosphokinase di una purificato sintetasi di ppGpp (p). Questo metodo è stato utilizzato in precedenza per caratterizzare l'attività della sintetasi del nucleotide e dinucleotide ciclici ed è ampiamente adatto per caratterizzare l'attività di ogni enzima che idrolizza un legame di nucleotide trifosfato o trasferisce un terminale fosfato da un donatore di fosfato ad un'altra molecola.
Introduzione
Gli enzimi della chinasi e pyrophosphokinase (o membrana in direzione extra-chinasi) trasferimento fosfati dai precursori di nucleotide trifosfato (NTP) per molecole substrato. I substrati possono includere altri nucleotidi, aminoacidi o proteine, carboidrati e lipidi1. Analisi bioinformatiche talvolta possono prevedere un enzima substrato affine o substrati basate sulla somiglianza agli enzimi caratterizzati, ma convalida sperimentale è ancora necessario. Allo stesso modo, l'affinità di un enzima per la sua overesprimessero e la velocità che esso catalizza la reazione di trasferimento di fosforo e gli effetti della co-fattori, inibitori, o altri effettori enzima devono essere determinati sperimentalmente. Per evitare lo svuotamento del precursore ATP da altri enzimi che consumano ATP presenti nel citoplasma batterico, analisi quantitativa attività richiedono proteina purificata.
Purificazione della proteina di cromatografia di affinità del metallo è stato coperto accuratamente in letteratura2,3. Tag di istidina composto da residui di istidina consecutivi sei accodati a N - o C-terminale di una proteina ricombinante consentono la rapida purificazione da cromatografia di affinità del metallo4,5,6. Queste sequenze sono piccole rispetto alle proteine modificano e in genere hanno un effetto minimo sulla funzione della proteina, anche se a volte possono alterare stabilità proteica e/o enzima cinetica7,8. Istidina Tag presso il N - e C-termini della stessa proteina può avere effetti differenti, che sono difficili da prevedere senza conoscere la struttura della proteina in questione. Tag di istidina sono incorporati in genere durante la clonazione di una proteina ricombinante di progettazione degli iniettori che codificano sei residui di istidina, sia immediatamente il codone di inizio ATG a 3' o immediatamente 5' per il codone di arresto del telaio di lettura aperto. Dopo l'amplificazione, il gene hexahistidine-contenenti è legato in un vettore sotto il controllo di un promotore inducibile ed espressa, in genere in un ceppo di laboratorio di e. coli. La proteina di ricombinazione può quindi essere isolata su una resina di affinità contenente cationi bivalenti immobilizzato (tipicamente nichel o cobalto)9. Contaminando native proteine metallo-leganti possa essere rimossi mediante titolazione con imidazolo, che sposta competitivi associata proteina2. Infine, la proteina dell'obiettivo è eluita dalla colonna con concentrazioni più elevate di imidazolo. Ci sono diverse fonti commerciali per resine immobilizzato catione del metallo, e i produttori forniscono raccomandazioni per le condizioni di buffer e le concentrazioni dell'imidazolo. Dopo eluizione, proteina può essere analizzata mediante elettroforesi su gel di sodio dodecil solfato-poliacrilammide (SDS-PAGE), dializzata o usata immediatamente in saggi funzionali.
Ci sono diversi metodi per monitorare indirettamente l'attività della chinasi di idrolisi dell'ATP fosfato legame di accoppiamento a una seconda reazione che rilascia o eccita un fluoroforo o genera chemiluminescenza, ma queste reazioni hanno più parti mobili e possono essere logisticamente impegnativo10. Il modo più semplice per misurare specificamente l'attività di fosforo-trasferimento è per monitorare direttamente il trasferimento di un gruppo fosfato radioattivo da un commercialmente disponibili γ -32-P precursore NTP di un substrato non-radioattivi11 , 12 , 13. miscele di substrati radioattivi e i prodotti possono essere separate e quantificate mediante cromatografia su strato sottile (TLC). TLC utilizza la mobilità differenziale di soluti in un dato solvente consentendo il solvente (fase liquida) eseguire la migrazione di azione capillare attraverso una superficie (fase solida) su cui una miscela di soluti è stato adsorbito14. Soluti che sono piccole e/o mancano di interazioni favorevoli con la fase solida migrerà distanze più lunghe dalla loro posizione iniziale del soluti con pesi molecolari superiori o grande affinità per il solido. Per l'esame di fosforo-trasferimento, moiety fosfato aumentare il peso molecolare delle molecole vengono aggiunti a e aggiungere carica ionica negativa a pH neutro o acido11,12,14. Questo diminuisce la loro mobilità su una superficie di base quali PEI-cellulosa. Quando sviluppate in tampone fosfato acido di potassio, miscele di mono-, di-, tri-, tetra-e pentaphosphate specie possono essere facilmente separate su PEI-cellulosa, consentendo la quantificazione di ogni specie (Figura 2, figura 3). Tali dosaggi possono essere eseguite utilizzando lisati cellulari contenenti l'enzima di interesse, ma questo include il potenziale per l'attività di altre chinasi e fosfatasi ATPasi generale per vuotare il substrato e/o prodotto. Per una valutazione quantitativa in vitro dell'attività dell'enzima, è necessario purificare l'enzima di interesse.
Tetrafosfato di guanosina (ppGpp) e guanosina pentaphosphate (pppGpp) sono ribonucleotide di segnalazione molecole formate dal trasferimento di un pirofosfato di gruppo da un precursore di adenosina trifosfato (ATP) a, rispettivamente, un guanosina difosfato (GDP) o guanosina tetrafosfato (GTP) substrato15. Questi segnali di singolo ribonucleotide, collettivamente conosciuti come ppGpp (p), mediano una risposta cellulare allo stress ambientale, conosciuta come la risposta rigorosa in diverse specie batteriche15,16. Conservate due classi di enzimi catalizzano la formazione di (p) ppGpp15,17 Rel/Spo omologo (RSH) enzimi sono 'lungo' bifunzionale (p) ppGpp sintetasi/idrolasi denominato per la loro somiglianza con le relazioni ed il punto (p) ppGpp metabolica enzimi da Escherichia coli che contengono sintetasi, idrolasi e regolamentazione domini, mentre piccole alarmone sintetasi (SAS) gli enzimi sono breve monofunzionale sintetasi trovati esclusivamente a Gram positivi batteri15, 17 , 18. il batterio gram-positivo sporigeni Clostridium difficile codifica putativi geni RSH e SAS19. Qui, presentiamo analisi di attività iniziale che confermano che l'enzima di c. difficile RSH è una cataliticamente attiva sintetasi di ppGpp (p).
Protocollo
1. viscoelastica sovraespressione di una proteina istidina-etichettate
- Amplificare rsh da c. difficile R20291 DNA genomic.
- Utilizzare una polimerasi ad alta fedeltà e seguire le istruzioni del produttore.
- Amplificare il c. difficile rsh utilizzando primers
rsh_F (CAGGTACCGGTTATATGCATGATAAAGAATTACAAG) e
rsh_R (CCCTGCAGCTAATGGTGATGGTGATGGTGATTTGTCATTCTATAAATAC), che introduce un tag hexahistidine C-terminale.
Nota: Il KpnI e PstI tagliare siti nelle sequenze dell'iniettore sono in grassetto. - Digerire il vettore pMMBneo e il prodotto PCR di rsh-his6 con restrizione Pstl e KpnI tagliato siti a 37 ° C per 45 min.
- Purificare il vettore linearizzato e i frammenti PCR mediante elettroforesi su gel di agarosio e purificazione successiva con un kit di estrazione del gel del DNA.
- Misurare l'assorbanza nm 280 (un280) del vettore e il prodotto PCR amplificato. Utilizzare l'equazione
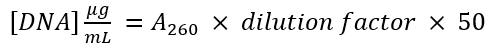 per determinare la concentrazione di DNA di ogni frammento di DNA.
per determinare la concentrazione di DNA di ogni frammento di DNA.
- Lega i rsh in pMMBneo per l'espressione.
- Combine 25 ng di digerito PMMBneo vettoriale, 125 ng del prodotto del gene di rsh-his6 , 2 μL di tampone di ligasi 10x e 1 μL di DNA ligasi. Utilizzare l'acqua gratuita di nucleasi per regolare il volume totale a 20 μL. Incubare a 16 ° C per 16 h.
Nota: L'efficacia della legatura dipende dalla dimensione del frammento e deve essere aggiustato in base a protocolli del produttore. - Trasformare il prodotto vettoriale in e. coli DH5-α o un altro ceppo recA - plasmide manutenzione. Selezionare per cellule trasformate su piastre LB con kanamicina di 100 μg/mL e incubare per 16-24 h a 37 ° C.
- Prendere quattro colonie dalla piastra di trasformazione (s) e rigare ciascuno su un piatto fresco che contiene 100 kanamicina μg/mL per isolamento del DNA. Incubare le nuove piastre a 37 ° C per 16 – 24 h e selezionare un isolato per l'espressione della proteina successive.
- Per confermare il successo legatura della regione di codificazione della proteina intatta rsh , prelevare una Colonia del scelto isolare in 20 μL di acqua, riscaldare a 95 ° C per 10 min e utilizzare come un modello per una PCR conferma utilizzando il primer stesso utilizzato per amplificare il gene.
- Combine 25 ng di digerito PMMBneo vettoriale, 125 ng del prodotto del gene di rsh-his6 , 2 μL di tampone di ligasi 10x e 1 μL di DNA ligasi. Utilizzare l'acqua gratuita di nucleasi per regolare il volume totale a 20 μL. Incubare a 16 ° C per 16 h.
- Trasformare il plasmide verificato secondo i protocolli standard di trasformazione per la trasformazione di plasmide batterico di Escherichia coli in e. coli BL21 sistema per la produzione di proteine ad alto rendimento.
- RSH-His6 espressa in e. coli.
- Selezionare una singola colonia di e. coli BL21 trasformato con pMMBneo::rsh e inoculare 2 mL di terreno LB con kanamicina di 50 μg/mL. Incubare a 37 ° C 12 – 16 h mentre si stringono a 250 giri/min.
- Inoculare 500 mL di mezzo LB contenente 50 kanamicina μg/mL con 0,5 mL della coltura durante la notte per la crescita delle cellule e l'espressione della proteina.
- Incubare la cultura di espressione a 37 ° C e 250 giri/min in un agitatore incubatore fino a quando la densità delle cellule raggiunge un OD600 0,167 a 37 ° C.
- Ridurre la temperatura dell'incubatore a 30 ° C e attendere 30 minuti per la temperatura di cultura a goccia.
- Inducono l'espressione di RSH aggiungendo isopropilico β-D-thiogalactoside (IPTG) ad una concentrazione finale di 0,5 mM. Consentire l'induzione che si terrà durante la notte (per h 16 – 18) a 30 ° C, agitando a 250 giri/min.
- A pellet mediante centrifugazione a 3.080 x g per 30 min a 4 ° C.
Nota: Il pellet possa essere immagazzinato durante la notte a-20 ° C per purificare la proteina bersaglio il giorno seguente senza notevole perdita di resa di proteine o di attività enzimatica.
2. proteina purificazione mediante cromatografia di affinità di nichel
Nota: Continuare direttamente con fasi di purificazione di proteine fornite di seguito dopo aver chiarito il lysate delle cellule. Memorizzazione di chiarito lisato a 4 ° C durante la notte per la purificazione della proteina successive riduce il rendimento della proteina.
- Purificare la proteina con 1 mL di resina nichel-Nitriloacetic acido (Ni-NTA) su una colonna di gravità.
- Il giorno prima di utilizzare, equilibrare la colonna durante la notte a 4 ° C con 2 mL di tampone di equilibrazione (10 mM Tris-HCl pH 7.79, 300 mM NaCl, 50mm NaH2PO4, lisozima di 0,5 mg/mL, 5 mM MgCl2, imidazolo di 10 mM, 0.25 mM DTT, sulfonil fenilmetano 5 mM fluoruro (PMSF), 10% glicerolo).
- Il giorno seguente portare la colonna da 4 ° C a RT prima di caricare il chiarificato lisato e lasciate stare per ~ 2 – 3 h.
Nota: Portare la colonna all'equilibrio termico è fondamentale per evitare bolle d'aria che formano all'interno della colonna. - Risospendere il pellet ottenuto nel passaggio 1.3.6 il tampone di lisi (10 mM Tris-HCl pH 7,8, 300 mM NaCl, 5 mM MgCl2, 50mm NaH2PO4, 10% glicerolo, lisozima di 0,5 mg/mL, imidazolo di 10 mM, 0.25 mM DTT e 5 mM PMSF).
- Sonicare cellule sul ghiaccio per intervalli di 8 x 10 s, pausa 30 s tra gli impulsi.
- Chiarire il lisato mediante centrifugazione a 3.080 x g per 30 min a 4 ° C, utilizzando una microcentrifuga.
- Preparare lisato con volumi uguali di tampone di lisi quindi applicare il preparato chiarito lisato alla colonna e raccogliere il flusso continuo.
- Riapplicare lisato chiarificato flusso-attraverso la colonna e raccogliere il passaggio secondario.
- Lavare la colonna con tampone di lavaggio 1 (10 mM Tris-HCl (pH 7.79), 300 mM NaCl, 5 mM MgCl2, 50mm NaH2PO4, imidazolo di 30 mM, 10% glicerolo). Raccogliere il flusso continuo.
Nota: L'inclusione di 5 mM MgCl2 nei buffer di lavaggio e di eluizione è importante per l'attività enzimatica della proteina purificata. - Lavare la colonna con tampone di lavaggio 2 (10 mM Tris-HCl (pH 7.79), 300 mM NaCl, 5 mM MgCl2, 50mm NaH2PO4 e imidazolo di 50 mM). Raccogliere il flusso continuo.
- Applicare 2 mL di tampone di eluizione (10 mM Tris-HCl (pH 7.79), 300 mM NaCl, 50mm NaH2PO4, 10% glicerolo e imidazolo 75 mM). Raccogliere flusso continuo in due frazioni di 1 mL ciascuno.
Nota: La colonna dopo questo passaggio possa essere memorizzata nel buffer di equilibrazione a 4 ° C se la stessa proteina sarà purificata nell'analisi della purificazione successiva. La colonna può essere utilizzata fino a 3 volte se conservati correttamente.
- Valutare le frazioni di colonna per la purezza da SDS-PAGE.
- Per valutare qualitativamente la purificazione della proteina, eseguire 20 aliquote del μL di tutte le frazioni di colonna su un gel di poliacrilammide del 4% / 10% per 60 min a 170 V.
- Macchia il gel con blue di Coomassie 0,1% a temperatura ambiente per 5 h, oscilla delicatamente su un rocker di benchtop.
- Decolorare il gel in metanolo al 40%, 10% di acido acetico glaciale durante la notte a temperatura ambiente, a dondolo su un rocker di benchtop.
Nota: Un gel rappresentativo è raffigurato nella Figura 1.
- Dializzare eluire frazione 2 durante la notte a 4 ° C.
- Dializzare contro il buffer di dialisi (15,7 mM Tris-HCl (pH 7.6), 471,9 mM NaCl, 15,69 mM MgCl2, 1,57 mM DTT, 1,5 mM PMSF e 15,7% glicerolo) con un rapporto di 200: 1 utilizza un dispositivo di dialisi 1 mL con un limite di peso molecolare 20 kDa (MWCO).
- Determinare la concentrazione di proteina dializzati campione misurando l'assorbanza a 280 nm e utilizzando l'estinzione molare calcolato coefficiente 82085 M-1cm-1 20.
- Memorizzare 5 aliquote del μL del campione della proteina dializzati in aliquote a-80 ° C fino all'utilizzo.
3. proteine attività analisi mediante cromatografia su strato sottile
- Preparare la piastra di cromatografia di strato sottile.
- Prima di eseguire la reazione, preparare polyethyleneimine (PEI)-piastrine di cellulosa mediante lavaggio con acqua deionizzata. Posizionare le piastre in una camera di vetro con doppia acqua distillata ad una profondità di cm ~0.5.
- Lasciare acqua a migrare verso la parte superiore della piastra.
Nota: I piatti di lavaggio non è strettamente necessario, come piastre possono essere utilizzati senza di essa, ma lavaggio aumentare la chiarezza delle immagini risultanti. I piatti di lavaggio in due direzioni perpendicolari ulteriormente garantisce che eventuali contaminanti presenti nella resina sono isolati in un angolo della piastra (Figura 2). - Portare le piastre nella camera di vetro e lasciare su una cremagliera di benchtop per asciugare durante la notte (12 – 18 h).
- Contrassegnare le piastre asciutte 2,0 cm da un bordo con una matita morbida per indicare dove i campioni saranno applicati per TLC. Per 2 campioni μL, applicare i campioni non meno di 1,0 cm di distanza (Figura 2).
Nota: Questo consente la netta separazione tra posto adiacente, che è fondamentale per la quantificazione del segnale. Come la resina di cellulosa non è graffiato, piccoli segni di matita sulla superficie non interferirà con solvente migrazione. - Durante la pianificazione di esperimenti, sempre lasciare uno spot su ogni piatto inutilizzati.
Nota: Ciò fornirà una corsia vuota per la quantificazione del campione (Figura 2). Un piatto di 20cm TLC avrà spazio per 19 punti.
- Enzymaktivitätsbestimmung
- Preparare una miscela di buffer 5x contenente 50 mM Tris-HCl (pH 7.5), acetato di ammonio di 25 mM, 10 mM KCl, 1 millimetro DTT e 0,6 mM ATP.
Nota: Questo mix può essere preparato in grandi quantità e congelato in 10 aliquote del μL per un uso successivo. Non sottoporre mix a più cicli di gelo-disgelo. - Preparare le reazioni individuali contenenti 3 μM RSH, mix di 1 x buffer, 0,6 mM PIL, 1,2 mM MgCl2. Aggiungere 1,0 μCi γ -32P-ATP per 10 μL di reazione e utilizzare acqua gratuita nucleasi per portare la reazione fino a un volume desiderato. Aggiungere il RSH dopo gli altri componenti sono stati mescolati, come l'aggiunta di RSH alla miscela del nucleotide-contenente avvia il dosaggio dell'attività enzimatica.
Nota: Volume di reazione finale dipenderà il numero di punti temporali campionate. Per campionare 2 μL/timepoint, assemblare 10 μL di miscela di reazione per ogni 4 punti temporali. - Per controllare per idrolisi dell'ATP da contaminanti attività nucleasi, assemblare una reazione μL 10 non contenente nessuna proteina e viene quindi incubato in parallelo. Spot 2 campioni μL a t = 0 e alla fine dell'esperimento per garantire che ATP non era idrolizzato in assenza di proteina.
- Rimuovere immediatamente con l'aggiunta di RSH, 2 μL e individuarlo sulla piastra di PEI-cellulosa etichettata come la t = 0 min campione.
- Incubare la reazione a 37 ° C, rimozione 2 aliquote del μL timepoints desiderata.
Nota: L'attività enzimatica cesserà quando il campione è adsorbito sulla piastra di cellulosa. Attendere 10 – 30 min dopo l'ultimo posto è aggiunto alla piastra prima di sviluppo per garantire completa adsorbimento ed essiccazione del campione.
- Preparare una miscela di buffer 5x contenente 50 mM Tris-HCl (pH 7.5), acetato di ammonio di 25 mM, 10 mM KCl, 1 millimetro DTT e 0,6 mM ATP.
- Cromatografia su strato sottile
- Riempire la camera di cromatografia con 1,5 M 1,5 M KH2PO4 (pH 3,64) ad una profondità di 0,5 cm.
Nota: Il volume necessario sarà dipendono dalle dimensioni del serbatoio cromatografia. Qualsiasi contenitore di vetro con un basso livello che è sufficientemente ampia per consentire l'inserimento della piastra TLC senza flessione può essere utilizzato come un tank di sviluppo con l'aggiunta di una copertura. La piastra di TLC può essere tagliata in strisce più stretti con un battistrada pulito per consentire lo sviluppo in un becher di vetro rivestito in film plastico. - Immergere il bordo inferiore della piastra in solvente. Consentire il solvente migrare nella parte superiore della piastra (~ 90 min).
Nota: Mentre solvente migrazione verrà interrotta nella parte superiore della piastra e campioni non verranno perso o correre insieme durante un'immersione più lunga, piastre dovrebbero non essere lasciati in solvente durante la notte. Immersioni più di 4 ore possono causare la resina staccare dalla piastra di appoggio e causare la perdita del segnale. - Rimuovere la piastra dal serbatoio di cromatografia e posizionarlo su un benchtop stendino.
- Lasciare asciugare la piastra all'aria durante la notte.
Nota: Essiccazione può essere accelerato tramite l'uso di un asciugacapelli. Secchezza può essere valutata dal colore della resina, che si scurisce quando è bagnato e ritorno al colore di una piastra inutilizzato quando è completamente asciutto. - Dopo che il piatto è asciutto, avvolgere la piastra in pellicola di plastica per evitare il trasferimento di materiale radioattivo per la cassetta di imaging e analizzare mediante autoradiografia (Figura 3).
- Riempire la camera di cromatografia con 1,5 M 1,5 M KH2PO4 (pH 3,64) ad una profondità di 0,5 cm.
- Analisi dei dati
- Esporre la piastra di PEI-cellulosa contenente le reazioni separate su una videocassetta phosphorimager per 4 h a temperatura ambiente.
Nota: Questa è l'esposizione sufficiente per produrre un'immagine molto chiara utilizzando le concentrazioni indicate di fresco γ -32P-ATP. Se vengono utilizzati gli importi più bassi di substrato radiomarcato, tempo di esposizione può essere aumentata a 12-16 h. - Immagine della cassetta su un phosphorimager.
- Utilizzando software di imaging con un'interfaccia utente grafica, disegnare regioni di interesse (ROI) selezionando Rettangolo disegnare e utilizzando il mouse per disegnare ROIs rettangolare intorno uno spot di lane e l'ATP e ppGpp intero contenuto all'interno di quella corsia (Figura 3 ).
- Utilizzare i comandi selezionare, copiare e incollare (o corrispondenti comandi basati sul software di imaging utilizzato) per disegnare ROIs identici all'interno le altre corsie per garantire che il ROIs sono misurando il segnale all'interno di aree identiche in ogni corsia. Sono ROIs da una corsia di inutilizzati, per essere utilizzati come spazi vuoti.
- Utilizzando il analizzare | Strumenti | ROI Manager | Aggiungi comandi del software di imaging, selezionare tutti i ROIs disegnata sul piatto cellulosa PEI.
- Utilizzando il analizzare | Impostare misure | Misura comandi, quantificare l'intensità del segnale all'interno di ogni ROI ed esportare le misurazioni come un foglio di calcolo (Figura 3). Sottrarre i valori vuoti di ROI da segnali sperimentali.
- Calcolare quale percentuale del segnale tranciate all'interno di ogni corsia è imputabile all'ATP e ppGpp utilizzando le formule

Nota: ROIs può essere disegnata e quantificata utilizzando software commerciali compatibile con il phosphorimager o liberamente disponibile software ImageJ (National Institutes of Health).
- Esporre la piastra di PEI-cellulosa contenente le reazioni separate su una videocassetta phosphorimager per 4 h a temperatura ambiente.
Risultati
Presentiamo un metodo per la purificazione di affinità di una sintetasi di ppGpp (p) da Clostridium difficile e la valutazione della sua attività enzimatica. Figura 1 dimostra la purificazione della proteina mediante cromatografia di affinità del metallo. La seconda frazione di eluizione (E2) da questa purificazione è stata dializzata e utilizzata per il dosaggio dell'attività enzimatica. Figura 2 in dettaglio i pas...
Discussione
Qui segnaliamo la purificazione della sua etichetta RSH da c. difficile e presentare un metodo per la quantificazione di attività mediante cromatografia su strato sottile radiomarcato. Questo metodo è stato utilizzato in precedenza per valutare l'attività di enzimi ciclasi diguanylate da c. difficile, come pure della sintetasi di ppGpp (p), ciclasi del nucleotide, chinasi ed enzimi fosfodiesterasi da altri organismi11,12 ,
Divulgazioni
Gli autori dichiarano senza concorrenti interessi finanziari o altri conflitti di interesse.
Riconoscimenti
Questo lavoro è stato finanziato dal NIAID 1K22AI118929-01. EBP è stata sostenuta da una sovvenzione di programma estate Research Fellowship dall'Office of Research presso Old Dominion University, Norfolk, Virginia, USA.
Materiali
| Name | Company | Catalog Number | Comments |
| Inducible overexpression of a histidine-tagged protein | |||
| Phusion polymerase | New England Biolabs (NEB) | M0530L | |
| QIAEX II DNA Gel Extraction Kit | Qiagen | 20021 | |
| KpnI restriction enzyme | NEB | R0142S | |
| PstI restriction enzyme | NEB | R0140S | |
| T4 DNA ligase | NEB | M0202 | |
| NEB® 5-alpha Competent E. coli (High Efficiency) | NEB | C2987I | |
| BL21 (DE3) Competent E. coli | NEB | C2527I | |
| IPTG | Sigma-Aldrich | 10724815001 | |
| JXN-26 centrifuge with JLA 10.500 rotor | Beckman Coulter Avanti | - | |
| Microcentrifuge with D3024/D3024R rotor | Scilogex | - | |
| MaxQ SHKE6000 Incubator | Thermo Scientific | - | |
| Ultrasonic processor | Sonics | VC-750 | |
| Protein purification by nickel affinity chromatography | |||
| Ni-NTA resin | G Biosciences | 786-940/941 | |
| Pierce Disposable Gravity columns, 10 mL | Thermo Scientific | 29924 | |
| 1 mL Spectra/ Por float-A-lyzer G2 dialysis device (MWCO: 20-kD) | Spectrum | G235033 | |
| Mini-Protean Electrophoresis Cell | BioRad | 1658004 | |
| Protein activity assay by thin layer chromatography | |||
| Thin layer chromatograph (TLC) development tank | General Glass Blowing Company | 80-3 | |
| Polyethylenimine (PEI)-cellulose plates (20 cm x 20 cm, 100 μm thickness) with polyester support | Sigma-Aldrich | Z122882-25EA | |
| ATP, [γ-32P]- 3000 Ci/mmol 10mCi/ml lead, 100 μCi | Perkin Elmer | NEG002A | |
| Adenosine 5’-triphosphate (ATP) 100 mM | Bio Basic Canada | AB0311 | |
| Guanosine-5’-diphosphate disodium salt (GDP) | Alfa Aesar | AAJ61646MC/E | |
| Storage phosphor screen | GE Healthcare Life Sciences | BAS-IP TR 2040 E Tritium Screen | |
| Storm 860 phosphorimager | GE Healthcare Life Sciences | - |
Riferimenti
- Cheek, S., Ginalski, K., Zhang, H., Grishin, N. V. A comprehensive update of the sequence and structure classification of kinases. BMC Structural Biology. 5 (1), 6 (2005).
- Porath, J. Immobilized metal ion affinity chromatography. Protein Expression and Purification. 3 (4), 263-281 (1992).
- Arnau, J., Lauritzen, C., Pedersen, J. Cloning strategy, production and purification of proteins with exopeptidase-cleavable His-tags. Nature Protocols. 1 (5), 2326-2333 (2006).
- Porath, J., Carlsson, J., Olsson, I., Belfrage, G. Metal chelate affinity chromatography, a new approach to protein fractionation. Nature. 258 (5536), 598-599 (1975).
- Smith, M. C., Furman, T. C., Ingolia, T. D., Pidgeon, C. Chelating peptide-immobilized metal ion affinity chromatography. A new concept in affinity chromatography for recombinant proteins. Journal of Biological Chemistry. 263 (15), 7211-7215 (1988).
- Graslund, S., et al. Protein production and purification. Nature Methods. 5 (2), 135-146 (2008).
- Booth, W. T., et al. Impact of an N-terminal Polyhistidine Tag on Protein Thermal Stability. ACS Omega. 3 (1), 760-768 (2018).
- Thielges, M. C., Chung, J. K., Axup, J. Y., Fayer, M. D. Influence of histidine tag attachment on picosecond protein dynamics. Biochemistry. 50 (25), 5799-5805 (2011).
- Chaga, G. S. Twenty-five years of immobilized metal ion affinity chromatography: past, present and future. Journal of Biochemical and Biophysical Methods. 49 (1-3), 313-334 (2001).
- Ma, H., Deacon, S., Horiuchi, K. The challenge of selecting protein kinase assays for lead discovery optimization. Expert opinion on drug discovery. 3 (6), 607-621 (2008).
- Tamayo, R., Tischler, A. D., Camilli, A. The EAL domain protein VieA is a cyclic diguanylate phosphodiesterase. Journal of Biological Chemistry. 280 (39), 33324-33330 (2005).
- Purcell, E. B., McKee, R. W., McBride, S. M., Waters, C. M., Tamayo, R. Cyclic Diguanylate Inversely Regulates Motility and Aggregation in Clostridium difficile. Journal of Bacteriology. 194 (13), 3307-3316 (2012).
- Purcell, E. B., Tamayo, R. Identification and characterization of cyclic nucleotide phosphodiesterases. Methods in Molecular Biology. 1016, 235-243 (2013).
- Ross, P., et al. Control of cellulose synthesis Acetobacter xylinum. A unique guanyl oligonucleotide is the immediate activator of the cellulose synthase. Carbohydrate Research. 149 (1), 101-117 (1986).
- Potrykus, K., Cashel, M. (p)ppGpp: still magical?. Annual Review of Microbiology. 62, 35-51 (2008).
- Boutte, C. C., Crosson, S. Bacterial lifestyle shapes stringent response activation. Trends in Microbiology. 21 (4), 174-180 (2013).
- Nanamiya, H., et al. Identification and functional analysis of novel (p)ppGpp synthetase genes in Bacillus subtilis. Molecular Microbiology. 67 (2), 291-304 (2008).
- Gaca, A. O., et al. Basal Levels of (p)ppGpp in Enterococcus faecalis: the Magic beyond the Stringent Response. mBio. 4 (5), (2013).
- Sebaihia, M., et al. The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome. Nature Genetics. 38 (7), 779-786 (2006).
- Gasteiger, E., et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis. Nucleic Acids Research. 31 (13), 3784-3788 (2003).
- Purcell, E. B., et al. A nutrient-regulated cyclic diguanylate phosphodiesterase controls Clostridium difficile biofilm and toxin production during stationary phase. Infection and Immunity. , (2017).
- Mechold, U., Murphy, H., Brown, L., Cashel, M. Intramolecular Regulation of the Opposing (p)ppGpp Catalytic Activities of RelSeq, the Rel/Spo Enzyme from Streptococcus equisimilis. Journal of Bacteriology. 184 (11), 2878-2888 (2002).
- Dalebroux, Z. D., Svensson, S. L., Gaynor, E. C., Swanson, M. S. ppGpp Conjures Bacterial Virulence. Microbiology and Molecular Biology Reviews. 74 (2), 171-199 (2010).
- Bartlett, J. G. Clostridium difficile: progress and challenges. Annals of the New York Academy of Sciences. 1213, 62-69 (2010).
- . . US Department of Health and Human Services. , (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon