JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
정화 및 체 외에서 활동 분석 결과 (p) ppGpp Clostridium 남과 어울리지 않 는에서 합성에 대 한
요약
여기, 히스티딘 태그 pyrophosphokinase 효소 정화 및 radiolabelled 기질 그리고 효소 활동 체 외에 대 한 분석 결과를 제품의 얇은 층 크로마토그래피를 이용 하는 방법을 설명 합니다. 효소 활동 분석 결과 모든 키, 뉴클레오티드 있고, 또는 인광 체 전송 반응의 메커니즘 포함 뉴클레오티드 3 인산 염 가수분해에 광범위 하 게 적용 됩니다.
초록
키 및 pyrophosphokinase 효소 phosphorylated 제품을 기판에 뉴클레오티드 3 인산 염 선구자에서 감마 인산 염 또는 베타-감마 파이 인산 moiety를 전송. 사용 하는 γ-32-P NTP 선구자를 표시 하실 수 있습니다 방사선에 의해 기판 사용률 및 제품 대형의 동시 모니터링. 셀 룰 로스 판에 얇은 층 크로마토그래피 (TLC) 신속한 분리 및 기질과 제품의 민감한 정량화를 허용 한다. 우리는 얇은 층 크로마토그래피 정제 (p) ppGpp 합성의 pyrophosphokinase 활동 분석 결과를 활용 하는 방법을 제시. 이 방법은 주기적인 뉴클레오티드 및 디뉴클레오티드 synthetases의 활동 특성 사용 되었습니다 이전 이며 뉴클레오티드 3 인산 염 유대 hydrolyzes 또는 터미널 전송 하는 모든 효소의 활동을 특성화 하는 데 광범위 하 게 적합 인산 다른 분자를 염 인산 염 기증자 로부터
서문
키 및 pyrophosphokinase (또는 diphospho 키) 효소 기질 분자 뉴클레오티드 3 인산 염 (NTP) 선구자에서 인산 염을 전송. 기판 다른 뉴클레오티드, 아미노산 이나 단백질, 탄수화물, 그리고 지질1포함할 수 있습니다. 효소의 동족 기판 또는 특징이 효소를 유사성에 따라 기판 Bioinformatic 분석 예측 가끔 수 있습니다 하지만 실험 유효성 검사 여전히 필요 합니다. 마찬가지로, 그것의 substrate(s) 및는 그것 catalyzes 형광체 전송 반응 속도 효소의 선호도 및 공동 요인, 억제제, 또는 다른 효소 이펙터의 효과 결정 되어야 합니다 실험적으로. 다른 ATP 소모 효소 세균성 세포질에 존재에 의해 ATP 전조의 소모를 피하기 위해, 양적 활동 분석 실험 순화 된 단백질을 필요로 합니다.
단백질 정화 금속 친화성 크로마토그래피에 의해 문학2,3에 철저 하 게 적용 되었습니다 했습니다. 히스티딘 태그 6 연속 히스티딘 잔류물에는 N-또는 C-말단 재조합 단백질의 추가 구성 된 금속 친화성 크로마토그래피4,,56에 의해 급속 한 정화를 수 있습니다. 이러한 시퀀스는 작은 그들은 때로는 단백질 안정성 및 효소 반응 속도 론7,8변경할 수 있지만 그들은 수정 하 고 일반적으로 단백질 기능에 미치는 영향을 최소화 하는 단백질에 비해. 히스티딘 태그에는 N-와 C-테르미니 같은 단백질에 단백질의 구조를 알지 못하고 예측 하기 어려운 다양 한 효과 가질 수 있습니다. 히스티딘 태그는 일반적으로 복제는 재조합 형 단백질의 중 즉시 6 히스티딘 잔류물, 인코딩할 뇌관을 설계 하 여 3' ATG 시작 codon에 또는 즉시 5', 열려있는 독서 프레임 정지 codon를 하는 동안 통합 된다. 증폭 후 포함 하는 hexahistidine 유전자는 유도할 수 있는 발기인의 통제 벡터에 출혈 이며 표현 하는, 일반적으로 대장균의 실험실 긴장에. 재조합 단백질 다음 고정된 divalent 양이온 (일반적으로 니켈 또는 코발트)9를 포함 하는 선호도 수 지에 고립 될 수 있다. 기본 금속-바인딩 단백질 오염 하는 것은 이미, 경쟁적으로 바인딩된 단백질2배 수량으로 적정 하 여 제거할 수 있습니다. 마지막으로, 대상 단백질은 eluted 이미의 높은 농도와 열에서. 고정된 금속 양이온 수 지에 대 한 몇 가지 상용 소스 고 버퍼 조건 및 이미 농도 대 한 권장 사항을 제공 하는 제조 업체. 차입, 후 단백질 수 수 나트륨 라우릴 황산 polyacrylamide 젤 전기 이동 법 (SDS 페이지)에 의해 분석, dialyzed, 또는 기능 분석에서 즉시 사용.
해제 또는 fluorophore는 흥분 하거나 생성 하는 화학, 두 번째 반응에 ATP 인산 염 결합 가수분해를 커플링 하 여 키 니 아 제 활동을 직접으로 모니터링 하는 여러 가지 방법이 있습니다 하지만이 반응은 여러 이동 부분 그리고 될 수 있는 물류 도전10. 특히 형광체 전송 활동을 측정 하는 가장 간단한 방법은 모니터링 하는 데 직접 상용 γ-32에서 방사선된 인산 염 그룹의 전송-P 비 방사선 기판11 에 NTP 전조 , 12 , 13. 방사선된 기판 및 제품의 혼합물을 분리 하 고 얇은 층 크로마토그래피 (TLC)에 의해 측정할 수 있다. TLC 용 매 (액체 단계) 모 세관 작용에 의해 표면 (단단한 단계)는 용액의 흡착된14되었습니다에서 마이그레이션할 수 있도록 하 여 주어진된 용 매에서 용액의 차동 이동성을 활용 합니다. 있는 작은 고체 단계와 호의 베푸는 상호 작용 부족 용액 마이그레이션됩니다 높은 분자 무게 또는 고체에 대 한 대단한 선호도 용액 보다 더 긴 거리 그들의 초기 위치에서. 인 전송의 시험, 인산 moieties 그들에, 추가 되 고 중성 또는 산 성 pH11,,1214에서 부정적인 이온 충전 추가 분자의 분자량 증가. 이 페이 셀 룰 로스 등 기본 표면에 그들의 기동성을 감소합니다. 신랄 한 칼륨 인산 염 버퍼에서 개발, 페이-셀 루 로스, 각 종 (그림 2, 그림 3)의 정량화를 허용에 모노, 디, 트라이-, tetra-및 pentaphosphate 종의 혼합물을 쉽게 분리 수 있다. 이러한 분석의 관심, 효소를 포함 하는 세포 lysates를 사용 하 여 수행할 수 있습니다 하지만이 다른 kinases, 가수분해, 및 일반적인 ATPases의 활동에 대 한 잠재력을 고갈 기판 및/또는 제품을 포함 합니다. 평가 대 한 양적 체 외에서 효소 활동의, 그것은 관심의 효소 정화 해야 합니다.
Guanosine tetraphosphate (ppGpp) 및 guanosine pentaphosphate (pppGpp)는 ribonucleotide 신호 분자는 파이 인산의 이동에 의해 형성 된 그룹 아데노신 3 인산 염 (ATP) 전조에서 각각, guanosine diphosphate (GDP) 또는 guanosine tetraphosphate (GTP) 기판15. 이러한 단일 ribonucleotide 신호, (p) ppGpp로 총칭 알려진 다양 한 세균성 종15,16에 엄격한 응답으로 알려진 환경 스트레스에 셀 전체 응답 중재. 두 가지 보존된 수준의 효소 촉매 형성 (p) ppGpp15,17 Rel/Spo의 체 (RSH) 효소는 '긴' bifunctional (p) ppGpp 합성/hydrolases 휴식 자리 (p)를 그들의 유사성에 대 한 명명 된 ppGpp 신진 대사 대장균 에서 합성, 가수분해 효소, 및 규제 도메인, 작은 alarmone (SAS) 합성 효소는 짧은 monofunctional synthetases 그램 긍정적인 박테리아15, 에서 독점적으로 포함 하는 효소 17 , 18. 포자 형성 그람 양성 박테리아 클로스 트리 남과 어울리지 않 는 putative RSH 및 SAS 유전자19인코딩합니다. 여기, 선물이 C. 남과 어울리지 않 는 RSH 효소 촉매로 활성 (p) ppGpp 합성 인지 확인 하는 초기 활동 분석 실험.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 유도할 수 있는 Overexpression 히스티딘 태그 단백질의
- Rsh C. 남과 어울리지 않 는 R20291 genomic DNA에서에서 증폭.
- 높은 중 합 효소를 사용 하 고 제조업체의 지침을 따르십시오.
- C. 남과 어울리지 않는 rsh 뇌관을 사용 하 여 증폭
rsh_F (CAGGTACCGGTTATATGCATGATAAAGAATTACAAG)와
rsh_R (CCCTGCAGCTAATGGTGATGGTGATGGTGATTTGTCATTCTATAAATAC), C 터미널 hexahistidine 태그를 소개 하.
참고: KpnI와 PstI 사이트 뇌관 순서에 잘라 굵게 표시 됩니다. - PMMBneo 벡터 소화 그리고 rsh his6 PCR 제품 Pstl 및 KpnI 제한으로 45 분 동안 37 ° C에 사이트.
- 선형화 벡터와 PCR 파편 agarose 젤 전기 이동 법 및 DNA 젤 추출 키트와 함께 후속 정화를 통해 정화.
- 280 nm의 흡수도 (280)는 벡터 및 증폭 된 PCR 제품을 측정 합니다. 방정식을 사용 하 여
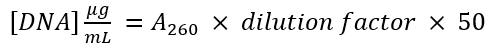 각 DNA 조각의 DNA 농도 결정 하.
각 DNA 조각의 DNA 농도 결정 하.
- PMMBneo 식으로 rsh 를 선.
- 소화 PMMBneo 벡터, 125의 결합 25 ng ng rsh his6 유전자 제품, 리가 버퍼, x 10의 2 μ 및 DNA 리가의 1 μ. Nuclease 무료 물을 사용 하 여 20 μ에 총 볼륨 조절. 16 h 16 ° C에서 품 어.
참고: 결 찰의 효능 그리고 조각 크기에 따라 제조 업체 프로토콜에 따라 조정 해야 합니다. - 대장균 DH5-α 또는 다른 recA- 플라스 미드 유지 보수 부담으로 합자 벡터 제품을 변환. 100 μ g/mL 대 파운드 접시에 변환 된 셀에 대 한 선택 하 고 37 ° c.에서 16-24 h에 대 한 품 어
- 변환 플레이트 (s)에서 4 개의 식민지를 선택 하 고 각 100 μ g/mL 대 DNA 격리를 포함 하는 신선한 접시에 행진. 16-24 h에 대 한 37 ° C에서 새로운 번호판을 품 어 고 후속 단백질 식에 대 한 분리를 선택 합니다.
- 확인 하려면 지역 코딩 그대로 rsh 단백질의 성공적인 결 찰, 선택 선택의 식민지, 물 20 μ로 격리 10 분 동안 95 ° C에가 열 하 고 확실 한 PCR 유전자를 증폭 하는 데 사용 하는 동일한 뇌관을 사용 하 여 템플릿으로 사용 합니다.
- 소화 PMMBneo 벡터, 125의 결합 25 ng ng rsh his6 유전자 제품, 리가 버퍼, x 10의 2 μ 및 DNA 리가의 1 μ. Nuclease 무료 물을 사용 하 여 20 μ에 총 볼륨 조절. 16 h 16 ° C에서 품 어.
- 고수익 단백질 생산을 위한 대장균 BL21 시스템으로 대장균 세균성 플라스 미드 전이 대 한 표준 변환 프로토콜에 따라 확인 된 플라스 미드를 변환.
- 대장균에서 RSH-His6 익스프레스.
- PMMBneo로 변형 대장균 BL21의 단일 식민지를 선택::rsh 50 μ g/mL 대와 파운드 매체의 2 개 mL를 접종 하 고. 37 ° C에서 품 어 12-16 h 250 rpm에서 떨고 있는 동안.
- 고 단백질 식 숙박 문화의 0.5 mL와 함께 50 μ g/mL 대를 포함 하는 파운드 매체의 500 mL 접종
- 셀 밀도는 OD600 0.167 37 ° c.에 도달할 때까지 37 ° C와 인큐베이터 셰이 커에 250 rpm에서 식 문화를 품 어
- 인큐베이터 온도 30 ° C를 감소 시키십시오 하 고 드롭 문화 온도 대 일 분을 기다립니다.
- 0.5 m m의 최종 농도에 이소프로필 β-D-thiogalactoside (IPTG)를 추가 하 여 RSH 식 유도. 유도 250 rpm 동요 30 ° C에서 하룻밤 (h를 위한 16-18) 자리를 차지할 수 있습니다.
- 4 ° c.에 30 분 동안 3,080 x g에서 원심 분리 하 여 펠 렛
참고: 펠 릿 정화 대상 단백질 단백질 수확량 또는 효소 활동의 눈에 띄는 손실 없이 다음 날-20 ° C에서 하룻밤 저장할 수 있습니다.
2. 단백질 정화 니켈 친화성 크로마토그래피에 의해
참고: 직접 세포 lysate를 명확히 후 아래 제공 된 단백질 정화 단계 계속 합니다. 저장 명확히 lysate 4 ° C에서 하룻밤 후속 단백질 정화 단백질 수확량 감소.
- 니켈-Nitriloacetic 산 (Ni-NTA) 수 지의 1 mL를 사용 하 여 중력 열에 단백질 정화.
- 하루 전에 사용 하 여, 재래식 버퍼 (10 mM Tris HCl pH 7.79, 300 m m NaCl, 50mm NaH2포4, 0.5 mg/mL lysozyme, 5 mM MgCl2, 이미 10 m m, 0.25 m m 5 mM phenylmethane sulfonyl DTT의 2 mL와 4 ° C에서 하룻밤 열 equilibrate 불 소 (PMSF), 10% 글리세롤).
- 다음 날 열가지고 명확히 하기 전에 rt 4 ° C에서 lysate와 서 ~ 2-3 h.
참고: 열 평형에 데 열은 열 내에서 형성 하는 기포를 피하기 위해 중요 합니다. - 1.3.6 세포의 용 해 버퍼 (10 mM Tris HCl pH 7.8, 300 m m NaCl, 5 mM MgCl2, 50mm NaH2포4, 10% 글리세롤, 0.5 mg/mL lysozyme, 10mm 이미, 0.25 mM DTT와 5 밀리미터 PMSF)에 단계에서 얻은 펠 릿 resuspend
- 8 x 10 s 간격으로, 30 일시 중지에 대 한 얼음에 셀 sonicate 펄스 사이 s.
- 명확히는 lysate를 microcentrifuge를 사용 하 여 4 ° C에서 30 분 동안 3,080 x g에서 원심 분리.
- 세포의 용 해 버퍼의 동등한 양으로 lysate 준비 다음 적용를 준비한 열 lysate 명확히 하 고 흐름 통해 수집.
- 명확히 lysate 자형 열에 다시 적용 하 고 보조 흐름 통해 수집 합니다.
- 워시 워시 버퍼 1 (10 mM Tris HCl (7.79 pH) 300 mM NaCl, 5 mM MgCl2, 50 m m NaH2포4, 30 mM 이미 10% 글리세롤) 열. 흐름 통해 수집 합니다.
참고: 세척 및 차입 버퍼에서 5 mM MgCl2 의 순화 된 단백질의 효소 활동에 대 한 중요 하다. - 워시 워시 버퍼 2 (10 mM Tris HCl (7.79 pH) 300 mM NaCl, 5 mM MgCl2, 50 m m NaH2포4 와 50 m m 이미) 열. 흐름 통해 수집 합니다.
- 2 mL 차입 버퍼 (10 mM Tris-HCl (7.79 pH) 300 m m NaCl, 50mm NaH2포4, 10% 글리세롤과 75mm 이미)를 적용 합니다. 1 mL의 두 분수에서 흐름 통해 수집 합니다.
참고:이 단계를 수행한 후 열 경우 동일한 단백질 다음 정화 분석 결과에 순화 될 것 이다 4 ° C에서 재래식 버퍼에 저장할 수 있습니다. 열 제대로 저장 하는 경우 최대 3 번까지 사용할 수 있습니다.
- SDS 페이지 순도 대 한 열 분수를 평가 합니다.
- 단백질 정화는 질적 평가, 170 V에서 60 분 동안 4%/10 %polyacrylamide 젤에 모든 열 분수의 20 μ aliquots를 실행 합니다.
- 벤치탑 로커에 부드럽게 락 5 h에 대 한 실 온에서 0.1 %Coomassie 파란 젤 얼룩.
- 40% 메탄올, 벤치탑 로커에 락 실 온에서 하룻밤 10% 빙 초 산에에서 젤 destain
참고: 대표적인 젤은 그림 1에서 그림입니다.
- Dialyze 분수 2 4 ° c.에 하룻밤을 elute
- 200: 1 비율 20 kDa 분자량 컷오프 (MWCO)와 함께 1 mL 투 석 장치를 사용 하 여 투 석 버퍼 (15.7 m m Tris HCl (pH 7.6), 471.9 m m NaCl, 15.69 m m MgCl2, 1.57 m m DTT, 1.5 밀리미터 PMSF, 15.7% 글리세롤)에 대 한 dialyze.
- 280에서 흡 광도 측정 하 여 dialyzed 단백질 샘플의 농도 결정 및 사용 하 여 계산 된 어 금 니 소멸 계수 82085 M-1c m-1 20.
- 사용까지-80 ° C에서 aliquots에 dialyzed 단백질 샘플의 5 μ aliquots를 저장 합니다.
3. 단백질 활동 분석 결과 얇은 층 크로마토그래피에 의해
- 얇은 층 크로마토그래피 접시를 준비 합니다.
- 반응 하기 전에, 준비 polyethyleneimine (페이)-이온된 물에 세척 하 여 셀 룰 로스 판. 두 배 증류수 ~0.5 cm의 깊이 유리 챔버에 접시를 놓습니다.
- 물 접시의 상단에 마이그레이션할 수 있습니다.
참고: 엄격 하 게 필요 하 고, 번호판 없이, 사용할 수 있습니다 하지만 세척 결과 이미지의 선명도 증가 하는가 격판덮개를 세척. 2 개의 수직 방향 더에서 격판덮개를 세척 접시 (그림 2)의 한 구석에 어떤 오염 물질 수 지에 격리 됩니다 보장 합니다. - 유리 챔버 밖으로 접시를 가져오고 건조 하룻밤 (12-18 h) 벤치탑 선반에 두고.
- 어디 샘플 TLC 적용 됩니다 나타내는 부드러운 연필로 건조 접시 한쪽 가장자리에서 2.0 c m를 표시 합니다. 2 μ 샘플에 대 한 적용 샘플 보다 1.0 c m 간격 (그림 2).
참고:이 신호 정량화에 대 한 중요 한 인접 한 자리 사이의 명확한 분리 수 있습니다. 셀 루 로스 수 지는 긁힌, 작은 표면에 연필 자국 용 마이그레이션 방해 하지 않습니다. - 실험을 계획할 때 항상 한 자리에 두고 각 플레이트 사용 하지 않는.
참고:이 샘플 정량화 (그림 2)에 대 한 빈 레인을 제공 합니다. 20 cm TLC 판 19 관광 명소에 대 한 공간이 됩니다.
- 효소 활동 분석 결과
- 50 mM Tris HCl (pH 7.5), 25mm 암모늄 아세테이트, 10mm KCl, 1mm DTT와 0.6 m m ATP 포함 하는 5 x 버퍼 믹스를 준비 합니다.
참고:이 혼합 대량에서 준비 고 나중 사용을 위해 10 μ aliquots에 동결 될 수 있습니다. 여러 freeze-thaw 주기를 믹스를 적용 하지 마십시오. - 3 μ RSH, 버퍼 믹스 1 개, 0.6 m m GDP, 1.2 m m MgCl2를 포함 하는 개별 반응 준비. Γ-32P-ATP 당 반응의 10 μ의 1.0 μCi를 추가 하 고 nuclease 무료 물을 사용 하 여 원하는 볼륨까지 반응 하기. RSH의 뉴클레오티드를 포함 하는 혼합 뿐만 아니라 효소 활동 분석 결과 시작으로 다른 구성 요소, 혼합 후는 RSH 추가 합니다.
참고: 최종 반응 볼륨 timepoints 샘플링의 수에 따라 달라 집니다. 2 μ/timepoint 샘플, 10 μ 각 4 timepoints에 대 한 반응 혼합물의 조립. - 오염 nuclease 활동에서 ATP 가수분해에 대 한 제어, 조립 10 μ 반응 없는 단백질을 포함 하 고 동시에 품 어. T 2 μ 샘플 자리 0 = ATP 단백질 결핍에서 분해 하지는 보장 하기 위해 실험의 끝에.
- 즉시 RSH의 추가, 시 2 μ를 제거 하 고 t로 레이블이 지정 된 페이 셀 루 로스 접시에 자리 = 0 분 샘플.
- 원하는 timepoints에서 2 μ aliquots를 제거 하는 37 ° C에서 반응을 품 어.
참고: 효소 활동 샘플 셀 루 로스 판에 흡착 될 때 중단 됩니다. 마지막 자리는 완전 한 흡착 및 샘플 건조 되도록 개발 전에 접시에 추가 됩니다 후 10-30 분을 기다립니다.
- 50 mM Tris HCl (pH 7.5), 25mm 암모늄 아세테이트, 10mm KCl, 1mm DTT와 0.6 m m ATP 포함 하는 5 x 버퍼 믹스를 준비 합니다.
- 얇은 층 크로마토그래피
- 크로마토그래피 챔버를 1.5 M 1.5 M KH2포4 (pH 3.64) 0.5 cm의 깊이 채워 넣어 라.
참고: 볼륨 필요한 크로마토그래피 탱크의 크기에 따라 달라 집니다. 굽 힘 없이 TLC 판의 삽입을 허용 하도록 충분히 넓은 수준 바닥 유리 컨테이너 개발 탱크 커버의 추가 함께 사용할 수 있습니다. TLC 판 플라스틱 필름으로 덮여 유리 비 커에 개발을 활성화 하는 깨끗 한 razorblade로 좁은 스트립으로 잘릴 수 있다. - 용 매에 접시의 아래쪽 가장자리를 담가. 용 플레이트 (~ 90 분)의 상단에 마이그레이션할 수 있습니다.
참고: 동안 용 마이그레이션 판의 상단에 멈추게 되 고 샘플 또는 분실 될 하지 더 이상 침수 동안 함께 실행, 접시 안 맡겨야 한다 용 매에서 하룻밤. 4h 이상 침수 백업 플레이트에서 분리 하 여 신호 손실 될 수를 발생할 수 있습니다. - 크로마토그래피 탱크에서 접시를 제거 하 고 건조 랙 벤치탑에 배치.
- 공기 접시를 허용 하룻밤 건조 합니다.
참고: 건조 헤어 드라이어를 사용 하 여 가속 수 있습니다. 건조는 젖은 때 어두워질 것 이다 수 지와 완전히 건조 때 사용 하지 않는 접시의 색상으로 돌아갑니다의 색상에 의해 평가 될 수 있습니다. - 접시 건조 후 랩 autoradiography (그림 3)에 의해 분석을 이미징 카세트에 방사성 물질의 양도 피하기 위해 플라스틱 필름에 접시.
- 크로마토그래피 챔버를 1.5 M 1.5 M KH2포4 (pH 3.64) 0.5 cm의 깊이 채워 넣어 라.
- 데이터 분석
- 실 온에서 4 h phosphorimager 카세트를 분리 된 반응을 포함 하 페이 셀 룰 로스 판 노출.
참고: 이것은 신선한 γ-32P-ATP의 표시 농도 사용 하 여 매우 깨끗 한 이미지를 충분 한 노출. Radiolabelled 기판의 낮은 양을 사용 하는 경우 12-16 h에 노출 시간을 늘릴 수 있습니다. - 이미지는 phosphorimager에 카세트입니다.
- 그릴 사각형 을 선택 하 고 약 한 전체 레인과 ATP ppGpp 관광 명소 그 레인 (그림 3에 포함 된 직사각형 ROIs를 그리는 마우스를 사용 하 여 관심 영역 (ROIs) 그릴 이미징 소프트웨어를 사용 하 여 그래픽 사용자 인터페이스와 함께, ).
- 선택, 복사 및 붙여넣기 명령 (또는 사용 중인 이미징 소프트웨어에 따라 해당 명령)를 사용 하 여는 ROIs 각 차선에서 동일 지역 내에서 신호를 측정 하는 보장 하기 위해 다른 차선 내에서 동일한 ROIs를 그릴. 공란으로 사용할 수 있는 사용 하지 않는 차선에서 ROIs를 포함 합니다.
- 분석은 를 사용 하 여 | 도구 | 투자 수익 관리자 | 추가 이미징 소프트웨어의 명령을 모든 페이 셀 룰 로스 판에 그려진 ROIs을 선택.
- 분석은 를 사용 하 여 | 측정을 설정 | 측정 명령, 각 ROI 내에서 신호 강도 계량 및 스프레드시트 (그림 3)으로 측정을 내보낼. 실험 신호에서 빈 ROI 값을 뺍니다.
- ATP와 수식을 사용 하 여 ppGpp에 기인 각 차선 내에서 숨겨진된 신호의 비율을 계산

참고: ROIs 얻을 수 있습니다 및 quantitated 상용 소프트웨어는 phosphorimager와 호환 또는 자유롭게 사용할 수 ImageJ 소프트웨어 (국립 보건원)를 사용 하 여.
- 실 온에서 4 h phosphorimager 카세트를 분리 된 반응을 포함 하 페이 셀 룰 로스 판 노출.
Access restricted. Please log in or start a trial to view this content.
결과
선물이 Clostridium 남과 어울리지 않는 그것의 효소 활동의 평가에서 (p) ppGpp 합성의 친 화력 정화 하는 방법. 그림 1 에 금속 친화성 크로마토그래피에 의해 달성 단백질 정화 방법을 보여 줍니다. 이 정화에서 두 번째 차입 (E2) 분수 dialyzed 고 효소 활동 분석 결과 대 한 사용. 그림 2 에 대 한 준비 하 고 얇은 층 크로마토그?...
Access restricted. Please log in or start a trial to view this content.
토론
여기 우리가 보고 그의 태그 RSH C. 남과 어울리지 않 는에서 정화 활동 정량화 방사선된 얇은 층 착 색 인쇄기를 사용 하 여에 대 한 방법을 제시 하 고. 이 방법은 이전 C. 남과 어울리지 않는로 (p) ppGpp 합성, 뉴클레오티드 있고, 키 니 아 제 diguanylate 있고 효소와 다른 유기 체11,12 포스 효소의 활동을 평가 하기 위해 사용 되었습니다. ,
Access restricted. Please log in or start a trial to view this content.
공개
저자 아무 경쟁 금융 관심사 또는 다른 충돌을 선언합니다.
감사의 말
이 작품은 NIAID 1K22AI118929-01에 의해 투자 되었다. EBP는 오래 된 판도 대학 노퍽, 버지니아, 미국에서 연구의 사무실에서 여름 연구 친교 프로그램 교부 금에 의해 지원 되었다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Inducible overexpression of a histidine-tagged protein | |||
| Phusion polymerase | New England Biolabs (NEB) | M0530L | |
| QIAEX II DNA Gel Extraction Kit | Qiagen | 20021 | |
| KpnI restriction enzyme | NEB | R0142S | |
| PstI restriction enzyme | NEB | R0140S | |
| T4 DNA ligase | NEB | M0202 | |
| NEB® 5-alpha Competent E. coli (High Efficiency) | NEB | C2987I | |
| BL21 (DE3) Competent E. coli | NEB | C2527I | |
| IPTG | Sigma-Aldrich | 10724815001 | |
| JXN-26 centrifuge with JLA 10.500 rotor | Beckman Coulter Avanti | - | |
| Microcentrifuge with D3024/D3024R rotor | Scilogex | - | |
| MaxQ SHKE6000 Incubator | Thermo Scientific | - | |
| Ultrasonic processor | Sonics | VC-750 | |
| Protein purification by nickel affinity chromatography | |||
| Ni-NTA resin | G Biosciences | 786-940/941 | |
| Pierce Disposable Gravity columns, 10 mL | Thermo Scientific | 29924 | |
| 1 mL Spectra/ Por float-A-lyzer G2 dialysis device (MWCO: 20-kD) | Spectrum | G235033 | |
| Mini-Protean Electrophoresis Cell | BioRad | 1658004 | |
| Protein activity assay by thin layer chromatography | |||
| Thin layer chromatograph (TLC) development tank | General Glass Blowing Company | 80-3 | |
| Polyethylenimine (PEI)-cellulose plates (20 cm x 20 cm, 100 μm thickness) with polyester support | Sigma-Aldrich | Z122882-25EA | |
| ATP, [γ-32P]- 3000 Ci/mmol 10mCi/ml lead, 100 μCi | Perkin Elmer | NEG002A | |
| Adenosine 5’-triphosphate (ATP) 100 mM | Bio Basic Canada | AB0311 | |
| Guanosine-5’-diphosphate disodium salt (GDP) | Alfa Aesar | AAJ61646MC/E | |
| Storage phosphor screen | GE Healthcare Life Sciences | BAS-IP TR 2040 E Tritium Screen | |
| Storm 860 phosphorimager | GE Healthcare Life Sciences | - |
참고문헌
- Cheek, S., Ginalski, K., Zhang, H., Grishin, N. V. A comprehensive update of the sequence and structure classification of kinases. BMC Structural Biology. 5 (1), 6(2005).
- Porath, J. Immobilized metal ion affinity chromatography. Protein Expression and Purification. 3 (4), 263-281 (1992).
- Arnau, J., Lauritzen, C., Pedersen, J. Cloning strategy, production and purification of proteins with exopeptidase-cleavable His-tags. Nature Protocols. 1 (5), 2326-2333 (2006).
- Porath, J., Carlsson, J., Olsson, I., Belfrage, G. Metal chelate affinity chromatography, a new approach to protein fractionation. Nature. 258 (5536), 598-599 (1975).
- Smith, M. C., Furman, T. C., Ingolia, T. D., Pidgeon, C. Chelating peptide-immobilized metal ion affinity chromatography. A new concept in affinity chromatography for recombinant proteins. Journal of Biological Chemistry. 263 (15), 7211-7215 (1988).
- Graslund, S., et al. Protein production and purification. Nature Methods. 5 (2), 135-146 (2008).
- Booth, W. T., et al. Impact of an N-terminal Polyhistidine Tag on Protein Thermal Stability. ACS Omega. 3 (1), 760-768 (2018).
- Thielges, M. C., Chung, J. K., Axup, J. Y., Fayer, M. D. Influence of histidine tag attachment on picosecond protein dynamics. Biochemistry. 50 (25), 5799-5805 (2011).
- Chaga, G. S. Twenty-five years of immobilized metal ion affinity chromatography: past, present and future. Journal of Biochemical and Biophysical Methods. 49 (1-3), 313-334 (2001).
- Ma, H., Deacon, S., Horiuchi, K. The challenge of selecting protein kinase assays for lead discovery optimization. Expert opinion on drug discovery. 3 (6), 607-621 (2008).
- Tamayo, R., Tischler, A. D., Camilli, A. The EAL domain protein VieA is a cyclic diguanylate phosphodiesterase. Journal of Biological Chemistry. 280 (39), 33324-33330 (2005).
- Purcell, E. B., McKee, R. W., McBride, S. M., Waters, C. M., Tamayo, R. Cyclic Diguanylate Inversely Regulates Motility and Aggregation in Clostridium difficile. Journal of Bacteriology. 194 (13), 3307-3316 (2012).
- Purcell, E. B., Tamayo, R. Identification and characterization of cyclic nucleotide phosphodiesterases. Methods in Molecular Biology. 1016, 235-243 (2013).
- Ross, P., et al. Control of cellulose synthesis Acetobacter xylinum. A unique guanyl oligonucleotide is the immediate activator of the cellulose synthase. Carbohydrate Research. 149 (1), 101-117 (1986).
- Potrykus, K., Cashel, M. (p)ppGpp: still magical? Annual Review of Microbiology. 62, 35-51 (2008).
- Boutte, C. C., Crosson, S. Bacterial lifestyle shapes stringent response activation. Trends in Microbiology. 21 (4), 174-180 (2013).
- Nanamiya, H., et al. Identification and functional analysis of novel (p)ppGpp synthetase genes in Bacillus subtilis. Molecular Microbiology. 67 (2), 291-304 (2008).
- Gaca, A. O., et al. Basal Levels of (p)ppGpp in Enterococcus faecalis: the Magic beyond the Stringent Response. mBio. 4 (5), (2013).
- Sebaihia, M., et al. The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome. Nature Genetics. 38 (7), 779-786 (2006).
- Gasteiger, E., et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis. Nucleic Acids Research. 31 (13), 3784-3788 (2003).
- Purcell, E. B., et al. A nutrient-regulated cyclic diguanylate phosphodiesterase controls Clostridium difficile biofilm and toxin production during stationary phase. Infection and Immunity. , (2017).
- Mechold, U., Murphy, H., Brown, L., Cashel, M. Intramolecular Regulation of the Opposing (p)ppGpp Catalytic Activities of RelSeq, the Rel/Spo Enzyme from Streptococcus equisimilis. Journal of Bacteriology. 184 (11), 2878-2888 (2002).
- Dalebroux, Z. D., Svensson, S. L., Gaynor, E. C., Swanson, M. S. ppGpp Conjures Bacterial Virulence. Microbiology and Molecular Biology Reviews. 74 (2), 171-199 (2010).
- Bartlett, J. G. Clostridium difficile: progress and challenges. Annals of the New York Academy of Sciences. 1213, 62-69 (2010).
- US Department of Health and Human Services. , Centers for Disease Control and Prevention, US Department of Health and Human Services. Atlanta, GA. (2013).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유