Method Article
Verwendung von Kapillarelektrophorese Immunoassay zur Suche nach potenziellen Biomarkern der Amyotrophen Lateralsklerose in menschlichen Thrombozyten
In diesem Artikel
Zusammenfassung
Blutbasierte Biomarker für neurodegenerative Erkrankungen sind für die Durchführung groß angelegter klinischer Studien unerlässlich. Ein zuverlässiger und validierter Bluttest sollte ein kleines Probenvolumen sowie eine weniger invasive Probenahmemethode erfordern, erschwinglich und reproduzierbar. Dieses Papier zeigt, dass der Immunoassay für kapillare Elektrophorese mit hohem Durchsatz Kriterien für die entwicklung potenzieller Biomarker erfüllt.
Zusammenfassung
Kapillarelektrophorese Immunoassay (CEI), auch bekannt als Kapillar-Western-Technologie, wird zu einer Methode der Wahl für das Screening von krankheitsrelevanten Proteinen und Medikamenten in klinischen Studien. Reproduzierbarkeit, Empfindlichkeit, geringer Probenvolumenbedarf, Multiplexing-Antikörper für die Mehrfach-Protein-Etikettierung in derselben Probe, automatisierte Hochdurchsatzfähigkeit zur Analyse von bis zu 24 Einzelproben und kurzes Zeitaufwand machen CEI vorteilhaft gegenüber dem klassischen Western Blot Immunoassay. Es gibt einige Einschränkungen dieser Methode, z. B. die Unfähigkeit, ein Gradientengel zu verwenden (4%–20%) Matrix, hoher Hintergrund mit unraffinierten biologischen Proben und kommerzieller Nichtverfügbarkeit einzelner Reagenzien. Dieses Papier beschreibt eine effiziente Methode zum Ausführen von CEI in einer Mehrfach-Assay-Einstellung, zur Optimierung der Proteinkonzentration und der primären Antikörpertitration in einer Assayplatte und zur Bereitstellung benutzerfreundlicher Vorlagen für die Probenvorbereitung. Ebenfalls beschrieben sind Methoden zur Messung von Pan TDP-43 und phosphoryliertem TDP-43-Derivat in Thrombozytosollysatcytosol als Teil der Initiative zur blutbasierten Biomarkerentwicklung bei neurodegenerativen Erkrankungen.
Einleitung
Das gesamtziel von CEI, wie hier beschrieben, ist es, ein aktualisiertes stepwise Protokoll für die Analyse von Zielproteinen in menschlichen Thrombozyten bereitzustellen. Die Zuordnung eines blutbasierten Signaturmoleküls ist eine der wichtigsten Aufgaben auf dem Gebiet der Biomarkerentwicklung bei menschlichen neurodegenerativen Erkrankungen wie Alzheimer(AD), amyotropher Lateralsklerose (ALS), frontotemporaler Lobar Degeneration (FTLD), Parkinson-Krankheit (PD), Inklusionskörpermyositis (IBM) und andere Protein-Aggregation relevante pathologische Bedingungen. Der Nachweis von winzigen Mengen solcher Signaturproteine in großen Blutmengen mit vielen störenden Wirkstoffen ist eine Herausforderung. Daher sind die Spezifität, Empfindlichkeit, die Fähigkeit, eine große Anzahl von Proben zu behandeln, und die Reproduzierbarkeit der ausgewählten Methode entscheidend.
Menschliche Thrombozyten können als Milieu dienen, um potenzielle Biomarkerproteine für neurodegenerative Erkrankungen zu identifizieren und zuzuweisen. Thrombozyten bieten die Möglichkeit, als Ersatz-Primärzellmodell zu dienen, das einige Merkmale der neuronalen Zellen1,2,3widerspiegelt. Es gibt bestimmte Merkmale, die Thrombozyten zu einem der bevorzugten Mittel machen, um Biomarker-Kandidatenproteine und ihre chemischen Derivate zu analysieren. Erstens können Blutplättchen leicht mit einem weniger invasiven Ansatz erworben werden, indem Blut von Spendern (d. h. Venipunktur) oder in großen Mengen von gemeinschaftlichen Blutbanken gesammelt wird. Zweitens können Blutplättchen leicht aus dem Vollblut isoliert werden, mit minimalen Vorarbeiten in minimal ausgestatteten Laboratorien4,5. Drittens haben Thrombozyten keine Kerne; Daher sind sie eine gute Modellzelle, um Veränderungen im Stoffwechsel ohne Transkriptionsregulation zu untersuchen. Viertens ist der Biomolekülgehalt der Blutplättchen verkapselt; Daher schützt die Thrombozytenmikroumgebung ihren Inhalt vor serumstörenden Substanzen (d. h. Proteasen). Fünftens kann mit Thrombozyten angereichertes Plasma 7–8 Tage lang bei Raumtemperatur gelagert werden, ohne die Stoffwechselaktivität zu verlieren. Daher bieten Thrombozyten ein Arbeitsmodell, bei dem externe Faktoren minimiert und kontrolliert werden.
Herkömmliche Immunoassay-Techniken wie Immunoblotting (z. B. Western Blotting) und enzymgebundener Immunsorbent-Assay (ELISA) werden in der spezifischen Proteinanalyse häufiger eingesetzt. Diese beiden Methoden haben jedoch mehrere Nachteile, darunter mehrere Assay-Schritte, Anforderungen an gefährliche Chemikalien und Reagenzien, große Probengröße, Probleme mit der Reproduzierbarkeit des Asses und Inter-Run-Datenvariabilitäten. Diese veranlassten die Entwicklung einer Methode, die mit weniger Schritten einfacher und in relativ kurzer Zeit erreichbar ist. Obwohl die klassische Western-Blot-Technik eine beliebte Labormethode bleiben wird, werden ihr mehrstufiges Verfahren, Vorräte, giftige Abfälle (d. h. Acrylamid, Methanol usw.) und die Testzeit bei quantitativer Durchführung mit hohem Durchsatz immer weniger wünschenswert. Proteinanalyse.
Ein automatisierter CEI-Ansatz entwickelt sich allmählich zu einer Methode der Wahl für Laboratorien, die Protein-Assays mit hohem Durchsatz durchführen6. CEI eliminiert die Notwendigkeit von Gelen, Gelelektrophoresegeräten, Membranen, Elektrophorese- und Elektrotransfergeräten und mehr physikalischen Handhabungsbeteiligungen. Wenn gut ausgelegt, sollte ein CEI-Test innerhalb von ca. 3,5 h abgeschlossen werden, einschließlich quantitativer Datenanalyse, Elektropherogramm in Publikationsqualität und Graphiken mit statistischer Analyse. Eine weitere Überlegenheit des CEI-Systems ist seine Anforderung von 10x–20x weniger Proteinkonzentration, was es ideal für den Einsatz in menschlichen Proben in klinischen Studien7,8macht.
Der kritischste Teil von CEI ist die Optimierung der Assay-Bedingungen für jeden Antikörper, der von verschiedenen Anbietern gekauft wurde, die Art des Antikörpers (monoklonal vs. polyklonal), optimale Proteinkonzentrationen, Probenvorbereitung, Probendenaturierungstemperatur und Elektrophoresespannung, die auf die Kapillaren angewendet wird. Wir haben eine Single-Assay-Formatoptimierungsmethode für das CEI entwickelt, die vor neuen Assays implementiert werden sollte, was Zeit und Ressourcen spart. Diesem Optimierungsschritt folgt eine automatisierte quantitative Bewertung sowohl des totalen als auch des phosphorylierten Derivats des Transaktivierungsreaktions-DNA/RNA-Bindungsproteins (TARDP). Aufgrund seiner Größe (43 kDa) wird das Akronym TDP-43 in diesem Papier verwendet. Hierbei wird das von ALS-Patienten gewonnene TDP-43-Protein in humanem Thrombozytenlysat bewertet, um den prädiktiven Phosphorylierungswert (PPV) als potentiellen prognostischen Biomarker zu entwickeln.
TDP-43 ist ein neuer potenzieller Biomarker-Kandidat für ALS. TDP-43 ist ein allgegenwärtiges Protein in allen nukleierten Zellen; daher wurden die Funktionen von TDP-43 bei verschiedenen normalen zellulären Ereignissen und bei neurodegenerativen Erkrankungen9,10,11,12,13,14untersucht. Obwohl TDP-43 ein Kernprotein15ist, hat es die Fähigkeit, zwischen dem Kern und Zytoplasma aufgrund des Vorhandenseins von nuklearen Lokalisierung und nuklearen Exportsequenzen16,17,18,19zu pendeln. Cytoplasmaic TDP-43 ist an verschiedenen zellulären Ereignissen beteiligt, wie mRNA-Stabilität und -Transport, die Stressreaktion, mitochondriale Funktion, Autophagominasie20. Über die Rolle von phosphorylierten Derivaten von TDP-43 ist jedoch nicht viel bekannt als ihre Beteiligung an der Pathogenese der neurodegenerativen Erkrankung21.
Dieses Protokoll veranschaulicht, wie die Assay-Bedingungen optimiert werden, um den Inhalt von TDP-43 und seiner phosphorylierten Derivatderivate in Thrombozyten nach dem CEI-Ansatz zu analysieren. Da phosphoryliertes TDP-43 nicht kommerziell erhältlich ist, wird vorgeschlagen, einen prädiktiven Phosphorylierungswert (PPV) zu verwenden, um TDP-43-Profile bei ALS-Patienten zu bewerten. Dieses CEI-System verwendet ein kleines Volumen an Probenmischungen (2,5–3,0 l pro Kapillare). Das Gesamtvolumen des Assayvolumens beträgt 8,0 l pro Kapillare auf der Grundlage des Herstellerprotokolls; Daher können Forscher eine Probenmischungspräparation für zwei getrennte Durchläufe verwenden. Der Hersteller hat das Assay-Protokoll so konzipiert, dass Pipettierfehler minimiert, wenn nicht gar vollständig beseitigt werden. Die 24 einzelnen menschlichen Thrombozytenlysat-Probengemische werden in Halbvolumina (d. h. 2,5–3,0 l pro Probe) unterteilt und nacheinander durch zwei verschiedene Antikörper innerhalb von 7 h analysiert. Das hier beschriebene CEI-System bietet eine wünschenswerte Modalität für den Test mit hohem Durchsatz. Anwender müssen Antikörper verschiedener Anbieter und Probenvorbereitungsmodalitäten für das Zielprotein testen, bevor sie ein großangelegtes Screening durchführen.
Protokoll
Alle Protokolle über die Verarbeitung menschlicher Thrombozyten folgen den Richtlinien der IRB-Ausschüsse der University of Kansas Medical Center und der Kansas City University of Medicine and Biosciences.
1. Herstellung von Puffern und Reagenzien
HINWEIS: Bereiten Sie alle Proben gemäß den Richtlinien des Herstellers vor. Tragen Sie während dieses Vorgangs persönliche Schutzausrüstung (Labormäntel, Handschuhe und Schutzbrillen).
- Herstellung des Citrat-Waschpuffers durch Kombination von 0,941 g Saccharose (11 mM Endkonzentration), 6,4 ml 5 M NaCl (128 mM Endlauf), 5,4 ml mit 0,2 m NaH2PO4 (4,3 m Finale), 9,4 ml mit 0,2 m Na2PHO4 (7,5 mM Finale), 0,352 g Natriumcitrat (4,8 mM Finale) und 0,115 g Zitronensäure (2,4 mM Finale). Stellen Sie das Gesamtvolumen auf 250 ml mit ddH2O. Filter durch eine 0,45 m Filterscheibe und stellen Sie den pH-Wert auf 6,5 ein. Bis zu 1 Jahr bei 4 °C lagern. Bringen Sie die Lösung Raumtemperatur (RT) vor der Verwendung22.
- Bereiten Sie den Bruchpuffer vor, indem Sie 250 mM Saccharose, 1 mM EDTA und 10 mM Tris-Cl (pH 7.4) in einem 100 ml Endvolumen kombinieren. Bis zu 1 Jahr bei 4 °C lagern. Fügen Sie 2 l Phosphatase-Inhibitoren-Cocktail (1:1000 final) und 1 l Protease-Inhibitoren-Cocktail (1:2000 end) in 2 ml Bruchpuffer ein. Auf Eis halten, bis es verwendet wird. Verwerfen Sie den nicht verwendeten Bruchpuffer.
2. Thrombozytenisolierung
- Sammeln Sie 8–10 ml menschliches Blut in Gelbkappen-Blutentnahmeröhrchen, das Säure-Citrat-Dextrose (ACD) Lösung enthält (75 mM Trinatriumcitrat, 124 mM Dextrose und 38 mM Zitronensäure, pH = 7,4; ACD:Blut = 1:9). Mischen Sie den Rohrinhalt 5x–6x invertieren von Hand.
- Zentrifugieren Sie die Rohre bei 200 x g in einem schwingenden Schaufelrotor für 20 min bei RT.
- Sammeln Sie plättchenreiches Plasma (PRP) (ca. 3–4 ml) in ein 15 ml konisches Bodenrohr und lassen Sie etwa 0,5 ml des PRP aus dem buffy Mantel (hazy-looking fraction) zu vermeiden Kontamination. Wenn eine Kontamination der roten Blutkörperchen auftritt, wiederholen Sie diesen Schritt.
- Zentrifugieren Sie die PRP-Proben bei 1.200 x g für 15 min bei RT.
- Waschen Sie die Plättchenpellets (P1) durch sanfte Resuspension in 1 ml Citrat-Waschpuffer und Pellet durch Zentrifugation bei 1.200 x g für 15 min bei RT.
- Sparen Sie das reine Plättigepellet. Entsorgen Sie den Überstand.
- Die Thrombozytenpellets in 600 l des Bruchpuffers, der Inhibitorcocktails enthält, wieder aufhängen.
- Beschallen Sie die Thrombozytenaufhängung mit einem Beschallungsmittel. Legen Sie die Probe in einen Mini-Eiskübel. Stellen Sie den Beschallungsgerät im kontinuierlichen Modus auf Einstellung 3 für 20 s ein.
HINWEIS: Achten Sie darauf, die Sonde mit 10% Bleichmittel gefolgt von destilliertem Wasser zu reinigen. - Zentrifugieren Sie die beschallten Proben bei 20.000 x g für 30 min bei 4 °C, um membranöse Fraktionen zu entfernen. Aliquot Überräuer in 60 l und lagern bei -80 °C. Vermeiden Sie wiederholte Auftau-/Gefrierzyklen für die zytosolischen Thrombozytosolfraktionen.
3. Vorbereitung auf CEI
ANMERKUNG: 100 l menschliches Thrombozytenlysat wurden von ALS-Patienten (n = 8–10) kombiniert, und gesunde Probanden (n = 8–10) wurden getrennt gepoolt und für die Assay-Optimierung verwendet.
- Ausfüllen intern generierter Vorlagen für das CEI-Layout (Tabelle 1) und die Probenvorbereitung (Tabelle 2). Die Probenmischungsvorbereitungstabelle ist dynamisch und berechnet automatisch, wie viel Volumen aus der Quelle entfernt werden muss.
HINWEIS: Bei Bedarf wird das in der dynamischen Tabelle-2 eingegebene 0,1-X-Probenpuffervolumen automatisch berechnet. - Voretikettieren Sie 25 0,2 ml PCR-Röhren mit Kapillaren #1-#25 und legen Sie sie in ein PCR-Rack. Auf Eis gesetzt.
- Voretikett0,6 ml Mikrozentrifugenröhrchen: eine für jeden zu verwendenden Primärantikörper und verdünnte (falls erforderlich), eine für den 0,1-fachen Probenpuffer, eine für das Luminol-S/Peroxid und eine für jede zu verdünnende Probe (falls erforderlich). Legen Sie sie auf Eis in Rohrrack.
- Nehmen Sie den Probenpuffer, den Waschpuffer, eine Platte und eine Patrone heraus, die im CEI-Trennmodul 12–230 kDa-Masterkit enthalten ist.
- Aus dem 4 °C-Kühlschrank den Antikörperverdünnungspuffer, primärantikörper, sekundäre Antikörper, Luminol, Wasserstoffperoxid und Standardpackung herausnehmen. Legen Sie alle Reagenzien auf Eis, mit Ausnahme der Standardpackung, die bei RT verbleibt.
HINWEIS: Die Reagenzien aus den Standardpackungen sind lyophilisiert und mit einer Folienabdeckung versiegelt. Diese sollten kurz mit einer Minizentrifuge vor dem Öffnen gesponnen werden, um den Produktverlust zu reduzieren. Zum Öffnen können die Reagenzrohre entweder durch eine Pipettenspitze durchbohrt oder aus der Ecke gezogen werden. - Um die 400 mM DTT vorzubereiten, fügen Sie 40 l entionisiertes Wasser in das Klarrohr, das den DTT enthält.
- Um 40 l fluoreszierenden 5x Master-Mix vorzubereiten, fügen Sie 20 l des 10-fachen Probenpuffers und 20 l der vorbereiteten 400 mM DTT-Lösung in das im Kit enthaltene rosa Rohr hinzu.
- Zur Vorbereitung der biotinylierten Leiter fügen Sie dem im Kit bereitgestellten weißen Rohr 16 l entionisiertes Wasser, 2 l 10x Probenpuffer und 2 l der vorbereiteten 400 mM DTT-Lösung hinzu. Mischen Sie vorsichtig und übertragen Sie in ein 0,2 ml PCR-Rohr für die Denaturierung.
- Bereiten Sie 0,1x Probenpuffer vor, indem Sie einem 0,6 ml Mikrozentrifugenrohr 1,5 l 10x Probenpuffer und 148,5 l entionisiertes Wasser hinzufügen. Wirbel zu mischen und auf Eis zu legen.
- Bereiten Sie die gewünschten Antikörperverdünnungen vor. Fügen Sie Antikörperverdünner in Volumen hinzu, die jedem vorbeschrifteten Mikrozentrifugenrohr zugewiesen sind. Wenn Volumes identisch sind, verwenden Sie die Reverse Pipetting-Technik23; wenn nicht, spülen Sie die Pipettenspitze vor dem Dosieren vor.
HINWEIS: In diesem Test wurden a-TDP-43 Pan Antikörper und a-p(S409/410-2) TDP-43 Antikörper verwendet. Anti-ERK-Antikörper wurde für eine interne Kontrolle verwendet, um sicherzustellen, dass Assay-Komponenten funktionieren. - Führen Sie die umgekehrte Pipettierung für die Verdünnung von Antikörpern durch, wie unten beschrieben. Alternativ finden Sie weitere Informationen in der Literatur24.
HINWEIS: Reverse Pipetting-Technik wird bevorzugt, wenn kleine sequenzielle Volumina von Lösungen23. Diese Technik bietet einige Vorteile: (i) Bereitstellung eines präzisen Volumens, (ii) Beseitigung des Reagenzschäumens in der Spitzenöffnung und (iii) ideal für kleine Volumen (<5 l) Reagenzien, viskose Lösungen, Tensidlösungen und Lösungen mit hohem Dampfdruck.- Legen Sie eine richtige Spitze in eine Pipette und drücken Sie den Kolben bis zum zweiten Anschlag (Schritt-2). Tauchen Sie die Pipettenspitze ein paar Millimeter in die Lösung ein. Lösen Sie den Kolben langsam los, um die Pipettenspitze mit der Lösung zu füllen, während die Spitze noch in die Lösung eingetaucht ist. Entfernen Sie die Spitze aus der Lösung und berühren Sie vorsichtig gegen die Kante des Reagenzbehälters, so dass überschüssige Flüssigkeit, die auf der Außenseite der Spitze verbleibt, entfernt wird.
- Geben Sie die Lösung, indem Sie den Kolben bis zum ersten Stopp nach unten drücken (Schritt-1). Geben Sie die restliche Lösung nicht in die Spitze.
- Entleeren Sie die restliche Lösung in der Spitze zum Reagenzbehälter, indem Sie den Kolben auf den zweiten Anschlag drücken (Schritt-2). Lassen Sie den Kolben in die bereite Position für den nächsten Pipettierschritt.
- Fügen Sie den erforderlichen Antikörper in Volumen hinzu, die zu jedem vorbeschrifteten Mikrozentrifugenrohr bestimmt sind (Tabelle 1) Spülen Sie die Pipettenspitze nicht vor: Fügen Sie sie direkt in das Verdünnungsmittel ein und spülen Sie die Spitze mehrmals, um Antikörper zu entfernen. Legen Sie die Rohre auf Eis.
- Um den CEI-Probenmix vorzubereiten, führen Sie die unten aufgeführten Schritte für PCR-Röhren mit der Bezeichnung Cap 2 bis Cap 25 aus: Dies ist in der gleichen Reihenfolge, wie sie in Tabelle 1angezeigt wird.
- Öffnen Sie alle Röhren, fügen Sie jedem Rohr mit einer Reverse Pipettiertechnik 1,6 L-Aliquots fluoreszierenden 5x-Probenpuffer hinzu, und schließen Sie dann jede PCR-Röhre nach Zugabe des 5-fachen Puffers, um den Probenverlust zu minimieren.
- Öffnen Sie alle Rohre, fügen Sie 0,1x Probenpuffer in Volumen hinzu, die in Tabelle 2 zu jedem Rohr angegeben sind, und schließen Sie dann unmittelbar danach. Wenn Volumes identisch sind, verwenden Sie eine Reverse Pipetting-Technik. Wenn nicht, vorspülen Pipetspitze vor, bevor 0,1x Probenpuffer.
- Öffnen Sie alle Röhren, fügen Sie die Proteinprobe in den in Tabelle 2 bezeichneten Mengen zu jeder Röhre hinzu, und schließen Sie sie unmittelbar danach. Wenn Volumes identisch sind, verwenden Sie die Reverse Pipetting-Technik. Wenn nicht, vorspülen Pipetspitze vor, bevor 0,1x Probenpuffer.
- Kurz zentrifugieren Sie alle PCR-Röhren in einer Tischzentrifuge (13.000 x g für 30 s), Flick/Wirbel-PCR-Röhren zum Mischen, dann wiederholen Sie die Zentrifugation.
- Übertragen Sie alle PCR-Rohre mit einem beheizten Deckel in den Thermocycler. Denaturproben bei definierter Temperatur und Dauer (d. h. 95 °C für 5 min; 70 °C für 10 min).
HINWEIS: Die Denaturierungstemperatur und -dauer müssen für das Zielprotein optimiert werden. - Wiederholen Sie Schritt 3.12.4.
- Bringen Sie alle PCR-Rohre in das Rohrgestell zurück und legen Sie sie auf Eis.
- Bereiten Sie während des Denaturierungsschritts die Entwicklungslösung (1:1 luminol-S:Peroxid-Lösung) vor, und fügen Sie dann 200 l L-L-Luminol-S und 200 l Peroxid hinzu. Auf Eis legen.
- Um eine CEI-Fertigplatte mit der oben herbeigefertigten Probe zu beladen, geben Sie Reagenzien und Proben in die im Assay-Layout gezeigte Assay-Platte(Abbildung 1). Vermeiden Sie das Einführen von Luftblasen.
HINWEIS: Wenn Volumes und Lösung identisch sind, verwenden Sie eine Reverse Pipetting-Technik. Wenn nicht, spülen Sie die Pipettenspitze vor dem Dosieren vor und vertreiben Sie den Rest nicht mit dem zweiten Tabstopp an der Pipette in die Platte. Ein 12–230 kDa-Trennmodul kann eine farbcodierte Plattenladeführung enthalten. Platzieren Sie diese Anleitung unter die Platte, während Sie reagenzien und Proben in den Brunnen geben, was bei der Probenbelastung optisch hilft. Die Plattenladeanleitung kann auch von der Unternehmenswebsite heruntergeladen werden.- In Zeile E fügen Sie jedem Brunnen 15 L L L:Peroxid-Mischung hinzu.
HINWEIS: Im Idealfall bereiten Sie dieses Reagenz kurz vor der Anwendung vor und fügen Sie es jedem Brunnen hinzu. Wenn dies nicht bequem ist, darf diese Mischung nicht mehr als 30 min vor der Plattenbeladung zubereitet werden. - In Zeile D, um gut D1, fügen Sie 10 l Streptavidin-HRP hinzu.
- In Zeile D, zu Den Brunnen D2–D25, fügen Sie 10 l des bezeichneten sekundären Antikörpers hinzu.
- In Zeile B, zu jedem Brunnen, fügen Sie 10 l Antikörperverdünnung.
- In Zeile C, bis gut C1, fügen Sie 10 l Antikörperverdünnung hinzu.
- In Zeile C, zu den Brunnen C2–C25, fügen Sie 10 L des bezeichneten primären Antikörpers hinzu.
- In Zeile A, um gut A1, fügen Sie 5 l biotinylierte Leiter aus PCR-Rohr #1.
- In Zeile A, zu den Bohrungen A2–A25, fügen Sie 3 l der Probe, PCR-Röhren #2–#25 in entsprechende Bohrungen #2–#25 hinzu.
- Fügen Sie jedem vorgesehenen Waschpuffer 500 l Waschpuffer hinzu.
- Zentrifugieren Sie die Platte für 5 min bei 1.000 x g bei RT.
- In Zeile E fügen Sie jedem Brunnen 15 L L L:Peroxid-Mischung hinzu.
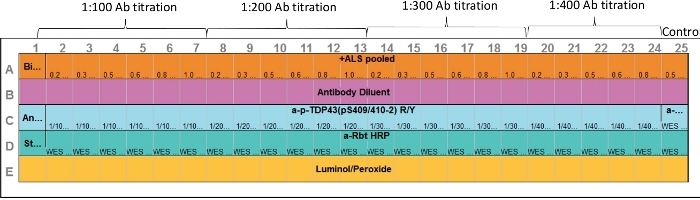
Abbildung 1: Assay-Layout. Sowohl primärer Antikörper als auch Zielproteinprobenoptimierung kann in einem Test durchgeführt werden. Die Kapillaren 2–7, 8–13, 14–19 und 20–24 stellen verschiedene Proteinkonzentrationen und den primären Antikörperbereich dar. Kapillare 25 stellt eine positive Kontrolle dar. Anti-ERK-Antikörper wurde verwendet; eine geeignete Positivkontrolle kann jedoch einbezogen werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Durchführung des CEI auf Platte 1
- Schalten Sie zuerst den CEI- (Table of Materials)-Analysator ein, und schalten Sie dann den Computer ein. Öffnen Sie die Software (Tabelle der Materialien)
- Verbinden Sie den Analysator mit dem Online-System (Tabelle der Materialien). Dies ist ein notwendiger Schritt, um die Laufdaten für Fehlerbehebungszwecke und Datenwiederherstellung zu sammeln.
- Klicken Sie im Menü oben links auf Instrument und dann auf Verbinden. Wählen Sie die Seriennummer des Instruments aus, die als Popup-Menü angezeigt wird. Klicken Sie auf Verbinden.
- Wählen Sie die Registerkarte "Assay" und wählen Sie New Assay oder wählen Sie eine gespeicherte Vorlage aus.
- Geben Sie Assay-Parameter ein (Tabelle 1) oder ändern Sie die Vorlage derzeit. Speichern Sie den Dateinamen und den Speicherort.
- Stellen Sie sicher, dass die blinkende blaue Farbanzeige im Analyzer durchgezogen blau bleibt.
- Berühren Sie den silbernen Metallknopf auf der Oberseite der orangen Tür, um sich zu öffnen.
- Entfernen Sie die Kapillarpatrone vorsichtig aus der Verpackung. Setzen Sie die Kapillarpatrone gemäß dem Herstellerprotokoll ein. Bei richtiger Installation schaltet sich das Innenlicht in "blau" um.
- Entfernen Sie die Schutzdichtung von der Assayplatte. Beobachten Sie die vorgefüllten Brunnen für Luftblasen visuell. Wenn beobachtet, pop sie mit einer kleinen Pipettenspitze (Langwelle P10 Pipettenspitze funktioniert gut).
- Laden Sie den Plattenhalter, indem Sie die Assayplatte platzieren und schließen Sie die Tür. Klicken Sie im Computer auf Starttaste.
- Eisschale mit temperaturempfindlichen Reagenzien und Proben bei 4 °C in die Dunkelheit geben, bis sie bereit sind, eine zweite fertiggefüllte Platte vorzubereiten.
- Reagenzien/Versorgungen bei Raumtemperatur für die zweite Platte abgeben.
5. Durchführung des CEI auf Platte 2
HINWEIS: Diese Platte ist für die Analyse der phosphorylierten TDP-43-Spiegel eingestellt.
- Entfernen Sie die bei 4 °C gelagerte Eisschale und legen Sie sie 1 h vor der geschätzten Fertigstellungszeit der ersten Platte auf die Bank. Holen Sie sich eine zweite Platte und Patrone.
- Bereiten Sie alle Antikörperverdünnungen vor, die für den zweiten Lauf benötigt werden, und lagern Sie sie auf Eis. Bereiten Sie frische 1:1 Luminol-S:Peroxid-Lösung (Schritt 3.12.8 oben).
- Den Probenmix und die Reagenzien, die zum Beladen der fertiggefüllten Platte benötigt werden, remixen und kurz neu zentrieren. Laden Sie die zweite Platte gemäß Abbildung 1. Laden Sie a-p(S409-410-2) TDP-43 Antikörperlösung in Reihe C-Bohrungen.
- Wenn der erste Durchlauf abgeschlossen ist, entsorgen Sie die erste Platte und die erste Patrone. Entfernen Sie die Patrone und legen Sie sie zur Entsorgung in einen scharfen Behälter. Bewahren Sie die Aufkleber von der Platte und der Patrone zu Referenzzwecken auf.
- Schließen Sie die Softwaredatei, und wählen Sie dieselbe Vorlage erneut aus. Die Software wird sich die Einstellungen aus dem vorherigen Lauf merken. Nehmen Sie bei Bedarf Änderungen an der Anmerkung vor (d. h. ändern Sie den primären Antikörper).
- Wiederholen Sie die Schritte 4.8–4.10. Alle 12–230 kDa Master Kit Trennmodul-Reagenzien und -Zubehörteile
- Nachlassen von CEI-Probenmischungen, Antikörperverdünnungen, 0,1x Probenpuffer und Luminol-S:Peroxid-Mischungen gemäß den universitären Vorschriften entsorgen.
6. Datenanalyse
- Stellen Sie nach Abschluss der Ausführung sicher, dass die folgenden Qualitätsprüfungen durchgeführt werden.
- Wählen Sie in der Software das Symbol Standards anzeigen und die Registerkarte Diagrammansicht aus. Überprüfen Sie alle 25 Kapillaren auf die Spitzenausrichtungen auf interne fluoreszierende Markergrößen. Korrigieren Sie die Fehlausrichtungen, indem Sie Force Standard auswählen oder mit der rechten Maustaste auf die falsche Spitze klicken und dann Not a Standardauswählen. Führen Sie diese Prüfung für jede neue Kapillare durch.
- Klicken Sie auf Samples und das Symbol Einzelansicht. Wählen Sie Kapillar#1 (biotinylierte Leiter) in der Registerkarte Experiment. Überprüfen Sie die Spitzenausrichtungen zu Molekulargewichtsmarkern. Klicken Sie in der Graph-Ansicht auf die Spitze und wählen Sie Peak entfernen, wenn die Software eine falsche Auswahl der Spitze vornimmt.
HINWEIS: Die Biotinylleiter 12–230 KDa zeigt beispielsweise Größenspitzen bei 12 kDa, 40 kDa, 66 kDa, 116 kDa, 180 kDa und 230 kDa. Die Größe der Stichprobenspitzen ist falsch, wenn dieser Schritt nicht ausgeführt wird und ungenaue Ergebnisse generiert. - Sehen Sie sich den elektrophoretischen Film an und beachten Sie, ob während des Laufs eine ungewöhnliche Migration aufgetreten ist.
- Ableitungsdaten (z. B. Peaks-Tabelle, einschließlich Molekulargewicht, Spitzenfläche, Spitzenhöhe und Signal-Rausch [S/N]), je nach Bedarf für weitere Berechnungen. In der oberen rechten Ecke des Diagrammfensters befinden sich Diagrammanmerkungswerkzeuge, um weitere Informationen zum Diagramm bereitzustellen.
Ergebnisse
Optimierung der zytosolischen Proteinkonzentration und der primärantikörpertitration
Es ist wichtig, einen linearen dynamischen Bereich von zytosolischen Thrombozytosolproteinen im Test zu etablieren, da Signaländerungen direkt proportional zu Proteinveränderungen im Thrombozytosol sind. Die Verwendung von ganzem Thrombozytenlysatgemisch im Assay kann die Signalintensität der Zielproteine (TDP-43 und P(S409-412) TDP-43) reduzieren und zu einem hohen Hintergrundsignal beitragen. Daher wurde bei diesem Test der klare Überstand (zytosolische Fraktion) nach dem Bruch der Thrombozyten verwendet (Abbildung 2).
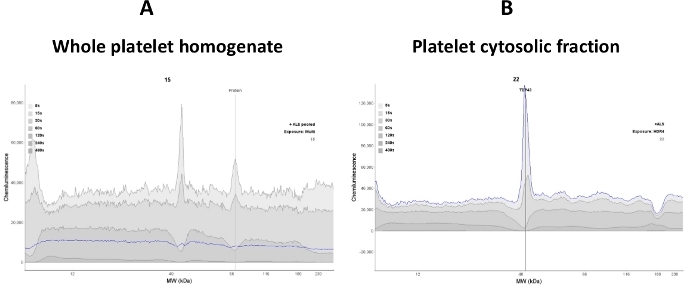
Abbildung 2: Die Signalklarheit hängt von der Probenqualität ab. (A) Das homogenate Blutplättliche Glysat stört die Anti-TDP-43-Antikörperbindung; daher wurde ein lautes Elektropherogramm beobachtet. (B) Die zytosolische Thrombozytosolfraktion wurde aus der Zentrifugation des gesamten Lysats (16.000 x g für 30 min) gewonnen. Die meisten membranösen Proteine wurden entfernt; Daher wurde die Anti-TDP-43-Antikörperbindung an das TDP-43-Protein verbessert (blaue Linienspur). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ein linearer Dynamikbereich für die Thrombozytosolproteinkonzentration wurde bei 0,2–0,8 mg/ml festgelegt. Es wurde eine Assay-Vorlage angenommen, damit sowohl die Proteinkonzentration als auch die primäre Antikörpertitration in einem Test durchgeführt werden konnten (Abbildung 3).
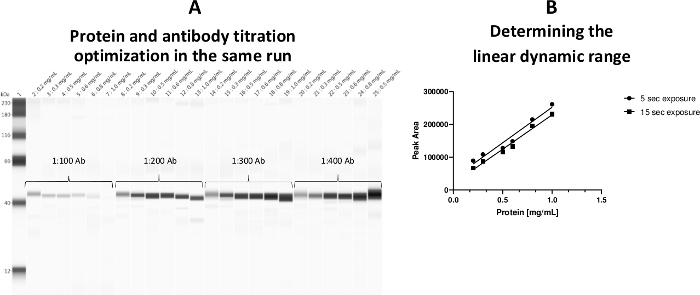
Abbildung 3: Linearer Dynamikbereich für die Thrombozytosolproteinkonzentration. (A) Sowohl die Proteinkonzentrationen als auch die Antikörpertitrationen wurden im selben Lauf auf einer Platte optimiert. (B) Der lineare Arbeitsbereich (0,2–0,8 mg/ml) für Proteine wurde festgelegt. Eine Proteinlast von 0,5 mg/ml wurde durch a-ERK-Antikörper als Positivkontrolle (Kapillare 25) gekennzeichnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Es sollte beachtet werden, dass der Glyzeringehalt im Probenvorbereitungsröhrchen weniger als 20 % (endgültig) betragen sollte, da sich die hohe Glycerinkonzentration sonst negativ auf die primäre Antikörperbindung auswirkt.
Bestimmung der optimalen Belichtungszeit
In der älteren Version der Software musste die optimale Belichtungszeit durch Plotten von Spitzenflächen gegen Dieproteinkonzentration (mg/ml) bestimmt werden. Die neue Version bietet ein neues Tool namens HDR-Erkennungsprofil (High Dynamic Range)(Abbildung 4). Über das Bildpanel wurde die Möglichkeit geboten, alle Belichtungszeiten (d. h. 5 s, 15 s, 30 s, 60 s, 120 s, 240 s, 480 s) zusammen anzuzeigen. Computersoftware analysierte alle Belichtungszeiten und identifizierte automatisch die beste Belichtungszeit (HDR). Das HDR-Erkennungsprofil lieferte aufgrund der höheren Empfindlichkeit von CEI einen deutlich größeren Dynamikbereich, was eine bessere Detektion und Quantifizierung über einen größeren Probenkonzentrationsbereich bedeutet. Benutzer haben jedoch immer noch die Möglichkeit, eine Belichtungszeit auszuwählen, die dem experimentellen Ziel entspricht. Mit dieser Funktion wurde eine optimale Belichtungszeit für TDP-43-Protein gefunden. Der Peak stellt die optimale Belichtungszeit dar (Abbildung 4A). Für diesen Antikörper wurde eine einzige Expositionszeit (4 s) definiert, nachdem alle neun Expositionszeiten zwischen 1–512 s überprüft wurden (Abbildung 4B).

Abbildung 4: HDR-Erkennungsprofil (High Dynamic Range) für ein Zielprotein. (A) Der TDP-43-Proteinpeak stellt die optimale Belichtungszeit für das Zielprotein dar. a-TDP-43 Abtitration betrug 1:300, und die Thrombozytosolproteinkonzentration betrug 0,5 mg/ml. Die softwaredefinierte blaue Linie zeigt die optimale Belichtungszeit an. (B) Diese Abbildung stellt eine benutzerdefinierte Einzelbelichtungszeit (4 s) nach Überprüfung aller neun Belichtungszeiten zwischen 1–512 s dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
TDP-43-Spiegel bei humanen Thrombozytosol-Spiegeln von ALS-Patienten
Eine Blutbasis Biomarker Entwicklung durchgeführt wurde. Unter Verwendung optimierter Assay-Bedingungen wurden zytosolische Thrombozysatfraktionen von ALS-Patienten mit zwei Antikörpersätzen analysiert (d.h. Anti-TDP-43 [Pan]-Antikörper, ein Antikörper, der phosphorylierte Derivate des TDP-43-Proteins erkennt; hier wurde a-P [S409/410-2] TDP-43 verwendet). In dieser Demonstration werden krankheitsspezifisches TDP-43 und sein phosphoryliertes Derivat vorgestellt (Abbildung 5).
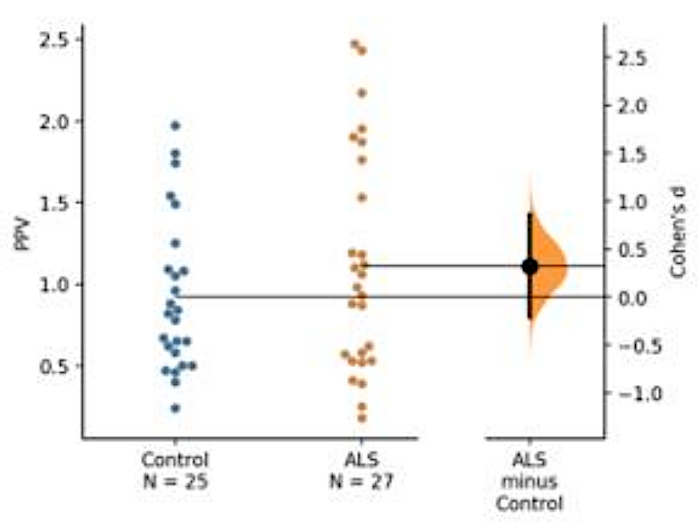
Abbildung 5: Eine Darstellung des prädiktiven Phosphorylierungswerts (PPV) von TDP-43. Die absolute Menge an phosphoryliertem TDP-43 und Pan TDP-43 allein zeigte keinen großen Unterschied zwischen den ALS- und Kontrollgruppen. PPV deutete jedoch auf einen leichten Anstieg der ALS-Kohorte hin, obwohl es keinen statistischen Unterschied zwischen den beiden Gruppen aufgrund unzureichender Anzahl von Probanden gab (ALS = 25, Kontrolle = 27). Ein niedriger Cohen-d-Wert zwischen den Mitteln von ALS und der Kontrollgruppe zeigte aufgrund der geringen Stichprobengröße eine geringe Effektgröße zwischen den beiden Gruppen (Kontrolle = 25, ALS = 27). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Gesamtmenge TDP-43 wurde anhand der Kalibrierkurve quantifiziert (Abbildung 6)

Abbildung 6: Standardkalibrierungskurve. Kommerziell erworbene rekombinante TDP-43-Proteinkonzentrationen wurden verwendet, um eine Standardkurve zu konstruieren. Jede Datenmenge stellt den Durchschnitt der Triplicate dar. Die Proteinbandintensitäten waren konzentrationsabhängig (Inset). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Quantifizierung des phosphorylierten TDP-43-Proteins war aufgrund der kommerziellen Nichtverfügbarkeit dieses Proteins nicht möglich. Stattdessen wurde ein vorhergesagter Phosphorylierungswert (PPV) festgelegt, der den Prozentsatz der phosphorylierten Arten von TDP-43 definiert. PPV wurde anhand der folgenden Gleichung aus zwei sequenziellen CEI-Assays für dieselbe Probe ermittelt.

Intra- und Inter-Run-Assay-Variabilität wurden in gepoolten menschlichen ALS-Thrombozytosolfraktionen getestet (Abbildung 7)
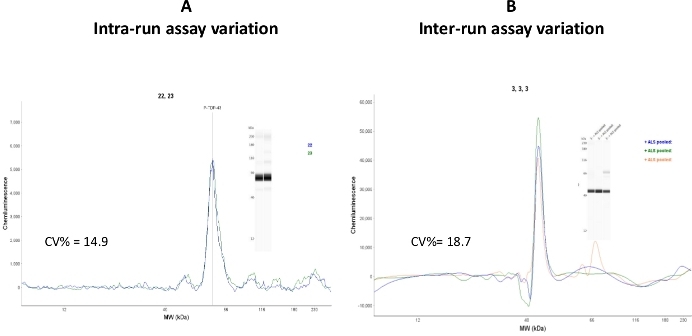
Abbildung 7: Assay-Variationen. (A) Zwei Kapillaren, die mit derselben Probe beladen waren, wurden von CEI analysiert, und die Intra-Run-Assay-Variation wurde als CV% = 14,9 berechnet. (B) Die gleiche Stichprobe wurde an drei verschiedenen Assay-Tagen und in drei verschiedenen Assay-Läufen analysiert. Die Differenz zwischen den durchläufen Assaywurde als CV% = 18,7 berechnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Obwohl die Variationswerte für den Intra-Run-Wert (Kapillar-zu-Kapillar-Variation) im akzeptablen Bereich (CV% = 14,9) fielen, war der Inter-Run-Assay-Wert relativ hoch (CV% = 18,7). Es wird interpretiert, dass diese Variation auf die Verwendung von Kapillarpatronen und Probenplatten aus verschiedenen Losen zurückzuführen sein kann. Es wird empfohlen, Reproduzierbarkeitsstudien an CEI-Komponenten mit der gleichen Chargennummer durchzuführen.
Tabelle 1: CEI-Assayplattenladescha. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Vorlage für die Herstellung von probenmischen. Nach eingabeder Proteinkonzentration aus unbekannten Proben berechnen interaktive Zellen automatisch, wie viel Volumen für die Vorbereitung des Probenmixes verwendet werden muss. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Der kapillare elektrophoretische Immunoassay ist jetzt die Methode der Wahl für Probenanalysen mit hohem Durchsatz und Arzneimittelscreenings25. Kleine Probenvolumina, gut optimierte Assay-Komponenten, benutzerfreundliche Assay-Plattform und Instrumentierung, Reagenzienaufwand und geringer CV-Prozentsatz sind primäre Vorteile26,27. Obwohl es mehrere Methoden zur Trennung von Proteinen in verschiedenen Assay-Modalitäten gibt, kann das hier beschriebene Antikörper-basierte CEI von kleinen Laboratorien angepasst werden, die sich mit der Entwicklung blutbasierter Biomarker beschäftigen. Die hier eingesetzte CEI-Assay-Technologie liefert zuverlässige, reproduzierbare und empfindliche Messungen für TDP-4328 und sein phosphoryliertes Derivat5.
Das CEI-System bietet auch eine Multiplexing-Auswahl, um TDP-43 und seine phosphorylierten Derivate gleichzeitig zu analysieren und die direkte Quantifizierung des Zielproteins zu liefern, wenn gereinigtes oder rekombinantes Zielprotein verfügbar ist. Rekombinantes TDP-43-Protein in voller Länge ist im Handel erhältlich; rekombinantes phosphoryliertes TDP-43-Derivat ist dies jedoch nicht. Da phosphoryliertes TDP-43 nicht kommerziell erhältlich ist, wurde ein prädiktiver Phosphorylierungswert (PPV) implementiert, um das TDP-43-Profil bei ALS-Patienten zu bewerten. Pan TDP-43 und phosphorylierte TDP-43-Mengen wurden dauerhaft mit einem Fluorophor gekennzeichnet; Daher bleibt das TDP-43-Profil mit oder ohne quantitative Einheit (d. h. ng/mL, pg/mL usw.) gleich. Obwohl die Bestimmung der absoluten Menge an TDP-43 und seinen phosphorylierten Derivaten (d. h. P [S409-410-12] TDP-43) eine quantitativere Messung bietet, eliminiert die Berechnung von PPV die Notwendigkeit des rekombinanten phosphorylierten TDP-43 für die Standardisierung, da es nicht kommerziell verfügbar ist.
CEI stellt mehrere Checkpoints in der Assay-Plattform bereit, um das Problem genau zu identifizieren, falls ein Test fehlschlägt. Dies beseitigt Hindernisse und bietet ein besseres experimentelles Design. Das Assay-Verfahren ist vollautomatisch, mit Ausnahme der Befüllung der Probenplatte. Dies ist ein wichtiges Merkmal im Vergleich zur Standard-Western-Blotting-Analyse. Diese Funktion bietet Konsistenz von Der Durchlauf. Obwohl jedes Labor über einzigartige Standard-Betriebsverfahren verfügt, ist es wichtig, sich an Praktiken zu halten, die menschliches Versagen minimieren. Beispielsweise ist es wichtig, das Luminol-S/Peroxid-Gemisch kurz vor der Plattenbeladung vorzubereiten, da die Zugabe von Peroxid in Luminol die enzymatische Reaktion einleitet und das Luminolsubstrat verbraucht. Auch das Laden von Proben und primär-sekundären Antikörpern in Plattenbrunnen ohne Luftblasen sind wichtige Schritte.
Da die Plattenbrunnen klein sind und es keinen Platz zwischen den Brunnen gibt, sollten Benutzer beim Pipetieren Vorsicht walten lassen, was der wichtigste Schritt ist, da alles andere automatisiert ist. Die Ladereihenfolge der Proben, Antikörper und anderen Reagenzien ist wichtig für die Konsistenz des Assays (Abbildung 1). Der Prozess der Plattenvorbereitung dauert ca. 40–45 min. Daher wird empfohlen, die Platte zunächst mit den erforderlichen Assaykomponenten zu beladen und das Luminol-S/Peroxid-Gemisch kurz vor dem Pipetieren vorzubereiten. Auf diese Weise gibt es eine konsistente Abfolge der Reagenzzugabe, und eine konsistente Lumineszenzsignalstärke wird erreicht. Es wird nicht empfohlen, ein abgelaufenes Luminol-S/Peroxid-Reagenz zu verwenden, da es in erster Linie die Festigkeit des Peroxids beeinflusst. Die jüngsten Fortschritte bei der Einführung des Split-Buffer-Systems und einschließlich des Chemikalien- und Waschmittelkompatibilitätsbereichs haben die Testqualität verbessert und zu reproduzierbareren und vorhersehbareren Ergebnissen geführt. Nun verfügt ein neuer Combo-Analysator desselben Herstellers über eine Funktion zur Analyse der Proben, die mit Chemilumineszenz und fluoreszenzkonjugierten Antikörpern im gleichen Lauf gekennzeichnet sind. Diese neue Funktion macht es überflüssig, zwei einzelne Platten nacheinander auszuführen, und eliminiert Lauf-zu-Lauf-Variationen.
Die Assayplatten sollten bei Umgebungstemperatur gelagert werden. Wenn die Assayplatten in einem 4 °C-Kühlschrank aufbewahrt werden sollen, müssen die Platten in der Nacht vor dem Test herausgenommen und auf Umgebungstemperatur gebracht werden. Falsch geladene Probenbrunnen müssen vor dem Hinzufügen der richtigen Probe ausgiebig (4-5-mal) mit Puffer im Kit gewaschen werden. Jeder primäre Antikörper und jede biologische Probe sind einzigartig; Daher sollte die Antikörper-Protein-Optimierung vor der Analyse der Proben auf Zielproteine in biologischen Flüssigkeiten durchgeführt werden.
Hier wurde standardmäßig die primäre Antikörperinkubationszeit auf 30 min eingestellt. Wenn das Signal schwach ist, benutzergreifen Benutzer, die primäre Antikörperinkubationszeit zu erhöhen, bis die gewünschte Signalstärke ohne Fluoreszenzsignal-Burnout erreicht wird. Für menschliche Thrombozyten wurden gepoolte Proben von Patienten vorbereitet und für einen Optimierungstest verwendet. Die Probenpooling besser stellt die Variation zwischen den Zielbiomolekülen dar. Es wird empfohlen, klare Überstande anstelle von Gesamtlysat oder Gesamthomogenat für das CEI zu verwenden.
Die hohe Konzentration von Protein in der ganzen Thrombozytenlysatmischung kann das Signal-Rausch-Verhältnis verringern (Abbildung 2). Wiederholte Frost-Tau-Zyklen von Proben sollten vermieden werden, da dies die primäre Antikörperbindung beeinträchtigt. Die Inhaltsstoffe des Lysatpuffers sind wichtig, da einige Reagenzien nicht mit CEI29kompatibel sind. Es wird empfohlen, die Liste der kompatiblen Reagenzien, die auf der Website des Herstellers bereitgestellt werden, vor der Probenvorbereitung zu überprüfen. Dies ist eine Einschränkung des Systems, die keine hohen Stringenzbedingungen für die Probenvorbereitung toleriert. Es wird empfohlen, die Parameter für den Testlauf (d. h. primäre Antikörperverdünnung, Proteinkonzentration, Primärantikörperinkubationszeit usw.) mithilfe von gepoolten Proben zu optimieren, um anschließend die einzelnen Proben zu analysieren.
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen außer ProteinSimple, Inc. deckte die Veröffentlichungskosten dieses Manuskripts.
Danksagungen
Diese Forschung wurde durch ein intramurales Stipendium für A.A. gefördert. Diese Arbeit wurde durch ein CTSA-Stipendium von NCATS unterstützt, das an das University of Kansas Medical Center for Frontiers: The Heartland Institute for Clinical and Translational Research (#UL1TR606381) vergeben wurde. Die Inhalte liegen allein in der Verantwortung der Autoren und stellen nicht unbedingt die offiziellen Ansichten des NIH oder NCATS dar. Wir sind dankbar für die University of Kansas Medical Center ALS Klinik persönlich für die Erlangung der IRB-Zulassung für die Blutentnahme von gesunden Freiwilligen und ALS-Patienten. Die Autoren danken Emre Agbas für das Korrekturlesen des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| 12-230 kDa Separation kit | ProteinSimple | SM-W004 | Contains pre-filled assay plate and 25-channel capillary cartridge |
| 3000G Thermocycler | Techne | FTC3G/02 | We used this thermocylcer for heating the sample mix |
| Anti-Mouse detection kit | ProteinSimple | 042-205 | Includes HRP-conjugated secondary antibody, buffer, luminol reagent, molecular weight marker |
| Anti-P(S409-410) TDP-43 antibody | ProteinTech | 22309-1-AP | Primary antibody that recognizes phosphorylated TDP-43 |
| Anti-P(S409-412) TDP-43 antibody | CosmoBio-USA | TIP-PTD-P02 | Primary antibody that recognizes phosphorylated TDP-43 |
| Anti-Rabbit detection kit | ProteinSimple | DM-001 | Includes HRP-conjugated secondary antibody, buffer, luminol reagent, molecular weight marker |
| Anti-TDP-43 (pan) antibody | ProteinTech | 10782-2-AP | Primary antibody that recognizes whole TDP-43 protein |
| Compass for SimpleWestern (SW) | ProteinSimple | Ver.4.0.0. | Compass for SW is the control and data analysis application for SimpleWestern instruments |
| Sonic Dismembrator; Model100 | Fisher Scientific | Sonicator. Used to rupture the cell membrane. This model is discontinued (Model XL2000-350) | |
| Table top centrifuge | Eppendorf | 22625004 | Model# 5810 with swinging plate bucket |
| Wes analyzer | ProteinSimple | 55892-WS-2203 | Performs the capillary gel electrophoresis |
Referenzen
- Blair, P., Flaumenhaft, R. Platelet alpha-granules: basic biology and clinical correlates. Blood Reviews. 23 (4), 177-189 (2009).
- Mercado, C. P., Kilic, F. Molecular mechanisms of SERT in platelets: regulation of plasma serotonin levels. Molecular Interventions. 10 (4), 231-241 (2010).
- Goubau, C., et al. Regulated granule trafficking in platelets and neurons: a common molecular machinery. European Journal of Paediatric Neurology. 17 (2), 117-125 (2013).
- Basu, S. S., et al. Human platelets as a platform to monitor metabolic biomarkers using stable isotopes and LC-MS. Bioanalysis. 5 (24), 3009-3021 (2013).
- Wilhite, R., et al. Platelet phosphorylated TDP-43: an exploratory study for a peripheral surrogate biomarker development for Alzheimer's disease. Future Science OA. 3 (4), 238 (2017).
- Worth, A. J., et al. LC-MS Analysis of Human Platelets as a Platform for Studying Mitochondrial Metabolism. Journal of Visualized Experiments. (110), e53941 (2016).
- Statland, J. M., et al. Rasagiline for amyotrophic lateral sclerosis: A randomized, controlled trial. Muscle and Nerve. 59 (2), 201-207 (2019).
- Charytan, D. M., et al. Safety and cardiovascular efficacy of spironolactone in dialysis-dependent ESRD (SPin-D): a randomized, placebo-controlled, multiple dosage trial. Kidney International. 95 (4), 973-982 (2019).
- Ugras, S. E., Shorter, J. RNA-Binding Proteins in Amyotrophic Lateral Sclerosis and Neurodegeneration. Neurology Research International. , 432780 (2012).
- Amador-Ortiz, C., et al. TDP-43 immunoreactivity in hippocampal sclerosis and Alzheimer's disease. Annal of Neurology. 61 (5), 435-445 (2007).
- Baloh, R. H. TDP-43: the relationship between protein aggregation and neurodegeneration in amyotrophic lateral sclerosis and frontotemporal lobar degeneration. FEBS Journal. 278 (19), 3539-3549 (2011).
- Buratti, E., Baralle, F. E. The molecular links between TDP-43 dysfunction and neurodegeneration. Advances in Genetics. 66, 1-34 (2009).
- Guo, W., et al. An ALS-associated mutation affecting TDP-43 enhances protein aggregation, fibril formation and neurotoxicity. Nature Structural Molecular Biology. 18 (7), 822-830 (2011).
- Geser, F., et al. Motor neuron disease clinically limited to the lower motor neuron is a diffuse TDP-43 proteinopathy. Acta Neuropathologica. 121 (4), 509-517 (2011).
- Neumann, M., et al. Ubiquitinated TDP-43 in frontotemporal lobar degeneration and amyotrophic lateral sclerosis. Science. 314 (5796), 130-133 (2006).
- Buratti, E., Baralle, F. E. TDP-43: gumming up neurons through protein-protein and protein-RNA interactions. Trends in Biochemical Sciences. 37 (6), 237-247 (2012).
- Fallini, C., Bassell, G. J., Rossoll, W. The ALS disease protein TDP-43 is actively transported in motor neuron axons and regulates axon outgrowth. Human Molecular Genetics. 21 (16), 3703-3718 (2012).
- Ayala, Y. M., et al. Structural determinants of the cellular localization and shuttling of TDP-43. Journal of Cell Sciences. 121, 3778-3785 (2008).
- Fiesel, F. C., Kahle, P. J. TDP-43 and FUS/TLS: cellular functions and implications for neurodegeneration. FEBS Journal. 278 (19), 3550-3568 (2011).
- Birsa, N., Bentham, M. P., Fratta, P. Cytoplasmic functions of TDP-43 and FUS and their role in ALS. Seminars in Cell and Development Biology. , (2019).
- Liachko, N. F., et al. CDC7 inhibition blocks pathological TDP-43 phosphorylation and neurodegeneration. Annals of Neurology. 74 (1), 39-52 (2013).
- Qureshi, A. H., et al. Proteomic and phospho-proteomic profile of human platelets in basal, resting state: insights into integrin signaling. PLoS One. 4 (10), 7627 (2009).
- Suominen, I., Koivisto, S. Increasing Precision When Pipetting Protein Samples: Assessing Reliability of the Reverse Pipetting Technique. American Laboratory. , (2011).
- . Pipetting tool box for life sciences Available from: https://www.mt.com/us/en/home/library/guides/laboratory-division/life-science/pipetting-toolbox-for-life-sciences.html (2019)
- Hale, L. J., et al. 3D organoid-derived human glomeruli for personalised podocyte disease modelling and drug screening. Nature Communication. 9 (1), 5167 (2018).
- Chen, J. Q., Wakefield, L. M., Goldstein, D. J. Capillary nano-immunoassays: advancing quantitative proteomics analysis, biomarker assessment, and molecular diagnostics. Journal of Translational Medicine. 13, 182 (2015).
- Moser, A. C., Hage, D. S. Capillary electrophoresis-based immunoassays: principles and quantitative applications. Electrophoresis. 29 (16), 3279-3295 (2008).
- Fourier, A., et al. Development of an automated capillary nano-immunoassay-Simple Western assay-to quantify total TDP43 protein in human platelet samples. Analytical and Bioanalytical Chemistry. 411 (1), 267-275 (2019).
- . Compatibility, S.W.S.A.B Available from: https://www.proteinsimple.com/technical_library.html (2017)
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten