Method Article
Agrobacterium- Vermitteltune unreife Embryo-Transformation von widerspenstigen Mais Inzuchtlinien mit morphogenen Genen
In diesem Artikel
Zusammenfassung
Pflanzenmorphogene können verwendet werden, um die genetische Transformation widerspenstiger Genotypen zu verbessern. Beschrieben wird hier ein Agrobacterium-vermittelte genetische Transformation (QuickCorn) Protokoll für drei wichtige öffentliche Mais-Inzuchtlinien.
Zusammenfassung
Gezeigt wird hier ein detailliertes Protokoll für Agrobacterium-vermittelte genetische Transformation von Mais-Inzuchtlinien mit morphogenen Genen Baby boom (Bbm) und Wuschel2 (Wus2). Bbm wird durch den Maisphospholipid-Transferase-Gen (Pltp) Promotor reguliert, und Wus2 ist unter der Kontrolle eines Mais-Auxin-induzierbaren (Axig1) Promotors. Ein Agrobacterium-Stamm, der diese morphogenen Gene auf Transfer-DNA (T-DNA) und Extrakopien von Agrobacterium-Virulen (vir) trägt, wird verwendet, um maisunreife Embryo-Explanten zu infizieren. Somatische Embryonen bilden sich auf den Scutella infizierter Embryonen und können durch Herbizidresistenz ausgewählt und in Pflanzen gekeimt werden. Ein wärmeaktiviertes Cre/LoxP-Rekombinationssystem, das in das DNA-Konstrukt integriert ist, ermöglicht die Entfernung morphogener Gene aus dem Maisgenom in einem frühen Stadium des Transformationsprozesses. Transformationsfrequenzen von ca. 14%, 4% und 4% (Anzahl unabhängiger transgener Ereignisse pro 100 infizierte Embryonen) können für W22, B73 und Mo17 mit diesem Protokoll erreicht werden.
Einleitung
Die Transformation ist ein grundlegendes Instrument zur Bewertung der Fremdgenexpression in Mais und zur Herstellung genetisch veränderter Maislinien sowohl für Forschungs- als auch für kommerzielle Zwecke. Der Zugang zu einer Transformation mit hohem Durchsatz kann den erhöhten Bedarf an molekularen und zellulären Biologiestudien für Mais erleichtern1. Die Fähigkeit, Pflanzenarten genetisch zu transformieren, ist sowohl für öffentliche als auch für private Laboratorien von entscheidender Bedeutung. Dies ermöglicht sowohl ein grundlegendes Verständnis der Genregulationsmechanismen als auch eine verbesserung der Kulturen auf globaler Ebene, um eine ständig wachsende Bevölkerung zu unterstützen.
Die Entdeckung, dass unreife Embryonen aus Mais zur Herstellung von regenerierbarem Callus verwendet werden könnten, entstand 19752. Seit dieser Enthüllung erforderten die meisten skalierbaren Maistransformationsprotokolle die Bildung und Selektion von Callus vor der Regeneration3. Während des Prozesses der genetischen Transformation werden Agrobacterium-infizierteoder biolistisch-bombardierte unreife Embryonen auf Medien für embryogene Callus-Induktion kultiviert. Induzierte Calli werden dann auf selektiven Medien kultiviert (z.B. mit einem Herbizid), so dass nur transformierte Callusstücke überleben können. Diese herbizidresistenten, vermeintlichen transgenen Calli werden aufgeschüttet und zu Pflanzen regeneriert. Während diese Methode wirksam ist, ist der Prozess lang und arbeitsintensiv, und es kann bis zu 3 Monate dauern, um4abzuschließen. Noch wichtiger ist, dass herkömmliche Maistransformationsprotokolle eine viel größere Begrenzung besitzen, d.h. nur eine begrenzte Anzahl von Maisgenotypen kann5,6transformiert werden.
Lowe et al.7,8 berichtete zuvor über eine "QuickCorn"-Transformationsmethode, die nicht nur die Dauer des Transformationsprozesses stark reduzierte, sondern auch die Liste der transformierbaren Genotypen erweiterte. Die QuickCorn-Methode verwendet Mais-Orthologs (Zm-Bbm und Zm-Wus2) der Arabidopsis-Transkriptionsfaktoren BABY BOOM (BBM)9 und WUSCHEL (WUS)10. Wenn sie in das Transformationsvektorsystem integriert sind, arbeiten diese Gene synergistisch, um das embryogene Wachstum zu stimulieren7.
Das in dieser Arbeit beschriebene QuickCorn-Protokoll basierte auf dem Protokoll in Jones et al11, das eine weitere Verbesserung der von Lowe et al7berichtetenMethodedarstellte, 8. In der vorliegenden Studie wird ein Agrobacterium-Stamm LBA4404(Thy-) verwendet, der ein binäres Vektorkonstrukt PHP81430 (Abbildung 1) und zubehörplasmaPHdes PHP7153912 beherbergt. Die T-DNA von PHP81430 enthält die folgenden molekularen Komponenten. (1) Das transformationsselektive Markergen Hra-Expressionskassette. Das Mais-Hra- (Zm-Hra)-Gen ist ein modifiziertes Acetolactase-Synthase-Synthase-Gen (ALS), das tolerant gegenüber ALS-hemmenden Herbiziden wie Sulfonylharnstoff und Imidazolinonen13,14ist. Das Zm-Hra-Gen wird durch den Sorghum ALS-Promotor8 und den Kartoffelproteinase-Inhibitor II(PinII) Terminator15reguliert. Die T-DNA enthält auch (2) eine Expressionskassette, die das transformationsbildbare Markergen ZsGreenbesitzt. Dieses grüne fluoreszierende Proteingen ZsGreen aus Zoanthus sp. riffkoralle16 wird durch einen Sorghum-Ubiquitin-Promotor/Intron- und Reis-Ubiquitin-Terminator reguliert.
Zusätzlich enthält die T-DNA (3) das morphogene Gen Bbm Expressionskassette. Bbm ist ein Transkriptionsfaktor im Zusammenhang mit der Embryoentwicklung9,17. Bbm wird durch das Maisphospholipid Transferase Protein (Pltp) Promoter8 und Reis T28 Terminator18reguliert. Zm-Pltp ist ein Gen mit starker Expression im Embryo-Scutellar-Epithel, Seidenhaaren und Blattnebenzellen (flankierend in den Wachzellen), geringer Expression in Fortpflanzungsorganen und ohne Expression in den Wurzeln8. Es enthält auch (4) das morphogene Gen Wus2 Expressionskassette. Wus2 ist ein weiterer Transkriptionsfaktor, der mit der Aufrechterhaltung des apikalen Meristes19verbunden ist. Zm-Wus2 steht unter der Kontrolle eines Mais-Auxin-induzierbaren Promotors (Zm-Axig1)20 und Mais In2-1 Terminator21. Schließlich enthält die T-DNA (5) das Cre- LoxP-Rekombinationssystem. Das Cre-Rekombinatoase-Gen22 steht unter der Kontrolle des Mais-Wärmeschockproteins 17.7 (Hsp17.7)23 Promoter und KartoffelpinII Terminator. Zwei loxP-Standorte (in der gleichen Ausrichtung)24 flankieren vier Genexpressionskassetten einschließlich ZsGreen, cre, Bbm und Wus2.
Da das Vorhandensein der morphogenen Gene für die Pflanzenreife und die nachfolgende Nachkommennichten nicht erwünscht ist, wurde das wärmeinduzierte Cre-LoxP-Rekombinationssystem in die T-DNA integriert, um morphogene Gene aus dem Maisgenom zu entfernen, um eine normale Callusregeneration und Pflanzenentwicklung zu ermöglichen. Bei der Wärmebehandlung entfernt die Expression des CRE-Proteins alle Transgene mit Ausnahme des Hra-Selektionsgens. Erfolgreiche Transformanten sollten herbizidresistent, aber ZsGreen-negativsein. Um die Transformationshäufigkeit weiter zu verbessern, enthält der Agrobacterium-Stamm auch ein zusätzliches Zubehörplasmid (PHP71539), das zusätzliche Kopien der Agrobacterium-Virulen (vir) Gene12enthält.
Die QuickCorn-Methode unterscheidet sich von herkömmlichen Maistransformationsprotokollen, da sie während der Transformation keinen Callus-Induktionsschritt beinhaltet. In der ersten Woche nach der Infektion mit Agrobacteriumentwickeln sich auf dem Scutellar-Epithel der infizierten unreifen Embryonen somatische Embryonen. Die Embryonen werden dann auf ein Medium mit Hormonen übertragen, die die Embryoreifung und Triebbildung fördern. Die schnelle Übertragung der somatischen Embryonen auf das Reifungs-/Triebbildungsmedium überspringt das traditionelle Callusstadium, das bisher für die Maisumwandlung verwendet wurde, und ermöglicht die direkte Erzeugung von T0-Pflanzen8. Im Vergleich zu den zuvor veröffentlichten Maistransformationsmethoden6ist die QuickCorn-Methode schneller, effizienter und weniger genotypabhängig. Mit dieser Methode sind verwurzelte Pflanzen in der Regel bereit, in nur 5-7 Wochen auf den Boden zu übertragen, anstatt die drei oder mehr Monate, die von traditionellen Protokollen erforderlich sind. Der Zweck dieses Artikels besteht darin, eine ausführliche Beschreibung und Demonstration der Methode bereitzustellen, um eine einfachere Replikation in einer Laborumgebung zu ermöglichen, die typischerweise in den meisten akademischen Einrichtungen zu finden ist.
Protokoll
1. Vorbereitung der Wachstumsmedien
- Genaue Rezepturen für dieses Protokoll finden Sie in Tabelle 1.
- Für die Vorbereitung von 1 L Medien, legen Sie einen 2 L Becher auf eine Rührplatte und legen Sie eine Rührstange innen.
- Becher mit 900 ml destilliertem Wasser füllen und die Rührplatte einschalten. Die Rührstange sollte sich mit mittlerer Geschwindigkeit drehen.
- Alle pulverförmigen Inhaltsstoffe wiegen und in einem Becher auflösen.
- Messen Sie alle flüssigen Zutaten, falls vorhanden, und fügen Sie das Becherglas hinzu.
- Bringen Sie das Endvolumen mit destilliertem Wasser auf 1 L.
- Messen Sie den pH-Wert und passen Sie sich den Rezeptspezifikationen an.
- Wenn ein flüssiges Wachstumsmedium formuliert wird, wird kein Agar hinzugefügt. Befestigen Sie einen Filtersterilisator an einer Vakuumpumpe und gießen Sie das flüssige Wachstumsmedium durch den Filter. Schalten Sie die Pumpe ein und warten Sie, bis die gesamte Flüssigkeit durchgezogen ist. Legen Sie eine Kappe auf den Behälter und befestigen Sie ein Etikett.
- Wenn Sie ein festes Wachstumsmedium formulieren, nachdem der pH-Wert angepasst wurde, fügen Sie Agar direkt in eine Flasche oder einen Kolben ein.
- Gießen Sie das 1 L flüssige Wachstumsmedium in einen 2 L Erlenmeyer Kolben oder teilen Sie es in zwei 1 L autoklavierbare Flaschen (je 500 ml) auf. Wenn zwei Flaschen verwendet werden, teilen Sie den Agar und fügen Sie direkt zu den Flaschen.
- Abdeckung Kolben mit einer atmungsaktiven Abdeckung, wie zwei Schichten von Aluminiumfolie, damit Dampf entweichen. Wenn Sie eine Flasche verwenden, den Schraubendeckel auf der Oberseite lose verkappen.
- Autoklav bei 121 °C für 25 min.
- Nach dem Autoklavieren das Wachstumsmedium aus dem Autoklaven entfernen und auf 55-60 °C abkühlen lassen (ein Auf 55 °C eingestelltes Wasserbad kann dies erleichtern). Halten Sie das Wachstumsmedium in einem flüssigen Zustand für ein paar Stunden, bis es bequem ist, Platten zu gießen.
- Nach dem Abkühlen alle Poststerilisationszusätze hinzufügen (siehe Tabelle 1) und gründlich mischen.
- Nachdem alle Zutaten zugegeben sind, gießen Sie das vorgesehene Volumen in den Behälter der Wahl in einer laminaren Strömungshaube.
- Wachstumsmedium kann manuell oder mit einem Flüssigkeitsdosiergerät in gewünschte Petrischalen gegossen werden. Beim manuellen Gießen wird empfohlen, ein großes Volumen autoklaviertes Medium in ein kleineres steriles Becherglas (500 ml) zu übertragen, um die Handhabung zu vereinfachen.
- Wachstumsmedium abkühlen und erstarren lassen.
- Das Wachstumsmedium wird für den Einsatz zur Verfügung stehen, sobald es fest wird und wird am besten am nächsten Tag nach dem Trocknen leicht über Nacht in einer sterilen Durchflusshaube als Stapel von Deckelplatten verwendet. Nach dem Trocknen über Nacht die Platten in Kunststoffhülsen geben, über das lose Ende falten und mit einem kleinen Stück Klebeband an Ort und Stelle halten. Dies verhindert eine übermäßige Trocknung. Medium kann bis zu 1 Monat in einer kühlen, dunklen und sauberen Umgebung (4-16 °C) gelagert werden.
2. Anbau von Spenderpflanzen und Ernte unreifer Ohren
- Wachsen Sie alle öffentlich verfügbaren Maisinzucht (z. B. B73, Mo17 oder W22) in einem Gewächshaus in 1,5 Gallonen (5,9 l) Töpfen, die ein bodenloses Substrat enthalten. Verwenden Sie eine 16/8 (Tag/Nacht) Fotoperiode, mit einer durchschnittlichen Temperatur von 25.5 °C während des Tages und 20 °C in der Nacht.
- Die Pflanzen werden nach Bedarf bewässert und mit einem kontrollierten Freisetzungsdünger (N-P-K von 15-9-12) gedüngt, der entweder in den Bodenmix eingearbeitet oder nach der Pflanzung an die Oberfläche gegeben werden kann.
- Es dauert in der Regel etwa 70-90 Tage nach der Samenkeimung für Die Ohren entstehen. Wenn Ohrsprossen auftauchen, bedecken Sie sie mit einem Schießbeutel, um eine unkontrollierte Bestäubung zu verhindern.
- Etwa 2-3 Tage, nachdem Seiden entstanden sind und wenn Pollen am nächsten Tag verfügbar sein werden, schneiden Sie die Seiden mit einer Schere, die in 70% Ethanol sterilisiert wurde. Schneiden Sie die Seiden und Schale etwa 2,5 cm unter dem Ende der Schalenblätter, wo die Seiden entstehen. Die Bestäubung kann am nächsten Tag durchgeführt werden. Achten Sie darauf, die Schere zwischen jedem Ohr zu resterilisieren.
- Sobald Anthers aus einer Quaste auftauchen, bedecken Sie die Quaste mit einer Quastetasche und einer rutschigen Büroklammer an der Basis der Tasche um den Stiel.
- Am nächsten Morgen, beugen Sie die Pflanze sanft um und tippen Sie auf den Beutel, um Pollen freizulassen.
- Entfernen Sie den Quastenbeutel und falten Sie die Oberseite des Beutels um, um zu verhindern, dass Pollen entweichen. Es ist in der Regel am besten, die Quaste 1 Tag vor der Verwendung zu packen (um anfallende Pollen und Schuppenanhäupter zu vermeiden). Frische Pollen können für ca. 3-5 Tage aus Quasten gesammelt werden. Wenn Anther aus den inneren Blüten an der Basis der Quaste auftauchen, wird diese Quaste wahrscheinlich am nächsten Tag keine lebensfähigen Pollen produzieren.
- Verwenden Sie die Pollen aus der gleichen Pflanze (selbst) oder von einer anderen Pflanze der gleichen inbred (sibbing).
- Entfernen Sie die Ohrtasche oder schneiden Sie das Ende der Tasche, um die Seide freizulegen, dann schnell Pollen aus der Quaste Tasche auf die Seide gießen.
- Bedecken Sie das Ohr sofort mit dem Quastenbeutel und heften Sie die Basis der Tasche um den Stiel, um ihn zu sichern. Es kann hilfreich sein, die Pflanze während der Bestäubung physisch von blühenden Pflanzen verschiedener Genotypen zu isolieren, um Kreuzbestäubung zu verhindern. Lassen Sie den Quastenbeutel am Ohr, bis das unreife Ohr erntereif ist.
- 9-12 Tage nach der Bestäubung, Siebohren für Embryogröße. Schieben Sie die Bestäubungstasche auf den Stiel, um das Ohr zu belichten. Ziehen Sie die Schale vorsichtig nach unten, um Kerne auf etwa einem Drittel bis zu einem Viertel des Umfangs des Ohres und etwa ein Drittel des Abstands nach unten das Ohr freizulegen. Kerne in der Nähe der Spitze sind nicht repräsentativ für die durchschnittliche Embryogröße.
- Schneiden Sie mit einem Skalpell die Kappe eines einzelnen Kernels ab, der in Größe und Farbe ähnlich wie die meisten anderen Kernel aussieht.
- Verwenden Sie einen Spachtel (mit einem Lineal), um den Embryo zu entfernen, wie in Schritt 4.6 beschrieben. Messen Sie die Länge des Embryos mit einem eingebauten Lineal auf dem Spachtel oder einem digitalen Sättel. Wenn der Embryo zwischen 1,5-2,0 mm liegt, ernten Sie das Ohr. Wenn es 1,3 mm beträgt, kann das Ohr später am Tag erntebereit sein und kann in ca. 7-8 h erneut überprüft werden.
3. Vorbereitung Der Agrobacterium Suspension kultur für Infektionen
HINWEIS: Der Agrobacterium-Stamm LBA4404(Thy-) mit PHP81430 (Abbildung 1) und PHP7153912 wird als Glycerinbestand bei -80 °C gespeichert. Diese Materialien können von Corteva Agriscience über eine Materialtransfervereinbarung bezogen werden. LBA4404 (Thy-) ist ein auxotropher Stamm, der Thymidin benötigt, das in den Wachstumsmedien geliefert wird. Der Hauptnutzen des Auxotroph Agro-Stamms ist für Biocontainment-Zwecke. Es hat den zusätzlichen Vorteil, das Überwucherungswachstum der Agro zu verringern. Die auxotrophe Agro-Sorte wächst nicht ohne zusätzliches Thymidin. Dennoch kann Thymidin (vermutlich) durch absterbendes Pflanzengewebe in der Kultur zugeführt werden. Daher besteht nach wie vor die Notwendigkeit, ein Antibiotikum im Medium zur Verfügung zu stellen, um die auxotrophe Agro vollständig zu kontrollieren. Jedoch, Es wird einfacher sein, aufgrund des beeinträchtigten Wachstums der auxotrophen Sorte in Abwesenheit von Thymidin zu kontrollieren.
- Vier Tage vor dem Infektionsdatum eine "Mutterplatte" aus dem Glycerinbestand initiieren, indem Sie die Bakterien auf einer YP-Platte mit 50 mg/L Thymidin, 50 mg/L Gentamicin und 50 mg/L Spectinomycin(Tabelle 1) streichen. Inkubieren Sie die "Mutterplatte" 3 Tage lang in einem 20 °C-Inkubator.
- Einen Tag vor dem Infektionsexperiment bereiten Sie eine "Arbeitsplatte" vor, indem Sie ein bis fünf Kolonien von der "Mutter"-Platte auswählen und die Bakterien von der "Mutter"-Platte auf eine neue YP-Platte (mit Thymidin, Gentamycin und Spektinomycin) sättigen; Tabelle 1).
- Streichen Sie die tägliche "Arbeitsplatte" in sequenziellen Quadranten und führen Sie die Schleife 1x durch den gerade gestreiften Bereich in den aufeinanderfolgenden Quadranten, wobei sie sich zu Quadranten wiederholt, die seriell verdünnt wurden. Inkubieren Sie die "Arbeitsplatte" über Nacht in einem 27 °C Inkubator.
- Nach Abschluss der Embryosektion (Schritt 4.8) verwenden Sie eine Schleife oder ein ähnliches Werkzeug, um Agrobacterium aus einem Bereich der "arbeitsfähigen" Platte zu sammeln, wo das bakterienwachstum als dünne Streifen von Kolonien sichtbar ist.
HINWEIS: Vermeiden Sie Bereiche der Platte mit einem dichten Rasen von bakteriellem Wachstum. Das Agrobacterium-Wachstum hat wahrscheinlich bereits begonnen, in dichten Gebieten zu sinken, während in den Gebieten mit sichtbaren Kolonien die Bakterien in der richtigen Wachstumsphase für Infektionen sind. - Die gesammelten Bakterien in einem 50 ml-Rohr mit 10 ml 700A flüssigem Medium aussetzen (Tabelle 1). Wirbel, um die Bakterienkultur vollständig auszusetzen.
- Messen Sie die optische Dichte bei einer Wellenlänge von 550 nm. Passen Sie die Lautstärke an, bis der OD zwischen 0,35 und 0,45 liegt, wobei 0,4 der optimale Wert ist.
HINWEIS: Wenn der OD höher als 0,45 ist, fügen Sie mehr 700A flüssiges Medium hinzu. Wenn die OD unter 0,35 liegt, impfen Sie mehr Agro-Kolonien in der Suspensionskultur.
4. Embryonizieren, Infektionen und Kokultivierung
- Wählen Sie geeignete Ohren für Transformationsexperimente; diese sollten einen guten Samensatz haben und Embryonen haben, die eine Größe von 1,5-2,0 mm haben. Sie werden in der Regel zwischen 9-12 Tage nach der Bestäubung geerntet. Geerntete Ohren können frisch oder für 1-4 Tage bei 4 °C gelagert werden, obwohl die Qualität der Reaktion wahrscheinlich allmählich bei längerer Lagerung über den ersten Tag hinaus abschwächen wird.
- Entfernen Sie die Schalen und Seiden. Legen Sie einen Griff entweder in die Basis oder die Oberseite des Ohres ein. Der Griff kann ein Paar Zangen, Schraubendreher, etc. sein.
- Ohren in einen großen Behälter legen (z.B. 2 L Becher mit dem Griff nach oben, den Behälter mit Desinfektionslösung füllen. Desinfektionslösung ist 1,8 L von 20% kommerzielle Bleichmittel (1,65% Natriumhypochlorit) und ein paar Tropfen Tensid Tween 20.
- Sterilisieren Sie die Ohren in einer laminaren Strömungsbank. Nach 20 min die Bleichlösung entleeren und die Ohren 3x (je 5 min) mit einer großzügigen Menge sterilen destillierten Wassers abspülen. Entfernen Sie das Wasser und lassen Sie die Ohren für einige Minuten trocknen.
HINWEIS: Es ist wichtig, dass die Ohren 20 min vollständig in die Bleichlösung getaucht werden. Bewegen Sie die Ohren vorsichtig in der Bleichlösung, um Luftblasen zu lösen. - Bereiten Sie ein 2 ml Mikrozentrifugenrohr vor, das mit 700A flüssigem Medium gefüllt ist. Dieses Rohr wird verwendet, um die unreifen Embryonen zu sammeln.
- Nehmen Sie das Ohr und mit einem sterilen Skalpell, entfernen Sie die oberen 1-2 mm der Kernkronen, um das Endosperm freizulegen. Verwenden Sie einen Mikrospachtel, um den unreifen zygoten Embryo (IZE) zu entfernen. Die IZE befindet sich innerhalb des Kerns, auf der Seite mit Blick auf die Ohrspitze und in der Nähe der Befestigung an der Cob. Mit dem Spachtel, legen Sie es in das Endosperm in der Pericarp am weitesten weg vom Embryo, dann sanft nach oben drehen, um das Endosperm zu vertreiben und ermöglichen die Entfernung des Embryos (Abbildung 2).
- Übertragen Sie den Embryo mit dem Spachtel in die Röhre, die das flüssige Medium 700A enthält. Fahren Sie so lange fort, bis bis bis zu 100 Embryonen gesammelt wurden. Mehrere Röhren können gefüllt werden (ca. 100 Embryonen/Rohr), bevor Sie mit dem nächsten Schritt fortfahren. An dieser Stelle sollte die Agrobacterium-Aufhängung vorbereitet werden (siehe Schritt 3.5).
- Entfernen Sie das flüssige Medium 700A mit einer 1 ml Pipette aus dem Embryorohr. Fügen Sie frische 700A Medium, um die Embryonen zu waschen, dann entfernen Sie diese Medien als auch.
- Fügen Sie 1 ml der Agrobacterium Suspension und Wirbel auf einer niedrigen Einstellung (3/10) für 30 s oder invertieren Rohr 12x-15x zu mischen. Lassen Sie dieses Rohr horizontal auf der Bank für 5 min ruhen.
- Nach 5 min die gesamte Röhre von Embryonen und Agrobacterium Suspension auf eine Platte von 562V Anbaumedium übertragen (Tabelle 1). Dies kann erreicht werden, indem man die Platte auf die Bank legt und den Rohrinhalt schnell auf die Platte gießt. Wirbeln Sie die Platte vorsichtig, um die Embryonen zu verteilen und entfernen Sie die Agrobacterium Suspension mit einer 1 ml Pipette.
- Stellen Sie sicher, dass die Embryonen mit der scutellum (runden) Seite nach oben platziert werden. Verwenden Sie bei Bedarf eine Lupe oder einen Sezierenvon. Teller in Plastikboxen (19 cm x 28 cm x 5,1 cm) aufstellen und über Nacht 16-18 h bei 21 °C im Dunkeln bebrüten. Eine individuelle Plattenumhüllung mit Paraffinfolie oder Entlüftungsband ist nicht erforderlich.
- Nach der nächtlichen Kokultivierung die infizierten Embryonen, scutellum seiteig nach oben, auf das ruhende Medium 605T (Tabelle 1) bewegen. Legen Sie etwa 30 Embryonen pro Platte. Inkubieren Sie die Platten bei 26-28 °C im Dunkeln.
- Inkubieren für 4-10 Tage (7 Tage wird bevorzugt). Zu diesem Zeitpunkt kann die Entwicklung somatischer Embryonen auf der Oberfläche des zygotischen Scutellums beobachtet werden (Abbildung 3).
5. Auswahl, Wärmebehandlung und Regeneration
- Nach der Ruhezeit schockieren die Embryonen. Legen Sie die Box mit den Platten der Embryonen in einen 45 °C-Inkubator mit 70% relativer Luftfeuchtigkeit für 2 h. Dann die Box aus dem 45 °C-Inkubator entfernen und 1-2 h in den dunklen Inkubator 26-28 °C legen.
HINWEIS: Wenn Sie nicht in der Lage sind, 70 % Luftfeuchtigkeit in einem Inkubator zu erreichen, fügen Sie eine doppelte Schicht autoklavierter Papiertücher an den Boden der Plattenbox und mit autoklaviertem Wasser einweichen, um die Feuchtigkeit in der Box aufrechtzuerhalten. Geben Sie die Platten auf die Box auf den Papiertüchern zurück und versiegeln Sie den Deckel, bevor Sie ihn bei 45 °C platzieren. Verwenden Sie ein kleines digitales Hygrometer/Thermometer, um Temperatur und Luftfeuchtigkeit zu überwachen. - Übertragen Sie die wärmebehandelten IZEs vom Ruhemedium auf das Triebbildungsmedium (13329A) mit 0,05 mg/L Imazapyr als selektives Mittel (Tabelle 1). Entfernen Sie bei der Übertragung Die Kolotile, falls vorhanden, mit feinen Spitzenzangen oder chirurgischen Scheren.
- Platzieren Sie 10-15 Embryonen pro Platte, um eine Überbelegung zu vermeiden. Bewahren Sie die Embryonen in diesem Medium 2 Wochen im 26 °C dunklen Inkubator auf.
- Übertragung der Embryonen auf das Wurzelmedium (13158; Tabelle 1) für 1-2 Wochen. Legen Sie etwa acht Stück pro Platte und inkubieren Sie in einem HellenRaum oder lichtkammer (16 Tag/ 8 Nacht, 20-150 mol/m2/s) bei 27 °C.
- Wenn sich Pflanzenentwickeln entwickeln, legen Sie stärkere Pflanzen, die sowohl Triebe als auch kräftige Wurzeln enthalten, auf neue Platten des Wurzelmediums, legen Sie eine Pflanze pro Platte. Dies wird ein stärkeres Pflanzenwachstum ermöglichen. Legen Sie die Platten für weitere 7-14 Tage in den Hellenraum oder die Lichtkammer.
HINWEIS: Entfernen Sie sorgfältig alle Callus-Stücke, die mit dem Plantlet verbunden sind, um sicherzustellen, dass es in gutem Kontakt mit dem Medium ist. - Wenn die Pflanze kräftiger wird, entfernen Sie die Pflanze aus dem Verwurzelungsmedium und spülen Sie die Wurzeln mit Leitungswasser, um Agar zu entfernen.
- Verpflanzen Sie einzelne Pflanzen in 3 in2 (ca. 19 cm2) Töpfe, die ein vorbenetztes bodenloses Substrat enthalten. Legen Sie die Töpfe in ein Tablett (27 cm x 54 cm) mit Ablauflöchern und bedecken Sie die Wohnung mit einer Kunststoff-Feuchtigkeitskuppel. Dieser Akklimatisierungsschritt kann entweder in der Wachstumskammer oder im Gewächshaus mit den in Abschnitt 2 (Schritt 2.1) beschriebenen Wachstumsbedingungen erreicht werden.
6. Verpflanzung in das Gewächshaus und Herstellung von T1-Samen
- Überprüfen Sie die Pflanzen 2x pro Tag. Wasser nach Bedarf. Stellen Sie sicher, dass die Pflanzen weder ausgetrocknet noch überwässert werden. Die Aufrechterhaltung eines leicht trockenen Substrats fördert das Wurzelwachstum.
HINWEIS: Die Feuchtigkeitskuppel kann 4-7 Tage nach der Transplantation entfernt werden. Pflanzen sollten in diesen kleinen Töpfen angebaut werden, bis sie sich sichtbar vom Stress der Transplantation auf den Boden erholt haben. Dies sollte etwa 9-14 Tage dauern. - Den gesamten bodenlosen Stecker und die Pflanze in einen 1,5-Gallonen-Topf (5,9 l) verpflanzen. Bewahren Sie im Gewächshaus und Wasser, wenn sich der Boden bis zur Berührung trocken anfühlt.
- Fügen Sie einen kontrollierten Trenndünger mit N-P-K von 15-9-12 in den Topf, der entweder in den Substratmix eingearbeitet oder auf die Oberfläche aufgetragen werden kann.
- Wenn Ohrsprossen aus der Pflanze zu entstehen beginnen, verwenden Sie eine Schießtasche, um die Ohrsprossen zu bedecken. Achten Sie darauf, eine Tasche zu verwenden, die halbtransparent ist, so dass die entstehenden Seiden ohne Entfernung der Tasche beobachtet werden können. Der Schießbeutel ermöglicht eine kontrollierte Bestäubung. Es ist wichtig, immer transgene Quasten zu packen.
- Nachdem die Seiden auftauchen (1-2 Tage), trimmen Sie die entstandenen Seiden auf eine einheitliche Länge. Dieser wird etwa 2,5 cm unter der Oberseite der Schalenblätter sein. Verwenden Sie eine saubere Schere, die in 70% Ethanol sterilisiert wurde. Durch Das Trimmen der Seiden entwickelt sich am nächsten Tag ein gleichmäßiger Büschel, damit die Bestäubung auftritt.
- Bei der Mehrheit der Maisgenotypen beträgt die optimale Zeit für die Bestäubung 2-3 Tage nach der Entstehung von Quasten oder Seiden.
- Sammeln Sie die Pollen entweder von der gleichen Pflanze (wenn sie selbst bestäuben) oder von einem Wildentyp der gleichen Inzucht (wenn outcrossing oder incrossing).
- Sammeln Sie die Pollen in einem Quastenbeutel und tragen Sie ihn auf den Seidenbüschel der T0-Pflanze auf. Wenn der Pollen von einer wilden (nicht-transgenen) Pflanze stammt, legen Sie den Pollen in einen schlichten braunen Quastenbeutel. Wenn der Pollen aus einer transgenen Pflanze stammt, legen Sie den Pollen in einen grün gestreiften Beutel, um anzuzeigen, dass der Pollen transgen ist.
- Befolgen Sie die Schritte 2.5-2.10 für Bestäubungsdetails.
- Etwa 2 Wochen nach der Bestäubung, entfernen Sie die Quastenbeutel von den Ohren, und lassen Sie zum Trocknen beginnen. Um beim Trocknen zu helfen, beenden Sie die Bewässerung der Pflanze 21-25 Tage nach der Bestäubung. Sie können auch die Schalenblätter zurückziehen, um den Samen freizulegen. Diese Praxis hilft auch Schimmel zu verhindern.
- Etwa 45 Tage nach der Bestäubung Samen ernten und bei 4-12 °C in der Kühllagerung lagern.
Ergebnisse
Hier wird ein Schritt-für-Schritt-Protokoll für Agrobacterium-vermittelte genetische Transformation von drei öffentlichen Mais-Inzuchtlinien (B73, Mo17 und W22) demonstriert, die im Bereich der Maisgenetik signifikant waren. Die Transformation der drei Inzuchtlinien konnte mit den herkömmlichen Maistransformationsprotokollen5nicht erreicht werden. Abbildung 1 und Abbildung 2 zeigen die hier verwendeten Konstruktions- bzw. Ausgangsmaterialien. Ohren werden in der Regel 9-12 Tage nach der Bestäubung geerntet. IZEs mit Längen zwischen 1,5 und 2,0 mm sind die besten Explanten für die Transformation für dieses Protokoll (Abbildung 2).
Acht Tage nach der Infektion wurden ZsGreen-extierendesomatische Embryonen unter dem GFP-Kanal eines Fluoreszenzmikroskops visualisiert (Abbildung 3). Infizierte IZEs wurden 8 Tage nach der Infektion einer Wärmebehandlung unterzogen (Schritte 5.1 und 5.2). Diese Behandlung induzierte die Expression von CRE-Rekombinat, die die Bbm- Wus2, creund ZsGreen Ausdruck Kassetten flankiert zwischen den beiden loxP-Standorten (Abbildung 1). Die wärmebehandelten Gewebe wurden dann auf einem Triebbildungsmedium kultiviert, das das Herbizid Imazapyr zur Selektion des transformierten Gewebes nach morphogener Genentfernung enthält.
Vermehrende Gewebe mit reifenden Embryonen oder Triebknospen, die gegen Imazapyr resistent waren, wurden etwa 3-4 Wochen nach der Infektion beobachtet (Abbildung 4). Einige Imazapyr-resistente Gewebe waren negativ für ZsGreen, was darauf hindeutet, dass in diesen Geweben wahrscheinlich eine kre-vermittelteExzision aufgetreten ist ( Abbildung4). Nachdem das Gewebe in die Wurzelmedium- und Lichtinkubation verschoben wurde, begannen sich die Triebe zu entwickeln (Abbildung 5). Gesunde und kräftig wachsende Triebe mit gut entwickelten Wurzeln wurden geerntet (Abbildung 5). Einige Gewebe schienen mehrere Triebe zu haben (Abbildung 5E,F,G). Diese Art von "grasigem" Regenerant kann auf klonale Pflanzen mit identischen Transgenintegrationsmustern zurückzuführen sein. Molekularbiologische Analysen sind erforderlich, um diese Pflanzen zu genotypisieren.
Alle drei öffentlichen Inzuchtlinien reagierten gut mit diesem Protokoll sowie dem in dieser Arbeit verwendeten Konstrukt. W22 produzierte die höchste Häufigkeit von Imazapyr-resistenten Trieben mit einer Häufigkeit von etwa 14% (ca. 14 transgene Triebe pro 100 infizierte unreife Embryonen). Sowohl B73 als auch Mo17 produzierten etwa 4% transgene Triebe. Diese Frequenzen zeigen alle transgenen Triebe an, einschließlich der beiden Pflanzen, die die morphogenen Gene tragen, und Pflanzen mit dem morphogenen Gen, das durch die CRE-vermittelte Exzision entfernt wurde.

Abbildung 1: Schematische Darstellung der T-DNA-Region des binären Plasmids PHP81430. RB = rechte T-DNA-Grenze; loxP = CRE-Rekombinator-Zielstandort; Axig1pro:Wus2 = Mais-Auxin-induzierbarer Promotor (Zm-Axig1) + Zm-Wus2 + Mais In2-1 Terminator; Pltppro:Zm-Bbm = Maisphospholipid Transferase Protein (Zm-Pltp) Promoter + Zm-Bbm + Reis T28 Terminator (Os-T28); Hsppro:cre = Mais-Wärmeschockprotein 17,7 Promotor (Zm-Hsp17.7) + Cre-Rekombinator-Gen + Kartoffelproteinase-Inhibitor II (pinII) Terminator; Ubipro:ZsGreen = sorghum ubiquitin promoter/intron (Sb-Ubi) + grünes fluoreszierendes Protein ZsGreen Gen + Reis Ubiquitin Terminator (Os-Ubi); Hra Kassette = Sorghum acetolactase synthase (Sb-Als) Promoter + Mais Hra (Zm-Hra) gen + pinII Terminator; LB = linke T-DNA-Grenze; colE1, Replikationsursprung des Plasmids ColE125; SpecR = spectinomycinresistentes Gen aadA1 von Tn21 zur Bakteriumauswahl26; Rep A,B,C = Replikationsursprung aus pRiA4 von Agrobacterium rhizogenes27. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
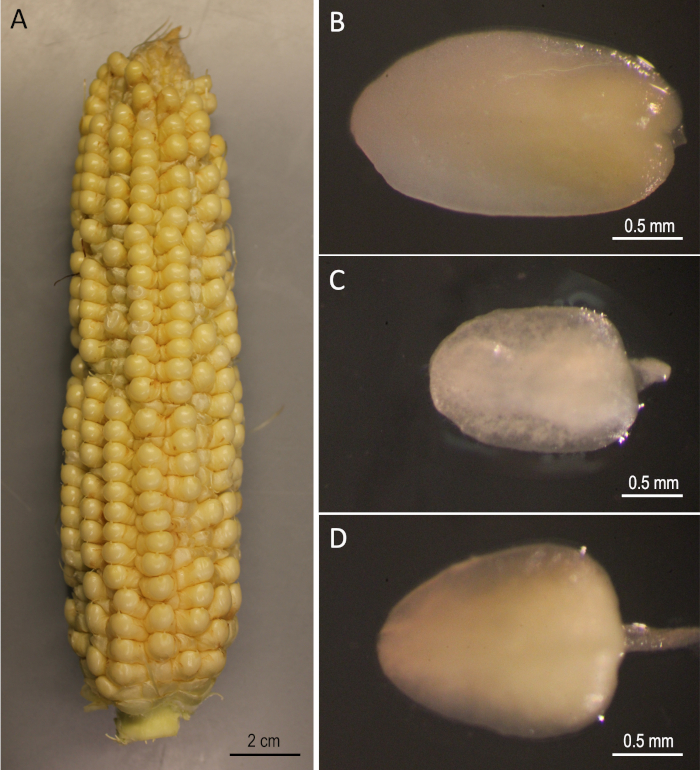
Abbildung 2: Ausgangsmaterialien. B73 Ohren geerntet 12 Tage nach der Bestäubung (A). Unreife Embryonen von B73 (B), Mo17 (C) und W22 (D). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
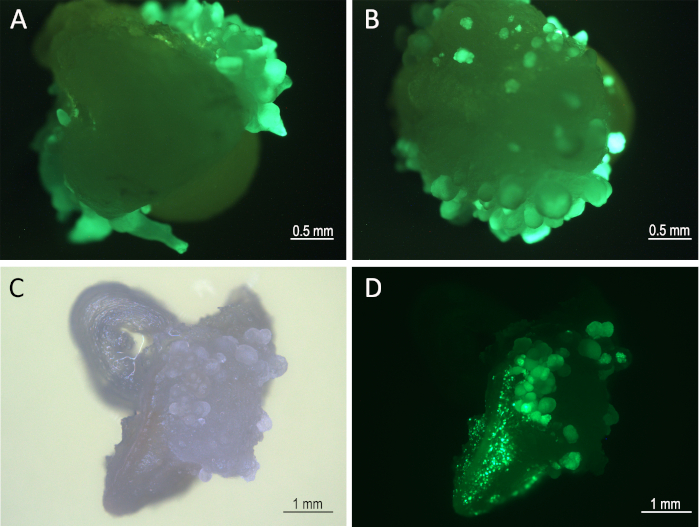
Abbildung 3: Gewebeentwicklung auf Ruhemedium 1 Woche nach der Infektion. Embryonen (8 Tage nach der Infektion) unter einem Blütenmikroskop (GFP-Filter) mit GFP-Exzessen somatischer Embryonen von Mo17 (A) und W22 (B). Entwicklung von Gewebe (B73) unter Hellem Feld (C) und GFP-Filter (D). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Gewebeentwicklung auf Reifungsmedium mit Auswahl. Eine W22 Reifungsplatte (A). Entwicklung von Gewebe (Mo17, 15 Tage nach der Infektion) unter Hellem Feld (B) und GFP-Filter (C). Entwicklung von Gewebe (Mo17, 28 Tage nach der Infektion) unter Hellem Feld (D) und GFP-Filter (E). Pfeile weisen auf regenerierende Gewebe hin, denen die GFP-Expression fehlt, was auf die Exzision des ZsGreen-Gens zwischen den LoxP-Standorten nach hitzeinduzierter CRE-Proteinaktivität hindeutet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Gewebeentwicklung auf Verwurzelungsmedien. Triebe von W22 (A), B73 (B) und Mo17 (C,D). Event mit mehreren Trieben (grasige Regeneranten) von B73 (E) und W22 (F,G). Triebe mit Wurzeln von B73 (H) und W22 (I). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Medienzusammensetzungen für die Maisumwandlung. Bitte klicken Sie hier, um diese Tabelle anzuzeigen (Rechtsklick zum Herunterladen).
Diskussion
Traditionelle Protokolle für die Maistransformation folgen dem Paradigma der Isolierung unreifer zygotischer Embryonen zur Herstellung von transgenem Kalusgewebe, das zu fruchtbaren Pflanzen regeneriert wird4,6. Während dies wirksam ist, können Callus-basierte Protokolle zeitaufwändig sein, und es dauert oft bis zu 3 Monate, bis der Gewebekulturprozess Pflanzen produziert. Was die hier vorgestellte Methode signifikant macht, ist, dass sie callusfrei, effizient, schnell ist und die Regeneration von T0-Pflanzen in etwa der Hälfte des Zeitrahmens ermöglicht. Es scheint auch weniger genotypabhängig zu sein und kann somit für die meisten öffentlich verfügbaren Inzuchten wirksam sein8,11.
Während alle Schritte effektiv befolgt werden sollten, ist eine korrekte Vorbereitung der Wachstumsmedien unerlässlich. Wachstumsmedienkomponenten müssen in den richtigen Stadien hinzugefügt werden, sowohl vor als auch nach dem Autoklaven, um sicherzustellen, dass das Pflanzenmaterial die richtige Konzentration von Chemikalien erhält. Dadurch wird sichergestellt, dass empfindliche Verbindungen wie Antibiotika nicht abbauen. Es ist auch wichtig, dass Pflanzenmaterial in jeder Phase auf das richtige Wachstumsmedium gelegt wird, wie im Protokoll angegeben. Das Nicht-Aufstellen von Material auf das richtige Wachstumsmedium kann zum materiellen Tod führen. Darüber hinaus sollte vermieden werden, zu viele Embryonen oder die Entwicklung von Geweben auf Platten zu platzieren. Während das Platzieren von doppelt so vielen Gewebestücken die Kosten für Chemikalien und Petrischalen (und sogar inkubatorRaum) sparen kann, kann das Wachstum von Gewebe in überfüllten Platten ernsthaft gehemmt werden. Bei der Durchführung der Infektion sollte sichergestellt werden, dass die optische Dichte der Agrobacterium Suspension angemessen ist. Wenn die bakterielle Suspensionsdichte zu niedrig ist, kann eine richtige Infektion nicht auftreten.
Die Qualität der Ausgangsmaterialien ist entscheidend für den Erfolg von Transformationsprotokollen. Ohren, die für die Embryosektion verwendet werden, müssen gesund sein, was bedeutet, dass die Pflanze, die sie produziert, gesund ist. Sie müssen auch über ein angemessenes Saatgutset verfügen und schädlings- und krankheitsfrei sein. Auch sollte altes Agrobacterium nicht verwendet werden. Die "Mutter"-Platte sollte nicht mehr als 2 Wochen alt sein. Danach sollte eine neue "Mutter"-Platte gestreift werden, um neue Experimente zu beginnen.
Obwohl sich gezeigt hat, dass diese Methode weniger genotypabhängig ist, kann nicht davon ausgegangen werden, dass alle Linien gleichermaßen erfolgreich sein werden. Es kann immer noch Unterschiede zwischen den Linien sowie Unterschiede im Erfolg basierend auf dem verwendeten Konstrukt geben. Ohr-zu-Ohr-Variabilität ist auch bei der Arbeit mit unreifen Embryonen unvermeidbar, daher sollten Experimente idealerweise mehrere Ohren verwenden, um dies zu berücksichtigen. In dieser Arbeit schnitt die inzuchtw22 am besten ab, mit einer Transformationshäufigkeit von über 14 %, gefolgt von B73 und Mo17 (jeweils 4 %). Lowe et al.8 berichteten mit dem QuickCorn-Protokoll für die B73- und Mo17-Transformation. In dieser Arbeit reichten die Transformationsfrequenzen von 9%-50% für B73 und 15%-35% für Mo17.
Eine Möglichkeit für die niedrigeren Transformationsfrequenzen für B73 und Mo17, die in dieser Arbeit beobachtet wurden, kann auf saisonale Ohrqualitätsschwankungen zurückgeführt werden. Ein weiterer Unterschied zwischen dieser Arbeit und der von Lowe et al.8 ist, dass hier verschiedene Vektorkonstrukte verwendet wurden. In Lowes Arbeit wurden morphogene Gene nicht aus den transformierten Pflanzen entfernt, sondern in den späteren Stadien entwicklungsfördernd. In dieser Arbeit wurden die morphogenen Gene 8 Tage nach der Infektion entfernt. Es ist möglich, dass B73 und Mo17 eine längere Präsenz von Bbm/Wus2 für die Entwicklung somatischer Embryonen benötigen.
Mit dieser Methode besteht die Möglichkeit, nicht-transgene Entweichungspflanzen, multimere Einfügungen und nicht verbrauchende Transgene zu erhalten. Diese Pflanzen haben keinen deutlich anderen Phänotyp, daher ist die Detektion durch PCR erforderlich, um festzustellen, ob eine Pflanze transgen ist. Um dies zu erreichen, können PCR-Primer innerhalb der verbraucheten Region und Primer, die die verbrauchsteuernde Region flankieren, verwendet werden. Mehrere unabhängige Transformationen können auch Pflanzen aus demselben unreifen Embryo produzieren, was die Bestimmung einer völlig unabhängigen transformativen Rückgewinnungsrate erschwert. Unser Standard war es, eine Transformationsrate auf der Grundlage der Probenahme einer Pflanze aus jedem unreifen Embryo, der Pflanzen produzierte, zu berechnen und diese durch die Anzahl der infizierten Embryonen zu dividieren. Diese Methode unterschätzt mit ziemlicher Sicherheit die tatsächliche Anzahl unabhängiger Ereignisse, die als Pflanzen zurückgewonnen werden. Die Diskriminierung unabhängiger Ereignisse desselben Embryos erfordert die Sequenzierung von Grenzregionen um Transgene, und dies wird für die meisten Anwendungen unerschwinglich teuer und zeitaufwändig sein; Es kann jedoch Fälle geben, in denen diese Daten nützlich sind.
Diese Methode der Gewebekulturtransformation hat sich als sehr effektiv erwiesen, aber Es können immer noch Probleme auftreten. Wenn Pflanzenmaterial nicht reagiert, ist es möglich, dass es ein Problem mit der jeweiligen Inzuchtlinie gibt, was darauf hindeutet, dass Variablen wie die Zusammensetzung der Wachstumsmedien und der Zeitpunkt der Subkultivierung Anpassungen erfordern. Eine weitere Variable ist das richtige Vektordesign und die genaue Vektorkonstruktion, wenn der ursprüngliche Vektor geändert wird. Es kann auch Probleme mit imazapyr Empfindlichkeit sein, da einige Linien empfindlicher als andere sind, und die Konzentration von Imazapyr muss möglicherweise angepasst werden, um erfolgreich transformierte Pflanzen zu erreichen.
In den letzten 30 Jahren haben sich die Maisgewebekultur und die Transformationsprotokolle verändert und sind vorangekommen; und es wird angenommen, dass dieses verkürzte Protokoll diese Progression weiter vorantreiben wird. Diese Methode ist für akademische Umgebungen wirksam, da sie weniger zeitaufwändig ist als herkömmliche Methoden. Darüber hinaus verlangt sie keine hochqualifizierten Bediener, was eine breitere Verbreitung im Vergleich zu herkömmlichen Methoden ermöglicht. In Zukunft kann diese Methode mit neuen Technologien wie genome engineering kombiniert werden.
Offenlegungen
Alicia Masters, William Gordon-Kamm und Todd Jones sind Mitarbeiter von Corteva Agriscience, die das Protokoll und die Maisohren von B73, Mo17 und W22 geliefert haben, die in diesem Artikel verwendet werden. Die Autoren Minjeong Kang, Morgan McCaw, Jacob Zobrist und Kan Wang haben nichts zu verraten.
Danksagungen
Wir danken dem Corteva Gewächshausteam für die Bereitstellung von Mais unreifen Ohren, dem Corteva Media Prep Lab für die Bereitstellung von Medien, Ning Wang aus Corteva für die Unterstützung beim Agrobacterium-Konstrukt und Keunsub Lee von der Iowa State University für Unterstützung. Dieses Projekt wurde teilweise unterstützt durch das National Science Foundation Plant Genome Research Program Grant 1725122 und 1917138 an K.W., durch Predictive Plant Phenomics Research Traineeship Program (National Science Foundation Grant DGE-1545453) an J.Z., durch das USDA NIFA Hatch Projekt #IOW04341, durch State of Iowa Fonds und durch Crop Bioengineering Center der Iowa State University.
Materialien
| Name | Company | Catalog Number | Comments |
| 2,4-D | Millipore Sigma | D7299 | |
| 6-Benzylaminopurine (BAP) | Millipore Sigma | B3408 | |
| Acetosyringone | Millipore Sigma | D134406 | |
| Agar | Millipore Sigma | A7921 | |
| Aluminum foil | To cover the flask | ||
| Ammonium Sulfate | Millipore Sigma | A4418 | |
| Analytical balance | To weigh small quantities of chemicals | ||
| Autocalve | Primus (Omaha, NE) | PSS5-K | To autoclave media and tools |
| Bacterial culture loop (10 µl) | Fisher scientific | 22-363-597 | Collects Agrobacterium from plate to transfer to liquid |
| Bactoagar | BD bioscience | 214030 | |
| Beakers (1 L, 2 L, 4 L) | To mix the chemicals for media | ||
| Benomyl | Millipore Sigma | #45339 | |
| Bleach (8.25% Sodium Hypochlorite) | Clorox | For seed sterilization | |
| Boric Acid | Millipore Sigma | B6768 | |
| Calcium Chloride Dihydrate | Millipore Sigma | C7902 | |
| Carbenicillin | Millipore Sigma | C3416 | |
| Casein Hydrolysate | Phytotech | C184 | |
| Cefotaxime | Phytotech | C380 | |
| Conical tube (50 mL) | Fisher scientific | 06-443-19 | Contain liquid medium and Agro suspension |
| Cuvette (Semi-micro) | Fisher scientific | 14955127 | To hold liquid for measuring OD |
| Dicamba | Phytotech | D159 | |
| Digital hygrometer | Checking temperature and humidity for heat treatment | ||
| EDTA, Disodium Salt, Dihydrate | Millipore Sigma | 324503 | |
| Eppendorf tube (2.0 mL) | ThermoFischer Scientific | AM12475 | |
| Eriksson's Vitamins | Phytotech | E330 | 1000x in liquid |
| Ethanol (70%) | Sterilizing tools and surfaces | ||
| Ferrous Sulfate Heptahydrate | Millipore Sigma | F8263 | |
| Fertilizer, Osmocote Plus 15-9-12 | ICL Specialty Fertilizers (Dublin, OH) | A903206 | Fertilizer |
| Flask (2 L) | Pyrex | 10-090E | To autoclave media and tools |
| Flats (Standard 1020, open w/holes, 11"W x 21.37"L x 2.44"D) | Hummert International (Earth City, Mo) | 11300000 | Tray to hold soil and pot insert, fits Humidome |
| Forceps (fine-tipped and large) | Fine for handling embryos; larger for large plant materials and use as ear holders | ||
| Gentamicin | Gold Biotechnologies | G-400 | |
| Glass bottle (1 L) | Pyrex | 06-414-1D | To autoclave medium |
| Graduated cylinder | To adjust volume of media | ||
| Imazapyr | Millipore Sigma | 37877 | |
| Incubator, 20 °C | Percival Scientific | Model I-36NL | To grow mother plate and incubate embryos during Agro infection |
| Incubator, 27 °C | Percival Scientific | Model I-36NL | To grow co-cultivation plate and maize embryo culture |
| Incubator, 45 °C | Heat shock treatment | ||
| Insert TO Standard, pots | Hummert International (Earth City, Mo) | 11030000 | For transplanting plants from rooting to soil, fits flat and Humidome |
| Laminar flow hood | Maintains sterile conditions | ||
| L-proline | Phytotech | P698 | |
| Magnesium Sulfate Heptahydrate | Millipore Sigma | M1880 | |
| Maize inbred seed B73 | U.S National Plant Germplasm | id=47638 | |
| Maize inbred seed Mo17 | U.S National Plant Germplasm | id=15785 | |
| Maize inbred seed W22 | U.S National Plant Germplasm | id=61755 | |
| Manganese Sulfate Monohydrate | Millipore Sigma | M7899 | |
| Milli-Q Water purification systems | Millipore sigma | MILLIQ | For tissue culture grade water |
| MS Basal Medium | Millipore Sigma | M5519 | |
| MS Basal Salt Mixture | Millipore Sigma | M5524 | |
| N6 Basal Salt Mixture | Millipore Sigma | C1416 | |
| Paperclips, non-skid | Holding on tassel bags | ||
| Peptone | BD bioscience | 211677 | |
| Petri dish (100x15 mm) | Fisher scientific | FB0875713 | For bacteria culture medium |
| Petri dish (100x25 mm) | Fisher scientific | FB0875711 | For the plant tissue culture medium |
| pH meter | Fisher scientific | AB150 | To adjust pH of media |
| Pipette (1 mL) | ThermoFischer Scientific | 4641100N | |
| Plastic Boxes | The Container Store | 10048430 | For tissue culture storage and incubation |
| Plastic humidy dome (Humi-Dome) | Hummert International (Earth City, Mo) | 14385100 | Plastic cover for soil flat |
| Potassium Iodide | Millipore Sigma | 793582 | |
| Potassium Nitrate | Millipore Sigma | P8291 | |
| Potassium Phosphate Monobasic | Millipore Sigma | P5655 | |
| Scale | To weigh chemicals for media | ||
| Scalpel Blade (No. 11, 4 cm) | Thermo Scientific | 3120030 | remove the top of the kernel crowns for embryo dissection |
| Scalpel handle | Holding scalpel blades | ||
| Schenk & Hildebrandt Vitamin (S&H vitamin) | Phytotech | S826 | 100x powder |
| Scissors | Cutting ear shoots | ||
| Shoot bag (Canvasback- semi-transparent) | Seedburo (Des Plaines, IL) | S26 | Semi-transparent bag to cover ear shoots |
| Silver Nitrate | Millipore Sigma | S7276 | |
| Sodium Molybdate Dihydrate | Millipore Sigma | M1651 | |
| Soiless substrate LC1 | SunGro Horticulture (Agawam, Ma) | #521 | For growing maize plants |
| Spatula (Double Ended Micro-Tapered) | Fischer Scientific | 2140110 | Dissecting embryos from kernels |
| Spatula (with spoon) | Fisher scientific | 14-375-10 | To measure chemicals for media |
| Spectinomycin | Millipore Sigma | S4014 | |
| Spectrophotometer (Genesys 10S UV-Vis) | Thermo Scientific | 840-300000 | Measure OD of Agro suspension |
| Stirring bar | Fisher scientific | 14-513-67 | To mix media |
| Stirring hotplates | To mix media | ||
| Syringe (without needle, 60 mL) | Fisher scientific | 14-823-43 | For filter sterilization |
| Syringe filter (0.22 µm) | Fisher scientific | 09-720-004 | For filter sterilization |
| Tassel bag (Canvasback- brown) | Seedburo (Des Plaines, IL) | T514 | Bag to cover tassels of non-transgenic plants |
| Tassel bag (Canvasback-green stripe) | Seedburo (Des Plaines, IL) | T514G | Bag to cover tassels of transgenic plants |
| Thiamine HCl | Phytotech | T390 | |
| Thidiazuron | Phytotech | T888 | |
| Thymidine | Millipore Sigma | T1895 | |
| Timentin | Phytotech | T869 | |
| Tween 20 | Fisher Scientific | Cas #9005-64-5 | surfactant |
| Vortex Genie 2 | Scientific Industries | SI0236 | Homogenizes liquids (Agro suspension) |
| Water bath (large - Precision model 186) | Fisher scientific | any that can fit 4+ 2L flasks and reach 55 °C | Keeps autoclaved media at optimal temperature |
| Weigh dish | Fisher scientific | 08-732-112 | To measure chemicals for media |
| Weighing paper | Fisher scientific | 09-898-12A | To measure chemicals for media |
| Yeast Extract | Fisher Scientific | BP14222 | |
| Zeatin | Millipore Sigma | Z0164 |
Referenzen
- Zhao, Z. Y., et al. High throughput genetic transformation mediated by Agrobacterium tumefaciens in maize. Molecular Breeding. 8 (4), 323-333 (2002).
- Green, C. E., Phillips, R. L. Plant regeneration from tissue cultures of maize. Crop Science. 15 (3), 417-421 (1975).
- Ji, Q., Xu, X., Wang, K. Genetic transformation of major cereal crops. International Journal of Developmental Biology. 57, 495-508 (2013).
- Frame, B., Warnberg, K., Main, M., Wang, K., Wang, K. Maize (Zea mays, L). Agrobacterium Protocols. , 101-117 (2015).
- Frame, B., et al. Improved Agrobacterium-mediated transformation of three maize inbred lines using MS salts. Plant Cell Reports. 25 (1), 1024-1034 (2006).
- Que, Q., et al. Maize transformation technology development for commercial event generation. Frontiers in Plant Science. 5 (379), (2014).
- Lowe, K. S., et al. Morphogenic regulators Baby boom and Wuschel improve monocot transformation. The Plant Cell. 28 (9), 1998-2015 (2016).
- Lowe, K. S., et al. Rapid genotypes independent maize transformation via direct somatic embryogenesis. In Vitro Cellular & Developmental Biology - Plant. 54 (3), 240-252 (2018).
- Boutilier, K., et al. Ectopic expression of BABY BOOM triggers a conversion from vegetative to embryonic growth. The Plant Cell. 14 (8), 1737-1749 (2002).
- Zuo, J., Niu, Q. W., Frugis, G., Chua, N. H. The WUSCHEL gene promotes vegetative-to-embryonic transition in Arabidopsis. The Plant Journal. 30 (3), 349-359 (2002).
- Jones, T. J., Kumar, S., Barone, P., Smith, M., et al. Maize transformation using the morphogenic genes Baby Boom and Wuschel2. Transgenic Plants. , 81-93 (2019).
- Anand, A., et al. An improved ternary vector system for Agrobacterium-mediated rapid maize transformation. Plant Molecular Biology. 97 (1-2), 187-200 (2018).
- Ray, K., et al. Mutant acetolactate synthase gene is an efficient in vitro selectable marker for the genetic transformation of Brassica juncea (Oilseed Mustard). Journal of Plant Physiology. 161 (9), 1079-1083 (2004).
- Green, J. M., Hale, T., Pagano, M. A., Andreassi, J. L., Gutteridge, S. A. Response of 98140 corn with gat4621 and hra transgenes to glyphosate and ALS-inhibiting herbicides. Weed Science. 57 (2), 142-148 (2009).
- An, G., et al. Functional analysis of the 3' control region of the potato wound-inducible proteinase inhibitor II gene. The Plant Cell. 1 (1), 115-122 (1989).
- Matz, M. V., et al. Fluorescent proteins from nonbioluminescent Anthozoa species. Nature Biotechnology. 17 (10), 969-973 (1999).
- Passarinho, P., et al. Target Genes Provide Diverse Entry Points into Cell Proliferation and Cell Growth Pathways. Plant Molecular Biology. 68 (3), 225-237 (2008).
- Bhyri, P., Khrishnamurthy, N., Narayanan, E., Nott, A., Sarangi, R. R. Novel plant terminator sequences. Patent Number US2014/0130205. , (2014).
- Laux, T., Mayer, K. F., Berger, J., Jürgens, G. The WUSCHEL gene is required for shoot and floral meristem integrity in Arabidopsis. Development. 122 (1), 87-96 (1996).
- Garnaat, C., Lowe, K., Roth, B. Zm-AXIG1-specific polynucleotides and methods of use. Patent Number WO2002006499. , (2002).
- Hershey, H. P., Stoner, T. D. Isolation and characterization of cDNA clones for RNA species induced by substituted benzenesulfonamides in corn. Plant Molecular Biology. 17 (4), 679-690 (1991).
- Abremski, K., Hoess, R. Bacteriophage P1 site-specific recombination. Purification and properties of the Cre recombinase protein. Journal of Biological Chemistry. 259 (3), 1509-1514 (1984).
- Sun, A. Q., et al. Cloning and Function Analysis of Small Heat Shock Protein Gene ZmHSP17.7 from Maize. ACTA Agronomica Sinica. 41 (3), 414 (2015).
- Sauer, B., Henderson, N. Site-specific DNA recombination in mammalian cells by the Cre recombinase of bacteriophage P1. Proceedings of the National Academy of Sciences of the United States of America. 85 (14), 5166-5170 (1988).
- Hershfield, V., Boyer, H. W., Yanofsky, C., Lovett, M. A., Helinski, D. R. Plasmid ColEl as a molecular vehicle for cloning and amplification of DNA. Proceedings of the National Academy of Sciences of the United States of America. 71 (9), 3455-3459 (1974).
- Liebert, C. A., Hall, R. M., Summers, A. O. Transposon Tn21, flagship of the floating genome. Microbiology and Molecular Biology Reviews. 63 (3), 507-522 (1999).
- Nishiguchi, R., Takanami, M., Oka, A. Characterization and sequence determination of the replicator region in the hairy-root-inducing plasmid pRiA 4b. Molecular and General Genetics. 206 (1), 1-8 (1987).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten