Method Article
Agrobacterium-Transformação de embrião imaturo mediado de linhas de milho recalcitrante usando genes morfogênicos
Neste Artigo
Resumo
Genes morfogênicos vegetais podem ser usados para melhorar a transformação genética de genótipos recalcitrantes. Descrito aqui está um protocolo de transformação genética mediado pela Agrobacterium(QuickCorn) para três importantes linhas de milho público.
Resumo
Demonstrado aqui está um protocolo detalhado para a transformação genética mediada pela Agrobacteriumde linhas de milho usando genes morfogênicos Baby boom (Bbm)e Wuschel2 (Wus2). O BBM é regulado pelo promotor do gene transferase fosfolipid de milho(Pltp),e Wus2 está o controle de um promotor de auxin-indutor de milho(Axig1). Uma cepa agrobacterium que carrega esses genes morfogênicos no DNA de transferência (T-DNA) e cópias extras de virulence agrobacterium (vir)são usadas para infectar explantas de embriões imaturos de milho. Embriões somáticos formam-se na scutella de embriões infectados e podem ser selecionados pela resistência ao herbicida e germinados em plantas. Um sistema de recombinação cre/loxP ativado pelo calor embutido na construção de DNA permite a remoção de genes morfogênicos do genoma do milho durante um estágio inicial do processo de transformação. Podem ser alcançadas frequências de transformação de aproximadamente 14%, 4% e 4% (número de eventos transgênicos independentes por 100 embriões infectados) para W22, B73 e Mo17, respectivamente, utilizando este protocolo.
Introdução
A transformação é uma ferramenta básica para avaliar a expressão genética estrangeira no milho e produzir linhas de milho geneticamente modificadas para fins de pesquisa e comerciais. O acesso à transformação de alta produtividade pode facilitar a maior necessidade de estudos de biologia molecular e celular de milho1. A capacidade de transformar geneticamente espécies culturais é vital para laboratórios públicos e privados. Isso permite tanto a compreensão fundamental dos mecanismos de regulação genética quanto a melhoria das culturas em escala global para apoiar uma população em constante crescimento.
A descoberta de que embriões imaturos do milho poderia ser usado para a produção de calo regenerado originado em 19752. Desde essa revelação, a maioria dos protocolos de transformação escalável do milho exigiu formação e seleção de calus antes da regeneração3. Durante o processo de transformação genética, embriões imaturos infectados pelo Agrobacteriumou biolísticos são cultivados na mídia para indução embrionária de calus. Calli induzido é então cultivado em mídia seletiva (por exemplo, contendo um herbicida) para que apenas peças de calo transformados sejam capazes de sobreviver. Estes calli transgênicos resistentes ao herbicida são ampliados e regenerados em plantas. Embora este método seja eficaz, o processo é longo e intensivo em mão-de-obra, e pode levar mais de 3 meses para completar4. Mais importante, os protocolos convencionais de transformação do milho possuem uma limitação muito maior, ou seja, apenas um número limitado de genótipos de milho pode ser transformado5,6.
Lowe et al.7,8 relataram anteriormente um método de transformação "QuickCorn" que não só reduziu muito a duração do processo de transformação, mas também ampliou a lista de genótipos transformáveis. O método QuickCorn utiliza ortologs de milho (Zm-Bbm e Zm-Wus2) dos fatores de transcrição da Arabidopsis BABY BOOM (BBM)9 e WUSCHEL (WUS)10. Quando incorporados no sistema vetor de transformação, esses genes trabalham sinergicamente para estimular o crescimento embrionário7.
O protocolo QuickCorn descrito neste trabalho foi baseado no protocolo em Jones et al11, que foi uma melhoria adicional do método relatado por Lowe et al7,8. No presente estudo, uma cepa de Agrobacterium LBA4404(Thy-) abrigando uma construção vetorial binária PHP81430(Figura 1) e o plasmídeo acessório PHP7153912 são usados para transformação. O T-DNA do PHP81430 contém os seguintes componentes moleculares. (1) O marcador seletivo de transformação gene Hra expressão. O gene hra de milho (Zm-Hra) é um gene modificado de sintetizador acetolactase (ELA) tolerante a herbicidas inibidoras da ELA, como sulfonylureas e imidazolinonas13,14. O gene Zm-Hra é regulado pelo promotor da ALS8 e inibidor de proteína de batata II(pinoII) exterminador15. O T-DNA também contém (2) um de expressão possuindo o gene de marcador de transformação zsGreen. Este gene de proteína fluorescente verde ZsGreen de Zoanthus sp. recife coral16 é regulado por um promotor de ubiquidade de sorgo/intron e ubiquilina de arroz.
Além disso, o T-DNA contém (3) o gene morfogênico de expressão bbm. Bbm é um fator de transcrição associado ao desenvolvimento de embriões9,17. O BBM é regulado pela proteína transferase fosfolipida de milho(Pltp)promotor8 e arroz T28 exterminador18. Zm-Pltp é um gene com forte expressão no epitélio de scutellar embrião, pelos de seda e células subsidiárias de folhas (flanqueando as células de guarda), baixa expressão em órgãos reprodutivos e nenhuma expressão nas raízes8. Também contém (4) o gene morfogênico wus2 fita de expressão Wus2. Wus2 é outro fator de transcrição associado à manutenção do meristem apical19. Zm-Wus2 está o controle de um promotor de auxin-indutor de milho (Zm-Axig1)20 e milho In2-1 exterminador21. Finalmente, o T-DNA contém (5) o sistema de recombinação cre-loxP. O cre recombinase gene22 está o controle da proteína de choque térmico do milho 17.7 (Hsp17.7)23 promotor e batata pinoII exterminador. Dois sites loxP (na mesma orientação)24 flanqueiam quatro fitas de expressão genética, incluindo ZsGreen, cre, Bbm e Wus2.
Como a presença dos genes morfogênicos não é desejada para a maturidade da planta e a prole subsequente, o sistema de recombinação cre-loxP induzido pelo calor foi construído no DNA T para remover genes morfogênicos do genoma do milho para permitir a regeneração normal do calus e o desenvolvimento de plantas. Após o tratamento térmico, a expressão da proteína CRE remove todos os transgenes, exceto o gene de seleção de Hra. Transformantes bem sucedidos devem ser resistentes ao herbicida, mas ZsGreen-negativo. Para aumentar ainda mais a frequência de transformação, a cepa Agrobacterium também abriga um plasmídeo adicional de acessórios (PHP71539) que possui cópias extras dos genes agrobacterium de virulence(vir)genes 12.
O método QuickCorn é diferente dos protocolos convencionais de transformação do milho, pois não envolve um passo de indução de calus durante a transformação. Durante a primeira semana após a infecção com agrobacterium,embriões somáticos desenvolvem-se no epitélio scutellar dos embriões imaturos infectados. Os embriões são então transferidos para um meio com hormônios que incentivam a maturação de embriões e a formação de tiros. Transferir rapidamente os embriões somáticos para o meio de formação de maturação/tiro ignora a etapa tradicional de calus anteriormente usada para a transformação do milho e permite a geração direta de plantas T08. Em comparação com os métodos de transformação de milho publicados anteriormente6,o método QuickCorn é mais rápido, eficiente e menos dependente do genótipo. Usando este método, as plantas enraizadas normalmente estão prontas para serem transferidas para o solo em apenas 5-7 semanas, em vez dos três ou mais meses exigidos pelos protocolos tradicionais. O objetivo deste artigo é fornecer uma descrição aprofundada e demonstração do método, permitindo uma replicação mais fácil em um ambiente de laboratório tipicamente encontrado na maioria das instituições acadêmicas.
Protocolo
1. Preparação da mídia de crescimento
- Para obter receitas médias de crescimento exatos para este protocolo, consulte a Tabela 1.
- Para preparar 1 L de mídia, coloque um béquer de 2 L em uma placa de agitação e coloque uma barra de agitação dentro.
- Encha o béquer com 900 mL de água destilada e ligue a placa de agitação. A barra de agitação deve estar girando a uma velocidade média.
- Pesar todos os ingredientes em pó e dissolver-se em um béquer.
- Meça todos os ingredientes líquidos, se houver, e adicione ao béquer.
- Leve o volume final para 1 L usando água destilada.
- Meça o pH e ajuste as especificações da receita.
- Se formular um meio de crescimento líquido, nenhum ágar é adicionado. Conecte um esterilizador de filtro a uma bomba de vácuo e despeje o meio de crescimento líquido através do filtro. Ligue a bomba e espere até que todo o líquido seja puxado. Coloque uma tampa no recipiente e conecte um rótulo.
- Se formular um meio de crescimento sólido, depois que o pH for ajustado, adicione ágar diretamente em uma garrafa ou frasco.
- Despeje o meio de crescimento líquido de 1 L em um frasco de 2 L Erlenmeyer, ou divida-o em duas garrafas autoclaváveis de 1 L (500 mL cada). Se duas garrafas forem usadas, divida o ágar e adicione diretamente às garrafas.
- Frasco de cobertura com uma tampa respirável, como duas camadas de papel alumínio, para permitir que o vapor escape. Se usar uma garrafa, cubra a tampa do parafuso na parte superior.
- Autoclave a 121 °C por 25 min.
- Após a autoclaving, remova o meio de crescimento do autoclave e esfrie para 55-60 °C (um banho de água definido para 55 °C pode facilitar isso). Mantenha o meio de crescimento em um estado líquido por algumas horas até que seja conveniente derramar placas.
- Uma vez resfriado, adicione todos os aditivos pós-esterilização (ver Tabela 1) e misture bem.
- Depois de todos os ingredientes serem adicionados, despeje o volume designado no recipiente de escolha em um capô de fluxo laminar.
- O meio de crescimento pode ser derramado em pratos de Petri desejados manualmente ou usando um aparelho de dispensação líquida. Ao derramar manualmente, recomenda-se transferir um grande volume de meio autocelava para um béquer estéril menor (500 mL) para facilitar o manuseio.
- Permita que o meio de crescimento esfrie e solidifique.
- O meio de crescimento estará disponível para uso uma vez que se torne sólido e é melhor utilizado no dia seguinte depois de secar ligeiramente durante a noite em um capô de fluxo estéril como pilhas de placas tampadas. Depois de secar durante a noite, transfira as placas para mangas plásticas, dobre sobre a ponta solta e mantenha isso no lugar com um pequeno pedaço de fita. Isso evita a secagem excessiva. O meio pode ser armazenado em um ambiente frio, escuro e limpo (4-16 °C) por até 1 mês.
2. Cultivar plantas doadoras e colher orelhas imaturas
- Cultivar qualquer milho disponível publicamente (ou seja, B73, Mo17 ou W22) em uma estufa em potes de 5,9 l de 1,5 galão contendo um substrato sem solo. Use um período fotográfico 16/8 (dia/noite), com temperaturas médias de 25,5 °C durante o dia e 20 °C à noite.
- As plantas são regadas conforme necessário e fertilizadas com um fertilizante de liberação controlada (N-P-K de 15-9-12), que pode ser incorporado na mistura do solo ou adicionado à superfície após o plantio.
- Normalmente leva cerca de 70-90 dias após a germinação de sementes para que as orelhas emergam. À medida que as filmagens de ouvido emergem, cubra-os com um saco de tiro para evitar que a polinização descontrolada ocorra.
- Cerca de 2-3 dias após a emerção de sedas e se o pólen estará disponível no dia seguinte, corte as sedas usando tesouras que foram esterilizadas em 70% de etanol. Corte as sedas e a casca cerca de 2,5 cm abaixo da extremidade das folhas de casca, onde as sedas emergem. A polinização pode ser realizada no dia seguinte. Certifique-se de reesterilizar tesouras entre cada orelha.
- Uma vez que os anteras emergem de um tassel, cubra o tassel com uma bolsa tassel e clipe de papel não-derrapado na base do saco ao redor do talo.
- Na manhã seguinte, dobre suavemente a planta e toque no saco para incentivar a liberação do pólen.
- Retire o saco tassel e dobre a parte superior do saco para evitar que o pólen escape. Geralmente é melhor ensacar o tassel 1 dia antes de ser usado (para evitar o acúmulo de pólen morto e anters derramados). O pólen fresco pode ser coletado de tassels por cerca de 3-5 dias. Quando os anteristas emergem dos floretes internos na base do tassel, esse tassel provavelmente não produzirá pólen viável no dia seguinte.
- Use o pólen da mesma planta (autoestima) ou de outra planta da mesma raça (escoamento).
- Retire o saco de ouvido ou corte a extremidade do saco para expor as sedas e, em seguida, despeje rapidamente pólen do saco tassel sobre as sedas.
- Cubra a orelha com a bolsa tassel imediatamente e grampeie a base do saco ao redor do talo para protegê-la. Pode ser útil isolar fisicamente a planta de plantas floradas de diferentes genótipos durante a polinização para ajudar a evitar a polinização cruzada. Deixe o saco tassel na orelha até que a orelha imatura esteja pronta para colher.
- 9-12 dias após a polinização, orelhas de tela para o tamanho do embrião. Deslize o saco de polinização até o talo para expor a orelha. Puxe suavemente a casca para baixo para expor os grãos em cerca de um terço a um quarto da circunferência da orelha e cerca de um terço da distância para baixo da orelha. Os grãos próximos à ponta não serão representativos do tamanho médio do embrião.
- Usando um bisturi, corte a tampa de um único kernel que aparece semelhante à maioria dos outros núcleos em tamanho e cor.
- Use uma espátula (com uma régua) para remover o embrião conforme descrito na etapa 4.6. Meça o comprimento do embrião usando uma régua embutida na espátula ou uma pinça digital. Se o embrião estiver entre 1,5-2,0 mm, colher a orelha. Se for ~1,3 mm, a orelha pode estar pronta para colher mais tarde e pode ser verificada novamente em cerca de 7-8 h.
3. Preparando cultura de suspensão agrobacterium para infecção
NOTA: A cepa agrobacterium LBA4404(Thy-) contendo PHP81430 (Figura 1) e PHP7153912 é armazenada como estoque de glicerol a -80 °C. Esses materiais podem ser obtidos da Corteva Agriscience por meio de um Contrato de Transferência de Materiais. LBA4404 (Thy-) é uma cepa auxotrófica que precisa de timmidina fornecida na mídia de crescimento. A utilidade primária da cepa auxotroph Agro é para fins de biocontenção. Tem o benefício adicional de reduzir o crescimento excessivo do Agro. A cepa auxotrófica agro não cresce sem timmidina suplementar. No entanto, a lata de timmidina (presumivelmente) pode ser fornecida pelo tecido vegetal moribundo na cultura. Portanto, ainda há a necessidade de fornecer um antibiótico no meio para controlar completamente o agro auxotrófico. No entanto, será mais fácil de controlar devido ao crescimento comprometido da cepa auxotrófica na ausência de timmidina.
- Quatro dias antes da data da infecção, inicie uma placa "mãe" do estoque glicerol, espalhando a bactéria em uma placa yp com 50 mg/L de thymidina, 50 mg/L gentamicina, e 50 mg/L spectinomicin(Tabela 1). Incubar a placa "mãe" em uma incubadora de 20 °C por 3 dias.
- Um dia antes do experimento de infecção, prepare uma placa de "trabalho" selecionando uma a cinco colônias da placa "mãe" e espalhando as bactérias da placa "mãe" para uma nova placa yp (com timmidina, gentamicina e espectinomicina; Tabela 1).
- Estique a placa diária de "trabalho" em quadrantes sequenciais e execute o loop 1x através da área apenas-listras para o quadrante sucessivo, repetindo para formar quadrantes que foram diluídos serialmente. Incubar a placa de "trabalho" durante a noite em uma incubadora de 27 °C.
- Após completar a dissecção de embriões (passo 4.8), use um loop ou ferramenta semelhante para coletar Agrobacterium de uma região da placa "funcionando", onde o crescimento bacteriano é visível como finas listras de colônias.
NOTA: Evite áreas da placa com um gramado denso de crescimento bacteriano. O crescimento do Agrobacterium provavelmente já começou a diminuir em áreas densas, enquanto nas áreas com colônias visíveis as bactérias estão em fase adequada de crescimento para infecção. - Suspender as bactérias coletadas em um tubo de 50 mL contendo 10 mL de 700A líquido médio(Tabela 1). Vórtice para suspender completamente a cultura das bactérias.
- Meça a densidade óptica a um comprimento de onda de 550 nm. Ajuste o volume até que o OD esteja entre 0,35-0,45, sendo 0,4 o valor ideal.
NOTA: Se o OD for superior a 0,45, adicione mais 700A de meio líquido. Se o OD for inferior a 0,35, inocule mais colônias agro na cultura suspensa.
4. Dissecação de embriões, infecção e co-cultivo
- Selecione ouvidos adequados para experimentos de transformação; estes devem ter um bom conjunto de sementes e ter embriões que variam em tamanho de 1,5-2,0 mm. Eles são tipicamente colhidos entre 9-12 dias após a polinização. Os ouvidos colhidos podem ser usados frescos ou armazenados por 1-4 dias a 4 °C, embora a qualidade da resposta provavelmente se degradará progressivamente com armazenamento prolongado além do primeiro dia.
- Remova as cascas e sedas. Insira uma alça na base ou na parte superior da orelha. A alça pode ser um par de fórceps, chave de fenda, etc.
- Coloque as orelhas em um recipiente grande (por exemplo, 2 L beaker com a alça para cima, encha o recipiente com solução de desinfecção. A solução de desinfecção é de 1,8 L de 20% de alvejante comercial (1,65% hipoclorito de sódio) e algumas gotas de Tween 20 surfactant.
- Esterilizar as orelhas dentro de um banco de fluxo laminar. Após 20 min, esvazie a solução de alvejante e enxágue as orelhas 3x (5 min cada) usando uma generosa quantidade de água destilada estéril. Retire a água e deixe as orelhas secarem por vários minutos.
NOTA: É importante que as orelhas fiquem completamente submersas na solução alvejante por 20 min. Mova as orelhas cuidadosamente ao redor da solução alvejante ocasionalmente para desalojar bolhas de ar. - Prepare um tubo de microcentrífuga de 2 mL preenchido com meio líquido 700A. Este tubo será usado para coletar os embriões imaturos.
- Pegue a orelha e use um bisturi estéril, remova o 1-2 mm superior das coroas do kernel para expor o endoesperma. Use uma micro espátula para remover o embrião zigótico imaturo (IZE). O IZE estará localizado dentro do kernel, na lateral de frente para a ponta da orelha, e perto do acessório à espiga. Usando a espátula, insira-a no endoesperma no pericarpo mais distante do embrião, em seguida, torça suavemente para cima para desalojar o endoesperma e permitir a remoção do embrião (Figura 2).
- Usando a espátula, transfira o embrião para o tubo contendo o meio líquido 700A. Continue fazendo isso até que até 100 embriões tenham sido coletados. Vários tubos podem ser preenchidos (~100 embriões/tubo) antes de seguir para o próximo passo. Neste ponto, a suspensão agrobacterium deve ser preparada (ver etapa 3.5).
- Remova o meio líquido 700A do tubo do embrião com uma pipeta de 1 mL. Adicione um meio fresco de 700A para lavar os embriões e, em seguida, remova essa mídia também.
- Adicione 1 mL da suspensão agrobacterium e vórtice em uma configuração baixa (3/10) para 30 s ou tubo invertido 12x-15x para misturar. Deixe este tubo descansar horizontalmente no banco por 5 min.
- Após 5 min, transfira todo o tubo de embriões e suspensão do Agrobacterium para uma placa de 562V de co-cultivo médio (Tabela 1). Isso pode ser alcançado colocando a placa no banco e rapidamente derramando o conteúdo do tubo na placa. Gire suavemente a placa para distribuir os embriões e remover a suspensão do Agrobacterium usando uma pipeta de 1 mL.
- Certifique-se de que os embriões são colocados com o lado scutellum (redondo) voltado para cima. Use uma lupa ou um escopo dissecando, se necessário. Coloque as placas em caixas plásticas (19 cm x 28 cm x 5,1 cm) e incuba as placas durante a noite 16-18 h a 21 °C no escuro. Não é necessário embrulhar placa individual usando filme parafina ou fita de ventilação.
- Após o cocultivo noturno, mova os embriões infectados, lado do scutellum para cima, para o meio de repouso 605T (Tabela 1). Coloque cerca de 30 embriões por prato. Incubar as placas de 26-28 °C no escuro.
- Incubar por 4 a 10 dias (7 dias é preferido). Neste momento, o desenvolvimento de embriões somáticos pode ser observado na superfície do scutellum zigótico (Figura 3).
5. Seleção, tratamento térmico e regeneração
- Após o período de descanso, o calor choca os embriões. Coloque a caixa contendo as placas de embriões em uma incubadora de 45 °C com 70% de umidade relativa por 2h. Em seguida, retire a caixa da incubadora de 45 °C e coloque na incubadora escura 26-28 °C por 1-2h.
NOTA: Se não conseguir atingir 70% de umidade em uma incubadora, adicione uma dupla camada de toalhas de papel autocelavadas na parte inferior da caixa e mergulhe com água autocelava para manter a umidade dentro da caixa. Devolva as placas para a caixa em cima das toalhas de papel e feche a tampa antes de colocar a 45 °C. Use um pequeno hygrometro/termômetro digital para monitorar a temperatura e a umidade. - Transfira os IZEs tratados com calor do meio de repouso para o meio de formação de tiro (13329A) contendo 0,05 mg/L imazapyr como agente seletivo (Tabela 1). Ao transferir, remova coleoptiles, se estiver presente, usando fórceps de ponta fina ou tesoura cirúrgica.
- Coloque 10-15 embriões por prato para evitar a superlotação. Mantenha os embriões neste meio por 2 semanas na incubadora escura de 26 °C.
- Transfira os embriões para o meio de raiz (13158; Tabela 1) por 1-2 semanas. Coloque cerca de oito peças por prato e incuba em uma sala de luz ou câmara de luz (16 dias/8 noite, 20-150 μmol/m2/s) a 27 °C.
- À medida que as plantas se desenvolvem, coloque plantas mais fortes contendo ambos os brotos e raízes vigorosas em novas placas de raiz média, coloque uma planta por prato. Isso permitirá um crescimento mais forte da plantação. Coloque as placas na sala de luz ou câmara de luz por mais 7-14 dias.
NOTA: Remova cuidadosamente quaisquer peças de calo associadas à plantapara garantir que ela esteja em bom contato com o meio. - À medida que a planta se torna mais vigorosa, remova a planta do meio de enraizamento e enxague as raízes com água da torneira para remover ágar.
- Transplante de plantas individuais em 3 em2 (~19 cm2) vasos contendo um substrato pré-molhado sem solo. Coloque os potes em uma bandeja (27 cm x 54 cm) com orifícios de drenagem e cubra o apartamento com uma cúpula de umidade plástica. Essa etapa de aclimatação pode ser alcançada tanto na câmara de crescimento quanto na estufa com condições de crescimento descritas na seção 2 (etapa 2.1) acima.
6. Transplante para a estufa e a produção de sementes T1
- Verifique as plantas 2x por dia. Água conforme necessário. Certifique-se de que as plantas não estão secas nem com água excessiva. Manter um substrato ligeiramente seco incentiva o crescimento das raízes.
NOTA: A cúpula de umidade pode ser removida de 4 a 7 dias após o transplante. As plantas devem ser cultivadas nesses pequenos vasos até que tenham se recuperado visivelmente do estresse do transplante para o solo. Isso deve levar cerca de 9-14 dias. - Transplante todo o plugue sem solo e plantação em uma panela de 1,5 gal (5,9 L). Mantenha na estufa e na água quando o solo se sentir seco ao toque.
- Adicione um fertilizante de liberação controlada com N-P-K de 15-9-12 ao pote, que pode ser incorporado na mistura de substrato ou aplicado à superfície.
- Quando as filmagens começarem a emergir da planta, use um saco de tiro para cobrir os ensaios auditivos. Certifique-se de usar um saco semi-transparente para que as sedas emergentes possam ser observadas sem a remoção do saco. O saco de tiro permite que a polinização controlada ocorra. É importante sempre empacotar tassels transgênicos.
- Depois que as sedas surgirem (1-2 dias), corte as sedas emergiu para um comprimento uniforme. Isso será cerca de 2,5 cm abaixo do topo das folhas de casca. Use uma tesoura limpa que foi esterilizada em 70% de etanol. Ao aparar as sedas, um tufo uniforme desenvolve-se no dia seguinte para que a polinização ocorra.
- Para a maioria dos genótipos de milho, o tempo ideal para a polinização é de 2-3 dias após o surgimento de tassel ou seda.
- Colete o pólen da mesma planta (se for autopolinada) ou de um tipo selvagem do mesmo íntado (se atravessar ou atravessar).
- Colete o pólen em um saco de tassel e aplique-o ao tufo de seda da planta T0. Se o pólen é de uma planta do tipo selvagem (não transgênica), coloque o pólen em um saco de tassel marrom simples. Se o pólen é de uma planta transgênica, coloque o pólen em um saco listrado verde para indicar que o pólen é transgênico.
- Siga as etapas 2.5-2.10 para detalhes de polinização.
- Cerca de 2 semanas após a polinização, remova os sacos tassel das orelhas e permita que a seca comece. Para ajudar a secar, pare de regar a planta 21-25 dias após a polinização. Você também pode puxar para trás as folhas de casca para expor a semente. Essa prática também ajuda a prevenir o.
- Cerca de 45 dias após a polinização, colher sementes e armazenar em armazenamento frio a 4-12 °C.
Resultados
Demonstrado aqui está um protocolo passo a passo para a transformação genética mediada pela Agrobacteriumde três linhas de milho público (B73, Mo17 e W22) que têm sido significativas no campo da genética do milho. A transformação das três linhas de raça não pôde ser alcançada utilizando os protocolos convencionais de transformação do milho5. A Figura 1 e a Figura 2 mostram a construção e os materiais iniciais, respectivamente, utilizados aqui. Os ouvidos são geralmente colhidos 9-12 dias após a polinização. Izes com comprimentos que variam entre 1,5-2,0 mm são as melhores explantas para transformação para este protocolo (Figura 2).
Oito dias após a infecção, os embriões somáticos zsGreen-expressando foram visualizados o canal GFP de um microscópio fluorescente(Figura 3). Izes infectados foram submetidos ao tratamento térmico 8 dias após a infecção (etapas 5.1 e 5.2). Este tratamento induziu a expressão de CRE recombinase que excisía os de expressão Bbm, Wus2, cree ZsGreen ladeados entre os dois sites loxP (Figura 1). Os tecidos tratados com calor foram então cultivados em meio de formação de tiros contendo o herbicida imazapyr para seleção de tecido transformado após a remoção de genes morfogênicos.
Foram observados tecidos proliferadores com embriões amadurecidos ou botões de disparo resistentes ao imazapyr cerca de 3-4 semanas após a infecção (Figura 4). Alguns tecidos resistentes a imazapyr foram negativos para zsGreen, sugerindo que a excisão mediada por creprovavelmente ocorreu nesses tecidos(Figura 4). Depois que os tecidos foram movidos para a incubação média e leve, as filmagens começaram a se desenvolver(Figura 5). Foram colhidos ensaios de crescimento saudáveis e vigorosos com raízes bem desenvolvidas (Figura 5). Alguns tecidos pareciam ter várias filmagens(Figura 5E,F,G). Esse tipo de regenerador "gramado" pode ser devido a plantas clonais com padrões idênticos de integração transgênica. A análise biológica molecular é necessária para genótipo dessas plantas.
Todas as três linhas públicas de raça responderam bem usando este protocolo, bem como a construção utilizada neste trabalho. O W22 produziu a maior frequência de brotos resistentes a imazapyr, com uma frequência de aproximadamente 14% (cerca de 14 brotos transgênicos por 100 embriões imaturos infectados). Tanto b73 quanto Mo17 produziram cerca de 4% de brotos transgênicos. Essas frequências indicam todos os brotos transgênicos, incluindo ambas as plantas que carregam os genes morfogênicos e plantas com o gene morfogênico removido pela excisão mediada pela CRE.

Figura 1: Representação esquemática da região de DNA T do plasmídeo binário PHP81430. RB = fronteira t-DNA direita; loxP = CRE recombinase site alvo; Axig1pro:Wus2 = promotor auxin-indutor de milho(Zm-Axig1) + Zm-Wus2 + milho In2-1 exterminador; Pltppro:Zm-Bbm = proteína transferase fosfolipid de milho (Zm-Pltp) promotor + Zm-Bbm + arroz T28 exterminador(Os-T28); Hsppro:cre = proteína de choque térmico de milho 17,7 promotor (Zm-Hsp17.7) + gene cre recombinase + inibidor de proteína de batata II (pinII) exterminador; Ubipro:ZsGreen = sorghum ubiquitin promoter/intron (Sb-Ubi) + proteína fluorescente verde ZsGreen gene + arroz ubiquitin terminator(Os-Ubi); Cassete de hra = sorgo acetolactase synthase(Sb-Als) promotor + milho Hra(Zm-Hra) gene + pinII terminator; LB = borda T-DNA esquerda; colE1, origem de replicação do plasmídeo ColE125; SpecR = gene resistente à espectinomicina aadA1 de Tn21 para seleção de bactérias26; Rep A,B,C = origem de replicação do pRiA4 de Agrobacterium rhizogenes27. Clique aqui para ver uma versão maior deste valor.
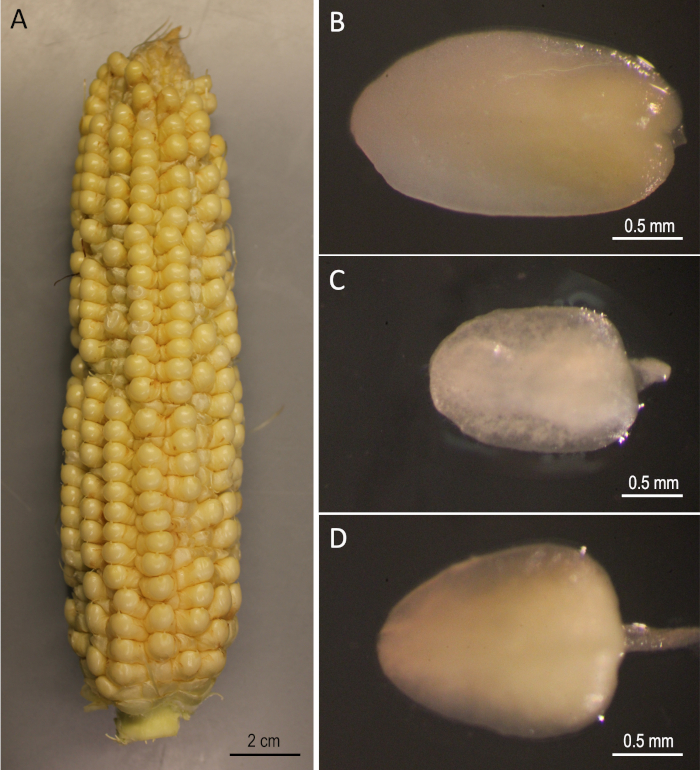
Figura 2: Materiais iniciais. Os ouvidos B73 colheram 12 dias após a polinização(A). Embriões imaturos de B73(B),Mo17(C)e W22(D). Clique aqui para ver uma versão maior deste valor.
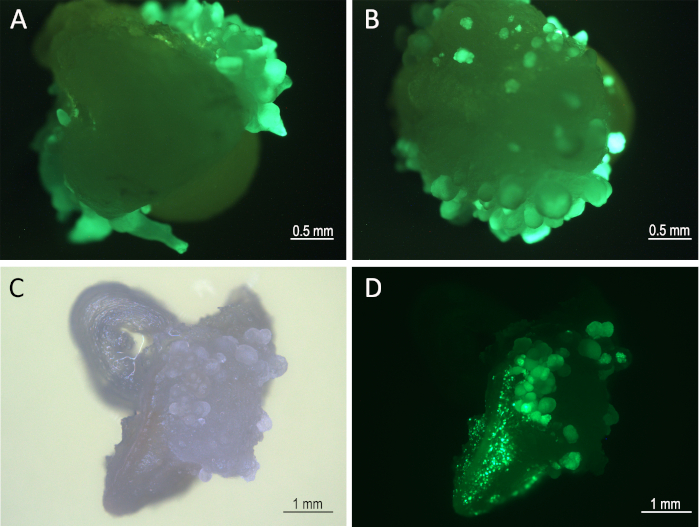
Figura 3: Desenvolvimento de tecidos em repouso médio 1 semana após a infecção. Embriões (8 dias após a infecção) um microscópio florescente (filtro GFP) mostrando GFP expressando embriões somáticos de Mo17 (A) e W22(B). Tecido em desenvolvimento (B73) o filtro de campo brilhante(C)e GFP(D). Clique aqui para ver uma versão maior deste valor.

Figura 4: Desenvolvimento tecidual em meio de maturação com seleção. Uma placa de maturação W22(A). Tecido em desenvolvimento (Mo17, 15 dias após a infecção) o filtro de campo brilhante(B)e GFP(C). Tecido em desenvolvimento (Mo17, 28 dias após a infecção) o filtro de campo brilhante(D) e GFP(E). As setas apontam para a regeneração de tecidos que não têm expressão GFP, sugerindo a excisão do gene ZsGreen entre os locais loxP após a atividade proteica CRE induzida pelo calor. Clique aqui para ver uma versão maior deste valor.

Figura 5: Desenvolvimento de tecidos na raiz da mídia. Filmagens de W22 (A),B73(B)e Mo17 (C.D). Evento com múltiplos ensaios (regeneradores gramados) de B73 (E) e W22 (F.G). Shoots com raízes de B73 (H) e W22 (I). Clique aqui para ver uma versão maior deste valor.
Tabela 1: Composições midiáticas para transformação do milho. Clique aqui para ver esta tabela (Clique para baixar).
Discussão
Os protocolos tradicionais para a transformação do milho seguem o paradigma de isolar embriões zigogoticos imaturos para produzir tecido calus transgênico, que é regenerado em plantas férteis4,6. Embora isso seja eficaz, os protocolos à base de calo podem ser demorados, e muitas vezes leva até 3 meses para o processo de cultura do tecido produzir plantas. O que torna o método aqui apresentado significativo é que ele é livre de calo, eficiente, rápido, e permite a regeneração de plantas T0 em cerca de metade do prazo. Ele também parece ser menos dependente do genótipo e, portanto, pode ser eficaz para a maioria dos incriados disponíveis publicamente8,11.
Embora todas as etapas devem ser efetivamente seguidas, a preparação correta da mídia de crescimento é imprescindível. Os componentes da mídia de crescimento precisam ser adicionados nos estágios corretos, tanto pré quanto pós-autoclave, para garantir que o material vegetal receba a concentração adequada de produtos químicos. Isso garantirá que compostos sensíveis como antibióticos não se quebrem. Também é importante que o material vegetal seja colocado no meio de crescimento correto em cada etapa, conforme indicado no protocolo. Não colocar material no meio de crescimento adequado pode resultar em morte material. Além disso, a colocação de muitos embriões ou o desenvolvimento de tecidos nas placas deve ser evitado. Embora colocar o dobro de peças de tecido pode economizar o custo de produtos químicos e pratos petri (e até mesmo espaço de incubadora), o crescimento do tecido em placas superlotadas pode ser seriamente inibido. Durante a realização da infecção, deve-se garantir que a densidade óptica da suspensão do Agrobacterium seja apropriada. Se a densidade de suspensão bacteriana for muito baixa, a infecção adequada pode não ocorrer.
A qualidade dos materiais iniciais é essencial para o sucesso nos protocolos de transformação. Os ouvidos utilizados para dissecção de embriões devem ser saudáveis, o que significa que a planta que as produz é saudável. Eles também devem possuir um conjunto adequado de sementes e ser livres de pragas e doenças. Além disso, o antigo Agrobacterium não deve ser usado. O prato "mãe" não deve ter mais do que 2 semanas de idade. Após este ponto, uma nova placa de "mãe" deve ser escada para iniciar novos experimentos.
Embora este método tenha se mostrado menos dependente do genótipo, não se pode supor que todas as linhas serão igualmente bem sucedidas. Ainda pode haver variação entre linhas, bem como diferenças de sucesso com base na construção que está sendo utilizada. A variabilidade de orelha a orelha também é inevitável quando se trabalha com embriões imaturos, por isso os experimentos idealmente devem usar várias orelhas para explicar isso. Neste trabalho, o Inbred W22 teve o melhor desempenho, com mais de ~14% de frequência de transformação, seguido por B73 e Mo17 (~4% cada). Lowe et al.8 relataram usar o protocolo QuickCorn para transformação B73 e Mo17. Neste trabalho, as frequências de transformação variaram de 9%-50% para B73 e 15%-35% para Mo17.
Uma possibilidade para as menores frequências de transformação para B73 e Mo17 observadas neste trabalho pode ser atribuída à flutuação sazonal da qualidade do ouvido. Outra diferença entre este trabalho e o de Lowe et al.8 é que diferentes construções vetoriais foram usadas aqui. No trabalho de Lowe, genes morfogênicos não foram removidos das plantas transformadas, mas sim silenciou-se nos estágios posteriores. Neste trabalho, os genes morfogênicos foram removidos 8 dias após a infecção. É possível que b73 e Mo17 possam precisar de uma presença mais longa de Bbm/Wus2 para o desenvolvimento de embriões somáticos.
Utilizando esse método, existe a possibilidade de obtenção de plantas de fuga não transgênicas, inserções multiméricas e transgenes não excidados. Essas plantas não terão um fenótipo notavelmente diferente, por isso a detecção por PCR é necessária para determinar se uma planta é transgênica. Para isso, primers PCR dentro da região excizada e primers flanqueando a região excisada podem ser empregadas. Múltiplas transformações independentes também podem produzir plantas a partir do mesmo embrião imaturo, dificultando a determinação da taxa total de recuperação transformadora independente. Nosso padrão tem sido calcular uma taxa de transformação baseada na amostragem de uma planta de cada embrião imaturo que produziu plantas e dividindo isso pelo número de embriões infectados. Este método quase certamente subestima o número real de eventos independentes recuperados como plantalets. A discriminação entre eventos independentes do mesmo embrião requer sequenciamento das regiões fronteiriças ao redor dos transgenes, e isso será proibitivamente caro e demorado para a maioria das aplicações; porém, pode haver casos em que esses dados são úteis.
Esse método de transformação da cultura tecidual provou ser muito eficaz, mas ainda podem ocorrer problemas. Se o material vegetal não está respondendo, é possível que haja um problema com a linha de raça particular, sugerindo que variáveis como composição de mídia de crescimento e tempo de subculação requerem ajustes. Outra variável é o design vetorial adequado e a construção vetorial precisa, se o vetor original for alterado. Também pode haver problemas com sensibilidade ao imazapyr, pois algumas linhas são mais sensíveis do que outras, e a concentração de imazapyr pode precisar ser ajustada para alcançar plantas transformadas com sucesso.
Nos últimos 30 anos, a cultura e os protocolos de transformação do tecido de milho mudaram e progrediram; e acredita-se que este protocolo encurtado irá promover essa progressão. Este método é eficaz para configurações acadêmicas porque é menos demorado do que os métodos tradicionais. Além disso, não exige operadores altamente treinados, tornando-o mais favorável à distribuição generalizada quando comparado aos métodos tradicionais. No futuro, esse método pode ser combinado com novas tecnologias, como a engenharia de genomas.
Divulgações
Alicia Masters, William Gordon-Kamm e Todd Jones são funcionários da Corteva Agriscience que forneceu o protocolo e as orelhas de milho de B73, Mo17 e W22 usadas neste artigo. Os autores Minjeong Kang, Morgan McCaw, Jacob Zobrist e Kan Wang não têm nada para revelar.
Agradecimentos
Agradecemos à equipe de estufa corteva por fornecer orelhas imaturas de milho, o laboratório preparatório de mídia Corteva por fornecer assistência para fazer mídia, Ning Wang da Corteva por ajuda com a construção da Agrobacterium, e Keunsub Lee da Universidade Estadual de Iowa para obter ajuda. Este projeto foi parcialmente apoiado pelo Programa de Pesquisa do Genoma Vegetal da Fundação Nacional de Ciência (Grant 1725122 e 1917138 para K.W., pelo Predictive Plant Phenomics Research Traineeship Program (National Science Foundation Grant DGE-1545453) para J.Z., pelo projeto USDA NIFA Hatch #IOW04341, pelos fundos do Estado de Iowa, e pelo Centro de Bioengenharia de Culturas da Universidade Estadual de Iowa.
Materiais
| Name | Company | Catalog Number | Comments |
| 2,4-D | Millipore Sigma | D7299 | |
| 6-Benzylaminopurine (BAP) | Millipore Sigma | B3408 | |
| Acetosyringone | Millipore Sigma | D134406 | |
| Agar | Millipore Sigma | A7921 | |
| Aluminum foil | To cover the flask | ||
| Ammonium Sulfate | Millipore Sigma | A4418 | |
| Analytical balance | To weigh small quantities of chemicals | ||
| Autocalve | Primus (Omaha, NE) | PSS5-K | To autoclave media and tools |
| Bacterial culture loop (10 µl) | Fisher scientific | 22-363-597 | Collects Agrobacterium from plate to transfer to liquid |
| Bactoagar | BD bioscience | 214030 | |
| Beakers (1 L, 2 L, 4 L) | To mix the chemicals for media | ||
| Benomyl | Millipore Sigma | #45339 | |
| Bleach (8.25% Sodium Hypochlorite) | Clorox | For seed sterilization | |
| Boric Acid | Millipore Sigma | B6768 | |
| Calcium Chloride Dihydrate | Millipore Sigma | C7902 | |
| Carbenicillin | Millipore Sigma | C3416 | |
| Casein Hydrolysate | Phytotech | C184 | |
| Cefotaxime | Phytotech | C380 | |
| Conical tube (50 mL) | Fisher scientific | 06-443-19 | Contain liquid medium and Agro suspension |
| Cuvette (Semi-micro) | Fisher scientific | 14955127 | To hold liquid for measuring OD |
| Dicamba | Phytotech | D159 | |
| Digital hygrometer | Checking temperature and humidity for heat treatment | ||
| EDTA, Disodium Salt, Dihydrate | Millipore Sigma | 324503 | |
| Eppendorf tube (2.0 mL) | ThermoFischer Scientific | AM12475 | |
| Eriksson's Vitamins | Phytotech | E330 | 1000x in liquid |
| Ethanol (70%) | Sterilizing tools and surfaces | ||
| Ferrous Sulfate Heptahydrate | Millipore Sigma | F8263 | |
| Fertilizer, Osmocote Plus 15-9-12 | ICL Specialty Fertilizers (Dublin, OH) | A903206 | Fertilizer |
| Flask (2 L) | Pyrex | 10-090E | To autoclave media and tools |
| Flats (Standard 1020, open w/holes, 11"W x 21.37"L x 2.44"D) | Hummert International (Earth City, Mo) | 11300000 | Tray to hold soil and pot insert, fits Humidome |
| Forceps (fine-tipped and large) | Fine for handling embryos; larger for large plant materials and use as ear holders | ||
| Gentamicin | Gold Biotechnologies | G-400 | |
| Glass bottle (1 L) | Pyrex | 06-414-1D | To autoclave medium |
| Graduated cylinder | To adjust volume of media | ||
| Imazapyr | Millipore Sigma | 37877 | |
| Incubator, 20 °C | Percival Scientific | Model I-36NL | To grow mother plate and incubate embryos during Agro infection |
| Incubator, 27 °C | Percival Scientific | Model I-36NL | To grow co-cultivation plate and maize embryo culture |
| Incubator, 45 °C | Heat shock treatment | ||
| Insert TO Standard, pots | Hummert International (Earth City, Mo) | 11030000 | For transplanting plants from rooting to soil, fits flat and Humidome |
| Laminar flow hood | Maintains sterile conditions | ||
| L-proline | Phytotech | P698 | |
| Magnesium Sulfate Heptahydrate | Millipore Sigma | M1880 | |
| Maize inbred seed B73 | U.S National Plant Germplasm | id=47638 | |
| Maize inbred seed Mo17 | U.S National Plant Germplasm | id=15785 | |
| Maize inbred seed W22 | U.S National Plant Germplasm | id=61755 | |
| Manganese Sulfate Monohydrate | Millipore Sigma | M7899 | |
| Milli-Q Water purification systems | Millipore sigma | MILLIQ | For tissue culture grade water |
| MS Basal Medium | Millipore Sigma | M5519 | |
| MS Basal Salt Mixture | Millipore Sigma | M5524 | |
| N6 Basal Salt Mixture | Millipore Sigma | C1416 | |
| Paperclips, non-skid | Holding on tassel bags | ||
| Peptone | BD bioscience | 211677 | |
| Petri dish (100x15 mm) | Fisher scientific | FB0875713 | For bacteria culture medium |
| Petri dish (100x25 mm) | Fisher scientific | FB0875711 | For the plant tissue culture medium |
| pH meter | Fisher scientific | AB150 | To adjust pH of media |
| Pipette (1 mL) | ThermoFischer Scientific | 4641100N | |
| Plastic Boxes | The Container Store | 10048430 | For tissue culture storage and incubation |
| Plastic humidy dome (Humi-Dome) | Hummert International (Earth City, Mo) | 14385100 | Plastic cover for soil flat |
| Potassium Iodide | Millipore Sigma | 793582 | |
| Potassium Nitrate | Millipore Sigma | P8291 | |
| Potassium Phosphate Monobasic | Millipore Sigma | P5655 | |
| Scale | To weigh chemicals for media | ||
| Scalpel Blade (No. 11, 4 cm) | Thermo Scientific | 3120030 | remove the top of the kernel crowns for embryo dissection |
| Scalpel handle | Holding scalpel blades | ||
| Schenk & Hildebrandt Vitamin (S&H vitamin) | Phytotech | S826 | 100x powder |
| Scissors | Cutting ear shoots | ||
| Shoot bag (Canvasback- semi-transparent) | Seedburo (Des Plaines, IL) | S26 | Semi-transparent bag to cover ear shoots |
| Silver Nitrate | Millipore Sigma | S7276 | |
| Sodium Molybdate Dihydrate | Millipore Sigma | M1651 | |
| Soiless substrate LC1 | SunGro Horticulture (Agawam, Ma) | #521 | For growing maize plants |
| Spatula (Double Ended Micro-Tapered) | Fischer Scientific | 2140110 | Dissecting embryos from kernels |
| Spatula (with spoon) | Fisher scientific | 14-375-10 | To measure chemicals for media |
| Spectinomycin | Millipore Sigma | S4014 | |
| Spectrophotometer (Genesys 10S UV-Vis) | Thermo Scientific | 840-300000 | Measure OD of Agro suspension |
| Stirring bar | Fisher scientific | 14-513-67 | To mix media |
| Stirring hotplates | To mix media | ||
| Syringe (without needle, 60 mL) | Fisher scientific | 14-823-43 | For filter sterilization |
| Syringe filter (0.22 µm) | Fisher scientific | 09-720-004 | For filter sterilization |
| Tassel bag (Canvasback- brown) | Seedburo (Des Plaines, IL) | T514 | Bag to cover tassels of non-transgenic plants |
| Tassel bag (Canvasback-green stripe) | Seedburo (Des Plaines, IL) | T514G | Bag to cover tassels of transgenic plants |
| Thiamine HCl | Phytotech | T390 | |
| Thidiazuron | Phytotech | T888 | |
| Thymidine | Millipore Sigma | T1895 | |
| Timentin | Phytotech | T869 | |
| Tween 20 | Fisher Scientific | Cas #9005-64-5 | surfactant |
| Vortex Genie 2 | Scientific Industries | SI0236 | Homogenizes liquids (Agro suspension) |
| Water bath (large - Precision model 186) | Fisher scientific | any that can fit 4+ 2L flasks and reach 55 °C | Keeps autoclaved media at optimal temperature |
| Weigh dish | Fisher scientific | 08-732-112 | To measure chemicals for media |
| Weighing paper | Fisher scientific | 09-898-12A | To measure chemicals for media |
| Yeast Extract | Fisher Scientific | BP14222 | |
| Zeatin | Millipore Sigma | Z0164 |
Referências
- Zhao, Z. Y., et al. High throughput genetic transformation mediated by Agrobacterium tumefaciens in maize. Molecular Breeding. 8 (4), 323-333 (2002).
- Green, C. E., Phillips, R. L. Plant regeneration from tissue cultures of maize. Crop Science. 15 (3), 417-421 (1975).
- Ji, Q., Xu, X., Wang, K. Genetic transformation of major cereal crops. International Journal of Developmental Biology. 57, 495-508 (2013).
- Frame, B., Warnberg, K., Main, M., Wang, K., Wang, K. Maize (Zea mays, L). Agrobacterium Protocols. , 101-117 (2015).
- Frame, B., et al. Improved Agrobacterium-mediated transformation of three maize inbred lines using MS salts. Plant Cell Reports. 25 (1), 1024-1034 (2006).
- Que, Q., et al. Maize transformation technology development for commercial event generation. Frontiers in Plant Science. 5 (379), (2014).
- Lowe, K. S., et al. Morphogenic regulators Baby boom and Wuschel improve monocot transformation. The Plant Cell. 28 (9), 1998-2015 (2016).
- Lowe, K. S., et al. Rapid genotypes independent maize transformation via direct somatic embryogenesis. In Vitro Cellular & Developmental Biology - Plant. 54 (3), 240-252 (2018).
- Boutilier, K., et al. Ectopic expression of BABY BOOM triggers a conversion from vegetative to embryonic growth. The Plant Cell. 14 (8), 1737-1749 (2002).
- Zuo, J., Niu, Q. W., Frugis, G., Chua, N. H. The WUSCHEL gene promotes vegetative-to-embryonic transition in Arabidopsis. The Plant Journal. 30 (3), 349-359 (2002).
- Jones, T. J., Kumar, S., Barone, P., Smith, M., et al. Maize transformation using the morphogenic genes Baby Boom and Wuschel2. Transgenic Plants. , 81-93 (2019).
- Anand, A., et al. An improved ternary vector system for Agrobacterium-mediated rapid maize transformation. Plant Molecular Biology. 97 (1-2), 187-200 (2018).
- Ray, K., et al. Mutant acetolactate synthase gene is an efficient in vitro selectable marker for the genetic transformation of Brassica juncea (Oilseed Mustard). Journal of Plant Physiology. 161 (9), 1079-1083 (2004).
- Green, J. M., Hale, T., Pagano, M. A., Andreassi, J. L., Gutteridge, S. A. Response of 98140 corn with gat4621 and hra transgenes to glyphosate and ALS-inhibiting herbicides. Weed Science. 57 (2), 142-148 (2009).
- An, G., et al. Functional analysis of the 3' control region of the potato wound-inducible proteinase inhibitor II gene. The Plant Cell. 1 (1), 115-122 (1989).
- Matz, M. V., et al. Fluorescent proteins from nonbioluminescent Anthozoa species. Nature Biotechnology. 17 (10), 969-973 (1999).
- Passarinho, P., et al. Target Genes Provide Diverse Entry Points into Cell Proliferation and Cell Growth Pathways. Plant Molecular Biology. 68 (3), 225-237 (2008).
- Bhyri, P., Khrishnamurthy, N., Narayanan, E., Nott, A., Sarangi, R. R. Novel plant terminator sequences. Patent Number US2014/0130205. , (2014).
- Laux, T., Mayer, K. F., Berger, J., Jürgens, G. The WUSCHEL gene is required for shoot and floral meristem integrity in Arabidopsis. Development. 122 (1), 87-96 (1996).
- Garnaat, C., Lowe, K., Roth, B. Zm-AXIG1-specific polynucleotides and methods of use. Patent Number WO2002006499. , (2002).
- Hershey, H. P., Stoner, T. D. Isolation and characterization of cDNA clones for RNA species induced by substituted benzenesulfonamides in corn. Plant Molecular Biology. 17 (4), 679-690 (1991).
- Abremski, K., Hoess, R. Bacteriophage P1 site-specific recombination. Purification and properties of the Cre recombinase protein. Journal of Biological Chemistry. 259 (3), 1509-1514 (1984).
- Sun, A. Q., et al. Cloning and Function Analysis of Small Heat Shock Protein Gene ZmHSP17.7 from Maize. ACTA Agronomica Sinica. 41 (3), 414 (2015).
- Sauer, B., Henderson, N. Site-specific DNA recombination in mammalian cells by the Cre recombinase of bacteriophage P1. Proceedings of the National Academy of Sciences of the United States of America. 85 (14), 5166-5170 (1988).
- Hershfield, V., Boyer, H. W., Yanofsky, C., Lovett, M. A., Helinski, D. R. Plasmid ColEl as a molecular vehicle for cloning and amplification of DNA. Proceedings of the National Academy of Sciences of the United States of America. 71 (9), 3455-3459 (1974).
- Liebert, C. A., Hall, R. M., Summers, A. O. Transposon Tn21, flagship of the floating genome. Microbiology and Molecular Biology Reviews. 63 (3), 507-522 (1999).
- Nishiguchi, R., Takanami, M., Oka, A. Characterization and sequence determination of the replicator region in the hairy-root-inducing plasmid pRiA 4b. Molecular and General Genetics. 206 (1), 1-8 (1987).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados