Method Article
Vollautomatische beinverfolgung in frei beweglichen Insekten mit Feature Learning Leg Segmentation and Tracking (FLLIT)
In diesem Artikel
Zusammenfassung
Wir beschreiben detaillierte Protokolle für die Verwendung von FLLIT, einer vollautomatischen maschinellen Lernmethode zur Beinkrallenbewegungsverfolgung in frei beweglichen Drosophila melanogaster und anderen Insekten. Diese Protokolle können verwendet werden, um subtile Gangbewegungen in wilden Fliegen, mutierten Fliegen und Fliegenmodellen der Neurodegeneration quantitativ zu messen.
Zusammenfassung
Das Drosophila-Modell war von unschätzbarem Wert für die Erforschung der neurologischen Funktion und für das Verständnis der molekularen und zellulären Mechanismen, die der Neurodegeneration zugrunde liegen. Während Fliegentechniken für die Manipulation und Untersuchung neuronaler Teilmengen immer ausgefeilter geworden sind, wurde der Reichtum der resultierenden Verhaltensphänotypen nicht an einem ähnlichen Detail erfasst. Um subtile Fliegenbeinbewegungen für den Vergleich unter Mutanten untersuchen zu können, ist die Fähigkeit erforderlich, hochgeschwindigkeits- und schnelle Beinbewegungen automatisch zu messen und zu quantifizieren. Daher haben wir einen maschinellen Lernalgorithmus für automatisiertes Beinkrallen-Tracking in frei gehenden Fliegen, Feature Learning-basierte Limb-Segmentierung und Tracking (FLLIT) entwickelt. Im Gegensatz zu den meisten Deep-Learning-Methoden ist FLLIT vollautomatisiert und generiert eigene Trainingssets ohne Benutzeranmerkungen, indem morphologische Parameter verwendet werden, die in den Lernalgorithmus integriert sind. Dieser Artikel beschreibt ein detailliertes Protokoll zur Durchführung von Ganganalysen mit FLLIT. Es beschreibt die Verfahren für Kamera-Setup, Arena-Konstruktion, Video-Aufnahme, Beinsegmentierung und Beinkrallen-Tracking. Es gibt auch einen Überblick über die von FLLIT produzierten Daten, die rohe Tracked Body- und Beinpositionen in jedem Videoframe, 20 Gangparameter, 5 Plots und ein nachverfolgtes Video enthalten. Um die Verwendung von FLLIT zu demonstrieren, quantifizieren wir relevante krankheitskranke Gangparameter in einem Fliegenmodell der Spinozerebellar-Ataxie 3.
Einleitung
In den letzten Jahrzehnten sind neurodegenerative Erkrankungen und Bewegungsstörungen in unseren alternden Bevölkerungsgruppen häufiger geworden. Obwohl unser Verständnis vieler neurodegenerativer Erkrankungen auf molekularer und zellulärer Ebene fortgeschritten ist, bleiben grundlegende Merkmale der betroffenen neuronalen Schaltkreiserkrankungen schlecht verstanden. Kürzlich entwickelte Verhaltens-Tracking-Tools1,2,3,4 ermöglichen es uns jetzt, Bewegungsanomalien in Tierseuchenmodellen zu untersuchen, um molekulare, zelluläre und Kreislaufdysregulation zugrunde liegende Erkrankungen zu identifizieren.
Molekulare Wege, die an vielen neurodegenerativen Erkrankungen beteiligt sind, werden in der Fruchtfliege Drosophila melanogasterkonserviert, und Drosophila-Krankheitsmodelle haben dazu beigetragen, grundlegende Mechanismen zu klären, die der Neurodegeneration zugrunde liegen5,6. Wir haben vor kurzem gezeigt, dass Fliegenmodelle der Parkinson-Krankheit (PD) und Der Spinozerebellar-Ataxie 3 (SCA3) deutliche, konservierte Gangsignaturen aufweisen, die denen der jeweiligen menschlichen Krankheiten ähneln1, was zeigt, dass das Fliegenmodell verwendet werden kann, um Schaltkreismechanismen zu verstehen, die Bewegungsstörungen bei bestimmten Bewegungsstörungen zugrunde liegen. Das reiche und ständig wachsende Arsenal an Werkzeugen im Fliegenmodell zur gezielten Manipulation und Visualisierung von Neuronen auf der Einzelgen- und Einzelzellebene7,8,9,10 macht die Fliege zu einem idealen Modell, um die Beziehung zwischen Krankheitswegen, neuronalen Schaltkreisen und verhaltensbedingten phänotychen Manifestationen in vivo zu untersuchen. Um eine präzise, automatisierte Insektenganganalyse zu ermöglichen, haben wir vor kurzem eine maschinelle Lernmethode entwickelt, Feature Lertragbasierte LImb Segmentierung und TRacking (FLLIT)1.
FLLIT besteht aus einem vollautomatischen mehrstufigen Algorithmus, der zunächst die Beinpixel segmentiert, die anschließend verwendet werden, um die entsprechenden Beinkrallen zu lokalisieren und zu verfolgen. FLLIT verwendet einen Steigerungsalgorithmus für die Segmentierung, im Gegensatz zu Deep Learning-Algorithmen, die in der letzten Arbeit verwendet wurden2,3. Es gibt einige Ähnlichkeiten mit konvolutionalen neuronalen Netzwerken, da für beide Frameworks die Merkmalsextraktion automatisch durch das Erlernen von faltigen Kernen erfolgt. Der erste Schritt in FLLIT besteht darin, morphologische Operationen (Kante und Skelettierung) zu verwenden, um automatisch positive (Pixel an den Beinen) und negative (Hintergrund oder Pixel auf dem Fliegenkörper) Trainingsproben mit hoher Sicherheit zu generieren. Daher ist FLLIT vollautomatisiert und erfordert keine vom Benutzer mit Anmerkungen benoteten Trainingsbeispiele. Anhand der oben genannten Trainingsbeispiele wird dann ein Klassifier im Rahmen eines Boost-Algorithmus trainiert. Ein Ensemble schwacher Klassifikatoren wird iterativ erlernt, wobei jeder aus einem Satz von Faltkernen für die Feature-Extraktion und einem Entscheidungsbaum besteht. Der letzte gelernte Klassifizierer wird dann für die Beinsegmentierung verwendet und ist in der Lage, schwierige Regionen/harte Proben besser zu erkennen als morphologische Operationen, wodurch eine insgesamt viel genauere Segmentierung für die Verfolgungvon 1erzeugt wird. Von den segmentierten Beinen aus suchen wir die Spitzen und verfolgen sie mit dem ungarischen Algorithmus: indem wir Spitzen über Rahmen hinweg so abgleichen, dass die Summe der von jeder Spitze bewegten Entfernung minimiert wird. FLLIT kann Okklusionsfälle verarbeiten, indem es sich an die zuletzt gesehene Position (in fliegenzentrierten Koordinaten) erinnert, so dass eine Beinspitze wiederhergestellt wird, sobald sie nicht mehr unter Okklusion ist.
Wir haben zuvor gezeigt, dass FLLIT die Beinbewegungen automatisch und genau verfolgen und Gang gang es in einer unmarkierten, frei beweglichen Fliege oder Spinne aus Highspeed-Video1analysieren kann; FLLIT sollte daher allgemein für die Arthropodenbeinverfolgung gelten. Durch das Extrahieren von Maschinell-Lerntrainingssätzen unter Verwendung morphologischer Parameter trainiert flLIT sich automatisch, um Insektenbeine zu segmentieren und zu verfolgen, ohne dass eine mühsame manuelle Anmerkung erforderlich ist, die für die meisten Deep-Learning-Methoden erforderlich ist. FLLIT ist somit voll automatisiert. Nach der Leg-Segmentierung und -Verfolgung erzeugt FLLIT automatisch rohe Tracked Body- und Beinpositionen in jedem Videorahmen, 20 Gangparameter, 5 Plots und ein trackiertes Video zur Ganganalyse und Visualisierung von Gangbewegungen. Dieses Protokoll bietet eine Schritt-für-Schritt-Anleitung zur Verwendung von FLLIT.
Protokoll
1. Systemeinrichtung
- Stellen Sie sicher, dass die Aufnahmestation über eine Hochgeschwindigkeitskamera und eine Bühne verfügt, um die Arenakammer zu halten (Abbildung 1). Stellen Sie die Kamera so ein, dass sie mindestens 250 Bilder pro Sekunde (fps) aufzeichnet, mit einer entsprechend schnellen Verschlusszeit (in diesem Fall wird die Aufnahme mit 1000 fps mit einer Verschlusszeit von 1 ms durchgeführt).
HINWEIS: Überprüfen Sie, ob das Video für die Verfolgung geeignet ist, indem Sie sicherstellen, dass es minimale oder keine Bewegungsunschärfe von beweglichen Beinen in allen Frames gibt. Wenn das bewegliche Bein so verschwommen ist, dass ein menschlicher Annotator es nicht verfolgen kann, muss die Kameraaufnahmegeschwindigkeit und/oder Verschlusszeit erhöht werden. - Platzieren Sie die Infrarot-LED-Leuchten oben auf der Bühne mit einem Diffusor (transluzentes Blatt) zwischen Kamera und Probe (Abbildung 1A,B).
- Machen Sie die Aufnahmekammer, indem Sie eine 1,6 mm dicke Acrylfolie schneiden. Verwenden Sie in diesem Experiment ein Sichtfeld von 11 mm x 11 mm. Platzieren Sie die Kammer zwischen zwei Glasgleitern (Abbildung 1C).
2. Vorbereitung von Fliegen für die Aufnahme
- Übertragen Sie die zu erfassenden Fliegen 24 h vor der Aufnahme in eine neue Lebensmitteldurchstechflasche.
HINWEIS: Verwenden Sie CO2 (in der Regel nicht verwendet, um die Fliegen bei der ersten Sammlung zu anästhesieren) auf den Fliegen weniger als 24 h vor der Aufnahme. - Ca. 40 min vor der Aufnahme, übertragen Sie die Fliegen in leere Fläschchen und halten Sie auf Eis für 5-7 min.
- In der Zwischenzeit wischen Sie die Arena und klare Glasrutschen mit Wasser und einem Wisch.
HINWEIS: Verwenden Sie kein Ethanol, um die Kammern und die Rutschen zu reinigen. - Bereiten Sie die Aufnahmekammer vor. Befestigen Sie einen der mikroskopischen Glasgleiter unter der Kammer mit Klebeband.
- Wenn die Fliegen auf Eis beäpft wurden, übertragen Sie eine Fliege in jeder Kammer mit einer sauberen Bürste.
HINWEIS: Sowohl männliche als auch weibliche Fliegen können in diesem Setup verwendet werden, und, soweit möglich, Sollten Fliegen beider Geschlechter analysiert werden, um geschlechtsspezifische Voreingenommenheit zu vermeiden. - Sichern Sie die Kammer mit einem weiteren mikroskopischen Glasschlitten mit Klebeband (Abbildung 1C).
- Halten Sie die kammerförmigen Fliegen bei Raumtemperatur für 15 - 20 min für die Akklimatisierung.
3. Generierung von Videos für die FLLIT-Analyse
HINWEIS: Dieser Schritt ist spezifisch für die verwendete Videokamera. In diesem Fall wird eine handelsübliche Videokamera verwendet (siehe Materialtabelle).
- Schalten Sie die Stromquelle ein. Warten Sie auf die grüne LED für Strom und die orange LED für die Ethernet-Schnittstellenverbindung zu stabilisieren. Schalten Sie die Infrarot-LED ein. Stellen Sie sicher, dass die Spannung bei 12,5 V bleibt.
- Öffnen Sie die Viewer-Anwendung auf dem angeschlossenen Computersystem.
- Ändern Sie die Aufnahmebildrate auf 1000 fps. Stellen Sie die Verschlusszeit auf 1/1000 s (1 ms) ein.
- Platzieren Sie die Kammer mit der Fliege auf der Aufnahmearena und wählen Sie die LIVE-Taste. Stellen Sie sicher, dass die Kamera auf die Beinspitzen fokussiert ist, wenn die Fliege aufrecht auf dem Boden der Kammer läuft; die Beinspitzen sollten scharf im Fokus stehen.
- Klicken Sie auf Datensatz (Abbildung 2).
- Zeichnen Sie die Fliege zu Fuß, um sicherzustellen, dass:
Die Fliege ging in einer relativ geraden Flugbahn, ohne den Rand der Arena zu berühren.
Die Fliege ging mindestens drei Schritte pro Bein.
Die Fliege hält während des Spaziergangs nicht an.
Die zurückgelegte Strecke entspricht mindestens einer Körperlänge.
HINWEIS: Wenn der Hintergrund sauber subtrahiert wird, ist dies für eine genaue Segmentierung von entscheidender Bedeutung. Der von FLLIT verwendete automatisierte Hintergrundsubtraktionsalgorithmus erfordert, dass die abgebildete Fliege mindestens eine Körperlänge in der Entfernung bewegt. - Klicken Sie auf Rec Done, um die Aufzeichnung zu beenden (Abbildung 2).
- Schneiden Sie das Video zu, um sicherzustellen, dass die Aufnahme nur einen geraden Spaziergang der Fliege umfasst (wie in Schritt 3.6 beschrieben).
- Klicken Sie auf Speichern (Abbildung 2). Speichern Sie die Dateien im Format '.mraw' oder '.tiff' in den entsprechenden Ordnern.
HINWEIS: Das '.mraw'-Format bietet mehr Flexibilität beim Ändern des Dateinamens (falls erforderlich) und beim Speichern von Videos im Vergleich zum Dateiformat ".tiff".
4. Installation des FLLIT-Programms
HINWEIS: Aktuelle Anweisungen finden Sie unter: https://github.com/BII-wushuang/FLLIT/blob/master/Compiled/Readme.pdf
- Download von FLLIT auf jedem Betriebssystem
- Laden Sie FLLIT vom folgenden Github-Link herunter: https://github.com/BII-wushuang/FLLIT/archive/master.zip. Extrahieren Sie den Inhalt der ZIP-Datei.
- Laden Sie Beispiel-Datasets aus dem folgenden Google Drive-Link herunter: https://bit.ly/2EibvNY. Erstellen Sie einen Ordner Daten unter FLLIT-master/Compiled, und platzieren Sie Datasetordner in diesem Datenverzeichnis.
- Installieren von FLLIT in Ubuntu
- Navigieren Sie zum Verzeichnis FLLIT/Compiled.
- Klicken Sie mit der rechten Maustaste und wählen Sie Öffnen in Terminal aus.
- Geben Sie den folgenden Befehl aus, um die MATLAB-Laufzeitbibliotheken auf $HOME/MCR herunterzuladen und zu installieren:
bash MCR_2016a.sh - Nachdem die Installation der MATLAB-Laufzeitbibliotheken abgeschlossen wurde, geben Sie den folgenden Befehl aus, um sicherzustellen, dass FLLIT ausführbare Rechte gewährt werden:
chmod +x FLLIT - Öffnen Sie ein Terminal im Verzeichnis FLLIT/Compiled, und geben Sie den folgenden Befehl aus, um FLLIT auszuführen:
bash run_FLLIT.sh $HOME/MCR/v901
- Installieren von FLLIT in Windows
- Installieren Sie Docker Toolbox für die 7. und 10. Home Edition unter:
(https://github.com/docker/toolbox/releases/download/v19.03.1/DockerToolbox-19.03.1.exe). - Installieren Sie Docker Desktop für Windows für Windows unter Windows 10 Pro oder Enterprise Edition unter: (https://download.docker.com/win/stable/Docker.%20Desktop-%20Installer.exe).
- Um die Ausführung von GUI-Anwendungen in einem Docker-Container unter Windows zu aktivieren, installieren Sie zuerst VcXSrV (https://sourceforge.net/projects/vcxsrv). Konfigurieren Sie beim Starten von VcXsrv die Einstellungen wie in Abbildung S1.
HINWEIS: Stellen Sie sicher, dass Docker und VcXsrv ausgeführt werden, bevor Sie FLLIT starten. - Doppelklicken Sie auf FLLIT.bat, um FLLIT auszuführen.
HINWEIS: Wenn Sie zum ersten Mal ausführen, wird es einige Zeit dauern, das Docker-Image aus Docker Hub zu ziehen.
- Installieren Sie Docker Toolbox für die 7. und 10. Home Edition unter:
- Installieren von FLLIT in MacOS
- Docker Desktop für MacOS unter https://download.docker.com/mac/stable/Docker.dmg herunterladen
- Installieren Sie socat, indem Sie ein Terminal öffnen und den folgenden Befehl ausgeben:
Brew installieren socat - Starten Sie socat mit:
socat TCP-LISTEN:6000,reuseaddr,fork UNIX-CLIENT:"$DISPLAY" & disown - Installieren Sie XQuartz (https://www.xquartz.org), um die Ausführung von GUI-Anwendungen in einem Docker-Container unter MacOS zu ermöglichen. Starten Sie XQuartz, und ändern Sie die Einstellungen, indem Sie die Option Verbindungen von Netzwerkclients zulassen auf der Registerkarte Sicherheit aktivieren, wie in Abbildung S2dargestellt.
HINWEIS: Stellen Sie sicher, dass Docker, Socat und XQuartz alle ausgeführt werden, bevor Sie FLLIT starten. - Öffnen Sie ein Terminal im Verzeichnis FLLIT/Compiled und führen Sie FLLIT mit folgendem Befehl aus:
bash FLLIT_Mac.sh
HINWEIS: Wenn Sie zum ersten Mal ausführen, wird es einige Zeit dauern, das Docker-Image aus Docker Hub zu ziehen.
5. FlLIT für automatisierte seichte Beinverfolgung
- Segmentierung
- Konvertieren Sie das Video in einzelne TIFF-Dateien und kopieren Sie es in den FLLIT-Datenordner.
- Führen Sie FLLIT (In Ubuntu, rechts klicken, um FLLIT in Terminal zu öffnen).
- Wählen Sie den Ordner aus, der die Frame-by-Frame-TIFF-Bilder des zu verfolgenden Videos enthält, und klicken Sie auf die Schaltfläche Hinzufügen.
- Wählen Sie im Pop-up-Fenster 0 für die Durchführung der Beinsegmentierung oder 1 für die Beinverfolgung mit Beinsegmentierung aus.
- Klicken Sie auf Fertig, um die Segmentierung und Nachverfolgung des ausgewählten Videos zu initiieren.
- Tracking
- Um die Genauigkeit der Nachverfolgung zu überprüfen und Fehlerkorrekturen (falls vorhanden) durchzuführen, klicken Sie auf Datenordner auswählen. Wählen Sie den nachzuverfolgenden Ordner aus, und klicken Sie auf Öffnen.
- Klicken Sie auf Tracking anzeigen.
HINWEIS: Überprüfen Sie, ob der Viewer-Modus beim Anzeigen der verfolgten Beinpositionen ausgewählt bleibt. Andernfalls werden alle vorherigen Korrekturen überlagert. - Überprüfen Sie die Beschriftung für alle Beine im ersten Rahmen
HINWEIS: Da die Beinetiketten entsprechend der Position auf dem Bildschirm platziert sind, wird die RECHTE Seite der Fliege als L1 (Vorbein), L2 (Mittelbein), L3 (Hinterbein) und die LINKE Seite der Fliege als R1 (Vorbein), R2 (Mittelbein), R3 (Hinterbein) bzw.(Bild 3)gekennzeichnet. Wenn die Fliege auf dem Kopf läuft, werden die Beinetiketten korrekt mit Anmerkungen versehen. - Wenn ein Bein falsch beschriftet ist und eine Korrektur erforderlich ist, klicken Sie auf Anzeige anhalten, gefolgt von Vorhersage anpassen ( Abbildung3).
- Wählen Sie auf der rechten Seite mit dem Bein zum Anpassendas Bein aus, das korrigiert werden muss.
- Doppelklicken Sie im Bildfenster auf die richtige Position für dieses Bein, klicken Sie auf Speichern, und beenden Siees . Um zum vorherigen oder nachfolgenden Frame zu wechseln, klicken Sie auf Ansicht anhalten, gefolgt von den Schaltflächen Vorwärts und Rückwärts bzw. I> (Abbildung 3).
- Um falsch nachverfolgte Videos zu korrigieren, öffnen Sie den Datenordner des Videos, das neu verfolgt werden soll, und wählen Sie Manuell tracking initiierenaus.
- Klicken Sie auf die Schaltfläche "Tracking", die dann die Bezeichnung in Initialändert.
- Klicken Sie auf Vorhersage anpassen und korrigieren Sie die Beinbeschriftungen, indem Sie auf jede Beinspitze doppelklicken und sie dann mit der richtigen Beinbeschriftung zuweisen. Klicken Sie auf Speichern und Beenden.
- Klicken Sie auf Fortsetzen, um die Nachverfolgung zu initiieren.
- Datenverarbeitung und Videogenerierung
- Klicken Sie auf Datenprozess. Geben Sie im Popup-Fenster die Anzahl der Bilder pro Sekunde (fps) ein, bei denen die Videos aufgenommen wurden (z. B. 1.000 fps).
- Verwenden Sie die folgende Gleichung, um das tatsächliche Sichtfeld des aufgenommenen Videos zu berechnen, sodass Gangparameter in Millimetern gemessen werden können:
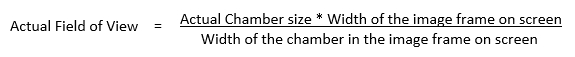
HINWEIS: Wenn z. B. die tatsächliche Kammergröße 7 mm beträgt, beträgt die Breite des Bildrahmens 137 mm, die Breite der Kammer im Bildrahmen auf dem Computerbildschirm 81 mm und die Breite des Sichtfeldes 11,83 mm(Abbildung S3). - Um die Tracking-Ergebnisse anzuzeigen, wechseln Sie zum Ordner "Tracking" unter dem Ordner Ergebnisse.
- Um ein Video der verfolgten Fliege zu generieren, wählen Sie Video erstellenaus. Das Video wird im gleichen Ergebnisordner wie die analysierten Ursprünglichen Videodaten gespeichert.
HINWEIS: Die Frames "Start" (erster) und "Ende" (letzter Punkt des Videos) können ausgewählt werden.
- Normalisierung auf Körperlänge jeder Fliege.
HINWEIS: Da jede Fliege leicht unterschiedlich groß sein kann, sollten bestimmte Gangparameter auf die Körperlänge jeder Fliege normalisiert werden, um Vergleiche zu erleichtern (z. B. kann die Schrittlänge bei größeren Fliegen länger und bei kleineren Fliegen kürzer sein).- Öffnen Sie drei Standbilder aus dem Video jeder Fliege (in der Regel erste, mittlere und letzte Frames) mit Bildsoftware.
- Vergrößern Sie jeden Bildrahmen auf 800 % und beschriften Sie das voritorigste Pixel des Kopfes und das hinterste Pixel des Bauches an der Mittellinie mit einer hellen Farbe (z. B. Gelb).
- Öffnen Sie die beschrifteten Bilder in ImageJ.
- Verwenden Sie Skalen festlegen, um den Maßstab entsprechend einzugeben: Abstand in Pixel einstellen: 512; Bekannter Abstand: Ist-Sichtfeld (mm) gemessen in Schritt 5.3.2.; Längeneinheit: mm.
- Zeichnen Sie eine gerade Linie zwischen dem beschrifteten Kopf und Bauchspitzenpixel, um die Körperlänge zu erhalten.
- Öffnen Sie Erneut Scale, um den Wert in einem bekannten Abstand zu erhalten, d. h. die Körperlänge in mm.
- Nehmen Sie einen Durchschnitt der Länge, die in jedem der drei Bilder bestimmt wird, um die durchschnittliche Körpergröße in mm zu erhalten.
Ergebnisse
Nach Der Leg-Segmentierung, Verfolgung und Datenverarbeitung generiert FLLIT automatisch Rohdaten für die Positionen des Körpers und jeder Beinkralle, 20 Gangparameter, 5 Plots und ein nachverfolgtes Video (Tabelle 1).
Hier zeigen wir diese Analysen mit einem Fliegenmodell der Spinozerebellar-Ataxie 3 (SCA3). Der panneuronale Treiber Elav-GAL4 wurde verwendet, um entweder den in voller Länge lebenden menschlichen SCA3 mit 27 Glutaminen im PolyQ-Trakt (UAS-SCA3-flQ27) oder einen mutierten menschlichen SCA3 in voller Länge mit 84 Glutaminen im PolyQ-Trakt (UAS-SCA3-flQ84)11zu fahren. SCA3 ist typisiert durch einen ataxic Gang mit Körperveering, unregelmäßige Fußplatzierung und kurzen, lauernden Schritten12,13 (Tabelle 2). Um den Gang von mutierten SCA3-Fliegen zu charakterisieren und zu untersuchen, ob sie einen ähnlichen Gang wie menschliche Patienten aufweisen, analysierten wir relevante Gangparameter, die von FLLIT generiert wurden, nämlich: Anzahl der Körperumdrehungen, Fußabdruck-Regelmäßigkeit, Beindomänenüberlappung und -größen sowie Beinschrittlängen (Tabelle 2).
Wir fanden heraus, dass SCA3-Q84 Fliegen mehr Drehungen aufwiesen (Abbildung 4A,A'),eine unregelmäßige Fußplatzierung, wie sie durch die regelmäßigitätsmäßigen Fußverhältnisse des Fußbedarfs (vergrößerte Standardabweichungen des AEP14) (Abbildung 4B), erhöhte Beindomänenüberlappung ( Abbildung4C-D), vergrößerte Beindomänen in Länge und Fläche (Abbildung 4E,F) und verringerte Schrittlänge (Abbildung 4G) zeigten.
FLLIT generiert auch ein Video, das die verfolgte Fliege und Beine in den Arena-zentrierten und körperzentrierten Ansichten, Körperbahn und Richtung sowie vertikale und seitliche Verschiebungen jedes Beins zeigt (Abbildung 5). Die verfolgten Videos ermöglichen einen nebeneinander liegenden Vergleich von Beinbewegungen in verschiedenen Fliegen. Repräsentative Videos von Elav-GAL4>SCA3-flQ27 ( Video 1 ) und Elav-GAL4>SCA3-flQ84 ( Video2) Fliegen zeigen, dass im Vergleich zu Elav-GAL4>UAS-SCA3-flQ27 Fliegen ( Abbildung5A), Elav-GAL4>UAS-SCA3-flQ84 Fliegen (Abbildung 5B) sich schneidenden Beindomänen unterschiedlicher Größe, was auf einen lauernden, ataxic Gang hinweist.Video 1
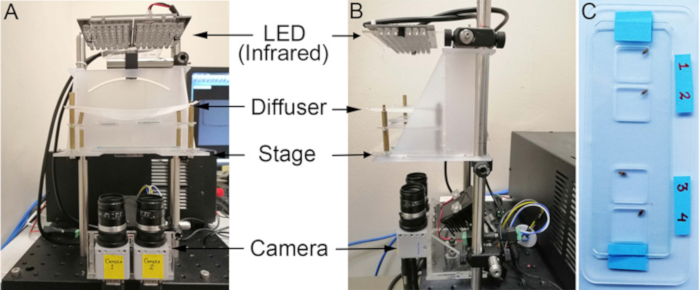
Abbildung 1. Einrichtung der Aufnahmestation und Arena. Aufnahmen aus den (A) Front- und (B) Seitenansichten. (C) Ein Beispiel für eine Arena, die für die Erstellung von Flugaufnahmen für FLLIT-Tracking verwendet wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
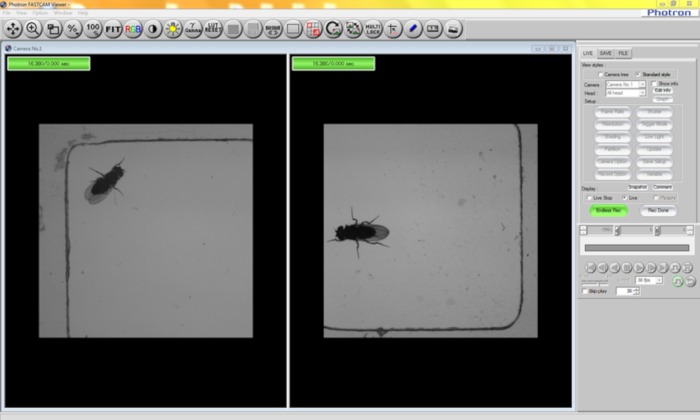
Abbildung 2: Ansicht des aktiven Fensters während der Fluggangaufnahme mit einer Dual-Head-Kamera, die die gleichzeitige Aufnahme von zwei Fliegen ermöglicht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
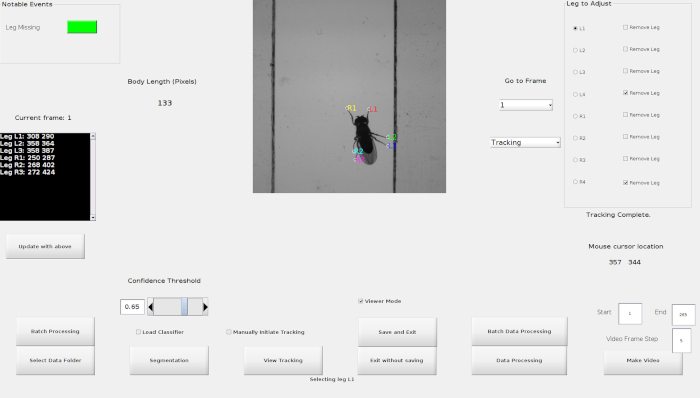
Abbildung 3: Aktives FLLIT-Fenster mit dem Schaltflächenfenster und beschrifteten Beinen nach Segmentierung und Nachverfolgung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
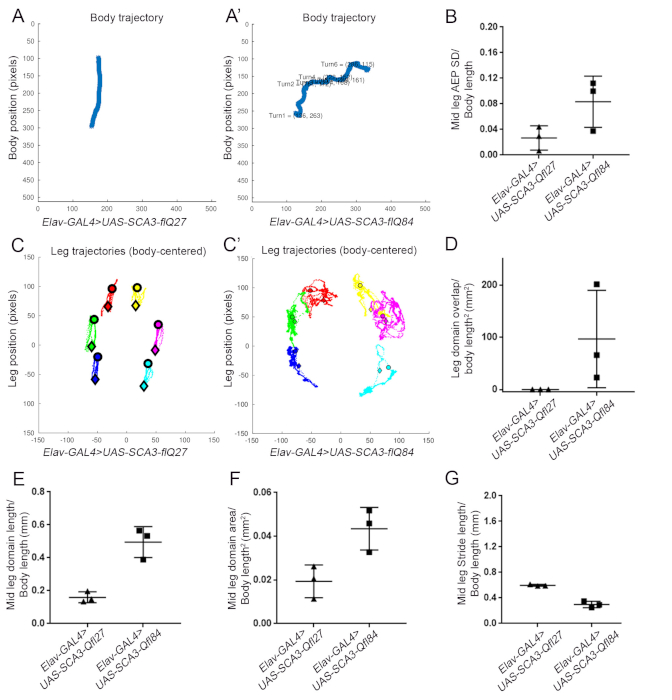
Abbildung 4: Repräsentative FLLIT-generierte Daten für relevante Gangparameter von Fliegen, die Wildtyp (SCA3-flQ27) vs. Mutant (SCA3-flQ84) SCA3 exdrücken. (A) Anzahl der Umdrehungen in der Körperbahn. (B) Mittelbein-Fußabdruck Regelmäßigkeit normalisiert auf Körperlänge. (C-C') Durchquerte Beindomänen jedes Beins. (D) Domänenüberlappung zwischen den Beinen. (E) Mittlere Domainlänge normalisiert auf Körperlänge. (F) Mid-Leg-Domänenbereich normalisiert auf Körperlänge2. (G) Mittlere Beinschrittlänge normalisiert auf Körperlänge. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
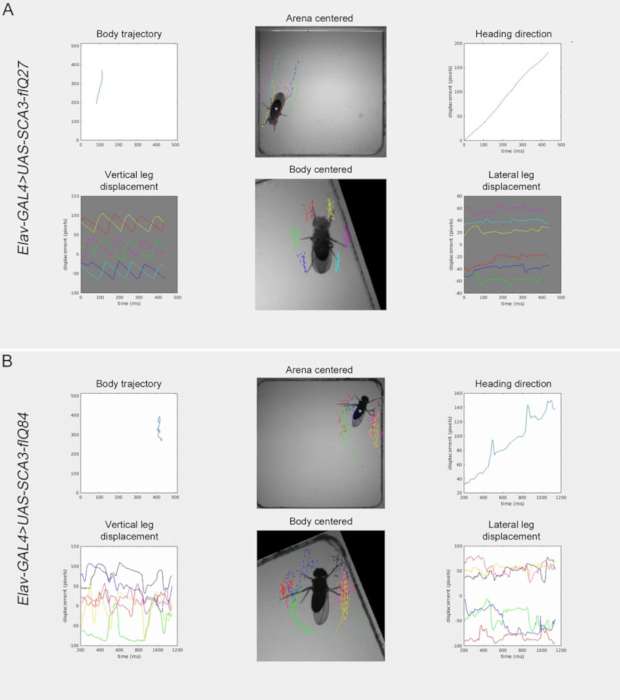
Abbildung 5: Snapshot von repräsentativen FLLIT-generierten Videos. (A) Elav-GAL4>UAS-SCA3-flQ27 und (B) Elav-GAL4>UAS-SCA3-flQ84 fliegt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Video 1: Repräsentatives Video einer Fliege, die panneuronale Wilde-Typ menschliche volle Länge SCA3 (Elav-GAL4>UAS-SCA3-flQ27) ausdrückt Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)

Video 2: Repräsentatives Video einer Fliege, die panneuronale Mutante menschliche in voller Länge SCA3 (Elav-GAL4>UAS-SCA3-flQ84) ausdrückt Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)
Ergänzende Abbildung 1: Konfigurationen für VcXSrv. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 2: Konfiguration für Xquartz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Ergänzende Abbildung 3: Bild mit den Fürdien erforderlichen Dimensionen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Kategorie | Parameter | Beschreibung | Datei/Plot (falls zutreffend) |
| Rohdaten | Körperposition | Positionskoordinaten des Körperzentroids in jedem Rahmen | Die ersten beiden Spalten von CoM.csv |
| Körperbahn | Drehwinkel der Körperachse in Grad (relativ zur y-Achse) | Dritte Spalte von CoM.csv | |
| Arena-zentrierte Beinkralle Positionen | Positionskoordinaten jeder Beinkralle in jedem Frame basierend auf Arena-Koordinaten | trajectory.csv | |
| Körperzentrierte Beinkralle Positionen | Positionskoordinaten jeder Beinkralle in jedem Rahmen basierend auf Arena-Koordinaten | norm_trajectory.csv | |
| Körperbewegung | Körperlänge (mm) | Länge des Probentiers, das in jedem Rahmen geschätzt wird (vordere Position auf dem Kopf bis zum hintersten Position auf den Flügeln) | bodylength.csv |
| Sofortige Körpergeschwindigkeit (mm/s) | Sofortige Geschwindigkeit des Körpers (Zentroid) im Probentier | BodyVelocity.csv; BodyVelocity.pdf | |
| Wendepunkte des Körpers Flugbahn | Um die Wendepunkte zu lokalisieren, wird die Flugbahn mit dem Dougl as'ASPeucker auf eine stückweise-lineare Kurve reduziert. Algorithmus, nach dem ein Wendeereignis als mit einem Winkel > 50 Grad zwischen zwei benachbarten lineare Segmente, die die vereinfachte Flugbahn bilden | BodyTrajectory.pdf | |
| Individuelle Schrittparameter | Schrittdauer (ms) | Die Dauer eines Schrittereignisses | StrideParameters.csv |
| Schrittperiode (ms) | Die Dauer von einem Schrittereignis zum nächsten | ||
| Schrittverschiebung (mm) | Verdrängung der Beinkralle während eines Schrittereignisses | ||
| Schrittweg abgedeckt (mm) | Gesamtweg, der während eines Schrittereignisses von der Beinkralle bedeckt wird | ||
| Vordere Extremlage (mm) | Landeposition (relativ zum Körper) einer Beinkralle am Ende eines Schrittereignisses | ||
| Hintere Extremlage (mm) | Startposition (relativ zum Körper) einer Beinkralle zu Beginn eines Schrittereignisses | ||
| Schrittamplitude (mm) | Verschiebung entlang der Bewegungsrichtung für ein Schrittereignis | ||
| Stance Linearität (mm) | Definiert als die Abweichung des Schrittwegs von einer Kurve, die (bei 20ms Intervallen) die entsprechende vordere und hintere extreme Positionen des Schritts | ||
| Schrittdehnung (mm) | Abstand der Beinkrallenposition vom Körperzentrum in der Mitte eines Schrittereignisses | ||
| Beinbewegung | Beingeschwindigkeit (mm/s) | Die momentane Geschwindigkeit jedes Beins | LegSpeed.csv; Gait.pdf |
| Gait-Index | Dies misst die Art der Gangkoordination, die das (sechsbeinige) Mustertier während seiner Bewegung an den Tag legte. Ein Gang Index von 1 entspricht einem Stativgang, _1 entspricht einem Tetrapodengang, während 0 einen nicht-kanonischen Gang darstellt. In unserer Implementierung wird der Gangindex durch einen gleitenden Durchschnitt über ein 120 ms-Fenster ermittelt. | GaitIndex.csv; GaitIndex.pdf | |
| Bewegungsprozentsatz | Prozentsatz der Zeit, in der ein Bein in Bewegung ist | LegParameters.csv | |
| Mittlere Schrittperiode (ms) | Durchschnittliche Dauer von einem Schrittereignis zum nächsten | LegParameters.csv | |
| Fußabgrundregularität (mm) | Gemessen als Standardabweichungen der hinteren und | LegParameters.csv | |
| vordere Extrempositionen eines Beins | |||
| Beinbahnbereich (mm2) | Der Bereich des minimalen konvexen Rumpfes, der die gesamte Beinbahn im körperzentrierten Bezugsrahmen enthält | LegParameters.csv; LegDomain.pdf | |
| Länge und Breite des Beinbahnbereich (mm) | Erreicht über den maximalen projizierten Abstand der Klauenpositionen auf die Haupt- (Domänenlänge) und (Domänenbreite) Hauptachsen | LegParameters.csv | |
| Schnittpunkt/Überlappung von Beindomänen (mm2) | Der Schnitt-/Überlappungspunkt zwischen den einzelnen möglichen | LegDomainOverlap.csv | |
| Haltungsbreite (mm) | Durchschnitt des Abstands zwischen AEP und PEP des linken und mittleren Beins | StanceWidth.csv |
Tabelle 1: Gait-Parameter, die von FLLIT generiert werden.
| Gait-Funktion | ||||
| Gait-Merkmale der Spinocerebellar-Ataxie 3 (SCA3) | Veering | Erratische Fußplatzierung und Beinüberquerung | Lurching-Schritte | Kurze Schritte |
| Messparameter | Anzahl der Body-Turn-Ereignisse | Fußabdrückerruhe | Größe der Beindomänen, Grad der Domänenüberlappung | Schrittlänge |
| FLLIT-Datei | BodyTrajectory.pdf | LegParameters.csv | LegDomainOverlap.csv | StrideParameters.csv |
Tabelle 2: Tabelle mit markanten SCA3-Gangmerkmalen bei menschlichen Patienten mit den entsprechenden FLLIT-Parametern und Ausgabedateien.
Diskussion
In diesem Manuskript beschreiben wir detailliert die Schritte bei der Verwendung von FLLIT, einem automatisierten maschinellen Lernprogramm1, um Gang in frei gehenden Drosophilazu analysieren. Nach der Verfolgung und Datenanalyse generiert FLLIT automatisch Rohdaten für die Positionsinformationen des Körpers und der Beinkrallen und erzeugt 20 Körper- und Gangfunktionen sowie ein Video der verfolgten Fliege, um die Gangvisualisierung zu ermöglichen.
Es gibt jetzt eine Reihe von Methoden für die Beinbewegungsverfolgung von Drosophila und anderen Tieren1,2,3,4,14,15,16, was Forschern eine breite Palette von Optionen je nach den Zielen des Experiments. Einige davon sind fußdruckbasierte Ansätze, die hochpräzise sind, aber nur Klauenkontaktpunkte mit der Erfassungsfläche4,14melden. Auf der anderen Seite sind die jüngsten Deep-Learning-Ansätze2,3,16 sehr vielseitig und ermöglichen die Analyse von Verhaltensweisen, die eine Verfolgung von Beingelenken und anderen Körperteilen bei jedem Tier erfordern, mit dem Vorbehalt, dass die Algorithmen zuerst mit benoteten Datensätzen des Benutzers trainiert werden müssen. Ein dritter Ansatz typisiert Morphologie oder bildkontrastbasierte Methoden1,15,17, um die Umrisse jedes Beins zu finden, um Klauenpositionen zu identifizieren. Im Allgemeinen gehen diese Methoden schlecht mit Verhaltensweisen um, bei denen sich die Beine kreuzen (z.B. während der Pflege). FLLIT kombiniert den zweiten und dritten Ansatz und verwendet morphologische Parameter, um einen Steigerungsalgorithmus für die Beinsegmentierung zu trainieren. Auf diese Weise kann FLLIT die mühsame Aufgabe der Benutzeranmerkung umgehen, um das Trainings-Dataset zu generieren, während gleichzeitig die Genauigkeit mithilfe von machine learning verbessert wird. Zukünftige Verbesserungen von FLLIT müssen sich mit Fällen befassen, in denen sich die Beine kreuzen, um eine Analyse komplexerer Verhaltensweisen zu ermöglichen.
FLLIT ist robust gegen leichte Änderungen der Beleuchtung, Aufnahmeauflösung und Rahmengeschwindigkeit1. Die Frame-Geschwindigkeit der aufgenommenen Videos sollte jedoch nicht unter 250 fps fallen, und FLLIT läuft optimal für Videos, die mit 1000 fps aufgenommen wurden. Wenn es Bewegungsunschärfe in den Bildern gibt, so dass es für einen menschlichen Annotator eine Herausforderung ist, die Beinposition zu identifizieren, wird FLLIT nicht in der Lage sein, Beinspitzen in diesen Rahmen genau zu identifizieren. Vor diesem Hintergrund ist es wichtig, dass die Kamera scharf auf die Beinspitzen fokussiert wird. Um Segmentierungsartefakte zu verhindern, sollte die Arena gründlich gereinigt und während der Aufnahme nicht verschoben werden. Für eine genaue Hintergrundsubtraktion und saubere Segmentierung sollte die Fliege während der Aufnahme mindestens eine Körperlänge bewegen, ohne zu pausieren. Nach der automatischen Segmentierung und Verfolgung muss die Beschriftung aller Beine überprüft werden. Wenn der Fliegengang nicht falsch verfolgt oder falsch verfolgt wird, sollte die Datei manuell mit der Option Manuelle Sordien (Schritt 5.2.7 – 5.2.10) manuell nachverfolgt werden.
Neurodegenerative Erkrankungen und Bewegungsstörungen sind in unseren alternden Gesellschaften immer häufiger. Seit mehr als 2 Jahrzehnten werden Fliegenmodelle der Neurodegeneration untersucht, in denen Fortschritte in Bezug auf die molekularen und zellulären Aspekte der Krankheitspathophysiologie gemacht wurden. Spezifische Verhaltensfolgen von Krankheiten waren jedoch technisch schwer einzuschätzen. Während beispielsweise Berichte über Zitterbewegungen in der Fliege gemacht wurden18,19, waren diese bis vor kurzem nicht quantitativ untersucht worden1. Der Klettertest war ein nützliches und quantitatives, aber relativ grobes Maß6. Dieses technische Defizit hat auch die hochauflösende Bewegungsanalyse in anderen Tiermodellen behindert. Das Aufkommen neuer Werkzeuge für die Verhaltensanalyse hat daher versprochen, das Feld der Bewegungsstörungen zu verjüngen, um Forschern zu ermöglichen, zu untersuchen, wie molekulare und zelluläre Mechanismen neuromuskulärer Erkrankungen zu spezifischen Verhaltensergebnissen in Tiermodellen führen. In diesem Beitrag und in unserer vorherigen Arbeit1zeigten wir mit FLLIT, dass Fliegenmodelle von SCA3 einen hyperkinetischen ataxic Gang aufweisen, während PD-Fliegenmodelle einen hypokinetischen starren Gang aufweisen, der Bewegungsmerkmale der jeweiligen menschlichen Krankheiten rekapituliert1. Die Gait-Analyse ermöglichte es uns auch, verschiedene neuronale Populationen zu identifizieren, die spezifischen Bewegungsstörungen zugrunde liegen. In Zukunft wird eine detaillierte Bewegungsanalyse, kombiniert mit den leistungsstarken bildgebenden und funktionalen Werkzeugen, die in der Fliege zur Verfügung stehen, es uns ermöglichen, neue Einblicke in Mechanismen der bewegungsbedingten Dysfunktion zu gewinnen, die unser Verständnis neurodegenerativer Erkrankungen in Bezug auf Schaltkreismechanismen beleuchten.
FLLIT sollte allgemein anwendbar sein, um Gang bei anderen kleinen Arthropoden zu studieren, da es sich zuvor als hochgenau für die Verfolgung von Spinnenbeinbewegungen1erwiesen hat. Während wir uns hier auf die Verwendung detaillierter Bewegungs-Phänotypisierung zur Quantifizierung pathogener Gangundungen und ihrer zugrunde liegenden Schaltkreise konzentrieren, haben die Fortschritte in der Bewegungsverfolgung das Verständnis der normalen Gehkoordination und Desgang und der zugrunde liegenden Schaltkreise, insbesondere in unzähligen verschiedenen Zweigen des Evolutionsbaums, bereits revolutioniert und werden sich weiterhin auf das Verständnis der normalen Gehkoordination und Gangart und ihrer zugrunde liegenden Schaltkreise auswirken.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Autoren danken Moumita Chatterjee und Alice Liu für die technische Unterstützung und dem Bloomington Drosophila Stock Centre (Indiana, USA) für die Bereitstellung der Drosophila-Stämme, die in diesem Werk verwendet werden. Diese Arbeit wurde vom Institute of Molecular and Cell Biology, Singapur, unterstützt; das Bioinformatics Institute, Singapur; die Organisation des Gemeinsamen Rates der Agentur für Wissenschaftstechnologie und Forschung (Zuschussnummer 15302FG149 an SA und LC); das Clinical Research Flagship Programme (Parkinson es Disease), das vom National Medical Research Council des Gesundheitsministeriums von Singapur (Grant-Nummer NMRC/TCR/013-NNI/2014 an SA), die University of Alberta (Startup-Stipendium an LC) und das Discovery Grant des Natural Sciences and Engineering Research Council of Canada (NSERC) (Grant-Nummer RGPIN-2019-04575 an LC) verwaltet wird.
Materialien
| Name | Company | Catalog Number | Comments |
| Acrylic Sheets | Dama | 1.6 mm thickness, Clear sheets | Singapore, Singapore |
| Clear Glass slides | Biomedia | BMH 880101 | Singapore, Singapore |
| High speed camera | Photron | Fastcam MC2.1 | Tokyo, Japan. A shutter speed of 1 msec or faster is ideal to reduce motion blur of captured images |
| Infra Red LED | Any - Generic from hardware store | 940nm Infrared Light Board | Singapore, Singapore |
| Kimwipe | Kimberly Clark | 34155-01LS | Irving, Texas, USA |
Referenzen
- Wu, S., et al. Fully automated leg tracking of Drosophila neurodegeneration models reveals distinct conserved movement signatures. PLoS Biology. 17 (6), 3000346 (2019).
- Mathis, A., et al. DeepLabCut: markerless pose estimation of user-defined body parts with deep learning. Nature Neuroscience. 19, 1281-1289 (2018).
- Pereira, T. D., et al. Fast animal pose estimation using deep neural networks. Nature Methods. 16 (1), 117-125 (2019).
- Machado, A. S., Darmohray, D. M., Fayad, J., Marques, H. G., Carey, M. R. A quantitative framework for whole-body coordination reveals specific deficits in freely walking ataxic mice. eLife. 4, (2015).
- Lu, B., Vogel, H. Drosophila models of neurodegenerative diseases. Annual Reviews in Pathology. 4, 315-342 (2009).
- McGurk, L., Berson, A., Bonini, N. M. Drosophila as an In Vivo Model for Human Neurodegenerative Disease. Genetics. 201 (2), 377-402 (2015).
- Dionne, H., Hibbard, K., Cavallaro, A., Kao, J. -. C., Rubin, G. M. Genetic reagents for making split-GAL4 lines in Drosophila. bioRxiv. , (2017).
- Cande, J., et al. Optogenetic dissection of descending behavioral control in Drosophila. eLife. 7, (2018).
- Nern, A., Pfeiffer, B. D., Rubin, G. M. Optimized tools for multicolor stochastic labeling reveal diverse stereotyped cell arrangements in the fly visual system. Proceedings of the National Academy of Sciences of the United States of America. 112 (22), 2967-2976 (2015).
- Xie, T., et al. A Genetic Toolkit for Dissecting Dopamine Circuit Function in Drosophila. Cell Reports. 23 (2), 652-665 (2018).
- Warrick, J. M., et al. Ataxin-3 suppresses polyglutamine neurodegeneration in Drosophila by a ubiquitin-associated mechanism. Molecular Cell. 18 (1), 37-48 (2005).
- Ebersbach, G., et al. Comparative analysis of gait in Parkinson's disease, cerebellar ataxia and subcortical arteriosclerotic encephalopathy. Brain. 122 (7), 1349-1355 (1999).
- Stolze, H., et al. Typical features of cerebellar ataxic gait. Journal of Neurology, Neurosurgery and Psychiatry. 73 (3), 310-312 (2002).
- Mendes, C. S., Bartos, I., Akay, T., Marka, S., Mann, R. S. Quantification of gait parameters in freely walking wild type and sensory deprived Drosophila melanogaster. eLife. 2, 00231 (2013).
- DeAngelis, B. D., Zavatone-Veth, J. A., Clark, D. A. The manifold structure of limb coordination in walking Drosophila. eLife. 8, (2019).
- Gunel, S., et al. DeepFly3D, a deep learning-based approach for 3D limb and appendage tracking in tethered, adult Drosophila. eLife. 8, (2019).
- Isakov, A., et al. Recovery of locomotion after injury in Drosophila melanogaster depends on proprioception. The Journal of Experimental Biology. 219, 1760-1771 (2016).
- Aw, S. S., Lim, I. K. H., Tang, M. X. M., Cohen, S. M. A Glio-Protective Role of mir-263a by Tuning Sensitivity to Glutamate. Cell Reports. 19 (9), 1783-1793 (2017).
- Eberl, D. F., Duyk, G. M., Perrimon, N. A genetic screen for mutations that disrupt an auditory response in Drosophila melanogaster. Proceedings of the National Academy of Sciences of the United States of America. 94 (26), 14837-14842 (1997).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten