Method Article
Querschnitt von reifem Reis (Oryza sativa L.) Kerne für die Rasterelektronenmikroskopie-Bildgebung mit Pipettenspitzen als Immobilisierungsunterstützung
In diesem Artikel
Zusammenfassung
Dieses Protokoll ermöglicht die Herstellung von Querschnitten von Getreidesamen (z. B. Reis) zur Analyse der Endosperm- und Stärkegranulatmorphologie mittels Rasterelektronenmikroskopie.
Zusammenfassung
Stärkegranulate (SGs) weisen je nach Pflanzenart unterschiedliche Morphologien auf, insbesondere im Endosperm der Familie der Poaceae. Die Endosperm-Phänotypisierung kann verwendet werden, um Genotypen basierend auf dem SG-Morphotyp unter Verwendung der rasterelektronenmikroskopischen (REM) Analyse zu klassifizieren. SGs können mit SEM visualisiert werden, indem man durch den Kern (Perikarp, Aleuronschichten und Endosperm) schneidet und den organellaren Inhalt freilegt. Aktuelle Methoden erfordern, dass der Reiskorn in Kunststoffharz eingebettet und mit einem Mikrotom oder in eine abgeschnittene Pipettenspitze eingebettet und von Hand mit einer Rasierklinge geschnitten wird. Die erste Methode erfordert spezielle Ausrüstung und ist zeitaufwendig, während die letztere je nach Reisgenotyp eine neue Vielzahl von Problemen mit sich bringt. Insbesondere Kalkreissorten stellen aufgrund der brüchigen Natur ihres Endospermgewebes ein Problem für diese Art der Schnitte dar. Hier wird eine Technik zur Vorbereitung von durchscheinenden und kalkhaltigen Reiskornschnitten für die Mikroskopie vorgestellt, die nur Pipettenspitzen und eine Skalpellklinge erfordern. Die Vorbereitung der Abschnitte innerhalb der Grenzen einer Pipettenspitze verhindert, dass das Reiskernendosperm zerbricht (für transluzente oder "glasartige" Phänotypen) und zerbröckelt (für kalkhaltige Phänotypen). Mit dieser Technik können Endospermzellmuster und die Struktur intakter SGs beobachtet werden.
Einleitung
Stärkegranulate (SGs) weisen je nach Pflanzenart unterschiedliche Morphologien auf, insbesondere im Endosperm der Poaceae-Familie 1,2. Die Endosperm-Phänotypisierung kann verwendet werden, um Genotypen basierend auf dem SG-Phänotyp unter Verwendung der rasterelektronenmikroskopischen Analyse zu klassifizieren. SGs können mittels Rasterelektronenmikroskopie (REM) visualisiert werden, indem der Kern geschnitten und die Endosperm-Zellwände abgehoben werden2.
Der Zweck dieser Technik besteht darin, quer verlaufende Reiskornabschnitte ausschließlich für die schnelle REM-Analyse einfach vorzubereiten. Die Entwicklung dieser Technik wurde durch die Notwendigkeit eines schnellen Querschnittsansatzes motiviert, bei dem Proben unmittelbar vor der Visualisierung mit minimaler Ausrüstung für die REM-Mikroskopie vorbereitet werden.
Diese Technik beinhaltet das Einführen des geschälten Reiskorns in die Pipettenspitze zur vollständigen Ruhigstellung. Dies ist besonders wichtig beim Querschnitt von kalkhaltigen Reiskorn-Phänotypen, die brüchig sind und unter Druck leicht zerbröckeln3. Kreide ist eine unerwünschte Eigenschaft bei Reis, da sie das Aussehen des Kerns beeinflusst und dazu führt, dass der Kern beim Polieren und Mahlen leichtbricht 3. Kreidegrad präsentiert sich als undurchsichtiger Bereich in einem Querschnitt des Kerns, der mit bloßem Auge beobachtet werden kann; auf mikroskopischer Ebene ist die Kreidezeit durch kleine, lose gepackte Stärkegranulate gekennzeichnet. Ursachen für Kreide können genetisch4,5 oder umweltbedingt6,7sein.
Getreidesaatgutquerschnitte wurden traditionell unter Verwendung chemischer Fixierungsmethoden und Schnitte nach dem Einbetten der Probe in Paraffinwachs oder eine andere feste Matrix4,8,9,10hergestellt . Im Jahr 2010 wurde die Matsushima-Methode eingeführt, um eine komplizierte und zeitaufwändige Vorbereitung von Reiskornproben zu vermeiden4. Diese Methode beinhaltete das Einführen des geschälten Reiskorns in eine abgeschnittene Pipettenspitze. Die Spitze wird von einem Blocktrimmer stationär gehalten, und dünne, partielle Endospermabschnitte werden mit einer Handrasierklinge geerntet. Eine weitere schnelle Technik, die 2016 entwickelt wurde, ermöglichte eine dünne Ganzschnitte einer Vielzahl von trockenen Samen, einschließlich kalkhaltiger Sorten10. Diese Methoden motivierten die Entwicklung der hier vorgestellten Schnelltechnik.
Diese neue Technik eignet sich für Forscher, die intakte Querquerschnitte von Reiskörnern für die Endosperm-Phänotypisierung und die Analyse der Stärkemorphologie mit REM erhalten möchten.
Dieses Protokoll stellt eine Anpassung der Matsushima-Pipettenspitzen-Methode4dar, mit mehreren bemerkenswerten Modifikationen: (1) Kerne werden an keiner Stelle der Technik aufgenommen; (2) Für die Vorbereitung der Abschnitte sind weder ein Blocktrimmer noch ein Ultramikrotom erforderlich. In dieser Studie wurden eine Wildtyp-'transluzente' Sorte(Oryza sativa L. ssp. japonica cv. Nipponbare) und eine mutagene 'kalkhaltige' Linie von Nipponbare (ssg1, minderwertiges Stärkekorn1)4 untersucht. Diese beiden Sorten wurden für die Analyse hier ausgewählt, um die technischen und visuellen Unterschiede bei der Verarbeitung von transluzenten und kalkhaltigen Reisabschnitten zu demonstrieren.
Protokoll
1. Vorbereitung des Querreisabschnitts
- Enthäuten Sie trockene, intakte Kerne, wie in Abbildung 1A dargestellt.
- Lösen Sie die Schalen und entfetten Sie die Reiskörner, indem Sie die Kerne zwischen zwei flachen Gummistopfen mahlen. Entfernen Sie die geschälten Reiskörner bei Bedarf aus der Rispe.
- Legen Sie einen einzelnen Kern auf einen flachen Gummistopfen auf einer Werkbank (Abbildung 1B). Stellen Sie sicher, dass dieser Stopper stehen bleibt.
- Verwenden Sie einen zweiten flachen Gummistopfen (Abbildung 1C), um den Kern abzuschleifen, indem Sie ihn mit ausreichendem Druck gegen den ersten Gummistopfen verdrehen (Abbildung 1D). Entfernen Sie die Schalen aus dem Kern und achten Sie darauf, das Endosperm nicht zu zerbrechen. Entfernen Sie die restliche Schale mit einer feinen Pinzette. Ein enthäuteter Kern ist in Abbildung 1E dargestellt.
- Führen Sie mit einer feinen Pinzette einen einzelnen geschälten Kern in eine Kunststoffpipettenspitze (250 μL Größe, ein Samen/Spitze) ein (Abbildung 1F). Stellen Sie sicher, dass das Embryonenende des Kerns zum (konischen) Ende der Pipettenspitze zeigt (Abbildung 1G).
HINWEIS: Wenn Sie den Kern auf diese Weise einfügen, wird sichergestellt, dass der Kern so eng wie möglich in die Pipettenspitze passt, da der Kern zu seinem proximalen Ende hin schmaler ist. - Setzen Sie eine zweite 250-μL-Pipettenspitze ein, um den Kern in die Pipettenspitze zu drücken und den Kern während des Schneidens unbeweglich zu halten, wobei darauf zu achten ist, dass der Kern nicht beschädigt oder die zweite Pipettenspitze gebogen wird (Abbildung 1H). Die richtige "Teleskop"-Assemblage ist in Abbildung 1Idargestellt.
- Legen Sie die Pipettenspitzenmontage flach auf eine Werkbank und halten Sie sie von Hand fest (Abbildung 1J). Verwenden Sie andererseits eine scharfe Skalpellklinge (Nr. 20), um durch die Mitte des Kerns zu schneiden und das Ende der Pipettenspitze abzuschneiden (Abbildung 1K). Schneiden Sie mit dem Skalpell 1 mm dicke Abschnitte des Reiskorns ab (Abbildung 1L).
HINWEIS: Der Kernabschnitt ist fest in einem Kunststoffring eingeschlossen (Abbildung 1M). Die mittlere Querschnittsdicke für drei exemplarische Genotypen ist in Tabelle 1 dargestellt. Abschnitte, die deutlich dünner als 1 mm sind, zerbrechen oder zerbröckeln. Es ist wichtig zu beachten, aus welchem Teil des Kerns jeder Abschnitt stammt, wenn dieses Experiment an mehreren Reissorten zum Vergleich durchgeführt wird, da die Stärkemorphologie im gesamten Endosperm11variiert.
2. Auflichtmikroskopie von Querreisschnitten
- Verwenden Sie eine feine Pinzette, um die Querreisabschnitte (in Abschnitt 1 vorbereitet) auf ein schwarzes Stück schweres schwarzes Papier zu legen.
- Erhalten Sie Lichtbilder von Querschnitten von Nipponbare mit einem Stereomikroskop mit montierten Schwanenhälsen für schräge Beleuchtung, wie in Abbildung 1N-Sgezeigt.
- Beobachten Sie die Endospermmorphologie unter mindestens 10-facher Vergrößerung.
HINWEIS: Jede Epilichtquelle ist der Hellfeldmikroskopie vorzuziehen, da die mit dieser Technik erhaltenen Schnitte nicht dünn genug sind, um das Licht durchzulassen.
3. Rasterelektronenmikroskopie von quer verlaufenden Reisschnitten
- Legen Sie die Proben auf eine Kohlenstoffscheibe, die an einem Aluminiumstutzen klebt, und legen Sie sie auf einen Probenhalter zur Ladungsreduzierung. Entfernen Sie den Kunststoffring mit einer feinen Pinzette von der Pipettenspitzenhalterung, um zu verhindern, dass der Kunststoff in die Vakuumvorrichtung des REM gelangt.
HINWEIS: Bilder von Endospermzellen, SGs und Subgranulaten werden mit einer Desktop-REM-Maschine aufgenommen, bei der keine Proben sputterbeschichtet werden müssen. - Erhalten Sie die Bilder mit einem hochempfindlichen Multimode-Rückstreuelektronendetektor (BSE) bei 10 kV.
Ergebnisse
Wildtyp Nipponbare (Abbildung 2A) und ssg1 Abschnitte (Abbildung 2B) wurden unter drei Vergrößerungen untersucht: 260x, 920x und 4200x. Diese Technik ermöglicht die Herstellung von Abschnitten von ausreichender Qualität, um die gesamte Endospermzelle (Abbildung 3A), zusammengesetzte Stärkegranulate (Abbildung 3B) und einzelne Untermoleküle (Abbildung 3C) zu beobachten. Geschälte Kerne brauchen länger in der Verarbeitung als polierte Kerne, da die trockenen Rümpfe vor dem Schneiden durch Abrieb entfernt werden müssen. Kreidekörner brauchen auch länger in der Verarbeitung als polierte transluzente Kerne, da darauf geachtet werden muss, dass der Kern während des Schneidens nicht zerbricht. Ein ordnungsgemäß zubereiteter Reisabschnitt sollte etwa 0,9 mm dick sein (Tabelle 1) mit minimalem bis keinem Zerbrechen des Endosperms (Abbildung 1N) und intakten Perikarp- und Aleuronschichten (Abbildung 1O). Unsachgemäße Platzierung des Skalpells auf der Pipettenspitze beim Schneiden kann zu "abgebrochenen" Abschnitten führen (Abbildung 1P). In ähnlicher Weise zeigten Hellfeldbilder optimaler Transversalschnitte von ssg1 (Abbildung 1Q) intakte Endosperm-, Perikarp- und Aleuronschichten intakt und zur Visualisierung verfügbar (Abbildung 1R). Ein gebrochener kalkhaltiger Kernabschnitt (Abbildung 1S) kann immer noch zur Visualisierung verwendet werden, wenn der einzige Zweck darin besteht, SGs zu beobachten, aber das Endospermenzellmuster ist nicht sichtbar. Ein unterbrochener Abschnitt kann für die Analyse schwierig zu handhaben sein. Beim Wildtyp Nipponbare wurde eine stärkere Scherung der Endospermzellwände beobachtet, da die Zellen dichter gepackt und weniger brüchig sind als die ssg1-Kerne. In den ssg1-Abschnitten wurde keine Scherung von Endospermzellen beobachtet, und die zusammengesetzten Stärkegranulate sind intakt.
Abbildung S1 zeigt die Zuverlässigkeit der Ergebnisse mit der "Teleskop"-Technik zum Schneiden von Reiskörnern. Reislinien, die als transluzente Kernproduzenten identifiziert wurden – Wildtyp-resistente Stärke (RS) Hybridlinie Xieyou 7954 (Oryza sativa L. ssp. indica)12,13,14 ( AbbildungS1A) und kobaltgenerierte Mutante RS11113,15 ( AbbildungS1B) erzeugten Abschnitte, durch die Licht mit einem Stereomikroskop sichtbar war. Die entsprechenden REM-Bilder zeigten, dass diese Linien den "normalen" Reis-Endosperm-Phänotyp produzieren: dicht gepackte, polyedrische Stärkegranulate. Kalkkernproduzenten, kommerzielle Sorten Yi-Tang16 (Abbildung S1C)und RS413,eine Mutante von RS11115 (Abbildung S1D),wiesen weiße, undurchsichtige Kernabschnitte auf. Die entsprechenden REM-Bilder zeigten eine deutlich andere Morphologie im Vergleich zur wildtypischen transluzenten RS-Hintergrundlinie: Stärkegranulate waren rund und lose gepackt. Wildtyp Xiushui 11 (Oryza sativa L. ssp. japonica) (Abbildung S1E) und seine Mutante KMD1 (Kemingdao1), die das Cry1Ab-Gen zur Hemmung der Insektenprädation exprimieren17,18,19 ( AbbildungS1F) zeigten Abschnitte und Endospermmorphotypen ähnlich den durchscheinenden RS-Linien.
Die hier vorgestellte Technik ist optimal für die Vorbereitung von Proben von kalkhaltigen Reiskörnern für die phänotypische Analyse, bietet aber auch Vorteile für das Schneiden von transluzenten Reiskernphänotypen20:Das Schneiden der Proben unter Druck von oben reduziert das Risiko des Zerbrechens des Endosperms und der Dislokation. Proben können einfach innerhalb von Sekunden vorbereitet werden (Tabelle 2). Mehrere Genotypen wurden mit dieser Technik analysiert, um ihre Wirksamkeit zu testen (Tabelle 3). Wie in Abbildung S2 gezeigt,kann diese Technik auf Samen anderer Arten angewendet werden. Das Modell-Monokotyledonen Brachypodium distachyon produziert sehr harte Samen, die nur B-Granulatstärke21enthalten, denen Puroindolin A fehlt, ein Protein, das Stärkegranulat22Weichheit verleiht. Es war noch möglich, einen intakten Querschnitt zu erhalten (Abbildung S2A). Die Gewinnung eines intakten Querschnitts aus weichem weißem Winterweizen (SWWW) war eine Herausforderung, kann aber durchgeführt werden (Abbildung S2B). SWWW-Samen sind reich an Puroindolin A und groß im Vergleich zu B. distachyon Samen und Reiskörnern. Diese Samen zerbröckeln häufig beim Schneiden mit der Teleskop-Assemblage.
| Genotyp | Mittlere Schnittbreite (μm) mit Teleskop-Assemblage | Mittlere Querschnittsbreite (μm) freihändig |
| Nipponbare (geschält) | 971,7 ± 152,4ab | 1059.571 ± 394,2ab |
| Xieyou 7954 | 825,1 ± 128,3b | 1306.187 ± 179,1a |
| RS4 | 910,6 ± 165,0ab | 1126.694 ± 395.3ab |
| Mittelwerte, gefolgt von den gleichen Buchstaben, unterscheiden sich bei P < 0,01 nicht signifikant unter Verwendung einer Einweganalyse der Varianz (ANOVA) und des Tukey-Tests (n = 10). Statistische Analysen wurden mit der Software JMP 15 durchgeführt. | ||
Tabelle 1: Mittlere Kernabschnittsdicke.
| Genotyp | Mittlere Zeit(en)* |
| Nipponbare (geschält) | 14,7 ± 1,36a |
| Xieyou 7954 | 9,81 ± 0,98b |
| RS4 | 11,9 ± 1,28c |
| * Verwendung der Teleskop-Assemblage. | |
| Mittelwerte, gefolgt von den gleichen Buchstaben, unterscheiden sich bei P < 0,01 nicht signifikant unter Verwendung einer Einweganalyse der Varianz (ANOVA) und des Tukey-Tests (n = 10). Statistische Analysen wurden mit der Software JMP 15 durchgeführt. | |
Tabelle 2: Mittlere Probenvorbereitungszeit.
| Genotyp | Hintergrund | Qualität |
| Nipponbare | Wildtyp | Durchscheinend |
| Minderwertiges Stärkekorn1 (ssg1) | Nipponbare | Kreidig |
| Widerstandsfähige Stärke (RS) Xieyou 7954 | Wildtyp | Durchscheinend |
| RS111 | Xieyou 7954 | Durchscheinend |
| RS4 | RS111 | Kreidig |
| Yi-Tang, 'New Life', Marke Lujuren | Xieyou 7954 | Kreidig |
| Xiushui 11 | Wildtyp | Durchscheinend |
| Kemingdao1 (KMD1) | Xiushui 11 | Durchscheinend |
Tabelle 3: In dieser Studie untersuchte Reisgenotypen.

Abbildung 1: Vorbereitung von Querreisabschnitten. (A) Wildtyp Nipponbare Kern mit intakter Schale. (B). Der Kern wird auf einen flachen Gummistopfen mit einem Durchmesser von vier Zoll gelegt. (C) Die Schalen wurden entfernt, indem der Kern zwischen zwei ansetzenden Gummistopfen geschliffen wurde. (D) Die Schale wurde vom Reiskorn getrennt. E) Nahaufnahme von geschältem Reiskorn. Das Embryonenende ist angezeigt. (F) Einsetzen des Kerns in die Pipettenspitze mit einer feinen Pinzette. (G) Der Kern wurde in das distale Ende der Pipettenspitze eingeklemmt. (H) Einsetzen der zweiten Pipettenspitze, um den Kern für das Schneiden zu immobilisieren (die "Teleskop"-Assemblage). (I) Der Reiskorn wurde eng in das distale Ende der Pipettenspitze eingepasst. ( J) Schneiden des Reiskorns innerhalb der Assemblage. (K) Nahaufnahme des Schnittschnitts. (L) Ein Abschnitt des Kerns, der vom Kunststoffring umschlossen ist. (M) Nahaufnahme des Querteils. (N) Querabschnitt des Wildtyps Nipponbare. (O) Nahaufnahme des Endosperms innerhalb des Wildtyp-Nipponbare-Abschnitts. (P) Armer, suboptimaler Abschnitt des Wildtyps Nipponbare Kernel. (Q) Querschnitt der Nipponbare-Mutante ssg14. (R) Nahaufnahme des Endosperms innerhalb des ssg1-Abschnitts. (S) Schlechter, suboptimaler Abschnitt von ssg1. Stange (Paneele A, N-S) = 1 mm. Ganze Reiskörner und Schnitte wurden mit einem Stereomikroskop mit einer digitalen Zoomkamera und Schwanenhalslichtern abgebildet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
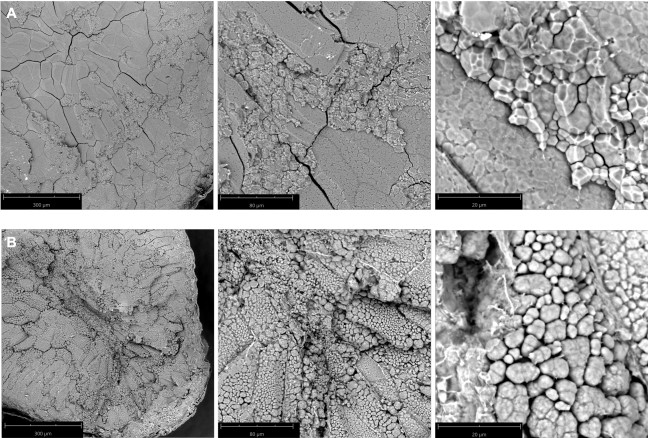
Abbildung 2: REM-Bilder von transversalen Kernschnitten. (A) Wildtyp Nipponbare, eine durchscheinende Sorte. Das zusammengesetzte Stärkegranulat wurde fest zueinander zementiert; (B) Nipponbare-Mutante ssg14, ein kalkhaltiger Phänotyp. Das zusammengesetzte Stärkegranulat war lose verpackt und hat nicht die zementartige Natur des Wildtyps Nipponbare Stärkemorphotyp. Vergrößerung von links nach rechts: 260x, 920x und 4200x. Die Länge des Balkens wird in Paneelen angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
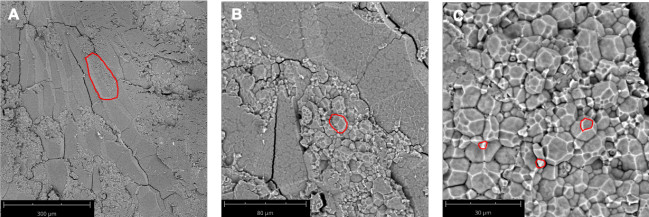
Abbildung 3: SEM-mikroskopische Anatomie eines transversalen Kernabschnitts von Xiushui 11. (A) Eine einzelne Endospermzelle ist rot umrandet. 260-fache Vergrößerung. (B) Ein zusammengesetztes Stärkegranulat ist rot umrandet. 920-fache Vergrößerung. (C) Mehrere Stärke-Subgranulate sind rot umrandet. 2250-fache Vergrößerung. Die Balkenlängen sind in den Paneelen angegeben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung S1: Querschnitte anderer Reisgenotypen, die mit dieser Technik für REM hergestellt wurden. (A) Beständige Stärke (RS) Xieyou 795412. (B) RS111, eine hoch-RS-transparente Mutante von 795413. (C) RS4, eine kalkhaltige Mutante von RS11115. (D)Yi-Tang, eine handelsübliche Sorte von Reis mit hohem Amylosegehalt16. (E) Xiushui 11. (F) KMD1 (Kemingdao1)17,18,19. 10-fache Vergrößerung für Hellefeldbilder. Weißer Balken = 1 mm. 2250-fache Vergrößerung für REM-Bilder. Die Balkenlängen sind in den Paneelen angegeben. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Abbildung S2: Die Technik ist für andere Samen nützlich. (A) Querschnitt aus falschem Purpurbrom (Brachypodium distachyon L. Accession Bd21) Samen. (B) Querschnitt aus weichem weißem Winterweizen (Triticum aestivum L. cv. Augusta) Samen. Helles Feld, 20-fache Vergrößerung. Balken = 1 mm. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Diskussion
Die hier vorgestellte Technik stellt einen schnellen, einfachen und scharfen Ansatz zur Vorbereitung von transversalen Reisquerschnitten für die Desktop-REM-Visualisierung dar. Diese Schnitttechnik ermöglicht die schnelle Beobachtung der Endospermstruktur, der Form, Größe und des Musters der Endospermzellen, des zusammengesetzten Granulats und der Stärkemorphologie. Für die Zwecke der Endosperm-Phänotypisierung und des Keimplasma-Screenings ist es entscheidend, einen ganzen Querschnitt des Reiskorns zu erhalten4,23,24. Es ist von größter Bedeutung, den Kern vollständig in die Pipettenspitze einzuführen, um zu verhindern, dass der Druck der Skalpellklinge das Endosperm zum Zerbröckeln oder Zerbrechen zwingt. Vorausgesetzt, die "Teleskop"-Assemblage ist ordnungsgemäß konstruiert, können Proben innerhalb von 15 Sekunden für die Visualisierung vorbereitet werden (Tabelle 2) unter Verwendung von Materialien, die bereits in einer typischen Laborumgebung zur Hand sind. Diese Technik ist auf den Querschnitt eines ellipsoidischen Samens mit einem Durchmesser von etwa vier Millimetern an seiner breitesten Stelle anwendbar. Samen des Modellgrases Brachypodium distachyon (Abbildung S2A) können ähnlich geschnitten werden, bleiben aber nicht im Ring eingeschlossen. Größere Samen, wie Weizen, brechen leicht und erfordern Pflege beim Schneiden (Abbildung S2B).
Es gibt jedoch mehrere Einschränkungen für die hier vorgestellte Technik. Schnitte, die mit dieser Technik erhalten werden, sind nicht dünn genug, damit das Licht hindurchgehen kann, was die Verwendung dieser Technik für durchgelassene mikroskopische Ansätze wie Hellfeld (500 μm maximale Probendicke für Reiskernabschnitte25)und Transmissionselektronenmikroskopie (TEM) (500 nm maximale Probendicke26) verbietet ). Die Verwendung einer Pipettenspitze als Schnittmatrix begrenzt auch die Größe des Saatguts, das mit dieser Technik geschnitten werden kann. Eine weitere Fehlerbehebung wäre erforderlich, um diese Technik für Arten anzupassen, die sich stark von Reis unterscheiden, und die Größe der "Matrix" ist durch die Größe der zum Kauf verfügbaren Pipettenspitzen begrenzt.
Ein weiterer entscheidender Vorteil, den diese Technik bietet, ist die Qualität der Proben, die aus kalkhaltigen Phänotyp-Reiskörnern hergestellt werden können. Es ist erwähnenswert, dass selbst die Matsushima-Studie zugab, dass es schwierig war, Querschnitte mit dieser speziellen Methode für kalkhaltige Phänotypen4zu erhalten, wie in dieser Studie zum Zwecke des Vergleichs repliziert (Abbildung 1S). In ihrem Fall wurde es notwendig, ihre kalkhaltigen Reisproben chemisch zu fixieren und sie zum Schneiden in Harz einzubetten. Die neue Technik in Verbindung mit der Desktop-REM-Bildgebung ermöglicht es dem Forscher, Querschnitte von Reiskörnern für die Mikroskopie mit mehr Konsistenz als ohne Immobilisierungsunterstützung einfach vorzubereiten (Tabelle 3).
In der neuen Ära der Phänomie und Metabolomik ist es wichtig, mutagene Linien und Transposon-markierte Bibliotheken zu überwachen, um die Funktion und Bedeutung von Stärke in Samen besser zu verstehen. Darüber hinaus hält die Internationale Reis-Genbank über 130 000 Reis-Beitritte27. Eine schnelle Samenphänotypisierungstechnik wie die hier vorgestellte würde die Klassifizierung und Probenahme für die Ernährungsqualität beschleunigen28. Schließlich kann diese Technik angesichts der sich ausbreitenden Auswirkungen des Klimawandels nützlich sein. Saisonaler Hochtemperaturstress während der Getreidefüllung wurde bereits als Hauptursache für Kalkablagerungen identifiziert6, aber neuere Studien haben steigende globale Temperaturen mit zunehmender Kreidehärte der Reiserträge in Verbindung gebracht7,29. Eine solche beschleunigte Endosperm-Phänotypisierung kann dazu beitragen, ein breites landwirtschaftliches Bild der Auswirkungen steigender globaler Temperaturen zu erhalten.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Die Autoren danken Systems for Research (SFR Corp.) für den Einsatz ihres Phenom ProX Desktop SEM-Instruments sowie für die technische Unterstützung durch Maria Pilarinos (Systems for Research (SFR) Corp.) und Chloë van Oostende-Triplet (Cell Biology and Image Acquisition Core Facility, Faculty of Medicine, University of Ottawa). Die Finanzierung erfolgte durch den Low Carbon Innovation Fund (LCIF) des Ministeriums für wirtschaftliche Entwicklung, Schaffung von Arbeitsplätzen und Handel der Regierung von Ontario sowie von Proteins Easy Corp.
Materialien
| Name | Company | Catalog Number | Comments |
| JMP 15 | SAS | N/A | N/A |
| Leit Adhesive Carbon Tabs 12 mm (Pack of 100) | Agar Scientific | AGG3347N | N/A |
| Phenom Pro Desktop SEM | Thermo Scientific | PHENOM-PRO | N/A |
| Pipette Tips RC UNV 250 µL | Rainin | 17001116 | N/A |
| SEM Pin Stub Ø12.7 Diameter Top, Standard Pin, Aluminium | Micro to Nano | 10-002012-50 | N/A |
| Shandon Microdissecting Fine Tips Thumb Forceps, Fine Tips, 12.7 cm | Thermo Scientific | 3120019 | N/A |
| Shandon Scalpel Blade No. 20, Sterile, 4.5 cm | Thermo Scientific | 28618256 | N/A |
| Shandon Stainless-Steel Scalpel Blade Handle | Thermo Scientific | 5334 | N/A |
| Zeiss V20 Discovery Stereomicroscope | Zeiss | N/A | N/A |
Referenzen
- James, M. G., Denyer, K., Myers, A. M. Starch synthesis in the cereal endosperm. Current Opinion in Plant Biology. 6 (3), 215-222 (2003).
- Shapter, F. M., Henry, R. J., Lee, L. S. Endosperm and starch granule morphology in wild cereal relatives. Plant Genetic Resources. 6 (2), 85-97 (2008).
- Ashida, K., Iida, S., Yasui, T. Morphological, physical, and chemical properties of grain and flour from chalky rice mutants. Cereal Chemistry. 86 (2), 225-231 (2009).
- Matsushima, R., Maekawa, M., Fujita, N., Sakamoto, W. A rapid, direct observation method to isolate mutants with defects in starch grain morphology in rice. Plant and Cell Physiology. 51 (5), 728-741 (2010).
- Zhao, X., et al. Identification of stable QTLs causing chalk in rice grains in nine environments. Theoretical and Applied Genetics. 129 (1), 141-153 (2016).
- Nagato, K., Ebata, M. Effects of high temperature during ripening period on the development and the quality of rice kernels. Japanese Journal of Crop Science. 34 (1), 59-66 (1965).
- Zhao, X., Fitzgerald, M. Climate change: implications for the yield of edible rice. PLoS One. 8 (6), 66218 (2013).
- Zhao, Z. K., Mu, T. H., Zhang, M., Richel, A. Effects of high hydrostatic pressure and microbial transglutaminase treatment on structure and gelation properties of sweet potato protein. LWT - Food Science and Technology. 115, 108436 (2019).
- Feiz, L., et al. Puroindolines co-localize to the starch granule surface and increase seed bound polar lipid content. Journal of Cereal Science. 50 (1), 91-98 (2009).
- Zhao, L., Pan, T., Guo, D., Wei, C. A simple and rapid method for preparing the whole section of starchy seed to investigate the morphology and distribution of starch in different regions of seed. Plant Methods. 14 (1), 16 (2018).
- Zhao, L., Pan, T., Cai, C., Wang, J., Wei, C. Application of whole sections of mature cereal seeds to visualize the morphology of endosperm cell and starch and the distribution of storage protein. Journal of Cereal Science. 71, 19-27 (2016).
- Li, C., Dong, S., Li, G., Yuan, G., Dong, W. Breeding and application of the new combination of hybrid rice "Xieyou 7954". Journal of Zhejiang University (Agriculture and Life Sciences). 19 (3), 179-181 (2002).
- Shu, X., Jia, L., Ye, H., Li, C., Wu, D. Slow digestion properties of rice different in resistant starch. Journal of Agricultural and Food Chemistry. 57 (16), 7552-7559 (2009).
- Zhou, H., et al. Critical roles of soluble starch synthase SSIIIa and granule-bound starch synthase Waxy in synthesizing resistant starch in rice. Proceedings of the National Academy of Sciences. 113 (45), 12844-12849 (2016).
- Yang, C. Z., et al. Starch properties of mutant rice high in resistant starch. Journal of Agricultural and Food Chemistry. 54, 523-528 (2006).
- Zhou, Y., Zou, Y., Jiang, Y., Li, B. Detection methods for resistance starch content of Yi-Tang rice and optimization of pretreatment. Food Science and Biotechnology. 36, 416-419 (2017).
- Cheng, X., Sardana, R., Kaplan, H., Altosaar, I. Agrobacterium-transformed rice plants expressing synthetic cryIA(b) and cryIA(c) genes are highly toxic to striped stem borer and yellow stem borer. Proceedings of the National Academy of Sciences of the United States of America. 95 (6), 2767-2772 (1998).
- Liu, H., et al. Rapid detection of P-35S and T-nos in genetically modified organisms by recombinase polymerase amplification combined with a lateral flow strip. Food Control. 107, 106775 (2020).
- Shu, Q., et al. Transgenic rice plants with a synthetic cry1Ab gene from Bacillus thuringiensis were highly resistant to eight lepidopteran rice pest species. Molecular Breeding. 6 (4), 433-439 (2000).
- Lisle, A. J., Martin, M., Fitzgerald, M. A. Chalky and translucent rice grains differ in starch composition and structure and cooking properties. Cereal Chemistry. 77 (5), 627-632 (2000).
- Chen, G., et al. Dynamic development of starch granules and the regulation of starch biosynthesis in Brachypodium distachyon: comparison with common wheat and Aegilops peregrina. BMC Plant Biology. 14 (1), 198 (2014).
- Giroux, M. J., Morris, C. F. Wheat grain hardness results from highly conserved mutations in the friabilin components puroindoline a and b. Proceedings of the National Academy of Sciences of the United States of America. 95 (11), 6262-6266 (1998).
- Matsushima, R., Hisano, H. Imaging amyloplasts in the developing endosperm of barley and rice. Scientific Reports. 9, 3745 (2019).
- Matsushima, R., et al. Amyloplast-localized SUBSTANDARD STARCH GRAIN4 protein influences the size of starch grains in rice endosperm. Plant Physiology. 164 (2), 623-636 (2014).
- Monjardino, P., et al. Development of flange and reticulate wall ingrowths in maize (Zea mays L.) endosperm transfer cells. Protoplasma. 250 (2), 495-503 (2013).
- Tizro, P., Choi, C., Khanlou, N. Sample preparation for transmission electron microscopy. Methods in Molecular Biology. 1897, 417-424 (2019).
- Liu, Q. H., Zhou, X. B., Yang, L. Q., Li, T. Effects of chalkiness on cooking, eating and nutritional qualities of rice in two indica varieties. Rice Science. 16 (2), 161-164 (2009).
- Morita, S., Wada, H., Matsue, Y. Countermeasures for heat damage in rice grain quality under climate change. Plant Production Science. 19 (1), 1-11 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten