Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
3D-zellgedruckter hypoxischer Krebs-on-a-Chip zur Rekapitulation des pathologischen Verlaufs von solidem Krebs
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hypoxie ist ein Kennzeichen der Tumormikroumgebung und spielt eine entscheidende Rolle beim Fortschreiten des Krebses. Dieser Artikel beschreibt den Herstellungsprozess eines hypoxischen Krebs-auf-einem-Chip basierend auf der 3D-Zelldrucktechnologie, um eine hypoxiebedingte Pathologie von Krebs zu rekapitulieren.
Zusammenfassung
Die Mikroumgebung von Krebs hat einen signifikanten Einfluss auf das Fortschreiten der Krankheit. Insbesondere Hypoxie ist der Schlüsselfaktor für das Überleben, die Invasion und die Chemoresistenz von Krebs. Obwohl mehrere In-vitro-Modelle entwickelt wurden, um die hypoxiebedingte Krebspathologie zu untersuchen, wurde das komplexe Zusammenspiel der in vivo beobachteten Krebsmikroumgebung aufgrund des Mangels an präziser räumlicher Kontrolle noch nicht reproduziert. Stattdessen wurden 3D-Biofabrikationsansätze vorgeschlagen, um mikrophysiologische Systeme für eine bessere Nachbildung der Krebsökologie und eine genaue Bewertung der Krebsbehandlung zu schaffen. Hier schlagen wir einen 3D-Zelldruckansatz vor, um einen hypoxischen Krebs-auf-einem-Chip herzustellen. Die Hypoxie-induzierenden Komponenten im Chip wurden auf Basis einer Computersimulation der Sauerstoffverteilung bestimmt. Krebs-Stroma-Konzentrische Ringe wurden mit Biotinten gedruckt, die Glioblastomzellen und Endothelzellen enthielten, um eine Art von solidem Krebs zu rekapitulieren. Der resultierende Chip realisierte zentrale Hypoxie und verschlimmerte maligne bei Krebs mit der Bildung repräsentativer pathophysiologischer Marker. Insgesamt wird erwartet, dass der vorgeschlagene Ansatz zur Schaffung eines solid-krebsmimetischen mikrophysiologischen Systems die Lücke zwischen In-vivo- und In-vitro-Modellen für die Krebsforschung schließen wird.
Einleitung
Die Mikroumgebung von Krebs ist ein kritischer Faktor, der das Fortschreiten des Krebses antreibt. Mehrere Komponenten, einschließlich biochemischer, biophysikalischer und zellulärer Hinweise, bestimmen die pathologischen Merkmale von Krebs. Unter diesen ist Hypoxie stark mit krebserhaltendem Überleben, Proliferation und Invasion verbunden1. Durch das unbegrenzte Wachstum und die Teilung von Krebszellen werden Nährstoffe und Sauerstoff kontinuierlich erschöpft und es entsteht ein hypoxischer Gradient. Unter sauerstoffarmen Bedingungen aktivieren Zellen eine mit Hypoxie induzierbare Transkriptionsfaktor (HIF) assoziierte molekulare Kaskade. Dieser Prozess induziert einen nekrotischen Kern, löst metabolische Veränderungen aus und initiiert Blutgefäßhyperplasie und Metastasierung2,3. Anschließend verursacht Hypoxie in Krebszellen die Zerstörung benachbarter normaler Gewebe. Darüber hinaus ist Hypoxie stark mit der therapeutischen Resistenz solider Tumoren in multifaktorieller Weise verbunden. Hypoxie kann die Strahlentherapie stark behindern, da die Strahlenempfindlichkeit aufgrund der reaktiven Sauerstoffspezies1,4begrenzt ist. Darüber hinaus senkt es den pH-Wert der Mikroumgebung von Krebs, was die Arzneimittelakkumulation verringert1. Daher ist die Reproduktion pathologischer Merkmale im Zusammenhang mit Hypoxie in vitro eine vielversprechende Strategie für wissenschaftliche und präklinische Befunde.
Die Modellierung einer spezifischen Mikroumgebung von Krebs ist für das Verständnis der Krebsentwicklung und die Erforschung geeigneter Behandlungen unerlässlich. Obwohl Tiermodelle aufgrund ihrer starken physiologischen Relevanz weit verbreitet sind, gibt es Fragen im Zusammenhang mit Artenunterschieden und ethischen Problemen5. Obwohl herkömmliche 2D- und 3D-Modelle die Manipulation und Echtzeit-Bildgebung von Krebszellen für eine eingehende Analyse ermöglichen, kann ihre architektonische und zelluläre Komplexität nicht vollständig rekapituliert werden. Zum Beispiel sind Krebs-Sphäroid-Modelle weit verbreitet, da die Aggregation von Krebszellen in einem Sphäroid auf natürliche Weise Hypoxie im Kern erzeugen kann. Darüber hinaus wurde eine große Anzahl von zellulären Sphäroiden einheitlicher Größe mit kunststoff- oder silikonbasierten Multi-Well-Systemenhergestellt 6,7. Die geringere Flexibilität in Bezug auf die Erfassung der genauen heterogenen Struktur von Krebsgewebe mit herkömmlichen Plattformen hat jedoch die Etablierung einer fortschrittlichen Biofabrikationstechnologie erfordert, um eine hochbiomimetische Plattform zur Verbesserung der Krebsforschung aufzubauen8.
Mikrophysiologische 3D-Systeme (MPS) sind nützliche Werkzeuge, um die komplexe Geometrie und das pathologische Fortschreiten von Krebszellen zu rekapitulieren9. Da Krebszellen den biochemischen Gradienten von Wachstumsfaktoren und Chemokinen und die auf dem System reproduzierte mechanische Heterogenität spüren, können wichtige Merkmale der Krebsentfaltung in vitro untersucht werden. Zum Beispiel wurden Krebslebensfähigkeit, metastasierende Malignität und Arzneimittelresistenz in Abhängigkeit von den unterschiedlichen Sauerstoffkonzentrationen mit MPSs10,11untersucht. Trotz der jüngsten Fortschritte beruht die Erzeugung hypoxischer Bedingungen von In-vitro-Modellen auf komplexen Herstellungsverfahren, einschließlich der Verbindung mit physikalischen Gaspumpen. Daher sind einfache und flexible Methoden zum Aufbau krebsspezifischer Mikroumgebungen erforderlich.
Die 3D-Zelldrucktechnologie hat aufgrund ihrer präzisen Kontrolle der räumlichen Anordnung von Biomaterialien zur Rekapitulation nativer biologischer Architekturen erhebliche Aufmerksamkeit erregt12. Insbesondere überwindet diese Technologie die bestehenden Einschränkungen von 3D-Hypoxiemodellen aufgrund ihrer hohen Kontrollierbarkeit und Machbarkeit für den Aufbau der räumlichen Merkmale der Krebsmikroumgebung. Der 3D-Druck erleichtert auch die computergestützte Fertigung durch einen Schicht-für-Schicht-Prozess und bietet so eine schnelle, genaue und reproduzierbare Konstruktion komplexer Geometrien, um tatsächliche Gewebearchitekturen nachzuahmen. Zusätzlich zu den Vorteilen bestehender Herstellungsstrategien für 3D-MPS können die pathophysiologischen Merkmale des Krebsprogressiones durch Musterung der biochemischen, zellulären und biophysikalischen Komponenten reproduziert werden13,14.
Hier stellen wir eine 3D-Zelldruckstrategie für einen hypoxischen Krebs-on-a-Chip vor, um die Heterogenität eines soliden Krebses zu rekapitulieren (Abbildung 1)15. Die Herstellungsparameter wurden durch eine computergestützte Simulation der zentralen Hypoxiebildung im System bestimmt. Krebs-Stroma-Konzentrische Ringe wurden mit Kollagen-Biotinten gedruckt, die Glioblastomzellen und Endothelzellen enthalten, um die Pathophysiologie des Glioblastoms, einer Art von solidem Krebs, nachzuahmen. Die Bildung eines radialen Sauerstoffgradienten verschlimmerte die Malignität des Krebses, was auf eine verstärkte Aggressivität hindeutet. Darüber hinaus zeigen wir Zukunftsperspektiven für die Anwendungen des Chips auf patientenspezifische präklinische Modelle auf. Der vorgeschlagene Ansatz zur Schaffung eines solid-krebsmimetischen mikrophysiologischen Systems soll die Lücke zwischen In-vivo- und In-vitro-Krebsmodellen schließen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Computersimulation der Sauerstoffgradientenbildung
-
Erstellung eines 3D-Geometriemodells für den hypoxischen Krebs-on-a-Chip-Druck
- Führen Sie eine 3D-CAD-Software aus.
- Skizzieren Sie das Geometriemodell von hypoxischem Krebs auf einem Chip. Klicken Sie auf Skizze und wählen Sie die gewünschte Ebene aus, um die Geometrie zu zeichnen. Die Detailskala der einzelnen Teile finden Sie in der Zeichnung (Abbildung 2A).
- Legen Sie die Dicke der Geometrie fest, indem Sie auf Feature-Protrusion Boss/Baseklicken. Geben Sie die gewünschte Dicke (siehe Abbildung 2A)in das leere Feld ein und wählen Sie das grüne Häkchensymbol aus, um die 3D-Geometrie zu bilden.
HINWEIS: Die Dimension des Krebs-auf-einem-Chips wird basierend auf den gewünschten Volumina von Medien und Hydrogel definiert. Im vorliegenden Experiment lagen die gewünschten Medien- und Hydrogelvolumina bei ca. 1.500 μL bzw. 500 μL, basierend auf den bisherigen praktischen Erfahrungen zur Auflösung von extrusionsbasierten Bioprintern. - Speichern Sie die Geometriedatei als 3D-CAD-Dateiformat (.prt oder .stl).
-
Bestimmung der Zelldichte zur Induktion des hypoxischen Kerns
- Führen Sie ein Simulationsprogramm für physikalische Diffusion aus.
- Klicken Sie auf LiveLink und wählen Sie das verwendete CAD-Programm aus. Klicken Sie auf Synchronisieren, um die Geometrie des hypoxischen Krebs-on-a-Chips in das Simulationsprogramm zu importieren. Da der innere Raum der Kammer in einer tatsächlichen experimentellen Umgebung mit einem Kulturmedium gefüllt wird, diffundiert Sauerstoff über den inneren Raum der Kammer und das zelluläre Konstrukt, das aus zellbeladenen Hydrogelen besteht.
HINWEIS: Einzelheiten zu den physikalischen Parametern finden Sie in der vorherigen Studie15. - Definieren Sie die importierte 3D-Geometrie als Kontrollvolumen des Raumes, in dem Sauerstoff diffundiert und die Zellen Sauerstoff verbrauchen (Abbildung 2B).
- Führen Sie eine Computeranalyse für die Gasdiffusionsanalyse nach einem Benutzerhandbuch und zuvor etablierten Methoden16,17durch.
- Exportieren Sie aus den Ergebnissen der Computeranalyse die geschätzten Sauerstoffkonzentrationsdaten über den Querschnitt A-A' zu jedem Zeitpunkt gemäß der Bedienungsanleitung. Die maßgebliche Gleichung basiert auf dem ersten Gesetz von, wie es in Gleichung (1) ausgedrückt ist (Abbildung 2C).
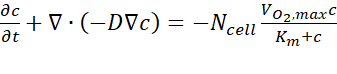
wobei c die Konzentration, D der Sauerstoffdiffusionskoeffizient, N-Zelle die Dichte der Zellen,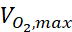 die maximale Aufnahmerate von Sauerstoff und Km die Michaelis-Menten-Konstante ist. Die Konstanten wurden wie in einer früheren Veröffentlichung15beschrieben angewendet.
die maximale Aufnahmerate von Sauerstoff und Km die Michaelis-Menten-Konstante ist. Die Konstanten wurden wie in einer früheren Veröffentlichung15beschrieben angewendet.
HINWEIS: Jeder Zeitpunkt bedeutet einen Schrittpunkt, um die Veränderung der Sauerstoffdiffusion im Laufe der Zeit zu beobachten. - Bewerten Sie, ob der minimale Sauerstoffgehalt eine Hypoxieschwelle erreicht, und wiederholen Sie den Computeranalyseprozess mit einem Inkrement oder einer Abnahme der Zelldichte.
HINWEIS: Definieren Sie, dass hypoxie Gradient im Konstrukt gebildet wird, wenn der Sauerstoffgehalt von 80% im Hydrogelbereich nach 24 h weniger als 0,02 mM beträgt. - Bestätigen Sie die Anzahl der Zellen, die erforderlich sind, um den Sauerstoffgradienten zu erzeugen, der Hypoxie in der zentralen Region induziert, aus erstem Gesetz in Schritt 1.2.5 und die Simulationsergebnisse aus Schritt 1.2.6.
HINWEIS: In diesem Protokoll war die Zellnummer 2 × 106 Zellen / jedes Konstrukt.
2. Zellkultur von Krebszellen und Stromazellen
-
Herstellung von Zellkulturmedien zur Vermeidung physiologischer Belastungen
- Für U-87 MG-Zellen (immortalisierte menschliche Glioblastom-Zelllinie) legen Sie 12 ml des modifizierten Eagle-Mediums von Dulbecco mit hohem Glukosespiegel, das 10% fetales Rinderserum, 100 U / ml Penicillin und 100 μg / ml Streptomycin enthält, in einen T-75-Zellkulturkolben in einem 37 ° C, 5% CO2 befeuchteten Inkubator für 30 minuten, um die thermischen und alkalischen Wirkungen des Mediums auf die Zellen zu minimieren.
HINWEIS: Glioblastom wurde als eine Art von solidem Krebs ausgewählt, weil es aggressive Eigenschaften in einer hypoxischen Umgebung hat. Andere verschiedene Krebsarten können auf dieses Modell angewendet werden. - Bei humanen Nabelschnurvenendothelzellen (HUVECs) werden 12 ml Endotheliale Wachstumsmedium in einen T-75-Zellkulturkolben in einen 5% CO2 befeuchteten Inkubator bei 37 °C für 30 min geben.
HINWEIS: HUVECs wurden ausgewählt, weil es eine der repräsentativsten Endothelzelllinien ist. Verschiedene Arten von Stromazellen können auch auf dieses Modell angewendet werden.
- Für U-87 MG-Zellen (immortalisierte menschliche Glioblastom-Zelllinie) legen Sie 12 ml des modifizierten Eagle-Mediums von Dulbecco mit hohem Glukosespiegel, das 10% fetales Rinderserum, 100 U / ml Penicillin und 100 μg / ml Streptomycin enthält, in einen T-75-Zellkulturkolben in einem 37 ° C, 5% CO2 befeuchteten Inkubator für 30 minuten, um die thermischen und alkalischen Wirkungen des Mediums auf die Zellen zu minimieren.
-
Schnelles Auftauen von kryokonservierten Krebszellen und Stromazellen und deren Erhaltung
- Bewegen Sie Kryoviale mit 5 x 105 U-87 MG-Zellen und HUVECs aus dem Flüssigstickstoffbehälter in einen Laminar-Flow-Schrank. Lösen Und verkleinern Sie die Kappe sofort, um den Innendruck freizugeben.
- Legen Sie die kryokonservierten Zellen vorsichtig für 2 min in ein Wasserbad bei 37 °C und halten Sie die Kappe aus dem Wasser. Spülen Sie die Fläschchen mit 70% Ethanol unter laminarem Durchfluss ab, um eine Kontamination zu vermeiden.
- Die aufgetauten Zellen werden in die Kolben mit den in Schritt 2.1 beschriebenen vorbereiteten Zellkulturmedien überführen und die zellhaltigen Kolben zur Zellrückgewinnung in einen 5%igen CO 2-Befeuchtungsinkubator bei 37 °C eingelegt.
- Aktualisieren Sie das Zellkulturmedium alle 2 Tage und erhalten Sie das Zellwachstum.
- Ersetzen Sie nach 24 Stunden Auftauen die Zellkulturmedien, um eine Zytotoxizität von Dimethylsulfoxid (DMSO) zu vermeiden, das zum Einfrieren der Zellen verwendet wurde. Verwenden Sie HUVECs, die weniger als 6 Passagen durchlaufen haben.
3. Herstellung von Kollagen-Pre-Gel-Lösung
-
Solubilisierung von Kollagenschwamm mit 0,1 N Salzsäure (HCl)
- Bereiten Sie eine Lösung von 0,1 N HCl vor und filtern Sie sie mit einem 0,2 μm Spritzenfilter.
- Für 3 ml einer 1% (w/ v) neutralisierten Kollagen-Pre-Gel-Lösung Kollagenschwämme in 5 x 5 mm2 Stück schneiden und 30 mg wiegen.
- Die geschnittenen Kollagenstücke in eine sterile 10-ml-Glasfläschchen geben.
HINWEIS: Bereiten Sie das 1,5-fache Volumen des erforderlichen Kollagenhydrogels vor, wobei der Verlust des Hydrogels aufgrund der klebrigen Eigenschaft der Kollagenlösung berücksichtigt wird. - 2,4 ml 0,1 N HCl in die kollagenhaltige Glasfläschchen geben und 3 Tage lang bei 15 U/min und 4 °C auf der Wippe inkubieren.
HINWEIS: Das Volumen der 0,1 N HCl-Lösung betrug vier Fünftel des endgefeinten Volumens des erforderlichen Kollagenhydrogels. In diesem Fall wurden 3 ml Kollagen hergestellt. - Nach der Verdauung die unverdauten Kollagenpartikel mit einem 40 μm Zellsieb sieben. Lagern Sie die saure Kollagenlösung bei 4 °C und verwenden Sie sie innerhalb von 7 Tagen.
-
pH-Einstellung für 1% neutralisierte Kollagen-Pre-Gel-Lösung
- Zentrifuge die saure Kollagenlösung bei 1224 x g für 5 min bei 4 °C.
- 30 μL Phenolrotlösung als pH-Indikator zu einer Endkonzentration von 1% (v/v) und 300 μL 10x phosphatgepufferter Kochsalzlösung (PBS) auf eine Endkonzentration von 10% (v/v) in der Kollagen-Pre-Gel-Lösung geben.
- Neutralisieren Sie den pH-Wert auf 7 mit 1 N Natriumhydroxid (NaOH) und überprüfen Sie die Farbänderung.
HINWEIS: Basierend auf der Formel, Mol H+ = Molarität H+ x Volumen H+ = Mol OH-= Molarität OH- x Volumen OH-, addieren Sie 240 μL NaOH. - Fügen Sie destilliertes Wasser hinzu, um ein Gesamtvolumen von 3 ml zu erhalten.
- Nach der pH-Einstellung die 1% (w/v) neutralisierte Kollagen-Pre-Gel-Lösung bei 4 °C lagern und innerhalb von 3 Tagen verwenden.
HINWEIS: Um die Gelierung der neutralisierten Kollagen-Pre-Gel-Lösung vorzuprüfen, machen Sie 50 μL Kollagentröpfchen auf einer kleinen Schüssel mit einer Verdrängerpipette und inkubieren Sie sie in einem 37 °C-Inkubator für 1 h. Beziehen Sie sich auf die folgenden drei Methoden, um die Vernetzung von Kollagentröpfchen zu überprüfen. - Überprüfen Sie, ob sich die Farbe des Kollagens von der transparenten Farbe in undurchsichtiges Weiß verwandelt hat.
- Kippen Sie den Behälter und überprüfen Sie, ob das Kollagen am Boden des Behälters haftet.
- Gießen Sie 1x PBS auf die Tröpfchen und prüfen Sie, ob das Kollagenkonstrukt in der Lösung nicht gebrochen ist.
4.3D Druck von gasdurchlässiger Barriere
-
3D-Druck einer Opfer-Polyform (Ethylen-Vinylacetat) (PEVA)
- Generieren Sie die 3D-Geometrie der in Schritt 1 definierten PEVA-Opferform mit einer 3D-CAD-Software (Abbildung 3A).
HINWEIS: Die 3D-Geometrie und der detaillierte Modellmaßstab einschließlich Bemaßung, Einheiten und Linientypen wurden in Abbildung 2A dargestellt. - Konvertieren Sie die 3D-CAD-Datei in ein STL-Dateiformat, indem Sie auf Datei | Dateityp als STL. Klicken Sie auch auf Option | Ausgabeformular als ASCII zur G-Code-Generierung.
- Klicken Sie auf Datei | Öffnen Sie die STL-Datei und wählen Sie die gespeicherte STL-Datei aus, um die generierte STL-Datei zu importieren. Klicken Sie auf Slice-Modell des STL-CAD-Austauschers, um automatisch den G-Code der PEVA-Opferform zu generieren (Abbildung 3B, C).
HINWEIS: Der Druckpfad wird durch die Verbindung von schnittigen Punkten zwischen der Grundfigur der STL-Datei und der Slicing-Ebene (d. h. Der Ebene) generiert. Grundsätzlich ist die grundlegende Figur eines Fragments in einer STL-Datei ein Dreieck, das die 3D-Koordinaten enthält. Nachdem die schnittigen Punkte zwischen dem Dreieck und der Ebene erhalten wurden, wird ein G-Code für den Druck generiert, indem jeder Punkt ohne überlappenden Pfad auf einer Ebene18verbunden wird. Jeder G-Code-Generierungsalgorithmus auf der Bordsoftware kann verwendet werden, um Druckpfade für die Chipherstellung zu generieren. - Bereiten Sie einen sterilen Klebstoff und einen hydrophilen Histologie-Objektträger vor.
HINWEIS: Das hydrophile Objektträgerglas ist entscheidend für die dauerhafte Bindung von Polydimethylsiloxan (PDMS) auf dem Glas und die Haftung der Kollagenkonstrukte, die Krebszellen und Stromazellen verkapseln. - Drucken Sie die PEVA-Opferform mit einer 50 G Präzisionsdüse bei einem pneumatischen Druck von 500 kPa bei 110 °C auf den Schlitten.
HINWEIS: Die Linienbreite wird durch die Vorschubgeschwindigkeit, die Düsenstärke und die Temperatur des Materials beeinflusst. Es wurde die 50 G Düse verwendet und eine Vorschubgeschwindigkeit von 400 angewendet, um 500 μm Linienbreite für die Opferwand zu erzeugen. Das Düsenmanometer, der pneumatische Druck und die Vorschubgeschwindigkeit werden mit praktischen Ergebnissen definiert19. Die Opferwand muss ausreichend dick sein, um die PDMS-Lösung zu halten, was der nächste Fertigungsschritt ist.
- Generieren Sie die 3D-Geometrie der in Schritt 1 definierten PEVA-Opferform mit einer 3D-CAD-Software (Abbildung 3A).
-
Gießen der Polydimethylsiloxan (PDMS)-Barriere
- 6 mL PDMS-Basiselastomer und 0,6 mL Härter homogen über 5 min in einem Kunststoffbehälter mischen. Dies kann 6 hypoxische Krebs-auf-Chips herstellen, wenn man den Verlust aufgrund der klebrigen Eigenschaft von PDMS berücksichtigt.
- Laden Sie die gemischte PDMS-Lösung in eine 10-ml-Einwegspritze und besinnen Sie den Spritzenkopf mit einer 20 G kunststoffverjüngten Dosierspitze.
- Füllen Sie die PEVA-Opferform mit der gemischten PDMS-Lösung in die Spritze. Das gemischte PDMS füllt die PEVA-Opferform mit einer konvexen Oberfläche. Die Höhe der PDMS-Barriere ist höher als die der PEVA-Form.
- Härten Sie die PDMS-Barriere in einem Ofen bei 40 °C für über 36 h aus, um das Schmelzen von PEVA zu vermeiden. Erhöhen Sie die Temperatur nicht auf über 88 °C, was die Schmelztemperatur von PEVA ist.
- Lösen Sie die PEVA-Opferform mit einer Präzisionspinzette und sterilisieren Sie die gasdurchlässige Barriere bei 120 °C in einem Autoklaven.
5. Herstellung von zellverkapselten Kollagen-Bio-Tinten
-
Ablösung der präparierten Krebszellen und Stromazellen
HINWEIS: Unter Berücksichtigung der Zelllebensfähigkeit sollte der gesamte Druckprozess so schnell wie möglich nach dem Ablösen der Zellen abgeschlossen sein.- Waschen Sie Krebs- und Stromazellen mit 10 ml 1x PBS mit einer serologischen Pipette; mit 2 mL 0,25% Trypsin-Ethylendiamintetraessigsäure (EDTA) mit einer Pipette behandeln und 3 min bei 37 °C inkubieren.
- Neutralisieren Sie die trypsinisierten Zellen mit 3 ml Zellkulturmedien; sammeln Sie die Suspensionen der Zellen in 15 mL konischen Röhrchen und zentrifugieren Sie bei 516 x g für 5 min bei 20 °C.
- Saugen Sie den Überstand langsam an; Resuspendieren Sie die Zellpellets in 5 ml Zellkulturmedien und zählen Sie die Anzahl der Zellen mit einem Hämozytometer.
- 5 x 106 Zellen jedes Zelltyps in neue 15 mL konische Röhrchen geben und bei 516 x g für 5 min bei 20 °C zentrifugieren.
- Saugen Sie den Überstand ab und legen Sie ihn auf nasses Eis.
-
Mischen jedes Zelltyps mit der 1% igen neutralisierten Kollagen-Pre-Gel-Lösung
HINWEIS: Um eine thermische Erstarrung der 1% igen neutralisierten Kollagen-Pre-Gel-Lösung zu vermeiden, sollte dieser Prozess auf nassem Eis durchgeführt werden.- Resuspend jede Art von Zellpellet, das in Schritt 5.1.4 gesammelt wurde, mit jeweils 20 μL Zellkulturmedien.
- Fügen Sie 1 ml der 1% igen neutralisierten Kollagen-Pre-Gel-Lösung in jede der resuspendierten Zellsuspensionen hinzu und mischen Sie sie homogen mit einer Verdrängerpipette. Die Endkonzentration jedes Zelltyps wird 5 x 106 Zellen/ml sein.
- Die zellverkapselten Kollagen-Biotinten mit einer positiven Einwegpipette in 3-ml-Einwegspritzen überführen und die Spritzen bis zum 3D-Zelldruck bei 4 °C lagern.
6.3D Zelldruck von krebs-stroma-konzentrischen Ringen
-
3D-Zelldruck von Kollagen-Biotinten, die Krebszellen und Stromazellen verkapseln
- Generieren Sie die 3D-Geometrie der in Schritt 1.2 definierten konzentrischen Krebs-Stroma-Ringe mit einer 3D-CAD-Software.
HINWEIS: Die Abmessungen der konzentrischen Krebsstromaringe werden über simulierte Parameter definiert. Die endgültigen Dimensionen der Dimensionsparameter sind in Abbildung 3A dargestellt. - Konvertieren Sie die 3D-CAD-Datei in ein STL-Dateiformat und generieren Sie einen G-Code der konzentrischen Krebs-Stroma-Ringe mit einem STL-CAD-Austauscher.
HINWEIS: Informationen zum G-Code-Generierungsalgorithmus finden Sie im Hinweis in Schritt 4.1.2. - Laden Sie die zellverkapselten Kollagen-Biotinten, die in 3-ml-Einwegspritzen enthalten sind, auf den Kopf des 3D-Druckers und stellen Sie die Temperatur von Kopf und Platte auf 15 °C ein.
HINWEIS: Erreicht die Temperatur von Kopf und Platte des Druckers über 37 °C, wird die Biotinte vernetzt und druckt nicht mehr. - Laden Sie den generierten Druckpfad in die Steuerungssoftware des 3D-Druckers.
- Drucken Sie mit einem Klick auf die Schaltfläche Start die Kollagen-Biotinten, die Krebszellen und Stromazellen auf der gasdurchlässigen Barriere nach dem geladenen G-Code verkapseln, mit einer 18 G-Kunststoffnadel bei pneumatischem Druck von ca. 20 kPa bei 15 °C.
- Legen Sie am Ende jedes Druckvorgangs manuell eine sterilisierte 22 mm x 50 mm Glasabdeckung auf die gasdurchlässige Barriere, um den hypoxischen Gradienten zu erzeugen.
HINWEIS: Vergleichen Sie zwei Gruppen in Abhängigkeit vom Vorhandensein einer Glasabdeckung (GR+) und dem Fehlen (GR-) dieser, um die Erzeugung des hypoxischen Gradienten zu überprüfen. - Nachdem drei hypoxische Krebs-auf-Chips erzeugt wurden, werden die Chips für 1 h in einen Inkubator bei 37 °C überführen, um die Kollagen-Biotinen zu vernetzen.
- Generieren Sie die 3D-Geometrie der in Schritt 1.2 definierten konzentrischen Krebs-Stroma-Ringe mit einer 3D-CAD-Software.
-
Abschluss des Herstellungsprozesses und Wartung des hypoxischen Krebs-on-a-Chips
- Nach Abschluss aller 3D-Zelldruckverfahren des hypoxischen Krebs-auf-einem-Chips reiben Sie die Deckbrille vorsichtig auf die gasdurchlässigen Barrieren mit dem Zell-Scrapper zur dichten Verklebung (Abbildung 4A,B).
HINWEIS: Das Deckglas und die gasdurchlässige Barriere werden durch hydrophobe Bindung ohne chemische Klebstoffe zusammengefügt, wobei das gebundene Teil einfach zwischen dem Deckglas und der PDMS-Barriere abgekratzt wird. - 1,5 ml Endotheliales Wachstumsmedium in jeden Chip einführen. Um eine Ablösung des Krebskonstrukts zu vermeiden, führen Sie Zellkulturmedium von einer Seite des Chips ein. Neigen Sie den Chip, damit das Zellkulturmedium mit einer Pipette fließen kann.
- Aktualisieren Sie die Zellkulturmedien eine Woche lang jeden Tag. Verwenden Sie eine Pipette, um das Zellkulturmedium abzusaugen; Verwenden Sie keine Druckpumpe.
- Nach Abschluss aller 3D-Zelldruckverfahren des hypoxischen Krebs-auf-einem-Chips reiben Sie die Deckbrille vorsichtig auf die gasdurchlässigen Barrieren mit dem Zell-Scrapper zur dichten Verklebung (Abbildung 4A,B).
7. Bewertung der Lebensfähigkeit der Post-Printing-Zelle
-
Probenvorbereitung und Behandlung mit Calcein AM und EthD-1 Lösung
- 1x PBS im Wasserbad bei 37 °C erwärmen.
- Die Assay-Lösung wird durch Zugabe von 0,75 μL Calceinacetoxymethyl (Calcein AM) und 3 μL Ethidiumhomdimer (EthD-1) zu 1,5 ml vorgewärmtem PBS zubereitet.
- Saugen Sie vorsichtig alle Medien mit einer Pipette vom Chip ab.
- Waschen Sie das Krebskonstrukt mit vorgewarmtem PBS. Füllen Sie 1,5 mL PBS mit einer Pipette in den Chip und lassen Sie ihn 10 min bei Raumtemperatur stehen. Um eine Verformung des Krebskonstrukts zu vermeiden, führen Sie 1x PBS von einer Seite der Chips ein und neigen Sie die Chips, damit 1x PBS fließen kann.
- Saugen Sie das PBS vom Chip ab; Behandeln Sie die 1,5 ml Assay-Lösung und inkubieren Sie den Chip bei 37 °C für 20 min mit einer Folie zum Schutz vor Licht. Verwenden Sie eine Pipette, um 1x PBS anzusaugen; Verwenden Sie keine Saugpumpe.
-
Abbildung der Zelllebensfähigkeit mit einem Fluoreszenzmikroskop
- Betrachten und erfassen Sie die markierten Zellen mit einem Fluoreszenzmikroskop (Abbildung 4C).
HINWEIS: Calcein AM markiert lebende Zellen mit grüner Fluoreszenz (Wellenlänge ~ 488 nm). EthD-1 stellt das Signal toter Zellen mit roter Fluoreszenz (Wellenlänge ~594 nm) dar. - Zählen Sie die Anzahl der lebenden und toten Zellen mit Bildgebungssoftware, einem Open-Source-Bildverarbeitungsprogramm, und berechnen Sie die Lebensfähigkeit mit den Zahlen.
- Betrachten und erfassen Sie die markierten Zellen mit einem Fluoreszenzmikroskop (Abbildung 4C).
8. Immunfluoreszenz zur Validierung der Bildung der zentralen Hypoxie und ihrer Wirkung auf die Malignität von Krebs
-
Fixierung, Permeabilisierung und Blockierung des Krebskonstrukts
- Bereiten Sie 1x PBS, 4% Paraformaldehyd (PFA), 0,1% (v/v) Triton X-100 und 2% (w/v) Rinderserumalbumin (BSA) bei Raumtemperatur vor.
- Saugen Sie vorsichtig alle Medien mit einer Pipette vom Chip ab und spülen Sie den Chip dreimal mit 1x PBS aus. Um eine Verformung des Krebskonstrukts zu vermeiden, führen Sie 1x PBS von einer Seite der Chips ein und neigen Sie die Chips, damit 1x PBS fließen kann. Lassen Sie den Chip zwischen jedem Waschschritt mit 1x PBS für 5 min stehen, um Restlösungen zu entfernen.
HINWEIS: 1x PBS wurde mit einer Pipette und nicht mit einer Druckpumpe aspiriert. - Fügen Sie 500 μL von 4% PFA mit einer Pipette zum Krebskonstrukt auf dem Chip hinzu; Lassen Sie es für 15 minuten und waschen Sie dreimal mit 1x PBS, um die Zellen im Krebskonstrukt zu fixieren.
- Behandeln Sie das Krebskonstrukt mit 500 μL 0,1% Triton X-100 mit einer Pipette bei Raumtemperatur für 5 min und waschen Sie dreimal mit 1x PBS, um die Zellmembran zu solubilisieren und zu permeabilisieren.
- Behandeln Sie das Krebskonstrukt mit 500 μL 2% BSA mit einer Pipette bei Raumtemperatur für 1 h, um reaktive Epitope zu blockieren.
HINWEIS: Decken Sie den Chip mit Paraffinfilm ab, um eine Verdunstung zu verhindern. - Nach 1 h den Chip dreimal mit 1x PBS waschen.
-
Behandlung mit primärem Antikörper, sekundärem Antikörper und DAPI und Abbildung der Struktur mit einem konfokalen Mikroskop.
- Bereiten Sie Isotyp-Kontrollantikörper und den Cocktail der primären Antikörper vor, indem Sie die Antikörper in 1x PBS auf jede gewünschte Arbeitskonzentration verdünnen.
HINWEIS: Die spezifischen Details der Antikörper sind in der Materialtabelleaufgeführt. Es sollten die gleichen Arbeitskonzentrationen von Isotyp-Kontrollantikörpern wie bei den primären Antikörpern verwendet werden. - Saugen Sie vorsichtig alle 1x PBS mit einer Pipette vom Chip ab und behandeln Sie den Chip über Nacht mit 200 μL primärer Antikörperlösung bei 4 °C. Decken Sie die Chips mit Paraffinfilm ab, um eine Verdunstung zu verhindern.
- Saugen Sie die primäre Antikörperlösung an und waschen Sie den Chip dreimal mit 1x PBS.
- Sekundäre Antikörper und DAPI in 1x PBS auf die gewünschte Arbeitskonzentration verdünnen.
HINWEIS: Ein grüner fluoreszenzkonjugierter sekundärer Antikörper wird in diesem Fall im Verhältnis 1:200 verwendet. DAPI wurde im Verhältnis 1:1000 verwendet. - Saugen Sie vorsichtig alle 1x PBS mit einer Pipette vom Chip ab und behandeln Sie den Chip mit 200 μL sekundärer Antikörper-DAPI-Lösung bei 4 °C für 3 h. Bedecken Sie den Chip mit Paraffinfilm, um eine Verdunstung zu verhindern, und wickeln Sie ihn dann mit Aluminiumfolie ein, um Einblendung zu verhindern.
- Saugen Sie die sekundäre Antikörper-DAPI-Lösung ab und waschen Sie den Chip dreimal mit 1x PBS.
- Nachdem Sie den Färbeschritt beendet haben, übertragen Sie das Krebskonstrukt in eine konfokale Schale, indem Sie vorsichtig mit einer Zette greifen.
- Visualisieren und erfassen Sie die markierten Zellen mit einem konfokalen Mikroskop (Abbildung 5).
HINWEIS: Die Wellenlänge des konfokalen Mikroskops wurde je nach Art der fluoreszierenden Marker angepasst. Die spezifischen Details der Antikörper sind in der Materialtabelle aufgeführt. Um die Zellposition effizient zu erfassen, wäre es besser, zunächst die DAPI-gefärbten Kerne des Konstrukts zu beobachten. Die Detektionsanregungs-/Emissionswellenlängen der fluoreszierenden Signale waren 358/461 nm (DAPI, Blau), 494/517 nm (Grün) und 590/617 nm (Rot). Die Vergrößerungen waren 4x, 10x und 20x, angepasst von der niedrigsten zur höchsten.
- Bereiten Sie Isotyp-Kontrollantikörper und den Cocktail der primären Antikörper vor, indem Sie die Antikörper in 1x PBS auf jede gewünschte Arbeitskonzentration verdünnen.
9. Statistische Auswertung
-
Zellzählung mit Bildverarbeitungsprogramm
- Führen Sie ein Bildverarbeitungsprogramm aus, um die Anzahl der lebenden und toten Zellen zu zählen.
- Öffnen Sie die fluoreszierenden Bilddateien. Klicken Sie auf Datei | Öffnen und importieren Sie die TIFF-Bilder.
- Konvertieren Sie die Bilder in 16-Bit-Graustufenbilder. Klicken Sie auf Image | Typ | 16-Bit-Graustufen.
- Passen Sie den Schwellenwert an, indem Sie auf Image | | anpassen Schwellenwert und wählen Sie dann die Farbe der Zellen aus, die schwarz sein sollen.
- Schneiden Sie zusammengeführte Zellen auseinander, indem Sie auf Prozess | Binäre | Wasserscheide für präzises Zellzählen.
- Zählen Sie die Anzahl der Zellen, indem Sie dreimal auf Analysieren und dann auf Partikel analysieren klicken. Berechnen Sie den Durchschnitt und präsentieren Sie die Daten als Mittelwert ± Standardfehlers.
HINWEIS: Immunfluoreszenzmarker wurden durch Vergleich der Fluoreszenzintensität analysiert.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Der hypoxische Krebs-auf-einem-Chip wurde mit computergestützter 3D-Zelldrucktechnologie entwickelt, um Hypoxie und krebsbedingte Pathologie zu rekapitulieren (Abbildung 1). Sauerstofftransport und -verbrauch wurden mit dem 3D-Geometriemodell simuliert. Der Chip wurde in Form von konzentrischen Ringen entwickelt, um die radiale Sauerstoffdiffusion und -erschöpfung in Krebsgeweben nachzuahmen (Abbildung 2A). Nach der Definition ...
Access restricted. Please log in or start a trial to view this content.
Diskussion
In dieser Studie beschreiben wir den Herstellungsprozess eines hypoxischen Krebs-auf-einem-Chips auf Basis der 3D-Zelldrucktechnologie. Die Bildung des hypoxischen Gradienten im entworfenen Chip wurde durch Computersimulationen vorhergesagt. Die Umgebung, die einen heterogenen hypoxischen Gradienten induzieren kann, wurde über eine einfache Strategie reproduziert, die die 3D-gedruckte gasdurchlässige Barriere und die Glasabdeckung kombiniert. Die hypoxiebedingten pathologischen Merkmale des Glioblastoms, einschließlic...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Angaben.
Danksagungen
Diese Forschung wurde von der National Research Foundation of Korea (NRF) unterstützt, die vom Bildungsministerium (Nr. 2020R1A6A1A03047902 und NRF-2018H1A2A1062091) und der koreanischen Regierung (MSIT) (Nr. NRF-2019R1C1C1009606 und NRF-2019R1A3A3005437).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Cells | |||
| Human umbilical vein endothelial cells | Promocell | C-12200 | |
| U-87 MG cells | ATCC | ATCC HTB-14 | |
| Disposable | |||
| 0.2 μm syringe filter | Sartorius | 16534-K | |
| 10 mL disposable syringe | Jung Rim | 10ml 21G32 | |
| 10 mL glass vial | Hubena | A0039 | |
| 10 mL Serological pipette tip | SPL lifescience | 91010 | |
| 15 mL conical tube | SPL lifescience | 50015 | |
| 18G plastic needle | Musashi engineering | PN-18G-B | |
| 20G plastic tapered dispense tip | Musashi engineering | TPND-20G-U | |
| 22x50 glass cover | MARIENFIELD | 0101142 | |
| 25 mL Serological pipette tip | SPL lifescience | 90125 | |
| 3 mL disposable syringes | HENKE-JET | 4020-X00V0 | |
| 40 µm cell strainer | Falcon | 352360 | |
| 5 mL Serological pipette tip | SPL lifescience | 91005 | |
| 50 mL conical tube | SPL lifescience | 50050 | |
| 50 mL Serological pipette tip | SPL lifescience | 90150 | |
| 50N precision nozzle | Musashi engineering | HN-0.5ND | |
| Aluminum foil | SINKWANG | ||
| Capillary tips | Gilson | CP1000 | |
| Cell-scrapper | SPL lifescience | 90030 | |
| Confocal dish | SPL lifescience | 200350 | |
| Parafilm | Bemis | PM996 | |
| Pre-coated histology slide | MATSUNAMI | MAS-11 | |
| Reservoir | SPL lifescience | 23050 | |
| T-75 cell culture flask | SPL lifescience | 70075 | |
| Equipment | |||
| 3DX printer | T&R Biofab | ||
| Autoclave | JEIOTECH | AC-12 | |
| Centrifuger | Cyrozen | 1580MGR | |
| Confocal laser microscopy | Olympus Life Science | FV 1000 | |
| Fluorescence microscope | FISHER SCEINTIFIC | O221S366 | |
| Forcep | Korea Ace Scientific | HC.203-30 | |
| Hand tally counter | KTRIO | ||
| Hemocytometer | MARIENFIELD | 0650030 | |
| Incubator | Panasonic | MCO-170AIC | |
| Laminar flow cabinet | DAECHUNG SCIENCE | CB-BMMS C-001 | |
| Metal syringe | IWASHITA engineering | SUS BARREL 10CC | |
| Operating Scissors | Hirose | HC.13-122 | |
| Oven | JEIOTECH | OF-12, H070023 | |
| Positive displacement pipette | GILSON | NJ05652 | |
| Refrigerator | SAMSUNG | CRFD-1141 | |
| Voltex Mixer | DAIHAN scientific | VM-10 | |
| Water bath | DAIHAN SCIENTIFIC | WB-11 | |
| Water purifier | WASSER LAB | DI-GR | |
| Materials | |||
| 0.25 % Trypsin-EDTA | Gibco | 25200-072 | |
| 10x PBS | Intron | IBS-BP007a | |
| 4% Paraformaldehyde | Biosesang | ||
| 70% Ethanol | Daejung | 4018-4410 | |
| Anti-CD31 antibody | Abcam | ab28364 | |
| Anti-HIF-1 alpha antibody | Abcam | ab16066 | |
| Anti-SHMT2/SHMT antibody | Abcam | ab88664 | |
| Anti-SOX2 antibody | Abcam | ab75485 | |
| Bovine Serum Albumin | Thermo scientific | J10857-22 | |
| Collagen from porcine skin | Dalim tissen | PC-001-1g | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Thermofisher | D1306 | |
| Endothelial Cell Growth Medium-2 | Promocell | C22011 | |
| Fetal bovine serum | Gibco | 12483-020 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Theromofisher | A-11001 | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 594 | Theromofisher | A-11012 | |
| High-glucose Dulbecco’s Modified Eagle Medium(DMEM) | Hyclone | SH30243-0 | |
| Hydrochloric acid | Sigma-Aldrich | 311413-100ML | |
| Live/dead assay kit | Invitrogen | L3224 | |
| Mouse IgG1, kappa monoclonal [15-6E10A7] - Isotype Control | Abcam | ab170190 | |
| Penicillin/streptomycin | Gibco | 15140-122 | |
| Phenol red solution | Sigma-Aldrich | P0290-100ML | |
| Poly(ethylene-vinyl acetate) | Poly science | 06108-500 | |
| Polydimethylsiloxane | Dowhitech | sylgard 184 | |
| Rabbit IgG, polyclonal - Isotype Control | Abcam | ab37415 | |
| Sodium hydroxide solution | Samchun | S0610 | |
| Triton X-100 | Biosesang | TRI020-500-50 | |
| Trypan Blue | Sigma-Aldrich | T8154 | |
| Software | |||
| COMSOL Multiphysics 3.5a | COMSOL AB | ||
| IMS beamer | in-house software | ||
| SolidWorks Package | Dassault Systems SolidWorks Corporation |
Referenzen
- Jing, X., et al. Role of hypoxia in cancer therapy by regulating the tumor microenvironment. Molecular Cancer. 18 (1), 157(2019).
- Al Tameemi, W., Dale, T. P., Al-Jumaily, R. M. K., Forsyth, N. R. Hypoxia-modified cancer cell metabolism. Frontiers in Cell and Developmental Biology. 7, 4(2019).
- Petrova, V., Annicchiarico-Petruzzelli, M., Melino, G., Amelio, I. The hypoxic tumour microenvironment. Oncogenesis. 7 (1), 1-13 (2018).
- Hockel, M., Vaupel, P. Tumor hypoxia: definitions and current clinical, biologic, and molecular aspects. Journal of the National Cancer Institute. 93 (4), 266-276 (2001).
- Kim, H., Lin, Q., Glazer, P. M., Yun, Z. The hypoxic tumor microenvironment in vivo selects the cancer stem cell fate of breast cancer cells. Breast Cancer Research. 20 (1), 16(2018).
- Jeong, G. S., Lee, J., Yoon, J., Chung, S., Lee, S. -H. Viscoelastic lithography for fabricating self-organizing soft micro-honeycomb structures with ultra-high aspect ratios. Nature Communications. 7 (1), 1-9 (2016).
- Razian, G., Yu, Y., Ungrin, M. Production of large numbers of size-controlled tumor spheroids using microwell plates. Journal of Visualized Experiments:JoVE. (81), e50665(2013).
- Nunes, A. S., Barros, A. S., Costa, E. C., Moreira, A. F., Correia, I. J. 3D tumor spheroids as in vitro models to mimic in vivo human solid tumors resistance to therapeutic drugs. Biotechnology and Bioengineering. 116 (1), 206-226 (2019).
- Wan, L., Neumann, C., LeDuc, P. Tumor-on-a-chip for integrating a 3D tumor microenvironment: chemical and mechanical factors. Lab on a Chip. 20 (5), 873-888 (2020).
- Nam, H., Funamoto, K., Jeon, J. S. Cancer cell migration and cancer drug screening in oxygen tension gradient chip. Biomicrofluidics. 14 (4), 044107(2020).
- Palacio-Castañeda, V., Kooijman, L., Venzac, B., Verdurmen, W. P., Le Gac, S. Metabolic switching of tumor cells under hypoxic conditions in a tumor-on-a-chip model. Micromachines. 11 (4), 382(2020).
- Ronaldson-Bouchard, K., Vunjak-Novakovic, G. Organs-on-a-chip: a fast track for engineered human tissues in drug development. Cell Stem Cell. 22 (3), 310-324 (2018).
- Mi, S., Du, Z., Xu, Y., Sun, W. The crossing and integration between microfluidic technology and 3D printing for organ-on-chips. Journal of Materials Chemistry B. 6 (39), 6191-6206 (2018).
- Yi, H. -G., Lee, H., Cho, D. -W. 3D printing of organs-on-chips. Bioengineering. 4 (1), 10(2017).
- Yi, H. -G., et al. A bioprinted human-glioblastoma-on-a-chip for the identification of patient-specific responses to chemoradiotherapy. Nature Biomedical Engineering. 3 (7), 509-519 (2019).
- Kang, T. -Y., Hong, J. M., Jung, J. W., Yoo, J. J., Cho, D. -W. Design and assessment of a microfluidic network system for oxygen transport in engineered tissue. Langmuir. 29 (2), 701-709 (2013).
- Woo Jung, J., et al. Evaluation of the effective diffusivity of a freeform fabricated scaffold using computational simulation. Journal of Biomechanical Engineering. 135 (8), (2013).
- Brown, A. C., De Beer, D. Development of a stereolithography (STL) slicing and G-code generation algorithm for an entry level 3-D printer. 2013 Africon (IEEE). , 1-5 (2013).
- Shim, J. -H., Lee, J. -S., Kim, J. Y., Cho, D. -W. Bioprinting of a mechanically enhanced three-dimensional dual cell-laden construct for osteochondral tissue engineering using a multi-head tissue/organ building system. Journal of Micromechanics and Microengineering. 22 (8), 085014(2012).
- Gillispie, G., et al. Assessment methodologies for extrusion-based bioink printability. Biofabrication. 12 (2), 022003(2020).
- Kim, B. S., Das, S., Jang, J., Cho, D. -W. Decellularized extracellular matrix-based bioinks for engineering tissue-and organ-specific microenvironments. Chemical Reviews. 120 (19), 10608-10661 (2020).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten