Method Article
Probenvorbereitung für metabolisches Profiling mittels MALDI-Massenspektrometrie-Bildgebung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Das Ziel dieses Protokolls ist es, detaillierte Anleitungen zur Probenvorbereitung bei der Planung von Experimenten mit MALDI MSI zur Maximierung des metabolischen und molekularen Nachweises in biologischen Proben bereitzustellen.
Zusammenfassung
Die Metabolomik, die Studie zur Identifizierung und Quantifizierung kleiner Moleküle und Metaboliten, die in einer experimentellen Probe vorhanden sind, hat sich als wichtiges Werkzeug zur Untersuchung der biologischen Aktivitäten während der Entwicklung und Krankheiten erwiesen. Metabolomik-Ansätze werden häufig bei der Untersuchung von Krebs, Ernährung / Ernährung, Diabetes und anderen physiologischen und pathologischen Zuständen mit Stoffwechselprozessen eingesetzt. Ein vorteilhaftes Werkzeug, das bei der metabolomischen Profilierung hilft, das in diesem Artikel befürwortet wird, ist die matrixgestützte Laserdesorption / Ionisations-Massenspektrometrie-Bildgebung (MALDI MSI). Seine Fähigkeit, Metaboliten in situ ohne Markierung, strukturelle Modifikationen oder andere spezialisierte Reagenzien, wie sie bei der Immunfärbung verwendet werden, nachzuweisen, macht MALDI MSI zu einem einzigartigen Werkzeug bei der Weiterentwicklung von Methoden, die auf dem Gebiet der Metabolomik relevant sind. Ein geeigneter Probenvorbereitungsprozess ist entscheidend, um optimale Ergebnisse zu erzielen, und wird im Mittelpunkt dieses Papiers stehen.
Einleitung
Metaboliten, die Zwischen- oder Endprodukte des Stoffwechsels, einschließlich Nukleotide, Aminosäuren oder organische Säuren, Lipide, sind Schlüsselkomponenten für biologische Funktionen und Prozesse. Die Metabolomik, die Untersuchung von Metaboliten, ermöglicht die Erforschung ihrer biochemischen Wechselwirkungen und das Verständnis ihrer Rolle im Kontext der Grundlagen-, translationalen und klinischen Forschung. Metaboliten sind stark mit den Phänotypen von Organismen assoziiert und geben Aufschluss über biochemische Aktivitäten, die während des Zellstoffwechsels auftreten1. Daher hat sich die Metabolomik neben der Genomik und Proteomik zu einem wichtigen Werkzeug zum Verständnis physiologischer und pathologischer Zustände entwickelt. Zum Beispiel wird die Metabolomik verwendet, um die Mechanismen hinter bestehenden Medikamenten sowie deren Toleranz zu klären. In der Arzneimittelentwicklung ist der xenobiotische Stoffwechsel nützlich, um die Aktivität oder Toxizität von Metaboliten über Spezies hinweg zu beurteilen, was später zur Unterstützung der personalisierten Medizinführt 2. Trotz der breiten Anwendung der Metabolomik kann die Bildgebung von Metaboliten aufgrund der chemischen Reaktivität, der strukturellen Heterogenität und des breiten Konzentrationsbereichs der Metaboliten eine Herausforderung darstellen3. Die Konzentrationen labiler Metaboliten, einschließlich hochenergetischer Verbindungen, Glukose, Laktat, Glykolytikum, Pentose-Shunt-Signalweg und TCA-Zyklus-Zwischenprodukte, Phospholipide, Neurotransmitter, Signalverbindungen, können sich jedoch innerhalb von Sekunden ändern und über Minuten fortschreiten, wenn Gewebeenzyme während der Gewebeentnahme aktiv sind, wie z. B. postmortale Ischämie bei der Gehirnernte4,5,6 . Um eine genaue Metabolomik-Datenerfassung zu gewährleisten, ist eine angemessene und sorgfältige Probenvorbereitung entscheidend7,8. Zu den derzeit etablierten Plattformen für die Messung von Metaboliten gehören NMR, Enzymassays und Massenspektrometrie (einschließlich Flüssigkeits- und Gaschromatographie), von denen die letzte weiter unten diskutiert wird.
MALDI-MSI ist eine hochmoderne Technik, die die Analyse komplexer Proben durch den Nachweis einzelner molekularer Spezies ermöglicht. MALDI MSI bietet den Vorteil, verschiedene molekulare Verbindungen in biologischen Proben schnell und reproduzierbar messen zu können. Die massenspektrometrische Bildgebung ermöglicht außerdem die Erstellung von Bildern, die die Gewebebiologie auf der Grundlage ihrer zusammengesetzten Metaboliten darstellen, und zwar unter Beibehaltung der räumlichen Verteilung der Metaboliten in der Probe9. Die Fähigkeit von MALDI, Analyten in einer Probe ohne die Verwendung von Antikörpermarkierung, strukturellen Modifikationen oder anderen spezialisierten Reagenzien, wie sie bei der Immunfärbung verwendet werden, nachzuweisen, in Verbindung mit seiner Fähigkeit, Hunderte von Molekülen innerhalb eines einzigen Experiments zu überwachen, sind nur einige der Vorteile, die ms imaging beim metabolischen Profiling gewährt10. ( 11) DER PRÄSIDENT. - Nach der 11 Neben häufig verwendeten Matrixen wie 2,5-Dihydroxybenzoesäure (DHB) und 9-Aminoacridin (9-AA) hat die kürzlich entdeckte neuartige Matrix N-(1-Naphthyl) Ethylendiamindihydrochlorid (NEDC), die sich gut für Analysen verschiedener niedermolekularer Metaboliten eignet, die Anwendung von MALDI MSI im metabolischen Profiling weiter verbessert12.
Trotz der breiten Anwendung von MALDI MSI verhindern die hohen Kosten des Instruments und die Komplexität des experimentellen Verfahrens seine breitere Implementierung in einzelnen Forschungslabors. Daher werden die meisten MALDI MSI-Studien durch gemeinsame Kerneinrichtungen unterstützt. Die Probenvorbereitung, einschließlich Objektträgervorbereitung und Matrixbeschichtung, ist der kritischste Schritt in MALDI MSI. Die Objektträgervorbereitung wird jedoch normalerweise im Labor eines einzelnen Forschers durchgeführt, was zu möglichen Variationen bei der späteren MALDI-MSI-Erfassung führt. Hier wollen wir ein detailliertes Protokoll für die Probenvorbereitung biologischer Proben bereitstellen, bevor wir mit MALDI MSI-Messungen fortfahren, und ein metabolomisches Profiling des Entwicklungsgehirns der Maus als Beispiel verwenden.
Protokoll
Das Protokoll folgt den Richtlinien des Institutional Animal Care and Use Committee (IACUC) des Advanced Science Research Center (ASRC) der City University of New York (CUNY).
1. Ernte das Gewebe
- Bereiten Sie das Aluminiumboot vor. Bereiten Sie ein Rechteck Aluminium 10cm x 20 cm vor, falten Sie es in der Mitte, um 10 cm Doppelschicht quadratisch zu machen. Beschriften Sie die Probeninformationen auf einer Seite und falten Sie die andere Seite, um ein Boot mit einer Unterseite von etwa 4 cm x 4 cm zu bilden.
- Das Boot auf dem flüssigen Stickstoff (LN2)in einer Styroporbox vorkühlen.
HINWEIS: Wenn das Boot zu klein ist, kann die Probe aus dem Boot fallen und durch direkten Kontakt mit LN2übergefroren werden, was zu einer Fragmentierung während der Kryosektion führen kann. - Euthanisieren Sie das Tier durch zervikale Dislokation nach institutionellen IACUC-Richtlinien, sezieren Sie sofort das Gewebe von Interesse.
- Halten Sie das Intervall zwischen Tieranästhesie und Schnappgefrieren so kurz wie möglich, um die Veränderung der Metaboliten während der Gewebeentnahme, insbesondere der labialen Metaboliten im Gehirn aufgrund einer postmortalen Ischämie, zu minimieren.
HINWEIS: Die Durchblutung des Tieres mit phosphatgepufferter Kochsalzlösung (PBS) erhöht die Dauer der Ischämie, verändert die Konzentrationen labiler Metaboliten weiter und übertreibt die künstlichen Ergebnisse. Daher wird das Perfusionieren oder Waschen des Gewebes mit PBS nicht empfohlen, es sei denn, die Kontamination von Blut oder Körperflüssigkeit ist von mehr Betroffener als die Verschlechterung der Metaboliten oder das Auswaschen für das individuelle Projekt.
- Halten Sie das Intervall zwischen Tieranästhesie und Schnappgefrieren so kurz wie möglich, um die Veränderung der Metaboliten während der Gewebeentnahme, insbesondere der labialen Metaboliten im Gehirn aufgrund einer postmortalen Ischämie, zu minimieren.
- Legen Sie das Gewebe sofort in das Aluminiumboot, das auf flüssigem Stickstoff schwimmt, schließen Sie den Deckel der Styroporbox und frieren Sie je nach Größe des Gewebes 2-10 Minuten ein: 2 min, 5 min, 7 min für den postnatalen Tag 1 (P1), P21, P60 Mausgehirn bzw. 10 min für P60 Rattengehirn.
HINWEIS: Frieren Sie das Gewebe nicht für längere Zeit ein (z. B. 5 Minuten für das P1-Mausgehirn), da das überfrorene Gewebe während der Kryosektion zu einer Gewebefragmentierung führen kann. - Entfernen Sie das Boot mit einer Zette, falten Sie die Folie, um das Gewebe einzuwickeln, transportieren Sie es auf Trockeneis und lagern Sie es bei -80 °C für den späteren Gebrauch. Wenn eine sofortige Schnittung folgt, tragen Sie die Proben auf Trockeneis zum Kryostaten.
HINWEIS: Um die Metaboliten besser zu erhalten, wird es bevorzugt, die Probe in intaktem Gewebe zu lagern und direkt vor der MALDI-Bildgebung zu abschnitten. Das Gewebe kann bei -80 °C bis zu 24 Monate gelagert werden.
2. Kryosektion des Gewebes
HINWEIS: Tragen Sie beim Umgang mit den Indium tinoxid (ITO)-Objektträgern jederzeit Handschuhe. Vermeiden Sie direktes Atmen auf dem Objektträger oder tragen Sie Masken (optional), um die Kontamination des menschlichen Speichels auf dem Gewebeabschnitt zu verhindern.
- Bevor Sie das Gewebe teilen, sammeln Sie die gewünschte Anzahl von MALDI-kompatiblen ITO-beschichteten Glasobjektträgern.
- Testen Sie die Leitfähigkeit des Objektträgers mit einem voltmeter, das auf Widerstand eingestellt ist. Beschriften Sie die Seite, auf der eine Widerstandsmessung gelesen wird: Dies ist die Seite, an der die Gewebeabschnitte haften. Legen Sie den Dia immer auf ein sauberes Papiertuch, um eine Kontamination zu vermeiden.
- Kühlen Sie die Dias in einem auf -20 °C eingestellten Kryostaten vor.
- Wenn Gewebeproben aus den -80 °C entfernt wurden, lassen Sie das Gewebe je nach Größe des Gewebes für ca. 45-60 min in der Kryostatkammer ausgleichen. Wenn Proben aus Trockeneis entfernt werden, gleichen Sie sie für ca. 30 Minuten aus.
- Reinigen Sie den Kryostaten gründlich mit 70% Ethanol. Kühlen Sie alle notwendigen Werkzeuge einschließlich dünnspitzigem Künstlerpinsel und Pinzette in der Kryostatkammer vor.
- Stellen Sie die Temperatur der Kryostatkammer und des Probenkopfes entsprechend der Art des Gewebes ein: -14 °C für Leber, -20 °C für Muskeln und -25 °C für Haut9.
- Montieren Sie das Gewebe mit Kryogewebe einbettender Verbindung OCT am Futter, um OCT aus dem interessierenden Bereich zu vermeiden.
- Legen Sie eine saubere Klinge in die Bühne und verriegeln Sie. Stellen Sie die Position der Bühne und den Winkel der Probe ein, um den gewünschten Schnittwinkel zu erreichen.
HINWEIS: Wenn verschiedene Arten / Genotypen von Gewebe geschnitten werden sollen, stellen Sie sicher, dass Sie entweder die Probe neu positionieren, so dass ein sauberer Teil der Klinge verwendet wird, oder vor dem Schneiden der nächsten Probe zu einer neuen Klinge wechseln, um eine Kreuzkontamination zu vermeiden. - Schneiden Sie weiter, bis die interessierende Region (z. B. Corpus callosum im Gehirn) gefunden ist. Achten Sie darauf, die Bühne sauber zu halten, indem Sie zusätzliche Stücke mit einem Künstlerpinsel abbürsten, der im Kryostaten ausgeglichen wurde.
- Sobald der gewünschte Bereich aufgedeckt ist, schneiden Sie kleinere Abschnitte von 10-12 μm Dicke. Wenn der Abschnitt dazu neigt, leicht zu flocken oder auseinanderzufallen, erhöhen Sie die Temperatur des Kryostaten und bleiben Sie im Bereich von -22 ° C bis -11 ° C. Wir haben festgestellt, dass die optimale Schnitttemperatur für Hirngewebe -15 °C bis - 18 °C beträgt.
- Sobald ein guter Abschnitt geschnitten wurde, kleben Sie ihn auf den ITO-Objektträger (in der Kryostatkammer arbeiten).
- Übertragen Sie einen Abschnitt des Gewebes mit der Spitze des Künstlerpinsels auf ITO-Dias.
- Erwärmen Sie den Abschnitt mit den Fingern, indem Sie einen Finger unter den Schlitten legen, um den Abschnitt aufzuwärmen, um eine sichere Montage zu gewährleisten. Der Gewebeabschnitt wird zuerst in 5-10 s transparent und dann in etwa 30-60 s undurchsichtig.
- Legen Sie den Schieber vorsichtig im Kryostaten beiseite.
- Wiederholen Sie die Schritte für andere Gewebeproben und stellen Sie sicher, dass jeder Abschnitt des Gewebes gleichmäßig auf dem Objektträger platziert und so ausgerichtet wie möglich ausgerichtet ist.
- Da das MALDI-Target bis zu zwei Objektträger aufnehmen kann, platzieren Sie die Abschnitte aus mehreren Proben derselben Kohorte auf einem einzigen Objektträger oder auf zwei Objektträgern, um sicherzustellen, dass sie gleichzeitig analysiert werden können.
- Wenn Sie fertig sind, legen Sie ITO-Dias in eine Vakuumbox und geben Sie sie mit Trockenmittel in einen Trockenmittel. Vakuumtrocknen Sie den Schlitten für 45-60 min.
HINWEIS: Wenn im Labor kein Vakuum-Aussauger verfügbar ist, halten Sie die Objektträger ständig unter -20 °C, um eine Verschlechterung der Metaboliten zu vermeiden. - Lagerung und Versand von Objektträgern: Sofern die Probe nicht von den MALDI-Bildgebungskerneinrichtungen vorbereitet wird, um direkt mit der MALDI-Bildgebung fortzufahren, lagern Sie die Objektträger bei -80 ° C oder versenden Sie sie an Kerneinrichtungen oder andere MALDI-Forschungslabors auf Trockeneis.
- Um die Proben bestmöglich zu konservieren, legen Sie die Dias in den Diatransporter, füllen Sie ihn mit Stickstoff (optional), versiegeln Sie ihn mit Parafilm, legen Sie ihn in einen Reißverschlussbeutel, der dann in einen anderen Reißverschlussbeutel mit Trockenmittel gelegt wird. Beschriften Sie den äußeren Reißverschlussbeutel.
- Lagerung bei -80 °C (bis zu 6 Monate) oder Versand mit ausreichend Trockeneis.
3. Matrixvorbereitung
- Bereiten Sie die Matrix vor.
HINWEIS: Alle Reagenzien müssen HPLC-Grad haben.- NEDC in einer Konzentration von 10 mg/ml zubereiten. 10 ml Matrix in einem Lösungsmittel aus 70% Methanol gelöst (100 mg NEDC, 7 ml Methanol, 3 ml H2O) werden10ml Methanol vorbereitet.
- Bereiten Sie zusätzlich eine zusätzliche 10 ml 70% ige Methanol: Wasserlösung vor, um das Sprühsystem vor dem Einfüllen der Matrix zu spülen.
- Sobald die Objektträger in Schritt 2.17 dehydriert sind, platzieren Sie "X" -Markierungen auf den leeren Raum des Glasobjektträgers außerhalb der Gewebeabschnitte mit einem fetten Punkt Silbermarker und legen Sie dann ein zweites "x" mit einem feinen Punkt schwarzen Marker auf das silberne "X". Das schwarze "X" mit einem scharfen Kontrast aus dem silbernen Hintergrund (das fette silberne "X") wird später als treuhänderischer Marker für die spätere Massenspektrenerfassung im MALDI-Instrument dienen.
- Laden Sie den Schlitten in das MALDI-Gleitmetallziel. Verwenden Sie eine Kunststoffabdeckung und zeichnen / umreißen Sie, wo sich die Proben auf der Kunststoffabdeckung befinden. Verwerfen.
- Scannen Sie das Bild des Dias zusammen mit dem MALDI-Target mit einem Flachbettscanner. Die Schrauben auf der Oberfläche des Ziels dienen als Abstandhalter, um die Beschädigung oder Kontamination des Gewebeabschnitts durch den Scanner zu verhindern. Zeigen Sie eine Vorschau des ausgewählten Folienbereichs in 16-Bit-Graustufen und 2400 dpi an und scannen Sie ihn. Speichern Sie das Bild für die spätere Verwendung in MALDI MSI.
4. Matrixabscheidung
HINWEIS: Es gibt mehrere Methoden, um eine gleichmäßige Matrixschicht in feiner Kristallgröße auf den MALDI-Dia aufzutragen, einschließlich Sublimation, Tröpfchen-Inkjet-Druck, automatisches Matrixsprühgerät und manuelles Spray mit Künstler-Airbush9. Wir werden in diesem Protokoll ein automatisches Matrix-Sprayer als Beispiel für seine hohe Reproduzierbarkeit verwenden.
- Start: Schalten Sie die Matrix-Sprüheinheit ein, stellen Sie sicher, dass das Ventil bei LOAD positioniert ist, und starten Sie die Sprühsoftware. Überprüfen Sie, ob der Abluftventilator gut funktioniert. Starten Sie die Lösungsmittelpumpe nicht, wenn die aktive Entlüftung nicht ordnungsgemäß funktioniert.
- Bestätigen Sie auf der Registerkarte Comms, dass alles kommuniziert, und starten Sie dann die Lösungsmittelpumpe bei 0,1 ml/min mit einem Gegendruck von ~500 psi (oder 3,4 MPa).
- Starten Sie den Druckluftstrom zum Matrixsprühgerät, indem Sie den Stickstofftank auf 30 psi einstellen. Stellen Sie dann den Druckregler an der Vorderseite des Sprühgeräts auf 10 psi ein und stellen Sie die Temperatur der Sprühdüse wie gewünscht ein.
HINWEIS: Wenn der Luftdruck im Gebet niedriger als 5 psi ist, kann sich die Sprühdüse zum Schutz nicht erwärmen. - Wenn sich das Ventil noch in der LOAD-Position befindet, verwenden Sie eine Spritze, um den Kreislauf mit 7 ml 70% Methanol zu spülen, und füllen Sie dann den Kreislauf mit 6 ml Matrix.
- Legen Sie die Gewebeschieber in die Halter im Sprühgerät und klebten Sie beide Enden ab, um Bewegungen zu verhindern und eine matrixfreie Kante zu erhalten, um die Kontamination der Metallklemme auf dem MALDI-Target durch die Matrix zu vermeiden.
HINWEIS: Es wird dringend empfohlen, zuerst Matrixspray auf einem blanken Objektträger zu testen, bevor Sie mit wertvollen Probenobjektträgern fortfahren. - Überprüfen Sie, ob die Durchflussrate und die Temperatur stabil sind, um mit dem Sprühen zu beginnen.
- Wählen Sie die gewünschte Methode vorgetestet mit leerem Glasträger.
- Drücken Sie Start. Dadurch wird die Düsentemperatur eingestellt und der Pumpendurchfluss an die gewählte Methode angepasst. Schalten Sie das Ventil von Last in Sprühposition und bestätigen Sie dann, indem Sie auf Weiterklicken.
- Lassen Sie das System laufen, was je nach Methode 5-20 Minuten pro Schlitten dauert. Schalten Sie das Ventil von Spray in Load Position und bestätigen Sie dann, indem Sie auf Weiter klicken, wenn Sie fertig sind.
- Entfernen Sie die Objektträger aus dem Sprühgerät, untersuchen Sie das Muster der Matrixbeschichtung unter dem Mikroskop, um eine gleichmäßige Schicht aus feinem Matrixkristall zu gewährleisten.
- Nach der Matrixabscheidung legen Sie den/die Schlitten zur sofortigen Verwendung in den MALDI-Metallhalter. Wenn nur ein Probenobjektträger vorhanden ist, fügen Sie einen weiteren leeren Objektträger hinzu, um die beiden Felder des MALDI-Halters zu füllen.
- Reinigen Sie das System sofort nach dem Gebrauch gemäß den Anweisungen des Herstellers, um das Verstopfen der Sprühdüse zu verhindern.
- Nachdem der Probenträger mit Matrix beschichtet wurde, fahren Sie entweder sofort mit dem MALDI-Bildgebungsinstrument fort oder versenden Sie es mit Trockeneis unter Verwendung der gleichen Doppelreißverschlussbeutelvorbereitung, die in Schritt 2.19 beschrieben wurde.
HINWEIS: Unter Notfallbedingungen, wie z. B. wenn das MALDI-Instrument nicht für den sofortigen Gebrauch verfügbar ist, lagern Sie den beschichteten Objektträger im Vakuumzustand oder bei -20 ° C für bis zu 24 Stunden, obwohl die Verschlechterung einiger Metaboliten während der Lagerung auftreten kann, die nicht gründlich untersucht wurde.
Ergebnisse
Das repräsentative Experiment wurde gemäß dem in Abbildung 1gezeigten Workflow durchgeführt. Die entwicklungsförderlichen C57BL Wildtyp-Mausgehirne des postnatalen Tages 1, 21, 60 (erwachsene) wurden wie oben beschrieben nach CUNY IACUC-Richtlinien geerntet und für 2, 5 bzw. 7 Minuten auf einem Aluminiumboot, das auf flüssigem Stickstoff schwimmt, eingefroren. Die gefrorenen Gewebe wurden bei 10 μm dicken Abschnitten bei −15 °C kryossektioniert, die sowohl für den Probenkopf als auch für die Kammer eingestellt waren. Die Gewebeschreibeschnitte wurden dann schonend auf die vorgekühlte leitfähige Seite von ITO-beschichteten Glasobjektträgern für die MALDI-Bildgebung übertragen. Montierte Kryosektionen auf ITO-Objektträgern wurden 45 min bei Raumtemperatur im Vakuum ausgetrocknet, gefolgt von der Matrixabscheidung mit einem automatischen Matrixsprühgerät. Matrix NEDC wurde verwendet, um Metaboliten nachzuweisen, und eine Matrixlösung von 10 mg/ml in Methanol/Wasser (70/30, v/v) wurde mit einer Durchflussrate von 0,1 ml/min und einer Düsentemperatur von 75 °C für 12 Zyklen mit 5 s Trocknung zwischen jedem Zyklus abgeschieden. Es wurde eine Sprühgeschwindigkeit von 1300 mm/min, ein Spurabstand von 2 mm, ein N2-Gasdruck von 10 psi und eine Durchflussrate von 3 L/min und eine Düsenhöhe von 40 mm verwendet.
MALDI-Massenspektren wurden im negativen Ionenmodus mit dem MALDI Time of Flight (TOF) MSI-Instrument aufgenommen. 0,5-1 μL rote Phosphoremulsion (Pn-Cluster mit n = 1 - 90) Emulsion in Methanol wurden auf den ITO-Objektträgern neben den montierten Geweben abgeschieden und verwendet, um das Instrument im Massenbereich von 100 - 1000 m/ z durch Anwendung der quadratischen Kalibrierkurve13zu kalibrieren. Die Laserspotdurchmesser wurden auf das modulierte Strahlprofil "Medium" für 50 μm Rasterbreite fokussiert. Spektren im Massenbereich von m/z 50 bis 1000 wurden bei 1000 Hz für 500 Aufnahmen aufgenommen. Massenspektrendaten wurden aufgezeichnet und die Bildgebung wurde mit der fortschrittlichen MALDI MSI-Datenanalysesoftware weiter analysiert. Ionenbilder wurden mit RMS-Normalisierung (Root-Mean Square) bei einer Behälterbreite von ±0,25 Da erzeugt.
Die Ergebnisse in Abbildung 2 zeigen Ausgangsbilder der MALDI MSI-Datenanalysesoftware von m/z-Spektren, die aus jedem 100-Dalton-Intervall ausgewählt wurden, und zeigen deutlich den Nutzen für die Identifizierung von Spektren von niedermolekularen Metaboliten bis hin zu hochmolekularen Lipiden. Jede Zeile zeigt die jeweiligen Ionenwärmekarten, die sowohl räumliche als auch spektrale Informationen einer bestimmten Metabolitenspezies über drei Gewebe enthalten, die am postnatalen Tag 1, 21 und 60 gesammelt wurden. Für jedes repräsentative m/z kann die Analyse der regionalen Verteilung und Ionenhäufigkeiten verwendet werden, um relative Mengen entsprechender Arten zwischen verschiedenen Altersgruppen zu vergleichen. Eine Stärke der MALDI MSI-Methodik ist die Fähigkeit, die Spezifität bestimmter von m/z identifizierter Arten auf Entwicklungsmeilensteine oder spezifische anatomische Strukturen zu erkennen. Es wird beobachtet, dass einige Metaboliten bei P1-Neugeborenen angereichert (m/z 320.1), bei P60-Erwachsenen angereichert (m/z 846.5) oder gleichmäßig über Dasalter verteilt sind (m/z 480.3); andere molekulare Spezies sind spezifisch angereichert mit grauer Substanz (m/z 117.1; 524.3; 765.1), weißer Substanz (m/z 673.4; 846.5) oder CSF/Ventrikeln (m/z 239.0)(Abbildung 2A). Die räumliche Verteilung repräsentativer Metaboliten einschließlich Hypoxanthin (m/z 135,0), N-Acetyl-L-Asparaginsäure (m/z 174,0), Arachidonsäure (m/z 303,2) und mehrere Lipide wie Lysophosphatidylethanolamin LPE (18:1) (m/z 478,3), LPE(20:4) (m/z 500,3), LPE (22:6) (m/z 524,3), Phosphatidylethanolamin PE (44:10) (m/z 838,5), Phosphatidylinositol PI(38:4) (m/z 885,6) sind ebenfalls dargestellt (Abbildung 2B).
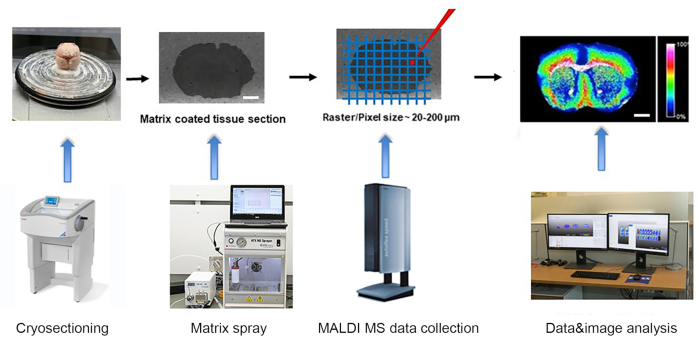
Abbildung 1. Workflow der MALDI- Time of Flight (TOF) Massenspektrometrie-Bildgebung. Das schnappgefrorene Gewebe wird im Kryostaten kryrosektioniert und auf einem ITO-Objektträger montiert. → Der Objektträger mit Gewebeschnitten wird mit einer feinen Matrixschicht mit einem automatischen Matrixsprüher beschichtet. → Massenspektren werden vom MALDI-TOF MSI-Instrument an einem Raster von 20-200um gesammelt. → Die Daten werden analysiert und die Bilder werden mit der fortschrittlichen MALDI MSI-Datenanalysesoftware generiert. Maßstabsleiste: 2 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
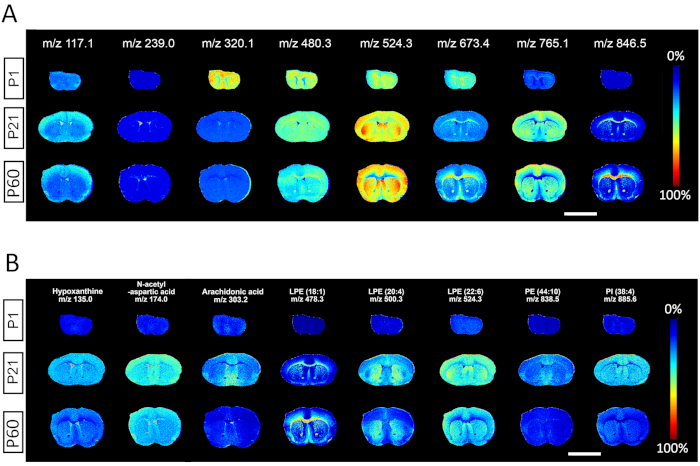
Abbildung 2. Repräsentativer Output mit ausgewählten m/z-Spektren aus der Massenspektrometrie bei 50 μm lateraler Auflösung. (A) Eine Heatmap zeigt die räumliche Verteilung spezifischer Metabolitenspezies, die aus jedem 100-Dalton-m/z-Intervall über die drei bei P1, P21 und P60 identifizierten Entwicklungsmeilensteine ausgewählt wurden. (B) Die räumliche Verteilung repräsentativer Metaboliten bei P1, P21 und P60, einschließlich: Hypoxanthin, N-Acetyl-L-Asparaginsäure, Arachidonsäure und verschiedene Lipidspezies wie Lysophosphatidylethanolamin (LPE), Phosphatidylethanolamin (PE), Phosphatidylinositol (PI). Maßstabsleiste, 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
MALDI-Imaging (MALDI-MSI) ist eine markierungsfreie Bildgebungstechnik, mit der Forscher die Verteilung verschiedener Biomoleküle und deren Modifikationen im Gewebe, der molekularen Grundlage der Pathologie, untersuchen können. Die kombinierte Verwendung von MALDI-MSI mit traditionellen LC-MS-Ansätzen für die Gewebeanalyse bietet die gleiche molekulare Tiefe wie herkömmliche Omics-Workflows, behält aber auch die räumliche Beziehung dieser Signale innerhalb des Mobilfunknetzes bei. Die Probenvorbereitung ist der kritischste Schritt in MALDI MSI und berücksichtigt die Variation der endgültigen Auslesungen von Metabolomik-Studien, die in verschiedenen Labors durchgeführt wurden4. Hier bieten wir ein umfassendes, aber praktisches Protokoll zur Standardisierung der Probenvorbereitung für die metabolomische Profilierung mit MALDI MSI, in der Hoffnung, dass es einer breiten Forschungsgemeinschaft zugute kommt, MALDI MSI in ihrer aktuellen und zukünftigen Forschung von der Grundlagenbiologie bis hin zu translationalen Studien zu implementieren.
Man muss immer alle Vorkehrungen beachten, um Veränderungen der molekularen Profile (sowohl Häufigkeiten als auch räumliche Verteilung) während der Probenvorbereitung zu minimieren und Kontaminationen zu vermeiden. Erstens, minimieren Sie die Zeit zwischen tierischer Euthanasie und Gewebeentnahme, wie z.B. in situ eingefroren oder durch Mikrowellenfixierung erhitzt, um Enzyme im Gehirn zu inaktivieren, um die Haftung für postmortale Ischämie zu reduzieren4,5,6. Zweitens ist der Schnappgefrierzustand der Probe kritisch. Unzureichendes Einfrieren führt zum Abbau und Verlust der Metaboliten, während ein Überfrieren zu einer Gewebefragmentierung während der Kryosektion führt. Die Gefrierzeit sollte immer zuerst nach früheren berichteten Studien getestet werden, und die in diesem Artikel vorgestellte Studie des Entwicklungsgehirns von Mäusen liefert die Referenzpunkte für nagetieriges Hirngewebe. Drittens erfordert das Schneiden von biologischem Gewebe und das Übertragen ihrer Abschnitte auf die ITO-Objektträger angemessene Übung. Es ist wichtig zu beachten, dass beim Verwenden des Pinsels, um den Abschnitt von der Bühne aufzunehmen, der Pinsel vorsichtig verwendet werden sollte. Lassen Sie die Borsten der Bürste nur mit den Rändern des Gewebeabschnitts in Kontakt kommen, um das Risiko einer Kontamination und Abschnittsfragmentierung zu verringern. Flachen Sie den Abschnitt auf dem Dia so weit wie möglich ab, dies verhindert das Kräuseln des Abschnitts während der Fingererwärmung. Viertens, stellen Sie bei der Montage auf dem ITO-Objektträger sicher, dass der gesamte Abschnitt gut mit dem ITO-Objektträger verbunden ist, da verschiedene Regionen des Gewebes eine unterschiedliche Zeit der Fingererwärmung erfordern können. Zum Beispiel benötigt Hirntumorgewebe eine längere Aufwärmzeit als normales Hirngewebe. Eine schlechte Montage kann zu einer Ablösung und Fragmentierung des Gewebes während des MALDI-MS-Scans führen. Denken Sie daran, dass die Fingererwärmung die Enzymwirkung und den Stoffwechsel ermöglichen kann, was zu künstlichen Veränderungen der Metaboliten führt. Fünftens spielt eine feine und gleichmäßige Abscheidung der MALDI-Matrix eine wichtige Rolle bei der Erzielung genauer räumlicher Informationen und eines starken MALDI-MS-Signals. Es wird empfohlen, das Matrixsprühen auf einem blinden Objektträger zu testen und das Kristallmuster unter einem Mikroskop zu beobachten, um die ordnungsgemäße Abdeckung zu überprüfen, bevor Sie mit der Beschichtung des wertvollen Probenobjektträgers fortfahren. Und schließlich, da ein einzelner Forscher die Gewebeentnahme und die Objektträgervorbereitung mit unterschiedlichen Geschwindigkeiten durchführt, wäre es ideal, eine Person zu haben, die die Probenvorbereitung für die Probe in derselben Kohorte übernimmt, um die Variation zu minimieren.
Das oben bereitgestellte Protokoll beschreibt Standardverfahren, die auf die Bedürfnisse bestimmter Experimente zugeschnitten werden können. Zum Beispiel kann ein Kryo-Schnittgel OCT, das normalerweise beim Kryo-Schnitten der Probe verwendet wird, weiter als Montagekleber auf dem Gewebefutter verwendet werden (wie in der oben beschriebenen Studie). Frühere Studien haben gezeigt, dass die Polymerkomponente in OCT eine starke Ionenunterdrückung verursacht14. Das Einbetten der Probe kann jedoch unvermeidlich sein, wenn das Gewebe zu zerbrechlich ist, um ohne zusätzliche Unterstützung durch ein Polymergel geschnitten zu werden. Um die Signalunterdrückung in diesen Fällen zu bekämpfen, müssen die Gewebe möglicherweise mit seriellem Waschen in 70% Ethanol und 95% Ethanol gewaschen werden, um Rest-OCT für den Nachweis von Proteinen oder Lipiden zu entfernen, während das Waschen für den Nachweis kleiner molekularer Metaboliten nicht empfohlen wird9.
MALDI MSI hat sowohl im Forschungslabor als auch in der klinischen Praxis zunehmend an Relevanz gewonnen. Zum Beispiel hat sich MALDI MSI kürzlich in Studien der Proteomik als nützlich erwiesen, um den phänotypischen Funktionsstatus eines Organismus zu charakterisieren15, und als Mittel zur mikrobiellen Identifizierung und Diagnose von Folgeerkrankungen16. Während MALDI MSI eine breite Palette von Anwendungen unterstützt, gibt es einige Einschränkungen, die damit verbunden sind, sich ausschließlich auf diese Technik zu verlassen, insbesondere wenn es um die Unterscheidung zwischen ähnlichen Spezies oder Metaboliten und die Identifizierung spezifischer Ziele geht. Eine weitere Herausforderung ist die Quantifizierung der Metabolitenkonzentration nach MALDI MSI-Signalen. Es wird oft angenommen, dass Ionenhäufigkeiten in MALDI MSI-Spektren und die räumliche Verteilung (oder relative Häufigkeiten) entsprechender molekularer Spezies über sezierte Gewebe gut korreliert sind. Man sollte jedoch immer bedenken, dass die Beziehung zwischen ionenintensität und der Menge der entsprechenden molekularen Spezies durch zahlreiche Faktoren kompliziert wird, einschließlich, aber nicht beschränkt auf Auswirkungen der Ionenunterdrückung, Veränderungen der Gewebestruktur und Ionenmolekülreaktionen17. Techniken, die interne Standards nutzen, können zur absoluten Quantifizierung (μmol/g Gewebe) in MALDI-MSI18implementiert werden. Diese beiden Herausforderungen werden typischerweise mit dem kombinierten Workflow von MALDI MSI mit Flüssigkeitschromatographie-Tandem-MS(LC-MS/MS)-Techniken angegangen, wobei MALDI-MS die Kartierung der interessierenden Region ermöglicht, die später einer Mikroextraktion und LC-MS/MS unterzogen wird, um mehr Informationen zur Identifizierung des Metaboliten zu liefern19.
MS-basierte Bildgebungsverfahren wurden in den letzten Jahren als alternative Modalität zu früheren Techniken zur Abbildung niedermolekularer Metaboliten entwickelt. Mit den Fortschritten und der wachsenden Popularität von MALDI MSI wird erwartet, dass die MALDI-Bildgebung zu einem neuen Standardwerkzeug für die Visualisierung kleiner Moleküle wird. Von besonderem Interesse sind die Bildgebung von Lipiden und körpereigenen kleinen Molekülen (z.B. Neurotransmitter und Metaboliten) im biologischen Kontext sowie die Bildgebung von Xenobiotika für die Entwicklung neuer pharmazeutischer Wirkstoffe. Es wird erwartet, dass diese drei Bereiche in naher Zukunft schnelle Fortschritte mit der Anwendung von MALDI MSI machen werden20.
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen.
Danksagungen
Ye He und Rinat Abzalimov werden vom Professional Staff Congress-City University of New York (PSC-CUNY) Research Award Program unterstützt. Yuki Chen und Kelly Veerasammy werden vom CuNY Summer Undergraduate Research Program der Alfred P. Sloan Foundation unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Andwin Scientific Tissue-Tek CRYO-OCT Compound | Fisher Scientific | 14-373-65 | |
| Artist brush MSC #5 1/8 X 9/16 TRIM RED SABLE | Fisher Scientific | 50-111-2302 | |
| Autoflex speed MALDI-TOF MS system | Bruker Daltonics Inc | MALDI-TOF MS instrument | |
| BD Syringe with Luer-Lok Tips | Fisher Scientific | 14-823-16E | |
| BD Vacutainer General Use Syringe Needles | Fisher Scientific | 23-021-020 | |
| Bruker Daltonics GLASS SLIDES MALDI IMAGNG | Fisher Scientific | NC0380464 | |
| Drierite, with indicator, 8 mesh, ACROS Organics | Fisher Scientific | AC219095000 | |
| Epson Perfection V600 Photo Scanner | Amazon | Perfection V600 | |
| Fisherbrand 5-Place Slide Mailer | Fisher Scientific | HS15986 | |
| Fisherbrand Digital Auto-Range Multimeter | Fisher Scientific | 01-241-1 | |
| FlexImaging v3.0 | Bruker Daltonics Inc | Bruker MS imaging analysis software | |
| HPLC Grade Methanol | Fisher Scientific | MMX04751 | |
| HPLC Grade Water | Fisher Scientific | W5-1 | |
| HTX M5 Sprayer | HTX Technologies, LLC | Automatic heated matrix sprayer | |
| Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers | Fisher Scientific | 06-666A | |
| MSC Ziploc Freezer Bag | Fisher Scientific | 50-111-3769 | |
| N -(1-Naphthyl) Ethylenediamine Dihydrochloride (NEDC) | Millipore Sigma Aldrich | 222488 | |
| SCiLS Lab (2015b) | SCiLS Lab | Advanced MALDI MSI data analysis software | |
| Thermo Scientific CryoStar NX50 Cryostat | Fisher Thermo Scientific | 95-713-0 | |
| Thermo Scientific Nalgene Transparent Polycarbonate Classic Design Desiccator | Fisher Scientific | 08-642-7 |
Referenzen
- Watkins, S. M., German, J. B. Metabolomics and biochemical profiling in drug discovery and development. Current Opinion in Molecular Therapeutics. 4, 224-228 (2002).
- Fernie, A. R., Trethewey, R. N., Krotzky, A. J., Willmitzer, L. Metabolite profiling: from diagnostics to systems biology. Nature Reviews Molecular Cell Biology. 5, 763-769 (2004).
- Theodoridis, G. A., Gika, H. G., Want, E. J., Wilson, I. D. Liquid chromatography-mass spectrometry based global metabolite profiling: a review. Analytica Chimica Acta. 711, 7-16 (2012).
- Dienel, G. A. Metabolomic and Imaging Mass Spectrometric Assays of Labile Brain Metabolites: Critical Importance of Brain Harvest Procedures. Neurochemistry Research. 45, 2586-2606 (2020).
- Dienel, G. A. Metabolomic Assays of Postmortem Brain Extracts: Pitfalls in Extrapolation of Concentrations of Glucose and Amino Acids to Metabolic Dysregulation In Vivo in Neurological Diseases. Neurochemistry Research. 44, 2239-2260 (2019).
- Wasek, B., Arning, E., Bottiglieri, T. The use of microwave irradiation for quantitative analysis of neurotransmitters in the mouse brain. Journal of Neuroscience Methods. 307, 188-193 (2018).
- Andres, D. A., et al. Improved workflow for mass spectrometry-based metabolomics analysis of the heart. Journal of Biological Chemistry. 295, 2676-2686 (2020).
- Lu, W., et al. Metabolite Measurement: Pitfalls to Avoid and Practices to Follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Norris, J. L., Caprioli, R. M. Analysis of tissue specimens by matrix-assisted laser desorption/ionization imaging mass spectrometry in biological and clinical research. Chemical Reviews. 113, 2309-2342 (2013).
- Miura, D., et al. Ultrahighly sensitive in situ metabolomic imaging for visualizing spatiotemporal metabolic behaviors. Analytical Chemistry. 82, 9789-9796 (2010).
- Han, J., et al. Towards high-throughput metabolomics using ultrahigh-field Fourier transform ion cyclotron resonance mass spectrometry. Metabolomics. 4, 128-140 (2008).
- Wang, J., et al. MALDI-TOF MS imaging of metabolites with a N-(1-naphthyl) ethylenediamine dihydrochloride matrix and its application to colorectal cancer liver metastasis. Analytical Chemistry. 87, 422-430 (2015).
- Sladkova, K., Houska, J., Havel, J. Laser desorption ionization of red phosphorus clusters and their use for mass calibration in time-of-flight mass spectrometry. Rapid Communication in Mass Spectrometry. 19, 3114-3118 (2019).
- Schwartz, S. A., Reyzer, M. L., Caprioli, R. M. Direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation. Journal of Mass Spectrometry. 38, 699-708 (2003).
- Greco, V., et al. Applications of MALDI-TOF mass spectrometry in clinical proteomics. Expert Review of Proteomics. 15, 683-696 (2018).
- Singhal, N., Kumar, M., Kanaujia, P. K., Virdi, J. S. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis. Frontiers in Microbiology. 6, 791 (2015).
- Hankin, J. A., Murphy, R. C. Relationship between MALDI IMS intensity and measured quantity of selected phospholipids in rat brain sections. Analytical Chemistry. 82 (20), 8476-8484 (2010).
- Prentice, B. M., Chumbley, C. W., Caprioli, R. M. Absolute Quantification of Rifampicin by MALDI Imaging Mass Spectrometry Using Multiple TOF/TOF Events in a Single Laser Shot. Journal of the American Society for Mass Spectrometry. 28 (1), 136-144 (2017).
- Quanico, J., Franck, J., Wisztorski, M., Salzet, M., Fournier, I. Combined MALDI Mass Spectrometry Imaging and Parafilm-Assisted Microdissection-Based LC-MS/MS Workflows in the Study of the Brain. Methods in Molecular Biology. 1598, 269-283 (2017).
- Trim, P. J., Snel, M. F. Small molecule MALDI MS imaging: Current technologies and future challenges. Methods. 104, 127-141 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten