Method Article
Preparação de amostras para perfil metabólico usando imagem de espectrometria de massa MALDI
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
O objetivo deste protocolo é fornecer orientação detalhada sobre a preparação da amostra ao planejar experimentos usando MALDI MSI para maximizar a detecção metabólica e molecular em amostras biológicas.
Resumo
Metabolômica, o estudo para identificar e quantificar pequenas moléculas e metabólitos presentes em uma amostra experimental, emergiu como uma importante ferramenta para investigar as atividades biológicas durante o desenvolvimento e doenças. As abordagens metabolômicas são amplamente utilizadas no estudo do câncer, nutrição/dieta, diabetes e outras condições fisiológicas e patológicas envolvendo processos metabólicos. Uma ferramenta vantajosa que auxilia no perfil metabolômico defendido neste artigo é a desorção/espectrometria de massa assistida por matriz /ionização (MALDI MSI). Sua capacidade de detectar metabólitos in situ sem rotulagem, modificações estruturais ou outros reagentes especializados, como os utilizados na imunossão, torna o MALDI MSI uma ferramenta única no avanço de metodologias relevantes no campo da metabolômica. Um processo adequado de preparação da amostra é fundamental para produzir resultados ideais e será o foco deste artigo.
Introdução
Metabólitos, intermediários ou produtos finais do metabolismo, incluindo nucleotídeos, amino ou ácidos orgânicos, lipídios, são componentes-chave para funções e processos biológicos. Metabolômica, o estudo dos metabólitos, permite a exploração de suas interações bioquímicas e a compreensão de seus papéis no contexto da pesquisa básica, translacional e clínica. Metabólitos estão fortemente associados aos fenótipos dos organismos e fornecem informações sobre atividades bioquímicas que ocorrem durante o metabolismo celular1. Portanto, além da genômica e da proteômica, a metabolômica surgiu como uma importante ferramenta na compreensão das condições fisiológicas e patológicas. Por exemplo, a metabolômica é usada para elucidar os mecanismos por trás das drogas existentes, bem como sua tolerância. No desenvolvimento de medicamentos, o metabolismo xenobiótico é útil na avaliação da atividade ou toxicidade de metabólitos entre espécies, o que mais tarde se traduz em apoiar a medicina personalizada2. Apesar da ampla aplicação da metabolômica, a imagem de metabólitos pode ser desafiadora devido à reatividade química dos metabólitos, heterogeneidade estrutural e ampla faixa de concentração3. No entanto, as concentrações de metabólitos labile, incluindo compostos de alta energia, glicose, lactato, glicóltico, pentose shunt pathway, e intermediários de ciclo TCA, fosfolipídios, neurotransmissores, compostos de sinalização, podem mudar em segundos e progredir ao longo de minutos quando enzimas teciduais estão ativas durante procedimentos de colheita de tecidos, como isquemia pós-morte na colheita cerebral4,5,6 . Para garantir a aquisição precisa de dados metabolômicos, a preparação adequada e cuidadosa da amostra é fundamental7,8. As plataformas estabelecidas atuais para medir metabólitos incluem RMN, ensaios enzimáticos e espectrometria de massa (incluindo cromatografia líquida e gasosa), a última das quais é discutida mais abaixo.
MALDI-MSI é uma técnica de última geração que permite a análise de amostras complexas através da detecção de espécies moleculares individuais. Maldi MSI concede o benefício de ser capaz de medir rapidamente e reprodutivelmente vários compostos moleculares em amostras biológicas. A imagem de espectrometria em massa permite ainda a produção de imagens que representam a biologia tecidual com base em seus metabólitos compostos, e o faz preservando a distribuição espacial dos metabólitos na amostra9. A capacidade do MALDI de detectar analitos em uma amostra sem o uso de rotulagem de anticorpos, modificações estruturais ou outros reagentes especializados, como os usados em imunossagens, juntamente com sua capacidade de monitorar centenas de moléculas dentro de um único experimento compreendem apenas algumas das vantagens que a imagem em MS concede quando se trata de perfil metabólico10, 11. Além de matriz comumente utilizada como ácido dihidroxibenómico 2,5 -DIhydroxybenzoic (DHB) e 9-aminoacridina (9-AA), recentemente descoberta nova matriz N -(1-Nafthil) Dihidrocloide de Etilladiamina (NEDC), que é bem adequado para análises de vários metabólitos de baixo peso molecular, melhorou ainda mais a aplicação do MALDI MSI no perfil metabólico12.
Apesar da ampla aplicação do MALDI MSI, o alto custo do instrumento e a complexidade do procedimento experimental impedem sua implementação mais ampla em laboratórios de pesquisa individuais. Portanto, a maioria dos estudos maldi MSI são suportados através de instalações de núcleo compartilhado. A preparação da amostra, incluindo preparação de slides e revestimento de matriz, é o passo mais crítico no MALDI MSI. No entanto, a preparação do slide é normalmente realizada no laboratório individual do pesquisador, o que cria potenciais variações na aquisição posterior do MSI maldi. Aqui, pretendemos fornecer um protocolo detalhado para a preparação amostral de amostras biológicas antes de prosseguir para as medições maldi MSI, e usar um perfil metabolômico do cérebro do rato de desenvolvimento como exemplo.
Protocolo
O protocolo segue as diretrizes do Centro de Pesquisa em Ciência Avançada (IACUC) da City University of New York (CUNY) Advanced Science Center (ASRC) institucional.
1. Colher o tecido
- Prepare o barco de alumínio. Prepare um retângulo de alumínio 10cm x 20 cm, dobre no meio para fazer 10 cm quadrado de camada dupla. Rotule as informações da amostra de um lado e dobre o outro lado para formar um barco com superfície inferior de cerca de 4 cm x 4 cm.
- Pré-covil do barco sobre o nitrogênio líquido (LN2) em uma caixa de isopor.
NOTA: Se o barco for muito pequeno, a amostra pode cair do barco e ficar congelada entrando em contato direto com a LN2, o que pode resultar em fragmentação durante a crioseção. - Eutanize o animal por deslocamento cervical seguindo as diretrizes institucionais da IACUC, disseca imediatamente o tecido de interesse.
- Mantenha o intervalo entre anestesia animal e congelamento de estalos o mais curto possível, para minimizar a alteração de metabólitos durante a colheita de tecidos, especialmente os metabólitos labiais no cérebro devido à isquemia pós-morte.
NOTA: A perfusão do animal com soro fisiológico tamponado de fosfato (PBS) aumentará a duração da isquemia, alterando ainda mais as concentrações de metabólitos labile e exagerando os resultados artefatos. Portanto, a perfusão ou lavagem do tecido com PBS não é recomendada, a menos que a contaminação do sangue ou fluido corporal esteja mais preocupada do que a deterioração ou lavagem de metabólitos para projeto individual.
- Mantenha o intervalo entre anestesia animal e congelamento de estalos o mais curto possível, para minimizar a alteração de metabólitos durante a colheita de tecidos, especialmente os metabólitos labiais no cérebro devido à isquemia pós-morte.
- Coloque imediatamente o tecido no barco de alumínio flutuando sobre nitrogênio líquido, feche a tampa da caixa de isopor e congele por 2-10 min dependendo do tamanho do tecido: 2 min, 5 min, 7 min para o dia pós-natal 1 (P1), P21, cérebro de rato P60, respectivamente, e 10 min para cérebro de rato P60.
NOTA: Não congele o tecido por tempo prolongado (por exemplo, 5 min para cérebro de rato P1) pois o excesso de congelado pode levar à fragmentação do tecido durante a criosecção. - Remova o barco por fórceps, dobre a folha para enrolar o tecido, transporte em gelo seco e armazene a -80 °C para uso posterior. Se seguido por secção imediata, leve as amostras em gelo seco para o criostat.
NOTA: Para preservar melhor os metabólitos, é preferível armazenar a amostra em tecido intacto e secioná-la logo antes de proceder à imagem MALDI. O tecido pode ser armazenado em -80 °C por até 24 meses.
2. Criosection o tecido
NOTA: Use luvas o tempo todo ao manusear os slides de tinóxido de índio (ITO). Evite respirar diretamente na lâmina ou usar máscaras (opcional) para evitar a contaminação da saliva humana na seção tecidual.
- Antes de seccionar o tecido, reúna o número desejado de lâminas de vidro revestidas de ITO compatíveis com MALDI.
- Teste a condutividade do slide usando um conjunto de voltímetros para resistência. Rotule o lado onde uma medição de resistência é lida: este será o lado a que as seções teciduais aderem. Coloque sempre o slide em uma toalha de papel limpa para evitar contaminação.
- Pré-esfrie os slides em um criostat definido para -20 °C.
- Se as amostras de tecido foram removidas do -80 °C, deixe o tecido equilibrar na câmara criostat por cerca de 45-60 min, dependendo do tamanho do tecido. Se as amostras estiverem se removendo do gelo seco, equilibre-a por cerca de 30 minutos.
- Limpe completamente o criostat com 70% de etanol. Pré-esfrie todas as ferramentas necessárias, incluindo pincel de artista de ponta fina e fórceps na câmara criostat.
- Ajuste a temperatura da câmara criostat e da cabeça da amostra de acordo com o tipo do tecido: -14 °C para fígado, -20 °C para músculo e -25 °C para pele9.
- Monte o tecido para o mandril usando tecido criográfico incorporando composto OCT, evitando OCT da região de interesse.
- Coloque uma lâmina limpa no palco e bloqueie. Ajuste a posição do estágio e o ângulo da amostra para alcançar o ângulo de corte desejado.
NOTA: Se diferentes tipos/genótipos de tecido forem cortados, certifique-se de reposicionar a amostra para que uma parte limpa da lâmina seja usada ou mudar para uma nova lâmina antes de cortar a próxima amostra para evitar contaminação cruzada. - Continue cortando até que a região de interesse (por exemplo, corpus calosum no cérebro) seja encontrada. Certifique-se de manter o palco limpo, escovando peças extras com um pincel de artista que foi equilibrado no criostat.
- Uma vez que a região desejada é revelada, corte seções menores de 10-12 μm de espessura. Se a seção tende a se desfazer ou desmoronar facilmente, aumente a temperatura do criostat, permanecendo na faixa de -22 °C a -11 °C. Descobrimos que a temperatura de corte ideal para o tecido cerebral é de -15 °C a - 18 °C.
- Uma vez que uma boa seção tenha sido cortada, adere ao slide ITO (opere na câmara criostat).
- Transfira uma seção do tecido para lâminas ITO usando a ponta da escova de artista.
- Aqueça a seção colocando um dedo sob o slide para aquecer a seção para garantir a montagem segura. A seção tecidual primeiro se transformará em transparente em 5-10 s e, em seguida, ficará opaca em cerca de 30-60 s.
- Coloque cuidadosamente o slide de lado no criostat.
- Repita passos para outras amostras de tecido, garantindo que cada seção do tecido seja colocada uniformemente na lâmina e esteja o mais alinhada possível.
- Uma vez que o alvo MALDI pode conter até dois slides, coloque as seções de várias amostras da mesma coorte em um único slide ou em dois slides, para garantir que eles possam ser analisados ao mesmo tempo.
- Quando terminar, coloque os slides da ITO em uma caixa de vácuo e transfira para um dessecante com dessecante. Seque o slide por 45-60 min.
NOTA: Se um desiccador de vácuo não estiver disponível no laboratório, mantenha os slides abaixo de -20 °C o tempo todo para evitar a deterioração dos metabólitos. - Armazenamento e envio de slides: a menos que a amostra seja preparada pelas instalações do núcleo de imagem MALDI para proceder diretamente à imagem MALDI, armazene os slides a -80 °C ou envie para instalações centrais ou outros laboratórios de pesquisa MALDI em gelo seco.
- Para melhor preservar as amostras, coloque os slides no transportador de slides, encha-os com nitrogênio (opcional), lacre com parafilm, coloque em um saco zip, que é então colocado em outro saco zip contendo dessecante. Rotule o saco zip externo.
- Prossiga para armazenamento a -80 °C (até 6 meses) ou transporte com gelo seco adequado.
3. Preparação matricial
- Prepare a matriz.
NOTA: Todos os reagentes devem ser de grau HPLC.- Prepare NEDC em uma concentração de 10 mg/mL. Prepare 10 mL de matriz dissolvida em um solvente de 70% de metanol (100 mg de NEDC, 7 mL de metanol, 3 mL de H2O).
- Além disso, prepare um adicional de 10 mL de 70% de solução de metanol:água para lavar o sistema de pulverizador antes de preencher a matriz.
- Uma vez que os slides estejam desidratados na etapa 2.17, coloque marcas "X" no espaço em branco do deslizamento de vidro fora das seções de tecido usando um marcador de prata de ponto ousado, e, em seguida, coloque um segundo "x" com um marcador preto de ponto fino em cima do "X" prata. O "X" preto com um contraste acentuado do fundo prateado (o prata ousado "X") mais tarde servirá como marcador fiduciário para a posterior aquisição de espectros em massa no instrumento MALDI.
- Carregue o slide no alvo de metal de slides MALDI. Use tampa plástica e desenhe/delineie onde as amostras estão na tampa de plástico. Reservar.
- Escaneie a imagem do slide juntamente com o alvo MALDI usando o scanner de cama plana. Os parafusos na superfície do alvo servirão como espaçador para evitar danos ou contaminação da seção tecidual pelo scanner. Visualize e escaneie a área de slides selecionada em escala de cinza de 16 bits e 2400 dpi. Salve a imagem para uso posterior no MSI MALDI.
4. Depoimento matricial
NOTA: Existem vários métodos para aplicar uma camada uniforme de matriz em tamanho de cristal fino no slide MALDI, incluindo sublimação, impressão de jato de tinta de gotícula, pulverizador de matriz automática e spray manual usando o artista airbush9. Usaremos o pulverizador de matriz automático como exemplo neste protocolo para sua alta reprodutibilidade.
- Inicie: Ligue a unidade do pulverizador de matriz, tendo certeza de que a válvula está posicionada no LOAD e inicie o software do pulverizador. Verifique se o ventilador de escape está funcionando bem. Não inicie a bomba de solvente se a ventilação ativa não estiver funcionando corretamente.
- Confirme na guia Comms que tudo está se comunicando e, em seguida, inicie a bomba de solvente a .1 mL/min, com uma pressão traseira de ~500 psi (ou 3,4 MPa).
- Inicie o fluxo de ar comprimido para o pulverizador de matriz, colocando o tanque de nitrogênio em 30 psi. Em seguida, ajuste o regulador de pressão na parte frontal do pulverizador para 10 psi, e ajuste a temperatura do bocal do pulverizador conforme desejado.
NOTA: Se a pressão de ar na oração for inferior a 5 psi, o bocal do pulverizador não será capaz de aquecer para proteção. - Com a válvula ainda na posição LOAD, use uma seringa para lavar o laço com 7 mL de 70% de metanol e, em seguida, encha o laço com 6 mL de matriz.
- Coloque os slides do tecido nos suportes do pulverizador, gravando as duas extremidades para evitar o movimento, e para preservar uma borda livre de matriz para evitar a contaminação do grampo metálico no alvo MALDI pela matriz.
NOTA: Testar o spray de matriz em um slide de microscópio em branco primeiro antes de proceder a slides de amostra preciosos é altamente recomendado. - Verifique se a vazão e a temperatura estão estáveis para começar a pulverizar.
- Selecione o método desejado pré-testado usando slide de vidro em branco.
- Pressione Start. Isso definirá a temperatura do bocal e ajustará a taxa de fluxo da bomba para corresponder ao método selecionado. Mude a válvula da posição Carregar para Spray e confirme clicando em Continuar.
- Permita que o sistema seja executado, o que levará de 5 a 20 minutos por slide, dependendo do método. Mude a válvula da posição Spray para Carregar e confirme clicando em Continuar quando terminar.
- Remova o slide(s) do pulverizador, examine o padrão de revestimento matricial sob microscópio para garantir uma camada uniforme de cristal de matriz fina.
- Após a deposição da matriz, coloque o slide(s) no suporte MALDI metálico para uso imediato. Se houver apenas um slide de amostra, adicione outro slide em branco para preencher os dois espaços do suporte MALDI.
- Limpe o sistema imediatamente após o uso seguindo as instruções do fabricante, para evitar o entupimento do bocal do pulverizador.
- Depois que o slide da amostra for revestido com matriz, proceda imediatamente ao instrumento de imagem MALDI, ou envie-o com gelo seco usando a mesma preparação de saco zip duplo descrito na etapa 2.19.
NOTA: Em circunstâncias emergenciais, como o instrumento MALDI não está disponível para uso imediato, armazene o slide revestido em condição de vácuo ou a -20 °C por até 24 horas, embora a deterioração de alguns metabólitos possa acontecer durante o armazenamento, o que não foi exaustivamente estudado.
Resultados
O experimento representativo foi realizado de acordo com o fluxo de trabalho mostrado na Figura 1. Os cérebros de camundongos de desenvolvimento C57BL do pós-natal dia 1, 21, 60 (adulto) foram colhidos conforme descrito acima seguindo as diretrizes do CUNY IACUC e foram congelados por 2, 5 e 7 minutos, respectivamente, em um barco de alumínio flutuando em nitrogênio líquido. Os tecidos congelados foram criosecionados a seções de espessura de 10 μm a -15 °C para a cabeça do espécime e para a câmara. As crioseções teciduais foram então gentilmente transferidas para o lado condutor pré-resfriado de lâminas de vidro revestidas de ITO para imagens MALDI. As crioseções montadas nos slides do ITO foram dessecadas no vácuo por 45 minutos à temperatura ambiente, seguidas pela deposição matricial usando pulverizador automático de matriz. Matrix NEDC foi utilizado para detectar metabólitos, e uma solução matricial de 10 mg/mL em metanol/água (70/30, v/v) foi depositada a uma taxa de fluxo de 0,1 mL/min e uma temperatura de bico de 75 °C para 12 ciclos com secagem de 5 s entre cada ciclo. Foi utilizada uma velocidade de pulverização de 1300 mm/min, espaçamento de 2 mm, pressão gasosa N2 de 10 psi e vazão de 3 L/min e altura do bocal de 40 mm.
Os espectros de massa MALDI foram adquiridos no modo íon negativo pelo instrumento MSI tempo de voo MALDI (TOF). 0,5-1 μL de fósforo vermelho (agrupamentos pn com n = 1 - 90) emulsão em metanol foi depositado nos slides do ITO, ao lado dos tecidos montados, e usado para calibrar o instrumento na faixa de massa de 100 - 1000 m/z, aplicando curva de calibração quadrática13. Os diâmetros da mancha de laser foram focados no perfil do feixe modulado "Médio" para largura de raster de 50 μm. Espectros dentro da faixa de massa de m/z 50 a 1000 foram adquiridos a 1000 Hz para 500 shots. Os dados de espectros em massa foram registrados, e a imagem foi ainda analisada utilizando-se de um software avançado de análise de dados MALDI MSI. As imagens de íon foram geradas com a normalização do quadrado de raiz (RMS) em uma largura de lixeira de ±0,25 Da.
Os resultados da Figura 2 mostram imagens de saída do software de análise de dados MALDI MSI de espectros m/z selecionados a cada intervalo de 100 Dalton, representando claramente a utilidade para identificação de espectros de metabólitos de moléculas pequenas a lipídios de alto peso molecular. Cada linha retrata os respectivos mapas de calor de íons contendo informações espaciais e espectrais de uma determinada espécie metabólica em três tecidos coletados nos dias 1, 21 e 60 pós-natal. Para cada representante m/z, a análise da distribuição regional e das abundâncias de íons pode ser utilizada para comparar quantidades relativas de espécies correspondentes entre diferentes idades. Uma força da metodologia MALDI MSI é a capacidade de discernir a especificidade de determinadas espécies identificadas por m/z para marcos de desenvolvimento ou estruturas anatômicas específicas. Alguns metabólitos são observados para serem enriquecidos em recém-nascidos P1 (m/z 320,1), enriquecidos em adultos P60 (m/z 846,5) ou distribuídos uniformemente entre idades (m/z 480,3); outras espécies moleculares são especificamente enriquecidas em matéria cinzenta (m/z 117,1; 524,3; 765,1), matéria branca (m/z 673,4; 846,5) ou CSF/ventrículos (m/z 239,0)(Figura 2A). A distribuição espacial de metabólitos representativos incluindo hipoxantina (m/z 135,0), ácido N-Acetil-L-aspartic (m/z 174.0), ácido aracidônico (m/z 303.2), e vários lipídios como lisofosphatidylethanolamina LPE (18:1) (m/z 478.3), LPE(2 00:4) (m/z 500.3), LPE (22:6) (m/z 524.3), fosfattidyletanolamina PE (44:6 10) (m/z 838,5), Fosphatidylinositol PI (38:4) (m/z 885,6) também são mostrados(Figura 2B).
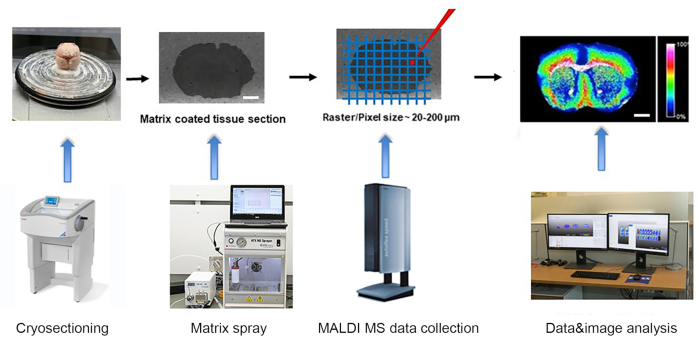
Figura 1. Fluxo de trabalho de imagens de espectrometria de massa MALDI-time of flight (TOF). O tecido congelado é criuado em criostat e montado no slide ITO. → O slide com seções de tecido é revestido com uma fina camada de matriz usando pulverizador de matriz automático. → espectros de massa é coletado pelo instrumento MALDI-TOF MSI em um raster de 20-200um. → Os dados são analisados e as imagens são geradas usando um software avançado de análise de dados MALDI MSI. Barra de escala: 2 mm. Clique aqui para ver uma versão maior desta figura.
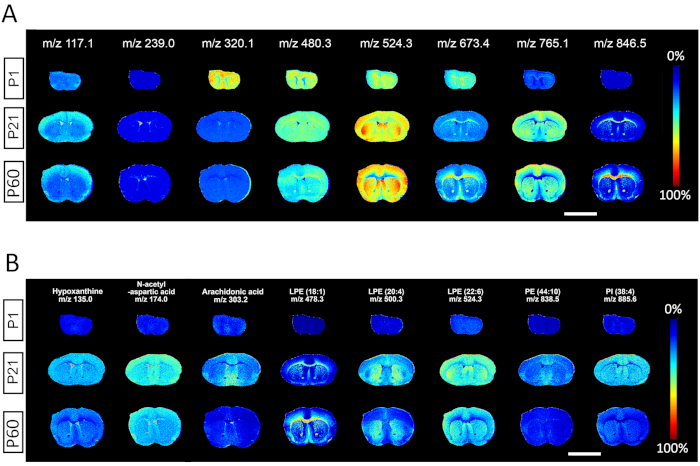
Figura 2. Saída representativa com espectrotrais m/z selecionados da espectrometria de massa adquirida a resolução lateral de 50 μm. (A) Um mapa de calor retrata a distribuição espacial de espécies metabólitos específicas selecionadas a cada intervalo de 100 Dalton m/z através dos três marcos de desenvolvimento identificados em P1, P21 e P60. (B) Distribuição espacial de metabólitos representativos em P1, P21 e P60, incluindo: hipoxantina, ácido N-Acetil-L-aspartic, ácido aracidonico e diferentes espécies lipídicas como lisofosofosphatidylethanolamina (LPE), fosfatidylethanolamina (PE), Fosfatidylinositol (PI). Barra de escala, 500 μm. Clique aqui para ver uma versão maior desta figura.
Discussão
MALDI-Imaging (MALDI-MSI) é uma técnica de imagem livre de rótulos que permite aos pesquisadores investigar a distribuição de várias biomoléculas e suas modificações no tecido, a base molecular da patologia. O uso combinado de MALDI-MSI com abordagens tradicionais de LC-MS para análise de tecidos fornece a mesma profundidade molecular que os fluxos de trabalho tradicionais de Omics, mas que também mantém a relação espacial desses sinais dentro da rede celular. A preparação da amostra é o passo mais crítico no MALDI MSI e explica a variação nas leituras finais de estudos metabolômicos realizados em diferentes laboratórios4. Aqui fornecemos um protocolo abrangente, porém prático, para padronizar a preparação da amostra para o perfil metabolômico usando o MALDI MSI, na esperança de que beneficie uma ampla comunidade de pesquisa para implementar o MALDI MSI em suas pesquisas atuais e futuras, desde a biologia básica até estudos translacionais.
Deve-se sempre ter em mente todas as precauções para minimizar as mudanças nos perfis moleculares (tanto abundâncias quanto distribuição espacial) durante as preparações amostrais e evitar contaminação. Em primeiro lugar, minimizar o tempo entre eutanásia animal e colheita de tecidos, como congelado in situ ou aquecido por fixação de micro-ondas para inativar enzimas no cérebro para reduzir a responsabilidade à isquemia pós-morte4,5,6. Em segundo lugar, a condição de congelamento da amostra é crítica. O congelamento inadequado causará a degradação e perda dos metabólitos, enquanto o congelamento excessivo levará à fragmentação tecidual durante a crioseção. O tempo de congelamento deve ser sempre testado primeiro de acordo com estudos relatados anteriormente, e o estudo do cérebro de camundongos de desenvolvimento apresentado neste artigo fornece os pontos de referência para o tecido cerebral de roedores. Em terceiro lugar, o corte de tecidos biológicos e a transferência de suas seções para os slides da OIT exigirão prática adequada. É importante notar que, ao usar o pincel para pegar a seção do palco, o pincel deve ser usado delicadamente. Permita que as cerdas da escova só entrem em contato com as bordas da seção tecidual para diminuir o risco de contaminação e fragmentação da seção. Achate a seção no slide o máximo possível, isso evitará o curling da seção durante o aquecimento dos dedos. Em quarto lugar, durante a montagem no slide ITO, certifique-se de que toda a seção esteja bem ligada ao slide da ITO, pois diferentes regiões do tecido podem exigir diferentes tempos de aquecimento dos dedos. Por exemplo, o tecido tumoral cerebral requer mais tempo de aquecimento do que o tecido cerebral normal. Uma montagem ruim pode levar ao desprendimento e fragmentação do tecido durante a varredura MALDI-MS. Tenha em mente que o aquecimento do dedo pode permitir a ação enzimada e o metabolismo causando alterações artefatosas de metabólitos. Em quinto lugar, uma deposição fina e uniforme da matriz MALDI desempenha um papel importante na obtenção de informações espaciais precisas e forte sinal MALDI-MS. Recomenda-se testar a matriz pulverizando em um slide em branco, e observar o padrão de cristal sob um microscópio para verificar a cobertura adequada, antes de prosseguir para o revestimento de precioso slide de espécime. E por último, uma vez que um pesquisador individual executa a colheita de tecidos e a preparação de slides em velocidades diferentes, seria ideal ter uma pessoa para lidar com a preparação da amostra para a amostra na mesma coorte, para minimizar a variação.
O protocolo fornecido acima detalha os procedimentos padrão, que podem ser adaptados para as necessidades de experimentos específicos. Por exemplo, um gel de crio secção OCT, que normalmente é usado na seção criobito da amostra, pode ser ainda mais utilizado como uma cola de montagem para o mandril de tecido (como no estudo descrito acima). Estudos anteriores mostraram que o componente polímero em OCT causa forte supressão deíons 14. No entanto, a incorporação da amostra pode ser inevitável nos casos em que o tecido é muito frágil para ser cortado sem suporte adicional de um gel de polímero. Para combater a supressão de sinal nesses casos, os tecidos podem precisar ser lavados com lavagem serial em 70% de etanol e 95% de etanol para remover OCT residuais para detecção de proteínas ou lipídios, enquanto a lavagem não é recomendada para a detecção de pequenos metabólitos moleculares9.
O MALDI MSI tem se tornado cada vez mais relevante tanto no laboratório de pesquisa quanto na prática clínica. Por exemplo, o MALDI MSI mostrou-se recentemente útil em estudos de proteômica, a fim de caracterizar o estado funcional fenotípico de um organismo15, e em atuar como agente de identificação e diagnóstico microbiano da doença subsequente16. Embora o MALDI MSI suporte uma ampla gama de aplicações, existem algumas limitações associadas a depender apenas dessa técnica, especialmente quando se trata de diferenciar entre espécies ou metabólitos semelhantes, e a identificação de alvos específicos. Outro desafio é a quantificação da concentração de metabólitos de acordo com os sinais maldi MSI. Presume-se frequentemente que as abundâncias de íons no espectro MALDI MSI e a distribuição espacial (ou abundâncias relativas) de espécies moleculares correspondentes através de tecidos dissecados estão bem correlacionadas. No entanto, deve-se sempre ter em mente que a relação entre a intensidade de íons e a quantidade de espécies moleculares correspondentes é complicada por inúmeros fatores, incluindo, mas não se limitando a, efeitos da supressão de íons, mudanças na estrutura tecidual e reações deion-molécula 17. Técnicas que fazem uso de padrões internos podem ser implementadas para quantificação absoluta (tecido μmol/g) no MALDI-MSI18. Esses dois desafios são tipicamente abordados com o fluxo de trabalho combinado do MALDI MSI com técnicas de cromatografia líquida tandem MS (LC-MS/MS), pelo qual o MALDI-MS permite o mapeamento da região de interesse, que é posteriormente submetida à microextração e LC-MS/MS para fornecer mais informações para identificar o metabólito19.
Os métodos de imagem baseados em MS têm sido desenvolvidos nos últimos anos como uma modalidade alternativa às técnicas anteriores para a imagem de metabólitos de pequenas moléculas. Com os avanços e a crescente popularidade do MALDI MSI, espera-se que a imagem MALDI se torne uma nova ferramenta padrão para visualizar pequenas moléculas. Imagens de pequenas moléculas lipídicas e endógenas (por exemplo, neurotransmissores e metabólitos) no contexto biológico, bem como imagens de xenobióticos para o desenvolvimento de novos agentes farmacêuticos são de particular interesse. Espera-se que essas três áreas tenham avanços rápidos com a aplicação do MALDI MSI no próximo futuro20.
Divulgações
Os autores não declaram interesses financeiros concorrentes.
Agradecimentos
Ye He e Rinat Abzalimov são apoiados pelo Programa de Prêmios de Pesquisa do Congresso da Cidade do Profissional da Universidade de Nova York (PSC-CUNY). Yuki Chen e Kelly Veerasammy são apoiados pelo Programa de Pesquisa de Graduação de Verão da Fundação Alfred P. Sloan CUNY.
Materiais
| Name | Company | Catalog Number | Comments |
| Andwin Scientific Tissue-Tek CRYO-OCT Compound | Fisher Scientific | 14-373-65 | |
| Artist brush MSC #5 1/8 X 9/16 TRIM RED SABLE | Fisher Scientific | 50-111-2302 | |
| Autoflex speed MALDI-TOF MS system | Bruker Daltonics Inc | MALDI-TOF MS instrument | |
| BD Syringe with Luer-Lok Tips | Fisher Scientific | 14-823-16E | |
| BD Vacutainer General Use Syringe Needles | Fisher Scientific | 23-021-020 | |
| Bruker Daltonics GLASS SLIDES MALDI IMAGNG | Fisher Scientific | NC0380464 | |
| Drierite, with indicator, 8 mesh, ACROS Organics | Fisher Scientific | AC219095000 | |
| Epson Perfection V600 Photo Scanner | Amazon | Perfection V600 | |
| Fisherbrand 5-Place Slide Mailer | Fisher Scientific | HS15986 | |
| Fisherbrand Digital Auto-Range Multimeter | Fisher Scientific | 01-241-1 | |
| FlexImaging v3.0 | Bruker Daltonics Inc | Bruker MS imaging analysis software | |
| HPLC Grade Methanol | Fisher Scientific | MMX04751 | |
| HPLC Grade Water | Fisher Scientific | W5-1 | |
| HTX M5 Sprayer | HTX Technologies, LLC | Automatic heated matrix sprayer | |
| Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers | Fisher Scientific | 06-666A | |
| MSC Ziploc Freezer Bag | Fisher Scientific | 50-111-3769 | |
| N -(1-Naphthyl) Ethylenediamine Dihydrochloride (NEDC) | Millipore Sigma Aldrich | 222488 | |
| SCiLS Lab (2015b) | SCiLS Lab | Advanced MALDI MSI data analysis software | |
| Thermo Scientific CryoStar NX50 Cryostat | Fisher Thermo Scientific | 95-713-0 | |
| Thermo Scientific Nalgene Transparent Polycarbonate Classic Design Desiccator | Fisher Scientific | 08-642-7 |
Referências
- Watkins, S. M., German, J. B. Metabolomics and biochemical profiling in drug discovery and development. Current Opinion in Molecular Therapeutics. 4, 224-228 (2002).
- Fernie, A. R., Trethewey, R. N., Krotzky, A. J., Willmitzer, L. Metabolite profiling: from diagnostics to systems biology. Nature Reviews Molecular Cell Biology. 5, 763-769 (2004).
- Theodoridis, G. A., Gika, H. G., Want, E. J., Wilson, I. D. Liquid chromatography-mass spectrometry based global metabolite profiling: a review. Analytica Chimica Acta. 711, 7-16 (2012).
- Dienel, G. A. Metabolomic and Imaging Mass Spectrometric Assays of Labile Brain Metabolites: Critical Importance of Brain Harvest Procedures. Neurochemistry Research. 45, 2586-2606 (2020).
- Dienel, G. A. Metabolomic Assays of Postmortem Brain Extracts: Pitfalls in Extrapolation of Concentrations of Glucose and Amino Acids to Metabolic Dysregulation In Vivo in Neurological Diseases. Neurochemistry Research. 44, 2239-2260 (2019).
- Wasek, B., Arning, E., Bottiglieri, T. The use of microwave irradiation for quantitative analysis of neurotransmitters in the mouse brain. Journal of Neuroscience Methods. 307, 188-193 (2018).
- Andres, D. A., et al. Improved workflow for mass spectrometry-based metabolomics analysis of the heart. Journal of Biological Chemistry. 295, 2676-2686 (2020).
- Lu, W., et al. Metabolite Measurement: Pitfalls to Avoid and Practices to Follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Norris, J. L., Caprioli, R. M. Analysis of tissue specimens by matrix-assisted laser desorption/ionization imaging mass spectrometry in biological and clinical research. Chemical Reviews. 113, 2309-2342 (2013).
- Miura, D., et al. Ultrahighly sensitive in situ metabolomic imaging for visualizing spatiotemporal metabolic behaviors. Analytical Chemistry. 82, 9789-9796 (2010).
- Han, J., et al. Towards high-throughput metabolomics using ultrahigh-field Fourier transform ion cyclotron resonance mass spectrometry. Metabolomics. 4, 128-140 (2008).
- Wang, J., et al. MALDI-TOF MS imaging of metabolites with a N-(1-naphthyl) ethylenediamine dihydrochloride matrix and its application to colorectal cancer liver metastasis. Analytical Chemistry. 87, 422-430 (2015).
- Sladkova, K., Houska, J., Havel, J. Laser desorption ionization of red phosphorus clusters and their use for mass calibration in time-of-flight mass spectrometry. Rapid Communication in Mass Spectrometry. 19, 3114-3118 (2019).
- Schwartz, S. A., Reyzer, M. L., Caprioli, R. M. Direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation. Journal of Mass Spectrometry. 38, 699-708 (2003).
- Greco, V., et al. Applications of MALDI-TOF mass spectrometry in clinical proteomics. Expert Review of Proteomics. 15, 683-696 (2018).
- Singhal, N., Kumar, M., Kanaujia, P. K., Virdi, J. S. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis. Frontiers in Microbiology. 6, 791 (2015).
- Hankin, J. A., Murphy, R. C. Relationship between MALDI IMS intensity and measured quantity of selected phospholipids in rat brain sections. Analytical Chemistry. 82 (20), 8476-8484 (2010).
- Prentice, B. M., Chumbley, C. W., Caprioli, R. M. Absolute Quantification of Rifampicin by MALDI Imaging Mass Spectrometry Using Multiple TOF/TOF Events in a Single Laser Shot. Journal of the American Society for Mass Spectrometry. 28 (1), 136-144 (2017).
- Quanico, J., Franck, J., Wisztorski, M., Salzet, M., Fournier, I. Combined MALDI Mass Spectrometry Imaging and Parafilm-Assisted Microdissection-Based LC-MS/MS Workflows in the Study of the Brain. Methods in Molecular Biology. 1598, 269-283 (2017).
- Trim, P. J., Snel, M. F. Small molecule MALDI MS imaging: Current technologies and future challenges. Methods. 104, 127-141 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados