Method Article
Preparazione del campione per la profilazione metabolica utilizzando la spettrometria di massa MALDI
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'obiettivo di questo protocollo è quello di fornire una guida dettagliata sulla preparazione del campione quando si pianificano esperimenti utilizzando MALDI MSI per massimizzare il rilevamento metabolico e molecolare in campioni biologici.
Abstract
La metabolomica, lo studio per identificare e quantificare piccole molecole e metaboliti presenti in un campione sperimentale, è emersa come uno strumento importante per indagare le attività biologiche durante lo sviluppo e le malattie. Gli approcci metabolomici sono ampiamente utilizzati nello studio del cancro, della nutrizione / dieta, del diabete e di altre condizioni fisiologiche e patologiche che coinvolgono i processi metabolici. Uno strumento vantaggioso che aiuta nella profilazione metabolomica sostenuta in questo articolo è il desorbimento laser assistito da matrice / spettrometria di massa a ionizzazione (MALDI MSI). La sua capacità di rilevare metaboliti in situ senza etichettatura, modifiche strutturali o altri reagenti specializzati, come quelli utilizzati nell'immuno colorazione, rende MALDI MSI uno strumento unico nell'avanzamento di metodologie rilevanti nel campo della metabolomica. Un processo di preparazione del campione appropriato è fondamentale per ottenere risultati ottimali e sarà al centro di questo documento.
Introduzione
I metaboliti, gli intermedi o i prodotti finali del metabolismo, compresi i nucleotidi, gli amminoacidi o gli acidi organici, i lipidi, sono componenti chiave delle funzioni e dei processi biologici. La metabolomica, lo studio dei metaboliti, consente l'esplorazione delle loro interazioni biochimiche e la comprensione dei loro ruoli nel contesto della ricerca di base, traslazionale e clinica. I metaboliti sono fortemente associati ai fenotipi degli organismi e forniscono informazioni sulle attività biochimiche che si verificano durante il metabolismo cellulare1. Pertanto, oltre alla genomica e alla proteomica, la metabolomica è emersa come uno strumento importante per comprendere sia le condizioni fisiologiche che patologiche. Ad esempio, la metabolomica viene utilizzata per chiarire i meccanismi alla base dei farmaci esistenti e la loro tolleranza. Nello sviluppo di farmaci, il metabolismo xenobiotico è utile per valutare l'attività o la tossicità dei metaboliti tra le specie, che in seguito si traduce nel sostenere la medicina personalizzata2. Nonostante l'ampia applicazione della metabolomica, l'imaging dei metaboliti può essere difficile a causa della reattività chimica dei metaboliti, dell'eterogeneità strutturale e dell'ampio intervallo di concentrazione3. Tuttavia, le concentrazioni di metaboliti labili tra cui composti ad alta energia, glucosio, lattato, glicolitico, via di shunt del pentoso e intermedi del ciclo TCA, fosfolipidi, neurotrasmettitori, composti di segnalazione, possono cambiare in pochi secondi e progredire in pochi minuti quando gli enzimi tissutali sono attivi durante le procedure di raccolta dei tessuti, come l'ischemia post mortem nella raccolta cerebrale4,5,6 . Per garantire un'accurata acquisizione dei dati metabolomici, è fondamentale un'adeguata e attenta preparazione del campione7,8. Le attuali piattaforme stabilite per la misurazione dei metaboliti includono NMR, saggi enzimatici e spettrometria di massa (compresa la cromatografia liquida e gassosa), l'ultima delle quali è discussa più avanti.
MALDI-MSI è una tecnica all'avanguardia che consente l'analisi di campioni complessi attraverso la rilevazione di singole specie molecolari. MALDI MSI garantisce il vantaggio di poter misurare in modo rapido e riproducibile vari composti molecolari in campioni biologici. L'imaging con spettrometria di massa consente inoltre la produzione di immagini che rappresentano la biologia tissutale basata sui suoi metaboliti compositi, e lo fa preservando la distribuzione spaziale dei metaboliti nel campione9. La capacità di MALDI di rilevare analiti in un campione senza l'uso di etichettatura anticorpale, modifiche strutturali o altri reagenti specializzati, come quelli utilizzati nell'immunostaining, insieme alla sua capacità di monitorare centinaia di molecole all'interno di un singolo esperimento comprendono solo alcuni dei vantaggi che l'imaging MS garantisce quando si tratta di profilazione metabolica10, 11. Oltre alla matrice comunemente usata come l'acido 2,5-diidrossibenzoico (DHB) e la 9-aminoacridina (9-AA), recentemente scoperta una nuova matrice N -(1-Naftil) Etilendiammina Dicloridrato (NEDC), che è adatta per l'analisi di vari metaboliti a basso peso molecolare, ha ulteriormente migliorato l'applicazione di MALDI MSI nel profilo metabolico12.
Nonostante l'ampia applicazione di MALDI MSI, l'elevato costo dello strumento e la complessità della procedura sperimentale ne impediscono una più ampia implementazione nei singoli laboratori di ricerca. Pertanto, la maggior parte degli studi MALDI MSI sono supportati attraverso strutture di base condivise. La preparazione del campione, compresa la preparazione del vetrino e il rivestimento della matrice, è la fase più critica in MALDI MSI. Tuttavia, la preparazione del vetrino viene normalmente eseguita nel laboratorio del singolo ricercatore, il che crea potenziali variazioni nella successiva acquisizione di MALDI MSI. Qui miriamo a fornire un protocollo dettagliato per la preparazione del campione di campioni biologici prima di procedere alle misurazioni MALDI MSI e utilizzare un profilo metabolomico del cervello del topo in via di sviluppo come esempio.
Protocollo
Il protocollo segue le linee guida del comitato istituzionale per la cura e l'uso degli animali (IACUC) dell'Advanced Science Research Center (ASRC) della City University of New York (CUNY).
1. Raccogli il tessuto
- Preparare la barca in alluminio. Preparare un rettangolo di alluminio 10cm x 20 cm, piegare al centro per fare 10 cm quadrato a doppio strato. Etichettare le informazioni del campione su un lato e piegare l'altro lato per formare una barca con una superficie inferiore di circa 4 cm x 4 cm.
- Preraffreddare la barca sull'azoto liquido (LN2) in una scatola di polistirolo.
NOTA: Se la barca è troppo piccola, il campione potrebbe cadere dalla barca e diventare troppo congelato contattando direttamente LN2, il che potrebbe causare frammentazione durante la criosezione. - Eutanasizzare l'animale per lussazione cervicale seguendo le linee guida istituzionali IACUC, sezionare immediatamente il tessuto di interesse.
- Mantenere l'intervallo tra l'anestesia animale e il congelamento a scatto il più breve possibile, per ridurre al minimo l'alterazione dei metaboliti durante la raccolta dei tessuti, in particolare i metaboliti labiali nel cervello a causa dell'ischemia post-mortem.
NOTA: La perfusione dell'animale con soluzione salina tamponata con fosfato (PBS) aumenterà la durata dell'ischemia, alterando ulteriormente le concentrazioni di metaboliti labili ed esagerando i risultati artefatti. Pertanto, la perfusione o il lavaggio del tessuto con PBS non è raccomandato, a meno che la contaminazione del sangue o del fluido corporeo non sia più interessata del deterioramento dei metaboliti o del washout per singolo progetto.
- Mantenere l'intervallo tra l'anestesia animale e il congelamento a scatto il più breve possibile, per ridurre al minimo l'alterazione dei metaboliti durante la raccolta dei tessuti, in particolare i metaboliti labiali nel cervello a causa dell'ischemia post-mortem.
- Posizionare immediatamente il tessuto nella barca di alluminio che galleggia su azoto liquido, chiudere il coperchio della scatola di polistirolo e congelare per 2-10 minuti a seconda delle dimensioni del tessuto: 2 minuti, 5 minuti, 7 minuti per il giorno 1 postnatale (P1), P21, cervello di topo P60, rispettivamente, e 10 minuti per il cervello di ratto P60.
NOTA: Non congelare il tessuto per un tempo prolungato (ad esempio, 5 minuti per il cervello del topo P1) poiché l'eccessivamente congelato potrebbe portare alla frammentazione dei tessuti durante la criosezione. - Rimuovere la barca con una pinze, piegare la lamina per avvolgere il tessuto, trasportare su ghiaccio secco e conservare a -80 °C per un uso successivo. Se seguito da un sezionamento immediato, portare i campioni su ghiaccio secco al criostato.
NOTA: Per preservare meglio i metaboliti, si preferisce conservare il campione in tessuto intatto e sezionarlo subito prima di procedere all'imaging MALDI. Il tessuto può essere conservato a -80 °C per un massimo di 24 mesi.
2. Criosezione del tessuto
NOTA: indossare guanti in ogni momento quando si maneggiano i vetrini di tinoxuro di indio (ITO). Evitare la respirazione diretta sul vetrino o indossare maschere (opzionali) per prevenire la contaminazione della saliva umana sulla sezione del tessuto.
- Prima di sezionare il tessuto, raccogliere il numero desiderato di vetrini rivestiti ITO compatibili MALDI.
- Testare la conduttività della slitta utilizzando un voltmetro impostato sulla resistenza. Etichettare il lato in cui viene letta una misurazione della resistenza: questo sarà il lato a cui aderiscono le sezioni di tessuto. Posizionare sempre la diapositiva su un tovagliolo di carta pulito per evitare contaminazioni.
- Pre-raffreddare i vetrini in un criostato impostato a -20 °C.
- Se i campioni di tessuto sono stati rimossi da -80 °C, lasciare che il tessuto si equilibra nella camera criostatica per circa 45-60 minuti a seconda delle dimensioni del tessuto. Se i campioni vengono rimossi dal ghiaccio secco, equilibrarlo per circa 30 minuti.
- Pulire accuratamente il criostato con il 70% di etanolo. Pre-raffreddare tutti gli strumenti necessari, tra cui pennello d'artista a punta sottile e pinna nella camera criostata.
- Impostare la temperatura della camera criostata e della testa del campione in base al tipo di tessuto: -14 °C per il fegato, -20 °C per il muscolo e -25 °C per la pelle9.
- Montare il tessuto sul mandrino utilizzando il composto di incorporamento criossutale OCT, evitando OCT dalla regione di interesse.
- Posizionare una lama pulita nel palco e bloccare. Regolare la posizione del palco e l'angolo del campione per ottenere l'angolo di taglio desiderato.
NOTA: se devono essere tagliati diversi tipi/genotipi di tessuto, assicurarsi di riposizionare il campione in modo da utilizzare una parte pulita della lama o passare a una nuova lama prima di tagliare il campione successivo per evitare contaminazioni incrociate. - Continuare a tagliare fino a quando non viene trovata la regione di interesse (ad esempio, il corpo calloso nel cervello). Assicurati di mantenere pulito il palco spazzolando via pezzi extra con un pennello d'artista che è stato bilanciato nel criostato.
- Una volta rivelata la regione desiderata, tagliare sezioni più piccole di 10-12 μm di spessore. Se la sezione tende a sfaldarsi o a sfaldarsi facilmente, aumentare la temperatura del criostato, rimanendo nell'intervallo da -22 °C a -11 °C. Abbiamo scoperto che la temperatura di taglio ottimale per il tessuto cerebrale è da -15 °C a - 18 °C.
- Una volta tagliata una buona sezione, aderisci alla slitta ITO (opera nella camera criostata).
- Trasferire una sezione del tessuto su vetrini ITO utilizzando la punta del pennello dell'artista.
- Riscaldare la sezione posizionando un dito sotto la diapositiva per riscaldare la sezione per garantire un montaggio sicuro. La sezione tissutale diventerà prima trasparente in 5-10 s e poi diventerà opaca in circa 30-60 s.
- Mettere da parte con attenzione la diapositiva nel criostato.
- Ripetere i passaggi per altri campioni di tessuto, assicurandosi che ogni sezione del tessuto sia posizionata uniformemente sul vetrino e sia il più allineata possibile.
- Poiché il target MALDI può contenere fino a due diapositive, posizionare le sezioni di più campioni della stessa coorte su una singola diapositiva o su due diapositive, per garantire che possano essere analizzate contemporaneamente.
- Al termine, posizionare le diapositive ITO in una scatola sottovuoto e trasferirle in un essiccatore con essiccante. Asciugare sottovuoto il vetrino per 45-60 min.
NOTA: Se un essiccatore sottovuoto non è disponibile in laboratorio, tenere i vetrini sotto i -20 °C per tutto il tempo per evitare il deterioramento dei metaboliti. - Stoccaggio e spedizione dei vetrini: a meno che il campione non sia preparato dalle strutture principali di imaging MALDI per procedere direttamente all'imaging MALDI, conservare i vetrini a -80 °C o spedirlo alle strutture principali o ad altri laboratori di ricerca MALDI su ghiaccio secco.
- Per preservare al meglio i campioni, posizionare i vetrini nel trasportatore di diapositive, riempirlo di azoto (opzionale), sigillare con parafilm, metterlo in un sacchetto con cerniera, che viene quindi inserito in un altro sacchetto con cerniera contenente essiccante. Etichettare il sacchetto esterno con zip.
- Procedere allo stoccaggio a -80 °C (fino a 6 mesi) o alla spedizione con ghiaccio secco adeguato.
3. Preparazione della matrice
- Preparare la matrice.
NOTA: Tutti i reagenti devono essere di grado HPLC.- Preparare NEDC ad una concentrazione di 10 mg/ml. Preparare 10 ml di matrice disciolta in un solvente al 70% di metanolo (100 mg di NEDC, 7 ml di metanolo, 3 ml di H2O).
- Preparare inoltre un extra di 10 ml di metanolo al 70%: soluzione acquosa per lavare il sistema di spruzzatura prima di riempire la matrice.
- Una volta disidratate le diapositive nel passaggio 2.17, posizionare i segni "X" sullo spazio vuoto del vetrino al di fuori delle sezioni di tessuto utilizzando un pennarello in argento a punta in grassetto, quindi posizionare una seconda "x" con un pennarello nero a punta fine sopra la "X" d'argento. La "X" nera con un netto contrasto fuori dallo sfondo argentato (la "X" in grassetto d'argento) servirà in seguito come marcatore fiduciario per la successiva acquisizione di spettri di massa nello strumento MALDI.
- Caricare la diapositiva nel target metallico della diapositiva MALDI. Usa una copertura di plastica e disegna / delinea dove i campioni si trovano sul coperchio di plastica. Accantonare.
- Scansiona l'immagine della diapositiva insieme al target MALDI utilizzando lo scanner piano. Le viti sulla superficie del bersaglio fungeranno da distanziatore per prevenire il danneggiamento o la contaminazione della sezione tissutale da parte dello scanner. Visualizza in anteprima ed esegui la scansione dell'area di scorrimento selezionata in scala di grigi a 16 bit e 2400 dpi. Salvare l'immagine per un uso successivo in MALDI MSI.
4. Deposizione della matrice
NOTA: Esistono diversi metodi per applicare uno strato uniforme di matrice in dimensioni di cristallo fine sulla diapositiva MALDI, tra cui sublimazione, stampa a getto d'inchiostro a goccia, spruzzatore automatico a matrice e spray manuale utilizzando artist airbush9. Useremo lo spruzzatore a matrice automatica come esempio in questo protocollo per la sua elevata riproducibilità.
- Avvio: accendere l'unità spruzzatrice a matrice, assicurandosi che la valvola sia posizionata a LOAD e avviare il software dello spruzzatore. Controllare che la ventola di scarico funzioni bene. Non avviare la pompa del solvente se lo sfiato attivo non funziona correttamente.
- Confermare nella scheda Comunicazione che tutto stia comunicando, quindi avviare la pompa del solvente a .1 mL / min, con una contropressione di ~ 500 psi (o 3,4 MPa).
- Avviare il flusso di aria compressa allo spruzzatore a matrice impostando il serbatoio dell'azoto a 30 psi. Quindi, regolare il regolatore di pressione sulla parte anteriore dello spruzzatore a 10 psi e impostare la temperatura dell'ugello dello spruzzatore come desiderato.
NOTA: Se la pressione dell'aria in preghiera è inferiore a 5 psi, l'ugello dello spruzzatore non sarà in grado di riscaldarsi per protezione. - Con la valvola ancora in posizione LOAD, utilizzare una siringa per lavare l'anello con 7 ml di metanolo al 70%, quindi riempire il ciclo con 6 mL di matrice.
- Posizionare i vetrini di tessuto nei supporti dello spruzzatore, nastrando entrambe le estremità per impedire il movimento e per preservare un bordo privo di matrice per evitare la contaminazione del morsetto metallico sul bersaglio MALDI da parte della matrice.
NOTA: Si consiglia vivamente di testare lo spray a matrice su un vetrino per microscopio bianco prima di procedere a preziosi vetrini campione. - Controllare che la portata e la temperatura siano stabili per iniziare la spruzzatura.
- Selezionare il metodo desiderato pre-testato utilizzando un vetrino vuoto.
- Premere Start. Questo regolerà la temperatura dell'ugello e regolerà la portata della pompa in modo che corrisponda al metodo selezionato. Commutare la valvola dalla posizione Carico a Quella Spruzzo, quindi confermare facendo clic su Continua.
- Consentire l'esecuzione del sistema, che richiederà 5-20 minuti per diapositiva a seconda del metodo. Spostare la valvola dalla posizione Spray alla posizione Load, quindi confermare facendo clic su Continua al termine.
- Rimuovere i vetrini dallo spruzzatore, esaminare il modello di rivestimento della matrice al microscopio per garantire uno strato uniforme di cristallo a matrice fine.
- Dopo la deposizione della matrice, posizionare le diapositive nel supporto MALDI metallico per un uso immediato. Se è presente un solo vetrino campione, aggiungere un'altra diapositiva vuota per riempire i due spazi del supporto MALDI.
- Pulire il sistema immediatamente dopo l'uso seguendo le istruzioni del produttore, per evitare l'intasamento dell'ugello dello spruzzatore.
- Dopo che il vetrino del campione è stato rivestito con matrice, procedere immediatamente allo strumento di imaging MALDI o spedirlo con ghiaccio secco utilizzando la stessa preparazione a doppia cerniera descritta nel passaggio 2.19.
NOTA: In circostanze di emergenza come lo strumento MALDI non è disponibile per l'uso immediato, conservare il vetrino rivestito sotto vuoto o a -20 °C per un massimo di 24 ore, anche se il deterioramento di alcuni metaboliti potrebbe verificarsi durante lo stoccaggio, che non è stato studiato a fondo.
Risultati
L'esperimento rappresentativo è stato eseguito in base al flusso di lavoro illustrato nella Figura 1. I cervelli di topo wildtype C57BL in via di sviluppo dei giorni postnatali 1, 21, 60 (adulti) sono stati raccolti come descritto sopra seguendo le linee guida CUNY IACUC e sono stati congelati a scatto per 2, 5 e 7 minuti, rispettivamente, su una barca di alluminio che galleggia su azoto liquido. I tessuti congelati sono stati criosezionati a sezioni di spessore di 10 μm a -15 °C impostate sia per la testa del campione che per la camera. Le criosezioni tissutali sono state quindi trasferite delicatamente sul lato conduttivo pre-raffreddato dei vetrini rivestiti itO per l'imaging MALDI. Le criosezioni montate su vetrini ITO sono state essiccate sotto vuoto per 45 minuti a temperatura ambiente, seguite dalla deposizione della matrice utilizzando lo spruzzatore automatico a matrice. La matrice NEDC è stata utilizzata per rilevare i metaboliti e una soluzione di matrice di 10 mg/mL in metanolo/acqua (70/30, v/v) è stata depositata a una portata di 0,1 ml/min e una temperatura dell'ugello di 75 °C per 12 cicli con 5 s di essiccazione tra ogni ciclo. È stata utilizzata una velocità di spruzzatura di 1300 mm/min, una spaziatura del binario di 2 mm,una pressione del gas N 2 di 10 psi e una portata di 3 L/min e un'altezza dell'ugello di 40 mm.
Gli spettri di massa MALDI sono stati acquisiti in modalità ioni negativi dallo strumento MALDI time of flight (TOF) MSI. 0,5-1 μL di emulsione di fosforo rosso (gruppi Pn con n = 1 - 90) in metanolo sono stati depositati sui vetrini ITO, accanto ai tessuti montati, e utilizzati per calibrare lo strumento nell'intervallo di massa 100 - 1000 m/z applicando la curva di calibrazione quadratica13. I diametri dello spot laser sono stati focalizzati su un profilo del fascio modulato "Medio" per una larghezza raster di 50 μm. Spettri all'interno della gamma di massa da m/ z 50 a 1000 sono stati acquisiti a 1000 Hz per 500 colpi. Sono stati registrati i dati degli spettri di massa e l'imaging è stato ulteriormente analizzato utilizzando il software avanzato di analisi dei dati MALDI MSI. Le immagini ioniche sono state generate con normalizzazione RMS (root-mean square) a una larghezza del bin di ±0,25 Da.
I risultati nella Figura 2 mostrano le immagini di output del software di analisi dei dati MALDI MSI di spettri m/z selezionati ogni intervallo di 100 Dalton, che descrivono chiaramente l'utilità per l'identificazione degli spettri dai metaboliti di piccole molecole ai lipidi ad alto peso molecolare. Ogni riga raffigura le rispettive mappe di calore ioiche contenenti informazioni spaziali e spettrali di una determinata specie di metaboliti attraverso tre tessuti raccolti ai giorni postnatali 1, 21 e 60. Per ogni m/z rappresentativo, l'analisi della distribuzione regionale e delle abbondanze di ioni può essere utilizzata per confrontare le quantità relative delle specie corrispondenti tra le diverse età. Un punto di forza della metodologia MALDI MSI è la capacità di discernere la specificità di alcune specie identificate da m/z a tappe dello sviluppo o a specifiche strutture anatomiche. Si osserva che alcuni metaboliti sono arricchiti nei neonati P1 (m/z 320,1), arricchiti negli adulti P60 (m/z 846,5) o distribuiti uniformemente tra le età (m/z 480,3); altre specie molecolari sono specificamente arricchite in materia grigia (m/z 117.1; 524.3; 765.1), sostanza bianca (m/z 673.4; 846.5) o CSF/ventricoli (m/z 239.0) (Figura 2A). Viene inoltre mostrata la distribuzione spaziale di metaboliti rappresentativi tra cui ipoxantina (m/z 135.0), acido N-acetil-L-aspartico (m/z 174.0), acido arachidonico (m/z 303.2) e diversi lipidi come lisofosfatidilethanolamine LPE (18:1) (m/z 478.3), LPE(20:4) (m/z 500.3), LPE (22:6) (m/z 524.3), fosfatidiletanolammina PE (44:10) (m/z 838.5), Fosfatidilinositolo PI(38:4) (m/z 885.6) (Figura 2B).
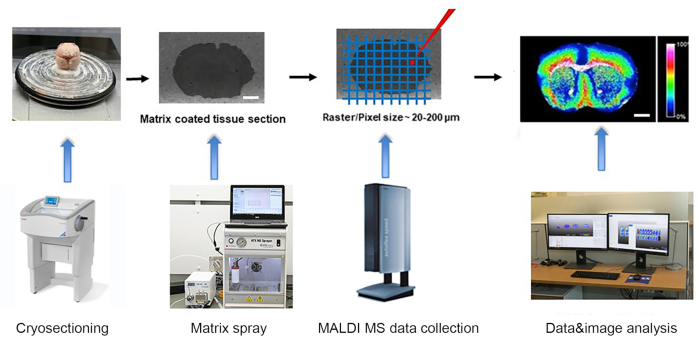
Figura 1. Flusso di lavoro dell'imaging della spettrometria di massa MALDI- time of flight (TOF). Il tessuto congelato a scatto viene criosciotto in criostato e montato su vetrino ITO. → Il vetrino con sezioni di tessuto è rivestito con un sottile strato di matrice utilizzando uno spruzzatore automatico a matrice. → spettri di massa sono raccolti dallo strumento MALDI-TOF MSI ad un raster di 20-200um. → I dati vengono analizzati e le immagini vengono generate utilizzando il software avanzato di analisi dei dati MALDI MSI. Barra della scala: 2 mm. Fare clic qui per visualizzare una versione più grande di questa figura.
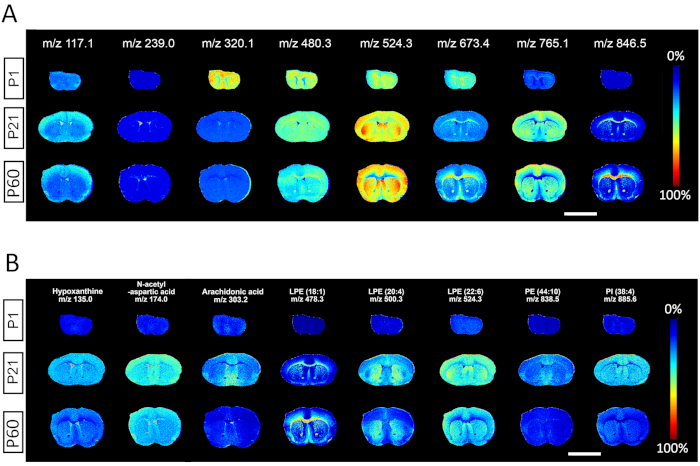
Figura 2. Uscita rappresentativa con spettri m/z selezionati dalla spettrometria di massa acquisita a risoluzione laterale di 50 μm. (A) Una mappa termica raffigura la distribuzione spaziale di specifiche specie di metaboliti selezionati da ogni intervallo di 100 Dalton m/z attraverso le tre tappe dello sviluppo identificate a P1, P21 e P60. (B) La distribuzione spaziale di metaboliti rappresentativi a P1, P21 e P60, tra cui: ipoxantina, acido N-acetil-L-aspartico, acido arachidonico e diverse specie lipidiche come lisofosfatidilethanolamine (LPE), fosfatidilfetanolammina (PE), fosfatidilinositolo (PI). Barra della scala, 500 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
MALDI-Imaging (MALDI-MSI) è una tecnica di imaging label-free che consente ai ricercatori di studiare la distribuzione di varie biomolecole e le loro modificazioni nei tessuti, la base molecolare della patologia. L'uso combinato di MALDI-MSI con i tradizionali approcci LC-MS per l'analisi dei tessuti fornisce la stessa profondità molecolare dei flussi di lavoro Omics tradizionali, ma che mantiene anche la relazione spaziale di tali segnali all'interno della rete cellulare. La preparazione del campione è la fase più critica in MALDI MSI e tiene conto della variazione delle lette finali degli studi di metabolomica condotti in diversi laboratori4. Qui forniamo un protocollo completo ma pratico per standardizzare la preparazione del campione per la profilazione metabolomica utilizzando MALDI MSI, nella speranza che sia vantaggioso per un'ampia comunità di ricerca implementare MALDI MSI nella loro ricerca attuale e futura dalla biologia di base agli studi traslazionali.
Bisogna sempre tenere a mente tutte le precauzioni per ridurre al minimo i cambiamenti nei profili molecolari (sia abbondanza che distribuzione spaziale) durante la preparazione del campione ed evitare la contaminazione. In primo luogo, ridurre al minimo il tempo tra l'eutanasia animale e la raccolta dei tessuti, come congelati in situ o riscaldati dalla fissazione a microonde per inattivare gli enzimi nel cervello per ridurre la responsabilità per l'ischemia post mortem4,5,6. In secondo luogo, la condizione di congelamento a scatto del campione è critica. Un congelamento inadeguato causerà la degradazione e la perdita dei metaboliti, mentre il congelamento eccessivo porterà alla frammentazione dei tessuti durante la criosezione. Il tempo di congelamento dovrebbe sempre essere testato prima secondo i precedenti studi riportati, e lo studio del cervello di topo dello sviluppo presentato in questo documento fornisce i punti di riferimento per il tessuto cerebrale dei roditori. In terzo luogo, il taglio dei tessuti biologici e il trasferimento delle loro sezioni ai vetrini ITO richiederanno una pratica adeguata. È importante notare che mentre si utilizza il pennello per raccogliere la sezione dal palco, il pennello deve essere usato delicatamente. Lasciare che le setole della spazzola entrino in contatto solo con i bordi della sezione tissutale per ridurre il rischio di contaminazione e frammentazione della sezione. Appiattire il più possibile la sezione sulla diapositiva, questo impedirà l'arricciamento della sezione durante il riscaldamento delle dita. In quarto luogo, durante il montaggio sulla diapositiva ITO, assicurarsi che l'intera sezione sia ben collegata alla diapositiva ITO poiché diverse regioni del tessuto potrebbero richiedere un tempo diverso di riscaldamento delle dita. Ad esempio, il tessuto tumorale cerebrale richiede un tempo di riscaldamento più lungo rispetto al normale tessuto cerebrale. Un montaggio scadente potrebbe portare al distacco e alla frammentazione del tessuto durante la scansione MALDI-MS. Tieni presente che il riscaldamento delle dita potrebbe consentire l'azione enzimatica e il metabolismo causando cambiamenti artefatti dei metaboliti. In quinto luogo, una deposizione fine e uniforme della matrice MALDI svolge un ruolo importante nel raggiungimento di informazioni spaziali accurate e di un forte segnale MALDI-MS. Si consiglia di testare la spruzzatura della matrice su un vetrino bianco e osservare il modello cristallino al microscopio per verificare la corretta copertura, prima di procedere al rivestimento del prezioso vetrino del campione. E infine, poiché un singolo ricercatore esegue la raccolta dei tessuti e la preparazione del vetrino a velocità diverse, sarebbe ideale avere una persona che gestisca la preparazione del campione per il campione nella stessa coorte, per ridurre al minimo la variazione.
Il protocollo fornito sopra dettaglia le procedure standard, che possono essere adattate alle esigenze di particolari esperimenti. Ad esempio, un OCT gel criosezionante, che viene normalmente utilizzato nella criosezione del campione, può essere ulteriormente utilizzato come colla di montaggio al mandrino del tessuto (come nello studio sopra descritto). Studi precedenti hanno dimostrato che il componente polimerico in OCT provoca una forte soppressione degli ioni14. Tuttavia, l'incorporamento del campione può essere inevitabile nei casi in cui il tessuto è troppo fragile per essere tagliato senza ulteriore supporto da un gel polimerico. Al fine di combattere la soppressione del segnale in questi casi, potrebbe essere necessario lavare i tessuti con lavaggio seriale in etanolo al 70% etanolo al 95% per rimuovere l'OCT residuo per la rilevazione di proteine o lipidi, mentre il lavaggio non è raccomandato per la rilevazione di piccoli metaboliti molecolari9.
MALDI MSI è diventato sempre più rilevante sia nel laboratorio di ricerca che nella pratica clinica. Ad esempio, MALDI MSI si è recentemente dimostrato utile negli studi di proteomica al fine di caratterizzare lo stato funzionale fenotipico di un organismo15e nell'agire come agente per l'identificazione microbica e la diagnosi della successiva malattia16. Mentre MALDI MSI supporta una vasta gamma di applicazioni, ci sono alcune limitazioni associate all'affidarsi esclusivamente a questa tecnica, specialmente quando si tratta di differenziare tra specie o metaboliti simili e l'identificazione di bersagli specifici. Un'altra sfida è la quantificazione della concentrazione dei metaboliti secondo i segnali MALDI MSI. Si presume spesso che le abbondanze di ioni negli spettri MALDI MSI e la distribuzione spaziale (o abbondanza relativa) delle corrispondenti specie molecolari tra i tessuti sezionati siano ben correlate. Tuttavia, si dovrebbe sempre tenere presente che la relazione tra l'intensità degli ioni e la quantità di specie molecolari corrispondenti è complicata da numerosi fattori tra cui, ma non solo, gli effetti della soppressione degli ioni, i cambiamenti nella struttura tissutale e le reazioni ione-molecola17. Le tecniche che fanno uso di standard interni possono essere implementate per la quantificazione assoluta (tessuto μmol/g) in MALDI-MSI18. Queste due sfide sono tipicamente affrontate con il flusso di lavoro combinato di MALDI MSI con tecniche di cromatografia liquida tandem MS (LC-MS / MS), in cui MALDI-MS consente la mappatura della regione di interesse, che viene successivamente sottoposta a microestrazione e LC-MS / MS per fornire maggiori informazioni per identificare il metabolita19.
Negli ultimi anni sono stati sviluppati metodi di imaging basati sulla SM come modalità alternativa alle tecniche precedenti per l'imaging di metaboliti di piccole molecole. Con i progressi e la crescente popolarità di MALDI MSI, si prevede che l'imaging MALDI diventerà un nuovo strumento standard per la visualizzazione di piccole molecole. Di particolare interesse sono l'imaging di piccole molecole lipidiche ed endogene (ad es. neurotrasmettitori e metaboliti) in ambito biologico, nonché l'imaging di xenobiotici per lo sviluppo di nuovi agenti farmaceutici. Queste tre aree dovrebbero avere rapidi progressi con l'applicazione di MALDI MSI nel prossimo futuro20.
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Ye He e Rinat Abzalimov sono supportati dal Professional Staff Congress-City University of New York (PSC-CUNY) Research Award Program. Yuki Chen e Kelly Veerasammy sono supportati dal programma di ricerca universitario estivo CUNY della Alfred P. Sloan Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Andwin Scientific Tissue-Tek CRYO-OCT Compound | Fisher Scientific | 14-373-65 | |
| Artist brush MSC #5 1/8 X 9/16 TRIM RED SABLE | Fisher Scientific | 50-111-2302 | |
| Autoflex speed MALDI-TOF MS system | Bruker Daltonics Inc | MALDI-TOF MS instrument | |
| BD Syringe with Luer-Lok Tips | Fisher Scientific | 14-823-16E | |
| BD Vacutainer General Use Syringe Needles | Fisher Scientific | 23-021-020 | |
| Bruker Daltonics GLASS SLIDES MALDI IMAGNG | Fisher Scientific | NC0380464 | |
| Drierite, with indicator, 8 mesh, ACROS Organics | Fisher Scientific | AC219095000 | |
| Epson Perfection V600 Photo Scanner | Amazon | Perfection V600 | |
| Fisherbrand 5-Place Slide Mailer | Fisher Scientific | HS15986 | |
| Fisherbrand Digital Auto-Range Multimeter | Fisher Scientific | 01-241-1 | |
| FlexImaging v3.0 | Bruker Daltonics Inc | Bruker MS imaging analysis software | |
| HPLC Grade Methanol | Fisher Scientific | MMX04751 | |
| HPLC Grade Water | Fisher Scientific | W5-1 | |
| HTX M5 Sprayer | HTX Technologies, LLC | Automatic heated matrix sprayer | |
| Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers | Fisher Scientific | 06-666A | |
| MSC Ziploc Freezer Bag | Fisher Scientific | 50-111-3769 | |
| N -(1-Naphthyl) Ethylenediamine Dihydrochloride (NEDC) | Millipore Sigma Aldrich | 222488 | |
| SCiLS Lab (2015b) | SCiLS Lab | Advanced MALDI MSI data analysis software | |
| Thermo Scientific CryoStar NX50 Cryostat | Fisher Thermo Scientific | 95-713-0 | |
| Thermo Scientific Nalgene Transparent Polycarbonate Classic Design Desiccator | Fisher Scientific | 08-642-7 |
Riferimenti
- Watkins, S. M., German, J. B. Metabolomics and biochemical profiling in drug discovery and development. Current Opinion in Molecular Therapeutics. 4, 224-228 (2002).
- Fernie, A. R., Trethewey, R. N., Krotzky, A. J., Willmitzer, L. Metabolite profiling: from diagnostics to systems biology. Nature Reviews Molecular Cell Biology. 5, 763-769 (2004).
- Theodoridis, G. A., Gika, H. G., Want, E. J., Wilson, I. D. Liquid chromatography-mass spectrometry based global metabolite profiling: a review. Analytica Chimica Acta. 711, 7-16 (2012).
- Dienel, G. A. Metabolomic and Imaging Mass Spectrometric Assays of Labile Brain Metabolites: Critical Importance of Brain Harvest Procedures. Neurochemistry Research. 45, 2586-2606 (2020).
- Dienel, G. A. Metabolomic Assays of Postmortem Brain Extracts: Pitfalls in Extrapolation of Concentrations of Glucose and Amino Acids to Metabolic Dysregulation In Vivo in Neurological Diseases. Neurochemistry Research. 44, 2239-2260 (2019).
- Wasek, B., Arning, E., Bottiglieri, T. The use of microwave irradiation for quantitative analysis of neurotransmitters in the mouse brain. Journal of Neuroscience Methods. 307, 188-193 (2018).
- Andres, D. A., et al. Improved workflow for mass spectrometry-based metabolomics analysis of the heart. Journal of Biological Chemistry. 295, 2676-2686 (2020).
- Lu, W., et al. Metabolite Measurement: Pitfalls to Avoid and Practices to Follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Norris, J. L., Caprioli, R. M. Analysis of tissue specimens by matrix-assisted laser desorption/ionization imaging mass spectrometry in biological and clinical research. Chemical Reviews. 113, 2309-2342 (2013).
- Miura, D., et al. Ultrahighly sensitive in situ metabolomic imaging for visualizing spatiotemporal metabolic behaviors. Analytical Chemistry. 82, 9789-9796 (2010).
- Han, J., et al. Towards high-throughput metabolomics using ultrahigh-field Fourier transform ion cyclotron resonance mass spectrometry. Metabolomics. 4, 128-140 (2008).
- Wang, J., et al. MALDI-TOF MS imaging of metabolites with a N-(1-naphthyl) ethylenediamine dihydrochloride matrix and its application to colorectal cancer liver metastasis. Analytical Chemistry. 87, 422-430 (2015).
- Sladkova, K., Houska, J., Havel, J. Laser desorption ionization of red phosphorus clusters and their use for mass calibration in time-of-flight mass spectrometry. Rapid Communication in Mass Spectrometry. 19, 3114-3118 (2019).
- Schwartz, S. A., Reyzer, M. L., Caprioli, R. M. Direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation. Journal of Mass Spectrometry. 38, 699-708 (2003).
- Greco, V., et al. Applications of MALDI-TOF mass spectrometry in clinical proteomics. Expert Review of Proteomics. 15, 683-696 (2018).
- Singhal, N., Kumar, M., Kanaujia, P. K., Virdi, J. S. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis. Frontiers in Microbiology. 6, 791 (2015).
- Hankin, J. A., Murphy, R. C. Relationship between MALDI IMS intensity and measured quantity of selected phospholipids in rat brain sections. Analytical Chemistry. 82 (20), 8476-8484 (2010).
- Prentice, B. M., Chumbley, C. W., Caprioli, R. M. Absolute Quantification of Rifampicin by MALDI Imaging Mass Spectrometry Using Multiple TOF/TOF Events in a Single Laser Shot. Journal of the American Society for Mass Spectrometry. 28 (1), 136-144 (2017).
- Quanico, J., Franck, J., Wisztorski, M., Salzet, M., Fournier, I. Combined MALDI Mass Spectrometry Imaging and Parafilm-Assisted Microdissection-Based LC-MS/MS Workflows in the Study of the Brain. Methods in Molecular Biology. 1598, 269-283 (2017).
- Trim, P. J., Snel, M. F. Small molecule MALDI MS imaging: Current technologies and future challenges. Methods. 104, 127-141 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon