Method Article
Effizientes Fragment-Screening in der XChem-Anlage an der Diamond Light Source
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt den kompletten XChem-Prozess für kristallbasiertes Fragment-Screening, beginnend mit der Beantragung des Zugangs und allen nachfolgenden Schritten bis hin zur Datenverbreitung.
Zusammenfassung
Bei der fragmentbasierten Wirkstoffforschung werden Hunderte oder oft Tausende von Verbindungen, die kleiner als ~300 Da sind, gegen das interessierende Protein getestet, um chemische Einheiten zu identifizieren, die zu potenten Wirkstoffkandidaten entwickelt werden können. Da die Verbindungen klein sind, sind die Wechselwirkungen schwach, und die Screening-Methode muss daher hochempfindlich sein. Darüber hinaus sind strukturelle Informationen in der Regel entscheidend, um diese Treffer in bleiähnliche Verbindungen umzuwandeln. Daher war die Proteinkristallographie schon immer ein Goldstandard, aber historisch gesehen zu schwierig, um eine breite Anwendung als primäres Screening zu finden.
Erste XChem-Experimente wurden 2014 demonstriert und dann mit akademischen und industriellen Mitarbeitern getestet, um den Prozess zu validieren. Seitdem haben ein großer Forschungsaufwand und eine beträchtliche Strahlzeit die Probenvorbereitung rationalisiert, eine Fragmentbibliothek mit schnellen Follow-up-Möglichkeiten entwickelt, die Fähigkeit der I04-1-Beamline für die unbeaufsichtigte Datenerfassung automatisiert und verbessert und neue Werkzeuge für das Datenmanagement, die Analyse und die Identifizierung von Treffern implementiert.
XChem ist jetzt eine Anlage für das Screening von kristallographischen Fragmenten in großem Maßstab, die den gesamten Prozess von der Kristallerzeugung bis zur Abscheidung unterstützt und für akademische und industrielle Anwender weltweit zugänglich ist. Das von Experten begutachtete akademische Nutzerprogramm wird seit 2016 aktiv weiterentwickelt, um Projekte mit einem möglichst breiten wissenschaftlichen Spektrum zu unterstützen, einschließlich gut validierter und explorativer Projekte. Der akademische Zugang wird durch halbjährliche Aufrufe zur Einreichung von Peer-Review-Vorschlägen vergeben, und proprietäre Arbeiten werden von der Industrial Liaison Group von Diamond arrangiert. Dieser Workflow wurde bereits routinemäßig auf über hundert Targets aus verschiedenen therapeutischen Bereichen angewendet und identifiziert effektiv schwache Bindemittel (1%-30% Trefferrate), die sowohl als hochwertige Ausgangspunkte für das Compound-Design dienen als auch umfangreiche strukturelle Informationen über Bindungsstellen liefern. Die Widerstandsfähigkeit des Prozesses wurde durch ein kontinuierliches Screening von SARS-CoV-2-Zielen während der COVID-19-Pandemie demonstriert, einschließlich einer 3-wöchigen Bearbeitungszeit für die Hauptprotease.
Einleitung
Fragment-Based Drug Discovery (FBDD) ist eine weit verbreitete Strategie für die Entdeckung von Leitstrukturen, und seit ihrer Entstehung vor 25 Jahren hat sie vier Medikamente für den klinischen Einsatz geliefert und mehr als 40 Moleküle wurden in klinische Studien überführt 1,2,3. Fragmente sind kleine chemische Einheiten, die in der Regel ein Molekulargewicht von 300 Da oder weniger haben. Sie werden aufgrund ihrer geringen chemischen Komplexität ausgewählt, die gute Ausgangspunkte für die Entwicklung von hochligandeneffizienten Inhibitoren mit hervorragenden physikalisch-chemischen Eigenschaften bietet. Aufgrund ihrer Größe beproben sie die Bindungslandschaft von Proteinen gründlicher als Bibliotheken größerer wirkstoff- oder bleiähnlicher Verbindungen und decken so auch Hot Spots und vermeintliche allosterische Stellen auf. In Kombination mit strukturellen Informationen liefern die Fragmente eine detaillierte Karte der möglichen molekularen Wechselwirkungen zwischen Protein und Ligand. Der zuverlässige Nachweis und die Validierung dieser Entitäten, die dazu neigen, schwach an das Zielprotein zu binden, erfordert jedoch eine Reihe robuster und empfindlicher biophysikalischer Screening-Methoden wie die Oberflächenplasmonenresonanz (SPR), die Kernspinresonanz (NMR) oder die isotherme Titrationskalorimetrie (ITC)4,5.
Die Röntgenkristallographie ist ein wesentlicher Bestandteil des FBDD-Toolkits: Sie ist empfindlich genug, um schwache Bindemittel zu identifizieren, und liefert direkt strukturelle Informationen über die Wechselwirkungen auf molekularer Ebene. Es ist komplementär zu anderen biophysikalischen Screenings und in der Regel unerlässlich für das Fortschreiten von Fragmenttreffern zu Bleiverbindungen. Es erfordert qualitativ hochwertige Kristallsysteme, was bedeutet, dass die Kristallisation hochgradig reproduzierbar ist und die Kristalle idealerweise mit einer Auflösung von mehr als 2,8 Å gebeugt werden.
In der Vergangenheit war es sehr schwierig, die Kristallographie als primäres Fragmentsieb zu verwenden 6,7,8, sei es in der Wissenschaft oder in der Industrie. Im Gegensatz dazu erzielten Synchrotrons Verbesserungen um Größenordnungen in der Robotik, Automatisierung 9,10,11 und Detektortechnologie 12,13, und in Kombination mit ebenso beschleunigter Rechenleistung und Algorithmen der Datenverarbeitung14,15,16 können vollständige Beugungsdatensätze in Sekundenschnelle und eine große Anzahl von ihnen völlig unbeaufsichtigt gemessen werden, wie es bei LillyCAT7 der Fall war und später MASSIF17,18 (European Synchrotron Radiation Facility (ESRF)). Dies führte dazu, dass Synchrotrons hochgradig optimierte Plattformen entwickelten, um das kristallbasierte Fragment-Screening als primären Bildschirm für eine breite Benutzergemeinschaft zugänglich zu machen (XChem bei Diamond; CrystalDirect auf der EMBL/ESRF19; BESSY am Helmholtz-Zentrum Berlin20; FragMax bei MaxIV21).
Dieser Artikel dokumentiert die Protokolle, die die XChem-Plattform für das Fragment-Screening durch Röntgenkristallographie ausmachen, von der Probenvorbereitung bis zu den endgültigen strukturellen Ergebnissen von 3D-modellierten Treffern. Die Pipeline (Abbildung 1) erforderte die Entwicklung neuer Ansätze für die Kristallidentifikation 22, das Einweichen23 und die Ernte24 sowie eine Datenmanagementsoftware 25 und einen algorithmischen Ansatz zur Identifizierung von Fragmenten 26, der heute in der Community weit verbreitet ist. Die Technologie zur Kristallernte wird jetzt von einem Anbieter verkauft (siehe Materialtabelle), und die offene Verfügbarkeit der Werkzeuge hat es anderen Synchrotrons ermöglicht, sie anzupassen, um gleichwertige Plattformen einzurichten21. Laufende Projekte befassen sich mit der Datenanalyse, Modellvervollständigung und Datenverbreitung über die Fragalysis-Plattform27. Das Probenvorbereitungslabor befindet sich neben der Beamline I04-1, was die Logistik für die Übertragung von Hunderten von gefrorenen Proben an die Beamline vereinfacht, und die dedizierte Strahlzeit auf I04-1 ermöglicht eine schnelle Röntgenrückkopplung zur Steuerung der Kampagne.
XChem ist ein integraler Bestandteil des Anwenderprogramms von Diamond, mit zwei Aufrufen pro Jahr (Anfang April und Oktober). Der Peer-Review-Prozess wurde in Absprache mit Experten für Arzneimittelforschung aus Wissenschaft und Industrie verfeinert. Neben einem überzeugenden wissenschaftlichen Argument verlangt das Antragsverfahren28 von den Antragstellern, dass sie nicht nur die Bereitschaft des Kristallsystems selbst bewerten, sondern auch ihr Fachwissen in biochemischen und orthogonalen biophysikalischen Methoden und ihre Fähigkeit, Screening-Treffer durch Follow-up-Chemie voranzutreiben. Auch die Zugriffsarten haben sich weiterentwickelt, um der multidisziplinären Nutzergemeinschaft gerecht zu werden:
Stufe 1 (Einzelprojekt ) ist für Projekte in der Sondierungsphase vorgesehen, und Hit-Validierungsinstrumente (biophysikalische oder biochemische Instrumente) und Follow-up-Strategien müssen nicht vorhanden sein. Wenn das Projekt angenommen wird, wird eine reduzierte Anzahl von Strahlzeitverschiebungen gewährt, die für den Proof of Concept ausreicht.
Stufe 2 (Einzelprojekt) ist für gut validierte Projekte gedacht und erfordert nachgelagerte Tools und Follow-up-Strategien. Wenn das Projekt angenommen wird, erhält es genügend Strahlzeit für eine vollständige Fragment-Screening-Kampagne. Einzelne Projekte (Tier 1 oder Tier 2) müssen innerhalb von 6 Monaten des Zuteilungszeitraums (entweder April bis September oder Oktober bis März) abgeschlossen werden.
Die Block Allocation Group (BAG) ist für Konsortien von Gruppen und Projekten gedacht, bei denen ein robuster Zielauswahl- und Priorisierungsprozess innerhalb des BAG sowie eine klare Follow-up-Pipeline vorhanden sind. BAGs müssen über mindestens einen vollständig XChem-geschulten Experten (Superuser) verfügen, der ihre Aktivitäten mit den Diamond-Mitarbeitern koordiniert und die BAG-Mitglieder schult. Die zugeteilte Anzahl der Strahlzeitverschiebungen wird durch die Anzahl der wissenschaftlich starken Projekte im BAG definiert und pro Vergabezeitraum auf Basis des Berichts des BAG neu bewertet. Der Zugang ist für 2 Jahre verfügbar.
Das XChem-Experiment ist in drei Phasen unterteilt, für die jeweils ein Entscheidungspunkt festgelegt ist: Lösungsmitteltoleranztest, Vorsieb und Hauptbildschirm (Abbildung 2). Der Lösungsmitteltoleranztest hilft dabei, die Einweichparameter, die Menge des Lösungsmittels (DMSO, Ethylenglykol oder andere Kryoprotektoren, falls erforderlich) zu definieren, die das Kristallsystem tolerieren kann und wie lange. Lösungsmittelkonzentrationen liegen in der Regel zwischen 5 % und 30 % über mindestens zwei Zeitpunkte. Beugungsdaten werden gesammelt und mit der Basisbeugung des Kristallsystems verglichen; Dadurch werden die Einweichparameter für die folgende Phase bestimmt. Für das Vorsieb werden 100-150 Verbindungen unter den im Lösungsmitteltest festgelegten Bedingungen eingeweicht, um zu bestätigen, dass die Kristalle die Verbindungen unter diesen Bedingungen vertragen können. Bei Bedarf wird das Kryoprotektivum anschließend zu den Tropfen gegeben, die bereits die Fragmente enthalten. Das Erfolgskriterium ist, dass 80 % oder mehr der Kristalle gut genug überleben, um Beugungsdaten von guter und gleichbleibender Qualität zu liefern. Wenn dies fehlschlägt, werden die Einweichbedingungen in der Regel durch Änderung der Einweichzeit oder der Lösungsmittelkonzentration überarbeitet. Nach einem erfolgreichen Pre-Screening können die restlichen für das Experiment ausgewählten Verbindungen anhand der finalen Parameter eingestellt werden.
Die DSI-Bibliothek (siehe Materialtabelle) wurde speziell entwickelt, um eine schnelle Nachverfolgung mit Hilfe der Chemie29 zu ermöglichen, und war das Arbeitspferd der Einrichtung. Es steht Anwendern in einer Konzentration von 500 mM in DMSO zur Verfügung. Akademische Nutzer können auch auf andere Bibliotheken zugreifen, die von Kooperationspartnern bereitgestellt werden (insgesamt über 2.000 Verbindungen) in Konzentrationen von 100-500 mM in DMSO (eine vollständige Liste finden Sie auf der Website28). Ein Großteil der Gesamtkollektion ist auch in Ethylenglykol erhältlich, für Kristallsysteme, die DMSO nicht vertragen. Benutzer können auch ihre eigenen Bibliotheken mitbringen, sofern sie sich in Platten befinden, die mit dem akustischen Liquid-Handling-System kompatibel sind (siehe Materialtabelle).
Für alle drei Schritte des Experiments (Lösungsmittelcharakterisierung, Pre-Screen oder Vollbild) sind die folgenden Probenvorbereitungsverfahren identisch (Abbildung 3): Auswahl des Abgabeortes der Verbindung durch Bildgebung und Targeting der Kristallisationstropfen mit TeXRank22; Abgabe in Tropfen unter Verwendung des akustischen Flüssigkeitsabgabesystems sowohl für Lösungsmittel als auch für Verbindungen23; effizientes Ernten der Kristalle mit dem Crystal Shifter24; und Hochladen von Probeninformationen in die Beamline-Datenbank (ISPyB). Die derzeitige Schnittstelle für die Versuchsplanung und -durchführung ist eine Excel-basierte Anwendung (SoakDB), die die notwendigen Eingabedateien für die verschiedenen Geräte der Plattform generiert und alle Ergebnisse in einer SQLite-Datenbank verfolgt und aufzeichnet. Barcode-Scanner werden in verschiedenen Phasen des Prozesses verwendet, um die Nachverfolgung von Proben zu unterstützen, und diese Daten werden der Datenbank hinzugefügt.
Die Beugungsdaten werden im unbeaufsichtigten Modus mit dedizierter Strahlzeit auf der Beamline I04-1 erfasst. Es stehen zwei Zentriermodi zur Verfügung, nämlich optisch und röntgenbasiert17. Für nadel- und stäbchenförmige Kristalle wird eine Röntgenzentrierung empfohlen, während klobigere Kristalle im Allgemeinen den optischen Modus unterstützen, der schneller ist und es daher ermöglicht, mehr Proben in der zugewiesenen Strahlzeit zu sammeln. Abhängig von der Auflösung der Kristalle (die vor dem Betreten der Plattform festgelegt wurde) kann die Datenerfassung entweder 60 s oder 15 s Gesamtbelichtung betragen. Die Datenerfassung während der Lösungsmitteltestphase gibt in der Regel Aufschluss darüber, welche Kombination am besten zur Leistung der Beamline I04-1 passt.
Das große Volumen der Datenanalyse wird über den XChemExplorer (XCE)25 verwaltet, der auch zum Starten des Trefferidentifikationsschritts mit PanDDA26 verwendet werden kann. XCE ist ein Datenmanagement- und Workflow-Tool, das die groß angelegte Analyse von Protein-Liganden-Strukturen unterstützt (Abbildung 4). Es liest alle Ergebnisse der automatischen Verarbeitung aus Daten, die an der Diamond Light Source (DIALS16, Xia214, AutoPROC30 und STARANISO31) gesammelt wurden, und wählt automatisch eines der Ergebnisse basierend auf der Datenqualität und der Ähnlichkeit mit einem Referenzmodell aus. Es ist wichtig, dass das Modell repräsentativ für das Kristallsystem ist, das für das XChem-Screening verwendet wird, und alle Wässer oder andere Lösungsmittelmoleküle sowie alle Cofaktoren, Liganden und alternativen Konformationen enthalten muss, die in Kristallen sichtbar sind, die nur mit Lösungsmittel getränkt sind. Die Qualität dieses Referenzmodells wirkt sich direkt auf den Arbeitsaufwand aus, der während der Modellerstellungs- und -verfeinerungsphase erforderlich ist. PanDDA wird verwendet, um alle Daten zu analysieren und Bindungsstellen zu identifizieren. Es richtet Strukturen an einer Referenzstruktur aus, berechnet die statistischen Karten, identifiziert Ereignisse und berechnet Ereigniskarten26,32. Im PanDDA-Paradigma ist es weder notwendig noch wünschenswert, ein vollständiges kristallographisches Modell zu erstellen. Was modelliert werden muss, ist nur die Ansicht des Proteins, an das ein Fragment gebunden ist (das Bound-State-Modell), so dass der Fokus nur auf dem Aufbau des Liganden und der umgebenden Reste/Lösungsmittelmoleküle gemäß der Ereigniskarte32 liegen muss.
Protokoll
1. Einreichung des Projektantrags
- Inhalt des Vorschlags: Da das XChem-Programm überzeichnet ist, sind gründliche und vollständige Informationen im Antrag entscheidend für das Bestehen des Peer-Review-Verfahrens.
- Machen Sie Argumente! Stellen Sie die Bedeutung des Ziels dar und stellen Sie es in den breiteren Kontext.
- Formulieren Sie die Strategie nach der Fragment-Screening-Kampagne: die orthogonalen Methoden zur Validierung der Treffer und wie sie weiterverarbeitet werden können. Ordnen Sie die Kollaborationen bei Bedarf an.
- Aufgrund des intensiven Labor- und Datenanalyseteils ist es sehr empfehlenswert, im Vorfeld einen erfahrenen Kristallographen zu beauftragen.
- Ein robustes Kristallsystem ist der Schlüssel, um technische Schwankungen zu vermeiden, und die Benutzer sollten diese wesentlichen Punkte berücksichtigen.
- Stellen Sie sicher, dass die Kristallisationsbedingungen reproduzierbare Tropfen mit ähnlicher Beugungsqualität in Platten liefern, die für die Verwendung auf der Plattform mit einem Reservoirvolumen von 30 μl (oder weniger) und einer Tropfengröße zwischen 200 und 500 nL geeignet sind. Im Idealfall weisen mehr als 50 % der Tropfen in einer Platte Kristalle von mindestens 35 μm Größe33 auf.
- Stellen Sie eine gleichbleibende Beugungsqualität der Kristalle sicher (2,6 Å oder besser).
- Überprüfen Sie die Eignung des Kristallsystems für das Fragment-Screening, einschließlich der Kristallpackung und der Zugänglichkeit bekannter Stellen. Frühere Beweise für ein Molekül, das an diesen Stellen gebunden ist, sind oft beruhigend.
- Machen Sie Argumente! Stellen Sie die Bedeutung des Ziels dar und stellen Sie es in den breiteren Kontext.
2. Vorbereitung des Besuchs
- Übertragung von Kristallisationsprotokollen für die Kristallisation vor Ort.
- Stellen Sie 2 x 50 ml Reservoirlösung zur Verfügung, gebrauchsfertig.
- Stellen Sie die Proteinlösung in der für die Kristallisation erforderlichen Konzentration gebrauchsfertig in Aliquoten von 30-50 μl bereit.
- Stellen Sie 10 ml der Proteinpufferlösung zur Verfügung.
- Stellen Sie Saatgut zur Verfügung (auch wenn es im Kristallisationsprotokoll nicht benötigt wird).
ANMERKUNG: Die Aussaat begünstigt die Reproduzierbarkeit der Kristallisation und beschleunigt die Keimbildungszeit33. - Füllen Sie das Formular mit Informationen zur Kristallisation aus, das auf der XChem-Websiteverfügbar ist 28.
- Geben Sie die Lagerinformationen im Versandformular an, das auf der XChem-Website verfügbar ist28.
- Installieren Sie NoMachine und richten Sie einen Remote-Desktop auf Diamond (https://www.diamond.ac.uk/Users/Experiment-at-Diamond/IT-User-Guide/Not-at-DLS/Nomachine.html ein.
- Generieren und übertragen Sie ein gutes Referenzmodell in Absprache mit einem erfahrenen Kristallographen oder XChem-Supportmitarbeitern.
3. Fragment-Screening-Experiment
- Definieren des Abgabeorts für die Mischung.
- Bildgebung von Kristallisationsplatten.
- Bilden Sie alle für das Experiment erforderlichen Kristallplatten (siehe Materialtabelle) in den Kristallplatten-Imagern ab (siehe Materialtabelle). Generieren Sie mit der Imager-Software Plattennamen im richtigen Verzeichnis für den Plattentyp im folgenden Format : Vorschlag Number_Plate Nummer.
- Drucken Sie die Barcodes aus (klicken Sie mit der rechten Maustaste auf den Plattennamen und wählen Sie sie aus dem Menü aus), platzieren Sie sie auf der gegenüberliegenden Seite des Schilds von den Zeilenbuchstaben, legen Sie die Platte (die Schilder) mit dem Barcode vom Benutzer abgewandt in die Ladeöffnung.
- Verwenden Sie die Imager-Steuerungssoftware, scannen Sie den Ladeanschluss, klicken Sie mit der rechten Maustaste auf die Platten und wählen Sie dann Speicherfolien aus.
- Sobald die Bildgebung abgeschlossen ist, entfernen Sie die Platten aus dem Imager.
- Auswahl der Kristalle und der Position der Verbindung
HINWEIS: Die Bilder der Kristallisationströpfchen werden innerhalb der Luigi-Pipeline mit dem textbasierten Algorithmus Ranker von TexRank verarbeitet, um die Tröpfchen nach dem wahrscheinlichen Vorhandensein von Kristallen22 zu ordnen. Dies dauert ca. 10 min und die Bilder stehen dann in TexRank zur Verfügung.- Öffnen Sie TeXRank von einem PC aus und wählen Sie das Kristallfach entweder aus der Liste unten rechts oder durch Eingabe des Barcodes in das Feld oben links aus.
- Wählen Sie das richtige Imager-Format und die Einzelwell-Ansicht aus. Bewegen Sie sich durch die Tropfenbilder, und wenn es einen Kristall gibt, der für die Verwendung in einem Experiment geeignet ist, klicken Sie mit der rechten Maustaste vom Kristall weg, aber innerhalb des Tropfens - das Ziel ist es, die Stelle im Tropfen anzuvisieren, an der Lösungsmittel/Verbindungen hinzugefügt werden sollen, so dass Sie den Kristall23 nicht direkt treffen möchten.
- Fahren Sie mit der gesamten Platte fort und wählen Sie am Ende die Echo 1 Target-Taste . Speichern Sie im Verzeichnis "Crystal Targets" unter dem entsprechenden Besuch. Ändern Sie den Dateinamen nicht.
- Wiederholen Sie den Vorgang für weitere Platten.
- Bildgebung von Kristallisationsplatten.
- Dosierung von Compounds
- Generieren von Dateien für die Compound-Dosierung
- Geben Sie in SoakDB Informationen zur Bibliotheksauswahl oder zum Lösungsmittel in die Tabelle Bibliothek/Lösungsmittel ein.
- Geben Sie das Tropfenvolumen und die Ladung in die Liste der Zielkristalle ein.
- Generieren Sie die erforderlichen Batches.
- Geben Sie die Einweichparameter ein. Klicken Sie auf Berechnen und dann auf die Schaltfläche Export ausstehend . Für Lösungsmittel geben Sie die verschiedenen Konzentrationen in die Tabelle ein. Dadurch werden die Dateien für die Verwendung im Akustikspender generiert.
- Wenn Sie Kryoprotektivum verwenden, geben Sie die Konzentration ein und erstellen Sie die Dateien auf die gleiche Weise.
- Dosierlösungen mit dem akustischen Spender (siehe Materialtabelle)
- Nehmen Sie die Ausgangsplatte (Verbindungen oder Lösungsmittel/Kryoprotektivum) und schleudern Sie die Platte 2 Minuten lang in der Zentrifuge bei 1.000 x g.
- Wenn Sie Lösungsmittel oder Kryoprotektivum abgeben, pipettieren Sie 30 μl in die entsprechende Vertiefung auf einer 384PP-Platte. Mit einer Mikrosiegelfolie abdecken und dann wie oben zentrifugieren.
- Öffnen Sie die Software. Wählen Sie Neu und wählen Sie die richtige Quellwellenplatte (384PP, 384LDV oder 1536LDV) und die Flüssigkeitsklasse (DMSO, CP, BP oder GP) aus. Stellen Sie sicher, dass der richtige Plattentyp als Zielplatte ausgewählt ist. Aktivieren Sie dann das Kontrollkästchen Benutzerdefiniert und fahren Sie fort.
- Klicken Sie auf Importieren und wählen Sie die entsprechende Batch-Datei aus. Führen Sie die Importschritte aus, wenn Sie von der Software dazu aufgefordert werden.
- Verwenden Sie die Plattenkarten, um die zu dosierende Lösung und die Bestimmungsorte zu überprüfen.
- Führen Sie das Protokoll aus und befolgen Sie die Eingabeaufforderungen, sobald sie angezeigt werden. Die Lösung(en) von der Ausgangsplatte werden in die ausgewählten Kristalltropfen abgegeben.
- Bewahren Sie die Platte für die erforderliche Zeit im Inkubator auf.
HINWEIS: Diese Parameter werden im Schritt der Lösungsmittelcharakterisierung bestimmt, die Temperatur beträgt je nach Kristallwachstumstemperatur entweder 4 °C oder 20 °C und die Zeiten liegen typischerweise zwischen 1 h und 3 h.
- Generieren von Dateien für die Compound-Dosierung
- Ernte von Kristallen mit dem halbautomatischen Kristallerntegerät (siehe Material-Tabelle).
HINWEIS: Wenn ein Kryoschutz erforderlich ist, wiederholen Sie Schritt 3.2.2 für die Zugabe von Kryoschutzlösungen auf die Kristalltropfen vor der Entnahme der Proben.- Vorbereitung für die Ernte
- Bereiten Sie die Dateien vor, die für das Harvesting in SoakDB erforderlich sind. Wenn Sie dazu aufgefordert werden, bestätigen Sie, dass die Einweichvorgänge abgeschlossen und die Chargen abgeschlossen sind.
- Scannen Sie die Anzahl der für das Experiment erforderlichen Pucks unter der korrekten Vorschlagsnummer aus.
- Wählen Sie ein Fach mit den Schleifen in der richtigen Größe für die Kristalle (35 μm, 75 μm oder 150 μm). Wichtig ist, dass Sie eine Schleifengröße wählen, die der Größe des Kristalls so genau wie möglich entspricht, um die automatische Zentrierung auf der Beamline genauer zu gestalten, die Datenqualität durch Reduzierung des Hintergrunds zu verbessern und die Notwendigkeit von Kryoprotektiven zu eliminieren.
- Öffnen Sie die entsprechende Software und öffnen Sie die Registerkarte Workflow.
- Scannen Sie die Pucks in die Software und scrollen Sie zurück zum Anfang der Liste, wobei Sie den ersten Puck markieren.
- Legen Sie die Pucks in einen Schaumdewar und kühlen Sie sie mit flüssigem Stickstoff ab.
- Wählen Sie Datei aus SoakDB importieren und wählen Sie den Stapel aus, der geerntet werden soll. Prüfen Sie, ob die Charge dem linken Halter zugeordnet ist. Sie gelangen auf einen Arbeitsvorrat.
- Nehmen Sie die Kristallplatte, entfernen Sie das Siegel und setzen Sie die linke Halterung ein. Bringen Sie die Platte in die Parkposition.
- Kristalle ernten
- Machen Sie es sich bequem und drücken Sie die Schaltfläche "Workflow starten" (der Bildschirm ist ein Touchscreen), um zur ersten ausgewählten Well-Position zu gelangen.
- Wenn der Kristall überlebt hat, montieren Sie den Kristall in der Schleife und tauchen Sie in den flüssigen Stickstoff ein, indem Sie ihn auf Position 1 im ersten Puck in der Liste platzieren.
- Wählen Sie auf der Benutzeroberfläche die entsprechende Beschreibung für den Kristall aus (normal, geschmolzen, gerissen, gelee oder farbig).
- Wenn es sich bei dem Tropfen um ein zusammengesetztes Einweichen handelt, notieren Sie die Beschreibung des Verbindungszustands (klar, kristallin, gefällt, schlecht dosiert oder Phasentrennung).
- Wenn der Quarz erfolgreich gemountet wurde, wählen Sie Mounted , andernfalls Fail.
- Die Platte wird in die nächste ausgewählte Vertiefung verschoben. Füllen Sie alle Puckpositionen nacheinander aus (lassen Sie keine Lücke, wenn ein Kristall ausgefallen ist). Fahren Sie bis zum Ende des Workflows fort.
- Laden Sie am Ende des Workflows alle zusätzlichen Chargen und füllen Sie die Pucks in der richtigen Reihenfolge. Es ist nicht erforderlich, einen neuen Puck für eine neue Charge zu starten.
- Barcode-Tracking der Ernteergebnisse
- Sobald alle Kristalle geerntet sind, bringen Sie die Pucks zum Barcode-Scanner, legen Sie einen nach dem anderen in die Halterung, um den Puck zu scannen und die Barcodes anzuheften.
- Wenn dies abgeschlossen ist, legen Sie die Deckel auf die Pucks und lagern Sie sie in einem Flüssigstickstoff-Lagerdewar.
- Laden Sie die Ausgabedatei in die SoakDB-Schnittstelle.
- Aufzeichnen von Beispielinformationen in ISPyB34,35,36
- Hochladen von Beispieldaten in ISPyB
- Aktualisieren Sie in SoakDB die Beamline, besuchen Sie Update für ISPyB und klicken Sie auf Exportieren, um die Datei zu erstellen, die in ISPyB hochgeladen werden soll.
- Offene Spachtelmasse. Melden Sie sich an und navigieren Sie zum folgenden Verzeichnis: dls/labxchem/data/year/lbXXXX-1/processing/lab36/ispyb.
- Führen Sie das Skript csv2ispyb (csv2ispyb lbXXXX-1-date.csv) aus.
HINWEIS: Die Beispiele werden jetzt in ISPyB geladen.
- Zeichnen Sie die Position des Pucks und die Datenerfassungsstrategie auf.
- Zeichnen Sie die Details und die Position der Pucks auf
HINWEIS: Es ist wichtig, die Details und die Position der Pucks aufzuzeichnen, damit sie lokalisiert und auf die Beamline geladen werden können.- Öffnen Sie in SoakDB die zweite Registerkarte mit der Bezeichnung Pucks.
- Geben Sie die Details in die Felder oben ein. Insbesondere die Position der Pucks (Lagerdewar und Stöcke), die Parameter der Datenerfassung, einschließlich der erwarteten Auflösung und der Vorschlagsnummer.
- Klicken Sie auf die Schaltfläche Speichern und eine Liste aller Pucks wird in der Tabelle angezeigt. Kopieren Sie die zuletzt gefüllten Pucks.
- Öffnen Sie die XChem-Warteschlangentabelle (Verknüpfung auf dem Desktop) und fügen Sie die Informationen ein. Geben Sie alle zusätzlichen relevanten Informationen ein.
- Zeichnen Sie die Details und die Position der Pucks auf
- Hochladen von Beispieldaten in ISPyB
- Vorbereitung für die Ernte
4. Datenerhebung
HINWEIS: Die Daten werden im unbeaufsichtigten Modus erfasst und vom XChem/Beamline-Team verwaltet.
- Erinnern von falsch zentrierten Proben.
HINWEIS: Diese sind erforderlich, wenn bei der Datenerfassung für bestimmte Proben Probleme aufgetreten sind, die höchstwahrscheinlich dadurch verursacht werden, dass die Stifte nicht richtig zentriert wurden.- Sehen Sie sich die Sample-Wechsler-Ansicht in ISPyB an, wählen Sie Rank by AP aus, um die Samples nach automatisch verarbeiteter Auflösung in einer Farbabstufung von Grün bis Rot zu bewerten.
- Klicken Sie auf die Proben, um zu prüfen, ob rote oder gelbe Proben vorhanden sind.
HINWEIS: Dadurch wird die Datenerfassung angezeigt. - Überprüfen Sie die Kristall-Schnappschüsse, um zu sehen, ob der Kristall zentriert ist.
- Notieren Sie sich alle, die nicht zentriert wurden, und senden Sie sie an den Ansprechpartner vor Ort, der die fehlenden Proben abholen wird.
5. Datenanalyse
- Abrufen und Analysieren der Ergebnisse der automatischen Verarbeitung von Diamond über XChemExplorer (XCE)25.
- Gehen Sie in einem Terminal in den Unterordner Processing: cd /dls/labxchem/data/year/visit/processing oder für XChem BAGs: cd /dls/labxchem/data/year/visit/processing/project/processing/.
- Verwenden Sie den Alias xce, um XChemExplorer zu öffnen.
- Wählen Sie die Schaltfläche Tabellen aus Datenquelle aktualisieren aus.
- Auf der Registerkarte Übersicht finden Sie eine Zusammenfassung der experimentellen Daten. Fügen Sie zusätzliche Kategorien mit der Option " Anzuzeigende Spalten auswählen" im Menü " Datenquelle" hinzu.
- Wählen Sie auf der Registerkarte Einstellungen das Datenerfassungsverzeichnis (/dls/i04-1/data/year/visit/) aus.
- Öffnen Sie die Registerkarte Datensätze, wählen Sie das Ziel aus dem Dropdown-Menü Ziel auswählen aus, wählen Sie Get Neue Ergebnisse aus dem Dropdown-Menü Automatische Verarbeitung aus Datensätze und klicken Sie auf Ausführen.
HINWEIS: XCE analysiert nun den Datenerfassungsbesuch für die automatische Verarbeitung von Ergebnissen. Dies kann bei der ersten Ausführung einige Zeit in Anspruch nehmen, abhängig von der Anzahl der Datasets/Verzeichnisse, die analysiert werden. - Überprüfen Sie die Konsistenz und Qualität der Daten, indem Sie die Auflösung, die Speichergruppe und Rmerge überprüfen. Schließen Sie Daten mit einer Auflösung von weniger als 2,8 Å aus.
HINWEIS: Standardmäßig basiert die Datensatzauswahl auf einem Score, der aus I/sigI, Vollständigkeit und Anzahl eindeutiger Reflexionen berechnet wird, aber es können auch andere Verarbeitungsergebnisse für die Verwendungausgewählt werden 25. - Um ein anderes Verarbeitungsergebnis für einzelne Datensätze auszuwählen, klicken Sie auf Beispiel-ID und wählen Sie das gewünschte Programm/den gewünschten Lauf aus. Um die Verarbeitungspipeline für alle Datensätze zu ändern, wählen Sie im Menü "Voreinstellungen" die Option "Voreinstellungen bearbeiten" und ändern Sie den Mechanismus zur Datensatzauswahl.
- Verarbeiten Sie die Daten bei Bedarf erneut über ISPyB37.
- Wenn keine verarbeiteten Daten für eine Stichprobe akzeptabel sind, kennzeichnen Sie sie als Fehlgeschlagen , um sie von der weiteren Analyse auszuschließen.
- Wenn Sie fertig sind, klicken Sie auf Tabellen aus Datenquelle aktualisieren , um Daten zu nachfolgenden Tabellen hinzuzufügen.
- Berechnung der Anfangskarten mit DIMPLE38.
- Öffnen Sie die Registerkarte Maps , wählen Sie das Referenzmodell aus dem Dropdown-Menü aus und wählen Sie die gewünschten Datensätze aus, gefolgt von DIMPLE für ausgewählte MTZ-Dateien ausführen.
- XCE führt zahlreiche DIMPLE-Jobs gleichzeitig auf dem Cluster bei Diamond aus. Suchen Sie den Status dieser Aufträge in der Spalte Dimple Status und aktualisieren Sie ihn mit der Schaltfläche Tabellen aus Datenquelle aktualisieren oder mit dem Befehl qstat unter Linux.
- Überprüfen Sie nach Abschluss des Prozesses, ob die Werte für Dimple Rcryst, Dimple Rfree und Space Group akzeptabel sind. Ändern Sie bei Bedarf (hoher Rfree/falsche Leerzeichengruppe/große Differenz im Einheitszellenvolumen) die Ergebnisse der automatischen Verarbeitung wie zuvor beschrieben, und wiederholen Sie die Kartengenerierung für diese Datensätze.
- Erzeugung von Ligandenbindungen mit Grad39, AceDRG40 oder phenix.eLBOW41.
- Wählen Sie das gewünschte Programm aus (Einstellungen, Voreinstellungen bearbeiten, Programm zur Generierung von Beschränkungen) und wählen Sie dann Datensätze auf der Registerkarte " Zuordnungen " aus, gefolgt von der Ausführung von CIF/PDB/PNG-Datei mit AUSGEWÄHLTEN Verbindungen erstellen aus der Dropdown-Liste "Karten und Beschränkungen ".
- Aktualisieren Sie den Status dieser Aufträge, die sich in der Spalte Verbundstatus befinden, mit der Schaltfläche Tabellen aus Datenquelle aktualisieren .
- Erstellen des Grundzustandsmodells (Pre-Run)
ANMERKUNG: Der Begriff Grundzustandsmodell stellt die Struktur des Proteins in seiner ligandenfreien Form dar, wie sie in 100 Datensätzen beobachtet wurde (diese Zahl ist willkürlich gewählt). Da das Grundzustandsmodell als Referenz für den Aufbau des ligandengebundenen Zustands verwendet wird, ist es wichtig, vor der Analyse der gesamten Fragment-Screening-Kampagne ein genaues Grundzustandsmodell zu erstellen, das alle Lösungsmittel- und Wassermoleküle enthält. In diesem Schritt werden die ersten hundert Datensätze mit der höchsten Auflösung, die von PanDDA als nicht interessant (und daher wahrscheinlich ligandenfrei) markiert wurden, verwendet, um die Karte des Grundzustandsmittelwerts zu erstellen, während der Datensatz mit dem niedrigstenfreien R für die Verfeinerung ausgewählt wird. Die Karte des Grundzustandsmittelwerts ist keine kristallographische Karte, es ist jedoch wichtig, diese Karte nur für die Erstellung des Grundzustandsmodells zu verwenden.- Öffnen Sie die Registerkarte PanDDAs, und aktualisieren Sie bei Bedarf Tabellen aus der Datenquelle.
- Definieren Sie das Ausgabeverzeichnis (/dls/labxchem/data/year/visit/processing/analysis/panddas).
- Wählen Sie Vorlauf für Grundzustandsmodell und klicken Sie auf Ausführen.
HINWEIS: Datasets mit hohem Rfree und unerwarteten Leerzeichengruppen sollten automatisch aus der Analyse ausgeschlossen werden. - Um Datasets mit hohem Rfree-Wert und unerwarteten Speicherplatzgruppen manuell auszuschließen, wählen Sie Vollständig ignorieren aus.
- Überprüfen Sie den Status des Vorabauftrags mit qstat in einem Terminalfenster.
- Wenn Sie fertig sind, wählen Sie Grundzustandsmodell erstellen und klicken Sie auf Ausführen.
HINWEIS: Dadurch wird Coot mit der PanDDA-Mittelwertkarte und einem Referenzmodell/2Fo-Fc/Fo-Fc-Karten aus dem Datensatz mit der besten Qualität für die Neumodellierung und Verfeinerung mit Coot geöffnet. Es ist von größter Bedeutung, dass nur die PanDDA-Mittelwertkarte für die Modellierung verwendet wird.
- Identifizieren von Treffern mit PanDDA26
- PanDDA-Analyse
HINWEIS: Die Ausführung auf dem Cluster kann einige Zeit in Anspruch nehmen, wenn viele Datasets vorhanden sind, die Einheitszelle groß ist und mehrere Kopien des Proteins in der asymmetrischen Einheit vorhanden sind.- Wiederholen Sie die zuvor beschriebenen Schritte für die automatische DLS-Analyse der Ergebnisse und die anfängliche Kartenberechnung. Verwenden Sie für die Kartenberechnung das Grundzustandsmodell als Referenz: Aktualisieren Sie die Referenzdateiliste > legen Sie eine neue Referenz fest , und generieren Sie die für die neuen Daten erforderlichen Ligandenbeschränkungen (Schritte 6.1-6.3).
- Stellen Sie auf der Registerkarte PanDDAs sicher, dass das Ausgabeverzeichnis wie zuvor eingestellt ist, und führen Sie pandda.analyse aus dem Dropdown-Menü Trefferidentifikation aus.
- Überprüfen Sie den Status des Auftrags im Linux-Terminal mit dem Befehl qstat.
- PanDDA-Inspektion - Überprüfen/Erstellen von Bindungsereignissen
- Führen Sie auf der Registerkarte PanDDAs in XCE pandda.inspect aus dem Dropdown-Menü Hit Identification aus, um Coot42 mit der PanDDA-Systemsteuerung zu öffnen.
HINWEIS: Die Systemsteuerung pandda.inspect bietet eine Zusammenfassung der PanDDA-Statistiken und ermöglicht es Benutzern, durch Bindungsereignisse/Websites zu navigieren. Eine zusammenfassende HTML-Datei mit den Ergebnissen wird ebenfalls generiert und kann während der Inspektion aktualisiert werden, indem HTML aktualisieren ausgewählt wird. - Um einen Liganden zu modellieren, klicken Sie auf Ligand mit Modell zusammenführen und Modell speichern , bevor Sie zu einem anderen Ereignis navigieren, um zu vermeiden, dass Änderungen am gebundenen Zustandsmodell verloren gehen.
HINWEIS: Nur Modelle, die aktualisiert und gespeichert wurden, werden exportiert, um sie zu einem späteren Zeitpunkt zu verfeinern. - Verwenden Sie das Feld Ereigniskommentar , um das Bindungsereignis mit Anmerkungen zu versehen, und die Informationen zur Aufzeichnungswebsite , um Bindungssites mit Anmerkungen zu versehen.
- Lastdurchschnitt und 2mFo-DFc-Karten (von DIMPLE) zum Vergleich mit der Ereigniskarte und dem Modell.
- Nachdem alle lebensfähigen Liganden modelliert, zusammengeführt und basierend auf der Ereigniszuordnung gespeichert wurden, schließen Sie pandda.inspect.
- Führen Sie auf der Registerkarte PanDDAs in XCE pandda.inspect aus dem Dropdown-Menü Hit Identification aus, um Coot42 mit der PanDDA-Systemsteuerung zu öffnen.
- PanDDA-Export und -Verfeinerung
HINWEIS: Im Anschluss an PanDDA werden die Inspektionsmodelle zurück in das Projektverzeichnis exportiert und eine erste Runde der Verfeinerung gestartet. Derzeit stehen dafür zwei Pipelines unter der Registerkarte PANDDAs in XCE zur Verfügung.- Beim Exportieren von NEUEN/ALLEN/AUSGEWÄHLTEN PANDDA-Modellen wird ein Ensemble der gebundenen und ungebundenen Modelle zur Verfeinerung und der Belegungsbeschränkungsparameter für Refmac43 generiert.
HINWEIS: Das Ensemble-Modell wird für die Verfeinerung verwendet, aber nur das gebundene Zustandsmodell wird in Blässhuhn aktualisiert und in der PDB hinterlegt. Diese Pipeline eignet sich am besten für Datensätze mit geringer Belegung von Fragmenten und signifikanten Änderungen am Proteinmodell. - Verfeinerung von NEW/ALL-Bound-State-Modellen mit BUSTER verfeinert den Bound-State nur mit Buster44.
HINWEIS: Dies wird am besten bei Liganden/Datensätzen mit hoher Belegung und minimalen Änderungen am Proteinmodell verwendet.
- Beim Exportieren von NEUEN/ALLEN/AUSGEWÄHLTEN PANDDA-Modellen wird ein Ensemble der gebundenen und ungebundenen Modelle zur Verfeinerung und der Belegungsbeschränkungsparameter für Refmac43 generiert.
- PanDDA-Analyse
- Verfeinerung der Treffer (alle für die Verfeinerung ausgewählten Datensätze sind jetzt auf der Registerkarte Verfeinerung sichtbar). Wählen Sie Open COOT - BUSTER Refinement oder Open COOT - REFMAC Refinement aus dem Dropdown-Menü Refinement, um Coot mit der XCE Refinement-Systemsteuerung zu öffnen.
- Wählen Sie den Status der zu verfeinernden Proben aus dem Dropdown-Menü Proben auswählen (normalerweise 3 - in Verfeinerung) und klicken Sie auf GO.
HINWEIS: Das XCE-Bedienfeld bietet eine Zusammenfassung der Anzahl der Datensätze für diese Kategorie und ermöglicht die Navigation zwischen Datensätzen, während eine Zusammenfassung der Verfeinerungsstatistiken bereitgestellt wird. - Kommentieren Sie die Ligandenkonfidenz in der XCE-Systemsteuerung: 0 - kein Ligand vorhanden - Fragment hat nicht gebunden; 1 - Niedriges Konfidenz- Fragment hat möglicherweise gebunden, ist aber nicht besonders überzeugend; 2 - Korrekter Ligand, schwache Dichte - Der Benutzer ist zuversichtlich, dass das Fragment gebunden ist, aber es ist eine geringe Belegung / es gibt einige Probleme mit den Karten; 3 - Klare Dichte, unerwarteter Ligand- Karten zeigen eindeutig eine Ligandenbindung, die nicht mit der bereitgestellten chemischen Struktur korreliert; 4 - Hohes Konfidenz- Der Ligand ist eindeutig gebunden.
- Nehmen Sie in dieser Phase alle erforderlichen Änderungen am Modell vor, und leiten Sie die weitere Verfeinerung über die Schaltfläche Verfeinern ein.
- Verwenden Sie die Schaltfläche MolProbity-To-Do-Liste anzeigen , um auf die MolProbity45-Analyse zuzugreifen, die für alle Verfeinerungszyklen ausgeführt wird.
- Fügen Sie bei Bedarf Verfeinerungsparameter hinzu, z. B. für anisotrope Temperaturfaktoren, Zwillingsdaten oder Belegungsverfeinerung, indem Sie die Schaltfläche Verfeinerungsparameter auswählen.
HINWEIS: Datenverarbeitungsstatistiken werden auch in XCE unter der Registerkarte "Verfeinerung" bereitgestellt, und wenn die Verfeinerung mit der Buster-Pipeline durchgeführt wird, werden Buster-Berichte, einschließlich der MOGUL-Analyse46, bereitgestellt. - Ändern Sie den Status eines Datensatzes während der Verfeinerung sowohl im XCE-Hauptfenster auf der Registerkarte "Verfeinerung " als auch in der Coot XCE-Systemsteuerung . Wenn Sie davon überzeugt sind, dass das Modell um den Liganden herum genau ist und für die weitere Analyse freigegeben werden kann, ändern Sie den Status in CompChem Ready. Wenn die Verfeinerung abgeschlossen ist und das Modell für das Hochladen in die PDB bereit ist, ändern Sie den Status in Ablage bereit.
- Wählen Sie den Status der zu verfeinernden Proben aus dem Dropdown-Menü Proben auswählen (normalerweise 3 - in Verfeinerung) und klicken Sie auf GO.
6. Hinterlegung der Daten
HINWEIS: Alle Datensätze aus einem Fragmentbildschirm und dem Grundzustandsmodell, das zum Generieren der PanDDA-Ereigniskarten verwendet wird, können mithilfe von Gruppenablagerungen in der PDB abgelegt werden.
- Konvertieren Sie alle PanDDA-Ereigniszuordnungen in das MTZ-Format, indem Sie Ereigniszuordnung ->SF aus dem Menü Trefferidentifikation ausführen.
- Geben Sie zusätzliche Metadaten wie Autoren und Methoden an, indem Sie Deposition > Informationen bearbeiten auswählen. Füllen Sie alle erforderlichen Elemente aus und klicken Sie auf In Datenbank speichern und speichern Sie diese Informationen dann für die Hinterlegung des Grundzustandsmodells. Tun Sie dies, nachdem der Modellstatus in "Abscheidungsbereit" geändert wurde.
- Wählen Sie auf der Registerkarte " Ablagerung " die Schaltfläche " mmcif vorbereiten" aus, um mmcif-Dateien mit Strukturfaktor für alle abscheidungsbereiten Datensätze zu generieren. Die folgende Meldung wird im Terminalfenster angezeigt, wenn dieser Vorgang abgeschlossen ist: Vorbereiten der mmcif-Dateien für die wwPDB-Ablage abgeschlossen.
- Wählen Sie die Schaltfläche mmcif kopieren aus, um alle diese Dateien in ein einzelnes gezipptes tar-Archiv im Group Deposition Directory des Besuchs zu kopieren.
- Gehen Sie zu https://deposit-group-1.rcsb.rutgers.edu/groupdeposit; Melden Sie sich mit dem Benutzernamen: GroupTester und dem Passwort: !2016RCSBPDB an. Erstellen Sie eine Sitzung, und laden Sie die ligandengebundene Datei .tar.bz2 aus dem Gruppenablagerungsverzeichnis hoch.
- Nach erfolgreicher Einreichung der ligandengebundenen Strukturen wird eine E-Mail mit den PDB-Codes versendet. Wählen Sie " Datenbank mit PDB-Codes aktualisieren " aus dem Menü " Ablage" . Kopieren Sie die Informationen aus dieser E-Mail, fügen Sie sie in das Popup-Fenster ein und klicken Sie auf Datenbank aktualisieren , um PDB-IDs hinzuzufügen.
- Um das von PanDDA verwendete Grundzustandsmodell zu hinterlegen, wählen Sie das entsprechende PanDDA-Verzeichnis in XCE aus und führen Sie apo->mmcif aus dem Menü Trefferidentifikation aus.
HINWEIS: XCE wählt willkürlich eine hochauflösende Struktur mit niedrigem Rfree als Modell für das Abscheidungsbündel aus und kompiliert dann alle mmcif-Dateien des Strukturfaktors in eine einzige Datei. - Wählen Sie auf der Registerkarte "Ablagerung" die Schaltfläche "Zur Datenbank hinzufügen" unterhalb des Abschnitts "Ablagerung des Grundzustandsmodells gruppieren" aus.
- Geben Sie die Metadaten für das Grundzustandsmodell ein (erneut durch Auswahl von Ablagerung > Informationen bearbeiten), laden Sie die vorherige Datei und speichern Sie sie in Datenbank.
- Bereiten Sie die mmcif-Datei für den Grundzustand vor, indem Sie mmcif vorbereiten im Abschnitt Group Deposition of Ground-State Model ausführen und kopieren Sie die mmcif-Datei anschließend in das Verzeichnis Group Deposition, indem Sie im selben Abschnitt die Schaltfläche mmcif kopieren auswählen.
- Gehen Sie wie zuvor zu https://deposit-group-1.rcsb.rutgers.edu/groupdeposit; Melden Sie sich mit dem Benutzernamen: GroupTester und dem Passwort: !2016RCSBPDB an. Erstellen Sie eine Sitzung, und laden Sie die Datei ground_state_structures.tar.bz2 aus dem Gruppenablageverzeichnis hoch.
Ergebnisse
Die XChem-Pipeline für das Fragment-Screening durch Röntgenkristallographie wurde umfassend gestrafft, so dass sie von der wissenschaftlichen Gemeinschaft übernommen werden kann (Abbildung 5). Dieser Prozess wurde bei über 150 Screening-Kampagnen mit einer Trefferquote zwischen 1 % und 30 % validiert47,48,49,50,51,52 und von vielen Wiederholungsnutzern. Kristallsysteme, die nicht geeignet sind (geringe Auflösung, inkonsistent in der Kristallisation oder in der Beugungsqualität) oder weder DMSO noch Ethylenglykol vertragen, werden frühzeitig im Prozess eliminiert, was Zeit, Aufwand und Ressourcen spart. Erfolgreiche Kampagnen liefern eine dreidimensionale Karte potenzieller Interaktionsstellen auf dem Zielprotein; Ein typisches Ergebnis ist das XChem-Screening der Hauptprotease von SARS-CoV-2 (Abbildung 6). Typischerweise werden Fragmenttreffer gefunden in: (a) bekannten Stellen von Interesse, wie z. B. enzymaktiven Zentren und Untertaschen48; (b) mutmaßliche allosterische Stellen, z. B. bei Protein-Protein-Wechselwirkungen53; (c) Kristallpackungsschnittstellen, die im Allgemeinen als falsch positiv angesehen werden (Abbildung 6). Diese Strukturdaten bilden im Allgemeinen die Grundlage für die Verschmelzung, Verknüpfung oder Vergrößerung von Fragmenttreffern zu bleiartigen kleinen Molekülen 1,3.
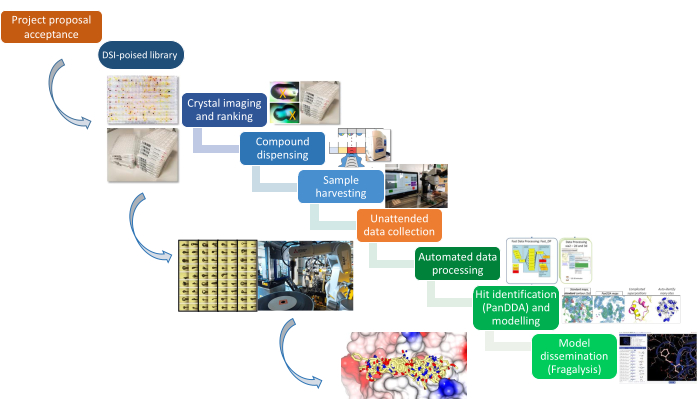
Abbildung 1: Die XChem-Pipeline. Die Plattform wird schematisch vom Projektvorschlag über die Probenvorbereitung und Datenerfassung bis hin zur Trefferidentifikation dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
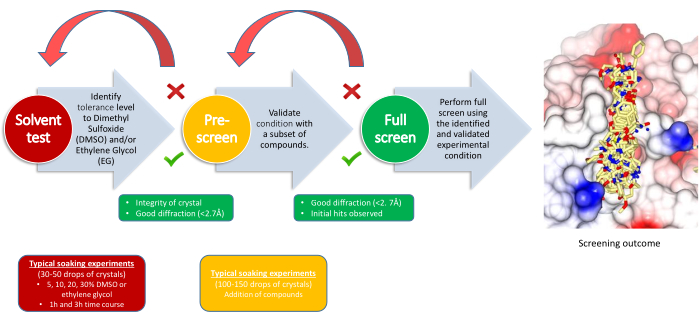
Abbildung 2: Screening-Strategie. Der Workflow gibt den Zweck jedes Meilensteins, die Anforderungen des Experiments und die Entscheidungspunkte an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
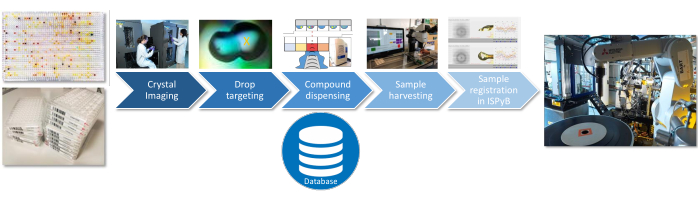
Abbildung 3: Arbeitsablauf bei der Probenvorbereitung. Kritische Schritte für die Probenvorbereitung werden dargestellt, wobei Informationen aus jedem Schritt in einer SQLite-Datenbank aufgezeichnet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
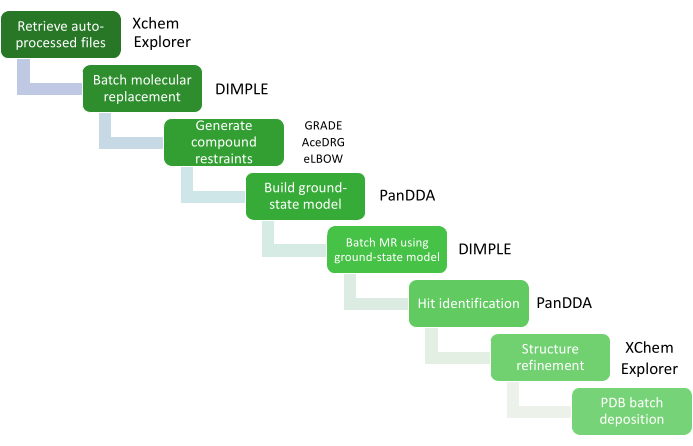
Abbildung 4: Datenanalyse mit XCE. Kritische Schritte in der Datenanalyse werden durch ein Workflow-Diagramm mit den entsprechenden Softwarepaketen dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
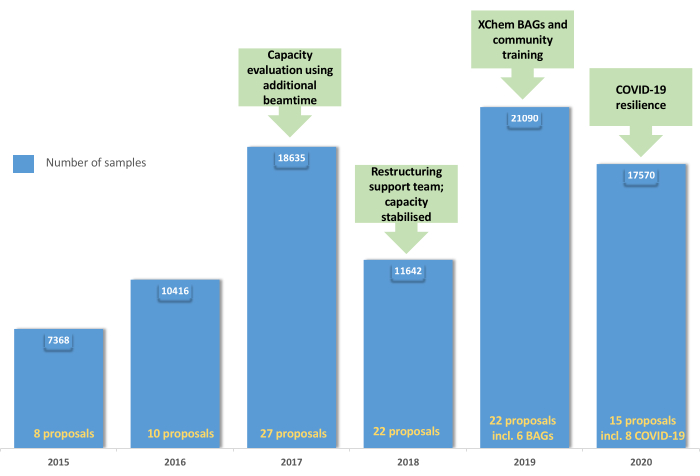
Abbildung 5: Entwicklung des XChem-Benutzerprogramms: Die Grafik zeigt die Akzeptanz und Konsolidierung des Benutzerprogramms von 2015 bis 2019 mit der Schaffung von BAGs im Jahr 2019 und die Widerstandsfähigkeit der Plattform während der COVID-19-Pandemie im Jahr 2020. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
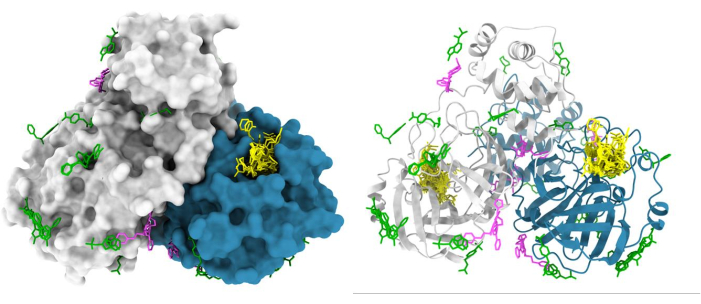
Abbildung 6: Repräsentative Ergebnisse des XChem-Fragment-Screenings. Das Dimer der SARS-CoV2-Hauptprotease (Mpro) ist in der Oberfläche dargestellt, wobei die Treffer des aktiven Zentrums gelb, die mutmaßlichen allosterischen Treffer in Magenta und die Oberflächen-/Kristallpackungsartefakte grün dargestellt sind. Die Abbildung wurde unter Verwendung von Chimera- undM-pro-PDB-Einträgen aus der Gruppenablagerung G_1002156 erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Der in diesem Dokument beschriebene Prozess wurde von der Benutzergemeinschaft ausgiebig getestet, und die Anpassungsfähigkeit der hier beschriebenen Protokolle ist der Schlüssel für die Handhabung der Vielzahl von Projekten, die typischerweise auf der Plattform auftreten. Es sind jedoch einige Voraussetzungen für das Kristallsystem notwendig.
Für jede Fragment-Screening-Kampagne, die mittels Röntgenkristallographie durchgeführt wird, ist ein reproduzierbares und robustes Kristallsystem von entscheidender Bedeutung. Da das Standard-XChem-Protokoll die Zugabe des Fragments direkt zum Kristalltropfen vorsieht, sollte sich die Optimierung auf die Anzahl der Tropfen konzentrieren, die hochwertige Kristalle enthalten, und nicht auf die Gesamtzahl der Kristalle. Wenn Tropfen mehrere Kristalle enthalten, sind sie effektiv überflüssig, obwohl sie den Ernteprozess erleichtern können. Darüber hinaus kann die Übertragung des Kristallisationsprotokolls vom Heimatinstitut auf die Einrichtungen vor Ort eine Herausforderung darstellen. Dies wird im Allgemeinen am besten durch Kristallaussaat erreicht, um eine reproduzierbare Keimbildung zu fördern54, und daher ist es eine gute Praxis für die Anwender, Saatgut zusammen mit ihren Protein- und Kristallisationslösungen bereitzustellen.
Um eine gute Löslichkeit und Unterstützung der Verbindung zu gewährleisten, werden die hohen Einweichkonzentrationen, die die Bindung schwacher Fragmente vorantreiben sollen, Fragmentbibliotheken in organischen Lösungsmitteln, insbesondere DMSO und Ethylenglykol, bereitgestellt. Die Bereitstellung von zwei verschiedenen Lösungsmitteln bietet Anwendern eine Alternative für Kristalle, die DMSO überhaupt nicht vertragen oder bei denen es die Bindung von Fragmenten an einer interessierenden Stelle verhindert. Anwender können alternative Bibliotheken in wässrigem Puffer liefern: Verbindungen lassen sich gut dosieren, sofern sie vollständig aufgelöst und in Platten formatiert sind, die mit dem Flüssigkeitsdosierroboter kompatibel sind.
Für Projekte, bei denen es nicht möglich ist, ein geeignetes organisches Lösungsmittel zu finden, das sowohl die Bibliothek auflöst als auch vom Kristallsystem toleriert wird, besteht ein alternatives Verfahren darin, getrocknete Verbindungen zu verwenden, wie sie an BESSY55 etabliert sind.
In der Gemeinschaft gibt es seit langem die Frage, ob Verbindungen in Kristalle eingeweicht werden können, die unter Kristallisationsbedingungen mit hohen Salzkonzentrationen gezüchtet wurden. In der Praxis wird eine stärkere Ausfällung der Verbindungen und eine schnelle Bildung von Salzkristallen in der Erntephase beobachtet, die durch die Anwendung einer feuchten Umgebung um den Erntebereich herum reduziert wird. Im Allgemeinen liefern Screening-Kampagnen in Kristallsystemen unter Bedingungen mit hoher Salzkristallisation eine vergleichbare Trefferquote wie unter Bedingungen mit niedrigem Salzgehalt.
Die Anfangsphasen des XChem-Prozesses (Lösungsmitteltoleranzprüfung und Pre-Screening) sind relativ kleine und schnelle Experimente, ermöglichen aber eine klare Go/No-Go-Entscheidung für das Projekt. Am schmerzhaftesten ist, dass alternative Kristallsysteme gefunden werden müssen, wenn keines der beiden Lösungsmittel vertragen wird oder das Pre-Screening zu einer sehr niedrigen Trefferquote führt. Im Gegensatz dazu geben die Ergebnisse, wenn sie erfolgreich sind, direkt Aufschluss über die Einweichbedingung, die für das Screening-Experiment verwendet werden soll, und über die beste Strategie für die Datenerfassung. Da die Qualität der Daten, insbesondere die Auflösung, die Qualität der Elektronendichte für die Trefferidentifikation und -analyse beeinflusst, ist es das Ziel, bei der höchstmöglichen Verbindungskonzentration zu tränken, die sich nicht nachteilig auf die Beugungsqualität auswirkt (wobei die Mehrheit der Datensätze (~80%) mit einer Auflösung von 2,8 Å oder besser beugt).
Der Datenanalyseprozess wird im XChemExplorer optimiert, der sich auf die PanDDA-Software zur Erkennung schwacher Bindemittel stützt und es den Benutzern ermöglicht, die Ergebnisse der Screening-Kampagne schnell zu visualisieren und zu überprüfen. XChemExplorer importiert Datenverarbeitungsergebnisse aus den bei Diamond verfügbaren Paketen (DIALS 16, autoPROC 30, STARANISO31 und Xia214) mit Auflösungsgrenzen, die durch die Standardmethode für jedes Paket bestimmt werden (d. h. CC1/2 = 0,3). Standardmäßig basiert die Datensatzauswahl auf einem Score, der aus I/sigI, Vollständigkeit und einer Anzahl eindeutiger Reflexionen berechnet wird, aber spezifische Verarbeitungsergebnisse können sowohl global als auch für einzelne Stichproben ausgewählt werden25. Die Daten werden auch von der Analyse durch PanDDA ausgeschlossen, basierend auf Kriterien wie Auflösung,R-frei und Unterschied im Einheitszellvolumen zwischen Referenz- und Zieldaten (Standardwerte sind 3,5 Å, 0,4 bzw. 12 %), so dass schlecht beugende, falsch zentrierte oder falsch indizierte Kristalle die Analyse nicht beeinträchtigen.
Der PanDDA-Algorithmus nutzt die beträchtliche Anzahl von Datensätzen, die während einer Fragmentkampagne gesammelt werden, um Liganden der Teilbelegung zu erkennen, die in kristallographischen Standardkarten nicht sichtbar sind. Anfänglich verwendet PanDDA Daten, die während der Lösungsmitteltoleranztests und der Pre-Screening-Schritte gesammelt wurden, um eine Karte der durchschnittlichen Dichte zu erstellen, die dann zur Erstellung eines Grundzustandsmodells verwendet wird. Da dieses Modell für alle nachfolgenden Analyseschritte verwendet wird, ist es wichtig, dass es das unligandierte Protein unter den für das Fragment-Screening verwendeten Bedingungen genau repräsentiert. PanDDA verwendet dann eine statistische Analyse, um gebundene Liganden zu identifizieren und eine Ereigniskarte für den gebundenen Zustand des Kristalls zu erstellen. Eine Ereigniskarte wird generiert, indem der ungebundene Anteil des Kristalls aus dem Teilbelegungsdatensatz subtrahiert wird, und stellt dar, was beobachtet würde, wenn der Ligand bei voller Belegung gebunden wäre. Selbst Fragmente, die in herkömmlichen 2mFo-DF c-Karten klar erscheinen, können falsch modelliert werden, wenn die Ereigniskarten nicht konsultiert werden32. Während PanDDA eine leistungsfähige Methode zur Identifizierung von Datensätzen ist, die sich von den durchschnittlichen Karten unterscheiden (was in der Regel auf die Fragmentbindung hinweist) und Metriken wie RSCC, RSZD, B-Faktor-Verhältnis und RMSD während der Verfeinerung zum Nutzen des Benutzers bereitgestellt werden, ist der Benutzer letztendlich dafür verantwortlich, zu entscheiden, ob die beobachtete Dichte den erwarteten Liganden und die am besten geeignete Konformation genau abbildet.
Nach der Datenanalyse und -verfeinerung ist es allen Anwendern möglich, mit dem XChemExplorer gleichzeitig mehrere Strukturen in der Proteindatenbank (PDB) zu hinterlegen. Für jedes Fragment-Screening werden zwei Gruppenablagerungen vorgenommen. Die erste Ablagerung enthält alle fragmentgebundenen Modelle mit Koeffizienten für die Berechnung von PanDDA-Ereigniszuordnungen, die in MMCIF-Dateien enthalten sind. Die zweite Ablagerung liefert das begleitende Grundzustandsmodell, zusammen mit den gemessenen Strukturfaktoren aller Datensätze des Experiments: Diese Daten können zur Reproduktion der PanDDA-Analyse und zur Entwicklung zukünftiger Algorithmen verwendet werden. Was die Strukturen der Treffer betrifft, so ist die Verfeinerung bei geringer Fragmentbelegung besser, wenn die Modelle eine Zusammensetzung der ligandengebundenen und verwirrenden Grundzustandsstrukturensind 32; Nichtsdestotrotz ist es üblich, nur die Bruchteile des gebundenen Zustands zu hinterlegen, da die vollständigen zusammengesetzten Modelle im Allgemeinen komplex und schwer zu interpretieren sind. Infolgedessen sind einige Qualitätsindikatoren, die von der HVE neu berechnet werden (insbesondere R/Rfree), manchmal leicht erhöht. Es ist auch möglich, alle Rohdaten über Plattformen wie Zenodo56 bereitzustellen, obwohl dies derzeit nicht von der XChem-Pipeline unterstützt wird.
Insgesamt konnten seit dem Betrieb im Jahr 2016 in über 95 % der Targets mit diesem Verfahren Fragmentliganden identifiziert werden. Die Erfahrungen aus den vielen Projekten, die XChem unterstützt hat, wurden zu Best Practices für die Kristallpräparation33 destilliert, während eine Fragmentbibliothek entwickelt wurde, die das Konzept zur Unterstützung der Fragmentprogression29 implementierte und auch dazu beitrug, die Praxis der Veröffentlichung der Bibliothekskomposition zu etablieren. Die Plattform hat gezeigt, wie wichtig eine gut gewartete Infrastruktur und dokumentierte Prozesse sind, die hier detailliert beschrieben werden, und hat es ermöglicht, andere Fragmentbibliotheken57,58 zu evaluieren, Bibliotheken48 zu vergleichen und das Design der kollaborativen EUOpenscreen-DRIVE-Bibliothek 59,60 zu informieren.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit stellt eine große gemeinsame Anstrengung zwischen der Diamond Light Source und dem Structure Genomic Consortium dar. Die Autoren möchten den verschiedenen Unterstützungsgruppen und der MX-Gruppe von Diamond für ihren Beitrag zur Automatisierung der i04-1-Beamline und für die Bereitstellung optimierter Datenerfassungs- und Auto-Processing-Pipelines danken, die üblicherweise über alle MX-Beamlines hinweg ausgeführt werden. Sie möchten sich auch bei der SGC PX-Gruppe für ihre Widerstandsfähigkeit bedanken, die ersten Anwender zu sein, die das Setup getestet haben, und bei Evotec dafür, dass sie der erste ernsthafte industrielle Anwender waren. Diese Arbeit wurde durch iNEXT-Discovery (Grant 871037) gefördert, gefördert durch das Horizon 2020 Programm der Europäischen Kommission.
Materialien
| Name | Company | Catalog Number | Comments |
| DSI-poised library | Enamine | DSI-896 | fragment library |
| Echo 550 and 650 series | Beckman-Coulter | acoustic dispensing system | |
| Echo microplates | Beckman-Coulter | 001-12380; 001-8768; 001-6025 | 1536-well and 384-well microplates |
| Shifter | Oxford Lab Technology | harvesting device | |
| Microplate centrifuge with a swing-out rotor | Sigma | model 11121 | microplate centrifuge |
| 3-drops crystallisation plates | Swissci | 3W96T-UVP | Crystallisation plates |
| Formulatrix plate imager and Rockmaker software | Formulatrix | Crystallisation plates imaging device |
Referenzen
- Erlanson, D. A., Fesik, S. W., Hubbard, R. E., Jahnke, W., Jhoti, H. Twenty years on: The impact of fragments on drug discovery. Nature Reviews Drug Discovery. 15 (9), 605-619 (2016).
- Jacquemard, C., Kellenberger, E. A bright future for fragment-based drug discovery: what does it hold. Expert Opinion on Drug Discovery. 14 (5), 413-416 (2019).
- Jahnke, W., et al. Fragment-to-lead medicinal chemistry publications in 2019. Journal of Medicinal Chemistry. 63 (24), 15494-15507 (2019).
- Li, Q. Application of fragment-based drug discovery to versatile targets. Frontiers in Molecular Biosciences. 7, 180 (2020).
- Kirsch, P., Hartman, A. M., Hirsch, A. K. H., Empting, M. Concepts and core principles of fragment-based drug design. Molecules. 24 (23), 4309 (2019).
- Patel, D., Bauman, J. D., Arnold, E. Advantages of crystallographic fragment screening: functional and mechanistic insights from a powerful platform for efficient drug discovery. Progress in Biophysics and Molecular Biology. 116 (2-3), 92-100 (2014).
- Wasserman, S., et al. Automated synchrotron crystallography for drug discovery: the LRL-CAT beamline at the APS. Acta Crystallographica Section A Foundations of Crystallography. 67 (1), 46-47 (2011).
- Hartshorn, M. J. Fragment-based lead discovery using X-ray crystallography. Journal of Medicinal Chemistry. 48 (2), 403-413 (2005).
- Arzt, S., et al. Automation of macromolecular crystallography beamlines. Progress in Biophysics and Molecular Biology. 89 (2), 124-152 (2005).
- Beteva, A. High-throughput sample handling and data collection at synchrotrons: Embedding the ESRF into the high-throughput gene-to-structure pipeline. Acta Crystallographica Section D, Biological Crystallography. 62, 1162-1169 (2006).
- Papp, G., et al. FlexED8: The first member of a fast and flexible sample-changer family for macromolecular crystallography. Acta Crystallographica. Section D, Structural Biology. 73, 841-851 (2017).
- Casanas, A., et al. EIGER detector: Application in macromolecular crystallography. Acta Crystallographica Section D, Structural Biology. 72, 1036-1048 (2016).
- Henrich, B., et al. PILATUS: A single photon counting pixel detector for X-ray applications. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 607 (1), 247-249 (2009).
- Winter, G., Lobley, C. M. C., Prince, S. M. Decision making in xia2. Acta Crystallographica Section D, Biological Crystallography. 69, 1260-1273 (2013).
- Winter, G., McAuley, K. E. Automated data collection for macromolecular crystallography. Methods. 55 (1), 81-93 (2011).
- Winter, G., et al. DIALS: Implementation and evaluation of a new integration package. Acta Crystallographica Section D, Structural Biology. 74, 85-97 (2018).
- Bowler, M. W. MASSIF-1: A beamline dedicated to the fully automatic characterization and data collection from crystals of biological macromolecules. Journal of Synchrotron Radiation. 22 (6), 1540-1547 (2015).
- Von Stetten, D., et al. ID30A-3 (MASSIF-3) - A beamline for macromolecular crystallography at the ESRF with a small intense beam. Journal of Synchrotron Radiation. 27, 844-851 (2020).
- Cipriani, F., et al. CrystalDirect: a new method for automated crystal harvesting based on laser-induced photoablation of thin films. Acta Crystallographica. Section D, Biological Crystallography. 68, 1393-1399 (2012).
- . Helmholtz Zentrum Berlin Available from: https://www.helmholtzberlin.de/forschung/oe/np/gmx/fragment-screening/index_en.html (2021)
- Lima, G. M. A., et al. FragMAX: the fragment-screening platform at the MAX IV Laboratory. Acta crystallographica. Section D, Structural biology. 76 (8), 771-777 (2020).
- Ng, J. T., Dekker, C., Kroemer, M., Osborne, M., Von Delft, F. Using textons to rank crystallization droplets by the likely presence of crystals. Acta Crystallographica. Section D, Biological Crystallography. 70, 2702-2718 (2014).
- Collins, P. M., et al. Gentle, fast and effective crystal soaking by acoustic dispensing. Acta Crystallographica. Section D, Structural Biology. 73, 246-255 (2017).
- Wright, N. D., et al. The low-cost Shifter microscope stage transforms the speed and robustness of protein crystal harvesting. Acta Crystallographica. Section D, Structural Biology. 77, 62-74 (2021).
- Krojer, T., et al. The XChemExplorer graphical workflow tool for routine or large-scale protein-ligand structure determination. Acta Crystallographica. Section D, Structural Biology. 73, 267-278 (2017).
- Pearce, N. M., et al. A multi-crystal method for extracting obscured crystallographic states from conventionally uninterpretable electron density. Nature Communications. 8, 15123 (2017).
- . Fragalysis Available from: https://fragalysis.diamond.ac.uk (2021)
- . Diamond Light Source Ltd Available from: https://www.diamond.ac.uk/Instruments/Mx/Fragment-Screening.html (2021)
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7 (3), 2322-2330 (2016).
- Vonrhein, C., et al. Data processing and analysis with the autoPROC toolbox. Acta Crystallographica. Section D, Biological Crystallography. 67, 293-302 (2011).
- Vonrhein, C., et al. Advances in automated data analysis and processing within autoPROC , combined with improved characterisation, mitigation and visualisation of the anisotropy of diffraction limits using STARANISO. Acta Crystallographica Section A: Foundations and Advances. 74 (1), 360 (2018).
- Pearce, N. M., Krojer, T., Von Delft, F. Proper modelling of ligand binding requires an ensemble of bound and unbound states. Acta Crystallographica. Section D, Structural Biology. 73, 265-266 (2017).
- Collins, P. M., et al. Achieving a good crystal system for crystallographic x-ray fragment screening. Methods in Enzymology. 610, 251-264 (2018).
- Delageniere, S., et al. ISPyB: an information management system for synchrotron macromolecular crystallography. Bioinformatics. 27 (22), 3186-3192 (2011).
- Fisher, S. J., Levik, K. E., Williams, M. A., Ashton, A. W., McAuley, K. E. SynchWeb: a modern interface for ISPyB. Journal of Applied Crystallography. 48, 927-932 (2015).
- Ginn, H. M., et al. SynchLink: an iOS app for ISPyB. Journal of Applied Crystallography. 47, 1781-1783 (2014).
- . Diamond Light Source Ltd Available from: https://www.diamond.ac.uk/Instruments/Mx/Common/Common-Manual/Data-Analysis/Reprocessing-in-ISPyB.html (2021)
- Wojdyr, M., Keegan, R., Winter, G., Ashton, A. DIMPLE - a pipeline for the rapid generation of difference maps from protein crystals with putatively bound ligands. Acta Crystallographica. Section A, Foundations of Crystallography. 69, 299 (2013).
- Long, F., et al. AceDRG: A stereochemical description generator for ligands. Acta Crystallographica. Section D, Structural Biology. 73, 112-122 (2017).
- Moriarty, N. W., Grosse-Kunstleve, R. W., Adams, P. D. Electronic ligand builder and optimization workbench (eLBOW): A tool for ligand coordinate and restraint generation. Acta Crystallographica. Section D, Biological Crystallography. 65, 1074-1080 (2009).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallographica. Section D, Biological Crystallography. 66, 486-501 (2010).
- Murshudov, G. N., Vagin, A. A., Dodson, E. J. Refinement of macromolecular structures by the maximum-likelihood method. Acta Crystallographica. Section D, Biological Crystallography. 53, 240-255 (1997).
- Bricogne, G., et al. Buster version 2.10.3. Global Phasing Ltd. , (2017).
- Chen, V. B., et al. MolProbity: All-atom structure validation for macromolecular crystallography. Acta Crystallographica. Section D, Biological Crystallography. 66, 12-21 (2010).
- Bruno, I. J., et al. Retrieval of crystallographically-derived molecular geometry information. Journal of Chemical Information and Computer Sciences. 44 (6), 2133-2144 (2004).
- Delbart, F., et al. An allosteric binding site of the α7 nicotinic acetylcholine receptor revealed in a humanized acetylcholine-binding protein. TheJournal of Biological Chemistry. 293, 2534-2545 (2018).
- Douangamath, A., et al. Crystallographic and electrophilic fragment screening of the SARS-CoV-2 main protease. Nature Communications. 11 (1), 5047 (2020).
- Guo, J., et al. In crystallo-screening for discovery of human norovirus 3C-like protease inhibitors. Journal of Structural Biology: X. 4, 100031 (2020).
- Keedy, D. A., et al. An expanded allosteric network in PTP1B by multitemperature crystallography, fragment screening, and covalent tethering. eLife. 7, 36307 (2018).
- McIntyre, P. J., et al. Characterization of three druggable hot-spots in the aurora-a/tpx2 interaction using biochemical, biophysical, and fragment-based approaches. ACS Chemical Biology. 12 (11), 2906-2914 (2017).
- Thomas, S. E., et al. Structure-guided fragmentbased drug discovery at the synchrotron: Screening binding sites and correlations with hotspot mapping. Philosophical Transactions. Series A, Mathematical, Physical and Engineering Sciences. 377 (2147), 20180422 (2019).
- Nichols, C., et al. Mining the PDB for tractable cases where x-ray crystallography combined with fragment screens can be used to systematically design protein-protein inhibitors: Two test cases illustrated by IL1β-IL1R and p38α-TAB1 complexes. Journal of Medicinal Chemistry. 63 (14), 7559-7568 (2020).
- D'Arcy, A., Bergfors, T., Cowan-Jacob, S. W., Marsh, M. Microseed matrix screening for optimization in protein crystallization: What have we learned. Acta Crystallographica. Section F, Structural Biology Communications. 70, 1117-1126 (2014).
- Wollenhaupt, J., et al. F2X-Universal and F2X-Entry: Structurally diverse compound libraries for crystallographic fragment screening. Structure. 28 (6), 694-706 (2020).
- . Zenodo Available from: https://zenodo.org (2021)
- Foley, D. J., et al. Synthesis and demonstration of the biological relevance of sp(3) -rich scaffolds distantly related to natural product frameworks. Chemistry. 23 (60), 15227-15232 (2017).
- Kidd, S. L., et al. Demonstration of the utility of DOS-derived fragment libraries for rapid hit derivatisation in a multidirectional fashion. Chemical Science. 11 (39), 10792-10801 (2020).
- . EU-openscreen ERIC Available from: https://www.eu-openscreen.eu/ (2021)
- Schuller, M., et al. Fragment binding to the Nsp3 macrodomain of SARS-CoV-2 identified through crystallographic screening and computational docking. bioRxiv. 393405, (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten