Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Bakterielle Expression und Aufreinigung der humanen Matrix-Metalloproteinase-3 mittels Affinitätschromatographie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
His-Tag-Reinigung, Dialyse und Aktivierung werden eingesetzt, um die Ausbeute an löslicher, aktiver Matrix-Metalloproteinase-3-katalytischer Domänenproteinexpression in Bakterien zu erhöhen. Proteinfraktionen werden über SDS-PAGE-Gele analysiert.
Zusammenfassung
Matrix-Metalloproteinasen (MMPs) gehören zur Familie der Metzincinproteasen mit zentralen Rollen beim Abbau und Remodeling der extrazellulären Matrix (ECM) sowie bei Wechselwirkungen mit mehreren Wachstumsfaktoren und Zytokinen. Die Überexpression spezifischer MMPs ist für verschiedene Krankheiten wie Krebs, neurodegenerative Erkrankungen und Herz-Kreislauf-Erkrankungen verantwortlich. MMPs standen in letzter Zeit im Mittelpunkt der Aufmerksamkeit als Ziele für die Entwicklung von Therapeutika, die Krankheiten behandeln können, die mit MMP-Überexpression korrelieren.
Um den MMP-Mechanismus in Lösung zu untersuchen, sind einfachere und robustere rekombinante Proteinexpressions- und Reinigungsmethoden für die Herstellung von aktiven, löslichen MMPs erforderlich. Der katalytische Bereich der meisten MMPs kann jedoch in Escherichia coli (E. coli) aufgrund fehlender posttranslationaler Mechanismen nicht in löslicher Form exprimiert werden, während Expressionssysteme für Säugetiere in der Regel teuer sind und geringere Erträge aufweisen. MMP-Einschlussgremien müssen den langwierigen und mühsamen Prozess der umfangreichen Reinigung und Wiederfaltung durchlaufen, wodurch die Ausbeute an MMPs in der nativen Konformation erheblich reduziert wird. Dieses Papier stellt ein Protokoll vor, das Rosetta2 (DE3) pLysS-Zellen (im Folgenden als R2DP bezeichnet) verwendet, um eine katalytische Matrix-Metalloproteinase-3-Domäne (MMP-3cd) zu erzeugen, die einen N-terminalen His-Tag gefolgt von einer Pro-Domäne (Hisx6-pro-MMP-3cd) für die Verwendung in der Affinitätsreinigung enthält. R2DP-Zellen verstärken die Expression eukaryotischer Proteine durch ein Chloramphenicol-resistentes Plasmid, das Codons enthält, die normalerweise in bakteriellen Expressionssystemen selten sind. Im Vergleich zur traditionellen Zelllinie der Wahl für die rekombinante Proteinexpression, BL21(DE3), verbesserte die Aufreinigung mit diesem neuen Stamm die Ausbeute an gereinigtem Hisx6-pro-MMP-3cd. Nach der Aktivierung und Entsalzung wird die Pro-Domäne zusammen mit dem N-terminalen His-Tag gespalten, wodurch aktives MMP-3cd für den sofortigen Einsatz in unzähligen In-vitro-Anwendungen bereitgestellt wird. Diese Methode erfordert keine teure Ausrüstung oder komplexe Fusionsproteine und beschreibt die schnelle Produktion von rekombinanten menschlichen MMPs in Bakterien.
Einleitung
Die meisten komplexen eukaryotischen Proteine durchlaufen nach der Expression ausgeklügelte posttranslationale Modifikationen, die eine hochunterstützte Proteinfaltung und Co-Faktoren erfordern, um funktionsfähig zu sein1. Die Herstellung großer Mengen löslichen menschlichen Proteins in einem bakteriellen Wirt bleibt aufgrund der hohen Kosten und des Mangels an robusten Expressions- und Reinigungsmethoden eine erhebliche Herausforderung, selbst für kleinere Laborexperimente2,3. MMPs, humane Endopeptidasen mit großem Molekulargewicht, werden normalerweise als unlösliche Einschlusskörper exprimiert, wenn sie in E. coli exprimiert werden. Die Extraktion von löslichen humanen MMPs führt oft zu einem mühsamen, zeitaufwändigen Solubilisierungs- und Umfaltungsprozess4.
MMPs spielen eine entscheidende Rolle sowohl in physiologischen als auch in pathogenen Prozessen. Humane MMPs sind eine Familie von 23 Zink-Endopeptidasen, kategorisiert nach Struktur- und Substratspezifität und differentiell exprimiert trotz einer hochkonservierten katalytischen Domäne5,6. MMPs werden als inaktive Zymogene sezerniert, reguliert durch posttranslationale Aktivierung und ihre endogenen Inhibitoren, Gewebeinhibitoren von Metalloproteinasen (TIMPs)7,8,9,10. Obwohl MMPs ursprünglich für ihre Rolle beim ECM-Umsatz anerkannt waren, waren sie auch an der Entwicklung, Morphogenese, Gewebereparatur und -umgestaltung beteiligt8. Die Dysregulation von MMPs wurde unter anderem mit Krebs sowie neurodegenerativen, kardiovaskulären und fibrotischen Erkrankungen in Verbindung gebracht5,7.
Die Entwicklung robuster MMP-Produktionsmethoden im großen Maßstab ist entscheidend, um den Erfolg zukünftiger Studien von MMP-Mechanismen durch biochemische und zellbasierte Assays sicherzustellen. Verschiedene MMPs wurden zuvor in Bakterien exprimiert11, einschließlich Hisx6-markierter MMPs, ohne die MMP-Aktivität zu verändern12,13,14,15. Diese Methoden umfassen jedoch mühsame, lange Schritte, die möglicherweise schwer zu replizieren sind.
Säugetierzellen können auch verwendet werden, um viele verschiedene menschliche Proteine zu exprimieren und gleichzeitig die richtigen posttranslationalen Modifikationen zu gewährleisten16. Obwohl das Expressionssystem für Säugetiere eine ideale Wahl ist, um rekombinante menschliche Proteine mit geeigneten posttranslationalen Modifikationen herzustellen, sind die Hauptnachteile dieser Methode anfänglich niedrige Ausbeuten, kostspielige Wachstumsmedien und Reagenzien, lange Zeiträume zum Erreichen stabiler Mimiklinien und das Risiko einer Kontamination mit anderen Spezies wie Pilzen oder Bakterien2,11 . Darüber hinaus führt die MMP-Produktion in Zelllinien von Säugetieren zu Verunreinigungen aus assoziierten zellulären Proteinen wie TIMPs oder Fibronektinen11. Im Gegensatz zu dem langsamen Zellwachstum, das in Säugetierzellen beobachtet wird, bietet das bakterielle Expressionssystem eine groß angelegte Proteinproduktion in kurzer Zeit sowie einfachere Medien- und Wachstumsanforderungen. Aufgrund des Fehlens anderer assoziierter zellulärer Proteine (d. h. TIMPs) in bakteriellen Expressionssystemen unterliegen aktive MMPs in höheren Konzentrationen jedoch dem Abbau durch Autoproteolyse, was zu einer schlechten MMP-Ausbeute führt17.
Dieses Papier beschreibt eine detaillierte Methode zur bakteriellen Expression, Reinigung und Aktivierung von rekombinantem Hisx6-pro-MMP-3cd unter Verwendung von E. coli als Ausdruckshost aufgrund seiner Erschwinglichkeit, Einfachheit und seines Erfolgs bei der Erzeugung höherer Ausbeuten von MMPs2,3,18. Da E. coli nicht über die Proteinfaltungsmaschinerie und die posttranslationale Verarbeitung verfügt, die für rekombinante MMPs und andere komplexe Proteine erforderlich sind, wurden viele E. coli-Stämme entwickelt, um diese Einschränkungen zu überwinden, was E. coli zu einem geeigneteren Wirt für die Expression von rekombinantem humanem MMP-3cd macht,19,20 . Zum Beispiel verbessert der in dieser Studie verwendete R2DP-Stamm die eukaryotische Expression, indem er ein chloramphenicolresistentes Plasmid liefert, das Codons enthält, die selten in E. coli verwendet werden.
Wie in diesem Protokoll beschrieben, werden nach Überexpression von relativ reinen Einschlusskörpern aus dem pET-3a-Vektor (Abbildung 1) in R2DP-Zellen Hisx6-pro-MMP-3-Proteine (MMP-3cd) extrahiert und denaturiert4. Hisx6-pro-MMP-3cd3,19 wurde mittels Affinitäts-Tag-Chromatographie gereinigt. Nach der Rückfaltung und Dialyse wurde das Pro-MMP-3cd (Zymogen) durch 4-Aminophenylquecksilberacetat (APMA) aktiviert, und die SDS-PAGE-Analyse wird verwendet, um die Ausbeuten und die Notwendigkeit einer weiteren Reinigung zu bewerten5,21. Dieses Protokoll beschreibt als Beispiel den Ausdruck, die Reinigung und die Aktivierung von löslichem MMP-3cd. Es kann jedoch auch als Leitfaden für die Expression anderer MMPs und menschlicher Proteasen mit ähnlicher Expression und Aktivierungsmechanismen verwendet werden (Abbildung 2). Für andere Proteine als MMP-3cd wird dem Leser empfohlen, optimale Pufferzusammensetzungen und -methoden für sein Zielprotein zu bestimmen, bevor er dieses Protokoll versucht.
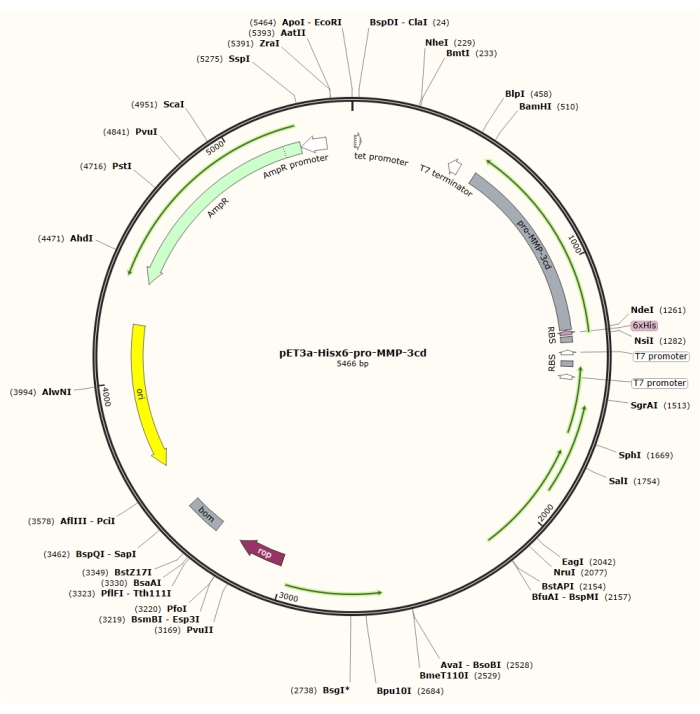
Abbildung 1: Plasmidabbildung des pET-3a-Hisx6-pro-MMP-3cd-Plasmids. Der pET-3a-Vektor enthält ein Ampicillin-Resistenz-Gen. Eine N-terminale Hisx6-Tag-Sequenz wird in den pET-3a-basierten Vektor, einschließlich pro-MMP-3cd, geklont, um das pET-3a-Hisx6-pro-MMP-3cd-Konstrukt unter Kontrolle des T7-Promotors zwischen BamHI- und NdeI-Restriktionssites zu erhalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Bakterielle Expression von Pro-MMP-3cd, Reinigung, Rückfaltung und Aktivierung . 1.1: pET-3a-Hisx6-pro-MMP-3cd-Plasmid wurde in BL21(DE3)- oder R2DP-Zellen umgewandelt. 1.2: Die Pro-MMP-3cd-Proteinexpression wurde mittels IPTG induziert. 1.3: Chemische Lyse und Beschallung werden verwendet, um Hisx6-pro-MMP-3cd-Proteine zu extrahieren, die hauptsächlich unlöslich sind und in den Einschlusskörpern vorkommen. Harnstoff wurde verwendet, um Protein aus Einschlusskörpern zu denaturieren und zu lösen. 2.1. Das denaturierte Hisx6-pro-MMP-3cd-Protein wurde durch affinitätschromatographische Reinigung gereinigt. 3. Die eluierte Hisx6-pro-MMP-3cd wurde während der Dialyse durch allmähliche Entfernung von Harnstoff aus dem Puffer langsam wieder gefaltet. 4. Schließlich wurde das gefaltete MMP-3cd-Protein mit APMA aktiviert, indem die N-terminale Pro-Peptid-Domäne entfernt wurde. APMA wird später durch Entsalzen aus der Lösung entfernt. Die Zahlen entsprechen den Protokollabschnitten, die diese Schritte beschreiben. Abkürzungen: MMP-3cd = Matrix-Metalloproteinase-3-katalytische Domäne; APMA = 4-Aminophenylquecksilberacetat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
1. MMP-Ausdruck
- Klonen und Transformation von pET-3a-Hisx6-pro-MMP-3cd in R2DP-Zellen
- Fermentieren Sie das pET-3a-Plasmid (siehe Materialtabelle) mit NdeI- und BamHI-Restriktionsenzymen im Digest-Puffer (siehe Materialtabelle). Bei einem Gesamtreaktionsvolumen von 40 μL werden 4 μL Digest-Puffer, 33 μL 100 ng/μL Plasmid und 1,5 μL jedes Restriktionsenzyms zugegeben und die Reaktion ~2 h bis zum Abschluss bei 37 °C fortgesetzt.
- Führen Sie eine PCR-Reaktion auf der MMP-3cd-Sequenz durch, um einen N-terminalen His-Tag einzufügen. Verwenden Sie 25 μL PCR-Mix (siehe Materialtabelle), 2,5 μL 10 μM Primer (Ergänzende Abbildung S1) und 1,25 μL der 100 ng/μL Einlegesequenz. Steriles Wasser zu einem endgültigen Reaktionsvolumen von 50 μL hinzufügen.
- Führen Sie das PCR-Produkt und den verdauten Vektor auf einem 1% igen Agarosegel aus. Reinigen Sie die Gelbänder mit einem Gel Recovery Kit (siehe Materialtabelle) gemäß dem Protokoll des Herstellers.
- Klonen Sie das amplifizierte PCR-Produkt mit DNA Assembly Mix in den verdauten Vektor zwischen den NdeI- und BamHI-Restriktionsstellen (siehe Materialtabelle). Verwenden Sie Online-Tools, um das erforderliche Volumen des Insert- und Cut-Vektors für ein Gesamtreaktionsvolumen von 15 μL zu bestimmen.
- Tauen Sie ein 50 μL Aliquot von Zellen mit hohem Transformationswirkungsgrad (siehe Materialtabelle) auf Eis auf, bis es aufgetaut ist. Vorgewärmtes SOC-Wachstumsmedium (siehe Materialtabelle) bis 37 °C und LB-Ampicillinplatten (LB Ampere) (siehe Materialtabelle).
- Fügen Sie 1-2 μL der pET-3a-Hisx6-pro-MMP-3cd-Montagereaktion zum 50 μL Aliquot hinzu. 30 min auf Eis inkubieren.
- Hitzeschock der Zellen durch Inkubation bei 42 °C für 30 s. 2 min auf Eis inkubieren.
- 950 μL SOC-Wachstumsmedium zu jeder Transformatorenmischung hinzufügen. 1 h bei 250 U/min und 37 °C schütteln.
- Platte 100 μL der Transformatoren auf LB-Ampereplatten und Inkubation über Nacht bei 37 °C.
- Beimpfen Sie jede isolierte Kolonie in 10 ml LB Ampere-Medium. Über Nacht bei 250 U/min und 37 °C schütteln.
- Extrahieren Sie Plasmid-DNA gemäß dem Protokoll des Herstellers für das Miniprep-Kit (siehe Materialtabelle). Bestätigen Sie die Reihenfolge des Konstrukts mit T7-Vorwärts- und Rückwärtsprimern (Ergänzende Abbildung S1).
HINWEIS: Die pET-3a-Hisx6-pro-MMP-3cd-Konstrukt-DNA kann bei -20 °C gespeichert werden. Wenn Sie fertig sind, fahren Sie mit der Umwandlung in R2DP-Zellen fort. - Ein 20 μL Aliquot R2DP-Zellen (siehe Materialtabelle) auf Eis für 2-5 min auftauen. Vorwärmendes SOC-Wachstumsmedium auf Raumtemperatur und LB Amp CamR-Platten auf 37 °C (siehe Materialtabelle).
- Fügen Sie 1 μL 100 ng/μL sequenzbestätigtes pET-3a-Hisx6-pro-MMP-3cd zum 50 μL Aliquot hinzu. Rühren Sie vorsichtig um, um zu mischen und die Tube wieder zu Eis zu machen.
- Brüten Sie die Röhre auf Eis für 5 min.
- Hitzeschock der Zellen durch Inkubation bei 42 °C für genau 30 s. Nicht schütteln.
- Legen Sie die Zellen für 2 min auf Eis.
- Zugabe von 80 μL SOC-Medium bei Raumtemperatur zu der Transformatormischung. 1 h bei 250 U/min und 37 °C schütteln.
- Die Transformatoren werden auf LB Amp CamR-Platten plattiert und über Nacht bei 37 °C inkubiert.
- Wachstum und Induktion
- Beimpfen Sie eine einzelne, isolierte Kolonie R2DP pET-3a-Hisx6-pro-MMP-3cd-Transformant aus einer LB Amp CamR-Platte in 10 ml LB Amp CamR-Medien bei 37 °C. Über Nacht (~16 h) bei 250 U/min schütteln. Sparen Sie Aliquots aus jeder Kultur und bereiten Sie auf Wunsch 40% (v/v) Glycerin (siehe Materialtabelle) vor.
- Pro Nachtkultur wird ein 1-L-Kolben mit 500 ml LB Amp CamR-Medium auf eine optische Dichte bei 600 nm (OD600) von 0,05-0,1 geimpft.
HINWEIS: Dies sollte die Zellen zum logarithmischen Wachstum zurückbringen. - Messen Sie den OD600 an mehreren Zeitpunkten, typischerweise für 3-4 h, bis er zwischen 0,4 und 0,6 fällt.
- Vor der Induktion aliquotieren Sie einen Teil der Kultur in ein 1,5-ml-Mikrofugenröhrchen (siehe Materialtabelle) und kennzeichnen Sie es als uninduzierte Fraktion. Lagern Sie es bei -80 ° C für die Gelanalyse. Wenn Sie kein SDS-PAGE-Gel ausführen, überspringen Sie diesen Schritt und fahren Sie mit Schritt 1.2.5 fort.
- Bringen Sie die Kulturen zu einer Endkonzentration von 1 mM unter Verwendung von 1 M Isopropyl-ß-D-thiogalactopyranosid (IPTG)-Material (siehe Materialtabelle) induzieren. Inkubieren Sie im 37 °C Shaker für weitere 3-4 h weiter.
HINWEIS: Während der Expression sollte das Lesegerät den optimalen OD600 zum Zeitpunkt der Induktion und die IPTG-Konzentration bestimmen. Wenn die Ausbeute nach der Reinigung erheblich abnimmt, muss die Imidazolkonzentration in Reinigungspuffern möglicherweise angepasst werden, oder das Zellpellet muss möglicherweise weiter beschallt werden. - Bevor Sie die Kulturen zentrifugieren, aliquotieren Sie einen Teil der Kultur in ein zweites 1,5-ml-Mikrofugenröhrchen und kennzeichnen Sie es als induzierte Fraktion. Lagern Sie es bei -80 ° C für die Gelanalyse. Wenn Sie kein SDS-PAGE-Gel ausführen, überspringen Sie diesen Schritt und fahren Sie mit Schritt 1.2.7 fort.
- Zentrifugieren Sie die Zellkultur in 250 ml konischen Flaschen (siehe Materialtabelle) bei maximaler Geschwindigkeit und 4 °C für 10 min.
- Wiederholen Sie Schritt 1.2.7, bis die Kulturen vollständig pelletiert sind.
HINWEIS: PAUSE: Zellpellets können bei -80 °C eingefroren und später zur Weiterverarbeitung aufgetaut werden. Überspringen Sie andernfalls diesen Schritt und fahren Sie mit Schritt 1.3.1 fort.
- Einschlusskörperextraktion und Solubilisierung
HINWEIS: Frischen 10 M Harnstoff zubereiten, vorzugsweise frühestens einen Tag im Voraus, gründlich umrühren, bis er vollständig aufgelöst ist. Erhitzen oder autoklavieren Sie Harnstoff nicht; Lagern Sie es bei Raumtemperatur.- Resuspendiert das Pellet (ab Schritt 1.2.8) im Lysepuffer (siehe Materialtabelle). Pro Gramm Pellet 3 ml Lysepuffer hinzufügen und durch Vortexen oder Pipettieren resuspendieren. Über Nacht bei 4 °C schütteln.
- Fügen Sie 1,25 ml 10% (w/v) Natriumdesoxycholat (siehe Materialtabelle) pro 1 l Kultur hinzu. Bei Raumtemperatur 30 min bei 150 U/min schütteln.
- Fügen Sie 10 μL DNase I (siehe Materialtabelle) pro 1 L Kultur hinzu. Bei Raumtemperatur 30 min bei 150 U/min schütteln.
- Zentrifuge für 10 min bei 13.000 × g und 4 °C.
- Legen Sie einen Bruchteil von Lysed MMP für die Gelanalyse beiseite. Lagern Sie es bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 1.3.6 fort.
HINWEIS: Nach der Zentrifugation kann das Pellet strähnig und nicht kompakt verpackt sein, was es riskant macht, den Überstand zu entsorgen. Wenn dies der Fall ist, überspringen Sie Schritt 1.3.6 und fahren Sie mit Schritt 1.3.7 fort. - Entsorgen Sie den Überstand aus den zentrifugierten Proben.
HINWEIS: Das Protokoll kann an dieser Stelle pausiert und die Zellpellets bei -80 °C eingefroren und später aufgetaut werden. Überspringen Sie andernfalls diesen Schritt und fahren Sie mit Schritt 1.3.7 fort. - Resuspendiert das Pellet in 100 ml/L Kultur des Einschlusskörperpuffers (siehe Materialtabelle) durch Pipettieren nach oben und unten.
- Halten Sie die Proben während der Beschallung auf Eis, um eine Überhitzung zu vermeiden. Beschallen Sie jede Probe für 6 Zyklen von 15 s, Ausgabe 5 und 50% Puls. Erlauben Sie 15 s Ruhezeiten für die Kühlung zwischen den Zyklen.
HINWEIS: Falls erforderlich, geben Sie die Proben zur weiteren Zentrifugation in konische 50-ml-Röhrchen (siehe Materialtabelle). Zentrifuge für 10 min bei 13.000 × g und 4 °C. - Legen Sie einen Bruchteil des beschallten MMP für die Gelanalyse beiseite. Lagern Sie es bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 1.3.11 fort.
- Überprüfen Sie das Pellet. Wiederholen Sie bei Stringy die Schritte 1.3.8-1.3.10. Wenn das Pellet kompakt ist, entsorgen Sie den Überstand und fahren Sie mit Schritt 1.3.12 fort.
HINWEIS: Die Beschallung im Einschlusskörperpuffer kann wiederholt werden, um mehr Protein aus den lysierten Zelltrümmern zurückzugewinnen. Zu viel Beschallung kann jedoch zu Scherungen führen, was die MMP-Ausbeute beeinträchtigt. Das Protokoll kann in diesem Stadium pausiert werden, und die Zellpellets können bei -80 ° C eingefroren und später aufgetaut werden. - Resuspendiert jedes Pellet aus einer 1-L-Kultur in 5 ml Solubilisierungspuffer (siehe Materialtabelle) durch Pipettieren. Inkubieren Sie für mindestens 30 Minuten auf Eis, damit die Proteine auflösen können.
- Legen Sie einen Bruchteil des löslichen MMP für die Gelanalyse beiseite. Lagern Sie es bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 1.3.14 fort.
- Zentrifugieren Sie die Zellen für 10 min bei 13.000 × g und 4 °C. VERWERFEN SIE DEN ÜBERSTAND NICHT.
- Wenn sich nach der Zentrifugation ein Pellet bildet / verbleibt, gießen Sie den Überstand in ein separates konisches 50-ml-Rohr. Resuspendiert das Pellet in weiteren 5 ml Lösungsmittelpuffer (pro 1 l Kultur), indem es nach oben und unten pipettiert wird.
- Zentrifuge für 10 min bei 13.000 × g und 4 °C. VERWERFEN SIE DEN ÜBERSTAND NICHT.
- Wiederholen Sie die Schritte 1.3.13 und 1.3.14, bis sich nach der Zentrifugation etwas bis gar kein Pellet bildet oder nur noch grauer Niederschlag übrig bleibt. Poolen Sie die Überstände. Entsorgen oder lagern Sie das Pellet bei -80 °C zur zusätzlichen Beschallung.
2. MMP-Reinigung und -Rückfaltung
- His-Tag (HT) Affinitätsreinigung
- Füllen Sie gemäß dem Protokoll des Herstellers eine Schwerkraftflusssäule (siehe Materialtabelle) mit gut gemischtem Ni-NTA-Harz (siehe Materialtabelle). Lassen Sie das Harz absetzen und trennen Sie sich vom Speicherpuffer, so dass sich eine deutliche Linie zwischen den beiden Schichten bildet.
HINWEIS: Lassen Sie das Harz niemals trocknen, da Luft in das Harz eindringen und die Proteinausbeute beeinträchtigen wird. Führen Sie zwischen den Anwendungen das in Abschnitt 2.2 beschriebene Verfahren zur Harzregeneration durch. - Lassen Sie den Speicherpuffer entleeren. Füllen Sie die Spalte mit zwei Harzbettvolumina des HT-Gleichgewichtspuffers.
- Entleeren Sie den HT-Gleichgewichtspuffer und verwerfen Sie ihn. Während die Säule entwässert, zentrifugieren Sie den Proteinextrakt bei 13.000 × g für 1 min und filtersterilisieren Sie ihn mit einem 0,22-μm-Filter (siehe Materialtabelle).
- Tauschen Sie den Abfallbehälter gegen ein konisches 50-ml-Rohr mit der Bezeichnung HT Flowthrough aus. Fügen Sie den vorbereiteten Proteinextrakt zur Säule hinzu.
- Wenden Sie den Durchfluss erneut an, um die Bindung zu maximieren.
- Legen Sie einen Bruchteil der Durchflussfraktion für die Gelanalyse beiseite. Lagern Sie es bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 2.1.7 fort.
- Waschen Sie das Harz sofort mit 15 ml HT Wash Buffer (siehe Materialtabelle). Sammeln Sie den Durchfluss in 15 ml konischen Röhrchen (siehe Materialtabelle) mit der Aufschrift HT Wash.
HINWEIS: Absorptionswerte bei 280 nm (A280) wurden mittels Spektrophotometrie ermittelt und zusammen mit Molekulargewicht und Extinktionskoeffizient ε verwendet, um die Proteinkonzentrationen abzuschätzen. Für denaturierte Hisx6-pro-MMP-3cd beträgt das Molekulargewicht 29,86 kDa und ε 34,38 M-1 cm-1. - Blanking gegen HT Wash Buffer, messen und aufzeichnen Sie den A280. Wiederholen Sie die Schritte 2.1.7 und 2.1.8 mit zusätzlichen Waschfraktionen. Sobald sich der A280 dem Ausgangswert genähert hat und die Verunreinigungen minimiert wurden, fahren Sie mit Schritt 2.1.9 fort.
- Legen Sie einen Teil der Waschfraktion für die Gelanalyse beiseite. Wiederholen Sie dies für mehrere Waschfraktionen. Lagern Sie die Fraktionen bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 2.1.10 fort.
- Sofort eluieren Sie His-markierte Proteine durch Zugabe von 5 ml HT Elution Buffer (siehe Materialtabelle). Sammeln Sie den Durchfluss als 0,5-1 ml-Fraktionen in Mikrofugenröhrchen, die mit HT Elution gekennzeichnet sind.
- Legen Sie eine Fraktion der Elution-Fraktion für die Gelanalyse beiseite. Wiederholen Sie dies für mehrere Elutionsfraktionen. Lagern Sie die Fraktionen bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 2.1.12 fort.
- Wenn der A280 >0,3 mg/ml beträgt, verdünnen Sie die Fraktion mit HT-Gleichgewichtspuffer (siehe Materialtabelle).
HINWEIS: Die elastische Fraktion muss auf einen A280 von 0,3 mg / ml oder weniger verdünnt werden, um eine Ausfällung während der Dialyse zu verhindern. Hier kann das Protokoll pausiert und die gepoolten Fraktionen bei -80 °C eingefroren und später aufgetaut werden. Überspringen Sie andernfalls diesen Schritt und fahren Sie mit Schritt 2.2.1 fort.
- Füllen Sie gemäß dem Protokoll des Herstellers eine Schwerkraftflusssäule (siehe Materialtabelle) mit gut gemischtem Ni-NTA-Harz (siehe Materialtabelle). Lassen Sie das Harz absetzen und trennen Sie sich vom Speicherpuffer, so dass sich eine deutliche Linie zwischen den beiden Schichten bildet.
- Harz-Regeneration
- Waschen Sie das Harz mit zehn Harzbettvolumina HT-Regenerationspuffer (siehe Materialtabelle) und zehn Harzbettvolumina mit sterilem Wasser.
- Lagern Sie das Harz als 50% ige Aufschlämmung in 20% (v / v) Ethanol in Wasser.
3. Protein-Refolding
HINWEIS: Für kleinere Volumina können Dialysekassetten mit einem geringeren Risiko von Probenverlust verwendet werden. Dialyseschläuche sind erforderlich, wenn größere Volumina verwendet werden (siehe Materialtabelle).
- Dialyse
HINWEIS: Die optimale Proteinkonzentration für die Dialyse beträgt ~ 0,3 mg / ml. Wenn während der Dialyse signifikante Ausfällungen auftreten, reduzieren Sie den Harnstoffkonzentrationsgradienten zwischen den einzelnen Dialysen mit einer schrittweisen Dialysemethode und fügen Sie weitere Zwischenschritte hinzu (z. B. von 6 M bis 5 M und dann 5 M bis 4 M, anstatt die 5 M-Stufe zu überspringen). Da Gefrier-Tau-Zyklen die Zell- und Proteinstruktur schädigen, ist es wichtig, die Pausen im Protokoll zu minimieren.- Verwenden Sie gemäß dem Protokoll des Herstellers die entsprechende Menge an Dialyseschläuchen entsprechend dem Volumen der Elution Fraction-Proben .
- Tauchen Sie die elastischen MMP-Fraktionen in Dialyseschläuche in 1 L Dialysepuffer 1 (siehe Materialtabelle). Rühren Sie den Schlauch und seinen Inhalt auf einem Magnetrührer für mindestens 8 h bei 4 °C um.
- Transfer in 1 l Dialysepuffer 2 (siehe Materialtabelle). Rühren Sie den Schlauch und seinen Inhalt auf einem Magnetrührer für mindestens 8 h bei 4 °C um.
- Transfer in 1 l Dialysepuffer 3 (siehe Materialtabelle). Rühren Sie den Schlauch und seinen Inhalt auf einem Magnetrührer für mindestens 8 h bei 4 °C um.
- Die Probe in neue konische 50-ml-Röhrchen überführen und als dialysiertes MMP kennzeichnen.
- Untersuchen Sie das Rohr auf Niederschlag. Wenn sich Niederschlag gebildet hat, zentrifugieren Sie die Probe für 1 min bei 13.000 × g und 4 °C.
- Überfüllen Sie den Überstand in neue 15 ml konische Röhrchen und kennzeichnen Sie diese als Refolded MMP.
- Legen Sie eine Fraktion für die Gelanalyse beiseite und kennzeichnen Sie sie als Refolded MMP. Lagern Sie es bei -80 °C. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 3.1.9 fort.
HINWEIS: Wenn die Ausbeute gering ist, kann der Niederschlag im HT-Gleichgewichtspuffer aufgelöst und die Schritte in Abschnitt 3.1 mit Dialyseschläuchen wiederholt werden. Soll keine Gelanalyse durchgeführt werden oder muss hier das Protokoll pausiert werden, frieren Sie die Proben bei -80 °C ein und tauen Sie sie später auf. Wenn die Erträge im gewünschten Bereich liegen, fahren Sie mit Schritt 3.2.1 fort.
- Rekonzentration
HINWEIS: Die Extinktionskoeffizienten für gefaltete und denaturierte Hisx6-pro-MMP-3cd werden voraussichtlich gleich sein; Daher sind A280-Berechnungen nicht betroffen.- Die Probe wird auf bis zu 0,5 mg/ml neu konzentriert. Verwenden Sie eine 400 ml Rührzelle (siehe Materialtabelle), um die Probe auf 15 ml zu konzentrieren. Um Schaumbildung zu vermeiden, verwenden Sie ein 50-ml-Rekonzentrationsröhrchen, um sich bei Bedarf weiter zu konzentrieren.
HINWEIS: Wenn sich ein Niederschlag bildet, kann er pelletiert und im HT-Gleichgewichtspuffer gelöst werden. Wiederholen Sie dann die Abschnitte 3.1 und 3.2.1. Fahren Sie andernfalls mit Schritt 3.2.2 fort. - Legen Sie eine Fraktion für die Gelanalyse beiseite und kennzeichnen Sie sie als Concentrated MMP.
HINWEIS: Das Protokoll kann hier pausiert und die Proben bei -80 °C eingefroren und später aufgetaut werden.
- Die Probe wird auf bis zu 0,5 mg/ml neu konzentriert. Verwenden Sie eine 400 ml Rührzelle (siehe Materialtabelle), um die Probe auf 15 ml zu konzentrieren. Um Schaumbildung zu vermeiden, verwenden Sie ein 50-ml-Rekonzentrationsröhrchen, um sich bei Bedarf weiter zu konzentrieren.
4. Aktivierung
- 4-Aminophenylquecksilberacetat (APMA) Aktivierung
HINWEIS: APMA ist hochgiftig. Stellen Sie vor der Aktivierung eine frische Stammlösung von 20 mM APMA her und arbeiten Sie bei der Verwendung von APMA immer unter einem Abzug. Entsorgen Sie den APMA-Abfall in seinen Behälter.- Pro 1 mL Aliquot MMP (1 mg/ml) sind 50 μL 20 mM APMA (siehe Materialtabelle) hinzuzufügen, um eine endgültige APMA-Konzentration von 1 mM zu erreichen. Über Nacht bei 37 °C inkubieren.
- Wenn sich ein Niederschlag bildet, zentrifugieren Sie ihn mit maximaler Geschwindigkeit für 10 min bei 4 °C. Lagern Sie den Überstand in einem 1,5-ml-Mikrofugenröhrchen mit der Bezeichnung Activated MMP. Entsorgen Sie den Niederschlag in einen für APMA-Abfälle gekennzeichneten Behälter.
- Legen Sie eine Fraktion für die Gelanalyse beiseite und kennzeichnen Sie sie als aktiviertes MMP.
HINWEIS: Das Protokoll kann hier pausiert und die Proben bei -80 °C eingefroren und später aufgetaut werden. Wenn Sie keine Gelanalyse durchführen, fahren Sie mit Schritt 4.2.1 fort. Nach der Aktivierung betragen das Molekulargewicht und der Extinktionskoeffizient von MMP-3cd 19,40 kDa bzw. 28,42 M-1 cm-1.
- Entsalzung
- Entfernen Sie APMA aus der aktivierten MMP-3cd-Probe mit einer 2-ml-Entsalzungssäule (siehe Materialtabelle) gemäß dem Protokoll des Herstellers.
- Legen Sie eine Fraktion für die Gelanalyse beiseite und kennzeichnen Sie sie als entsalztes MMP. Lagern Sie es bei -80 °C. Wenn keine Gelanalyse durchgeführt wird, fahren Sie mit Abschnitt 4.3 mit den verbleibenden Proben fort.
HINWEIS: Das Protokoll kann hier pausiert und die Proben bei -80 °C eingefroren und später aufgetaut werden.
- Ausführen der SDS-PAGE Gele
- Führen Sie alle Proteinfraktionen auf SDS-PAGE-Gelen aus: Uninduzierte Fraktion, Induzierte Fraktion, Lysed MMP, Sonicated MMP, Solubilized MMP, Flowthrough Fraction, Wash Fraction, Elution Fraction, Refolded MMP, Concentrated MMP, Activated MMP und Desalted MMP.
- Langzeitlagerung von MMP-3
- Geben Sie 0,05% (v/v) nichtionisches Tensid (siehe Materialtabelle) zu den entsalzten MMP-3cd-Proben und lagern Sie sie bei -80 °C.
Ergebnisse
Wenn Proben auf SDS-PAGE ausgeführt werden, da das Protein in Form von unlöslichen Einschlusskörpern exprimiert wird, sollten die lysierten und beschallten Fraktionen wenig bis gar keinen Hisx6-pro-MMP-3cd-Extrakt enthalten, da das Protein noch nicht in Harnstoff resobilisiert wurde. Abbildung 3 vergleicht die His-tag-Reinigungselutionsfraktionen von Hisx6-pro-MMP-3cd aus BL21(DE3)-Zellen und R2DP-Zellen. Elution-Fraktionen wurden vor der Dialyse sowohl für BL21(DE3)- als auch für R2DP-...
Diskussion
Die großtechnische Produktion von löslichen, humanen, rekombinanten MMPs bleibt eine anspruchsvolle Aufgabe. Säugetierzellen können funktionelle MMPs mit hohen Kosten und langen Wartezeiten exprimieren, während E. coli schnell hohe Mengen an MMP-Einschlusskörpern produzieren, die gereinigt und wieder gefaltet werden müssen11,16. R2DP-Zellen erhöhen die Ausbeute von MMP-Einschlusskörpern signifikant und ermöglichen einen kostengünstigeren und p...
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Die Autoren möchten Dr. Evette Radisky und Alexandra Hockla von der Mayo Clinic in Jacksonville, Florida, für die Bereitstellung des pET-3a-pro-MMP-3cd-Plasmids als Vorlage für die Klonierung des Hisx6pro-MMP-3cd-Gens und ihre Kommentare sowie Dr. Paul Hartley vom Nevada Genomics Center an der University of Nevada, Reno, für die DNA-Sequenzierung danken. Die Autoren möchten auch Cassandra Hergenrader dafür danken, dass sie bei einem Teil der Proteinexpression geholfen hat. M.R.-S. möchte dem NIH-P20 GM103650-COBRE Integrative Neuroscience Grant und dem UNR R&D mICRO SEED Grant Award danken.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22 µm sterile filter | Sigma Aldrich | SLGP033RS | Used to remove some contaminants from the protein extract before purification, and prevent the Ni-NTA column from clogging |
| 1 L Erlenmeyer flasks | Thermo Fisher Scientific | S76106F | n/a |
| 1 L glass bottles | Thermo Fisher Scientific | 06-414-1D | n/a |
| 1.5 mL microfuge tubes | Thermo Fisher Scientific | 02-682-002 | n/a |
| 15 mL conical tubes | Thermo Fisher Scientific | 339650 | n/a |
| 18 G, 1-in. beveled needle | Amazon | B07S7VBHM2 | Used in combination with the dialysis casette |
| 2 mL desalting column | Thermo Fisher Scientific | 89890 | Removes APMA following activation |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Thermo Fisher Scientific | AAA1610422 | n/a |
| 250 mL conical bottle cushions | Thermo Fisher Scientific | 05-538-53A | Stabilize conical bottles during large-volume centrifugation |
| 250 mL conical bottles | Thermo Fisher Scientific | 05-538-53 | n/a |
| 400 mL stirred cell | Sigma Aldrich | UFSC40001 | Re-concentrates a much larger volume than the centrifugal filter unit. Rosetta2(DE3)pLysS cells produce high volumes of protein that may exceed the 15 mL limit of the centrifugal filter unit |
| 4-aminophenylmercuric acetate (APMA) | Sigma Aldrich | A9563-5G | Activates MMP-3 by cleaving the propeptide |
| 5 mL syringe | Thermo Fisher Scientific | NC0829167 | Used in combination with the dialysis casette |
| 50 mL conical tubes | Thermo Fisher Scientific | 339650 | Used for storage in many purification steps |
| 50 mL re-concentration tube | Sigma Aldrich | UFC901024D | Used for re-concentrating protein samples after dialysis or removing contaminants |
| Agar | Thermo Fisher Scientific | BP1423-500 | Buffer ingredient that solidifies autoclaved LB media upon cooling |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | Antibiotic used with pET3a vector; used at 100 µg/mL in LB media |
| BamHI | NEB | R3136S | Restriction enzyme to be used with the pET3a vector |
| Calcium chloride (CaCl2) | Thermo Fisher Scientific | 600-30-23 | The calcium ion stabilizes MMP structure |
| Cell spreaders | Thermo Fisher Scientific | 50-189-7544 | Can be used to spread cells across a petri dish after transformation |
| Chloramphenicol | Thermo Fisher Scientific | 22-055-125GM | Antibiotic used with pET3a vector; used at 34 µg/mL in LB media |
| Dialysis Buffer 1 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 4 M Urea. |
| Dialysis Buffer 2 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 2 M Urea. |
| Dialysis Buffer 3 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2 , 1 µM ZnCl2. |
| Dialysis clips | Thermo Fisher Scientific | 68011 | Used in combination with snakeskin dialysis tubing |
| Dialysis tubing | Thermo Fisher Scientific | 88243 | Alternative dialysis method that holds much larger sample volumes, but with higher risk of sample loss |
| Digest buffer | NEB | B7204S | Buffer used in digesting the pET3a vector |
| Disposable cuvettes | Thermo Fisher Scientific | 21-200-257 | Used to measure the bacterial culture OD during growth and expression |
| Dithiothreitol (DTT) | Thermo Fisher Scientific | D107125G | Assists with protein denaturation by reducing any disulfide bonds |
| DNA assembly mix | NEB | E2621S | Used to ligate the Hisx6-pro-MMP-3cd PCR product and digested pET3a vector |
| DNase I | NEB | M0303S | Endonuclease for degrading unfavorable DNA contaminants that could later affect protein purification |
| Ethanol | Thermo Fisher Scientific | A995-4 | n/a |
| Ethylenediaminetetraacetic acid (EDTA) | Thermo Fisher Scientific | J15694-AE | Used in denaturation. Prevents oxidation and subsequent formation of disulfide bonds |
| Gel recovery kit | Promega | A9281 | Isolates and purifies DNA from agarose gels |
| Glycerol | Thermo Fisher Scientific | G33-500 | Used for making glycerol stocks, which are frozen at -80 °C |
| Gravity flow column | BioRad | 7321010 | Used for Ni-NTA purification of recombinantly His-tagged proteins |
| High-transformation efficiency cells | NEB | C2987 | High-transformation efficiency cells with greater chance of success for cloning the N-terminal His-tag into the pET3a-pro-MMP-3cd construct |
| HT Elution Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 250 mM imidazole. Adjust pH to 7.4 |
| HT Equilibration Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea. Adjust pH to 7.4 |
| HT Regeneration Buffer | n/a | n/a | 20 mM MES, 0.1 M NaCl. Adjust pH to 5.0 |
| HT Wash Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 25 mM imidazole. Adjust pH to 7.4 |
| Hydrochloric acid (HCl) | Thermo Fisher Scientific | A144C-212 | Used to pH buffers |
| Imidazole | Thermo Fisher Scientific | AAA1022122 | Mimics the histidine side group. Used to separate non-specifically binding proteins from the his-tagged target protein |
| Inclusion Body Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 5 mM DTT, 2% v/v Triton X 100, 0.5 M Urea. Adjust pH to 8.0 |
| Isopropyl-ß-D-thiogalactopyranoside (IPTG) | Thermo Fisher Scientific | FERR0392 | A reagent that induces target gene expression in pET3a. Make 0.5 mL 1 M aliquots, filter sterilize and store in -20 °C |
| LB Amp CamR media | n/a | n/a | To be poured into a sterible 1 L bottle or 1 L flask. For 1 L, add 25 g LB Broth. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Amp CamR plates | n/a | n/a | To be poured into sterile petri dishes. Pour until the petri dish lid is completely covered. 1 L of media yields 40-60 plates. For 1 L: 25 g LB Broth, 16 g Agar. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Broth | Thermo Fisher Scientific | BP1426-2 | Pre-mixed with tryptone, yeast extract, and sodium chloride |
| Lysis Buffer | n/a | n/a | 50 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 0.133 g/mL lysozyme, 0.49% v/v Triton X-100. Adjust pH to 8.0 |
| Lysozyme | MP Biomedicals | 195303 | Used in protein extraction. Enzyme that lyses bacterial cell walls |
| Miniprep kit | Promega | A1330 | If successful, extracts the pET3a-pro-MMP-3cd construct from transformants |
| NdeI | NEB | R0111S | Restriction enzyme to be used with the pET3a vector |
| Ni-NTA resin | Thermo Fisher Scientific | PI88221 | Used to bind recombinant his-tagged proteins. This strong interaction can be displaced with higher concentrations of imidazole |
| Nonionic surfactant | Thermo Fisher Scientific | PI28316 | Storage detergent for preventing MMP aggregation. Minimizes interactions between hydrophobic residues on the MMP surface and water molecules, without disrupting catalytic activity. |
| PCR mix | NEB | M0492S | A PCR reagent for inserting an N-terminal his-tag into the pET3a-pro-MMP-3cd vector |
| pET plasmid | Addgene | n/a | The pET3a vector offers ampicillin resistance, inducible expression of a target gene, and sequencing with T7 primers |
| Petri dishes | VWR | 25384-342 | Used for plating transformants on LB agar media |
| R2DP cells | Novagen | 714033 | BL21 derivatives with enhanced expression of eukaryotic proteins. Contain tRNAs of codons found to be rare in e. coli |
| SOC growth media | NEB | B9020S | Non-selective growth media for rapid growth during transformation |
| Sodium chloride (NaCl) | Thermo Fisher Scientific | BP358-1 | Used in buffers and helps with protein stability |
| Sodium deoxycholate | Thermo Fisher Scientific | PI89905 | Detergent used in protein extraction. Lyses cell walls |
| Solubilization Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 10 mM DTT, 6 M Urea. Adjust pH to 8.0 |
| Tris base | Thermo Fisher Scientific | BP152-1 | Common buffer used in the physiological pH range. Temperature-sensitive |
| Triton X-100 | Thermo Fisher Scientific | M1122980101 | Detergent used for cell lysis |
| Urea | Thermo Fisher Scientific | AAJ75826A7 | First chaotropic agent for disrupting protein secondary structure |
| Zinc chloride (ZnCl2) | Thermo Fisher Scientific | AAA162810E | Stabilizes MMP structure. The zinc ion is found in the catalytic site of MMP-3 |
Referenzen
- Portolano, N., et al. Recombinant protein expression for structural biology in HEK 293F suspension cells: A novel and accessible approach. Journal of Visualized Experiments: JoVE. (92), e51897 (2014).
- Subedi, G. P., Johnson, R. W., Moniz, H. A., Moremen, K. W., Barb, A. High yield expression of recombinant human proteins with the transient transfection of HEK293 cells in suspension. Journal of Visualized Experiments: JoVE. (106), e53568 (2015).
- Nilvebrant, J., Alm, T., Hober, S. Orthogonal protein purification facilitated by a small bispecific affinity tag. Journal of Visualized Experiments: JoVE. (59), e3370 (2012).
- Yang, Z., et al. Highly efficient production of soluble proteins from insoluble inclusion bodies by a two-step-denaturing and refolding method. PLoS One. 6 (7), 22981 (2011).
- Hu, X., Beeton, C. Detection of functional matrix metalloproteinases by zymography. Journal of Visualized Experiments: JoVE. (45), e2445 (2010).
- Radisky, E. S., Raeeszadeh-Sarmazdeh, M., Radisky, D. C. Therapeutic potential of matrix metalloproteinase inhibition in breast cancer. Journal of Cellular Biochemistry. 118 (11), 3531-3548 (2017).
- Raeeszadeh-Sarmazdeh, M., Do, L. D., Hritz, B. G. Metalloproteinases and their inhibitors: Potential for the development of new therapeutics. Cells. 9 (5), 1313 (2020).
- Nagase, H., Visse, R., Murphy, G. Structure and function of matrix metalloproteinases and TIMPs. Cardiovascular Research. 69 (3), 562-573 (2006).
- Raeeszadeh-Sarmazdeh, M., et al. Directed evolution of the metalloproteinase inhibitor TIMP-1 reveals that its N- and C-terminal domains cooperate in matrix metalloproteinase recognition. Journal of Biological Chemistry. 294 (24), 9476-9488 (2019).
- Batra, J., et al. Matrix metalloproteinase-10 (MMP-10) interaction with tissue inhibitors of metalloproteinases TIMP-1 and TIMP-2. Journal of Biological Chemistry. 287 (19), 15935-15946 (2012).
- Singh, K. K., Jain, R., Ramanan, H., Saini, D. K., Galea, C. A. Expression and purification of matrix metalloproteinases in Escherichia coli. Matrix Metalloproteases. , 3-16 (2017).
- Manka, S. W., et al. Structural insights into triple-helical collagen cleavage by matrix metalloproteinase 1. Proceedings of the National Academy of Sciences of the United States of America. 109 (31), 12461-12466 (2012).
- Gomis-Ruth, F. X., et al. Mechanism of inhibition of the human matrix metalloproteinase stromelysin-1 by TIMP-1. Nature. 389 (6646), 77-81 (1997).
- Shirian, J., et al. Converting a broad matrix metalloproteinase family inhibitor into a specific inhibitor of MMP-9 and MMP-14. FEBS Letters. 592 (7), 1122-1134 (2018).
- Li, C., et al. Purification of recombinant histidine-tagged catalytic domain of MMP-13 in one step using affinity column and renaturation of it with histidine tag. Journal of Liquid Chromatography & Related Technologies. 37 (15), 2118-2130 (2014).
- Aydin, H., Azimi, F. C., Cook, J. D., Lee, J. E. A convenient and general expression platform for the production of secreted proteins from human cells. Journal of Visualized Experiments: JoVE. (65), e4041 (2012).
- McNiff, M. L., Haynes, E. P., Dixit, N., Gao, F. P., Laurence, J. S. Thioredoxin fusion construct enables high-yield production of soluble, active matrix metalloproteinase-8 (MMP-8) in Escherichia coli. Protein Expression and Purification. 122, 64-71 (2016).
- Maity, R., et al. GST-His purification: A two-step affinity purification protocol yielding full-length purified proteins. Journal of Visualized Experiments: JoVE. (80), e50320 (2013).
- Stefan, A., Ceccarelli, A., Conte, E., Montón Silva, A., Hochkoeppler, A. The multifaceted benefits of protein co-expression in Escherichia coli. Journal of Visualized Experiments: JoVE. (96), e52431 (2015).
- Yadavalli, R., Sam-Yellowe, T. HeLa based cell free expression systems for expression of Plasmodium rhoptry proteins. Journal of Visualized Experiments: JoVE. (100), e52772 (2015).
- Zeytuni, N., Zarivach, R. Purification of the M. magneticum strain AMB-1 magnetosome associated protein MamAΔ41. Journal of Visualized Experiments: JoVE. (37), e1844 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten