È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Espressione batterica e purificazione della metalloproteinasi-3 della matrice umana mediante cromatografia di affinità
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La purificazione, la dialisi e l'attivazione di His-tag sono impiegate per aumentare le rese di espressione proteica del dominio catalitico solubile e a matrice attiva metalloproteinasi-3 nei batteri. Le frazioni proteiche vengono analizzate tramite gel SDS-PAGE.
Abstract
Le metalloproteinasi della matrice (MMP) appartengono alla famiglia delle proteasi della metzincina con ruoli centrali nella degradazione e nel rimodellamento della matrice extracellulare (ECM), nonché nelle interazioni con diversi fattori di crescita e citochine. La sovraespressione di MMP specifiche è responsabile di diverse malattie come il cancro, le malattie neurodegenerative e le malattie cardiovascolari. Le MMP sono state recentemente al centro dell'attenzione come bersagli per sviluppare terapie in grado di trattare malattie correlate alla sovraespressione di MMP.
Per studiare il meccanismo MMP in soluzione, sono necessari metodi di espressione e purificazione delle proteine ricombinanti più facili e robusti per la produzione di MMP attive e solubili. Tuttavia, il dominio catalitico della maggior parte delle MMP non può essere espresso in Escherichia coli (E. coli) in forma solubile a causa della mancanza di macchinari post-traduzionali, mentre i sistemi di espressione dei mammiferi sono solitamente costosi e hanno rese inferiori. Gli organismi di inclusione MMP devono subire il noioso e laborioso processo di purificazione e ripiegamento estensivo, riducendo significativamente la resa delle MMP in conformazione nativa. Questo documento presenta un protocollo che utilizza le cellule Rosetta2(DE3)pLysS (di seguito denominate R2DP) per produrre il dominio catalitico della metalloproteinasi-3 della matrice (MMP-3cd), che contiene un N-terminal His-tag seguito da pro-domain (Hisx6-pro-MMP-3cd) da utilizzare nella purificazione dell'affinità. Le cellule R2DP migliorano l'espressione delle proteine eucariotiche attraverso un plasmide resistente al cloramfenicolo contenente codoni normalmente rari nei sistemi di espressione batterica. Rispetto alla tradizionale linea cellulare scelta per l'espressione proteica ricombinante, BL21 (DE3), la purificazione con questo nuovo ceppo ha migliorato la resa di Hisx6-pro-MMP-3cd purificato. Al momento dell'attivazione e della desalinizzazione, il dominio pro viene scisso insieme all'N-terminal His-tag, fornendo MMP-3cd attivo per l'uso immediato in innumerevoli applicazioni in vitro . Questo metodo non richiede attrezzature costose o proteine di fusione complesse e descrive la rapida produzione di MMP umane ricombinanti nei batteri.
Introduzione
La maggior parte delle proteine eucariotiche complesse subisce elaborate modificazioni post-traduzionali dopo l'espressione, che richiedono un ripiegamento proteico altamente assistito e co-fattori per essere funzionali1. La produzione di grandi quantità di proteine umane solubili in un ospite batterico rimane una sfida significativa a causa dei costi elevati e della mancanza di solidi metodi di espressione e purificazione, anche per esperimenti di laboratorio su scala ridotta2,3. Le MMP, endopeptidasi umane con grande peso molecolare, sono solitamente espresse come corpi di inclusione insolubili quando espresse in E. coli. L'estrazione di MMP umane solubili porta spesso a un laborioso e dispendioso processo di solubilizzazione e ripiegamento4.
Le MMP hanno ruoli critici sia nei processi fisiologici che in quelli patogenetici. Le MMP umane sono una famiglia di 23 endopeptidasi di zinco, classificate per struttura e specificità del substrato, ed espresse in modo differenziale nonostante un dominio catalitico altamente conservato5,6. Le MMP sono secrete come zimogeni inattivi, regolati tramite attivazione post-traduzionale e loro inibitori endogeni, inibitori tissutali delle metalloproteinasi (TIMP)7,8,9,10. Sebbene inizialmente riconosciute per il loro ruolo nel turnover ECM, le MMP sono state anche implicate nello sviluppo, nella morfogenesi, nella riparazione dei tessuti e nel rimodellamento8. La disregolazione delle MMP è stata in particolare collegata al cancro insieme a malattie neurodegenerative, cardiovascolari e fibrotiche, tra le altre malattie5,7.
Lo sviluppo di solidi metodi di produzione MMP su larga scala è fondamentale per garantire il successo di studi futuri sui meccanismi MMP attraverso saggi biochimici e cellulari. Varie MMP sono state precedentemente espresse in batteri11, comprese le MMP con tag Hisx6, senza alterare l'attività MMP12,13,14,15. Tuttavia, questi metodi includono passaggi noiosi e lunghi che potrebbero essere difficili da replicare.
Le cellule di mammifero possono anche essere utilizzate per esprimere molte proteine umane diverse, garantendo al contempo le corrette modifiche post-traduzionali16. Sebbene il sistema di espressione dei mammiferi sia la scelta ideale per produrre proteine umane ricombinanti con adeguate modifiche post-traduzionali, i principali svantaggi di questo metodo sono le basse rese iniziali, i costosi mezzi di crescita e reagenti, le lunghe tempistiche per raggiungere linee di espressione stabili e il rischio di contaminazione con altre specie come funghi o batteri2,11 . Inoltre, la produzione di MMP nelle linee cellulari di mammiferi produce impurità da proteine cellulari associate come TIMP o fibronectine11. A differenza della lenta crescita cellulare osservata nelle cellule di mammifero, il sistema di espressione batterica offre una produzione proteica su larga scala in un breve periodo insieme a mezzi e requisiti di crescita più semplici. Tuttavia, a causa della mancanza di altre proteine cellulari associate (ad esempio, TIMP) nei sistemi di espressione batterica, le MMP attive a concentrazioni più elevate sono soggette a degradazione attraverso l'autoproteolisi, con conseguente scarsa resa MMP17.
Questo documento descrive un metodo dettagliato per l'espressione batterica, la purificazione e l'attivazione di Hisx6-pro-MMP-3cd ricombinante utilizzando E. coli come ospite di espressione grazie alla sua convenienza, semplicità e successo nel produrre rese più elevate di MMP2,3,18. Poiché E. coli non ha il meccanismo di ripiegamento delle proteine e l'elaborazione post-traduzionale necessari per le MMP ricombinanti e altre proteine complesse, molti ceppi di E. coli sono stati progettati per superare queste limitazioni, rendendo E. coli un ospite più adatto per l'espressione di MMP-3cd umano ricombinante,19,20 . Ad esempio, il ceppo R2DP utilizzato in questo studio migliora l'espressione eucariotica fornendo un plasmide resistente al cloramfenicolo contenente codoni raramente usati in E. coli.
Come descritto in questo protocollo, dopo la sovraespressione di corpi di inclusione relativamente puri dal vettore pET-3a (Figura 1) nelle cellule R2DP, le proteine del dominio catalitico Hisx6-pro-MMP-3 (MMP-3cd) vengono estratte e denaturate4. Hisx6-pro-MMP-3cd3,19 è stato purificato utilizzando la cromatografia con tag di affinità. Dopo il ripiegamento e la dialisi, il pro-MMP-3cd (zimogeno) è stato attivato dall'acetato di 4-aminofenilmercurio (APMA) e l'analisi SDS-PAGE viene utilizzata per valutare le rese e la necessità di un'ulteriore purificazione5,21. Questo protocollo descrive l'espressione, la purificazione e l'attivazione di MMP-3cd solubile come esempio. Tuttavia, può anche essere usato come guida per l'espressione di altre MMP e proteasi umane con espressione simile e meccanismi di attivazione (Figura 2). Per altre proteine diverse da MMP-3cd, si consiglia al lettore di determinare le composizioni tampone e i metodi ottimali per la loro proteina bersaglio prima di tentare questo protocollo.
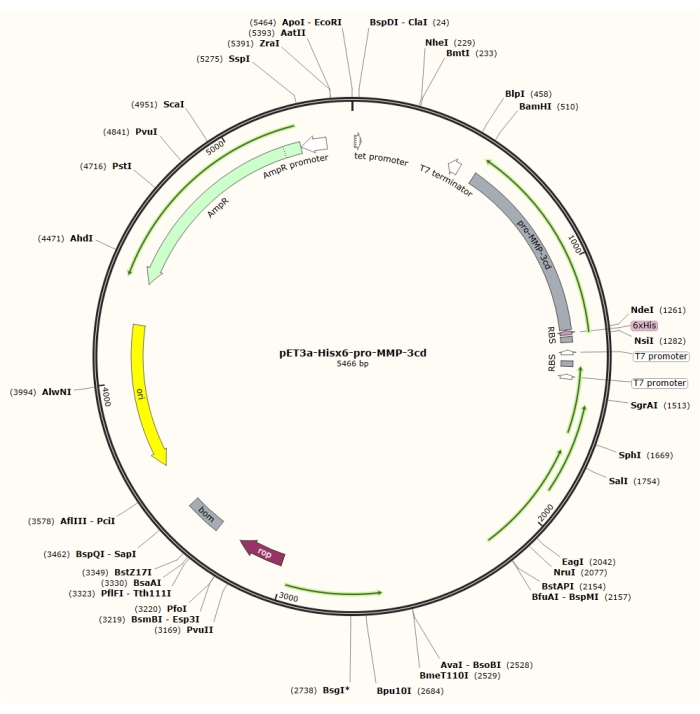
Figura 1: Mappa plasmidica del plasmide pET-3a-Hisx6-pro-MMP-3cd. Il vettore pET-3a include un gene di resistenza all'ampicillina. Una sequenza di tag Hisx6 N-terminale viene clonata nel vettore basato su pET-3a, incluso pro-MMP-3cd, per produrre il costrutto pET-3a-Hisx6-pro-MMP-3cd sotto il controllo del promotore T7 tra i siti di restrizione BamHI e NdeI. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Espressione batterica del plasmide pro-MMP-3cd, purificazione, ripiegamento e attivazione. 1.1: il plasmide pET-3a-Hisx6-pro-MMP-3cd è stato trasformato in cellule BL21(DE3) o R2DP. 1.2: L'espressione proteica Pro-MMP-3cd è stata indotta utilizzando IPTG. 1.3: La lisi chimica e la sonicazione vengono utilizzate per estrarre le proteine Hisx6-pro-MMP-3cd che sono principalmente insolubili e si trovano nei corpi di inclusione. L'urea è stata utilizzata per denaturare e solubilizzare le proteine dai corpi di inclusione. 2.1. La proteina Hisx6-pro-MMP-3cd denaturata è stata purificata tramite purificazione cromatografica di affinità. 3. L'Hisx6-pro-MMP-3cd eluito è stato lentamente ripiegato durante la dialisi attraverso la graduale rimozione dell'urea dal tampone. 4. Infine, la proteina MMP-3cd ripiegata è stata attivata utilizzando APMA rimuovendo il dominio pro-peptide N-terminale. L'APMA viene successivamente rimosso dalla soluzione attraverso la desalinizzazione. I numeri corrispondono alle sezioni di protocollo che descrivono questi passaggi. Abbreviazioni: MMP-3cd = Matrix metalloproteinase-3 catalytic domain; APMA = acetato di 4-aminofenilmercurio. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Espressione MMP
- Clonazione e trasformazione di pET-3a-Hisx6-pro-MMP-3cd in celle R2DP
- Digerire il plasmide pET-3a (vedere la tabella dei materiali) con gli enzimi di restrizione NdeI e BamHI nel digest buffer (vedere la tabella dei materiali). In un volume totale di reazione di 40 μL, aggiungere 4 μL di Digest Buffer, 33 μL di plasmide 100 ng/μL e 1,5 μL di ciascun enzima di restrizione e lasciare che la reazione proceda per ~2 h fino al completamento a 37 °C.
- Eseguire una reazione PCR sulla sequenza MMP-3cd per inserire un His-tag N-terminale. Utilizzare 25 μL di PCR Mix (vedere la Tabella dei materiali), 2,5 μL di primer da 10 μM (Figura supplementare S1) e 1,25 μL della sequenza di inserto da 100 ng/μL. Aggiungere acqua sterile a un volume di reazione finale di 50 μL.
- Eseguire il prodotto PCR e il vettore digerito su un gel di agarosio all'1%. Purificare le bande di gel utilizzando un kit di recupero del gel (vedere la tabella dei materiali) secondo il protocollo del produttore.
- Clonare il prodotto PCR amplificato nel vettore digerito tra i siti di restrizione NdeI e BamHI utilizzando DNA Assembly Mix (vedere la Tabella dei materiali). Utilizzare gli strumenti online per determinare il volume richiesto del vettore di inserto e taglio per un volume di reazione totale di 15 μL.
- Scongelare un'aliquota di 50 μL di celle ad alta efficienza di trasformazione (vedi la Tabella dei materiali) sul ghiaccio fino allo scongelamento. Terreno di crescita SOC prebellico (vedi tabella dei materiali) a 37 °C e piastre LB-ampicillina (LB Amp) (vedi tabella dei materiali).
- Aggiungere 1-2 μL della reazione di assemblaggio pET-3a-Hisx6-pro-MMP-3cd all'aliquota 50 μL. Incubare sul ghiaccio per 30 min.
- Shock termico delle celle mediante incubazione a 42 °C per 30 s. Incubare su ghiaccio per 2 min.
- Aggiungere 950 μL di terreno di coltura SOC a ciascuna miscela trasformante. Agitare per 1 ora a 250 giri/min e 37 °C.
- Piastra 100 μL dei trasformatori su piastre LB Amp e incubare durante la notte a 37 °C.
- Inoculare ogni colonia isolata in 10 ml di LB Amp medio. Agitare per una notte a 250 giri/min e 37 °C.
- Estrarre il DNA plasmidico secondo il protocollo del produttore per il kit miniprep (vedere la Tabella dei materiali). Confermare la sequenza del costrutto utilizzando i primer avanti e indietro T7 (Figura supplementare S1).
NOTA: Il DNA del costrutto pET-3a-Hisx6-pro-MMP-3cd può essere conservato a -20 °C. Quando è pronto, procedere con la trasformazione in celle R2DP. - Scongelare un'aliquota di 20 μL di celle R2DP (vedi Tabella dei Materiali) sul ghiaccio per 2-5 min. Crescita SOC prebellica da media a temperatura ambiente e piastre LB Amp CamR a 37 °C (vedere la Tabella dei materiali).
- Aggiungere 1 μL di pET-3a-Hisx6-pro-MMP-3cd confermato dalla sequenza 100 ng/μL all'aliquota da 50 μL. Mescolare delicatamente per mescolare e riportare il tubo sul ghiaccio.
- Incubare il tubo sul ghiaccio per 5 min.
- Shock termico delle celle mediante incubazione a 42 °C per esattamente 30 s. Non agitare.
- Mettere le cellule sul ghiaccio per 2 minuti.
- Aggiungere 80 μL di mezzo SOC a temperatura ambiente alla miscela trasformante. Agitare per 1 ora a 250 giri/min e 37 °C.
- Placcare i trasformatori su piastre LB Amp CamR e incubare durante la notte a 37 °C.
- Crescita e induzione
- Inoculare una singola colonia isolata di trasformatore R2DP pET-3a-Hisx6-pro-MMP-3cd da una piastra LB Amp CamR in 10 mL di supporto LB Amp CamR a 37 °C. Agitare a 250 giri/min durante la notte (~16 h). Risparmiare aliquote da ciascuna coltura e preparare il 40% (v/v) di scorte di glicerolo (vedi tabella dei materiali), se lo si desidera.
- Per coltura durante la notte, inoculare un pallone da 1 L contenente 500 mL di LB Amp CamR medio ad una densità ottica a 600 nm (OD600) di 0,05-0,1.
NOTA: questo dovrebbe riportare le cellule alla crescita logaritmica. - Misurare l'OD600 in diversi punti temporali, in genere per 3-4 ore, fino a quando non cade tra 0,4 e 0,6.
- Prima dell'induzione, aliquotare una frazione di coltura in un tubo di microfuga da 1,5 ml (vedere la Tabella dei materiali) ed etichettarla Frazione non indotta. Conservare a -80 °C per l'analisi del gel. Se non si esegue un gel SDS-PAGE, ignorare questo passaggio e procedere al passaggio 1.2.5.
- Indurre le colture ad una concentrazione finale di 1 mM utilizzando 1 M di stock di isopropil-ß-D-tiogalattopiranoside (IPTG) (vedere la Tabella dei materiali). Continuare a incubare nello shaker a 37 °C per ulteriori 3-4 ore.
NOTA: Durante l'espressione, il lettore deve determinare l'OD600 ottimale al momento dell'induzione e la concentrazione di IPTG. Se la resa diminuisce sostanzialmente dopo la purificazione, la concentrazione di imidazolo nei tamponi di purificazione può richiedere un aggiustamento o il pellet cellulare potrebbe dover essere ulteriormente sonicato. - Prima di centrifugare le colture, aliquotare una frazione di coltura in un secondo tubo di microfuga da 1,5 ml ed etichettarla Frazione indotta. Conservare a -80 °C per l'analisi del gel. Se non si esegue un gel SDS-PAGE, ignorare questo passaggio e procedere al passaggio 1.2.7.
- Centrifugare la coltura cellulare in flaconi conici da 250 ml (vedi Tabella dei Materiali) alla massima velocità e 4 °C per 10 min.
- Ripetere il passaggio 1.2.7 fino a quando le colture non sono completamente pellettizzate.
NOTA: PAUSA: I pellet cellulari possono essere congelati a -80 °C e scongelati successivamente per un'ulteriore lavorazione. In caso contrario, ignorare questo passaggio e procedere al passaggio 1.3.1.
- Inclusione corpo estrazione e solubilizzazione
NOTA: Preparare l'urea fresca da 10 M, preferibilmente non prima di un giorno in anticipo, mescolando accuratamente fino a completa dissoluzione. Non riscaldare o autoclave urea; conservarlo a temperatura ambiente.- Risospesso il pellet (dal punto 1.2.8) nel tampone di lisi (vedere la tabella dei materiali). Per grammo di pellet, aggiungere 3 ml di tampone di lisi e risospesso mediante vortice o pipettaggio. Agitare per una notte a 4 °C.
- Aggiungere 1,25 ml di desossicolato di sodio al 10% (p/v) (vedere la tabella dei materiali) per 1 L di coltura. Agitare a temperatura ambiente per 30 minuti a 150 giri/min.
- Aggiungere 10 μL di DNasi I (vedi tabella dei materiali) per 1 L di coltura. Agitare a temperatura ambiente per 30 minuti a 150 giri/min.
- Centrifuga per 10 min a 13.000 × g e 4 °C.
- Mettere da parte una frazione di MMP lisato per l'analisi del gel. Conservare a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 1.3.6.
NOTA: Dopo la centrifugazione, il pellet può essere filante e non imballato in modo compatto, rendendo rischioso scartare il surnatante. In questo caso, saltare il passaggio 1.3.6 e procedere al passaggio 1.3.7. - Scartare il surnatante dai campioni centrifugati.
NOTA: Il protocollo può essere sospeso a questo punto e i pellet cellulari congelati a -80 °C e scongelati in seguito. In caso contrario, ignorare questo passaggio e procedere al passaggio 1.3.7. - Risospesso il pellet in coltura da 100 mL/L di Inclusion Body Buffer (vedi tabella dei materiali) mediante pipettaggio su e giù.
- Durante la sonicazione, tenere i campioni sul ghiaccio per evitare il surriscaldamento. Sonicare ogni campione per 6 cicli di 15 s, uscita 5 e impulso del 50%. Consentire periodi di riposo di 15 s per il raffreddamento tra i cicli.
NOTA: Se necessario, trasferire i campioni in tubi conici da 50 ml per un'ulteriore centrifugazione (vedere la tabella dei materiali). Centrifuga per 10 min a 13.000 × g e 4 °C. - Mettere da parte una frazione di MMP sonicato per l'analisi del gel. Conservare a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 1.3.11.
- Controllare il pellet. Se rigoroso, ripetere i passaggi 1.3.8-1.3.10. Se il pellet è compatto, scartare il surnatante e procedere al passaggio 1.3.12.
NOTA: la sonicazione in Inclusion Body Buffer può essere ripetuta per recuperare più proteine dai detriti cellulari lisati. Tuttavia, troppa sonicazione può causare la cesoiatura, che danneggia la resa MMP. Il protocollo può essere sospeso in questa fase e i pellet cellulari possono essere congelati a -80 °C e scongelati in seguito. - Risospesso ogni pellet da una coltura di 1 L in 5 mL di tampone di solubilizzazione (vedi la tabella dei materiali) mediante pipettaggio. Incubare per almeno 30 minuti sul ghiaccio per consentire alle proteine di solubilizzarsi.
- Mettere da parte una frazione di MMP solubilizzato per l'analisi del gel. Conservare a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 1.3.14.
- Centrifugare le celle per 10 min a 13.000 × g e 4 °C. NON SCARTARE IL SURNATANTE.
- Se un pellet si forma/rimane dopo la centrifugazione, versare il surnatante in un tubo conico separato da 50 ml. Risospesciare il pellet in altri 5 mL di tampone di solubilizzazione (per 1 L di coltura) pipettando su e giù.
- Centrifugare per 10 min a 13.000 × g e 4 °C. NON SCARTARE IL SURNATANTE.
- Ripetere i passaggi 1.3.13 e 1.3.14 fino a quando non si forma poco o nessun pellet dopo la centrifugazione o rimane solo precipitato grigio. Metti in comune i supernatanti. Scartare o conservare il pellet a -80 °C per un'ulteriore sonicazione.
2. Purificazione e ripiegamento MMP
- Purificazione dell'affinità His-tag (HT)
- Secondo il protocollo del produttore, riempire una colonna di flusso gravitazionale (vedere la tabella dei materiali) con resina Ni-NTA ben miscelata (vedere la tabella dei materiali). Lasciare che la resina si depositi e si separi dal tampone di stoccaggio in modo tale che si formi una linea distinta tra i due strati.
NOTA: non lasciare mai asciugare la resina, poiché l'aria penetrerà nella resina e danneggerà la resa proteica. Tra un utilizzo e l'altro, eseguire la procedura di rigenerazione della resina descritta al punto 2.2. - Lasciare scaricare il buffer di stoccaggio. Riempire la colonna con due volumi a letto di resina di HT Equilibration Buffer.
- Scolare il tampone di equilibrio HT e scartare. Mentre la colonna è drenante, centrifugare l'estratto proteico a 13.000 × g per 1 minuto e filtrare-sterilizzare utilizzando un filtro da 0,22 μm (vedere la tabella dei materiali).
- Sostituire il contenitore dei rifiuti con un tubo conico da 50 ml etichettato HT Flowthrough. Aggiungere l'estratto proteico preparato alla colonna.
- Riapplicare il flusso per massimizzare l'associazione.
- Mettere da parte una frazione di Flowthrough Fraction per l'analisi del gel. Conservare a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 2.1.7.
- Lavare immediatamente la resina con 15 mL di tampone di lavaggio HT (vedere la tabella dei materiali). Raccogliere il flowthrough in tubi conici da 15 ml (vedere la Tabella dei materiali) etichettati HT Wash.
NOTA: I valori di assorbanza a 280 nm (A280) sono stati ottenuti tramite spettrofotometria e utilizzati insieme al peso molecolare e al coefficiente di estinzione, ε, per stimare le concentrazioni proteiche. Per Hisx6-pro-MMP-3cd denaturato, il peso molecolare è di 29,86 kDa e ε è di 34,38 M-1 cm-1. - Blanking contro HT Wash Buffer, misurare e registrare l'A280. Ripetere i passaggi 2.1.7 e 2.1.8 con frazioni di lavaggio aggiuntive. Una volta che l'A280 si avvicina alla linea di base e le impurità sono state ridotte al minimo, procedere al passaggio 2.1.9.
- Mettere da parte una frazione di Wash Fraction per l'analisi del gel. Ripetere l'operazione per più frazioni di lavaggio. Conservare le frazioni a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 2.1.10.
- Eluire immediatamente le proteine his-tagged aggiungendo 5 ml di HT Elution Buffer (vedere la tabella dei materiali). Raccogliere il flusso attraverso frazioni di 0,5-1 ml in tubi di microfuga etichettati HT Elution.
- Mettere da parte una frazione di frazione di Elution per l'analisi del gel. Ripetere l'operazione per più frazioni di eluizione. Conservare le frazioni a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 2.1.12.
- Se l'A280 è >0,3 mg/mL, diluire la frazione con HT Equilibration Buffer (vedere la Tabella dei materiali).
NOTA: La frazione eluita deve essere diluita ad un A280 di 0,3 mg/mL o inferiore per evitare precipitazioni durante la dialisi. Il protocollo può essere messo in pausa qui e le frazioni raggruppate congelate a -80 ° C e scongelate in seguito. In caso contrario, ignorare questo passaggio e procedere al passaggio 2.2.1.
- Secondo il protocollo del produttore, riempire una colonna di flusso gravitazionale (vedere la tabella dei materiali) con resina Ni-NTA ben miscelata (vedere la tabella dei materiali). Lasciare che la resina si depositi e si separi dal tampone di stoccaggio in modo tale che si formi una linea distinta tra i due strati.
- Rigenerazione della resina
- Lavare la resina con dieci volumi di HT Regeneration Buffer a letto di resina (vedere la Tabella dei materiali) e dieci volumi di acqua sterile a letto di resina.
- Conservare la resina come liquame al 50% in etanolo al 20% (v/v) in acqua.
3. Ripiegamento delle proteine
NOTA: per volumi più piccoli, le cassette per dialisi possono essere utilizzate a minor rischio di perdita del campione. I tubi per dialisi sono necessari se vengono utilizzati volumi maggiori (vedere la tabella dei materiali).
- Dialisi
NOTA: La concentrazione ottimale di proteine per la dialisi è di ~0,3 mg/ml. Se si verifica una precipitazione significativa durante la dialisi, ridurre il gradiente di concentrazione dell'urea tra ogni dialisi utilizzando un metodo di dialisi graduale e aggiungere più passaggi intermedi (ad esempio, da 6 M a 5 M e quindi da 5 M a 4 M, piuttosto che saltare lo stadio di 5 M). Poiché i cicli di congelamento-disgelo danneggiano la struttura cellulare e proteica, è fondamentale ridurre al minimo le pause nel protocollo.- Secondo il protocollo del produttore, utilizzare la quantità appropriata di tubi di dialisi in base al volume dei campioni di frazione di eluizione .
- Immergere le frazioni MMP eluite in tubi di dialisi in 1 L di Tampone di Dialisi 1 (vedere la Tabella dei Materiali). Mescolare il tubo e il suo contenuto su un agitatore magnetico per non meno di 8 ore a 4 °C.
- Trasferire a 1 L di Tampone di Dialisi 2 (vedere la Tabella dei Materiali). Mescolare il tubo e il suo contenuto su un agitatore magnetico per non meno di 8 ore a 4 °C.
- Trasferire a 1 L di Tampone di Dialisi 3 (vedere la Tabella dei Materiali). Mescolare il tubo e il suo contenuto su un agitatore magnetico per non meno di 8 ore a 4 °C.
- Trasferire il campione in nuovi tubi conici da 50 ml ed etichettarli come MMP dializzato.
- Esaminare il tubo per qualsiasi precipitato. Se si è formato un precipitato, centrifugare il campione per 1 minuto a 13.000 × g e 4 °C.
- Trasferire il surnatante in nuovi tubi conici da 15 ml ed etichettarli come MMP ripiegato.
- Mettere da parte una frazione per l'analisi del gel ed etichettarla come MMP ripiegato. Conservare a -80 °C. Se non si esegue l'analisi del gel, procedere al passaggio 3.1.9.
NOTA: Se le rese sono basse, il precipitato può essere sciolto nel tampone di equilibrio HT e i passaggi nel paragrafo 3.1 ripetuti con tubi di dialisi. Se l'analisi del gel non deve essere eseguita o se il protocollo deve essere messo in pausa qui, congelare i campioni a -80 °C e scongelarli in un secondo momento. Se le rese sono nell'intervallo desiderato, procedere al passaggio 3.2.1.
- Riconcentrazione
NOTA: i coefficienti di estinzione per Hisx6-pro-MMP-3cd ripiegato e denaturato dovrebbero essere gli stessi; pertanto, i calcoli A280 non sono interessati.- Riconcentrare il campione fino a 0,5 mg/ml. Utilizzare una cella agitata da 400 ml (vedere la tabella dei materiali) per concentrare il campione a 15 ml. Per evitare la formazione di schiuma, utilizzare un tubo di riconcentrazione da 50 ml per concentrarsi ulteriormente, se necessario.
NOTA: Se si forma un precipitato, può essere pellettato e disciolto in HT Equilibration Buffer. Quindi, ripetere i paragrafi 3.1 e 3.2.1. In caso contrario, continuare con il passaggio 3.2.2. - Mettere da parte una frazione per l'analisi del gel ed etichettarla come MMP concentrato.
NOTA: Il protocollo può essere messo in pausa qui e i campioni congelati a -80 °C e scongelati in seguito.
- Riconcentrare il campione fino a 0,5 mg/ml. Utilizzare una cella agitata da 400 ml (vedere la tabella dei materiali) per concentrare il campione a 15 ml. Per evitare la formazione di schiuma, utilizzare un tubo di riconcentrazione da 50 ml per concentrarsi ulteriormente, se necessario.
4. Attivazione
- Attivazione dell'acetato di 4-aminofenilmercurico (APMA)
NOTA: APMA è altamente tossico. Preparare una soluzione madre fresca di 20 mM APMA prima dell'attivazione e lavorare sempre sotto una cappa aspirante quando si utilizza APMA. Scartare i rifiuti APMA nel suo contenitore.- Per aliquota di 1 mL di MMP (1 mg/mL), aggiungere 50 μL di APMA da 20 mM (vedere la Tabella dei materiali) per raggiungere una concentrazione finale di APMA di 1 mM. Incubare durante la notte a 37 °C.
- Se si forma un precipitato, centrifugarlo alla massima velocità per 10 minuti a 4 °C. Conservare il surnatante in un tubo di microfuga da 1,5 ml etichettato MMP attivato. Gettare il precipitato in un contenitore contrassegnato per i rifiuti APMA.
- Mettere da parte una frazione per l'analisi del gel ed etichettarla MMP attivato.
NOTA: Il protocollo può essere messo in pausa qui e i campioni congelati a -80 °C e scongelati in seguito. Se non si esegue l'analisi del gel, procedere al passaggio 4.2.1. Dopo l'attivazione, il peso molecolare e il coefficiente di estinzione di MMP-3cd sono rispettivamente 19,40 kDa e 28,42 M-1 cm-1.
- Dissalazione
- Rimuovere APMA dal campione MMP-3cd attivato con una colonna di desalinizzazione da 2 ml (vedere la tabella dei materiali), seguendo il protocollo del produttore.
- Mettere da parte una frazione per l'analisi del gel ed etichettarla Come Disalted MMP. Conservare a -80 °C. Se non si esegue l'analisi del gel, procedere al paragrafo 4.3 con i campioni rimanenti.
NOTA: Il protocollo può essere messo in pausa qui e i campioni congelati a -80 °C e scongelati in seguito.
- Esecuzione dei gel SDS-PAGE
- Esegui tutte le frazioni proteiche su gel SDS-PAGE: frazione non indotta, frazione indotta, MMP lisato, MMP sonicato, MMP solubilizzato, frazione passante, frazione di lavaggio, frazione di eluizione, MMP ripiegato, MMP concentrato, MMP attivato e MMP dissalato.
- Archiviazione a lungo termine di MMP-3
- Aggiungere lo 0,05% (v/v) di tensioattivo non ionico (vedere la tabella dei materiali) ai campioni MMP-3cd dissalati e conservarli a -80 °C.
Risultati
Quando si eseguono campioni su SDS-PAGE, poiché la proteina è espressa sotto forma di corpi di inclusione insolubili, le frazioni lisate e sonicate dovrebbero contenere poco o nessun estratto di Hisx6-pro-MMP-3cd, poiché la proteina non è ancora stata risolubilizzata nell'urea. La Figura 3 confronta le frazioni di eluizione di purificazione His-tag di Hisx6-pro-MMP-3cd da celle BL21(DE3) e celle R2DP. Le frazioni di eluizione sono state raggruppate separatamente per entrambe le cellule B...
Discussione
La produzione su larga scala di MMP solubili, umane e ricombinanti rimane un compito impegnativo. Le cellule di mammifero possono esprimere MMP funzionali a costi elevati e lunghi tempi di attesa, mentre E. coli produce rapidamente elevate quantità di corpi di inclusione MMP che devono essere purificati e ripiegati11,16. Le celle R2DP aumentano significativamente la resa degli organismi di inclusione MMP, consentendo un processo di ripiegamento MMP più...
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori vorrebbero ringraziare la dott.ssa Evette Radisky e Alexandra Hockla della Mayo Clinic di Jacksonville, in Florida, per aver fornito il plasmide pET-3a-pro-MMP-3cd come modello per la clonazione del gene Hisx6pro-MMP-3cd e i loro commenti, insieme al Dr. Paul Hartley del Nevada Genomics Center presso l'Università del Nevada, Reno, per il sequenziamento del DNA. Gli autori vorrebbero anche ringraziare Cassandra Hergenrader per aver aiutato con parte dell'espressione proteica. M.R.-S. vorrei ringraziare il NIH-P20 GM103650-COBRE Integrative Neuroscience grant e l'UNR R&D mICRO SEED Grant Award.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 µm sterile filter | Sigma Aldrich | SLGP033RS | Used to remove some contaminants from the protein extract before purification, and prevent the Ni-NTA column from clogging |
| 1 L Erlenmeyer flasks | Thermo Fisher Scientific | S76106F | n/a |
| 1 L glass bottles | Thermo Fisher Scientific | 06-414-1D | n/a |
| 1.5 mL microfuge tubes | Thermo Fisher Scientific | 02-682-002 | n/a |
| 15 mL conical tubes | Thermo Fisher Scientific | 339650 | n/a |
| 18 G, 1-in. beveled needle | Amazon | B07S7VBHM2 | Used in combination with the dialysis casette |
| 2 mL desalting column | Thermo Fisher Scientific | 89890 | Removes APMA following activation |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Thermo Fisher Scientific | AAA1610422 | n/a |
| 250 mL conical bottle cushions | Thermo Fisher Scientific | 05-538-53A | Stabilize conical bottles during large-volume centrifugation |
| 250 mL conical bottles | Thermo Fisher Scientific | 05-538-53 | n/a |
| 400 mL stirred cell | Sigma Aldrich | UFSC40001 | Re-concentrates a much larger volume than the centrifugal filter unit. Rosetta2(DE3)pLysS cells produce high volumes of protein that may exceed the 15 mL limit of the centrifugal filter unit |
| 4-aminophenylmercuric acetate (APMA) | Sigma Aldrich | A9563-5G | Activates MMP-3 by cleaving the propeptide |
| 5 mL syringe | Thermo Fisher Scientific | NC0829167 | Used in combination with the dialysis casette |
| 50 mL conical tubes | Thermo Fisher Scientific | 339650 | Used for storage in many purification steps |
| 50 mL re-concentration tube | Sigma Aldrich | UFC901024D | Used for re-concentrating protein samples after dialysis or removing contaminants |
| Agar | Thermo Fisher Scientific | BP1423-500 | Buffer ingredient that solidifies autoclaved LB media upon cooling |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | Antibiotic used with pET3a vector; used at 100 µg/mL in LB media |
| BamHI | NEB | R3136S | Restriction enzyme to be used with the pET3a vector |
| Calcium chloride (CaCl2) | Thermo Fisher Scientific | 600-30-23 | The calcium ion stabilizes MMP structure |
| Cell spreaders | Thermo Fisher Scientific | 50-189-7544 | Can be used to spread cells across a petri dish after transformation |
| Chloramphenicol | Thermo Fisher Scientific | 22-055-125GM | Antibiotic used with pET3a vector; used at 34 µg/mL in LB media |
| Dialysis Buffer 1 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 4 M Urea. |
| Dialysis Buffer 2 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 2 M Urea. |
| Dialysis Buffer 3 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2 , 1 µM ZnCl2. |
| Dialysis clips | Thermo Fisher Scientific | 68011 | Used in combination with snakeskin dialysis tubing |
| Dialysis tubing | Thermo Fisher Scientific | 88243 | Alternative dialysis method that holds much larger sample volumes, but with higher risk of sample loss |
| Digest buffer | NEB | B7204S | Buffer used in digesting the pET3a vector |
| Disposable cuvettes | Thermo Fisher Scientific | 21-200-257 | Used to measure the bacterial culture OD during growth and expression |
| Dithiothreitol (DTT) | Thermo Fisher Scientific | D107125G | Assists with protein denaturation by reducing any disulfide bonds |
| DNA assembly mix | NEB | E2621S | Used to ligate the Hisx6-pro-MMP-3cd PCR product and digested pET3a vector |
| DNase I | NEB | M0303S | Endonuclease for degrading unfavorable DNA contaminants that could later affect protein purification |
| Ethanol | Thermo Fisher Scientific | A995-4 | n/a |
| Ethylenediaminetetraacetic acid (EDTA) | Thermo Fisher Scientific | J15694-AE | Used in denaturation. Prevents oxidation and subsequent formation of disulfide bonds |
| Gel recovery kit | Promega | A9281 | Isolates and purifies DNA from agarose gels |
| Glycerol | Thermo Fisher Scientific | G33-500 | Used for making glycerol stocks, which are frozen at -80 °C |
| Gravity flow column | BioRad | 7321010 | Used for Ni-NTA purification of recombinantly His-tagged proteins |
| High-transformation efficiency cells | NEB | C2987 | High-transformation efficiency cells with greater chance of success for cloning the N-terminal His-tag into the pET3a-pro-MMP-3cd construct |
| HT Elution Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 250 mM imidazole. Adjust pH to 7.4 |
| HT Equilibration Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea. Adjust pH to 7.4 |
| HT Regeneration Buffer | n/a | n/a | 20 mM MES, 0.1 M NaCl. Adjust pH to 5.0 |
| HT Wash Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 25 mM imidazole. Adjust pH to 7.4 |
| Hydrochloric acid (HCl) | Thermo Fisher Scientific | A144C-212 | Used to pH buffers |
| Imidazole | Thermo Fisher Scientific | AAA1022122 | Mimics the histidine side group. Used to separate non-specifically binding proteins from the his-tagged target protein |
| Inclusion Body Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 5 mM DTT, 2% v/v Triton X 100, 0.5 M Urea. Adjust pH to 8.0 |
| Isopropyl-ß-D-thiogalactopyranoside (IPTG) | Thermo Fisher Scientific | FERR0392 | A reagent that induces target gene expression in pET3a. Make 0.5 mL 1 M aliquots, filter sterilize and store in -20 °C |
| LB Amp CamR media | n/a | n/a | To be poured into a sterible 1 L bottle or 1 L flask. For 1 L, add 25 g LB Broth. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Amp CamR plates | n/a | n/a | To be poured into sterile petri dishes. Pour until the petri dish lid is completely covered. 1 L of media yields 40-60 plates. For 1 L: 25 g LB Broth, 16 g Agar. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Broth | Thermo Fisher Scientific | BP1426-2 | Pre-mixed with tryptone, yeast extract, and sodium chloride |
| Lysis Buffer | n/a | n/a | 50 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 0.133 g/mL lysozyme, 0.49% v/v Triton X-100. Adjust pH to 8.0 |
| Lysozyme | MP Biomedicals | 195303 | Used in protein extraction. Enzyme that lyses bacterial cell walls |
| Miniprep kit | Promega | A1330 | If successful, extracts the pET3a-pro-MMP-3cd construct from transformants |
| NdeI | NEB | R0111S | Restriction enzyme to be used with the pET3a vector |
| Ni-NTA resin | Thermo Fisher Scientific | PI88221 | Used to bind recombinant his-tagged proteins. This strong interaction can be displaced with higher concentrations of imidazole |
| Nonionic surfactant | Thermo Fisher Scientific | PI28316 | Storage detergent for preventing MMP aggregation. Minimizes interactions between hydrophobic residues on the MMP surface and water molecules, without disrupting catalytic activity. |
| PCR mix | NEB | M0492S | A PCR reagent for inserting an N-terminal his-tag into the pET3a-pro-MMP-3cd vector |
| pET plasmid | Addgene | n/a | The pET3a vector offers ampicillin resistance, inducible expression of a target gene, and sequencing with T7 primers |
| Petri dishes | VWR | 25384-342 | Used for plating transformants on LB agar media |
| R2DP cells | Novagen | 714033 | BL21 derivatives with enhanced expression of eukaryotic proteins. Contain tRNAs of codons found to be rare in e. coli |
| SOC growth media | NEB | B9020S | Non-selective growth media for rapid growth during transformation |
| Sodium chloride (NaCl) | Thermo Fisher Scientific | BP358-1 | Used in buffers and helps with protein stability |
| Sodium deoxycholate | Thermo Fisher Scientific | PI89905 | Detergent used in protein extraction. Lyses cell walls |
| Solubilization Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 10 mM DTT, 6 M Urea. Adjust pH to 8.0 |
| Tris base | Thermo Fisher Scientific | BP152-1 | Common buffer used in the physiological pH range. Temperature-sensitive |
| Triton X-100 | Thermo Fisher Scientific | M1122980101 | Detergent used for cell lysis |
| Urea | Thermo Fisher Scientific | AAJ75826A7 | First chaotropic agent for disrupting protein secondary structure |
| Zinc chloride (ZnCl2) | Thermo Fisher Scientific | AAA162810E | Stabilizes MMP structure. The zinc ion is found in the catalytic site of MMP-3 |
Riferimenti
- Portolano, N., et al. Recombinant protein expression for structural biology in HEK 293F suspension cells: A novel and accessible approach. Journal of Visualized Experiments: JoVE. (92), e51897 (2014).
- Subedi, G. P., Johnson, R. W., Moniz, H. A., Moremen, K. W., Barb, A. High yield expression of recombinant human proteins with the transient transfection of HEK293 cells in suspension. Journal of Visualized Experiments: JoVE. (106), e53568 (2015).
- Nilvebrant, J., Alm, T., Hober, S. Orthogonal protein purification facilitated by a small bispecific affinity tag. Journal of Visualized Experiments: JoVE. (59), e3370 (2012).
- Yang, Z., et al. Highly efficient production of soluble proteins from insoluble inclusion bodies by a two-step-denaturing and refolding method. PLoS One. 6 (7), 22981 (2011).
- Hu, X., Beeton, C. Detection of functional matrix metalloproteinases by zymography. Journal of Visualized Experiments: JoVE. (45), e2445 (2010).
- Radisky, E. S., Raeeszadeh-Sarmazdeh, M., Radisky, D. C. Therapeutic potential of matrix metalloproteinase inhibition in breast cancer. Journal of Cellular Biochemistry. 118 (11), 3531-3548 (2017).
- Raeeszadeh-Sarmazdeh, M., Do, L. D., Hritz, B. G. Metalloproteinases and their inhibitors: Potential for the development of new therapeutics. Cells. 9 (5), 1313 (2020).
- Nagase, H., Visse, R., Murphy, G. Structure and function of matrix metalloproteinases and TIMPs. Cardiovascular Research. 69 (3), 562-573 (2006).
- Raeeszadeh-Sarmazdeh, M., et al. Directed evolution of the metalloproteinase inhibitor TIMP-1 reveals that its N- and C-terminal domains cooperate in matrix metalloproteinase recognition. Journal of Biological Chemistry. 294 (24), 9476-9488 (2019).
- Batra, J., et al. Matrix metalloproteinase-10 (MMP-10) interaction with tissue inhibitors of metalloproteinases TIMP-1 and TIMP-2. Journal of Biological Chemistry. 287 (19), 15935-15946 (2012).
- Singh, K. K., Jain, R., Ramanan, H., Saini, D. K., Galea, C. A. Expression and purification of matrix metalloproteinases in Escherichia coli. Matrix Metalloproteases. , 3-16 (2017).
- Manka, S. W., et al. Structural insights into triple-helical collagen cleavage by matrix metalloproteinase 1. Proceedings of the National Academy of Sciences of the United States of America. 109 (31), 12461-12466 (2012).
- Gomis-Ruth, F. X., et al. Mechanism of inhibition of the human matrix metalloproteinase stromelysin-1 by TIMP-1. Nature. 389 (6646), 77-81 (1997).
- Shirian, J., et al. Converting a broad matrix metalloproteinase family inhibitor into a specific inhibitor of MMP-9 and MMP-14. FEBS Letters. 592 (7), 1122-1134 (2018).
- Li, C., et al. Purification of recombinant histidine-tagged catalytic domain of MMP-13 in one step using affinity column and renaturation of it with histidine tag. Journal of Liquid Chromatography & Related Technologies. 37 (15), 2118-2130 (2014).
- Aydin, H., Azimi, F. C., Cook, J. D., Lee, J. E. A convenient and general expression platform for the production of secreted proteins from human cells. Journal of Visualized Experiments: JoVE. (65), e4041 (2012).
- McNiff, M. L., Haynes, E. P., Dixit, N., Gao, F. P., Laurence, J. S. Thioredoxin fusion construct enables high-yield production of soluble, active matrix metalloproteinase-8 (MMP-8) in Escherichia coli. Protein Expression and Purification. 122, 64-71 (2016).
- Maity, R., et al. GST-His purification: A two-step affinity purification protocol yielding full-length purified proteins. Journal of Visualized Experiments: JoVE. (80), e50320 (2013).
- Stefan, A., Ceccarelli, A., Conte, E., Montón Silva, A., Hochkoeppler, A. The multifaceted benefits of protein co-expression in Escherichia coli. Journal of Visualized Experiments: JoVE. (96), e52431 (2015).
- Yadavalli, R., Sam-Yellowe, T. HeLa based cell free expression systems for expression of Plasmodium rhoptry proteins. Journal of Visualized Experiments: JoVE. (100), e52772 (2015).
- Zeytuni, N., Zarivach, R. Purification of the M. magneticum strain AMB-1 magnetosome associated protein MamAΔ41. Journal of Visualized Experiments: JoVE. (37), e1844 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon