Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Bestimmung der in vitro und zellulären Einschaltkinetik für fluorogene RNA-Aptamere
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Das Protokoll stellt zwei Methoden vor, um die Kinetik der fluorogenen RNA-Aptamere Spinat2 und Brokkoli zu bestimmen. Die erste Methode beschreibt, wie die fluorogene Aptamerkinetik in vitro mit einem Plattenleser gemessen werden kann, während die zweite Methode die Messung der fluorogenen Aptamerkinetik in Zellen durch Durchflusszytometrie beschreibt.
Zusammenfassung
Fluorogene RNA-Aptamere wurden in lebenden Zellen eingesetzt, um RNAs zu markieren und sichtbar zu machen, über die Genexpression zu berichten und fluoreszierende Biosensoren zu aktivieren, die Konzentrationen von Metaboliten und Signalmolekülen erkennen. Um dynamische Änderungen in jedem dieser Systeme zu untersuchen, ist es wünschenswert, Echtzeitmessungen zu erhalten, aber die Genauigkeit der Messungen hängt davon ab, ob die Kinetik der fluorogenen Reaktion schneller ist als die Probenahmefrequenz. Hier beschreiben wir Methoden zur Bestimmung der in vitro und zellulären Einschaltkinetik für fluorogene RNA-Aptamere mit einem Plattenleser, der mit einem Probeninjektor bzw. einem Durchflusszytometer ausgestattet ist. Wir zeigen, dass die in vitro Kinetik für die Fluoreszenzaktivierung der Spinat2- und Brokkoli-Aptamere als zweiphasige Assoziationsreaktionen modelliert werden kann und unterschiedliche schnelle Phasenratenkonstanten von 0,56 s−1 bzw. 0,35 s−1 aufweist. Darüber hinaus zeigen wir, dass die zelluläre Kinetik für die Fluoreszenzaktivierung von Spinat2 in Escherichia coli, die durch die Farbstoffdiffusion in die gramnegativen Bakterien weiter begrenzt wird, immer noch schnell genug ist, um eine genaue Probenhäufigkeit auf der Minutenskala zu ermöglichen. Diese Methoden zur Analyse der Fluoreszenzaktivierungskinetik sind auf andere fluorogene RNA-Aptamere anwendbar, die entwickelt wurden.
Einleitung
Fluorogene Reaktionen sind chemische Reaktionen, die ein Fluoreszenzsignal erzeugen. Fluorogene RNA-Aptamere erfüllen diese Funktion typischerweise, indem sie einen kleinmolekularen Farbstoff binden, um seine Fluoreszenzquantenausbeute zu erhöhen (Abbildung 1A)1. Verschiedene fluorogene RNA-Aptamersysteme wurden entwickelt und bestehen aus spezifischen RNA-Aptamersequenzen und den entsprechenden Farbstoffliganden1. Fluorogene RNA-Aptamere wurden an RNA-Transkripte als fluoreszierende Tags angehängt, die die Bildgebung lebender Zellen von mRNAs und nicht-kodierenden RNAsermöglichen 2,3,4. Sie wurden auch nach Promotorsequenzen als fluoreszierende Reporter der Genexpression platziert, ähnlich der Verwendung von grün fluoreszierendem Protein (GFP) als Reporter, außer dass die Berichtsfunktion auf der RNA-Ebene 5,6 liegt. Schließlich wurden fluorogene RNA-Aptamere in RNA-basierte fluoreszierende Biosensoren eingebaut, die die fluorogene Reaktion als Reaktion auf ein bestimmtes kleines Molekül auslösen sollen. RNA-basierte fluoreszierende Biosensoren wurden für die Lebendzellbildgebung verschiedener nicht-fluoreszierender Metaboliten und Signalmoleküle 7,8,9,10,11 entwickelt.
Es besteht ein wachsendes Interesse an der Entwicklung fluorogener RNA-Aptamere zur Visualisierung dynamischer Veränderungen der RNA-Lokalisierung, der Genexpression und der niedermolekularen Signale. Für jede dieser Anwendungen ist es wünschenswert, Echtzeitmessungen zu erhalten, aber die Genauigkeit der Messungen hängt davon ab, ob die Kinetik der fluorogenen Reaktion schneller ist als die Probenahmefrequenz. Hier beschreiben wir Methoden zur Bestimmung der in vitro Kinetik für fluorogene RNA-Aptamere Spinat212 und Brokkoli13 mit einem mit einem Probeninjektor ausgestatteten Plattenleser und zur Bestimmung der zellulären Einschaltkinetik für Spinat2 exprimiert in Escherichia coli mittels Durchflusszytometer. Diese beiden RNA-Aptamere wurden ausgewählt, weil sie zur Untersuchung der RNA-Lokalisierung 2,3,4 eingesetzt wurden, sie wurden in den Reportern5,6 und Biosensoren 7,8,9,10,11 verwendet und die entsprechenden Farbstoffliganden (DFHBI oder DFHBI-1T) sind kommerziell erhältlich. Eine Zusammenfassung ihrer in der Literatur ermittelten In-vitro-Eigenschaften ist in Tabelle 1 4,13,14 enthalten, die in die Entwicklung des Protokolls einflossen (z. B. die verwendeten Wellenlängen und Farbstoffkonzentrationen). Diese Ergebnisse zeigen, dass die fluorogenen Reaktionen, die von RNA-Aptameren beeinflusst werden, schnell sind und genaue Messungen für die gewünschten zellbiologischen Anwendungen nicht behindern sollten.
Protokoll
1. In-vitro-Kinetik-Experiment
- Herstellung von DNA-Templates mittels PCR
- PCR-Reaktion(en) einrichten: Zur Vorbereitung von PCR-Reaktionen kombinieren Sie die folgenden Reagenzien in einem dünnwandigen PCR-Röhrchen:
33 μL doppelt destilliertes Wasser (ddH2O)
10 μL 5x Puffer für High-Fidelity-DNA-Polymerase
5 μL je 2 mM Desoxyribonukleosidtriphosphat (dNTP)
0,5 μL 40 μM Vorwärtsprimer
0,5 μL 40 μM Reverse-Primer
0,5 μL (10-100 ng) DNA-Vorlage (nur für Spinat2-PCR; Brokkoli-Primer überlappen)
0,5 μL High-Fidelity-DNA-Polymerase (zuletzt hinzufügen)
HINWEIS: Synthetische Oligonukleotide werden oft trocken verschifft. Um Stammlösungen herzustellen, fügen Sie ein bekanntes Volumen (100 μL empfohlen) von ddH2Ohinzuund messen Sie das A260 dieser Lösung, um die Konzentration nach dem Beerschen Gesetz zu bestimmen, wobei der Extinktionskoeffizient durch Nearest-Neighbor-Regeln online berechnet werden kann. Diese Stammlösung kann dann verwendet werden, um Verdünnungen vorzunehmen, die für die Verwendung in der PCR geeignet sind. - Führen Sie das Thermocycler-Protokoll aus.
- Verwenden Sie das folgende Thermocycler-Protokoll, um Spinat2- und Brokkoli-DNA-Vorlagen in voller Länge zu amplifizieren:
Erste Denaturierung:98 °C für 2 min
35 Zyklen:
Denaturierung:98 °C Denaturierung für 15 s
Glühen:72 °C für 30 s
Verlängerung:72 °C für 30 s
Endverlängerung: 72 °C für 5 min. - Analysieren Sie nach der Reaktion ein kleines Aliquot des Produkts mit 2% Agarosegel zusammen mit einer Leiter mit niedrigem Molekulargewicht (Größenbereich: 25-766 Nukleotide), um das Vorhandensein des gewünschten DNA-Produkts zu bestätigen.
- Reinigen Sie das Produkt durch handelsübliche Gelextraktion oder PCR-Reinigungskits und eluieren Sie mitddH2Ooder dem vom Hersteller bereitgestellten Puffer.
HINWEIS: Stellen Sie bei der Auswahl eines PCR-Aufreinigungskits sicher, dass der Molekulargewichtsgrenzwert niedrig genug ist, um T7-Brokkoli (81 Nukleotide) zurückzuhalten, da sonst das PCR-Produkt verloren geht.
HINWEIS: Optionaler Pausenpunkt: Speichern Sie die DNA bei −20 °C.
- Verwenden Sie das folgende Thermocycler-Protokoll, um Spinat2- und Brokkoli-DNA-Vorlagen in voller Länge zu amplifizieren:
- PCR-Reaktion(en) einrichten: Zur Vorbereitung von PCR-Reaktionen kombinieren Sie die folgenden Reagenzien in einem dünnwandigen PCR-Röhrchen:
- Herstellung von Spinat2 und Brokkoli-RNA durch in vitro Transkription (IVT)
- Richten Sie die Transkriptionsreaktion(en) ein.
- Um eine 100-μL-Transkriptionsreaktion herzustellen, kombinieren Sie die folgenden Reagenzien in einem 1,5-ml-Mikrozentrifugenröhrchen: 10 μL 10x Transkriptionspuffer + 20 μL 10 mM Ribonukleosidtriphosphate (rNTPs) + 1-64 μL DNA-Vorlage (insgesamt 1 μg) + 2 μL anorganische Pyrophosphatase +ddH2O bis 98 μL + 2 μL T7-RNA-Polymerase (zuletzt hinzufügen).
- Diese Reaktion wird für 4 h bei 37 °C inkubiert. Löschen Sie die Reaktion durch Zugabe von 100 μL 2x Harnstoff-Gel-Ladepuffer (2x ULB), bestehend aus 20% Saccharose, 0,1% Natriumdodecylsulfat (SDS), 1x Tris-Borat-EDTA (1x FSME) Puffer und ~18 M Harnstoff.
HINWEIS: Optionaler Pausenpunkt: Lagern Sie die gelöschte Reaktion bei −20 °C.
- Richten Sie die Transkriptionsreaktion(en) ein.
- Polyacrylamid-Gelelektrophorese (PAGE) Reinigung von RNA
- PAGE Reinigung von Spinat2 und Brokkoli-RNA
ACHTUNG: Nicht polymerisiertes (flüssiges oder pulverförmiges) Acrylamid ist extrem giftig. Wenn Sie pulverförmiges Acrylamid wiegen, tun Sie dies in einem Abzug. Tragen Sie immer die richtige Schutzausrüstung und entfernen Sie sofort Handschuhe, die mit Acrylamidpulver oder -lösung kontaminiert sind, und waschen Sie sich gründlich die Hände. Wenn Acrylamid in direkten Kontakt mit der Haut kommt, waschen Sie die exponierte Stelle mindestens 15 Minuten lang mit Wasser und Seife. Wenn Acrylamid in direkten Kontakt mit den Augen kommt, spülen Sie sie 15 min lang mit Wasser aus.- Bereiten Sie das PAGE-Gel vor: Um unerwünschte abortive Transkripte und nicht reagierte rNTPs aus dem Produkt in voller Länge zu entfernen, bereiten Sie ein 6% Harnstoff-Polyacrylamid-Gel vor. Im Allgemeinen können 28 cm x 16,5 cm x 1,5 mm Gele mit einem 8-Well-Kamm verwendet werden. Richten Sie die Gel- und Elektrophoreseausrüstung mit 1x FSME-Puffer ein, um die Reservoirs zu füllen.
- Laden Sie die RNA-Probe(n) in das PAGE-Gel: Laden Sie das Gel mit einer abgeschreckten 200-μL-Reaktion pro Bahn. In einer separaten Spur 2x ULB mit den Tracker-Farbstoffen Xylolcyanol und Bromphenolblau beladen, die im Gel bei 106 Nukleotiden bzw. 26 Nukleotiden bzw.15 migrieren. Lassen Sie eine leere Spur zwischen den einzelnen Proben, um eine mögliche Kontamination in den nächsten Schritten zu vermeiden.
- Führen Sie das PAGE-Gel aus: Um 95-nt-Spinat2 und 49-nt-Brokkoli von ihren jeweiligen abgeschnittenen Produkten zu trennen, lassen Sie das Gel 1,5-2 h bei 25 W laufen, woraufhin der Bromphenolblau-Farbstoff ~ 5/6 der Gellänge migriert ist.
- Visualisieren Sie die RNA-Probe(n) im PAGE-Gel: Zerlegen Sie die Glasplatten um das Gel herum und bedecken Sie das Gel auf beiden Seiten mit Plastikfolie und beschriften Sie die Bahnen auf der Folie. Visualisieren Sie RNA-Banden in einem dunklen Raum durch UV-Abschattung, indem Sie das eingewickelte Gel auf eine fluoreszierende TLC-Platte unter UV-Licht legen. Umreißen Sie schnell den Rand der RNA-Banden, die dem Produkt entsprechen, mit einem Marker und schalten Sie die UV-Lampe aus, um Schäden durch UV-Exposition zu minimieren.
- Schneiden und extrahieren Sie die RNA-Probe(n) aus dem PAGE-Gel: Mit einer frischen Rasierklinge für jede Probe die gewünschten Produktbänder herausschneiden, in ~1 mm Würfel würfeln und die Gelstücke in ein 2-ml-Mikrozentrifugenröhrchen mit 500 μL Crush-Einweichpuffer geben, um RNA auf einem Rotator entweder für 2 h bei Raumtemperatur (RT) oder über Nacht bei 4 °C zu extrahieren.
HINWEIS: Optionaler Pausenpunkt: Die Probe kann bei −20 °C gelagert werden. - Fällung der RNA
- Um die Gelstücke von der extrahierten RNA im Puffer zu trennen, zentrifugieren Sie die Probe bei 13.000 x g für 20 min bei 4 °C und verwenden Sie dann eine Pipette mit schmaler Spitze, um den Überstand zu extrahieren und in ein neues 2-ml-Mikrozentrifugenröhrchen zu laden.
- Um die RNA auszufällen, fügen Sie 1,5 ml eiskaltes Ethanol und 1 μL 20 mg / ml Glykogen hinzu, Wirbel und lagern Sie für mindestens 1 h bei -20 ° C oder -80 ° C.
HINWEIS: Optionaler Pausenpunkt: Die RNA kann für einige Monate bei −20 °C gelagert werden.
- Sammlung von RNA-Niederschlag: Pelletieren Sie die gefällte RNA durch Zentrifugation bei 13.000 x g für 20 min bei 4 °C. Entfernen Sie den Überstand und lassen Sie das verbleibende Ethanol unter freiem Himmel (~1 h) verdampfen, bevor Sie das Pellet in 30 μLddH2Ooder 1x TE-Puffer resuspendieren.
HINWEIS: Dieser Prozess führt typischerweise zu einer endgültigen RNA-Konzentration von ~10 μM.
HINWEIS: Optionaler Pausenpunkt: Die RNA-Probe kann einige Monate bei −20 °C gelagert werden.
- Bestimmen Sie die RNA-Bestandskonzentrationen.
- Bereiten Sie ein RNA-Aliquot für die Hydrolysereaktion vor.
- Um diesen Assay durchzuführen, verwenden Sie zunächst ein UV / Vis-Nano-Spektralphotometer, um das A260 der Stamm-RNA-Probe zu bestimmen und ein verdünntes Aliquot der Probe auf ~ 10 Absorptionseinheiten (AE) in ddH2O zu machen.
- Die folgende Reaktion wird in einem 0,5 mL PCR-Röhrchen hergestellt: 16 μLddH2O+ 2 μL 10x Na2CO3-Puffer+ 2 μLRNA-Aliquot verdünnt auf ~10 AE. Die Reaktion 90 min bei 95 °C inkubieren und dann auf RT abkühlen lassen.
- Bestimmung der RNA-Konzentration mit Hilfe von Nukleotidabsorptionen: Messen Sie das A260 dieser Probe mit einem UV/Vis-Nano-Spektralphotometer und berechnen Sie die RNA-Konzentration nach folgender Formel:

wobei c die Konzentration der RNA, b die Weglänge, i das spezifische Nukleotid (A, C, G oder U), n i die Frequenz des Nukleotids i in der RNA-Sequenz und ε i der molare Extinktionskoeffizient von Nukleotid i ist. Um die ursprüngliche Stamm-RNA-Konzentration zu bestimmen, multiplizieren Sie c mit dem Verdünnungsfaktor.
HINWEIS: Optionaler Pausenpunkt: Die RNA kann einige Monate bei −20 °C gelagert werden.
- Bereiten Sie ein RNA-Aliquot für die Hydrolysereaktion vor.
- PAGE Reinigung von Spinat2 und Brokkoli-RNA
- Ausführen einer in vitro Plattenleser-Kinetik Assay
- Einrichten des RNA-Renaturierungsprogramms: Erstellen Sie das folgende Thermocycler-Protokoll, indem Sie Neues Programm erstellen > Neue Phase hinzufügen > Neuen Schritt hinzufügen mehrmals auswählen, um jeden der folgenden Schritte hinzuzufügen, bevor Sie auf Speichern klicken:
70 °C für 3 min
65 °C für 45 s
60 °C für 45 s
55 °C für 45 s
50 °C für 45 s
45 °C für 45 s
40 °C für 45 s
35 °C für 45 s
30 °C für 45 s - Renaturierung der RNA: Um Spinat2- und Brokkoli-RNAs zu renaturieren, bereiten Sie 2 μM-Bestände jeder RNA inddH2Oin einem 0,5 mL dünnwandigen PCR-Röhrchen vor und fügen Sie dann ein gleiches Volumen von 2x Renaturierungspuffer (80 mM HEPES, pH 7,5 [KOH], 250 mM KCl, 6 mM MgCl2) hinzu, um 1 μM RNA-Lösungen herzustellen. Fügen Sie die Rohre in den Thermocycler ein, öffnen Sie das gespeicherte Renaturierungsprogramm und drücken Sie Ausführen.
HINWEIS: Wenn kein Thermocycler verfügbar ist, können die RNAs stattdessen auf einem 70 °C heißen Hitzeblock für 3 min inkubiert und dann langsam auf RT auf dem Prüfstand abkühlen gelassen werden. - Vorbereitung des Bindungsreaktionspuffers: Um den Puffer für eine Aptamer-Farbstoff-Bindungsreaktion vorzubereiten, wird eine Mastermischung mit Pufferkomponenten (69,5 μL ddH2O + 4 μL 1 M HEPES, pH 7,5 [KOH] + 6,2 μL 2 M KCl + 0,3 μL 1 MMgCl2) hergestellt. Je nachdem, wie viele Proben und Replikate benötigt werden, multiplizieren Sie diese Werte mit der Anzahl der Proben plus eins. Im Allgemeinen sind drei Replikate pro RNA-Probe zufriedenstellend.
- Bereiten Sie das Plattenlesegerät vor.
- Richten Sie das Injektorprogramm des Plattenlesers ein: Wählen Sie auf dem Fluoreszenzplattenleser Temp und stellen Sie die gewünschte Temperatur auf 37 °C ein, um sicherzustellen, dass sich die Temperatur rechtzeitig auf diesen Wert eingestellt hat, bevor Sie mit kinetischen Experimenten beginnen. Öffnen Sie die Plattenlesesoftware, wählen Sie Einstellungen > Erfassungsansicht, und geben Sie das folgende Programm für kinetische Messungen ein:
Schleife: Für jeden Brunnen
Baseline-Einstellung: 60 Baseline-Lesevorgänge
SmartInject-Einstellungen: 10-μL-Injektion (die nach dem Lesen der Baseline erfolgt)
Fluoreszenz (oder FL) lautet:
Anregung: 448 nm (Bandbreite: 9 nm)
Emission: 506 nm (Bandbreite: 15 nm)
Patrone: MONO (s/n 3297)
Timing:
Gesamtlesezeit: 10 min
Leseintervall: 0,5 s
PMT und Optik: 6 Blitze pro Lesevorgang
Schleife: Nächster Brunnen - Vorbereiten des Injektors
- Injektor waschen und absaugen: Um den Plattenleser-Injektor vorzubereiten, wählen Sie zuerst Injizieren auf dem Plattenleser, liefern Sie dem Plattenlesegerät eine Abfallsammelplatte, wenn Sie dazu aufgefordert werden, und wählen Sie dann Waschen und reinigen Sie das Injektionsrohr gemäß den Anweisungen auf dem Plattenleser mit 1 ml Volumen ddH2O, 75% Ethanol in ddH2O, und dann ddH2O. Als nächstes wählen Sie Aspirate, damit der Injektor überschüssige Flüssigkeit ausstoßen kann.
- Prime-Injektor: Wählen Sie nach dem Verlassen des vorherigen Bildschirms Prime aus, um den Injektor mit zwei 260-μL-Volumen Ligand zu grundieren, der injiziert werden soll, um sicherzustellen, dass während der Experimente reiner, konzentrierter Ligand zu Proben hinzugefügt wird - in diesem Fall Prime mit 100 μM DFHBI in ddH2O.
- Richten Sie das Injektorprogramm des Plattenlesers ein: Wählen Sie auf dem Fluoreszenzplattenleser Temp und stellen Sie die gewünschte Temperatur auf 37 °C ein, um sicherzustellen, dass sich die Temperatur rechtzeitig auf diesen Wert eingestellt hat, bevor Sie mit kinetischen Experimenten beginnen. Öffnen Sie die Plattenlesesoftware, wählen Sie Einstellungen > Erfassungsansicht, und geben Sie das folgende Programm für kinetische Messungen ein:
- Durchführung von In-vitro-Kinetik-Experimenten: Um ein Kinetik-Experiment durchzuführen, fügen Sie zuerst 80 μL zuvor vorbereitete Bindungspuffer-Mastermischung in eine Vertiefung einer 96-Well-Clear-Bottom-Platte hinzu, gefolgt von 10 μL renaturierter RNA. Lassen Sie diese Platte und die DFHBI-Lösung im Injektor für 15 min im Plattenleser auf 37 °C ausgleichen.
- Wählen Sie in den Einstellungen der Plattenlesersoftware unter Lesebereich die zu analysierende Vertiefung aus, und wählen Sie dann auf der Registerkarte Start die Option Ausführen aus, um das zuvor beschriebene Kinetik auszuführen. Wiederholen Sie diesen Vorgang, bis alle Experimente abgeschlossen sind.
HINWEIS: Entscheidend ist, dass kinetische Experimente nacheinander durchgeführt werden sollten, um sicherzustellen, dass die RNA-Kinetik unter identischen Bedingungen zwischen Replikaten und Proben gemessen wird. - Injektor waschen: Um die verbleibende DFHBI-Lösung aus dem Injektionsröhrchen zu entfernen, waschen Sie das Injektionsrohr wie in Schritt 1.4.4.2.1 beschrieben mit 1 ml Volumen ddH2O, 75% Ethanol inddH2Ound dannddH2O.
HINWEIS: Optionaler Pausenpunkt.
- Einrichten des RNA-Renaturierungsprogramms: Erstellen Sie das folgende Thermocycler-Protokoll, indem Sie Neues Programm erstellen > Neue Phase hinzufügen > Neuen Schritt hinzufügen mehrmals auswählen, um jeden der folgenden Schritte hinzuzufügen, bevor Sie auf Speichern klicken:
- Analyse der in vitro Kinetik fluorogener RNA-Aptamere.
- Daten in die Analysesoftware eingeben: Exportieren Sie experimentelle Daten als Tabelle, um die Daten zur Verarbeitung einfach zu kopieren. Erstellen Sie in der Analysesoftware eine neue Datentabelle im XY-Format. Geben Sie in der Spalte X jeden Lesezeitpunkt ein, wobei t = 0 der Zeitpunkt der DFHBI-Injektion ist. Geben Sie in der Spalte Y die durchschnittlichen Fluoreszenzwerte zwischen Replikaten zu diesem jeweiligen Zeitpunkt ein, beginnend bei t = 0.
- Normalisieren und grafisch darstellen: Um die Fluoreszenzwerte zu normalisieren, klicken Sie auf Analysieren > Datenverarbeitung > Normalisieren und dann auf OK. Wählen Sie aus, ob Sie die kleinsten und größten Werte des Datensatzes für die Normalisierung verwenden möchten, um die Ergebnisse als Brüche darzustellen und die Ergebnisse grafisch darzustellen, und klicken Sie dann auf OK. Um ein Diagramm der normalisierten gemittelten Fluoreszenz im Zeitverlauf zu erstellen, klicken Sie auf das Symbol Normalisieren von [Datensatzname] und wählen Sie Graphfamilie: XY nur mit Punkten.
HINWEIS: Fehlerindikatoren können nützlich sein, um in Fällen zu sehen, in denen eine kleine Anzahl von Zeitpunkten grafisch dargestellt wird. Wenn diese gewünscht sind, wählen Sie bei der Auswahl einer Datentabelle im XY-Format die Option unter "Y", um Replikationswerte in nebeneinander liegende Spalten einzugeben. Wenn Sie die resultierende Tabelle mit den ausgewählten Standardoptionen grafisch darstellen, wird ein Diagramm mit Fehlerindikatoren erstellt. - Kurvenanpassung durchführen, um kinetische Parameter zu erhalten: Um eine Kurve an die Kinetik anzupassen, klicken Sie auf Analysieren > Daten analysieren und wählen Sie auf der Registerkarte XY-Analysen die Option Nichtlineare Regression (Kurvenanpassung). Klicken Sie auf der Registerkarte Modell auf Exponentielle > Zweiphasenassoziation, um die Kinetik mit der folgenden Zwei-Phasen-Assoziationsgleichung anzupassen:
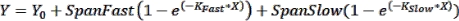
wobei Y die Fluoreszenz zum Zeitpunkt X ist, Y 0 die Fluoreszenz bei t = 0, K Fast und K Slow schnelle bzw. langsame Geschwindigkeitskonstanten sind und SpanFast und SpanSlow die Bereiche der Fluoreszenzeinschaltung sind, die durch die schnellen bzw. langsamen Raten berücksichtigt werden (siehe repräsentative Ergebnisse, Abbildung 1). Klicken Sie auf die Registerkarte Nonlin Fit, um Ratenkonstanten, t1/2-Werte und PercentFast-Werte anzuzeigen.
HINWEIS: Um eine Standardabweichung für alle diese Werte zu erhalten, können einzelne Plattenleserexperimente auf die gleiche Weise wie oben beschrieben verarbeitet werden.
2. Zelluläres Kinetik Experiment
- Herstellung von E. coli-Stämmen
- Transformieren Sie BL21 Star (DE3) E. coli Zellen mit ~100 ng pET31b tRNA-Spinat2 Konstrukt nach dem Protokoll des Herstellers.
HINWEIS: Das Plasmidkonstrukt ist im Handel erhältlich (Plasmid #79783). - Die Zellen auf LB-Agar, das Carbenicillin (Carb: 50 mg/ml) enthält, platten und bei 37 °C für 12-16 h inkubieren. Zellen, die Plasmide enthalten, wachsen als Kolonien auf der Platte.
HINWEIS: Optionaler Pausenpunkt: Transformierte BL21 Star-Zellen auf Platten können 1 Woche lang bei 4 °C in Parafilm verpackt gelagert werden.
- Transformieren Sie BL21 Star (DE3) E. coli Zellen mit ~100 ng pET31b tRNA-Spinat2 Konstrukt nach dem Protokoll des Herstellers.
- Wachsende Zellen und Induktion der Expression von fluorogenem RNA-Aptamer
- Beimpfen Sie eine 2-ml-Kultur aus nichtinduzierenden Medien (NI), die Carbenicillin (Kohlenhydrat: 50 mg/ml) enthalten, mit einer einzigen Kolonie der transformierten BL21 Star-Zellen. Wiederholen Sie dies für mindestens drei biologische Replikate. Die Kulturen bei 37 °C in einem Inkubator/Shaker bei 250 U/min für 22-24 h inkubieren.
HINWEIS: Optionaler Pausenpunkt: In NI-Medien gezüchtete Zellen behalten das Plasmid und können 1 Woche lang bei 4 °C gelagert werden. - Nach dem Wachstum in NI-Medien wird die Kultur 100x in frische 3 ml ZYP-5052-Autoinduktionsmedien (AI) mit Carbenicillin (Kohlenhydrat: 50 mg/ml) verdünnt. Züchten Sie die Zellen bei 37 °C in einem Inkubator/Shaker bei 250 U/min für 16-18 h, um die Expression zu induzieren.
HINWEIS: Der typische OD600 für Kulturen liegt nach 18 Stunden Wachstum zwischen 2,0 und 3,3. Der optimale Zelldichtebereich liegt zwischen 2,5-3,0.
- Beimpfen Sie eine 2-ml-Kultur aus nichtinduzierenden Medien (NI), die Carbenicillin (Kohlenhydrat: 50 mg/ml) enthalten, mit einer einzigen Kolonie der transformierten BL21 Star-Zellen. Wiederholen Sie dies für mindestens drei biologische Replikate. Die Kulturen bei 37 °C in einem Inkubator/Shaker bei 250 U/min für 22-24 h inkubieren.
- Durchführung des zellulären Kinetik-Experiments
- Richten Sie das Durchflusszytometer ein.
- Schalten Sie das Durchflusszytometer und den an das Gerät angeschlossenen Computer ein. Sobald Sie sich bei der Software für das Durchflusszytometer angemeldet haben, klicken Sie auf der Registerkarte Instrument auf das Startsymbol . Befolgen Sie die auf dem Softwarebildschirm angezeigten Schritte, um die ordnungsgemäße Initialisierung der Instrumentierung sicherzustellen.
HINWEIS: Einige Durchflusszytometer nennen die Startsequenz des Instruments Priming. Stellen Sie sicher, dass Sie das Protokoll des Herstellers für das Durchflusszytometer befolgen, das für das Experiment verwendet wird. - Führen Sie ggf. einen Leistungstest aus. Klicken Sie auf der Registerkarte Hauptmenü auf Leistungstest. Fügen Sie in einem Kulturröhrchen drei Tropfen der Leistungsverfolgungsperlen des Herstellers in 3 ml Fokussierflüssigkeit hinzu.
- Legen Sie das Kulturröhrchen in den Probenschlauchheber und heben Sie den Heber an. Bevor Sie auf Leistungstest ausführen klicken, stellen Sie sicher, dass das Los # des Röhrchens der Tracking-Perlen mit dem auf dem Bildschirm Leistungstest-Setup angegebenen übereinstimmt. Klicken Sie auf Leistungstest, um den Test auszuführen.
- Richten Sie die Durchflusszytometer-Software für dieses Experiment mit den folgenden Erfassungsparametern für Einzelzellfluoreszenz ein:
Anregungslaser: 488 nm
Emissionskanal: GFP (auch FITC genannt)
Erfassungsvolumen: 40 μL (mit einem Gesamtziehvolumen von 90 μL)
Durchflussrate: 200 μL/ min
Zellzahlen für jede Messung: 30.000
Geräteeinstellungen:
Spannung:
FSC: 480 V
SSC: 400 V
BL1: 540 V
BL2: 392 V
BL3: 422 V
- Schalten Sie das Durchflusszytometer und den an das Gerät angeschlossenen Computer ein. Sobald Sie sich bei der Software für das Durchflusszytometer angemeldet haben, klicken Sie auf der Registerkarte Instrument auf das Startsymbol . Befolgen Sie die auf dem Softwarebildschirm angezeigten Schritte, um die ordnungsgemäße Initialisierung der Instrumentierung sicherzustellen.
- Richten Sie die experimentellen Dateien ein.
- Erstellen Sie eine neue Experimentdatei auf der Registerkarte Experiment-Explorer, indem Sie mit der rechten Maustaste auf den Benutzernamen des Durchflusszytometers klicken. Wählen Sie im Dropdown-Fenster Neues Experiment aus. Wenn ein neues Fenster auf dem Computerbildschirm angezeigt wird, wählen Sie OK.
- Klicken Sie in der neuen Experimentdatei mit der rechten Maustaste auf den Ordner "Gruppe" und wählen Sie Neues Probenröhrchen hinzufügen. Beschriften Sie die Probenröhrchen für jeden bestimmten Zeitpunkt und replizieren Sie, indem Sie mit der rechten Maustaste auf Probe klicken und im Dropdown-Menü Umbenennen auswählen. Wiederholen Sie diesen Schritt für die Gesamtzahl der Wiederholungen und Zeitpunkte für den beabsichtigten Studienverlauf.
- Bereiten Sie eine verdünnte Zelllösung vor: In einem neuen Kulturröhrchen 1,5 ml 1x PBS-Lösung hinzufügen. Als nächstes fügen Sie 3 μL induzierte Zellen in AI-Medien in die 1x PBS-Lösung hinzu, um eine verdünnte Zelllösung herzustellen. Wiederholen Sie diesen Schritt für jede biologische Replikation in verschiedenen Kulturröhrchen.
- Messen Sie die Hintergrundfluoreszenz der Zellen: Bevor Sie Farbstoff hinzufügen, messen Sie jedes biologische Replikationskulturröhrchen, das Zellen in 1x PBS-Lösung enthält. Dies ist so, dass der fluoreszierende Hintergrund der Zellen gemessen wird, um zu beobachten, wie sich die Falte im Laufe der Zeit anschaltet, sobald der Farbstoff hinzugefügt wird.
- Um eine Messung durchzuführen, legen Sie das Kulturröhrchen in den Probenröhrchenheber und heben Sie den Heber von Hand zur Probeninjektionsnadel an. Wählen Sie auf der Registerkarte "Sammelbereich " die richtige Beispieldatei aus und klicken Sie auf "Aufzeichnen".
- Wenn der Lauf abgeschlossen ist, senken Sie den Probenschlauchheber mit dem Kulturröhrchen von Hand. Dadurch wird ein Spülschritt eingeleitet, der das fluidische System spült und die Verschleppung zwischen jeder biologischen Replikationsprobe minimiert. Die Daten werden nach Abschluss des Laufs automatisch auf dem Computer gespeichert.
- Wiederholen Sie die Schritte innerhalb von 2.3.4, um den zellulären fluoreszierenden Hintergrund für mindestens drei biologische Replikate zu messen. Um zur nächsten Probendatei zu wechseln, wählen Sie die nächste Probendatei aus, indem Sie auf das Pfeilsymbol nach rechts neben dem Namen des Probenröhrchens unter dem Symbol "Aufnahme" klicken.
- Messen Sie die Fluoreszenz zu bestimmten Zeitpunkten für Zellen mit Farbstoff.
- 1,4 μL eines konzentrierten Farbstoffmaterials (50 mM DFHBI-1T in DMSO) in 1x PBS-Lösung mit Zellen geben, um eine Endkonzentration von 50 μM DFBHI-1T zu erhalten. Als nächstes sichern Sie den Deckel des Kulturröhrchens und kehren Sie dann die Kulturröhrchen 3x-5x um, um die Lösung gleichmäßig zu mischen, bevor Sie den ersten Zeitpunkt lesen.
HINWEIS: Der Gesamtprozentsatz von DMSO in den Kulturröhrchen für E. coli sollte 10% nicht überschreiten, da dies die Zelllebensfähigkeit beeinträchtigen kann16. - Entfernen Sie den Deckel und legen Sie das Kulturröhrchen in den Probenröhrchenheber. Heben Sie den Halter von Hand auf die Probeninjektionsnadel und klicken Sie unter der richtigen Probendatei auf das Symbol Aufnahme . Starten Sie außerdem einen Timer, indem Sie für das Experiment auf Start drücken.
- Senken Sie den Schlauchheber nach Abschluss des Laufs von Hand ab und wiederholen Sie die Schritte 2.3.5.1-2.3.5.2. (bei laufendem Timer) durch Zugabe von 1,4 μL des konzentrierten DFHBI-1T, Invertieren der Kulturröhrchen und Messen für alle verbleibenden biologischen Replikate. Diese ersten Aufzeichnungen werden die Messwerte sein, die nach 0 min für alle biologischen Replikate erhalten wurden. Tun Sie dies nacheinander für jede biologische Replikation.
HINWEIS: Notieren Sie sich den Zeitpunkt, zu dem die Erfassung der Datensatzdurchflusszytometrie gedrückt wird. Halten Sie sich an diese Zeitstaffelung für Zeitpunkte im Laufe des Experiments. - Fahren Sie mit den Messungen fort, indem Sie die Kulturröhrchen im Probenröhrchenheber zur Probeninjektionsnadel anheben, die richtige Probendatei auswählen, auf Aufzeichnen klicken und den Lifter nach Abschluss jedes Durchlaufs von Hand absenken. Tun Sie dies für alle zusätzlichen Zeitpunkte und biologischen Replikate, die getestet werden. Wiederholen Sie die Schritte, bis der Test abgeschlossen ist.
HINWEIS: Halten Sie die Proben vor Licht, um das Photobleichen von DFHBI-1T in Lösung zu vermeiden, indem Sie die Proben mit Aluminiumfolie abdecken.
- 1,4 μL eines konzentrierten Farbstoffmaterials (50 mM DFHBI-1T in DMSO) in 1x PBS-Lösung mit Zellen geben, um eine Endkonzentration von 50 μM DFBHI-1T zu erhalten. Als nächstes sichern Sie den Deckel des Kulturröhrchens und kehren Sie dann die Kulturröhrchen 3x-5x um, um die Lösung gleichmäßig zu mischen, bevor Sie den ersten Zeitpunkt lesen.
- Fluoreszenz zu Zeitpunkten für Zellen ohne Farbstoff messen (Control).
- Führen Sie die entsprechenden Reinigungsprotokolle für das Durchflusszytometer durch, bevor Sie den Experiment mit Negativkontrollen gemäß dem Protokoll des Herstellers wiederholen. Dies geschieht, um jegliche Übertragung aus dem vorherigen Experiment in das Kontrollzeitpunktanalyseexperiment zu minimieren. Im Folgenden sind die Schritte aufgeführt, die für das Durchflusszytometer zwischen den Experimenten ausgeführt wurden:
- Legen Sie ein leeres Kulturrohr von Hand in den Schlauchheber, heben Sie den Schlauchhalter an und klicken Sie auf das Symbol Unclog in der Registerkarte Instrument . Dadurch wird eine Rückspülung im Fluidiksystem ausgeführt, um klebrige Proben zu reinigen. Senken Sie den Tubenhalter von Hand ab und entfernen Sie den Schlauch, sobald die Unclog-Sequenz abgeschlossen ist.
- Bei einem neuen Kulturröhrchen 3 ml einer 10%igen Bleichlösung hinzufügen, das Kulturröhrchen in den Röhrchenhalter geben und den Halter von Hand zur Probeninjektionsnadel anheben. Legen Sie zusätzlich eine saubere 96-Well-Platte in den Autosampler, falls für das Durchflusszytometer geeignet.
- Klicken Sie auf das Symbol Sanitize SIP /Sanitize Autosampler SIP , um eine Reinigungssequenz mit 10% Bleichmittel im gesamten Fluidiksystem durchzuführen. Senken Sie den Schlauchhalter ab, um die Reinigungssequenz abzuschließen.
- Legen Sie die Beispieldateien für den Kontrollzeitpunktanalyselauf gemäß den Anweisungen in Schritt 2.3.3 fest.
- In einem neuen Kulturröhrchen eine verdünnte Zelllösung in 1,5 ml 1x PBS-Lösung herstellen. Geben Sie 3 μL der induzierten Zellen in AI-Medien in die 1x PBS-Lösung, um eine verdünnte Zelllösung herzustellen. Wiederholen Sie diesen Schritt für jede biologische Replikation.
- Geben Sie 1,4 μL DMSO nacheinander in das Kulturröhrchen in die 1x PBS-Lösung mit Zellen und testen Sie die gleichen Zeitpunkte. Befestigen Sie den Deckel des Kulturröhrchens und invertieren Sie dann die Kulturröhrchen 3x-5x, um die Lösung gleichmäßig zu mischen, bevor Sie die erste Zeitmessung durchführen. Tun Sie dies nacheinander für jede biologische Replikation.
- Befolgen Sie für die Analyse von Kontrollzellen dasselbe Protokoll wie für Zellen mit Farbstoff gemäß den Schritten 2.3.5.2-2.3.5.4.
- Führen Sie die entsprechenden Reinigungsprotokolle für das Durchflusszytometer durch, bevor Sie den Experiment mit Negativkontrollen gemäß dem Protokoll des Herstellers wiederholen. Dies geschieht, um jegliche Übertragung aus dem vorherigen Experiment in das Kontrollzeitpunktanalyseexperiment zu minimieren. Im Folgenden sind die Schritte aufgeführt, die für das Durchflusszytometer zwischen den Experimenten ausgeführt wurden:
- Herunterfahren des Durchflusszytometers: Befolgen Sie das Protokoll des Herstellers für das ordnungsgemäße Herunterfahren der Instrumentierung. Für das Durchflusszytometer ist das Gerät wie folgt für die Abschaltung vorbereitet:
- Führen Sie das Erstreinigungsprotokoll für das Durchflusszytometer gemäß den Schritten 2.3.6.1.1-2.3.6.1.3 durch.
- Ersetzen Sie das Kulturröhrchen mit 10% Bleichlösung durch ein Kulturröhrchen mit 3 ml Fokussierflüssigkeit. Heben Sie den Röhrenhalter von Hand an, klicken Sie unter dem Symbol Herunterfahren auf das Dropdown-Menü und wählen Sie Gründlich aus.
HINWEIS: Optionaler Pausenpunkt.
- Richten Sie das Durchflusszytometer ein.
- Analyse der Durchflusszytometriedaten
- Exportieren Sie alle FCS-Dateien zur Analyse. Öffnen Sie die FCS-Dateien mit einer Durchflusszytometrie-Analysesoftware.
- Generieren Sie mit einer der FCS-Dateien nur Zellen ein Tor aus dem Vorwärtsstreu- (FSC) und Seitenstreupunktdiagramm (SSC) (FSC-Area/SSC-Area), wobei beide Log-Achsen verwendet werden, um Signale von Trümmern auszuschließen. Um dieses Tor zu erstellen, klicken Sie auf das AutoGate-Symbol in der Durchflusszytometrie-Analysesoftware und nennen Sie es Gate 1. Wenden Sie dasselbe Gate auf alle getesteten Samples auf der Registerkarte All Samples im Arbeitsbereich Datenverarbeitung an. Dies führt dazu, dass sich Gate 1 unter allen FCS-Dateien befindet, die verarbeitet werden.
- Erstellen Sie eine neue Teilmengendatei mit der in Schritt 2.4.2 verwendeten FCS-Datei nur Zelle, indem Sie darauf doppelklicken. Ändern Sie die Achseneinstellungen in FSC-Area/FSC-Height, wobei beide Protokollachsen verwenden. Klicken Sie auf das AutoGate-Symbol in der Durchflusszytometrie-Analysesoftware, um ein ovales Tor mit dem Namen Gate 2 zu generieren. Wenden Sie dieses Gate als Teilmenge unterhalb des in Schritt 2.4.2 festgelegten Gattersatzes auf alle getesteten Proben an. Dies führt dazu, dass allen Beispielen Gate 1 > Gate 2 zugeordnet ist.
- Erstellen Sie eine weitere Teilmengendatei, bei der beide Gates in Schritt 2.4.2 und Schritt 2.4.3 festgelegt sind, indem Sie auf Gate 2 doppelklicken. Ändern Sie die Achseneinstellungen in FSC-Area/Histogram. Wenden Sie dieses Histogramm-Gate als Teilmenge auf alle getesteten Proben an, was dazu führt, dass allen Proben Gate 1 > Gate 2 > Gate 2 zugeordnet ist.
HINWEIS: Die Histogramme können von Gate 2 in Histogramm umbenannt werden, um das Verschieben der Histogramme in das Layoutfenster zu erleichtern und mehr Organisation bei der Datenverarbeitung zu schaffen. - Um die mittleren Fluoreszenzintensitätswerte (MFI) zu analysieren, öffnen Sie das Layoutfenster. Klicken Sie auf die Histogramm-Gates für jeden Zeitpunkt und ziehen Sie sie in das Layoutfenster.
- Führen Sie für jede getestete Stichprobe eine statistische Analyse für "∑ Mittelwert: BL1-A" (GFP) durch, um die MFI-Ergebnisse im Layoutfenster anzuzeigen.
- Berechnen Sie die Standardabweichung für die MFI-Werte pro Zeitpunktanalyse für mindestens drei unabhängige biologische Replikate.
- Speichern Sie die Histogramme und MFI-Werte für jeden Zeitpunkt, indem Sie das Layoutfenster als PDF-Datei exportieren.
HINWEIS: Optionaler Pausenpunkt.
- Graphische Durchflusszytometrie-Daten
- Öffnen Sie die PDF-Datei mit den Histogrammen und MFI-Werten für jeden Zeitpunkt. Die MFI-Werte werden in eine Datenanalysesoftware kopiert. Erstellen Sie in der Datengrafiksoftware eine neue Datentabelle im XY-Format.
- Wählen Sie diese Option, um eine XY-Tabelle mit ausgewählter Option zu erstellen:
Datentabelle: Eingeben oder Importieren von Daten in eine neue Tabelle
Optionen:
X: Verstrichene Zeiten
Y: Geben Sie (drei bis vier) Replikationswerte in nebeneinander liegenden Unterspalten ein. - Beschriften Sie auf der X-Achse alle Zeitpunkte für die Experiment- und Kontrollläufe.
- Geben Sie in Gruppe A die MFI-Werte für alle biologischen Replikate in jeden Zeitpunkt für die Fluoreszenzanalyse der Zellen mit dem hinzugefügten Farbstoff ein.
- Geben Sie in Gruppe B die MFI-Werte für alle biologischen Replikate in jeden Zeitpunkt ein, um die Zellen ohne Farbstoff (DMSO) zu analysieren.
- Um die Ergebnisse zu beobachten, klicken Sie auf der Registerkarte Diagramme auf [Datensatzname einfügen]. Dadurch werden die Datenpunkte als Mittelwert angezeigt, wobei Fehlerbalken die Standardabweichung (s.d.) zu jedem Zeitpunkt darstellen. Die X-Achse stellt die verstrichene Zeit dar, und die Y-Achse stellt die MFI-Werte dar.
- Wählen Sie diese Option, um eine XY-Tabelle mit ausgewählter Option zu erstellen:
- Öffnen Sie die PDF-Datei mit den Histogrammen und MFI-Werten für jeden Zeitpunkt. Die MFI-Werte werden in eine Datenanalysesoftware kopiert. Erstellen Sie in der Datengrafiksoftware eine neue Datentabelle im XY-Format.
Ergebnisse
In-vitro-Kinetik
Die Sequenzen der DNA-Vorlagen und Primer, die als synthetische Oligonukleotide gekauft werden, sind in Tabelle 2 und die Reagenzienrezepte in der Zusatzdatei 1 aufgeführt. Die PCR-Amplifikation wird verwendet, um die Menge der DNA-Vorlage mit dem T7-Promotor zu erhöhen, die für die nachfolgende In-vitro-Transkriptionsreaktion (IVT) erforderlich ist. Darüber hinaus kann die PCR-Amplifikation für zwei Zwecke in...
Diskussion
Für das In-vitro-Kinetik-Experiment kann das gleiche allgemeine Protokoll modifiziert werden, um die In-vitro-Kinetik eines RNA-basierten fluoreszierenden Biosensors zu messen, der sowohl eine ligandenbindende als auch eine fluorophorbindende Domäne8 enthält. In diesem Fall sollte die RNA vor Messungen nach Injektion des Liganden mit dem Fluorophor inkubiert werden, um eine Ligandenantwortkinetik zu erhalten. Wenn eine hohe Variabilität zwischen den Replikaten beobachtet wir...
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Diese Arbeit wurde durch die folgenden Zuschüsse an MCH unterstützt: NSF-BSF 1815508 und NIH R01 GM124589. MRM wurde teilweise durch das Ausbildungsstipendium NIH T32 GM122740 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
Referenzen
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295 (2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139 (2016).
- Paige, J. S., Thinh, N. -. D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194 (2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002 (2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten