É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinação da Cinética de Ativação In Vitro e Celular para Aptamers de RNA Fluorogênico
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
O protocolo apresenta dois métodos para determinar a cinética dos aptâmeros de RNA fluorogênico Espinafre2 e Brócolis. O primeiro método descreve como medir a cinética do aptâmero fluorogênico in vitro com um leitor de placas, enquanto o segundo método detalha a medição da cinética do aptâmero fluorogênico nas células por citometria de fluxo.
Resumo
Aptâmeros de RNA fluorogênicos têm sido aplicados em células vivas para marcar e visualizar RNAs, relatar a expressão gênica e ativar biossensores fluorescentes que detectam níveis de metabólitos e moléculas de sinalização. Para estudar as mudanças dinâmicas em cada um desses sistemas, é desejável obter medições em tempo real, mas a precisão das medições depende da cinética da reação fluorogênica ser mais rápida do que a frequência de amostragem. Aqui, descrevemos métodos para determinar a cinética de ativação in vitro e celular para aptâmeros de RNA fluorogênicos usando um leitor de placas equipado com um injetor de amostra e um citômetro de fluxo, respectivamente. Mostramos que a cinética in vitro para a ativação por fluorescência dos aptâmeros Espinafre2 e Brócolis pode ser modelada como reações de associação bifásica e tem diferentes constantes de taxa de fase rápida de 0,56 s−1 e 0,35 s−1, respectivamente. Além disso, mostramos que a cinética celular para a ativação por fluorescência do Espinafre2 em Escherichia coli, que é ainda mais limitada pela difusão do corante nas bactérias Gram-negativas, ainda é suficientemente rápida para permitir uma frequência de amostragem precisa na escala de tempo minuto. Esses métodos para analisar a cinética de ativação por fluorescência são aplicáveis a outros aptâmeros de RNA fluorogênicos que foram desenvolvidos.
Introdução
Reações fluorogênicas são reações químicas que geram um sinal de fluorescência. Os aptâmeros de RNA fluorogênicos normalmente desempenham essa função ligando um corante de molécula pequena para aumentar seu rendimento quântico de fluorescência (Figura 1A)1. Diferentes sistemas de aptâmeros de RNA fluorogênicos foram desenvolvidos e consistem em sequências específicas de aptâmeros de RNA e os ligantes corantes correspondentes1. Aptâmeros de RNA fluorogênicos têm sido anexados a transcritos de RNA como etiquetas fluorescentes que permitem a imagem de células vivas de mRNAs e RNAs não codificantes 2,3,4. Eles também foram colocados após sequências promotoras como repórteres fluorescentes de expressão gênica, semelhante ao uso de proteína fluorescente verde (GFP) como repórter, exceto que a função de relato está no nível de RNA 5,6. Finalmente, os aptâmeros de RNA fluorogênicos foram incorporados em biossensores fluorescentes baseados em RNA, que são projetados para desencadear a reação fluorogênica em resposta a uma pequena molécula específica. Biossensores fluorescentes baseados em RNA foram desenvolvidos para imagens de células vivas de vários metabólitos não fluorescentes e moléculas sinalizadoras 7,8,9,10,11.
Há um interesse crescente no desenvolvimento de aptâmeros de RNA fluorogênicos para visualizar mudanças dinâmicas na localização do RNA, expressão gênica e sinais de moléculas pequenas. Para cada uma dessas aplicações, é desejável obter medições em tempo real, mas a precisão das medições depende da cinética da reação fluorogênica ser mais rápida do que a frequência de amostragem. Aqui, descrevemos métodos para determinar a cinética in vitro para os aptâmeros de RNA fluorogênico Espinafre2 12 e Brócolis13 usando um leitor de placas equipado com um injetor de amostra e para determinar a cinética de ativação celular para Espinafre2 expressa em Escherichia coli usando um citômetro de fluxo. Esses dois aptâmeros de RNA foram escolhidos por terem sido aplicados para estudar a localização do RNA 2,3,4, terem sido utilizados em repórteres 5,6 e biossensores 7,8,9,10,11, e os ligantes corantes correspondentes (DFHBI ou DFHBI-1T) estarem comercialmente disponíveis. Um resumo de suas propriedades in vitro determinadas na literatura é apresentado na Tabela 1 4,13,14, que informou o desenvolvimento do protocolo (por exemplo, os comprimentos de onda e as concentrações de corantes utilizados). Esses resultados demonstram que as reações fluorogênicas afetadas pelos aptâmeros de RNA são rápidas e não devem impedir medições precisas para as aplicações biológicas celulares desejadas.
Protocolo
1. Experimento de cinética in vitro
- Preparação de moldes de ADN por PCR
- Configurar reação(ões) de PCR: Para preparar reações de PCR, combine os seguintes reagentes em um tubo de PCR de paredes finas:
33 μL de água duplamente destilada (ddH2O)
10 μL de tampão 5x para DNA polimerase de alta fidelidade
5 μL de 2 mM de cada trifosfato desoxirribonucleosídeo (dNTP)
0,5 μL de 40 μM de primer para a frente
0,5 μL de primer reverso de 40 μM
0,5 μL (10-100 ng) de molde de DNA (apenas para Spinach2 PCR; Sobreposição de primers de brócolis)
0,5 μL de DNA polimerase de alta fidelidade (adicionar por último)
NOTA: Os oligonucleotídeos sintéticos são frequentemente enviados secos. Para preparar soluções-estoque, adicione um volume conhecido (recomenda-se 100 μL) de ddH2O e meça o A260 dessa solução para determinar a concentração pela lei de Beer, onde o coeficiente de extinção pode ser calculado pelas regras do vizinho mais próximo on-line. Esta solução-mãe pode então ser utilizada para efectuar diluições adequadas para utilização em PCR. - Execute o protocolo do termociclador.
- Use o seguinte protocolo termociclador para amplificar modelos completos de DNA de espinafre2 e brócolis:
Desnaturação inicial: 98 °C por 2 min
35 ciclos:
Desnaturação: 98 °C desnaturação para 15 s
Recozimento: 72 °C por 30 s
Extensão: 72 °C para 30 s
Extensão final: 72 °C por 5 min. - Após a reação, analise uma pequena alíquota do produto em gel de agarose a 2%, juntamente com uma escada de baixo peso molecular (faixa de tamanho: 25-766 nucleotídeos) para confirmar a presença do produto de DNA desejado.
- Purificar o produto através de kits de extracção em gel ou de limpeza por PCR comercialmente disponíveis e eluítar com ddH2O ou com o tampão fornecido pelo fabricante.
NOTA: Certifique-se de que, ao escolher um kit de limpeza de PCR, o ponto de corte do peso molecular da coluna seja baixo o suficiente para reter T7-Brócolis (81 nucleotídeos), ou então o produto de PCR será perdido.
NOTA: Ponto de pausa opcional: armazene o DNA a -20 °C.
- Use o seguinte protocolo termociclador para amplificar modelos completos de DNA de espinafre2 e brócolis:
- Configurar reação(ões) de PCR: Para preparar reações de PCR, combine os seguintes reagentes em um tubo de PCR de paredes finas:
- Preparação de RNA de espinafre2 e brócolis por transcrição in vitro (IVT)
- Configure a(s) reação(ões) de transcrição.
- Para preparar uma reação de transcrição de 100 μL, combine os seguintes reagentes em um tubo de microcentrífuga de 1,5 mL: 10 μL de tampão de transcrição 10x + 20 μL de trifosfatos ribonucleosídeos de 10 mM (rNTPs) + 1-64 μL de molde de DNA (total de 1 μg) + 2 μL de pirofosfatase inorgânica + ddH 2 O a 98 μL +2μL de RNA polimerase T7 (adicionar por último).
- Incubar esta reacção durante 4 h a 37 °C. Apague a reação adicionando 100 μL de 2x tampão de carga de gel de ureia (2x ULB), composto de 20% de sacarose, 0,1% de dodecil sulfato de sódio (SDS), 1x tampão Tris-Borato-EDTA (1x TBE) e ~18 M de ureia.
NOTA: Ponto de pausa opcional: armazene a reação extinta a -20 °C.
- Configure a(s) reação(ões) de transcrição.
- Eletroforese em gel de poliacrilamida (PAGE) purificação de RNA
- PAGE purificação de Espinafre2 e RNA de Brócolis
CUIDADO: A acrilamida não polimerizada (líquida ou em pó) é extremamente tóxica. Se pesar a acrilamida em pó, faça-o em um exaustor. Use sempre o equipamento de proteção adequado e remova imediatamente as luvas contaminadas com acrilamida em pó ou solução, lavando bem as mãos. Se a acrilamida entrar em contato direto com a pele, lave a área exposta por pelo menos 15 minutos com água e sabão. Se a acrilamida entrar em contato direto com os olhos, lave-os com água por 15 minutos.- Prepare o gel PAGE: Para remover transcritos abortivos indesejados e rNTPs não reagidos do produto de comprimento total, prepare um gel de ureia-poliacrilamida a 6%. Em geral, géis de 28 cm x 16,5 cm x 1,5 mm podem ser usados com um pente de 8 poços. Configure o equipamento de gel e eletroforese, usando 1x tampão TBE para encher os reservatórios.
- Carregar amostra(s) de RNA no gel PAGE: Carregue o gel com uma reação de 200 μL extinta por pista. Em uma pista separada, carregue 2x ULB com corantes rastreadores xileno cianol e azul de bromofenol, que migram no gel em 106 nucleotídeos e 26 nucleotídeos, respectivamente15. Deixe uma faixa vazia entre cada amostra para evitar possíveis contaminações nas próximas etapas.
- Execute o gel PAGE: Para separar o espinafre 95-nt 2 e o brócolis 49-nt de seus respectivos produtos truncados, execute o gel por 1,5-2 h a 25 W, momento em que o corante azul de bromofenol terá migrado ~ 5/6 do comprimento do gel.
- Visualize a(s) amostra(s) de RNA no gel PAGE: Desmonte as placas de vidro ao redor do gel e cubra o gel em filme plástico em ambos os lados, rotulando as faixas no invólucro. Visualize bandas de RNA em uma sala escura por sombreamento UV, colocando o gel embrulhado em uma placa TLC fluorescente sob luz UV. Contorne rapidamente a borda das bandas de RNA correspondentes ao produto com um marcador e desligue a lâmpada UV para minimizar os danos causados pela exposição aos raios UV.
- Extraia a(s) amostra(s) de RNA do gel PAGE: Com uma lâmina de barbear fresca para cada amostra, expiregue as bandas de produto desejadas, cubos de ~1 mm e adicione os pedaços de gel a um tubo de microcentrífuga de 2 mL com 500 μL de tampão de imersão de esmagamento para extrair RNA em um rotador por 2 h à temperatura ambiente (RT) ou durante a noite a 4 °C.
NOTA: Ponto de pausa opcional: a amostra pode ser armazenada a -20 °C. - Precipitar o RNA
- Para separar os pedaços de gel do RNA extraído em tampão, centrifugar a amostra a 13.000 x g por 20 min a 4 °C e, em seguida, usar uma pipeta de ponta estreita para extrair o sobrenadante e carregá-lo em um novo tubo de microcentrífuga de 2 mL.
- Para precipitar o RNA, adicione 1,5 mL de etanol gelado e 1 μL de 20 mg/mL de glicogênio, vórtice, e armazene por pelo menos 1 h a -20 °C ou -80 °C.
NOTA: Ponto de pausa opcional: o RNA pode ser armazenado a -20 °C por alguns meses.
- Coleta de precipitado de RNA: Pellet o RNA precipitado por centrifugação a 13.000 x g por 20 min a 4 °C. Remova o sobrenadante e deixe o etanol restante evaporar ao ar livre (~1 h) antes de ressuspender o pellet em 30 μL de ddH2O ou 1x TE buffer.
NOTA: Este processo normalmente resulta em uma concentração final de RNA de ~10 μM.
NOTA: Ponto de pausa opcional: a amostra de ARN pode ser armazenada a -20 °C durante alguns meses.
- Determine as concentrações de estoque de RNA.
- Prepare uma alíquota de RNA para a reação de hidrólise.
- Para realizar este ensaio, primeiro, use um nano espectrofotômetro UV/Vis para determinar o A260 da amostra de RNA estoque e faça uma alíquota diluída da amostra para ~10 unidades de absorbância (UA) em ddH2O.
- Preparar a seguinte reação em um tubo de PCR de 0,5 mL: 16 μL de ddH 2 O + 2 μL de 10x Na 2 CO3 tampão+2μL de alíquota de RNA diluída a ~10 UA. Incubar a reacção durante 90 min a 95 °C e, em seguida, deixar arrefecer até ao RT.
- Determinar a concentração de ARN utilizando absorvâncias de nucleótidos: Medir o A260 desta amostra com um nanoespectrofotómetro UV/Vis e calcular a concentração de ARN utilizando a seguinte fórmula:

onde c é a concentração de RNA, b é o comprimento do caminho, i é o nucleotídeo específico (A, C, G ou U), n i é a frequência do nucleotídeo i na sequência de RNA e ε i é o coeficiente de extinção molar do nucleotídeo i. Para determinar a concentração de ARN de reserva original, multiplicar c pelo factor de diluição.
NOTA: Ponto de pausa opcional: O RNA pode ser armazenado a -20 °C por alguns meses.
- Prepare uma alíquota de RNA para a reação de hidrólise.
- PAGE purificação de Espinafre2 e RNA de Brócolis
- Executando um in vitro ensaio de cinética do leitor de placas
- Configure o programa de renaturação de RNA: Crie o seguinte protocolo de termociclador selecionando Criar Novo Programa > Adicionar Nova Fase > Adicionar Nova Etapa várias vezes para adicionar cada uma das seguintes etapas antes de pressionar Salvar:
70 °C durante 3 min
65 °C por 45 s
60 °C por 45 s
55 °C por 45 s
50 °C por 45 s
45 °C por 45 s
40 °C por 45 s
35 °C por 45 s
30 °C por 45 s - Renaturalizar o RNA: Para renaturalizar os RNAs de espinafre2 e brócolis, prepare 2 μM de cada RNA em ddH 2 O emum tubo de PCR de parede fina de 0,5 mL e, em seguida, adicione um volume igual de 2x tampão de renaturação (80 mM HEPES, pH 7,5 [KOH], 250 mM KCl, 6 mM MgCl2) para fazer soluções de RNA de 1 μM. Adicione os tubos ao termociclador, abra o programa de renaturação salvo e pressione Executar.
NOTA: Se um termociclador não estiver disponível, os ARN podem ser incubados num bloco de calor de 70 °C durante 3 minutos e, em seguida, deixados arrefecer lentamente para RT na bancada. - Preparar o tampão de reação de ligação: Para preparar o tampão para uma reação de ligação aptâmero-corante, faça uma mistura mestra contendo componentes tampão (69,5 μL de ddH2 O + 4 μL de 1 M HEPES, pH 7,5 [KOH] + 6,2 μL de 2 M KCl + 0,3 μL de 1 M MgCl2). Dependendo de quantas amostras e replicações são necessárias, multiplique esses valores pelo número de amostras mais uma. Geralmente, três repetições por amostra de RNA são satisfatórias.
- Prepare o leitor de placas.
- Configure o programa injetor do leitor de placas: No leitor de placas de fluorescência, selecione Temp e defina a temperatura desejada para 37 °C, certificando-se de que a temperatura tenha se equilibrado para esse valor bem antes de iniciar experimentos de cinética. Abra o software leitor de placas, selecione Configurações > Visualização de aquisição e insira o seguinte programa para medições cinéticas:
Loop: Para cada poço
Configuração da linha de base: 60 leituras da linha de base
Configurações do SmartInject: injeção de 10 μL (o que acontecerá após a leitura da linha de base)
A fluorescência (ou FL) diz:
Excitação: 448 nm (largura de banda: 9 nm)
Emissão: 506 nm (largura de banda: 15 nm)
Cartucho: MONO (s/n 3297)
Timing:
Tempo total de leitura: 10 min
Intervalo de leitura: 0,5 s
PMT e Óptica: 6 flashes por leitura
Loop: Próximo poço - Prepare o injetor
- Lave e aspirar o injetor: Para preparar o injetor leitor de placas, primeiro selecione Injetar no leitor de placas, forneça uma placa de coleta de resíduos ao leitor de placas quando solicitado e, em seguida, selecione Lavar e limpe o tubo de injeção seguindo as instruções no leitor de placas com 1 mL de volumes de ddH 2 O, etanol a 75% em ddH2O, e depois ddH2O. Em seguida, selecione Aspirar, permitindo que o injetor ejete o excesso de líquido.
- Prepare o injetor: Depois de sair para a tela anterior, selecione Prime para preparar o injetor com dois volumes de 260 μL de ligante a serem injetados para garantir que o ligante puro e concentrado seja adicionado às amostras durante os experimentos - neste caso, primo com 100 μM DFHBI em ddH2O.
- Configure o programa injetor do leitor de placas: No leitor de placas de fluorescência, selecione Temp e defina a temperatura desejada para 37 °C, certificando-se de que a temperatura tenha se equilibrado para esse valor bem antes de iniciar experimentos de cinética. Abra o software leitor de placas, selecione Configurações > Visualização de aquisição e insira o seguinte programa para medições cinéticas:
- Realizar experimentos de cinética in vitro : Para realizar um experimento de cinética, primeiro adicione 80 μL de mistura mestre de tampão de ligação previamente preparada a um poço de uma placa de fundo claro de 96 poços, seguida por 10 μL de RNA renaturado. Deixar que esta placa e a solução de DFHBI no injector se equilibrem a 37 °C durante 15 minutos no leitor de placas.
- Nas Configurações do software leitor de placas em Área de leitura, selecione o poço a ser analisado e, na guia Página Inicial , selecione Executar para executar o programa de cinética descrito anteriormente. Repita esse processo até que todos os experimentos estejam completos.
NOTA: Criticamente, os experimentos cinéticos devem ser realizados um poço de cada vez para garantir que a cinética do RNA seja medida em condições idênticas entre replicações e amostras. - Lavar o injetor: Para remover a solução DFHBI restante do tubo de injeção, lave o tubo de injeção conforme descrito na etapa 1.4.4.2.1 com 1 mL de volumes de ddH 2 O, etanol a 75% em ddH 2 Oe, em seguida, ddH2O.
NOTA: Ponto de pausa opcional.
- Configure o programa de renaturação de RNA: Crie o seguinte protocolo de termociclador selecionando Criar Novo Programa > Adicionar Nova Fase > Adicionar Nova Etapa várias vezes para adicionar cada uma das seguintes etapas antes de pressionar Salvar:
- Análise da cinética in vitro de aptâmeros de RNA fluorogênico.
- Insira dados no software de análise: exporte dados experimentais como uma planilha para copiar facilmente os dados para processamento. No software de análise, crie uma nova tabela de dados no formato XY. Na coluna X, insira cada ponto de tempo de leitura, com t = 0 sendo o tempo de injeção de DFHBI. Na coluna Y, insira os valores médios de fluorescência entre as replicações nesse respectivo ponto de tempo a partir de t = 0.
- Normalizar e representar graficamente os dados: Para normalizar os valores de fluorescência, clique em Analisar > Processamento de Dados > Normalizar e, em seguida, clique em OK. Escolha usar os menores e maiores valores do conjunto de dados para normalização, para apresentar os resultados como frações e para representar graficamente os resultados e, em seguida, clique em OK. Para criar um gráfico da fluorescência média normalizada ao longo do tempo, clique no ícone Normalizar de [Nome do Conjunto de Dados] e selecione Família de Gráficos: XY com Somente Pontos.
NOTA: As barras de erro podem ser úteis para ver nos casos em que um pequeno número de pontos de tempo são representados graficamente. Se desejar, ao escolher uma tabela de dados no formato XY, selecione a opção em "Y" para inserir valores de replicação em colunas lado a lado. Representar graficamente a tabela resultante com as opções padrão selecionadas produzirá um gráfico com barras de erro. - Realizar ajuste de curva para obter parâmetros cinéticos: Para ajustar uma curva aos dados cinéticos, clique em Analisar > Analisar Dados e selecione Regressão Não Linear (Ajuste de Curva) na guia Análises XY . Na guia Modelo , clique em Associação Exponencial > Duas Fases para ajustar os dados cinéticos com a seguinte equação de associação de duas fases:
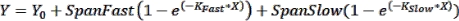
onde Y é a fluorescência no tempo X, Y 0 é a fluorescência em t = 0, K Fast e K Slow são constantes de taxa rápida e lenta, respectivamente, e SpanFast e SpanSlow são as faixas de ativação de fluorescência contabilizadas pelas taxas rápidas e lentas, respectivamente (ver resultados representativos, Figura 1). Clique na guia Nonlin Fit para exibir constantes de taxa, valores t1/2 e valores PercentFast.
NOTA: Para obter um desvio padrão para todos esses valores, replicações individuais do experimento do leitor de placas podem ser processadas da mesma maneira descrita acima.
2. Experimento de cinética celular
- Preparação de estirpes de E. coli
- Transforme células de E. coli BL21 Star (DE3) com ~100 ng de pET31b tRNA-Espinafre2 construindo seguindo o protocolo do fabricante.
NOTA: A construção do plasmídeo está comercialmente disponível (Plasmídeo #79783). - Chapear as células em placas de ágar LB contendo carbenicilina (Carb: 50 mg/mL) e incubar a 37 °C durante 12-16 h. As células que contêm plasmídeos crescerão como colônias na placa.
NOTA: Ponto de pausa opcional: As células BL21 Star transformadas em placas podem ser armazenadas a 4 °C envoltas em parafilme durante 1 semana.
- Transforme células de E. coli BL21 Star (DE3) com ~100 ng de pET31b tRNA-Espinafre2 construindo seguindo o protocolo do fabricante.
- Células em crescimento e indução da expressão do aptâmero de RNA fluorogênico
- Inocular uma cultura de 2 mL de meio não indutor (NI) contendo carbenicilina (Carb: 50 mg/mL) com uma única colônia das células BL21 Star transformadas. Repita isso por pelo menos três replicações biológicas. Incubar as culturas a 37 °C numa incubadora/agitadora a 250 rpm durante 22-24 h.
NOTA: Ponto de pausa opcional: As células cultivadas em meios de NI retêm o plasmídeo e podem ser armazenadas a 4 °C durante 1 semana. - Após o crescimento em meios de NI, diluir a cultura 100x em um novo 3 mL de meio de autoindução (IA) ZYP-5052 contendo carbenicilina (Carb: 50 mg/mL). Cultivar as células a 37 °C numa incubadora/agitadora a 250 rpm durante 16-18 h para induzir a expressão.
NOTA: O OD600 típico para culturas variará de 2,0-3,3 após 18 h de crescimento. A faixa de densidade celular ideal está entre 2,5-3,0.
- Inocular uma cultura de 2 mL de meio não indutor (NI) contendo carbenicilina (Carb: 50 mg/mL) com uma única colônia das células BL21 Star transformadas. Repita isso por pelo menos três replicações biológicas. Incubar as culturas a 37 °C numa incubadora/agitadora a 250 rpm durante 22-24 h.
- Realização do experimento de cinética celular
- Configure o citômetro de fluxo.
- Ligue o citômetro de fluxo e o computador conectado ao instrumento. Uma vez logado no software para o citômetro de fluxo, na guia Instrumento , clique no ícone Inicialização . Siga as etapas indicadas na tela do software para garantir a inicialização adequada da instrumentação.
NOTA: Alguns citômetros de fluxo chamam a sequência de inicialização do instrumento de Priming. Certifique-se de seguir o protocolo do fabricante para o citômetro de fluxo que estará em uso para o experimento. - Execute um teste de desempenho (se aplicável). Na guia Menu Principal , clique em Teste de Desempenho. Em um tubo de cultura, adicione três gotas das esferas de rastreamento de desempenho do fabricante em 3 mL de fluido de foco.
- Coloque o tubo de cultura no elevador do tubo de amostra e levante o elevador. Antes de clicar em Executar Teste de Desempenho, verifique se o Lote # do tubo dos grânulos de rastreamento é o mesmo indicado na tela Configuração do Teste de Desempenho . Clique em Teste de Desempenho para executar o teste.
- Configure o software do citômetro de fluxo para este experimento com os seguintes parâmetros de aquisição para fluorescência unicelular:
Laser de excitação: 488 nm
Canal de emissão: GFP (também chamado de FITC)
Volume de aquisição: 40 μL (com um volume total de desenho de 90 μL)
Taxa de fluxo: 200 μL / min
Contagens de células para cada medição: 30.000
Configurações do instrumento:
Voltagem:
FSC: 480 V
CCS: 400 V
BL1: 540 V
BL2: 392 V
BL3: 422 V
- Ligue o citômetro de fluxo e o computador conectado ao instrumento. Uma vez logado no software para o citômetro de fluxo, na guia Instrumento , clique no ícone Inicialização . Siga as etapas indicadas na tela do software para garantir a inicialização adequada da instrumentação.
- Configure os arquivos experimentais.
- Crie um novo arquivo de experimento na guia Explorador de Experimentos clicando com o botão direito do mouse no nome de usuário do citômetro de fluxo. Selecione Novo experimento na janela suspensa. Quando uma nova janela na tela do computador aparecer, selecione OK.
- No novo arquivo de experimento, clique com o botão direito do mouse na pasta "Grupo" e selecione Adicionar Novo Tubo de Amostra. Rotule os tubos de amostra para cada ponto de tempo específico e replique clicando com o botão direito do mouse em Amostra e selecionando Renomear no menu suspenso. Repita esta etapa para o número total de replicações e pontos de tempo para o curso de tempo pretendido do estudo.
- Preparar uma solução de células diluídas: Em um novo tubo de cultura, adicione 1,5 mL de 1x solução de PBS. Em seguida, adicione 3 μL de células induzidas em meios de IA na solução PBS 1x para fazer uma solução de célula diluída. Repita esta etapa para cada replicação biológica em diferentes tubos de cultura.
- Meça a fluorescência de fundo das células: Antes de adicionar corante, faça leituras de cada tubo de cultura biológico replicado contendo células em 1x solução de PBS. Isso é para que o fundo fluorescente das células seja medido para observar a dobra se ligar ao longo do tempo, uma vez que o corante é adicionado.
- Para fazer uma leitura, coloque o tubo de cultura no elevador do tubo de amostra e levante o elevador manualmente até a agulha de injeção de amostra. Selecione o arquivo de exemplo adequado na guia Painel de Coleta e clique em Gravar.
- Quando a corrida estiver concluída, abaixe o elevador do tubo de amostra com o tubo de cultura à mão. Isso iniciará uma etapa de enxágue que liberará o sistema fluídico e minimizará o transporte entre cada amostra replicada biológica. Os dados serão salvos automaticamente no computador após a conclusão da execução.
- Repetir os passos referidos no ponto 2.3.4 para medir o fundo fluorescente celular para pelo menos três repetições biológicas. Para ir para o próximo arquivo de exemplo, selecione o próximo arquivo de exemplo clicando no ícone de seta para a direita próximo ao nome do tubo de exemplo abaixo do ícone Gravar .
- Meça a fluorescência em pontos de tempo para células com corante.
- Adicionar 1,4 μL de um corante concentrado (50 mM DFHBI-1T em DMSO) em 1x solução de PBS com células para obter uma concentração final de 50 μM DFBHI-1T. Em seguida, prenda a tampa do tubo de cultura e, em seguida, inverta os tubos de cultura 3x-5x para misturar a solução uniformemente antes de fazer a primeira leitura do ponto de tempo.
NOTA: A porcentagem total de DMSO dentro dos tubos de cultura para E. coli não deve exceder 10%, pois isso pode afetar a viabilidade celular16. - Retire a tampa e coloque o tubo de cultura no elevador do tubo de amostra. Levante o suporte manualmente até a agulha de injeção de amostra e, sob o arquivo de amostra adequado, clique no ícone Gravar . Além disso, inicie um temporizador pressionando Iniciar para o experimento.
- Abaixe o elevador de tubos manualmente após a conclusão da corrida e repita as etapas 2.3.5.1-2.3.5.2. (com o temporizador ligado) adicionando 1,4 μL do DFHBI-1T concentrado, invertendo os tubos de cultura e fazendo leituras para todas as restantes replicações biológicas. Esses primeiros registros serão as leituras obtidas em 0 min para todas as replicações biológicas. Faça isso um de cada vez para cada replicação biológica.
NOTA: Anote o momento em que a aquisição da citometria de fluxo de registro é pressionada. Siga esse tempo escalonando para pontos de tempo ao longo do experimento. - Continue a fazer leituras elevando os tubos de cultura no elevador do tubo de amostra até a agulha de injeção de amostra, selecionando o arquivo de amostra adequado, clicando em Gravar e abaixando o elevador manualmente após a conclusão de cada execução. Faça isso para todos os pontos de tempo adicionais e replicações biológicas que estão sendo testadas. Repita as etapas até que o experimento seja concluído.
NOTA: Mantenha as amostras fora de luz para evitar o fotobranqueamento de DFHBI-1T em solução, cobrindo as amostras com papel alumínio.
- Adicionar 1,4 μL de um corante concentrado (50 mM DFHBI-1T em DMSO) em 1x solução de PBS com células para obter uma concentração final de 50 μM DFBHI-1T. Em seguida, prenda a tampa do tubo de cultura e, em seguida, inverta os tubos de cultura 3x-5x para misturar a solução uniformemente antes de fazer a primeira leitura do ponto de tempo.
- Meça a fluorescência em pontos de tempo para células sem corante (Controle).
- Execute os protocolos de limpeza apropriados para o citômetro de fluxo antes de repetir o experimento novamente com controles negativos seguindo o protocolo do fabricante. Isso é feito para minimizar qualquer transferência do experimento anterior para o experimento de análise do ponto de tempo de controle. Abaixo estão os passos seguidos para o citômetro de fluxo entre os experimentos:
- Coloque um tubo de cultura vazio no elevador do tubo à mão, levante o suporte do tubo e clique no ícone Desobstruir na guia Instrumento . Isso executará um backflush no sistema fluídico para limpar todas as amostras pegajosas. Abaixe o suporte do tubo à mão e remova o tubo assim que a sequência de desentupimento estiver concluída.
- Com um novo tubo de cultura, adicione 3 mL de uma solução de água sanitária a 10%, coloque o tubo de cultura no suporte do tubo e levante o suporte à mão até a agulha de injeção da amostra. Além disso, coloque uma placa limpa de 96 poços no amostrador automático, se aplicável ao citômetro de fluxo.
- Clique no ícone Sanitize SIP /Sanitize Autosampler SIP para executar uma sequência de limpeza com 10% de água sanitária em todo o sistema fluídico. Abaixe o suporte do tubo para completar a sequência de limpeza.
- Defina os arquivos de exemplo para a análise de ponto de tempo de controle executar seguindo as instruções dentro da etapa 2.3.3.
- Em um novo tubo de cultura, preparar uma solução de célula diluída em 1,5 mL de 1x solução de PBS. Adicione 3 μL das células induzidas em meios de IA na solução PBS 1x para fazer uma solução de célula diluída. Repita esta etapa para cada replicação biológica.
- Adicionar 1,4 μL de DMSO no tubo de cultura, um de cada vez, à solução de PBS 1x com células e testar os mesmos pontos de tempo. Prenda a tampa do tubo de cultura e, em seguida, inverta os tubos de cultura 3x-5x para misturar a solução uniformemente antes de fazer a primeira leitura pontual. Faça isso um de cada vez para cada replicação biológica.
- Siga o mesmo protocolo para a análise de células de controle como para células com corante, usando as etapas 2.3.5.2-2.3.5.4.
- Execute os protocolos de limpeza apropriados para o citômetro de fluxo antes de repetir o experimento novamente com controles negativos seguindo o protocolo do fabricante. Isso é feito para minimizar qualquer transferência do experimento anterior para o experimento de análise do ponto de tempo de controle. Abaixo estão os passos seguidos para o citômetro de fluxo entre os experimentos:
- Desligue o citômetro de fluxo: Siga o protocolo do fabricante para o desligamento adequado da instrumentação. Para o citômetro de fluxo, o instrumento é preparado para o desligamento da seguinte maneira:
- Execute o protocolo de limpeza inicial para o citômetro de fluxo seguindo as etapas 2.3.6.1.1-2.3.6.1.3.
- Substitua o tubo de cultura por solução de água sanitária a 10% por um tubo de cultura com 3 mL de fluido de foco. Levante o suporte do tubo à mão e, sob o ícone Desligar , clique no menu suspenso e selecione Completo.
NOTA: Ponto de pausa opcional.
- Configure o citômetro de fluxo.
- Análise dos dados da citometria de fluxo
- Exporte todos os arquivos FCS para análise. Abra os arquivos FCS com um software de análise de citometria de fluxo.
- Usando um dos arquivos FCS somente de célula, gere uma porta a partir do gráfico de pontos de dispersão frontal (FSC) e dispersão lateral (SSC) (FSC-Area/SSC-Area), usando ambos os eixos de log para excluir quaisquer sinais de detritos. Para criar esse portão, clique no ícone AutoGate no software de análise de citometria de fluxo e nomeie-o Portão 1. Aplique essa mesma porta a todos os exemplos testados na guia Todos os Exemplos no espaço de trabalho de processamento de dados. Isso resultará no Portão 1 abaixo de todos os arquivos FCS sendo processados.
- Crie um novo arquivo de subconjunto com o arquivo FCS somente de célula usado na etapa 2.4.2 clicando duas vezes nele. Altere as configurações do eixo para FSC-Area/FSC-Height, com ambos usando eixos de log. Clique no ícone AutoGate no software de análise de citometria de fluxo para gerar um portão oval, nomeando-o Portão 2. Aplicar esta porta como um subconjunto por baixo da porta definida no passo 2.4.2 a todas as amostras testadas. Isso resultará em todas as amostras com o Portão 1 > o Portão 2 associados a cada arquivo FCS.
- Crie outro arquivo de subconjunto com ambas as portas definidas na etapa 2.4.2 e na etapa 2.4.3 aplicadas clicando duas vezes na Porta 2. Altere as configurações do eixo para FSC-Area/Histogram. Aplique esta porta de histograma como um subconjunto a todas as amostras testadas, resultando em todas as amostras com a Porta 1 > a Porta 2 > a Porta 2 associadas a cada arquivo FCS.
NOTA: Os histogramas podem ser renomeados de Porta 2 para Histograma para ajudar a mover os histogramas para a janela de layout, bem como para criar mais organização com o processamento de dados. - Para analisar os valores médios de intensidade de fluorescência (IFM), abra a janela de layout. Clique e arraste as portas do histograma para cada ponto de tempo para a janela de layout.
- Execute uma análise estatística para "∑ Média: BL1-A" (GFP) para cada amostra testada para exibir os resultados da IFM na janela de layout.
- Calcular o desvio-padrão para os valores de IFM por análise de ponto de tempo para pelo menos três repetições biológicas independentes.
- Salve os histogramas e os valores de IFM para cada ponto de tempo exportando a janela de layout como um arquivo PDF.
NOTA: Ponto de pausa opcional.
- Dados de citometria de fluxo gráfica
- Abra o arquivo PDF que contém os histogramas e os valores de IFM para cada ponto de tempo. Os valores das IFM serão copiados para um software de análise de dados. No software de gráficos de dados, crie uma nova tabela de dados no formato XY.
- Selecione para criar uma Tabela XY com o seguinte selecionado:
Tabela de dados: insira ou importe dados para uma nova tabela
Opções:
X: Tempos decorridos
Y: Insira (três a quatro) valores de replicação em subcolunas lado a lado - Rotule no eixo X todos os pontos de tempo para as execuções do experimento e do controle.
- No Grupo A, insira os valores de IFM para todas as replicações biológicas em cada ponto de tempo para análise de fluorescência das células com o corante adicionado.
- No Grupo B, insira os valores de IFM para todas as replicações biológicas em cada ponto de tempo para análise das células sem adição de corante (DMSO).
- Para observar os resultados, clique em [Inserir Nome do Conjunto de Dados] na guia Gráficos . Isso exibirá os pontos de dados como médias, com barras de erro representando o desvio padrão (s.d.) em cada ponto de tempo. O eixo X representa o tempo decorrido e o eixo Y representa os valores de IFM.
- Selecione para criar uma Tabela XY com o seguinte selecionado:
- Abra o arquivo PDF que contém os histogramas e os valores de IFM para cada ponto de tempo. Os valores das IFM serão copiados para um software de análise de dados. No software de gráficos de dados, crie uma nova tabela de dados no formato XY.
Resultados
Cinética in vitro
As sequências dos moldes e primers de DNA, que são adquiridos como oligonucleotídeos sintéticos, são mostradas na Tabela 2, e as receitas de reagentes são mostradas no Arquivo Suplementar 1. A amplificação por PCR é usada para aumentar a quantidade de molde de DNA com o promotor T7, que é necessário para a reação subsequente de transcrição in vitro (IVT). Além disso, a amplificação por PCR pode s...
Discussão
Para o experimento de cinética in vitro , o mesmo protocolo geral pode ser modificado para medir a cinética in vitro de um biossensor fluorescente baseado em RNA contendo um domínio de ligação ao ligante e de ligação ao fluoróforo8. Neste caso, o RNA deve ser incubado com o fluoróforo antes das medições após a injeção do ligante, a fim de obter cinética de resposta do ligante. Se for observada alta variabilidade entre as replicações, pode-se solucionar problemas ...
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pelas seguintes subvenções à MCH: NSF-BSF 1815508 e NIH R01 GM124589. O MRM foi parcialmente apoiado pela bolsa de treinamento NIH T32 GM122740.
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
Referências
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295 (2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139 (2016).
- Paige, J. S., Thinh, N. -. D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194 (2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002 (2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados