Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Определение кинетики in vitro и клеточного включения фторогенных аптамеров РНК
В этой статье
Резюме
В протоколе представлены два метода определения кинетики фторогенных РНК аптамеров Spinach2 и Broccoli. Первый метод описывает, как измерить кинетику фторогенного аптамера in vitro с помощью считывателя пластин, в то время как второй метод детализирует измерение кинетики фторогенного аптамера в клетках с помощью проточной цитометрии.
Аннотация
Фторогенные аптамеры РНК были применены в живых клетках для маркировки и визуализации РНК, отчета о экспрессии генов и активации флуоресцентных биосенсоров, которые обнаруживают уровни метаболитов и сигнальных молекул. Для изучения динамических изменений в каждой из этих систем желательно получать измерения в режиме реального времени, но точность измерений зависит от кинетики фторогенной реакции, которая быстрее частоты выборки. Здесь мы описываем методы определения кинетики in vitro и клеточного включения фторогенных аптамеров РНК с использованием пластинчатого считывателя, оснащенного инжектором образца и проточным цитометром соответственно. Показано, что кинетика in vitro для флуоресцентной активации аптамеров Spinach2 и Broccoli может быть смоделирована как двухфазные ассоциативные реакции и иметь различные константы скорости быстрой фазы 0,56 с−1 и 0,35 с−1 соответственно. Кроме того, мы показываем, что клеточная кинетика для флуоресцентной активации Spinach2 в Escherichia coli, которая дополнительно ограничена диффузией красителя в грамотрицательные бактерии, все еще достаточно быстра, чтобы обеспечить точную частоту выборки на минутной шкале времени. Эти методы анализа кинетики активации флуоресценции применимы к другим фторогенным аптамерам РНК, которые были разработаны.
Введение
Флуорогенные реакции — это химические реакции, которые генерируют флуоресцентный сигнал. Фторогенные аптамеры РНК обычно выполняют эту функцию, связывая краситель с небольшой молекулой для повышения его квантового выхода флуоресценции (рисунок 1А)1. Разработаны различные фторогенные системы аптамеров РНК, состоящие из специфических последовательностей аптамеров РНК и соответствующих лигандов красителя1. Фторогенные аптамеры РНК были добавлены к транскриптам РНК в виде флуоресцентных меток, которые позволяют визуализировать живые клетки мРНК и некодирующих РНК 2,3,4. Они также были помещены после промоторных последовательностей в качестве флуоресцентных репортеров экспрессии генов, аналогично использованию зеленого флуоресцентного белка (GFP) в качестве репортера, за исключением того, что функция отчетности находится на уровне РНК 5,6. Наконец, фторогенные аптамеры РНК были включены в флуоресцентные биосенсоры на основе РНК, которые предназначены для запуска фторогенной реакции в ответ на конкретную небольшую молекулу. Флуоресцентные биосенсоры на основе РНК были разработаны для визуализации живых клеток различных нефлуоресцентных метаболитов и сигнальных молекул 7,8,9,10,11.
Растет интерес к разработке фторогенных РНК-аптамеров для визуализации динамических изменений локализации РНК, экспрессии генов и сигналов малых молекул. Для каждого из этих применений желательно получать измерения в режиме реального времени, но точность измерений зависит от того, что кинетика фторогенной реакции быстрее частоты отбора проб. Здесь мы описываем методы определения кинетики in vitro для фторогенных аптамеров РНК Spinach212 и Broccoli13 с использованием пластинчатого считывателя, оснащенного инъектором образца, и определения клеточной кинетики включения spinach2, экспрессируемой в Escherichia coli с использованием проточного цитометра. Эти два РНК-аптамера были выбраны потому, что они были применены для изучения локализации РНК 2,3,4, они были использованы в репортерах 5,6 и биосенсорах 7,8,9,10,11, а соответствующие лиганды красителя (DFHBI или DFHBI-1T) коммерчески доступны. Краткое изложение их свойств in vitro, определенных в литературе, приведено в таблице 1 4,13,14, которая послужила основой для разработки протокола (например, используемые длины волн и концентрации красителей). Эти результаты показывают, что фторогенные реакции, на которые влияют аптамеры РНК, являются быстрыми и не должны препятствовать точным измерениям для желаемых биологических применений клеток.
протокол
1. Эксперимент по кинетике in vitro
- Подготовка шаблонов ДНК методом ПЦР
- Настройка реакции (реакций) ПЦР: Чтобы подготовить реакции ПЦР, объедините следующие реагенты в тонкостенной ПЦР-трубке:
33 мкл двойной дистиллированной воды (ddH2O)
10 мкл 5-кратного буфера для высокоточной ДНК-полимеразы
5 мкл по 2 мМ каждый дезоксирибонуклеозидтрифосфат (dNTP)
0,5 мкл прямой грунтовки 40 мкМ
0,5 мкл обратной грунтовки 40 мкМ
0,5 мкл (10-100 нг) шаблона ДНК (только для ПЦР «Шпинат2»; Грунтовки для брокколи перекрываются)
0,5 мкл высокоточной ДНК-полимеразы (добавить последнюю)
ПРИМЕЧАНИЕ: Синтетические олигонуклеотиды часто поставляются сухими. Чтобы приготовить бульочные растворы, добавьте известный объем (рекомендуется 100 мкл) ddH2O и измерьте A260 этого раствора, чтобы определить концентрацию по закону Бира, где коэффициент вымирания может быть рассчитан по правилам ближайшего соседа онлайн. Этот исходный раствор затем может быть использован для получения разведений, подходящих для использования в ПЦР. - Запустите протокол термоциклера.
- Используйте следующий протокол термоциклера для амплификации полноразмерных шаблонов ДНК Spinach2 и Broccoli:
Начальная денатурация: 98 °C в течение 2 мин
35 циклов:
Денатурация: 98 °C денатурация в течение 15 с
Отжиг: 72 °C в течение 30 с
Расширение: 72 °C в течение 30 с
Окончательное расширение: 72 °C в течение 5 мин. - После реакции анализируют небольшую аликвоту продукта на 2% агарозный гель вместе с низкомолекулярной лестницей (диапазон размеров: 25-766 нуклеотидов) для подтверждения наличия желаемого продукта ДНК.
- Очистите продукт с помощью коммерчески доступных наборов для экстракции геля или ПЦР-очистки и элюируют с помощью ddH2O или буфера, предоставленного производителем.
ПРИМЕЧАНИЕ: При выборе комплекта для очистки ПЦР убедитесь, что отсечение молекулярной массы колонки достаточно низкое, чтобы сохранить T7-брокколи (81 нуклеотид), иначе продукт ПЦР будет потерян.
ПРИМЕЧАНИЕ: Необязательная точка паузы: храните ДНК при −20 °C.
- Используйте следующий протокол термоциклера для амплификации полноразмерных шаблонов ДНК Spinach2 и Broccoli:
- Настройка реакции (реакций) ПЦР: Чтобы подготовить реакции ПЦР, объедините следующие реагенты в тонкостенной ПЦР-трубке:
- Получение РНК шпината2 и брокколи методом транскрипции in vitro (IVT)
- Настройте реакцию (реакции) транскрипции.
- Чтобы получить реакцию транскрипции 100 мкл, соедините следующие реагенты в микроцентрифужной трубке объемом 1,5 мл: 10 мкл 10-кратного буфера транскрипции + 20 мкл рибонуклеозидтрифосфатов (rNTPs) + 1-64 мкл шаблона ДНК (всего 1 мкг) + 2 мкл неорганической пирофосфатазы + ddH2Oдо 98 мкл + 2 мкл РНК-полимеразы T7 (добавить последнюю).
- Инкубируют эту реакцию в течение 4 ч при 37 °C. Гасят реакцию путем добавления 100 мкл 2x буфера загрузки геля мочевины (2x ULB), состоящего из 20% сахарозы, 0,1% додецилсульфата натрия (SDS), 1x буфера Tris-Borate-EDTA (1x TBE) и ~18 M мочевины.
ПРИМЕЧАНИЕ: Необязательная точка паузы: храните закаленную реакцию при температуре −20 °C.
- Настройте реакцию (реакции) транскрипции.
- Полиакриламидный гель электрофорез (PAGE) очистка РНК
- PAGE очистка от РНК шпината2 и брокколи
ВНИМАНИЕ: Неполимеризованный (жидкий или порошкообразный) акриламид чрезвычайно токсичен. При взвешивании порошкообразного акриламида сделайте это в вытяжном шкафу. Всегда надевайте надлежащие защитные средства и немедленно снимайте перчатки, загрязненные акриламидным порошком или раствором, тщательно вымыв руки. Если акриламид вступает в непосредственный контакт с кожей, промойте открытый участок не менее 15 минут с мылом и водой. Если акриламид попадает в непосредственный контакт с глазами, промывайте их водой в течение 15 мин.- Подготовьте гель PAGE: Чтобы удалить нежелательные абортивные транскрипты и непрореагировавшие rNTP из полноразмерного продукта, приготовьте 6% мочевинно-полиакриламидный гель. В общем, гели 28 см х 16,5 см х 1,5 мм можно использовать с 8-луночным гребнем. Настройте оборудование для геля и электрофореза, используя 1x TBE буфер для заполнения резервуаров.
- Загрузка образца (образцов) РНК в гель PAGE: Загрузите гель одной закаленной реакцией 200 мкл на полосу. В отдельную полосу нагружают 2x ULB трекерными красителями ксилола цианола и бромфенола синего цвета, которые мигрируют в гель при 106 нуклеотидах и 26 нуклеотидах соответственно15. Оставьте пустую полосу между каждым образцом, чтобы избежать потенциального загрязнения на следующих этапах.
- Запустите гель PAGE: Чтобы отделить 95-nt Spinach2 и 49-nt Broccoli от их соответствующих усеченных продуктов, запустите гель в течение 1,5-2 ч при 25 Вт, после чего бромфенольный синий краситель будет мигрировать ~ 5/6 длины геля.
- Визуализируйте образец (образцы) РНК в геле PAGE: разберите стеклянные пластины вокруг геля и покройте гель полиэтиленовой пленкой с обеих сторон, маркируя полосы на упаковке. Визуализируйте полосы РНК в темной комнате путем ультрафиолетового затенения, помещая обернутый гель на флуоресцентную пластину TLC под ультрафиолетовым светом. Быстро очертите маркером края полос РНК, соответствующих продукту, и выключите УФ-лампу, чтобы свести к минимуму ущерб от воздействия ультрафиолета.
- Иссейте и извлеките образец (образцы) РНК из геля PAGE: Свежим лезвием бритвы для каждого образца иссейте желаемые полосы продукта, нарежьте кубиками ~ 1 мм и добавьте кусочки геля в микроцентрифужную трубку объемом 2 мл с 500 мкл буфера для измельчения для извлечения РНК на ротаторе в течение 2 ч при комнатной температуре (RT) или на ночь при 4 °C.
ПРИМЕЧАНИЕ: Необязательная точка паузы: образец может храниться при температуре −20°C. - Осаждение РНК
- Чтобы отделить кусочки геля от экстрагированной РНК в буфере, центрифугируют образец при 13 000 х г в течение 20 мин при 4 °C, а затем используют пипетку с узким наконечником для извлечения супернатанта и загрузки его в новую микроцентрифужную трубку объемом 2 мл.
- Чтобы вывести рнку в осадок, добавляют 1,5 мл ледяного этанола и 1 мкл 20 мг/мл гликогена, вихрь и хранят в течение не менее 1 ч при −20 °C или −80 °C.
ПРИМЕЧАНИЕ: Необязательная точка паузы: РНК может храниться при −20 °C в течение нескольких месяцев.
- Сбор РНК-осадка: Гранулирование осажденной РНК центрифугированием при 13 000 х г в течение 20 мин при 4 °C. Удалите супернатант и дайте оставшемуся этанолу испариться на открытом воздухе (~1 ч) перед повторным использованием гранулы в 30 мкл ddH2Oили 1x TE буфера.
ПРИМЕЧАНИЕ: Этот процесс обычно приводит к конечной концентрации РНК ~ 10 мкМ.
ПРИМЕЧАНИЕ: Необязательная точка паузы: образец РНК может храниться при температуре −20 °C в течение нескольких месяцев.
- Определение концентрации запаса РНК.
- Подготовьте Аликвоту РНК для реакции гидролиза.
- Для выполнения этого анализа, во-первых, используйте наноспектрофотометр UV / Vis для определения A260 исходного образца РНК и сделайте разбавленную аликвоту образца до ~ 10 единиц поглощения (AU) в ddH2O.
- Готовят следующую реакцию в ПЦР-трубке 0,5 мл: 16 мкл ddH2O+ 2 мкл 10xNa2CO3 буфера + 2 мкл Аликвоты РНК, разбавленной до ~10 а.е. Инкубировать реакцию в течение 90 мин при 95 °C, а затем дать ей остыть до RT.
- Определите концентрацию РНК с помощью нуклеотидных абсорбций: Измерьте A260 этого образца с помощью наноспектрофотометра UV / Vis и рассчитайте концентрацию РНК по следующей формуле:

где c — концентрация РНК, b — длина пути, i — специфический нуклеотид (A, C, G или U), ni — частота нуклеотида i в последовательности РНК, а εi — коэффициент молярного вымирания нуклеотида i. Чтобы определить исходную концентрацию РНК, умножьте c на коэффициент разбавления.
ПРИМЕЧАНИЕ: Необязательная точка паузы: РНК может храниться при −20 °C в течение нескольких месяцев.
- Подготовьте Аликвоту РНК для реакции гидролиза.
- PAGE очистка от РНК шпината2 и брокколи
- Выполнение in vitro анализ кинетики считывателя пластин
- Настройте программу ренатурации РНК: Создайте следующий протокол термоциклера, выбрав Создать новую программу > Добавить новую фазу > Добавить новый шаг несколько раз, чтобы добавить каждый из следующих шагов перед нажатием кнопки Сохранить:
70 °C в течение 3 мин
65 °C в течение 45 с
60 °C в течение 45 с
55 °C в течение 45 с
50 °C в течение 45 с
45 °C в течение 45 с
40 °C в течение 45 с
35 °C в течение 45 с
30 °C в течение 45 с - Реконструировать РНК: Чтобы реконструировать РНК Шпината2 и Брокколи, подготовьте 2 мкМ запасов каждой РНК в ddH2O в тонкостенной трубке ПЦР объемом 0,5 мл, а затем добавьте равный объем 2x буфера ренатурации (80 мМ HEPES, рН 7,5 [KOH], 250 мМ KCl, 6 мМ MgCl2), чтобы сделать 1 мкМ растворов РНК. Добавьте трубки в термоциклер, откройте сохраненную программу ренатурации и нажмите кнопку Выполнить.
ПРИМЕЧАНИЕ: Если термоциклер недоступен, РНК можно вместо этого инкубировать на тепловом блоке с температурой 70 °C в течение 3 минут, а затем дать медленно остыть до RT на скамейке. - Подготовка буфера реакции связывания: Чтобы подготовить буфер для одной реакции связывания аптамера и красителя, сделайте мастер-смесь, содержащую буферные компоненты (69,5 мкл ddH2O + 4 мкл 1 M HEPES, pH 7,5 [KOH] + 6,2 мкл 2 M KCl + 0,3 мкл 1 MMgCl2). В зависимости от того, сколько образцов и реплик необходимо, умножьте эти значения на количество образцов плюс один. Как правило, три реплики на образец РНК являются удовлетворительными.
- Подготовьте считыватель пластин.
- Настройте программу инжектора считывателя пластин: на считывателе флуоресцентных пластин выберите Temp и установите желаемую температуру на 37 °C, убедившись, что температура уравновешена до этого значения задолго до начала кинетических экспериментов. Откройте программное обеспечение для чтения пластин, выберите Настройки > Представление сбора и введите следующую программу для кинетических измерений:
Петля: Для каждой скважины
Базовая настройка: 60 считываний базовой линии
Настройки SmartInject: инъекция 10 мкл (которая произойдет после базового считывания)
Флуоресценция (или FL) гласит:
Возбуждение: 448 нм (полоса пропускания: 9 нм)
Эмиссия: 506 нм (полоса пропускания: 15 нм)
Картридж: MONO (s/n 3297)
Синхронизация:
Общее время чтения: 10 мин
Интервал считывания: 0,5 с
PMT и оптика: 6 вспышек на считывание
Петля: Следующая скважина - Подготовьте инжектор
- Промыть и аспирировать инжектор: Чтобы подготовить инжектор считывателя пластин, сначала выберите Впрыск на считыватель пластин, подайте пластину для сбора отходов в считыватель пластин по указанию, а затем выберите Промыть и очистите инъекционную трубку, следуя инструкциям на считывателе пластин с объемом 1 мл ddH2O, 75% этанола в ddH2O, а затем ddH2O. Затем выберите Аспират, позволяющий инжектору выбрасывать лишнюю жидкость.
- Прайм инжектора: После выхода на предыдущий экран выберите Prime, чтобы загрунтовать инжектор двумя объемами лиганда по 260 мкл, которые необходимо ввести, чтобы обеспечить добавление чистого концентрированного лиганда в образцы во время экспериментов - в этом случае прайм со 100 мкМ DFHBI в ddH2O.
- Настройте программу инжектора считывателя пластин: на считывателе флуоресцентных пластин выберите Temp и установите желаемую температуру на 37 °C, убедившись, что температура уравновешена до этого значения задолго до начала кинетических экспериментов. Откройте программное обеспечение для чтения пластин, выберите Настройки > Представление сбора и введите следующую программу для кинетических измерений:
- Выполнение экспериментов по кинетике in vitro : Чтобы выполнить один кинетический эксперимент, сначала добавьте 80 мкл ранее подготовленной связывающей буферной мастер-смеси в одну лунку пластины с прозрачным дном из 96 лунок, а затем 10 мкл ренатурированной РНК. Дайте этой пластине и раствору DFHBI в инжекторе уравновесить до 37 °C в течение 15 минут в считывателе пластин.
- В разделе Параметры программного обеспечения для чтения пластин в разделе Область чтения выберите анализируемую скважину, а затем на вкладке Главная выберите Выполнить , чтобы выполнить программу кинетики, описанную выше. Повторяйте этот процесс до тех пор, пока все эксперименты не будут завершены.
ПРИМЕЧАНИЕ: Критически важно, что кинетические эксперименты должны проводиться по одному за раз, чтобы гарантировать, что кинетика РНК измеряется в одинаковых условиях между репликами и образцами. - Промыть инжектор: Чтобы удалить оставшийся раствор DFHBI из инъекционной трубки, промыть инъекционную трубку, как описано на этапе 1.4.4.2.1, с объемами 1 мл ddH2O, 75% этанола в ddH2O, а затем ddH2O.
ПРИМЕЧАНИЕ: Необязательная точка паузы.
- Настройте программу ренатурации РНК: Создайте следующий протокол термоциклера, выбрав Создать новую программу > Добавить новую фазу > Добавить новый шаг несколько раз, чтобы добавить каждый из следующих шагов перед нажатием кнопки Сохранить:
- Анализ кинетики in vitro фторогенных аптамеров РНК.
- Ввод данных в аналитическое программное обеспечение: Экспорт экспериментальных данных в виде электронной таблицы для легкого копирования данных для обработки. В аналитическом программном обеспечении создайте новую таблицу данных в формате XY. В столбце X введите каждую точку времени считывания, где t = 0 — время инъекции DFHBI. В столбце Y введите средние значения флуоресценции между репликами в соответствующей точке времени, начиная с t = 0.
- Нормализуйте и отобразите данные: чтобы нормализовать значения флуоресценции, нажмите «Анализировать > обработку данных > нормализовать», а затем нажмите «ОК». Выберите использование наименьших и наибольших значений набора данных для нормализации, представления результатов в виде дробей и построения графика результатов, а затем нажмите кнопку ОК. Чтобы создать график нормализованной усредненной флуоресценции с течением времени, щелкните значок Нормализовать [Имя набора данных] и выберите Семейство графов: XY только с точками.
ПРИМЕЧАНИЕ: Полосы ошибок могут быть полезны для просмотра в тех случаях, когда отображается небольшое количество временных точек. Если это необходимо, при выборе таблицы данных формата XY выберите параметр в разделе «Y», чтобы ввести повторяющиеся значения в параллельные столбцы. Построение графика результирующей таблицы с выбранными параметрами по умолчанию приведет к созданию графика с полосами погрешностей. - Выполните подгонку кривой для получения кинетических параметров: Чтобы сопоставить кривую с кинетическими данными, нажмите «Анализировать > анализировать данные » и выберите «Нелинейная регрессия (соответствие кривой)» на вкладке «Анализ XY ». На вкладке Модель щелкните Экспоненциальная > двухфазная ассоциация , чтобы сопоставить данные кинетики со следующим двухфазным уравнением ассоциации:
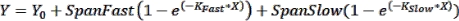
где Y — флуоресценция в момент времени X, Y0 — флуоресценция при t = 0, KFast и KSlow — константы быстрой и медленной скорости соответственно, а SpanFast и SpanSlow — диапазоны включения флуоресценции, учитываемые быстрыми и медленными скоростями соответственно (см. репрезентативные результаты, рисунок 1). Перейдите на вкладку Nonlin Fit , чтобы просмотреть константы скорости, значения t1/2 и значения PercentFast.
ПРИМЕЧАНИЕ: Для получения стандартного отклонения для всех этих значений отдельные реплики экспериментов с считывателем пластин могут быть обработаны таким же образом, как описано выше.
2. Эксперимент по клеточной кинетике
- Приготовление штаммов кишечной палочки
- Преобразование клеток BL21 Star (DE3) E. coli с ~100 нг pET31b tRNA-Spinach2 в соответствии с протоколом производителя.
ПРИМЕЧАНИЕ: Конструкция Plasmid коммерчески доступна (Plasmid #79783). - Обложите клетки на пластинах LB-агара, содержащего карбенициллин (углеводы: 50 мг/мл), и инкубируйте при 37 °C в течение 12-16 ч. Клетки, содержащие плазмиды, будут расти колониями на пластине.
ПРИМЕЧАНИЕ: Дополнительная точка паузы: Преобразованные ячейки BL21 Star на пластинах могут храниться при 4 °C в парапленке в течение 1 недели.
- Преобразование клеток BL21 Star (DE3) E. coli с ~100 нг pET31b tRNA-Spinach2 в соответствии с протоколом производителя.
- Растущие клетки и индуцирующие экспрессию фторогенного РНК-аптамера
- Инокулируют 2 мл культуры неиндуцирующих сред (NI), содержащих карбенициллин (carb: 50 мг/мл), одной колонией преобразованных клеток BL21 Star. Повторите это, по крайней мере, для трех биологических реплик. Инкубировать культуры при 37 °C в инкубаторе/шейкере при 250 об/мин в течение 22-24 ч.
ПРИМЕЧАНИЕ: Необязательная точка паузы: клетки, выращенные в среде NI, сохраняют плазмиду и могут храниться при 4 °C в течение 1 недели. - После роста в среде NI разводят культуру в 100 раз в свежие 3 мл автоиндукционной среды ZYP-5052 (AI), содержащей карбенициллин (карб: 50 мг/мл). Выращивайте клетки при 37 °C в инкубаторе/шейкере при 250 об/мин в течение 16-18 ч, чтобы индуцировать экспрессию.
ПРИМЕЧАНИЕ: Типичный OD600 для культур будет варьироваться от 2,0-3,3 после 18 ч роста. Оптимальный диапазон плотности клеток составляет 2,5-3,0.
- Инокулируют 2 мл культуры неиндуцирующих сред (NI), содержащих карбенициллин (carb: 50 мг/мл), одной колонией преобразованных клеток BL21 Star. Повторите это, по крайней мере, для трех биологических реплик. Инкубировать культуры при 37 °C в инкубаторе/шейкере при 250 об/мин в течение 22-24 ч.
- Проведение эксперимента по клеточной кинетике
- Настройте проточный цитометр.
- Включите проточный цитометр и компьютер, подключенный к прибору. После входа в программное обеспечение для проточного цитометра на вкладке «Инструмент» нажмите на значок «Запуск ». Выполните действия, указанные на экране программного обеспечения, чтобы обеспечить правильную инициализацию инструментария.
ПРИМЕЧАНИЕ: Некоторые проточные цитометры называют последовательность запуска прибора Priming. Обязательно следуйте протоколу производителя для проточного цитометра, который будет использоваться для эксперимента. - Запустите тест производительности (если применимо). На вкладке Главное меню щелкните Тест производительности. В культуральную трубку добавьте три капли шариков контроля производительности производителя в 3 мл фокусирующей жидкости.
- Поместите культуральную трубку в подъемник для пробоотборников и поднимите подъемник. Перед нажатием кнопки Выполнить тест производительности убедитесь, что номер партии трубки отслеживающих шариков совпадает с тем, что указано на экране настройки теста производительности . Щелкните Тест производительности , чтобы запустить тест.
- Настройте программное обеспечение проточного цитометра для этого эксперимента со следующими параметрами сбора для флуоресценции с одной ячейкой:
Лазер возбуждения: 488 нм
Эмиссионный канал: GFP (также называемый FITC)
Объем захвата: 40 мкл (с общим объемом потребления 90 мкл)
Расход: 200 мкл / мин
Количество ячеек для каждого измерения: 30 000
Настройки прибора:
Напряжение:
КФУ: 480 В
SSC: 400 В
БЛ1: 540 В
БЛ2: 392 В
БЛ3: 422 В
- Включите проточный цитометр и компьютер, подключенный к прибору. После входа в программное обеспечение для проточного цитометра на вкладке «Инструмент» нажмите на значок «Запуск ». Выполните действия, указанные на экране программного обеспечения, чтобы обеспечить правильную инициализацию инструментария.
- Настройте экспериментальные файлы.
- Создайте новый файл эксперимента на вкладке Обозреватель экспериментов , щелкнув правой кнопкой мыши имя пользователя проточного цитометра. Выберите Новый эксперимент в раскрывающемся окне. Когда на экране компьютера появится новое окно, нажмите кнопку ОК.
- В новом файле эксперимента щелкните правой кнопкой мыши папку «Группа» и выберите «Добавить новую пробу». Пометьте образцы для каждой конкретной точки времени и воспроизведите, щелкнув правой кнопкой мыши образец и выбрав Переименовать в раскрывающемся меню. Повторите этот шаг для общего числа реплик и временных точек для предполагаемого временного хода исследования.
- Приготовьте раствор разбавленных клеток: В новую культуральную трубку добавьте 1,5 мл 1x раствора PBS. Затем добавьте 3 мкл индуцированных клеток в среде ИИ в 1x раствор PBS, чтобы сделать раствор разбавленных клеток. Повторите этот шаг для каждого биологического реплицирования в разных культуральных трубках.
- Измерьте фоновую флуоресценцию клеток: Прежде чем добавлять краситель, снимите показания каждой биологической реплицированной культуральной трубки, содержащей клетки в 1x растворе PBS. Это делается для того, чтобы флуоресцентный фон клеток измерялся, чтобы наблюдать, как складка включается с течением времени после добавления красителя.
- Чтобы сделать показания, поместите культуральную трубку в подъемник пробоотборника и поднимите подъемник вручную к игле для инъекции образца. Выберите нужный образец файла на вкладке Панель коллекции и нажмите кнопку Запись.
- Когда пробег будет завершен, опустите подъемник пробоотборника с культуральной трубкой вручную. Это инициирует этап промывки , который будет промывать жидкостную систему и сводит к минимуму перенос между каждым биологическим реплицированным образцом. Данные будут автоматически сохранены на компьютере после завершения запуска.
- Повторите шаги в пределах 2.3.4, чтобы измерить клеточный флуоресцентный фон по крайней мере для трех биологических реплик. Чтобы перейти к следующему файлу образца, выберите следующий файл образца, щелкнув значок стрелки вправо рядом с именем пробы под значком Запись .
- Измерьте флуоресценцию в точках времени для клеток с красителем.
- Добавьте 1,4 мкл концентрированного запаса красителя (50 мМ DFHBI-1T в DMSO) в 1 раствор PBS с клетками для получения конечной концентрации 50 мкМ DFBHI-1T. Затем закрепите крышку культуральной трубки, а затем переверните культуральные трубки 3x-5x, чтобы равномерно перемешать раствор перед первым показанием точки времени.
ПРИМЕЧАНИЕ: Общий процент ДМСО в культуральных пробирках для E. coli не должен превышать 10%, так как это может повлиять на жизнеспособность клеток16. - Снимите крышку и поместите культуральную трубку в подъемник для пробоотборников. Поднимите держатель вручную к игле для инъекции образца и под соответствующим файлом образца щелкните значок Запись . Кроме того, запустите таймер, нажав кнопку Пуск для эксперимента.
- Опустите трубчатый подъемник вручную после завершения пробега и повторите шаги 2.3.5.1-2.3.5.2. (с работающим таймером) путем добавления 1,4 мкл концентрированного DFHBI-1T, инвертирования культуральных трубок и получения показаний для всех оставшихся биологических реплик. Эти первые записи будут показаниями, полученными через 0 мин для всех биологических реплик. Делайте это по одному для каждой биологической репликации.
ПРИМЕЧАНИЕ: Запишите время, когда нажата запись получения проточной цитометрии. Придерживайтесь этого времени, ошеломляющего по временным точкам в ходе эксперимента. - Продолжайте снимать показания, поднимая пробирки культуры в подъемнике пробоотборника к игле для инъекции образца, выбирая правильный файл образца, нажимая кнопку «Запись» и опуская подъемник вручную после завершения каждого прогона. Сделайте это для всех дополнительных временных точек и тестируемых биологических реплик. Повторяйте эти шаги, пока эксперимент не будет завершен.
ПРИМЕЧАНИЕ: Держите образцы подальше от света, чтобы избежать фотоотбеливания DFHBI-1T в растворе, покрыв образцы алюминиевой фольгой.
- Добавьте 1,4 мкл концентрированного запаса красителя (50 мМ DFHBI-1T в DMSO) в 1 раствор PBS с клетками для получения конечной концентрации 50 мкМ DFBHI-1T. Затем закрепите крышку культуральной трубки, а затем переверните культуральные трубки 3x-5x, чтобы равномерно перемешать раствор перед первым показанием точки времени.
- Измерение флуоресценции в точках времени для клеток без красителя (Контроль).
- Запустите соответствующие протоколы очистки для проточного цитометра, прежде чем повторить эксперимент снова с отрицательными элементами управления в соответствии с протоколом производителя. Это делается для минимизации любого переноса из предыдущего эксперимента в эксперимент по анализу контрольных временных точек. Ниже приведены шаги, выполняемые для проточного цитометра между экспериментами:
- Поместите пустую культуральную трубку в подъемник труб вручную, поднимите держатель трубки и нажмите на значок Unclog на вкладке «Инструмент ». Это запустит обратную промывку в системе жидкости, чтобы очистить любые липкие образцы. Опустите держатель трубки вручную и извлеките трубку, как только будет выполнена последовательность Unclog .
- С помощью новой культуральной трубки добавьте 3 мл 10% раствора отбеливателя, поместите культуральную трубку в держатель трубки и поднимите держатель вручную к игле для инъекции образца. Кроме того, поместите чистую 96-луночную пластину в автопробоотборник, если это применимо к проточному цитометру.
- Нажмите на значок Sanitize SIP / Sanitize Autosampler SIP, чтобы запустить последовательность очистки с 10% отбеливателем по всей системе жидкости. Опустите держатель трубки, чтобы завершить последовательность очистки.
- Задайте файлы образцов для выполнения анализа контрольных точек времени в соответствии с инструкциями, приведенными в шаге 2.3.3.
- В новой культуральной пробирке готовят раствор разбавленных клеток в 1,5 мл 1x раствора PBS. Добавьте 3 мкл индуцированных клеток в среде ИИ в 1x раствор PBS, чтобы сделать раствор разбавленных клеток. Повторите этот шаг для каждой биологической репликации.
- Добавляют 1,4 мкл ДМСО в культуральную трубку по одному к 1x раствору PBS с клетками и тестируют те же временные точки. Закрепите крышку культуральной трубки, а затем переверните культуральные трубки 3x-5x, чтобы равномерно перемешать раствор перед первым показанием точки времени. Делайте это по одному для каждой биологической репликации.
- Следуйте тому же протоколу анализа контрольных клеток, что и для клеток с красителем, используя шаги 2.3.5.2-2.3.5.4.
- Запустите соответствующие протоколы очистки для проточного цитометра, прежде чем повторить эксперимент снова с отрицательными элементами управления в соответствии с протоколом производителя. Это делается для минимизации любого переноса из предыдущего эксперимента в эксперимент по анализу контрольных временных точек. Ниже приведены шаги, выполняемые для проточного цитометра между экспериментами:
- Выключите проточный цитометр: следуйте протоколу производителя для правильного отключения контрольно-измерительных приборов. Для проточного цитометра прибор подготавливается к выключению следующим образом:
- Выполните протокол первоначальной очистки проточного цитометра, выполнив шаги 2.3.6.1.1-2.3.6.1.3.
- Заменить культуральную трубку 10% раствором отбеливателя культуральной трубкой с 3 мл фокусирующей жидкости. Поднимите держатель трубки вручную и под значком Shutdown нажмите на раскрывающееся меню и выберите Тщательно.
ПРИМЕЧАНИЕ: Необязательная точка паузы.
- Настройте проточный цитометр.
- Анализ данных проточной цитометрии
- Экспортируйте все файлы FCS для анализа. Откройте файлы FCS с помощью программного обеспечения для анализа проточной цитометрии.
- Используя один из файлов FCS, предназначенных только для ячеек, сгенерируйте затвор из диаграммы прямого рассеяния (FSC) и бокового рассеяния (SSC) (FSC-Area/SSC-Area), используя обе оси журнала, чтобы исключить любые сигналы от мусора. Чтобы создать этот шлюз, нажмите на значок AutoGate в программном обеспечении для анализа проточной цитометрии и назовите его Gate 1. Примените этот же шлюз ко всем образцам, протестированным на вкладке Все образцы в рабочей области обработки данных. Это приведет к тому , что Gate 1 находится под всеми обрабатываемыми файлами FCS.
- Создайте новый файл подмножества с файлом FCS Cell only, используемым на шаге 2.4.2, дважды щелкнув его. Измените настройки оси на FSC-Area/FSC-Height, используя обе оси журнала. Нажмите на значок AutoGate в программном обеспечении для анализа проточной цитометрии, чтобы сгенерировать овальные ворота, назвав их Gate 2. Примените этот затвор в качестве подмножества под затвором, установленным на шаге 2.4.2, ко всем тестируемым образцам. Это приведет к тому, что все образцы будут иметь Gate 1 > Gate 2 , связанные с каждым файлом FCS.
- Создайте другой файл подмножества с обоими шлюзами, установленными на шаге 2.4.2 и шаге 2.4.3, примененном двойным щелчком Gate 2. Измените настройки оси на FSC-область/гистограмму. Примените этот затвор гистограммы в качестве подмножества ко всем тестируемым образцам, в результате чего все образцы будут иметь Gate 1 > Gate 2 > Gate 2 , связанные с каждым файлом FCS.
ПРИМЕЧАНИЕ: Гистограммы могут быть переименованы из Gate 2 в Histogram , чтобы помочь с перемещением гистограмм в окно макета, а также создать большую организацию с обработкой данных. - Чтобы проанализировать значения средней интенсивности флуоресценции (MFI), откройте окно макета. Щелкните и перетащите затворы гистограммы для каждой точки времени в окно макета.
- Выполните статистический анализ для «среднего ∑: BL1-A» (GFP) для каждого тестируемого образца для отображения результатов MFI в окне макета.
- Рассчитайте стандартное отклонение для значений МФО на анализ временных точек по крайней мере для трех независимых биологических реплик.
- Сохраните гистограммы и значения MFI для каждой точки времени, экспортировав окно макета в виде PDF-файла.
ПРИМЕЧАНИЕ: Необязательная точка паузы.
- Данные графовой проточной цитометрии
- Откройте PDF-файл, содержащий гистограммы и значения MFI для каждой точки времени. Значения MFI будут скопированы в программное обеспечение для анализа данных. В программном обеспечении для построения графиков данных создайте новую таблицу данных в формате XY.
- Выберите, чтобы создать таблицу XY со следующим выбранным параметром:
Таблица данных: ввод или импорт данных в новую таблицу
Параметры:
X: Истекшее время
Y: Ввод (от трех до четырех) реплицированных значений в параллельных подколонках - Пометьте на оси X все точки времени для выполнения эксперимента и элемента управления.
- В группе А введите значения МФО для всех биологических реплик в каждую точку времени для флуоресцентного анализа клеток с добавлением красителя.
- В группе B введите значения MFI для всех биологических реплик в каждую точку времени для анализа клеток без добавления красителя (DMSO).
- Чтобы увидеть результаты, нажмите [Вставить имя набора данных] на вкладке Графики . Это будет отображать точки данных как средства, с полосами погрешностей, представляющими стандартное отклонение (s.d.) в каждой точке времени. Ось X представляет прошедшее время, а ось Y представляет значения MFI.
- Выберите, чтобы создать таблицу XY со следующим выбранным параметром:
- Откройте PDF-файл, содержащий гистограммы и значения MFI для каждой точки времени. Значения MFI будут скопированы в программное обеспечение для анализа данных. В программном обеспечении для построения графиков данных создайте новую таблицу данных в формате XY.
Результаты
Кинетика in vitro
Последовательности шаблонов ДНК и праймеров, которые приобретаются в виде синтетических олигонуклеотидов, приведены в таблице 2, а рецепты реагентов приведены в дополнительном файле 1. Амплификация ПЦР используется для увеличе...
Обсуждение
Для эксперимента по кинетике in vitro тот же общий протокол может быть модифицирован для измерения кинетики in vitro флуоресцентного биосенсора на основе РНК, содержащего как лигандсвязывающий, так и флуорофорсвязывающий домен8. В этом случае РНК следует инкубировать с...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Эта работа была поддержана следующими грантами MCH: NSF-BSF 1815508 и NIH R01 GM124589. MRM был частично поддержан учебным грантом NIH T32 GM122740.
Материалы
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
Ссылки
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295 (2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139 (2016).
- Paige, J. S., Thinh, N. -. D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194 (2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002 (2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены