Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détermination de la cinétique d’activation in vitro et cellulaire pour les aptamères d’ARN fluorogéniques
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le protocole présente deux méthodes pour déterminer la cinétique des aptamères d’ARN fluorogéniques Spinach2 et Brocoli. La première méthode décrit comment mesurer la cinétique des aptamères fluorogéniques in vitro avec un lecteur de plaques, tandis que la deuxième méthode détaille la mesure de la cinétique des aptamères fluorogéniques dans les cellules par cytométrie en flux.
Résumé
Des aptamères d’ARN fluorogéniques ont été appliqués dans des cellules vivantes pour marquer et visualiser les ARN, rendre compte de l’expression des gènes et activer des biocapteurs fluorescents qui détectent les niveaux de métabolites et de molécules de signalisation. Afin d’étudier les changements dynamiques dans chacun de ces systèmes, il est souhaitable d’obtenir des mesures en temps réel, mais la précision des mesures dépend de la cinétique de la réaction fluorogénique plus rapide que la fréquence d’échantillonnage. Ici, nous décrivons des méthodes pour déterminer la cinétique d’activation in vitro et cellulaire pour les aptamères d’ARN fluorogénique à l’aide d’un lecteur de plaques équipé respectivement d’un injecteur d’échantillon et d’un cytomètre en flux. Nous montrons que la cinétique in vitro pour l’activation de fluorescence des aptamères Spinach2 et Brocoli peut être modélisée comme des réactions d’association à deux phases et ont différentes constantes de vitesse de phase rapide de 0,56 s-1 et 0,35 s-1, respectivement. De plus, nous montrons que la cinétique cellulaire pour l’activation de fluorescence des épinards2 chez Escherichia coli, qui est encore limitée par la diffusion du colorant dans les bactéries à Gram négatif, est encore suffisamment rapide pour permettre une fréquence d’échantillonnage précise sur l’échelle de temps infime. Ces méthodes d’analyse de la cinétique d’activation de fluorescence sont applicables à d’autres aptamères d’ARN fluorogéniques qui ont été développés.
Introduction
Les réactions fluorogéniques sont des réactions chimiques qui génèrent un signal de fluorescence. Les aptamères d’ARN fluorogénique remplissent généralement cette fonction en liant un colorant à petite molécule pour améliorer son rendement quantique de fluorescence (Figure 1A)1. Différents systèmes d’aptamères d’ARN fluorogéniques ont été développés et se composent de séquences d’aptamères d’ARN spécifiques et des ligands colorants correspondants1. Des aptamères d’ARN fluorogéniques ont été ajoutés aux transcrits d’ARN sous forme de marqueurs fluorescents qui permettent l’imagerie de cellules vivantes d’ARNm et d’ARN non codants 2,3,4. Ils ont également été placés après des séquences promotrices en tant que rapporteurs fluorescents de l’expression génique, similaire à l’utilisation de la protéine fluorescente verte (GFP) comme rapporteur, sauf que la fonction de déclaration est au niveau de l’ARN 5,6. Enfin, des aptamères d’ARN fluorogéniques ont été incorporés dans des biocapteurs fluorescents à base d’ARN, conçus pour déclencher la réaction fluorogénique en réponse à une petite molécule spécifique. Des biocapteurs fluorescents à base d’ARN ont été développés pour l’imagerie de cellules vivantes de divers métabolites non fluorescents et molécules de signalisation 7,8,9,10,11.
Il y a un intérêt croissant pour le développement d’aptamères d’ARN fluorogéniques pour visualiser les changements dynamiques dans la localisation de l’ARN, l’expression des gènes et les signaux de petites molécules. Pour chacune de ces applications, il est souhaitable d’obtenir des mesures en temps réel, mais la précision des mesures dépend de la cinétique de la réaction fluorogénique plus rapide que la fréquence d’échantillonnage. Ici, nous décrivons des méthodes pour déterminer la cinétique in vitro des aptamères d’ARN fluorogéniques Spinach2 12 et Brocoli13 à l’aide d’un lecteur de plaques équipé d’un injecteur d’échantillon et pour déterminer la cinétique d’activation cellulaire pourSpinach2 exprimée dans Escherichia coli à l’aide d’un cytomètre en flux. Ces deux aptamères d’ARN ont été choisis parce qu’ils ont été appliqués pour étudier la localisation de l’ARN 2,3,4, ils ont été utilisés dans les rapporteurs5,6 et les biocapteurs 7,8,9,10,11, et les ligands colorants correspondants (DFHBI ou DFHBI-1T) sont disponibles dans le commerce. Un résumé de leurs propriétés in vitro déterminées dans la littérature est présenté dans le tableau 1 4,13,14, qui a éclairé l’élaboration du protocole (p. ex., les longueurs d’onde et les concentrations de colorant utilisées). Ces résultats démontrent que les réactions fluorogéniques affectées par les aptamères d’ARN sont rapides et ne devraient pas empêcher des mesures précises pour les applications biologiques cellulaires souhaitées.
Protocole
1. Expérience de cinétique in vitro
- Préparation de modèles d’ADN par PCR
- Configurer la ou les réactions PCR : Pour préparer les réactions PCR, combinez les réactifs suivants dans un tube PCR à paroi mince :
33 μL d’eau bidistillée (ddH2O)
10 μL de tampon 5x pour ADN polymérase haute fidélité
5 μL de 2 mM de désoxyribonucléoside triphosphate (dNTP)
0,5 μL d’amorce avant de 40 μM
0,5 μL d’amorce inverse 40 μM
0,5 μL (10-100 ng) de matrice d’ADN (pour la PCR Spinach2 seulement; Les apprêts pour le brocoli se chevauchent)
0,5 μL d’ADN polymérase haute fidélité (ajouter en dernier)
REMARQUE: Les oligonucléotides synthétiques sont souvent expédiés à sec. Pour préparer des solutions mères, ajoutez un volume connu (100 μL recommandé) de ddH2O et mesurez le A260 de cette solution pour déterminer la concentration par la loi de Beer, où le coefficient d’extinction peut être calculé par les règles du plus proche voisin en ligne. Cette solution mère peut ensuite être utilisée pour rendre les dilutions appropriées pour une utilisation en PCR. - Exécutez le protocole thermocycleur.
- Utilisez le protocole thermocycleur suivant pour amplifier les modèles d’ADN d’épinards2 et de brocoli sur toute la longueur :
Dénaturation initiale :98 °C pendant 2 min
35 cycles :
Dénaturation:98 °C dénaturation pendant 15 s
Recuit :72 °C pendant 30 s
Extension : 72 °C pendant 30 s
Extension finale : 72 °C pendant 5 min. - Après la réaction, analyser une petite partie aliquote du produit par un gel d’agarose à 2% avec une échelle de faible poids moléculaire (gamme de taille: 25-766 nucléotides) pour confirmer la présence du produit d’ADN désiré.
- Purifier le produit par extraction de gel disponible dans le commerce ou kits de nettoyage PCR et éluer avec ddH2O ou le tampon fourni par le fabricant.
REMARQUE: Assurez-vous lors du choix d’un kit de nettoyage PCR que le seuil de poids moléculaire de la colonne est suffisamment bas pour retenir T7-Brocoli (81 nucléotides), sinon le produit PCR sera perdu.
REMARQUE: Point de pause facultatif: stockez l’ADN à -20 ° C.
- Utilisez le protocole thermocycleur suivant pour amplifier les modèles d’ADN d’épinards2 et de brocoli sur toute la longueur :
- Configurer la ou les réactions PCR : Pour préparer les réactions PCR, combinez les réactifs suivants dans un tube PCR à paroi mince :
- Préparation d’épinards2 et d’ARN de brocoli par transcription in vitro (IVT)
- Configurez la ou les réactions de transcription.
- Pour préparer une réaction de transcription de 100 μL, combiner les réactifs suivants dans un tube microcentrifuge de 1,5 mL : 10 μL de tampon de transcription 10x + 20 μL de ribonucléosides triphosphates 10 mM (rNTP) + 1-64 μL de matrice d’ADN (total de 1 μg) + 2 μL de pyrophosphatase inorganique +ddH2Oà 98 μL + 2 μL d’ARN polymérase T7 (ajouter en dernier).
- Incuber cette réaction pendant 4 h à 37 °C. Éteignez la réaction en ajoutant 100 μL de 2x tampon de charge de gel d’urée (2x ULB), composé de 20% de saccharose, 0,1% de dodécylsulfate de sodium (SDS), 1x tampon Tris-Borate-EDTA (1x TBE) et ~18 M d’urée.
REMARQUE : Point de pause facultatif : stocker la réaction éteinte à −20 °C.
- Configurez la ou les réactions de transcription.
- Purification de l’ARN par électrophorèse sur gel polyacrylamide (PAGE)
- Purification PAGE des épinards2 et de l’ARN du brocoli
ATTENTION : L’acrylamide non polymérisé (liquide ou en poudre) est extrêmement toxique. Si vous pesez de l’acrylamide en poudre, faites-le dans une hotte. Portez toujours l’équipement de protection approprié et retirez immédiatement les gants contaminés par de la poudre ou de la solution d’acrylamide, en vous lavant soigneusement les mains. Si l’acrylamide entre en contact direct avec la peau, laver la zone exposée pendant au moins 15 minutes avec de l’eau et du savon. Si l’acrylamide entre en contact direct avec les yeux, rincez-les à l’eau pendant 15 min.- Préparez le gel PAGE : Pour éliminer les transcrits abortifs indésirables et les rNTP n’ayant pas réagi du produit complet, préparez un gel d’urée-polyacrylamide à 6 %. En général, des gels de 28 cm x 16,5 cm x 1,5 mm peuvent être utilisés avec un peigne à 8 puits. Installez l’équipement de gel et d’électrophorèse, en utilisant 1x tampon TBE pour remplir les réservoirs.
- Charger le(s) échantillon(s) d’ARN dans le gel PAGE : Charger le gel avec une réaction trempée de 200 μL par voie. Dans une voie séparée, charger 2x ULB avec des colorants trackers xylène cyanol et bleu de bromophénol, qui migrent dans le gel à 106 nucléotides et 26 nucléotides, respectivement15. Laissez une voie vide entre chaque échantillon pour éviter toute contamination potentielle aux étapes suivantes.
- Pour séparer les épinards 95-nt2 et 49-nt Brocoli de leurs produits tronqués respectifs, faites fonctionner le gel pendant 1,5-2 h à 25 W, après quoi le colorant bleu de bromophénol aura migré ~5/6 de la longueur du gel.
- Visualisez le(s) échantillon(s) d’ARN dans le gel PAGE : Démontez les plaques de verre autour du gel et couvrez le gel d’une pellicule plastique des deux côtés, en étiquetant les voies sur l’enveloppe. Visualisez les bandes d’ARN dans une pièce sombre par ombrage UV en plaçant le gel enveloppé sur une plaque TLC fluorescente sous lumière UV. Délimitez rapidement le bord des bandes d’ARN correspondant au produit à l’aide d’un marqueur et éteignez la lampe UV pour minimiser les dommages causés par l’exposition aux UV.
- Extraire le(s) échantillon(s) d’ARN du gel PAGE : Avec une lame de rasoir fraîche pour chaque échantillon, exciser les bandes de produit souhaitées, couper en dés en cubes de ~1 mm et ajouter les morceaux de gel dans un tube microcentrifuge de 2 ml avec 500 μL de tampon de trempage d’écrasement pour extraire l’ARN sur un rotateur pendant 2 h à température ambiante (RT) ou toute la nuit à 4 °C.
NOTE: Point de pause facultatif: l’échantillon peut être conservé à −20 °C. - Précipiter l’ARN
- Pour séparer les morceaux de gel de l’ARN extrait dans un tampon, centrifuger l’échantillon à 13 000 x g pendant 20 minutes à 4 °C, puis utiliser une pipette à pointe étroite pour extraire le surnageant et le charger dans un nouveau tube microcentrifuge de 2 mL.
- Pour précipiter l’ARN, ajouter 1,5 mL d’éthanol glacé et 1 μL de glycogène 20 mg/mL, vortex, et conserver pendant au moins 1 h à -20 °C ou -80 °C.
NOTE: Point de pause facultatif: l’ARN peut être stocké à -20 ° C pendant quelques mois.
- Collecte du précipité d’ARN : Enduit l’ARN précipité par centrifugation à 13 000 x g pendant 20 min à 4 °C. Retirer le surnageant et laisser l’éthanol restant s’évaporer à l’air libre (~1 h) avant de remettre la pastille en suspension dans 30 μL de tamponddH2Oou 1x TE.
REMARQUE: Ce processus entraîne généralement une concentration finale d’ARN de ~ 10 μM.
NOTE: Point de pause facultatif: l’échantillon d’ARN peut être conservé à -20 ° C pendant quelques mois.
- Déterminer les concentrations du stock d’ARN.
- Préparer une aliquote d’ARN pour la réaction d’hydrolyse.
- Pour effectuer ce test, utilisez d’abord un nanospectrophotomètre UV / Vis pour déterminer le A260 de l’échantillon d’ARN stock et faites une partie aliquote diluée de l’échantillon à ~10 unités d’absorbance (UA) dans ddH2O.
- Préparer la réaction suivante dans un tube de PCR de 0,5 mL : 16 μL deddH2O+ 2 μL de tampon 10xNa2CO3+ 2 μL d’ARN aliquote dilué à ~10 UA. Incuber la réaction pendant 90 min à 95 °C, puis laisser refroidir à TA.
- Déterminer la concentration d’ARN à l’aide d’absorbances nucléotidiques : Mesurer l’A260 de cet échantillon à l’aide d’un nanospectrophotomètre UV/Vis et calculer la concentration d’ARN à l’aide de la formule suivante :

où c est la concentration d’ARN, b est la longueur du trajet, i est le nucléotide spécifique (A, C, G ou U), n i est la fréquence du nucléotide i dans la séquence d’ARN et ε i est le coefficient d’extinction molaire du nucléotide i. Pour déterminer la concentration initiale d’ARN stock, multiplier c par le facteur de dilution.
REMARQUE: Point de pause facultatif: L’ARN peut être stocké à -20 ° C pendant quelques mois.
- Préparer une aliquote d’ARN pour la réaction d’hydrolyse.
- Purification PAGE des épinards2 et de l’ARN du brocoli
- Exécution d’une in vitro Test de cinétique du lecteur de plaques
- Configurer le programme de renaturation de l’ARN : Créez le protocole thermocycleur suivant en sélectionnant Créer un nouveau programme > Ajouter une nouvelle phase > Ajouter une nouvelle étape plusieurs fois pour ajouter chacune des étapes suivantes avant d’appuyer sur Enregistrer :
70 °C pendant 3 min
65 °C pendant 45 s
60 °C pendant 45 s
55 °C pendant 45 s
50 °C pendant 45 s
45 °C pendant 45 s
40 °C pendant 45 s
35 °C pendant 45 s
30 °C pendant 45 s - Renaturer l’ARN : Pour renaturer les ARN d’épinards2 et de brocoli, préparer 2 μM de chaque ARN dans ddH2O dans un tube de PCR à paroi mince de 0,5 mL, puis ajouter un volume égal de tampon de renaturation 2x (80 mM HEPES, pH 7,5 [KOH], 250 mM KCl, 6 mM MgCl2) pour faire des solutions d’ARN 1 μM. Ajoutez les tubes au thermocycleur, ouvrez le programme de renaturation enregistré et appuyez sur Exécuter.
REMARQUE: Si un thermocycleur n’est pas disponible, les ARN peuvent plutôt être incubés sur un bloc de chaleur à 70 ° C pendant 3 minutes, puis laissés refroidir lentement à TA sur le banc. - Préparer le tampon de réaction de liaison: Pour préparer le tampon pour une réaction de liaison aptamère-colorant, faire un mélange maître contenant des composants tampons (69,5 μL de ddH2O+ 4 μL de 1 M HEPES, pH 7,5 [KOH] + 6,2 μL de 2 M KCl + 0,3 μL de 1 M MgCl2). Selon le nombre d’échantillons et de répétitions nécessaires, multipliez ces valeurs par le nombre d’échantillons plus un. En général, trois répétitions par échantillon d’ARN sont satisfaisantes.
- Préparez le lecteur de plaques.
- Configurer le programme d’injecteur du lecteur de plaques : Sur le lecteur de plaques de fluorescence, sélectionnez Temp et réglez la température souhaitée à 37 °C, en vous assurant que la température s’est équilibrée à cette valeur bien avant de commencer les expériences de cinétique. Ouvrez le logiciel de lecture de plaques, sélectionnez Paramètres > vue d’acquisition et entrez le programme suivant pour les mesures cinétiques :
Boucle : Pour chaque puits
Paramètre de référence : 60 lectures de ligne de base
Paramètres SmartInject : injection de 10 μL (qui se produira après les lectures de la ligne de base)
La fluorescence (ou FL) se lit comme suit :
Excitation: 448 nm (bande passante: 9 nm)
Émission: 506 nm (bande passante: 15 nm)
Cartouche : MONO (s/n 3297)
Timing:
Temps de lecture total : 10 min
Intervalle de lecture: 0.5 s
PMT et optique : 6 flashs par lecture
Boucle : Puits suivant - Préparer l’injecteur
- Lavez et aspirez l’injecteur : Pour préparer l’injecteur de lecteur de plaques, sélectionnez d’abord Injecter sur le lecteur de plaques, fournissez une plaque de collecte des déchets au lecteur de plaques lorsqu’il est dirigé, puis sélectionnez Lavage et nettoyez le tube d’injection en suivant les instructions sur le lecteur de plaques avec 1 mL de volumes de ddH2O, éthanol à 75 % dans ddH2O, puis ddH2O. Ensuite, sélectionnez Aspirate, permettant à l’injecteur d’éjecter l’excès de liquide.
- Amorcer l’injecteur : Après avoir quitté l’écran précédent, sélectionnez Prime pour amorcer l’injecteur avec deux volumes de 260 μL de ligand à injecter pour s’assurer que le ligand pur et concentré est ajouté aux échantillons pendant les expériences - dans ce cas, amorcer avec 100 μM DFHBI dans ddH2O.
- Configurer le programme d’injecteur du lecteur de plaques : Sur le lecteur de plaques de fluorescence, sélectionnez Temp et réglez la température souhaitée à 37 °C, en vous assurant que la température s’est équilibrée à cette valeur bien avant de commencer les expériences de cinétique. Ouvrez le logiciel de lecture de plaques, sélectionnez Paramètres > vue d’acquisition et entrez le programme suivant pour les mesures cinétiques :
- Effectuer des expériences de cinétique in vitro : Pour effectuer une expérience cinétique, ajoutez d’abord 80 μL de mélange principal tampon de liaison préalablement préparé à un puits d’une plaque à fond clair de 96 puits, suivi de 10 μL d’ARN renaturé. Laisser cette plaque et la solution DFHBI dans l’injecteur s’équilibrer à 37 °C pendant 15 min dans le lecteur de plaques.
- Dans Paramètres du logiciel de lecture de plaques sous Zone de lecture, sélectionnez le puits à analyser, puis, sous l’onglet Accueil , sélectionnez Exécuter pour exécuter le programme cinétique décrit précédemment. Répétez ce processus jusqu’à ce que toutes les expériences soient terminées.
REMARQUE : De façon critique, les expériences de cinétique doivent être effectuées un puits à la fois pour s’assurer que la cinétique de l’ARN est mesurée dans des conditions identiques entre les répétitions et les échantillons. - Laver l’injecteur : Pour retirer la solution DFHBI restante du tube d’injection, laver le tube d’injection comme décrit à l’étape 1.4.4.2.1 avec 1 mL de volumes de ddH2O, d’éthanol à 75 % dans du ddH2O, puis duddH2O.
REMARQUE : Point de pause facultatif.
- Configurer le programme de renaturation de l’ARN : Créez le protocole thermocycleur suivant en sélectionnant Créer un nouveau programme > Ajouter une nouvelle phase > Ajouter une nouvelle étape plusieurs fois pour ajouter chacune des étapes suivantes avant d’appuyer sur Enregistrer :
- Analyse de la cinétique in vitro des aptamères d’ARN fluorogéniques.
- Entrée de données dans le logiciel d’analyse : exportez les données expérimentales sous forme de feuille de calcul pour les copier facilement en vue de leur traitement. Dans le logiciel d’analyse, créez une nouvelle table de données au format XY. Dans la colonne X, entrez chaque point de temps de lecture, t = 0 étant le moment de l’injection de DFHBI. Dans la colonne Y, entrez les valeurs de fluorescence moyennes entre les répétitions à ce point de temps respectif en commençant par t = 0.
- Normaliser et représenter graphiquement les données : Pour normaliser les valeurs de fluorescence, cliquez sur Analyser > Traitement des données > Normaliser, puis cliquez sur OK. Choisissez d’utiliser les valeurs les plus petites et les plus grandes du jeu de données pour la normalisation, de présenter les résultats sous forme de fractions et de représenter graphiquement les résultats, puis cliquez sur OK. Pour créer un graphique de la fluorescence moyenne normalisée au fil du temps, cliquez sur l’icône Normaliser de [Nom du jeu de données] et sélectionnez Famille de graphes : XY avec points uniquement.
Remarque : Les barres d’erreur peuvent être utiles à voir dans les cas où un petit nombre de points temporels sont représentés graphiquement. Si vous le souhaitez, lorsque vous choisissez une table de données au format XY, sélectionnez l’option sous « Y » pour entrer des valeurs répliquées dans des colonnes côte à côte. L’représentation graphique du tableau résultant avec les options par défaut sélectionnées produira un graphique avec des barres d’erreur. - Effectuer l’ajustement de courbe pour obtenir des paramètres cinétiques : Pour ajuster une courbe aux données cinétiques, cliquez sur Analyser > Analyser les données et sélectionnez Régression non linéaire (ajustement de la courbe) sous l’onglet Analyses XY . Sous l’onglet Modèle , cliquez sur Association exponentielle > deux phases pour ajuster les données cinétiques à l’équation d’association à deux phases suivante :
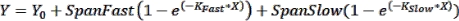
où Y est la fluorescence au temps X, Y 0 est la fluorescence à t = 0, K Fast et K Slow sont respectivement des constantes de vitesse rapide et lente, et SpanFast et SpanSlow sont les plages d’activation de la fluorescence expliquées respectivement par les vitesses rapides et lentes (voir les résultats représentatifs, Figure 1). Cliquez sur l’onglet Nonlin Fit pour afficher les constantes de taux, les valeurs t1/2 et les valeurs PercentFast.
REMARQUE : Pour obtenir un écart-type pour toutes ces valeurs, les répétitions expérimentales individuelles de lecteurs de plaques peuvent être traitées de la même manière que celle décrite ci-dessus.
2. Expérience de cinétique cellulaire
- Préparation des souches d’E. coli
- Transformez les cellules d’E. coli BL21 Star (DE3) avec ~100 ng de construction d’ARNt-épinards pET31b en suivant le protocole du fabricant.
REMARQUE: La construction plasmidique est disponible dans le commerce (Plasmide #79783). - Plaquer les cellules sur des plaques de gélose LB contenant de la carbénicilline (Carb : 50 mg/mL) et incuber à 37 °C pendant 12-16 h. Les cellules contenant des plasmides se développeront sous forme de colonies sur la plaque.
REMARQUE: Point de pause facultatif: Les cellules BL21 Star transformées sur les plaques peuvent être stockées à 4 ° C enveloppées dans un parafilm pendant 1 semaine.
- Transformez les cellules d’E. coli BL21 Star (DE3) avec ~100 ng de construction d’ARNt-épinards pET31b en suivant le protocole du fabricant.
- Croissance de cellules et induction de l’expression d’aptamères d’ARN fluorogénique
- Inoculer une culture de 2 mL de milieux non inducteurs (NI) contenant de la carbénicilline (Carb : 50 mg/mL) avec une seule colonie de cellules BL21 Star transformées. Répétez cette opération pour au moins trois réplications biologiques. Incuber les cultures à 37 °C dans un incubateur/agitateur à 250 rpm pendant 22-24 h.
REMARQUE : Point de pause facultatif : Les cellules cultivées dans des milieux NI retiennent le plasmide et peuvent être conservées à 4 °C pendant 1 semaine. - Après croissance dans un milieu NI, diluer la culture 100x dans un nouveau 3 mL de média d’autoinduction (AI) ZYP-5052 contenant de la carbénicilline (Carb : 50 mg/mL). Faire pousser les cellules à 37 °C dans un incubateur/agitateur à 250 rpm pendant 16-18 h pour induire l’expression.
NOTE: Le OD600 typique pour les cultures variera de 2,0 à 3,3 après 18 heures de croissance. La plage de densité cellulaire optimale se situe entre 2,5 et 3,0.
- Inoculer une culture de 2 mL de milieux non inducteurs (NI) contenant de la carbénicilline (Carb : 50 mg/mL) avec une seule colonie de cellules BL21 Star transformées. Répétez cette opération pour au moins trois réplications biologiques. Incuber les cultures à 37 °C dans un incubateur/agitateur à 250 rpm pendant 22-24 h.
- Réalisation de l’expérience de cinétique cellulaire
- Configurez le cytomètre en flux.
- Allumez le cytomètre en flux et l’ordinateur connecté à l’instrument. Une fois connecté au logiciel pour le cytomètre en flux, sous l’onglet Instrument , cliquez sur l’icône Démarrage . Suivez les étapes indiquées sur l’écran du logiciel pour assurer une initialisation correcte de l’instrumentation.
REMARQUE: Certains cytomètres en flux appellent la séquence de démarrage de l’instrument Amorçage. Assurez-vous de suivre le protocole du fabricant pour le cytomètre en flux qui sera utilisé pour l’expérience. - Exécutez un test de performance (le cas échéant). Sous l’onglet Menu principal , cliquez sur Test de performance. Dans un tube de culture, ajouter trois gouttes de billes de suivi du rendement du fabricant dans 3 ml de liquide de focalisation.
- Placez le tube de culture dans le tube de levage du tube d’échantillon et soulevez-le. Avant de cliquer sur Exécuter le test de performance, assurez-vous que le # de lot du tube des perles de suivi est identique à ce qui est indiqué sur l’écran Configuration du test de performance. Cliquez sur Test de performances pour exécuter le test.
- Configurez le logiciel de cytomètre en flux pour cette expérience avec les paramètres d’acquisition suivants pour la fluorescence unicellulaire :
Laser d’excitation: 488 nm
Canal d’émission: GFP (également appelé FITC)
Volume d’acquisition : 40 μL (avec un volume total de tirage de 90 μL)
Débit: 200 μL / min
Nombre de cellules pour chaque mesure : 30 000
Paramètres de l’instrument :
Tension:
FSC : 480 V
SSC : 400 V
BL1 : 540 V
BL2 : 392 V
BL3 : 422 V
- Allumez le cytomètre en flux et l’ordinateur connecté à l’instrument. Une fois connecté au logiciel pour le cytomètre en flux, sous l’onglet Instrument , cliquez sur l’icône Démarrage . Suivez les étapes indiquées sur l’écran du logiciel pour assurer une initialisation correcte de l’instrumentation.
- Configurez les fichiers expérimentaux.
- Créez un nouveau fichier d’expérience dans l’onglet Explorateur d’expériences en cliquant avec le bouton droit sur le nom d’utilisateur du cytomètre en flux. Sélectionnez Nouvelle expérience dans la fenêtre déroulante. Lorsqu’une nouvelle fenêtre s’affiche sur l’écran de l’ordinateur, sélectionnez OK.
- Dans le nouveau fichier d’expérience, cliquez avec le bouton droit de la souris sur le dossier « Group » et sélectionnez Ajouter un nouveau tube d’échantillonnage. Étiquetez les tubes d’échantillon pour chaque point de temps spécifique et répliquez-les en cliquant avec le bouton droit de la souris sur Échantillon et en sélectionnant Renommer dans le menu déroulant. Répétez cette étape pour le nombre total de répétitions et les points temporels pour le cours prévu de l’étude.
- Préparer une solution cellulaire diluée : Dans un nouveau tube de culture, ajouter 1,5 mL de 1x solution de PBS. Ensuite, ajoutez 3 μL de cellules induites dans un milieu d’IA dans la solution PBS 1x pour obtenir une solution cellulaire diluée. Répétez cette étape pour chaque réplication biologique dans différents tubes de culture.
- Mesurer la fluorescence de fond des cellules: Avant d’ajouter un colorant, prenez des lectures de chaque tube de culture de réplication biologique contenant des cellules dans 1x solution de PBS. C’est ainsi que le fond fluorescent des cellules est mesuré pour observer le pli s’allumer au fil du temps une fois le colorant ajouté.
- Pour prendre une lecture, placez le tube de culture dans le tube de levage du tube d’échantillon et soulevez le tube de levage à la main jusqu’à l’aiguille d’injection de l’échantillon. Sélectionnez l’exemple de fichier approprié dans l’onglet Panneau de collecte , puis cliquez sur Enregistrer.
- Lorsque l’exécution est terminée, abaissez le tube de levage du tube d’échantillon avec le tube de culture à la main. Cela déclenchera une étape de rinçage qui rincera le système fluidique et minimisera le transfert entre chaque échantillon de réplication biologique. Les données seront automatiquement enregistrées sur l’ordinateur une fois l’exécution terminée.
- Répéter les étapes décrites au point 2.3.4 pour mesurer le fond fluorescent cellulaire pour au moins trois réplicats biologiques. Pour passer au fichier d’exemple suivant, sélectionnez le fichier d’exemple suivant en cliquant sur l’icône en forme de flèche droite près du nom du tube d’échantillon sous l’icône Enregistrement .
- Mesurer la fluorescence aux points temporels des cellules avec colorant.
- Ajouter 1,4 μL d’un stock de colorant concentré (50 mM DFHBI-1T dans DMSO) dans 1x solution PBS avec des cellules pour obtenir une concentration finale de 50 μM DFBHI-1T. Ensuite, fixez le couvercle du tube de culture, puis inversez les tubes de culture 3x-5x pour mélanger la solution uniformément avant de prendre la première lecture de point temporel.
REMARQUE : Le pourcentage total de DMSO dans les tubes de culture pour E. coli ne doit pas dépasser 10 %, car cela peut affecter la viabilité cellulaire16. - Retirez le couvercle et placez le tube de culture dans le tube de levage du tube à échantillon. Soulevez le support à la main jusqu’à l’aiguille d’injection de l’échantillon et, sous le fichier d’échantillon approprié, cliquez sur l’icône Enregistrer . En outre, démarrez une minuterie en appuyant sur Démarrer pour l’expérience.
- Abaissez le tube de levage à la main une fois la course terminée et répétez les étapes 2.3.5.1-2.3.5.2. (avec la minuterie en marche) en ajoutant 1,4 μL de DFHBI-1T concentré, en inversant les tubes de culture et en prenant des lectures pour toutes les répétitions biologiques restantes. Ces premiers enregistrements seront les lectures obtenues à 0 min pour toutes les répliques biologiques. Faites-le un à la fois pour chaque répétition biologique.
REMARQUE: Notez l’heure à laquelle l’acquisition de la cytométrie de flux d’enregistrement est pressée. Respectez ce temps en échelonnant pour les points temporels au cours de l’expérience. - Continuez à prendre des lectures en soulevant les tubes de culture dans le tube de levage du tube d’échantillon jusqu’à l’aiguille d’injection de l’échantillon, en sélectionnant le fichier d’échantillon approprié, en cliquant sur Enregistrer et en abaissant le leveur à la main après chaque passage. Faites-le pour tous les points temporels supplémentaires et les réplications biologiques testées. Répétez les étapes jusqu’à ce que l’expérience soit terminée.
REMARQUE: Gardez les échantillons à l’abri de la lumière pour éviter le photoblanchiment du DFHBI-1T en solution en recouvrant les échantillons de papier d’aluminium.
- Ajouter 1,4 μL d’un stock de colorant concentré (50 mM DFHBI-1T dans DMSO) dans 1x solution PBS avec des cellules pour obtenir une concentration finale de 50 μM DFBHI-1T. Ensuite, fixez le couvercle du tube de culture, puis inversez les tubes de culture 3x-5x pour mélanger la solution uniformément avant de prendre la première lecture de point temporel.
- Mesurer la fluorescence à des points temporels pour les cellules sans colorant (contrôle).
- Exécutez les protocoles de nettoyage appropriés pour le cytomètre en flux avant de répéter l’expérience avec des contrôles négatifs en suivant le protocole du fabricant. Ceci est fait pour minimiser tout transfert de l’expérience précédente dans l’expérience d’analyse du point de temps de contrôle. Voici les étapes suivies pour le cytomètre en flux entre les expériences:
- Placez un tube de culture vide dans le tube élévateur à la main, soulevez le porte-tube et cliquez sur l’icône Déboucher dans l’onglet Instrument . Cela exécutera un rinçage à contre-courant dans le système fluidique pour nettoyer les échantillons collants. Abaissez le support du tube à la main et retirez le tube une fois la séquence de débouchage terminée.
- Avec un nouveau tube de culture, ajouter 3 mL d’une solution d’eau de Javel à 10 %, placer le tube de culture dans le porte-tube et soulever le support à la main jusqu’à l’aiguille d’injection de l’échantillon. De plus, placez une plaque propre de 96 puits dans l’échantillonneur automatique, le cas échéant, sur le cytomètre en flux.
- Cliquez sur l’icône SIP Sanitize / Sanitize Autosampler SIP pour exécuter une séquence de nettoyage avec 10% d’eau de Javel dans tout le système fluidique. Abaissez le porte-tube pour terminer la séquence de nettoyage.
- Définissez les fichiers d’exemple pour l’analyse du point temporel de contrôle exécutée en suivant les instructions de l’étape 2.3.3.
- Dans un nouveau tube de culture, préparer une solution cellulaire diluée dans 1,5 mL de 1x solution PBS. Ajouter 3 μL de cellules induites dans les milieux d’IA dans la solution 1x PBS pour obtenir une solution cellulaire diluée. Répétez cette étape pour chaque répétition biologique.
- Ajoutez 1,4 μL de DMSO dans le tube de culture un à la fois à la solution 1x PBS avec des cellules et testez les mêmes points temporels. Fixez le couvercle du tube de culture, puis inversez les tubes de culture 3x-5x pour mélanger la solution uniformément avant de prendre la première lecture de point temporel. Faites-le un à la fois pour chaque répétition biologique.
- Suivez le même protocole pour l’analyse des cellules témoins que pour les cellules avec colorant, en utilisant les étapes 2.3.5.2-2.3.5.4.
- Exécutez les protocoles de nettoyage appropriés pour le cytomètre en flux avant de répéter l’expérience avec des contrôles négatifs en suivant le protocole du fabricant. Ceci est fait pour minimiser tout transfert de l’expérience précédente dans l’expérience d’analyse du point de temps de contrôle. Voici les étapes suivies pour le cytomètre en flux entre les expériences:
- Arrêtez le cytomètre en flux : Suivez le protocole du fabricant pour l’arrêt correct de l’instrumentation. Pour le cytomètre en flux, l’instrument est préparé pour l’arrêt de la manière suivante:
- Effectuer le protocole de nettoyage initial du cytomètre en flux en suivant les étapes 2.3.6.1.1-2.3.6.1.3.
- Remplacer le tube de culture par une solution d’eau de Javel à 10 % par un tube de culture contenant 3 mL de liquide de focalisation. Soulevez le porte-tube à la main et, sous l’icône Arrêter , cliquez sur le menu déroulant et sélectionnez Complet.
REMARQUE : Point de pause facultatif.
- Configurez le cytomètre en flux.
- Analyse des données de cytométrie en flux
- Exportez tous les fichiers FCS pour analyse. Ouvrez les fichiers FCS avec un logiciel d’analyse de cytométrie en flux.
- À l’aide de l’un des fichiers FCS Cell only, générez une porte à partir du diagramme de points à diffusion avant (FSC) et à diffusion latérale (SSC) (FSC-Area/SSC-Area), en utilisant les deux axes logarithmiques pour exclure tout signal des débris. Pour créer cette porte, cliquez sur l’icône AutoGate sur le logiciel d’analyse de cytométrie en flux et nommez-la Gate 1. Appliquez cette même porte à tous les échantillons testés sous l’onglet Tous les échantillons de l’espace de travail pour le traitement des données. Cela se traduira par le traitement de la porte 1 sous tous les fichiers FCS.
- Créez un nouveau fichier de sous-ensemble avec le fichier FCS Cellule uniquement utilisé à l’étape 2.4.2 en double-cliquant dessus. Modifiez les paramètres de l’axe en FSC-Area/FSC-Height, les deux utilisant des axes logarithmiques. Cliquez sur l’icône AutoGate sur le logiciel d’analyse de cytométrie en flux pour générer une porte ovale, en la nommant Gate 2. Appliquer cette porte en tant que sous-ensemble sous la porte définie à l’étape 2.4.2 à tous les échantillons testés. Ainsi, tous les échantillons auront la porte 1 > la porte 2 associées à chaque fichier FCS.
- Créez un autre fichier de sous-ensemble avec les deux portes définies à l’étape 2.4.2 et à l’étape 2.4.3 appliquées en double-cliquant sur Porte 2. Définissez les paramètres de l’axe sur FSC-Area/Histogram. Appliquez cette porte d’histogramme en tant que sous-ensemble à tous les échantillons testés, ce qui permet à tous les échantillons d’avoir la porte 1 > la porte 2 > la porte 2 associées à chaque fichier FCS.
REMARQUE: Les histogrammes peuvent être renommés de la porte 2 à l’histogramme pour aider à déplacer les histogrammes dans la fenêtre de mise en page, ainsi que pour créer plus d’organisation avec le traitement des données. - Pour analyser les valeurs d’intensité de fluorescence moyenne (MFI), ouvrez la fenêtre de mise en page. Cliquez et faites glisser les portes de l’histogramme pour chaque point temporel dans la fenêtre de mise en page.
- Effectuez une analyse statistique pour « ∑ Mean: BL1-A » (GFP) pour chaque échantillon testé afin d’afficher les résultats de l’IMF dans la fenêtre de mise en page.
- Calculer l’écart type pour les valeurs de l’IMF par analyse ponctuelle pour au moins trois réplicas biologiques indépendants.
- Enregistrez les histogrammes et les valeurs MFI pour chaque point temporel en exportant la fenêtre de mise en page sous forme de fichier PDF.
REMARQUE : Point de pause facultatif.
- Données de cytométrie en flux graphique
- Ouvrez le fichier PDF contenant les histogrammes et les valeurs MFI pour chaque point de temps. Les valeurs de l’IMF seront copiées dans un logiciel d’analyse de données. Dans un logiciel de graphiques de données, créez une nouvelle table de données au format XY.
- Sélectionnez cette option pour créer une table XY avec les éléments suivants :
Table de données : entrez ou importez des données dans une nouvelle table
Options:
X : Temps écoulés
Y : entrez (trois à quatre) répliquer les valeurs dans des sous-colonnes côte à côte - Étiquetez sur l’axe X tous les points temporels de l’expérience et du contrôle.
- Dans le groupe A, entrez les valeurs d’IMF pour toutes les réplications biologiques dans chaque point temporel pour l’analyse de fluorescence des cellules avec le colorant ajouté.
- Dans le groupe B, entrez les valeurs d’IMF pour toutes les réplications biologiques dans chaque point temporel pour l’analyse des cellules sans colorant (DMSO) ajouté.
- Pour observer les résultats, cliquez sur [Insérer le nom de l’ensemble de données] sous l’onglet Graphiques . Cela affichera les points de données sous forme de moyennes, avec des barres d’erreur représentant l’écart type (s.d.) à chaque point de temps. L’axe des X représente le temps écoulé et l’axe des Y représente les valeurs de l’IMF.
- Sélectionnez cette option pour créer une table XY avec les éléments suivants :
- Ouvrez le fichier PDF contenant les histogrammes et les valeurs MFI pour chaque point de temps. Les valeurs de l’IMF seront copiées dans un logiciel d’analyse de données. Dans un logiciel de graphiques de données, créez une nouvelle table de données au format XY.
Résultats
Cinétique in vitro
Les séquences des matrices et des amorces d’ADN, qui sont achetées comme oligonucléotides synthétiques, sont présentées dans le tableau 2, et les recettes de réactifs sont présentées dans le fichier supplémentaire 1. L’amplification par PCR est utilisée pour augmenter la quantité de matrice d’ADN avec le promoteur T7, ce qui est nécessaire pour la réaction ultérieure de transcription in vitro
Discussion
Pour l’expérience de cinétique in vitro, le même protocole général peut être modifié pour mesurer la cinétique in vitro d’un biocapteur fluorescent à base d’ARN contenant à la fois un domaine de liaison au ligand et un domaine de liaison au fluorophore 8. Dans ce cas, l’ARN doit être incubé avec le fluorophore avant les mesures lors de l’injection du ligand afin d’obtenir la cinétique de réponse du ligand. Si une grande variabilité est observée entre l...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par les subventions suivantes à MCH: NSF-BSF 1815508 et NIH R01 GM124589. MRM a été partiellement soutenu par la subvention de formation NIH T32 GM122740.
matériels
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
Références
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295 (2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139 (2016).
- Paige, J. S., Thinh, N. -. D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194 (2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002 (2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon