Method Article
Im lebenden Organismus Ganzhirnbildgebung von Zebrafischlarven mittels dreidimensionaler Fluoreszenzmikroskopie
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll für die in vivo Ganzhirnbildgebung von Zebrafischlarven mittels dreidimensionaler Fluoreszenzmikroskopie vorgestellt. Das experimentelle Verfahren umfasst die Probenvorbereitung, die Bildaufnahme und die Visualisierung.
Zusammenfassung
Als Modelltier von Wirbeltieren sind Zebrafischlarven in den Neurowissenschaften weit verbreitet und bieten eine einzigartige Möglichkeit, die Aktivität des gesamten Gehirns mit zellulärer Auflösung zu überwachen. Hier stellen wir ein optimiertes Protokoll für die Durchführung der Ganzhirnbildgebung von Zebrafischlarven mittels dreidimensionaler Fluoreszenzmikroskopie zur Verfügung, einschließlich Probenvorbereitung und -immobilisierung, Probeneinbettung, Bildaufnahme und Visualisierung nach der Bildgebung. Das aktuelle Protokoll ermöglicht die In-vivo-Bildgebung der Struktur und neuronalen Aktivität eines Zebrafischlarvengehirns mit zellulärer Auflösung über 1 h unter Verwendung von konfokaler Mikroskopie und maßgeschneiderter Fluoreszenzmikroskopie. Die kritischen Schritte des Protokolls werden ebenfalls erörtert, einschließlich der Probenmontage und -positionierung, der Verhinderung von Blasenbildung und Staub im Agarosegel und der Vermeidung von Bewegungen in Bildern, die durch eine unvollständige Verfestigung des Agarosegels und eine Lähmung der Fische verursacht werden. Das Protokoll wurde in mehreren Einstellungen validiert und bestätigt. Dieses Protokoll kann leicht für die Bildgebung anderer Organe einer Zebrafischlarve angepasst werden.
Einleitung
Der Zebrafisch (Danio rerio) hat sich aufgrund seiner optischen Transparenz im Larvenstadium, seiner schnellen Entwicklung, seiner geringen Wartungskosten und der Verfügbarkeit verschiedener genetischerWerkzeuge als Modelltier in den Neurowissenschaften durchgesetzt 1,2,3,4. Insbesondere die optische Transparenz von Larven in Kombination mit genetisch kodierten fluoreszierenden Reportern biologischer Ereignisse 5,6,7,8,9 bietet eine einzigartige Möglichkeit, sowohl neuronale Aktivität als auch Struktur auf der Ebene des gesamten Gehirns abzubilden 10,11,12,13,14 . Doch selbst mit einem Mikroskop, das zelluläre Auflösung unterstützt, behalten die aufgenommenen Bilder die Informationen nicht unbedingt auf Einzelzellebene bei. Die optische Bildqualität kann aufgrund der Aberration, die durch das für die Probenmontage verwendete Agarosegel verursacht wird, verschlechtert werden, der Fisch kann schräg montiert sein, so dass die interessierenden Bereiche nicht vollständig im Sichtfeld des Mikroskops enthalten sind, und die Fische können sich während der Aufnahme bewegen, was zu Bewegungsartefakten in den Bildern führt oder eine genaue Signalextraktion aus den Bildern behindert.
Daher wird ein effektives und reproduzierbares Protokoll benötigt, um qualitativ hochwertige Bilddaten mit minimalem Rauschen und Bewegungen zu erfassen. Leider beschreiben öffentlich zugängliche Protokolle für die Bildgebung eines ganzen Gehirns von Zebrafischlarven in vivo 15,16,17,18,19 das Verfahren nur kurz und überlassen wesentliche Teile der Details, wie z. B. die Agaroseerstarrung, präzise Montagetechniken und die Probenpositionierung mit einer Pinzette, jedem Experimentator. Darüber hinaus können Inkonsistenzen in den Agarosekonzentrations- und Immobilisierungsmethoden 10,11,14,15,16,17,18,19 zu Herausforderungen führen, die sich aus der Bewegung der Fische während des Bildgebungsprozesses ergeben.
Hier wird ein detailliertes Protokoll für die Ganzhirnbildgebung von Zebrafischlarven mittels dreidimensionaler Fluoreszenzmikroskopie zur Verfügung gestellt. Abbildung 1 gibt einen grafischen Überblick über das Protokoll: Probenvorbereitung und -immobilisierung, Probeneinbettung, Bildaufnahme und Visualisierung nach der Bildgebung. Die strukturelle und funktionelle Bildgebung des Zebrafischlarvengehirns in vivo wird mit einem kommerziellen konfokalen Mikroskop und einem speziell entwickelten Fluoreszenzmikroskop demonstriert. Dieses Protokoll kann von Praktikern für die Bildgebung des Gehirns mit bestimmten Sinnesreizen oder Verhaltenskontexten angepasst werden, abhängig von den experimentellen Anforderungen und dem Design.
Protokoll
Alle Zebrafischexperimente wurden vom Institutional Animal Care and Use Committee (IACUC) des KAIST genehmigt (KA2021-125). Insgesamt 12 adulte Zebrafische mit pan-neuronaler Expression des Calciumindikators GCaMP7a [Tg(huc:GAL4); Tg(UAS:GCaMP7a)] auf einem Casper [mitfa(w2/w2);mpv17(a9/a9)] Hintergrund wurden für die Zucht verwendet. Diese Gruppe bestand aus acht Weibchen und vier Männchen im Alter von 3 bis 12 Monaten. Bildgebende Experimente wurden an Zebrafischlarven 3-4 Tage nach der Befruchtung (d.p.f.) durchgeführt, einem Stadium, in dem ihr Geschlecht nicht bestimmt werden kann.
1. Vorbereitung der Zebrafischprobe
- Entnahme von Embryonen nach der Zucht erwachsener Fische einer gewünschten transgenen Linie, wie z. B. Tg(huc:GAL4); Tg(UAS:GCaMP7a)20,21,22, in einer mit Eiwasser gefüllten Petrischale (siehe Materialtabelle). Die Embryonen werden in einen Inkubator bei 28 °C gegeben und auf 3-4 d.p.f. Larven 23,24,25 aufgezogen.
- Wenn der Zebrafischhintergrund kein Albino ist, werden die Embryonen 24 Stunden nach der Befruchtung (h.p.f.) in die Petrischale übertragen, die mit Eiwasser gefüllt ist, das 200 μM 1-Phenyl-2-thioharnstoff enthält (PTU; siehe Materialtabelle)25,26. Den Fisch alle 24 Stunden in eine neue Schüssel mit frischem Eiwasser umfüllen, das 200 μM PTU enthält.
HINWEIS: Die Zebrafische werden unter Standardbedingungen bei 28 °C und einem Hell-Dunkel-Zyklus von 14:10 h gehalten. Es ist bekannt, dass die PTU-Behandlung das Verhalten und die Schilddrüsenfunktion von Zebrafischlarven beeinflusst27,28. Daher ist es wichtig, die PTU mit Vorsicht zu verwenden und bei allen Experimenten sorgfältig auf mögliche Störfaktoren zu achten.
- Wenn der Zebrafischhintergrund kein Albino ist, werden die Embryonen 24 Stunden nach der Befruchtung (h.p.f.) in die Petrischale übertragen, die mit Eiwasser gefüllt ist, das 200 μM 1-Phenyl-2-thioharnstoff enthält (PTU; siehe Materialtabelle)25,26. Den Fisch alle 24 Stunden in eine neue Schüssel mit frischem Eiwasser umfüllen, das 200 μM PTU enthält.
- Um die Probe zu finden, die fluoreszierende Proteine von Interesse exprimiert (z. B. panneuronales GCaMP7a), wird die Probe unter einem Epi-Fluoreszenzmikroskop untersucht und eine Probe mit einer hellen Expression ausgewählt.
- Bereiten Sie die ausgewählte 3-4 d.p.f. Zebrafischprobe in der mit Eiwasser gefüllten Petrischale vor (Abbildung 2A).
HINWEIS: Um die spontane neuronale Aktivität aufzuzeichnen, wird empfohlen, Proben zu verwenden, die zwischen 80 und 100 h.p.f. alt sind, da das Niveau der spontanen Aktivität vor 80 h.p.f. gering ist und die Pigmentierungsentwicklung, selbst bei einem Casper-Hintergrund, die Bildqualität nach 100 h.p.f. verschlechtern kann. - Bereiten Sie eine 0,25 mg/ml Pancuroniumbromidlösung 14,19,29,30,31 vor, indem Sie 1 ml einer 2,5 mg/ml Stammlösung (siehe Materialtabelle) zu 10 ml des Eiwassers hinzufügen. Aliquotieren Sie die Pancuroniumbromidlösung in 1,5-ml-Mikrozentrifugenröhrchen.
- Übertragen Sie die vorgesiebte Probe mit einer Transferpipette in die Petrischale.
Anmerkungen: Versuchen Sie, ein Mindestmaß an Eiwasser mit der Probe mitzunehmen. - 0,1 ml der Pancuroniumbromidlösung zur Lähmung in die Petrischale geben.
HINWEIS: Pancuroniumbromid hat eine potenziell dämpfende Wirkung auf die neuronale Aktivität von Zebrafischlarven32. Es ist wichtig, die Konzentration und Dauer der Exposition gegenüber Pancuroniumbromid sorgfältig zu berücksichtigen.
2. 2% (Gew./vol) Agarose-Gel-Zubereitung
- Schalten Sie einen Heizblock ein und stellen Sie die Zieltemperatur auf 37 °C ein. Warten Sie, bis sich das Gerät aufgeheizt und auf die eingestellte Temperatur eingependelt hat.
- Lösen Sie 0,2 g Agarosepulver mit niedrigem Schmelzpunkt (siehe Materialtabelle) in 10 ml Eiwasser auf.
- Erhitzen Sie die Agaroselösung in der Mikrowelle und mischen Sie sie durch Schütteln und Wirbeln, bis sich die Agarose vollständig aufgelöst hat.
- Aliquotieren Sie das Agarose-Gel in 1,5-ml-Mikrozentrifugenröhrchen und lagern Sie die Mikrozentrifugenröhrchen auf dem Heizblock (Abbildung 2B).
Anmerkungen: Überprüfen Sie, ob die Blasen im Agarose-Gel verschwunden sind.
3. Probenmontage und -positionierung
- Überprüfen Sie die Probe unter einem Stereomikroskop, um sicherzustellen, dass die Larvenbewegung gestoppt wurde, und um den Gesundheitszustand der Probe visuell zu beurteilen, indem Sie ihren Herzschlag überprüfen (Abbildung 2C). Wenn der Herzschlag zu langsam ist, verwerfen Sie die Probe.
HINWEIS: Wenn der Herzschlag des abgebildeten Fisches zu langsam ist (z. B. unter 60 Schlägen pro Minute), ist es möglicherweise nicht möglich, eine Langzeitbildgebung durchzuführen. Um das Wohlbefinden des Fisches zu beurteilen, kann die Herzfrequenz visuell überprüft werden, indem sie mit anderen Fischen in derselben Petrischale verglichen wird. Dadurch wird sichergestellt, dass der abgebildete Fisch gesund und stabil genug für das Bildgebungsverfahren ist. - Geben Sie mit der Transferpipette eine einzelne Zebrafischlarve in das Agarosegel im 1,5-ml-Mikrozentrifugenröhrchen (Abbildung 2D).
Anmerkungen: Stellen Sie sicher, dass die Pipette nach dem Transfer der Probe entsorgt wird. - Gießen Sie das Agarose-Gel in die Petrischale, um eine 1-2 mm dicke Schicht zu erhalten. Überführen Sie die Probe in den Mikrozentrifugenröhrchen mit der Transferpipette in die Petrischale, so dass die Larve in die Mitte der Schale gelegt wird.
Anmerkungen: Wenn sich Staub und Blasen im Agarosegel befinden, entfernen Sie diese mit der Transferpipette. - Verwenden Sie eine Pinzette, um die Probe in der gewünschten Ausrichtung so zu positionieren, dass Kopf und Schwanz flach sind (Abbildung 2E).
- Drehen Sie die Probe mit einer Pinzette, so dass beide Augen waagerecht sind (Abbildung 2F).
Anmerkungen: Die Ausrichtungsvorgänge (Position und Rotation) sollten abgeschlossen sein, bevor das Agarosegel zu erstarren beginnt. - Warten Sie nach dem Alignment, bis sich das Agarosegel verfestigt hat (Abbildung 2G,H).
Anmerkungen: Die Wartezeit bis zum Erstarren des Agarosegels kann je nach Volumen und Größe des Gels zwischen 5 und 10 Minuten liegen. - Füllen Sie nach dem Erstarren des Agarosegels die Petrischale mit Eiwasser und stellen Sie die Petrischale mit der eingebetteten Probe auf den Mikroskoptisch (Abbildung 2I).
4. Bildaufnahme
- Schalten Sie das Mikroskopsystem ein (z. B. Laser, konfokale Controller, Mikroskop und Computer; siehe Materialtabelle) und überprüfen Sie, ob das gesamte System funktioniert.
- Wählen Sie ein Objektiv mit geringer Vergrößerung und lokalisieren Sie die Probe in der Mitte des Sichtfeldes.
- Wählen Sie ein Objektiv zum Eintauchen in Wasser oder zum Tauchen mit der richtigen Vergrößerung (z. B. 16-faches Objektiv mit numerischer Apertur (NA) mit 16-facher 0,8-facher numerischer Apertur (NA); siehe Materialtabelle). Nehmen Sie Feineinstellungen am Sichtfeld vor.
- Stellen Sie die Bildgebungsparameter (z. B. Bildgröße, Laserleistung, Belichtungszeit, Anzahl der Bilder) mit einer Bilderfassungssoftware ein.
HINWEIS: Stellen Sie die Bildgebungsparameter so ein, dass die bestmöglichen Ergebnisse für bestimmte Anforderungen erzielt werden (siehe die Einstellungen für die Bildaufnahme im Abschnitt Repräsentative Ergebnisse). Wenn das Bild gesättigt ist, reduzieren Sie die Laserleistung. - Finden Sie das Gehirn der Probe, indem Sie den Tisch bewegen, und bestimmen Sie seine Dicke im Live-View-Modus in der Software, indem Sie die Fokusebenen manuell nach oben und unten ändern. Legen Sie die untere und obere Grenze der Lautstärke fest.
Anmerkungen: Stellen Sie sicher, dass sich das gesamte Gehirn sowohl in lateraler als auch in axialer Richtung im Sichtfeld befindet. - Stellen Sie eine Z-Schrittweite unter Berücksichtigung der axialen Auflösung des Mikroskops ein.
HINWEIS: Die optimale Z-Schrittweite für die Bildgebung des Zebrafischlarvengehirns hängt von der Bildgebungsmodalität und der Auflösung des Mikroskops ab. Als Beispiel wurde eine z-Schrittweite von 5 μm unter Berücksichtigung der Dicke des Lichtblattes und des mittleren Durchmessers der Zellkörper10 verwendet. - Fahren Sie mit der Bildaufnahme für das eingestellte Sichtfeld fort.
- Für die volumetrische Strukturbildgebung erfassen Sie ein 3D-Bild (x,y,z) des gesamten Gehirns, indem Sie die Fokusebenen ändern und nacheinander 2D-Bilder jeder z-Ebene erhalten.
- Für die funktionelle Bildgebung einer einzelnen z-Ebene nehmen Sie Zeitreihenbilder (x,y,t) der neuronalen Aktivität des Gehirns in einer bestimmten Tiefe auf.
- Für die volumetrische funktionelle Bildgebung wird ein 4-D-Bild (x,y,z,t) der neuronalen Aktivität im gesamten Gehirn aufgenommen, indem 3D-Bilder sequenziell aufgenommen werden.
HINWEIS: Legen Sie die Anzahl der Frames unter Berücksichtigung der verfügbaren Speichergröße des Computers fest. Eine Erfassungszeit von weniger als 1 h wird aufgrund der Wirkungsdauer von Pancuroniumbromid empfohlen.
- Speichern Sie nach der Aufnahme der Bilder die Ergebnisse und zeichnen Sie die Bildparameter (z. B. Pixelgröße, Z-Schrittweite, Bildrate, Laserleistung) für die Bildanalyse auf.
- Exportieren Sie die Bilder in einem geeigneten Format für das Rendern und die Bildanalyse.
HINWEIS: Es wird empfohlen, Bilder im TIF-Format (Tagged Image File) zu exportieren. TIF unterstützt verlustfreie Bildkomprimierung und ist mit den meisten Bildverarbeitungsprogrammen und Programmiersprachen kompatibel. Darüber hinaus unterstützt das TIF-Format die Einbeziehung von Metadaten wie Erfassungsparametern, Bildauflösung und anderen relevanten Informationen, die bei der Dateninterpretation und Reproduzierbarkeit helfen können.
5. Einrichtung für Visualisierungen mit napari
HINWEIS: napari ist ein mehrdimensionaler Open-Source-Bildbetrachter in einer Python-Umgebung mit GPU-basiertem Rendering33. Das napari-animation Plugin bietet eine programmatische Erstellung von Filmen. Die Verwendung von Fidschi, einem Open-Source-Bildverarbeitungsprogramm, wird für die allgemeine Bildverarbeitung empfohlen, z. B. für Filterung und geometrische Transformation (siehe Materialtabelle). Der Quellcode, der für die Visualisierung mit napari verwendet wird, ist auf GitHub (https://github.com/NICALab/Zebrafish-brain-visualization) verfügbar.
- Installieren Sie napari und napari-animation entweder pip oder conda. Erstellen Sie nach der Installation eine neue Jupyter-Notebook-Datei.
Hinweis: Die Ausführung mit jupyter notebook, einem interaktiven Tool für Python, wird gegenüber dem Python-Skript empfohlen. - Importieren Sie napari und napari-animation.
6. Visualisierung von Strukturen mit napari
- Um Bilder zu visualisieren und Filme des gerenderten Zebrafischgehirns zu erstellen, laden Sie das 3D-Bild (x,y,z) und öffnen Sie das Napari-Fenster. Schließen Sie das napari-animation-Plug-in an (Abbildung 3A).
- Legen Sie Parameter wie Voxelgröße, Colormap und Kontrastgrenzen in den Ebenensteuerelementen fest.
- Passen Sie die Viewer-Einstellungen (z. B. Perspektive, Winkel) auf der Arbeitsfläche an.
- Um das gerenderte Bild aufzunehmen, drücken Sie die Aufnahmetaste im Animationsassistenten.
- Um Filme des gerenderten Volumes zu generieren, ändern Sie die Viewer-Einstellungen und fügen Sie Keyframes hinzu (Abbildung 3B).
- Legen Sie nach dem Hinzufügen von Keyframes die Anzahl der Frames (Schritte) zwischen Keyframes im Animationsassistenten fest. Speichern Sie die gerenderte Animation.
7. Bildverarbeitung und Visualisierung neuronaler Aktivität mit Napari
HINWEIS : Um die Zeitreihenbilder der neuronalen Aktivität als überlagerte Bilder eines statischen Hintergrunds und einer Aktivität zu visualisieren, muss ein Zerlegungsalgorithmus auf die Rohbilder angewendet werden. Verwenden Sie eine MATLAB-Implementierung eines Zerlegungsalgorithmus namens BEAR24. Die MATLAB-Version von BEAR ist auf GitHub (https://github.com/NICALab/BEAR) verfügbar.
- Um den statischen Hintergrund und die neuronale Aktivität zu zerlegen, wenden Sie BEAR auf die rohen Zeitreihenbilder (x,y,t oder x,y,z,t) an. Speichern Sie nach der Zerlegung die Bilder des Hintergrunds und der neuronalen Aktivität als TIF-Dateien (Abbildung 3C).
- Laden Sie die Hintergrund- und neuronalen Aktivitätsbilder und öffnen Sie das Napari-Fenster. Verbinden Sie das napari-animation Plugin.
- Verwenden Sie eine graue Colormap für Hintergrundbilder und eine Hot-Colormap für neuronale Aktivitätsbilder (Abbildung 3C).
- Legen Sie Parameter wie Deckkraft- und Kontrastgrenzen in den Ebenensteuerelementen fest.
- Um Filme mit neuronaler Aktivität zu erstellen, ändern Sie die Viewer-Einstellungen und fügen Sie Keyframes hinzu, um die Animation zu rendern.
- Legen Sie nach dem Hinzufügen von Keyframes die Anzahl der Frames (Schritte) zwischen Keyframes im Animationsassistenten fest. Speichern Sie die gerenderte Animation.
Ergebnisse
Die Struktur und neuronale Aktivität der Zebrafischlarven, die den Calciumindikator pan-neuronal GCaMP7a (Tg(huc:GAL4); Tg(UAS:GCaMP7a))20,21,22 mit einem Casper (mitfa(w2/w2);mpv17(a9/a9))34 Hintergrund wurde bei 3-4 d.p.f. unter Befolgung des beschriebenen Protokolls abgebildet.
Für die volumetrische Strukturbildgebung wurde die Probe mit einem handelsüblichen konfokalen Point-Scanning-Mikroskopiesystem abgebildet, das mit einer 16x 0,8 NA Wassertauchobjektivlinse ausgestattet war. Ein 488 nm Anregungslaser wurde sowohl für die strukturelle als auch für die funktionelle Bildgebung verwendet. Die Bildrate, die Bildauflösung, die Pixelgröße und die axiale Schrittweite betrugen 0,25 Hz, 2048 x 2048, 0,34 μm bzw. 1,225 μm. Die Bildaufnahme dauerte ca. 1 h und 20 min. Das volumetrische Gesichtsfeld des aufgenommenen Bildes umfasste die Hirnregionen des Vorderhirns, des Mittelhirns und des Hinterhirns (Abbildung 4A). Neuronale Zellkörper der Medulla oblongata, der Kleinhirnplatte, des optischen Tectums, der Habenula und des dorsalen Telencephalons im gesamten Gehirn von 4 d.p.f. Zebrafischlarven waren in den konfokalen Mikroskopiebildern deutlich sichtbar (Abbildung 4B). Das 3-D-Rendering der konfokalen Mikroskopiebilder wurde unter Verwendung von Napari27 unter Befolgung des oben genannten Protokolls durchgeführt (Abbildung 4C und Video 1).
Für die funktionelle Bildgebung in 2-D wurde die Probe mit dem gleichen konfokalen Mikroskopiesystem abgebildet, das mit einer 16x 0,8 NA Wassertauchobjektivlinse ausgestattet war. Die Bildrate, die Bildauflösung und die Pixelgröße betrugen 2 Hz, 512 x 256 bzw. 1,5 μm. Die neuronalen Zellkörper waren sowohl im Hintergrund als auch in der überlagerten neuronalen Aktivität deutlich zu erkennen (Abbildung 5A,B).
Für die funktionelle 3-D-Bildgebung wurde ein speziell entwickeltes 3-D-Mikroskopiesystem18 verwendet, das in der Lage war, die neuronale Aktivität eines gesamten Zebrafischgehirns mit einem Sichtfeld von 1.040 μm × 400 μm × 235 μm und lateralen und axialen Auflösungen von 1,7 μm bzw. 5,4 μm in vivo abzubilden. Die Bildrate betrug bis zu 4,2 Volumen pro Sekunde. Ähnlich wie bei der Darstellung der strukturellen Bildgebungsdaten wurde napari für das 3D-Rendering der Kalzium-Bildgebungsdaten des gesamten Gehirns verwendet (Abbildung 5C und Video 2).
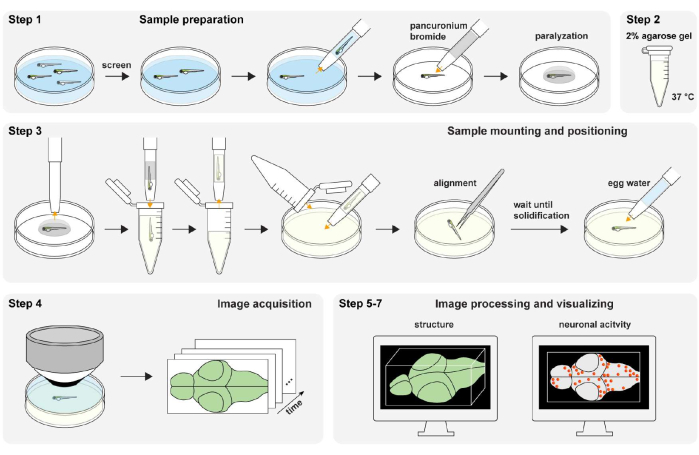
Abbildung 1: Überblick über den Versuchsablauf. Vorbereitung und Lähmung von Zebrafischproben (Schritt 1). Die 2%ige (wt/vol) Agarosegelzubereitung (Schritt 2). Probenmontage und -positionierung (Schritt 3). Bildaufnahme (Schritt 4). Bildverarbeitung und Visualisierung von Struktur und neuronaler Aktivität (Schritt 5-7). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Experimentelles Verfahren zur Präparation der Ganzhirnbildgebung . (A) Gescreente Zebrafischprobe, die panneuronales GCaMP7a exprimiert, in einer mit Eiwasser gefüllten Petrischale. (B) Ausrüstung und Material, die für die Probenmontage und -positionierung erforderlich sind. (1) Heizblock bei 37 °C; (2) 2 Gew.-% (vol) Agarosegel; (3) 1,5-ml-Mikrozentrifugenröhrchen; (4) Ei, Wasser; (5) 0,25 mg/ml Pancuroniumbromidlösung; (6) Petrischale; (7) Pinzetten; (8) Transferpipette. (C) Stereomikroskopische Aufnahme der gelähmten Probe. Der schwarze Pfeil zeigt auf das Herz der Probe. (D) Die Probe im 1,5-ml-Mikrozentrifugenröhrchen wird mit einer Pipette übertragen. Der Ausschnitt zeigt eine vergrößerte Ansicht des geschachtelten Bereichs. (E) Die Probe (schwarzer Pfeil) wird mit einer Pinzette in die Mitte der Petrischale gelegt. (F) Die Probe wird mit einer Pinzette ausgerichtet. (G) Ein Beispiel für eine falsch ausgerichtete eingebettete Probe. (H) Ein Beispiel für eine ausgerichtete eingebettete Probe. (I) Die Probe wird zur Bildaufnahme auf einen Mikroskoptisch unter die Objektivlinse gelegt. Maßstabsleiste: 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
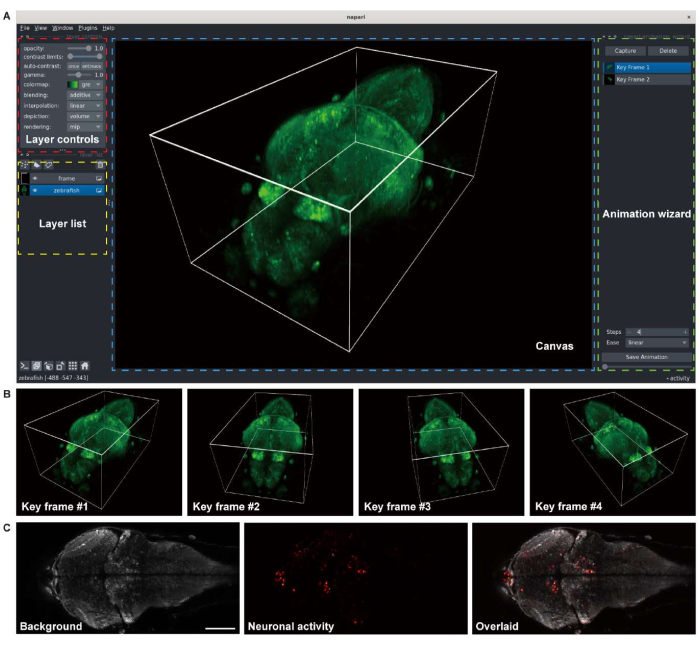
Abbildung 3: Visualisierung von Gehirnbildern von Zebrafischlarven . (A) Das 3-D-Rendering eines konfokalen Mikroskopiebildes eines Zebrafischlarven (4 d.p.f), das pan-neuronales GCaMP7a exprimiert. Für das Rendering wurde napari verwendet, ein quelloffener mehrdimensionaler Bildbetrachter in einer Python-Umgebung. Das Napari-Fenster umfasst Ebenensteuerelemente (rotes Feld), eine Ebenenliste (gelbes Feld), eine Arbeitsfläche (blaues Feld) und einen Animationsassistenten (grünes Feld). (B) Keyframes mit mehreren Viewer-Einstellungen werden zum Rendern einer Animation hinzugefügt. (C) Konfokalmikroskopische Aufnahme einer 2-D-Zeitrafferaufnahme der neuronalen Aktivität im Gehirn der Zebrafischlarve. Das Bild wird in den Hintergrund (links) und die neuronale Aktivität (Mitte) zerlegt und dann überlagert (rechts). Maßstabsleiste: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
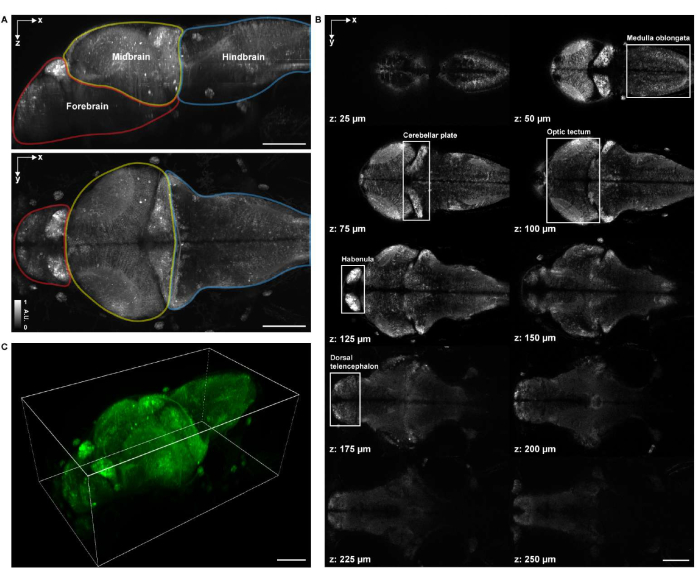
Abbildung 4: Bildgebende Struktur eines Zebrafischlarvengehirns. (A) Maximale Intensitätsprojektion (MIP) eines konfokalen Mikroskopiebildes eines Zebrafischlarven (4 d.p.f.), das pan-neuronales GCaMP7a exprimiert. Oben: seitlicher MIP. Unten: axial MIP. Jede Grenze enthält das Vorderhirn (rot), das Mittelhirn (gelb) und das Hinterhirn (blau). (B) Insgesamt 10 axiale Schnitte in mehreren Tiefen aus einem volumetrischen Bild des Gehirns (bei z = 25 μm, 50 μm, 75 μm, 100 μm, 125 μm, 150 μm, 175 μm, 200 μm, 225 μm, 250 μm, von oben nach unten gezählt; z = 0 μm bezeichnet die Oberseite des Gehirns). Jedes weiße Kästchen repräsentiert die Region des Gehirns (Medulla oblongata, Kleinhirnplatte, optisches Tectum, Habenula und dorsales Telencephalon). (C) Das ganze Gehirn wird mit Napari gerendert. Maßstabsleiste: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Abbildung 5: Bildgebung der neuronalen Aktivität einer Zebrafischlarve. (A) Konfokalmikroskopische Aufnahme der neuronalen Aktivität in einer Zebrafischlarve (4 d.p.f.), die pan-neuronales GCaMP7a exprimiert. Maßstabsleiste: 100 μm. (B) Vergrößerte Ansicht des gerahmten Bereichs in A, die die neuronale Aktivität im optischen Tectum zu mehreren Zeitpunkten zeigt. Maßstabsleiste: 20 μm. (C) Die 3-D-Darstellung der neuronalen Aktivität des gesamten Gehirns im Gehirn der Zebrafischlarve, aufgenommen mit einem speziell entwickelten Mikroskop (links: t = 133 s; rechts: t = 901 s). Die neuronale Aktivität wird dem statischen Hintergrund überlagert. Maßstabsleiste: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
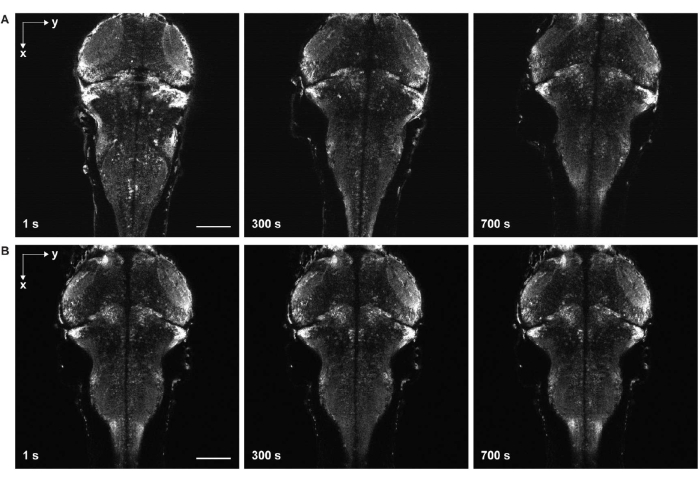
Abbildung 6: Zeitraffer-Bildgebung mit und ohne Probendrift. (A) Zeitraffer-Bildgebung des zebrafischlarven Gehirns, das pan-neuronales GCaMP7a exprimiert, mit Probendrift. Der Probe wurde vor der Erstarrung des Agarosegels Eiwasser zugesetzt (Schritt 3.6-3.7). Die Fische bewegten sich in lateraler und axialer Richtung. (B) Zeitrafferaufnahmen eines Zebrafischlarvengehirns ohne Probendrift. Der Probe wurde nach der Erstarrung des Agarosegels Eiwasser zugesetzt (Schritt 3.6-3.7). Maßstabsleiste: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Video 1: 3-D-Darstellung der Gesamthirnstruktur einer Zebrafischlarve (4 d.p.f.), die panneuronales GCaMP7a exprimiert. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Video 2: 3-D-Darstellung der neuronalen Aktivität des gesamten Gehirns in einer Zebrafischlarve (4 d.p.f.), die pan-neuronales GCaMP7a exprimiert. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
Das aktuelle Protokoll ermöglicht die in vivo Ganzhirnbildgebung von Zebrafischlarven über einen längeren Zeitraum (z.B. länger als 1 h) und die Visualisierung der erfassten strukturellen und funktionellen Bildgebungsdaten.
Die wichtigsten Schritte sind die Probenmontage und -positionierung. Bei der Einbettung der Probe ist es entscheidend, die Blasenbildung zu verhindern und Staub im Agarosegel zu vermeiden. Wenn das Gel Luftblasen und Staub enthält, kann die Bildqualität stark beeinträchtigt werden. Bei der Positionierung der Probe mit einer Pinzette ist darauf zu achten, dass die Probe sowohl horizontal als auch vertikal waagerecht ist. Andernfalls sind die interessierenden Bereiche möglicherweise nicht im Sichtfeld des Mikroskops enthalten. Darüber hinaus sollte diese Positionierung innerhalb eines kurzen Zeitfensters vor der Verfestigung des Agarosegels erfolgen, um Schäden an der Oberfläche zu vermeiden, da diese die Bildqualität beeinträchtigen.
Eine weitere große Herausforderung besteht darin, Bewegungen in Bildern zu vermeiden, die durch eine unvollständige Verfestigung des Agarosegels (Abbildung 6) und eine Lähmung des Zebrafisches entstehen. Wenn der Probe Eiwasser zugesetzt wird, bevor das Agorsegel vollständig erstarrt ist, bewegen sich die Fische langsam sowohl seitlich als auch axial, was sich in den Zeitreihenbildern als Probendrift manifestiert (Abbildung 6A). Wenn die Dosis der Lähmungsmittel nicht ausreicht oder die Wirkungsdauer endet, kann die Probe versuchen, sich zu bewegen, was in den Zeitrafferbildern als schnelles Zucken erscheint.
Trotz der Wichtigkeit der oben genannten Schritte für die Aufnahme qualitativ hochwertiger Bilder ohne Bewegungsartefakte bieten die öffentlich zugänglichen Protokolle 15,16,17,18,19 nur einen kurzen Überblick über das experimentelle Verfahren, da diese Details fehlen. Zum Beispiel leiden die aufgenommenen Bilder ohne Immobilisierungsprotokolle11 unter erheblichen Bewegungsartefakten, die die nachgeschaltete Bildanalyse zu einer Herausforderung machen. Durch die Integration wesentlicher Komponenten, wie z. B. der Verfestigung von Agarosegel und der Probenlähmung, verbessert unser Protokoll die Konsistenz in der Qualität der aufgenommenen Bilder erheblich und minimiert gleichzeitig Bewegungsartefakte.
Zusammenfassend wird ein optimiertes und reproduzierbares experimentelles Verfahren zur in vivo Bildgebung des zebrafischlarven Gehirns beschrieben. Die Validität und Reproduzierbarkeit dieses Protokolls für die In-vivo-Bildgebung der Hirnaktivität und -struktur wurde in mehreren Settings bestätigt 14,18,29,30,31. Der vorliegende Arbeitsablauf konzentriert sich auf die Bildgebung des gesamten Gehirns von Zebrafischlarven, kann aber leicht auf die Bildgebung anderer Organe von Zebrafischlarven angewendet werden35,36.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Die Zebrafischlinien, die für die Kalziumbildgebung verwendet werden, wurden vom Zebrafish Center for Disease Modeling (ZCDM), Korea, zur Verfügung gestellt. Diese Forschung wurde von der National Research Foundation of Korea unterstützt (2020R1C1C1009869, NRF2021R1A4A102159411, RS-2023-00209473).
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | SciLab | SL.Tub3513 | To aliquot agarose gel and pancuronium bromide solution |
| 15 mL Falcon tubes | Falcon | 352096 | To prepare agarose gel and pancuronium bromide solution |
| 16× 0.8NA water dipping objective lens | Nikon | CFI75 LWD 16×W | Objective lens for whole-brain imaging |
| 1-phenyl 2-thiourea (PTU) | Sigma-Aldrich | P7629-10G | 200 μM of 1-phenyl 2-thiourea (PTU) |
| 50 mL Falcon tubes | Falcon | 352070 | To prepare egg water |
| Disposable transfer pipette | SciLab | SL.Pip3032 | To transfer zebrafish larvae |
| Egg water | N/A | N/A | 0.6 g sea salt in 10 L deionized water |
| Forceps | Karl Hammacher GmbH | HSO 010-10 | Forceps used for sample positioning |
| Low melting point agarose | Thermo Scientific | R0801 | 2% (wt/vol) agarose gel |
| Napari | Napari | N/A | To visualize microscopy images in 3-D |
| NIS-Elements C | Nikon | N/A | Imaging software for confocal microscope |
| Pancuronium bromide | Sigma-Aldrich | P1918-10MG | 0.25 mg/mL of pancuronium bromide solution |
| Petri dish, 35 mm | SPL Life Sciences | 11035 | Petri dish used for sample embedding |
| Petri dish, 55 mm | SPL Life Sciences | 11050 | To prepare zebrafish larvae after screening |
| Point-scanning confocal microscopy system (C2 Plus) | Nikon | N/A | Confocal microscope for whole-brain imaging |
| Sea salt | Sigma-Aldrich | S9883-500G | Sea salt used for preparing egg water |
Referenzen
- Choi, T. -. Y., Choi, T. -. I., Lee, Y. -. R., Choe, S. -. K., Kim, C. -. H. Zebrafish as an animal model for biomedical research. Experimental & Molecular Medicine. 53 (3), 310-317 (2021).
- Ahrens, M. B., et al. Brain-wide neuronal dynamics during motor adaptation in zebrafish. Nature. 485 (7399), 471-477 (2012).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Frontiers in Neural Circuits. 7, 65 (2013).
- Bianco, I. H., Kampff, A. R., Engert, F. Prey capture behavior evoked by simple visual stimuli in larval zebrafish. Frontiers in Systems Neuroscience. 5, 101 (2011).
- Akerboom, J., et al. Optimization of a GCaMP calcium indicator for neural activity imaging. Journal of Neuroscience. 32 (40), 13819-13840 (2012).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Piatkevich, K. D., et al. A robotic multidimensional directed evolution approach applied to fluorescent voltage reporters. Nature Chemical Biology. 14 (4), 352-360 (2018).
- Tian, L., et al. Imaging neural activity in worms, flies and mice with improved GCaMP calcium indicators. Nature Methods. 6 (12), 875-881 (2009).
- Looger, L., et al. Fast and sensitive GCaMP calcium indicators for imaging neural populations. Biorxiv. , (2021).
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nature Methods. 10 (5), 413-420 (2013).
- Prevedel, R., et al. Simultaneous whole-animal 3D imaging of neuronal activity using light-field microscopy. Nature Methods. 11 (7), 727-730 (2014).
- Chhetri, R. K., et al. Whole-animal functional and developmental imaging with isotropic spatial resolution. Nature Methods. 12 (12), 1171-1178 (2015).
- Ahrens, M. B., Engert, F. Large-scale imaging in small brains. Current Opinion in Neurobiology. 32, 78-86 (2015).
- Yoon, Y. -. G., et al. Sparse decomposition light-field microscopy for high speed imaging of neuronal activity. Optica. 7 (10), 1457-1468 (2020).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nature Methods. 12 (11), 1039-1046 (2015).
- Cong, L., et al. Rapid whole brain imaging of neural activity in freely behaving larval zebrafish (Danio rerio). eLife. 6, e28158 (2017).
- Bruzzone, M., et al. Whole brain functional recordings at cellular resolution in zebrafish larvae with 3D scanning multiphoton microscopy. Scientific Reports. 11 (1), 11048 (2021).
- Cho, E. -. S., Han, S., Lee, K. -. H., Kim, C. -. H., Yoon, Y. -. G. 3DM: deep decomposition and deconvolution microscopy for rapid neural activity imaging. Optics Express. 29 (20), 32700-32711 (2021).
- Zhang, Z., et al. Imaging volumetric dynamics at high speed in mouse and zebrafish brain with confocal light field microscopy. NatureBiotechnology. 39 (1), 74-83 (2021).
- Muto, A., Ohkura, M., Abe, G., Nakai, J., Kawakami, K. Real-time visualization of neuronal activity during perception. Current Biology. 23 (4), 307-311 (2013).
- Köster, R. W., Fraser, S. E. Tracing transgene expression in living zebrafish embryos. Developmental Biology. 233 (2), 329-346 (2001).
- Park, H. C., et al. Analysis of upstream elements in the HuC promoter leads to the establishment of transgenic zebrafish with fluorescent neurons. Developmental Biology. 227 (2), 279-293 (2000).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: an introduction. Journal of Visualized Experiments. (69), e4196 (2012).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). 4th edition. , (2000).
- Morsch, M., et al. Triggering cell stress and death using conventional UV laser confocal microscopy. Journal of Visualized Experiments. (120), e54983 (2017).
- Antinucci, P., Hindges, R. A crystal-clear zebrafish for in vivo imaging. Scientific Reports. 6, 29490 (2016).
- Parker, M. O., Brock, A. J., Millington, M. E., Brennan, C. H. Behavioral phenotyping of casper mutant and 1-pheny-2-thiourea treated adult zebrafish. Zebrafish. 10 (4), 466-471 (2013).
- Elsalini, O. A., Rohr, K. B. Phenylthiourea disrupts thyroid function in developing zebrafish. Development Genes and Evolution. 212 (12), 593-598 (2003).
- Han, S., Cho, E. -. S., Park, I., Shin, K., Yoon, Y. -. G. Efficient neural network approximation of robust PCA for automated analysis of calcium imaging data. Medical Image Computing and Computer Assisted Intervention. , 595-604 (2021).
- Shin, C., et al. Three-dimensional fluorescence microscopy through virtual refocusing using a recursive light propagation network. Medical Image Analysis. 82, 102600 (2022).
- Eom, M., et al. Statistically unbiased prediction enables accurate denoising of voltage imaging data. bioRxiv. , (2022).
- Johnston, L., et al. Electrophysiological recording in the brain of intact adult zebrafish. Journal of Visualized Experiments. (81), e51065 (2013).
- Sofroniew, N., et al. napari: a multi-dimensional image viewer for Python. Zenodo. , 3555620 (2022).
- White, R. M., et al. Transparent adult zebrafish as a tool for in vivo transplantation analysis. Cell Stem Cell. 2 (2), 183-189 (2008).
- Mickoleit, M., et al. High-resolution reconstruction of the beating zebrafish heart. Nature Methods. 11 (9), 919-922 (2014).
- Voleti, V., et al. Real-time volumetric microscopy of in vivo dynamics and large-scale samples with SCAPE 2.0. Nature Methods. 16 (10), 1054-1062 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten