Method Article
In vivo Imágenes de todo el cerebro de larvas de pez cebra usando microscopía de fluorescencia tridimensional
En este artículo
Resumen
Aquí se presenta un protocolo para obtener imágenes in vivo de todo el cerebro de larvas de pez cebra utilizando microscopía de fluorescencia tridimensional. El procedimiento experimental incluye la preparación de muestras, la adquisición de imágenes y la visualización.
Resumen
Como animal modelo vertebrado, las larvas de pez cebra son ampliamente utilizadas en neurociencia y brindan una oportunidad única para monitorear la actividad de todo el cerebro a la resolución celular. Aquí, proporcionamos un protocolo optimizado para realizar imágenes de todo el cerebro de larvas de pez cebra utilizando microscopía de fluorescencia tridimensional, incluida la preparación e inmovilización de muestras, la incrustación de muestras, la adquisición de imágenes y la visualización después de la obtención de imágenes. El protocolo actual permite obtener imágenes in vivo de la estructura y la actividad neuronal de un cerebro larval de pez cebra a una resolución celular durante más de 1 h utilizando microscopía confocal y microscopía de fluorescencia diseñada a medida. También se discuten los pasos críticos en el protocolo, incluido el montaje y posicionamiento de la muestra, la prevención de la formación de burbujas y el polvo en el gel de agarosa, y evitar el movimiento en las imágenes causado por la solidificación incompleta del gel de agarosa y la paralización de los peces. El protocolo ha sido validado y confirmado en múltiples configuraciones. Este protocolo se puede adaptar fácilmente para obtener imágenes de otros órganos de una larva de pez cebra.
Introducción
El pez cebra (Danio rerio) ha sido ampliamente adoptado como animal vertebrado modelo en neurociencia, debido a su transparencia óptica en la etapa larvaria, su rápido desarrollo, su bajo costo de mantenimiento y la disponibilidad de diversas herramientas genéticas 1,2,3,4. En particular, la transparencia óptica de las larvas combinada con reporteros fluorescentes codificados genéticamente de eventos biológicos 5,6,7,8,9 proporciona una oportunidad única para obtener imágenes tanto de la actividad neuronal como de la estructura a nivel de todo el cerebro 10,11,12,13,14 . Sin embargo, incluso con un microscopio que admita la resolución celular, las imágenes adquiridas no necesariamente retienen la información a nivel de una sola célula; La calidad óptica de la imagen puede degradarse debido a la aberración introducida por el gel de agarosa utilizado para el montaje de muestras, los peces pueden montarse en ángulo, por lo que las regiones de interés no están completamente contenidas dentro del campo de visión del microscopio, y los peces pueden moverse durante la grabación, causando artefactos de movimiento en las imágenes o impidiendo la extracción precisa de señales de las imágenes.
Por lo tanto, se necesita un protocolo efectivo y reproducible para adquirir datos de imagen de alta calidad con un mínimo de ruido y movimiento. Desafortunadamente, los protocolos disponibles públicamente para obtener imágenes de un cerebro completo de larvas de pez cebra in vivo 15,16,17,18,19 solo describen brevemente el procedimiento, dejando partes sustanciales de los detalles, como la solidificación de la agarosa, las técnicas de montaje precisas y el posicionamiento de la muestra con fórceps, hasta cada experimentador. Además, las inconsistencias en la concentración de agarosa y los métodos de inmovilización 10,11,14,15,16,17,18,19 pueden conducir a desafíos derivados del movimiento de los peces durante el proceso de obtención de imágenes.
Aquí, se proporciona un protocolo detallado para obtener imágenes de todo el cerebro de larvas de pez cebra utilizando microscopía de fluorescencia tridimensional. La figura 1 proporciona una descripción gráfica del protocolo: preparación e inmovilización de muestras, incrustación de muestras, adquisición de imágenes y visualización después de la obtención de imágenes. Las imágenes estructurales y funcionales del cerebro larval del pez cebra in vivo se demuestran utilizando un microscopio confocal comercial y un microscopio de fluorescencia diseñado a medida. Este protocolo puede ser adaptado por profesionales para imágenes cerebrales con ciertos estímulos sensoriales o contextos de comportamiento dependiendo de las necesidades experimentales y el diseño.
Protocolo
Todos los experimentos con pez cebra fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) de KAIST (KA2021-125). Un total de 12 peces cebra adultos con expresión panneuronal del indicador de calcio GCaMP7a [Tg(huc:GAL4); Tg(UAS:GCaMP7a)] en un fondo de casper [mitfa(w2/w2);mpv17(a9/a9)] para la reproducción. Este grupo consistía en ocho mujeres y cuatro hombres, con edades que variaban de 3 a 12 meses. Se realizaron experimentos de imagen en larvas de pez cebra a los 3-4 días posteriores a la fertilización (d.p.f.), una etapa durante la cual no se puede determinar su sexo.
1. Preparación de muestras de pez cebra
- Recolectar embriones después de criar peces adultos de una línea transgénica deseada, como Tg(huc:GAL4); Tg(UAS:GCaMP7a)20,21,22, en una placa de Petri llena de agua de huevo (ver Tabla de Materiales). Colocar los embriones en una incubadora a 28 °C y elevarlos a 3-4 d.p.f. larvas23,24,25.
- Si el fondo del pez cebra no es albino, para inhibir la formación de pigmentación, transfiera los embriones a las 24 h después de la fertilización (h.p.f.) a la placa de Petri llena de agua de huevo que contiene 200 μM de 1-fenil 2-tiourea (PTU; ver Tabla de materiales)25,26. Cada 24 h, transfiera el pescado a un plato nuevo con agua de huevo fresca que contenga 200 μM de PTU.
NOTA: El pez cebra se mantiene en condiciones estándar a 28 °C y un ciclo de luz:oscuridad de 14:10 h. Se sabe que el tratamiento de la PTU afecta el comportamiento y la función tiroidea de larvas de pez cebra27,28. Por lo tanto, es importante usar la PTU con precaución y controlar cuidadosamente los posibles factores de confusión en cualquier experimento.
- Si el fondo del pez cebra no es albino, para inhibir la formación de pigmentación, transfiera los embriones a las 24 h después de la fertilización (h.p.f.) a la placa de Petri llena de agua de huevo que contiene 200 μM de 1-fenil 2-tiourea (PTU; ver Tabla de materiales)25,26. Cada 24 h, transfiera el pescado a un plato nuevo con agua de huevo fresca que contenga 200 μM de PTU.
- Para encontrar la muestra que expresa proteínas fluorescentes de interés (por ejemplo, GCaMP7a panneuronal), examine la muestra bajo un microscopio de epifluorescencia y seleccione una muestra con una expresión brillante.
- Prepare la muestra seleccionada de pez cebra de 3-4 d.p.f. en la placa de Petri llena de agua de huevo (Figura 2A).
NOTA: Para registrar la actividad neuronal espontánea, se recomienda utilizar muestras de entre 80-100 h.p.f., ya que el nivel de actividad espontánea es bajo antes de 80 h.p.f. y el desarrollo de pigmentación, incluso con un fondo de casper, puede degradar la calidad de imagen después de 100 h.p.f. - Prepare una solución de bromuro de pancuronio de 0,25 mg/ml 14,19,29,30,31 agregando 1 ml de una solución madre de 2,5 mg/ml (ver Tabla de materiales) a 10 ml de agua de huevo. Alícuota la solución de bromuro de pancuronio en tubos de microcentrífuga de 1,5 ml.
- Transfiera la muestra preseleccionada a la placa de Petri utilizando una pipeta de transferencia.
NOTA: Trate de llevar un volumen mínimo de agua de huevo con la muestra. - Transfiera 0,1 ml de la solución de bromuro de pancuronio a la placa de Petri para la parálisis.
NOTA: El bromuro de pancuronio tiene un efecto amortiguador potencial sobre la actividad neuronal en larvas de pez cebra32. Es esencial considerar cuidadosamente la concentración y la duración de la exposición al bromuro de pancuronio.
2. 2% (peso / vol) preparación de gel de agarosa
- Encienda un bloque de calor y ajuste la temperatura objetivo a 37 °C. Espere a que la unidad se caliente y se equilibre a la temperatura establecida.
- Disuelva 0,2 g de polvo de agarosa de bajo punto de fusión (consulte la Tabla de materiales) en 10 ml de agua de huevo.
- Caliente la solución de agarosa en un microondas y mézclela agitando y vórtice hasta que la agarosa se disuelva por completo.
- Alícuota el gel de agarosa en tubos de microcentrífuga de 1,5 ml y almacene los tubos de microcentrífuga en el bloque térmico (Figura 2B).
NOTA: Compruebe si las burbujas en el gel de agarosa han desaparecido.
3. Montaje y posicionamiento de la muestra
- Verifique la muestra bajo un microscopio estereoscópico para verificar que el movimiento de las larvas se haya detenido y para evaluar visualmente la salud de la muestra comprobando sus latidos cardíacos (Figura 2C). Si el latido del corazón es demasiado lento, deseche la muestra.
NOTA: Si el latido del corazón de los peces que se están fotografiando es demasiado lento (por ejemplo, por debajo de 60 latidos por minuto), es posible que no sea posible realizar imágenes a largo plazo. Para evaluar el bienestar de los peces, la frecuencia cardíaca se puede verificar visualmente comparándola con otros peces en la misma placa de Petri. Esto ayudará a garantizar que el pez que se está fotografiando esté lo suficientemente sano y estable para el procedimiento de imagen. - Usando la pipeta de transferencia, coloque una sola larva de pez cebra en el gel de agarosa en el tubo de microcentrífuga de 1,5 ml (Figura 2D).
NOTA: Asegúrese de desechar la pipeta después de transferir la muestra. - Vierta el gel de agarosa en la placa de Petri para hacer una capa de 1-2 mm. Transfiera la muestra en los tubos de microcentrífuga a la placa de Petri utilizando la pipeta de transferencia, de modo que la larva se coloque en el centro de la placa.
NOTA: Si hay polvo y burbujas en el gel de agarosa, retírelos con la pipeta de transferencia. - Utilice fórceps para colocar la muestra en la orientación deseada de modo que la cabeza y la cola queden planas (Figura 2E).
- Gire la muestra con fórceps para que ambos ojos estén nivelados (Figura 2F).
NOTA: Los procedimientos de alineación (posición y rotación) deben completarse antes de que el gel de agarosa comience a solidificarse. - Después de la alineación, espere hasta que el gel de agarosa se haya solidificado (Figura 2G, H).
NOTA: El tiempo de espera para que el gel de agarosa se solidifique puede variar de 5 a 10 minutos, dependiendo del volumen y tamaño del gel. - Después de la solidificación del gel de agarosa, llene la placa de Petri con agua de huevo y coloque la placa de Petri con la muestra incrustada en la etapa del microscopio (Figura 2I).
4. Adquisición de imágenes
- Encienda el sistema de microscopio (por ejemplo, láseres, controladores confocales, microscopio y computadora; consulte la Tabla de materiales) y verifique que todo el sistema esté funcionando.
- Seleccione una lente objetiva de bajo aumento y ubique la muestra en el centro del campo de visión.
- Seleccione una lente objetivo de inmersión en agua o de inmersión en agua con el aumento adecuado (por ejemplo, lente de inmersión en agua de 16x 0.8 de apertura numérica (NA); consulte la Tabla de materiales). Haga ajustes finos en el campo de visión.
- Establezca los parámetros de imagen (por ejemplo, tamaño de imagen, potencia del láser, tiempo de exposición, número de fotogramas) utilizando el software de adquisición de imágenes.
NOTA: Establezca los parámetros de imagen para lograr los mejores resultados posibles para necesidades específicas (consulte la configuración para la adquisición de imágenes en la sección de resultados representativos). Si la imagen está saturada, reduzca la potencia del láser. - Encuentra el cerebro de la muestra moviendo el escenario y determina su grosor usando el modo de visualización en vivo en el software cambiando los planos focales manualmente hacia arriba y hacia abajo. Establezca los límites inferior y superior del volumen.
NOTA: Asegúrese de que todo el cerebro esté contenido en el campo de visión a lo largo de las direcciones lateral y axial. - Establezca un tamaño de paso z teniendo en cuenta la resolución axial del microscopio.
NOTA: El tamaño óptimo del paso z para obtener imágenes del cerebro larval del pez cebra depende de la modalidad de imagen y la resolución del microscopio. Como ejemplo, se utilizó un tamaño de paso z de 5 μm considerando el grosor de la lámina de luz y el diámetro promedio de los cuerpos celulares10. - Continúe con la adquisición de imágenes para el campo de visión establecido.
- Para imágenes estructurales volumétricas, adquiera una imagen 3D (x, y, z) de todo el cerebro cambiando los planos focales y obteniendo imágenes 2D de cada plano z secuencialmente.
- Para obtener imágenes funcionales de un solo plano z, adquiera imágenes de series temporales (x, y, t) de la actividad neuronal del cerebro a cierta profundidad.
- Para imágenes funcionales volumétricas, adquiera una imagen 4-D (x,y,z,t) de la actividad neuronal en todo el cerebro mediante la obtención de imágenes 3-D secuencialmente.
Nota : establezca el número de fotogramas teniendo en cuenta el tamaño de memoria disponible del equipo. Se recomienda un tiempo de adquisición de menos de 1 h debido a la duración del efecto del bromuro de pancuronio.
- Después de adquirir imágenes, guarde los resultados y registre los parámetros de imagen (por ejemplo, tamaño de píxel, tamaño de paso z, velocidad de fotogramas, potencia láser) para el análisis de imágenes.
- Exporte las imágenes en un formato apropiado para la representación de datos y el análisis de imágenes.
NOTA: Se recomienda exportar imágenes en formato de archivo de imagen etiquetado (TIF). TIF admite la compresión de imágenes sin pérdida y es compatible con la mayoría de los programas de procesamiento de imágenes y lenguajes de programación. Además, el formato TIF admite la inclusión de metadatos, como parámetros de adquisición, resolución de imagen y otra información relevante que puede ayudar en la interpretación y reproducibilidad de los datos.
5. Configuración para visualizaciones usando napari
NOTA: napari es un visor de imágenes multidimensional de código abierto en un entorno Python con renderizado basado en la unidad de procesamiento de gráficos (GPU)33. El plugin napari-animation proporciona una creación programática de películas. Se recomienda el uso de Fiji, un programa de procesamiento de imágenes de código abierto, para el procesamiento de imágenes de propósito general, como el filtrado y la transformación geométrica (consulte la Tabla de materiales). El código fuente utilizado para la visualización usando napari está disponible en GitHub (https://github.com/NICALab/Zebrafish-brain-visualization).
- Instale napari y napari-animation usando pip o conda. Después de la instalación, cree un nuevo archivo de bloc de notas de Jupyter.
NOTA: Se recomienda ejecutar con Jupyter Notebook, que es una herramienta interactiva para Python, sobre el script de Python. - Importar napari y napari-animación.
6. Visualización de estructuras usando napari
- Para visualizar imágenes y crear películas del cerebro renderizado del pez cebra, cargue la imagen 3D (x, y, z) y abra la ventana napari. Conecte el plugin napari-animation (Figura 3A).
- Defina parámetros como el tamaño del vóxel, el mapa de colores y los límites de contraste en los controles de capa.
- Ajuste la configuración del visor (por ejemplo, perspectiva, ángulos) en el lienzo.
- Para capturar la imagen renderizada, pulse el botón de captura en el asistente de animación.
- Para generar películas del volumen renderizado, modifique la configuración del visor y agregue fotogramas clave (Figura 3B).
- Después de agregar fotogramas clave, establezca el número de fotogramas (pasos) entre fotogramas clave en el asistente para animación. Guarde la animación renderizada.
7. Procesamiento de imágenes y visualización de la actividad neuronal usando napari
NOTA: Para visualizar las imágenes de series temporales de actividad neuronal como imágenes superpuestas de un fondo estático y actividad, se debe aplicar un algoritmo de descomposición a las imágenes sin procesar. Utilice una implementación de MATLAB de un algoritmo de descomposición llamado BEAR24. La versión de BEAR de MATLAB está disponible en GitHub (https://github.com/NICALab/BEAR).
- Para descomponer el fondo estático y la actividad neuronal, aplique BEAR a las imágenes de series temporales sin procesar (x, y, t o x, y, z, t). Después de la descomposición, guarde las imágenes del fondo y la actividad neuronal como archivos TIF (Figura 3C).
- Cargue las imágenes de fondo y actividad neuronal y abra la ventana napari. Conecte el plugin napari-animation.
- Utilice un mapa de colores gris para las imágenes de fondo y un mapa de colores calientes para las imágenes de actividad neuronal (Figura 3C).
- Defina parámetros como límites de opacidad y contraste en los controles de capa.
- Para generar películas de actividad neuronal, modifique la configuración del visor y agregue fotogramas clave para renderizar la animación.
- Después de agregar fotogramas clave, establezca el número de fotogramas (pasos) entre fotogramas clave en el asistente para animación. Guarde la animación renderizada.
Resultados
La estructura y la actividad neuronal de los cerebros larvales del pez cebra que expresan el indicador de calcio panneuronal GCaMP7a (Tg(huc:GAL4); Tg(UAS:GCaMP7a))20,21,22 con un fondo de casper (mitfa(w2/w2);mpv17(a9/a9))34 fue fotografiado a 3-4 d.p.f. siguiendo el protocolo descrito.
Para las imágenes estructurales volumétricas, se obtuvo una imagen de la muestra utilizando un sistema de microscopía confocal de barrido puntual comercial equipado con una lente objetivo de inmersión en agua de 16x 0.8 NA. Se utilizó un láser de excitación de 488 nm para imágenes estructurales y funcionales. La velocidad de fotogramas, la resolución de imagen, el tamaño de píxel y el tamaño de paso axial fueron 0,25 Hz, 2048 x 2048, 0,34 μm y 1,225 μm, respectivamente. La adquisición de la imagen tomó aproximadamente 1 h y 20 min. El campo de visión volumétrico de la imagen adquirida cubrió las regiones cerebrales del cerebro anterior, mesencéfalo y cerebro posterior (Figura 4A). Los cuerpos celulares neuronales del bulbo raquídeo, la placa cerebelosa, el tectum óptico, la habénula y el telencéfalo dorsal en todo el cerebro de 4 d.p.f. larva de pez cebra fueron claramente visibles en las imágenes de microscopía confocal (Figura 4B). La representación 3D de las imágenes de microscopía confocal se realizó utilizando napari27 siguiendo el protocolo mencionado anteriormente (Figura 4C y Video 1).
Para imágenes funcionales en 2-D, la muestra se obtuvo una imagen utilizando el mismo sistema de microscopía confocal, equipado con una lente objetivo de inmersión en agua de 16x 0.8 NA. La velocidad de fotogramas, la resolución de imagen y el tamaño de píxel fueron 2 Hz, 512 x 256 y 1,5 μm, respectivamente. Los cuerpos celulares neuronales eran claramente visibles tanto en el fondo como en la actividad neuronal superpuesta (Figura 5A, B).
Para la obtención de imágenes funcionales 3D, se utilizó un sistema de microscopía 3D18 diseñado a medida, que era capaz de obtener imágenes in vivo de la actividad neuronal de un cerebro de pez cebra larval completo con un campo de visión de 1.040 μm × 400 μm × 235 μm y resoluciones laterales y axiales de 1,7 μm y 5,4 μm, respectivamente. La velocidad de imagen fue de hasta 4,2 volúmenes por segundo. De manera similar a la representación de los datos de imágenes estructurales, napari se utilizó para la representación en 3D de los datos de imágenes de calcio de todo el cerebro (Figura 5C y Video 2).
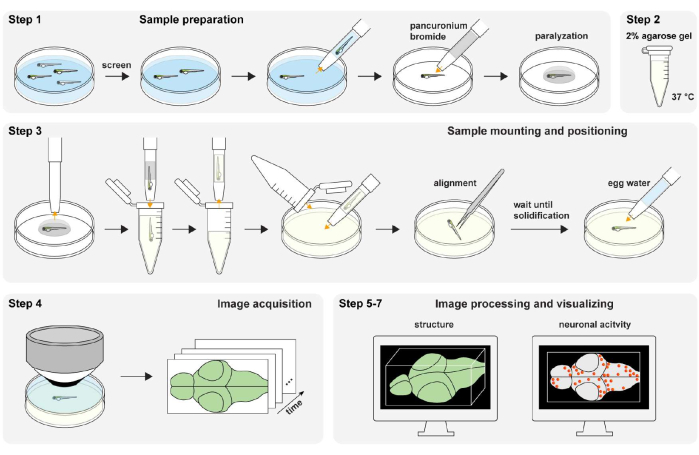
Figura 1: Visión general del procedimiento experimental. Preparación y paralización de muestras de pez cebra (paso 1). La preparación de gel de agarosa al 2% (peso / vol) (paso 2). Montaje y posicionamiento de la muestra (paso 3). Adquisición de imágenes (paso 4). Procesamiento de imágenes y visualización de la estructura y la actividad neuronal (paso 5-7). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Procedimiento experimental para preparar imágenes de todo el cerebro . (A) Muestra de pez cebra examinada que expresa GCaMP7a panneuronal en una placa de Petri llena de agua de huevo. (B) Equipo y material requerido para el montaje y posicionamiento de muestras. (1) Bloque de calor a 37 °C; (2) 2% (peso / vol) gel de agarosa; (3) tubo de microcentrífuga de 1,5 ml; (4) agua de huevo; (5) solución de bromuro de pancuronio de 0,25 mg/ml; (6) Placa de Petri; (7) fórceps; (8) Pipeta de transferencia. (C) Imagen de estereomicroscopio de la muestra paralizada. La flecha negra apunta al corazón de la muestra. (D) La muestra en el tubo de microcentrífuga de 1,5 ml se transfiere utilizando una pipeta. El recuadro muestra una vista ampliada del área en caja. (E) La muestra (flecha negra) se coloca en el centro de la placa de Petri usando fórceps. (F) La muestra se alinea usando fórceps. (G) Un ejemplo de una muestra incrustada desalineada. (H) Un ejemplo de una muestra incrustada alineada. (I) La muestra se coloca en una plataforma de microscopio debajo de la lente del objetivo para la adquisición de imágenes. Barra de escala: 500 μm. Haga clic aquí para ver una versión más grande de esta figura.
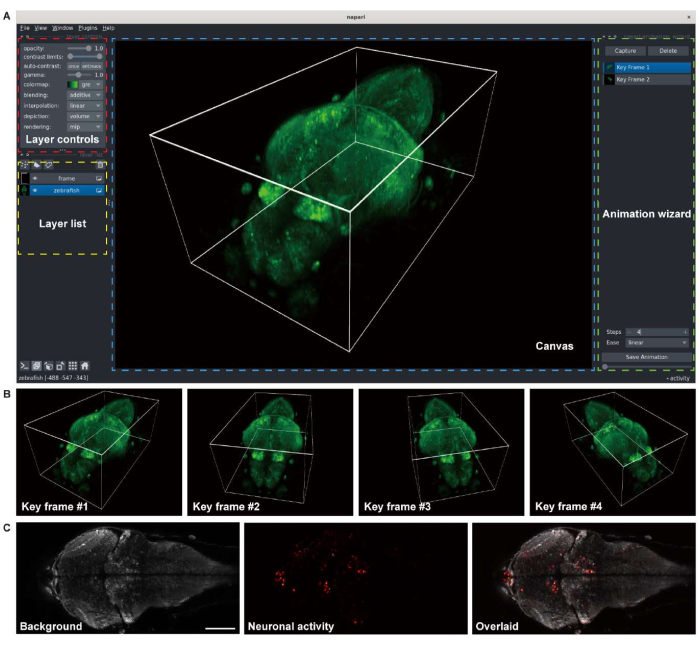
Figura 3: Visualización de imágenes cerebrales larvales de pez cebra. (A) La representación 3D de una imagen de microscopía confocal de un cerebro larval de pez cebra (4 d.p.f) que expresa GCaMP7a panneuronal. napari, un visor de imágenes multidimensionales de código abierto en un entorno Python, se utilizó para la representación. La ventana napari comprende controles de capa (cuadro rojo), una lista de capas (cuadro amarillo), un lienzo (cuadro azul) y un asistente de animación (cuadro verde). (B) Se agregan fotogramas clave con múltiples configuraciones de visor para renderizar una animación. (C) Imagen de microscopía confocal de imágenes de lapso de tiempo 2-D de la actividad neuronal en el cerebro larval del pez cebra. La imagen se descompone en el fondo (izquierda) y la actividad neuronal (centro), luego se superpone (derecha). Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
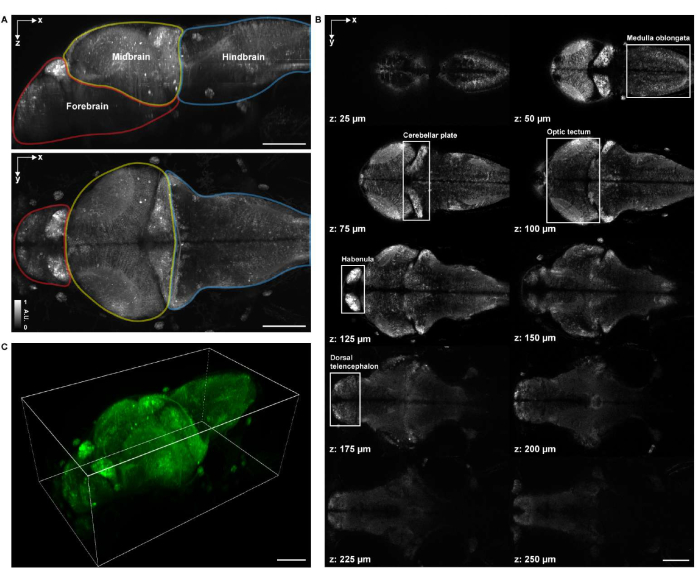
Figura 4: Estructura de imágenes del cerebro de una larva de pez cebra. (A) Proyección de intensidad máxima (MIP) de una imagen de microscopía confocal de un pez cebra larval (4 d.p.f.) cerebral que expresa GCaMP7a panneuronal. Arriba: MIP lateral. Abajo: MIP axial. Cada límite contiene el prosencéfalo (rojo), el mesencéfalo (amarillo) y el cerebro posterior (azul). (B) Un total de 10 cortes axiales a múltiples profundidades de una imagen volumétrica del cerebro (en z = 25 μm, 50 μm, 75 μm, 100 μm, 125 μm, 150 μm, 175 μm, 200 μm, 225 μm, 250 μm, contados hacia arriba de arriba a abajo; z = 0 μm indica la superficie superior del cerebro). Cada caja blanca representa la región del cerebro (bulbo raquídeo, placa cerebelosa, tectum óptico, habénula y telencéfalo dorsal). (C) Todo el cerebro renderizado usando napari. Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
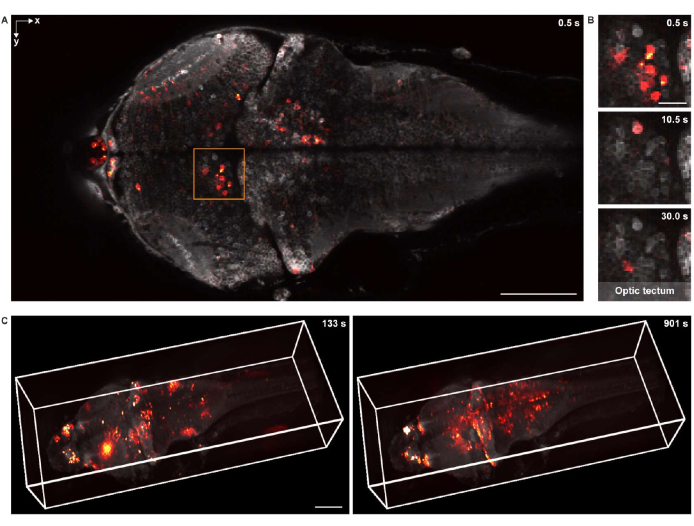
Figura 5: Obtención de imágenes de la actividad neuronal de un cerebro larval de pez cebra. (A) Imagen de microscopía confocal de la actividad neuronal en un cerebro larval de pez cebra (4 d.p.f.) que expresa GCaMP7a panneuronal. Barra de escala: 100 μm. (B) Vista ampliada del área en caja en A que muestra la actividad neuronal en el tectum óptico en múltiples puntos de tiempo. Barra de escala: 20 μm. (C) La representación 3D de la actividad neuronal de todo el cerebro en el cerebro larval del pez cebra adquirida usando un microscopio diseñado a medida (izquierda: t = 133 s; derecha: t = 901 s). La actividad neuronal se superpone al fondo estático. Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
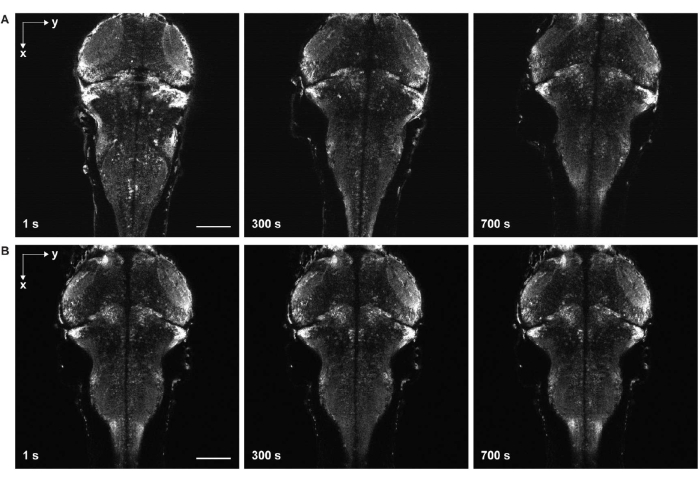
Figura 6: Imágenes de lapso de tiempo con y sin deriva de la muestra. (A) Imágenes de lapso de tiempo del cerebro larval de pez cebra que expresa GCaMP7a panneuronal con deriva de muestra. Se añadió agua de huevo a la muestra antes de la solidificación del gel de agarosa (paso 3.6-3.7). Los peces se movían en las direcciones lateral y axial. (B) Imágenes de lapso de tiempo de un cerebro larval de pez cebra sin deriva de muestra. Se añadió agua de huevo a la muestra después de la solidificación del gel de agarosa (paso 3.6-3.7). Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
Video 1: Representación en 3D de la estructura cerebral completa de un cerebro larval de pez cebra (4 d.p.f.) que expresa GCaMP7a panneuronal. Haga clic aquí para descargar este video.
Video 2: Representación en 3D de la actividad neuronal de todo el cerebro en un cerebro larval de pez cebra (4 d.p.f.) que expresa GCaMP7a panneuronal. Haga clic aquí para descargar este video.
Discusión
El protocolo actual permite obtener imágenes in vivo de todo el cerebro de larvas de pez cebra durante un período prolongado (por ejemplo, más de 1 h) y visualizaciones de los datos de imágenes estructurales y funcionales adquiridos.
Los pasos más críticos son el montaje y posicionamiento de muestras. Durante la incrustación de muestras, es crucial prevenir la formación de burbujas y evitar el polvo en el gel de agarosa. Si el gel contiene burbujas de aire y polvo, la calidad de la imagen puede verse gravemente degradada. Al colocar la muestra con fórceps, es importante asegurarse de que la muestra esté nivelada tanto horizontal como verticalmente. De lo contrario, las regiones de interés pueden no estar contenidas dentro del campo de visión del microscopio. Además, este posicionamiento debe realizarse dentro de un corto período de tiempo antes de la solidificación del gel de agarosa para evitar causar daños a su superficie, ya que dicho daño compromete la calidad de la imagen.
Otro desafío importante es evitar el movimiento en las imágenes que provienen de la solidificación incompleta del gel de agarosa (Figura 6) y la paralización del pez cebra. Cuando se agrega agua de huevo a la muestra antes de la solidificación completa del gel de agorse, los peces se mueven lentamente tanto lateral como axialmente, manifestándose como deriva de la muestra en las imágenes de series temporales (Figura 6A). Si la dosis de paralíticos es insuficiente o la duración del efecto termina, la muestra puede intentar moverse, lo que aparece como contracción rápida en las imágenes de lapso de tiempo.
A pesar de la importancia de los pasos antes mencionados para adquirir imágenes de alta calidad sin artefactos de movimiento, los protocolos disponibles públicamente 15,16,17,18,19 proporcionan solo una breve descripción del procedimiento experimental, careciendo de estos detalles. Por ejemplo, las imágenes adquiridas sin protocolos de inmovilización11 sufren de artefactos de movimiento sustanciales que dificultan el análisis de imágenes aguas abajo. Al integrar componentes esenciales, como la solidificación en gel de agarosa y la paralización de muestras, nuestro protocolo mejora significativamente la consistencia en la calidad de las imágenes adquiridas al tiempo que minimiza los artefactos de movimiento.
En resumen, se describe un procedimiento experimental optimizado y reproducible para la obtención de imágenes in vivo del cerebro larval del pez cebra. La validez y reproducibilidad de este protocolo para la obtención de imágenes in vivo de la actividad y estructura cerebral han sido confirmadas en múltiples entornos 14,18,29,30,31. El presente flujo de trabajo se centró en la obtención de imágenes de todo el cerebro de larvas de pez cebra, pero se puede aplicar fácilmente a la obtención de imágenes de otros órganos de larvas de pez cebra35,36.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Las líneas de pez cebra utilizadas para obtener imágenes de calcio fueron proporcionadas por el Centro de Modelado de Enfermedades de Pez Cebra (ZCDM), Corea. Esta investigación fue apoyada por la Fundación Nacional de Investigación de Corea (2020R1C1C1009869, NRF2021R1A4A102159411, RS-2023-00209473).
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | SciLab | SL.Tub3513 | To aliquot agarose gel and pancuronium bromide solution |
| 15 mL Falcon tubes | Falcon | 352096 | To prepare agarose gel and pancuronium bromide solution |
| 16× 0.8NA water dipping objective lens | Nikon | CFI75 LWD 16×W | Objective lens for whole-brain imaging |
| 1-phenyl 2-thiourea (PTU) | Sigma-Aldrich | P7629-10G | 200 μM of 1-phenyl 2-thiourea (PTU) |
| 50 mL Falcon tubes | Falcon | 352070 | To prepare egg water |
| Disposable transfer pipette | SciLab | SL.Pip3032 | To transfer zebrafish larvae |
| Egg water | N/A | N/A | 0.6 g sea salt in 10 L deionized water |
| Forceps | Karl Hammacher GmbH | HSO 010-10 | Forceps used for sample positioning |
| Low melting point agarose | Thermo Scientific | R0801 | 2% (wt/vol) agarose gel |
| Napari | Napari | N/A | To visualize microscopy images in 3-D |
| NIS-Elements C | Nikon | N/A | Imaging software for confocal microscope |
| Pancuronium bromide | Sigma-Aldrich | P1918-10MG | 0.25 mg/mL of pancuronium bromide solution |
| Petri dish, 35 mm | SPL Life Sciences | 11035 | Petri dish used for sample embedding |
| Petri dish, 55 mm | SPL Life Sciences | 11050 | To prepare zebrafish larvae after screening |
| Point-scanning confocal microscopy system (C2 Plus) | Nikon | N/A | Confocal microscope for whole-brain imaging |
| Sea salt | Sigma-Aldrich | S9883-500G | Sea salt used for preparing egg water |
Referencias
- Choi, T. -Y., Choi, T. -I., Lee, Y. -R., Choe, S. -K., Kim, C. -H. Zebrafish as an animal model for biomedical research. Experimental & Molecular Medicine. 53 (3), 310-317 (2021).
- Ahrens, M. B., et al. Brain-wide neuronal dynamics during motor adaptation in zebrafish. Nature. 485 (7399), 471-477 (2012).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Frontiers in Neural Circuits. 7, 65(2013).
- Bianco, I. H., Kampff, A. R., Engert, F. Prey capture behavior evoked by simple visual stimuli in larval zebrafish. Frontiers in Systems Neuroscience. 5, 101(2011).
- Akerboom, J., et al. Optimization of a GCaMP calcium indicator for neural activity imaging. Journal of Neuroscience. 32 (40), 13819-13840 (2012).
- Chen, T. -W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Piatkevich, K. D., et al. A robotic multidimensional directed evolution approach applied to fluorescent voltage reporters. Nature Chemical Biology. 14 (4), 352-360 (2018).
- Tian, L., et al. Imaging neural activity in worms, flies and mice with improved GCaMP calcium indicators. Nature Methods. 6 (12), 875-881 (2009).
- Looger, L., et al. Fast and sensitive GCaMP calcium indicators for imaging neural populations. Biorxiv. , (2021).
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nature Methods. 10 (5), 413-420 (2013).
- Prevedel, R., et al. Simultaneous whole-animal 3D imaging of neuronal activity using light-field microscopy. Nature Methods. 11 (7), 727-730 (2014).
- Chhetri, R. K., et al. Whole-animal functional and developmental imaging with isotropic spatial resolution. Nature Methods. 12 (12), 1171-1178 (2015).
- Ahrens, M. B., Engert, F. Large-scale imaging in small brains. Current Opinion in Neurobiology. 32, 78-86 (2015).
- Yoon, Y. -G., et al. Sparse decomposition light-field microscopy for high speed imaging of neuronal activity. Optica. 7 (10), 1457-1468 (2020).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nature Methods. 12 (11), 1039-1046 (2015).
- Cong, L., et al. Rapid whole brain imaging of neural activity in freely behaving larval zebrafish (Danio rerio). eLife. 6, e28158(2017).
- Bruzzone, M., et al. Whole brain functional recordings at cellular resolution in zebrafish larvae with 3D scanning multiphoton microscopy. Scientific Reports. 11 (1), 11048(2021).
- Cho, E. -S., Han, S., Lee, K. -H., Kim, C. -H., Yoon, Y. -G. 3DM: deep decomposition and deconvolution microscopy for rapid neural activity imaging. Optics Express. 29 (20), 32700-32711 (2021).
- Zhang, Z., et al. Imaging volumetric dynamics at high speed in mouse and zebrafish brain with confocal light field microscopy. NatureBiotechnology. 39 (1), 74-83 (2021).
- Muto, A., Ohkura, M., Abe, G., Nakai, J., Kawakami, K. Real-time visualization of neuronal activity during perception. Current Biology. 23 (4), 307-311 (2013).
- Köster, R. W., Fraser, S. E. Tracing transgene expression in living zebrafish embryos. Developmental Biology. 233 (2), 329-346 (2001).
- Park, H. C., et al. Analysis of upstream elements in the HuC promoter leads to the establishment of transgenic zebrafish with fluorescent neurons. Developmental Biology. 227 (2), 279-293 (2000).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: an introduction. Journal of Visualized Experiments. (69), e4196(2012).
- Westerfield, M. The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). 4th edition. , University of Oregon Press. Eugene. (2000).
- Morsch, M., et al. Triggering cell stress and death using conventional UV laser confocal microscopy. Journal of Visualized Experiments. (120), e54983(2017).
- Antinucci, P., Hindges, R. A crystal-clear zebrafish for in vivo imaging. Scientific Reports. 6, 29490(2016).
- Parker, M. O., Brock, A. J., Millington, M. E., Brennan, C. H. Behavioral phenotyping of casper mutant and 1-pheny-2-thiourea treated adult zebrafish. Zebrafish. 10 (4), 466-471 (2013).
- Elsalini, O. A., Rohr, K. B. Phenylthiourea disrupts thyroid function in developing zebrafish. Development Genes and Evolution. 212 (12), 593-598 (2003).
- Han, S., Cho, E. -S., Park, I., Shin, K., Yoon, Y. -G. Efficient neural network approximation of robust PCA for automated analysis of calcium imaging data. Medical Image Computing and Computer Assisted Intervention. , Springer International Publishing. 595-604 (2021).
- Shin, C., et al. Three-dimensional fluorescence microscopy through virtual refocusing using a recursive light propagation network. Medical Image Analysis. 82, 102600(2022).
- Eom, M., et al. Statistically unbiased prediction enables accurate denoising of voltage imaging data. bioRxiv. , (2022).
- Johnston, L., et al. Electrophysiological recording in the brain of intact adult zebrafish. Journal of Visualized Experiments. (81), e51065(2013).
- Sofroniew, N., et al. napari: a multi-dimensional image viewer for Python. Zenodo. , 3555620(2022).
- White, R. M., et al. Transparent adult zebrafish as a tool for in vivo transplantation analysis. Cell Stem Cell. 2 (2), 183-189 (2008).
- Mickoleit, M., et al. High-resolution reconstruction of the beating zebrafish heart. Nature Methods. 11 (9), 919-922 (2014).
- Voleti, V., et al. Real-time volumetric microscopy of in vivo dynamics and large-scale samples with SCAPE 2.0. Nature Methods. 16 (10), 1054-1062 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados