Method Article
Verbesserte Methoden zur Präparation von Querschnitten und abgerollten ganzen Mounts von Maisblattprimordien für die Fluoreszenz- und konfokale Bildgebung
In diesem Artikel
Zusammenfassung
Maisblatt-Primordien sind tief umhüllt und gerollt, was es schwierig macht, sie zu untersuchen. Hier stellen wir Methoden zur Herstellung von Querschnitten und abgerollten ganzen Mounts von Maisblattprimordien für die Fluoreszenz- und konfokale Bildgebung vor.
Zusammenfassung
Bei Mais (Zea mays) und anderen Gräsern (Poaceae) sind die Blattprimordien tief umhüllt und im Blattquirl eingerollt, was es schwierig macht, die frühe Blattentwicklung zu untersuchen. Hier beschreiben wir Methoden zur Präparation von Querschnitten und abgerollten ganzen Mounts von Maisblattprimordien für die Fluoreszenz- und konfokale Bildgebung. Bei der ersten Methode wird eine Abisolierzange verwendet, um die oberen Teile älterer Blätter zu entfernen, wodurch die Spitze des Blattprimordiums freigelegt wird und seine Messung für eine genauere Querschnittsprobenahme ermöglicht wird. Bei der zweiten Methode wird durchsichtiges, doppelseitiges Nanoklebeband verwendet, um ganzblättrige Primordien für die Bildgebung abzurollen und zu montieren. Wir zeigen den Nutzen der beiden Methoden bei der Visualisierung und Analyse fluoreszierender Proteinreporter in Mais. Diese Methoden bieten eine Lösung für die Herausforderungen, die sich aus der charakteristischen Morphologie von Maisblattprimordien ergeben, und werden für die Visualisierung und Quantifizierung von anatomischen und entwicklungsbedingten Merkmalen von Blättern bei Mais und anderen Grasarten nützlich sein.
Einleitung
Graspflanzen sind eine wichtige Nahrungsquelle und Biokraftstoffquelle für die Weltbevölkerung1, und die Verbesserung der Blattanatomie hat das Potenzial, ihre Produktivität zu steigern 2,3. Unser derzeitiges Verständnis darüber, wie die Blattanatomie bei Gräsern reguliert wird, ist jedoch begrenzt4 und erfordert die Analyse von Blattprimordien, da viele anatomische und physiologische Merkmale des Blattes früh in der Entwicklung vorgegeben sind 5,6,7. Zelluläre Bildgebungstechniken wie Fluoreszenz und konfokale Bildgebung sind unverzichtbar für die Untersuchung der Anatomie und der zellulären Merkmale von Grasblättern, aber diese Techniken lassen sich nur schwer auf Grasblattprimordien anwenden, da sie tief in den Blattquirl eingehüllt und gerollt sind. Wir haben dieses Problem angegangen, indem wir Methoden zur Herstellung von Querschnitten und abgerollten Ganzblatthalterungen für die Fluoreszenz- und konfokale Analyse von Maisblattprimordien entwickelt haben, einem Modellsystem zur Untersuchung der Anatomie und Entwicklung von Grasblättern 2,8.
Das Maisblatt besteht wie alle Grasblätter aus einer riemenartigen Halme mit einer Scheide, die sich um den Stängel wickelt und den Trieb 9,10,11,12,13 entwickelt. Die Blätter entwickeln sich aus dem apikalen Meristem (SAM) des Sprosses in einem distichösen Muster, bei dem jedes neue Blatt in der entgegengesetzten Position des vorherigen Blattes beginnt, was zu zwei Blattreihen entlang der vertikalen Achse führt (Abbildung 1A)14 . Das Entwicklungsstadium jedes Blattprimordiums wird durch seine Position relativ zum SAM identifiziert, wobei das nächstgelegene Primordium als Plastochron1 (P1) und die folgenden Primordien als P2, P3 usw. bezeichnet werden (Abbildung 1B,C)2. Während der Entwicklung (Abbildung 1D) erscheint das Blattprimordium zunächst als halbmondförmiger Strebepfeiler um die Basis des SAM (P1) und wächst dann zu einem haubenförmigen Primordium heran, das sich über das Meristem erstreckt (P2)9,10,11. Die basalen Ränder der Haube dehnen sich dann seitlich aus und überlappen sich, wenn die Spitze nach oben wächst, und bilden ein kegelförmiges Primordium (P3-P5)10. Das Primordium nimmt dann rasch an Länge zu, und die Scheiden-Blatt-Grenze an der Basis wird mit der Bildung des Blatthäutchens, dem fransenartigen Vorsprung auf der adaxialen Seite des Blattes (P6/P7), deutlicher. Schließlich entfaltet sich das Blatt, wenn es während des stationären Wachstums aus dem Wirtel austritt, bei dem die sich teilenden Zellen in der kleinen basalen Region der Spreite eingeschränkt sind und einen Gradienten mit expandierenden und differenzierenden Zellen entlang der proximal-distalen Achse bilden (P7/P8)15. Die Sprossspitze eines Maiskeimlings enthält mehrere Primordien in unterschiedlichen Entwicklungsstadien, was sie zu einem hervorragenden Modell für die Untersuchung der Blattentwicklung macht8.
Eine genaue Analyse der frühen Blattentwicklung erfordert eine Stadieneinteilung oder die Verwendung standardisierter Kriterien, um verschiedene Stadien der Primordiumentwicklung in Bezug auf andere Wachstums- oder morphologische Parameter zu definieren. Da die Blattprimordien im Grastrieb verborgen sind, verwenden die Forscher in der Regel Parameter wie das Alter der Pflanze oder die Größe der austreibenden Blätter als Prädiktoren für die Stadien und Größen der Blattprimordien 9,16. Bei Mais wird das chronologische Alter der Pflanze entweder durch die Anzahl der Tage nach der Pflanzung oder nach der Keimung (DAP/DAG) bestimmt17,18. Das vegetative Stadium (V-Stadium) wird durch das oberste Blatt mit sichtbarem Kragen, eine blasse Linie auf der abaxialen Seite zwischen der Blattspreite und der Scheide, die der Position des Blatthäutchens und der Ohrmuscheln entspricht, ein Paar keilförmiger Regionen an der Basis der Spreite bestimmt (Abbildung 1A,B)17,19 . Zwischen 20 und 25 DAG geht das SAM in ein Blütenstandsmeristem über und hört auf, neue Blätter zu produzieren20. Die Wachstumsraten von Maisblattprimordien können je nach Umgebung und Genotyp der Pflanze variieren. Aus diesem Grund können das Alter der Pflanzen und die Größe der austreibenden Blätter die Größe der Blattprimordien nicht genau vorhersagen. Die Verwendung dieser Parameter kann jedoch dazu beitragen, den Bereich der Primordia-Stadien und -Größen für experimentelle Zwecke vorherzusagen.
Die Querschnittsanalyse ist eine beliebte Methode zur Untersuchung der Blattanatomie und -entwicklung bei Mais und anderen Gräsern, da sie die Entnahme mehrerer Plastochronen in einem einzigen Schnitt über den Sprossermöglicht 21,22,23. Diese Methode eignet sich auch für die zelluläre Bildgebung frischer Proben, da die umgebenden Blätter als Gerüst dienen, das die Blattprimordien während des Schneidens und Montierens an Ort und Stelle hält24. Ein Nachteil dieser Methode besteht jedoch darin, dass es schwierig sein kann, den Zielplastochron und die Zielregion innerhalb des Primordiums genau zu lokalisieren, wenn aus einem intakten Spross geschnitten wird. Da das Blattwachstum zwischen den Plastochronen und entlang der proximal-distalen Achsevariiert 2,5, kann eine ungenaue Probenahme zu einer falschen Interpretation des Entwicklungsstadiums und der Region des Primordiums in einem bestimmten Abschnitt führen. Daher ist die Entwicklung einer Methode zur präzisen Querschnittsprobenahme von entscheidender Bedeutung, um die Genauigkeit und Reproduzierbarkeit von anatomischen und entwicklungsbedingten Analysen von Grasblattprimordien zu gewährleisten.
Die Ganzblattanalyse ermöglicht die umfassende und integrative Untersuchung von Gewebe- und Zellprozessen, die auf der Ebene des gesamten Organs ablaufen, wie z. B. proliferatives Wachstum25 und Venenmusterung26,27,28. Die Methode bietet einen paradermalen Überblick über das Blatt und ermöglicht die Entdeckung unterschiedlicher Prozesse und Muster, die sonst mit Hilfe der Querschnittsanalyse schwer zu erkennen wären24,27. Anders als bei Arabidopsis, wo es bereits etablierte Methoden zur Bildgebung ganzblättriger Halterungen gibt29,30, gibt es derzeit keine Standardmethode zur Abbildung von ungerollten ganzblättrigen Halterungen in Gräsern. Ein früheres Protokoll zum Abrollen isolierter Maisblattprimordien betraf ungewöhnliche Materialien und war für die zelluläre Bildgebung nicht geeignet31. Fortschrittliche bildgebende Verfahren wie Computertomographie (CT) und Magnetresonanztomographie (MRT) können anatomische 3D-Informationen erfassen, ohne die Primordien zu isolieren und abzurollen 11,32,33, aber sie sind teuer und erfordern spezielle Geräte. Die Entwicklung einer Technik zur Überwindung der Einschränkungen, die durch die gerollte und konische Morphologie von Blattprimordien bei Mais und anderen Gräsern auferlegt werden, würde die Untersuchung ihrer anatomischen und entwicklungsbedingten Merkmale vorantreiben.
Hier stellen wir Methoden zur Herstellung von Querschnitten und abgerollten ganzen Mounts von Maisblattprimordien für die Fluoreszenz- und konfokale Bildgebung vor. Wir haben diese Methoden verwendet, um die Venenzahl zu quantifizieren und die räumlich-zeitliche Hormonverteilung in Maisblattprimordien mit fluoreszierenden Proteinen (FPs) zu kartieren24. Bei der ersten Methode wird der obere Teil älterer Blätter von Maissämlingen mit einer Abisolierzange entfernt (Abbildung 1E). Durch die Freilegung der Spitze des Primordiums (P5-P7) wird es möglich, seine Länge zu bestimmen, ohne die älteren umgebenden Blätter vollständig entfernen zu müssen, was ein einfaches und genaues Schneiden ermöglicht. Die zweite Methode besteht darin, ganzblättrige Primordien (P3-P7) mit durchsichtigem, doppelseitigem Nanoband abzurollen und zu montieren (Abbildung 1F). Diese Methoden eignen sich für die Visualisierung verschiedener FPs24, müssen jedoch für die Verwendung von Fluoreszenzfarbstoffen und Clearingreagenzien optimiert werden. Darüber hinaus beschreiben wir einige Verfahren zum Reduzieren von Z-Stapeln, zum Zusammenfügen von Bildern und zum Zusammenführen von Kanälen in ImageJ/FIJI34, die für die mit den beiden Methoden erzeugten Bilder gelten. Diese Methoden sind nützlich für die routinemäßige Fluoreszenz- oder konfokale Bildgebung von Maisblättern, können aber auch für andere Modellgrasarten wie Reis, Setaria und Brachypodium angepasst werden.
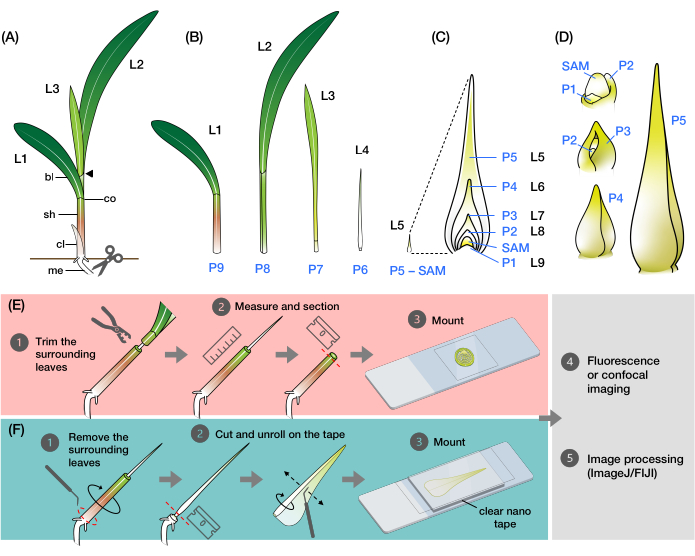
Abbildung 1: Organisation und Morphologie von Maisblattprimordien und Überblick über die Methoden . (A) Schematische Darstellung eines Maiskeimlings. Mais hat eine distichige Phyllotaxie, wobei das neue Blatt an der entgegengesetzten Position des vorherigen Blattes beginnt. Die Blattnummer gibt die chronologische Reihenfolge an, in der die Blätter aus der Keimung hervorgegangen sind (d. h. erstes Blatt, L1, zweites Blatt, L2, drittes Blatt, L3 usw.). Jedes Blatt hat eine distale Spreite und eine basale Scheide, die durch einen Kragen abgegrenzt wird, der dem Blatthäutchen und der Ohrmuschel entspricht. Das oberste Blatt mit sichtbarem Kragen kennzeichnet das vegetative Stadium. Der Sämling in diesem Beispiel befindet sich im V2-Stadium, wobei der L2-Kragen (Pfeilspitze) sichtbar ist. Das Scherensymbol zeigt die Stelle am Mesokotyl (Me) an, an der der Sämling geschnitten werden muss, um gesammelt zu werden. (B) Schematische Darstellung des präparierten Sprosses mit isolierten L1 bis L4, wobei die Blattprimordien L5 bis L9 als vergrößertes Bild in (C) dargestellt sind. Die Plastochronzahl gibt die Position des Primordiums relativ zum SAM an, wobei das jüngste Blattprimordium (P1) dem SAM am nächsten ist und die älteren Blattprimordien (P2, P3, P4 usw.) sukzessive weiter entfernt sind2. (D) Schematische Darstellung der Morphologie von Maisblattprimordien von P1 bis P5. (E) Schematische Darstellung des Verfahrens zur Querschnittsanalyse der Maisblattprimordien. (1) Schneiden Sie die älteren Blätter mit einer Abisolierzange ab. (2) Messen Sie das Primordium und schneiden Sie den Trieb. (3) Montieren Sie den Schnitt zur Bildgebung und Verarbeitung auf einen Objektträger (4, 5). (F) Schematische Übersicht über das Verfahren zur Whole-Mount-Analyse der Maisblatt-Primordien. (1) Entfernen Sie die umgebenden Blätter, um das Primordium zu extrahieren. (2) Schneiden Sie das Primordium ab und rollen Sie es flach auf dem Nanoband ab. (3) Montieren Sie die Probe für die Bildgebung und Verarbeitung (4, 5). Abkürzungen: L = Blatt; bl = Klinge; sh = Scheide; co = Kragen; me = Mesokotyl; V = vegetativ; P = Plastochron; SAM = apikales Meristem schießen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
1. Inszenierung der Maisblattentwicklung und Versuchsplanung
- Aufzucht der Pflanzen und Stadienbau der Blattentwicklung
- Bestimmen Sie das Alter und das Entwicklungsstadium der Pflanzen, die für das Experiment verwendet werden sollen, und führen Sie eine detaillierte Inszenierung der Blattentwicklung durch.
HINWEIS: Es wird empfohlen, die Stadieneinteilung an Pflanzen durchzuführen, die den gleichen genetischen Hintergrund haben wie die mutierten oder transgenen Linien, die in den Experimenten verwendet werden. Der SAM hört auf, neue Blätter zwischen 20 und 25 DAG zu produzieren, abhängig vom genetischen Hintergrund und den Wachstumsbedingungen. Aus diesem Grund sind die hier beschriebenen Methoden ideal für Maissämlinge im Alter von 7-14 DAG. - Ziehen Sie die Maispflanzen in einem Gewächshaus oder einer Wachstumskammer nach einem geeigneten Pflanzenpflegeprotokoll35.
- Sammeln Sie die Pflanzen im gewünschten Alter oder V-Stadium mit einem kleinen Messer oder einer Schere, indem Sie am Mesokotyl, dem Stängel unter der Bodenoberfläche, schneiden (Abbildung 1A). Führen Sie das Schneidwerkzeug vorsichtig in die Erde neben dem Sämling ein und schieben Sie es in einem Winkel von 45° zur Basis, um das Mesokotyl zu schneiden.
- Hebe den Sämling aus der Erde und entstaube den restlichen Schmutz oder die Bodenpartikel, die an der Pflanze haften bleiben. Entfernen Sie die Koleoptile, die Schutzhülle, die den austreibenden Trieb bei jungen Sämlingen bedeckt, von Hand.
- Notieren Sie für jede Pflanze das V-Stadium, die Anzahl der Blätter und die Länge des letzten Blattes, das aus dem Wirtel austritt. Machen Sie Fotos von den Pflanzen, um später darauf zurückgreifen zu können.
- Entfernen Sie die älteren Blätter nacheinander. Halten Sie dazu die Pflanze an den Resten des Mesokotyls oder Stängels fest, schneiden Sie jedes Blatt von der Basis der Scheide ab und entfalten Sie die Hülle vorsichtig kreisförmig mit einer Zahnsonde (Abbildung 1B).
Anmerkungen: In Schritt 2.1 finden Sie eine schnelle Methode zum Entfernen der umgebenden Blätter. - Präparieren Sie unter einem Stereomikroskop bei etwa 2-facher Vergrößerung vorsichtig den Rest der Triebspitze auf feuchten Tüchern, um ein Austrocknen der Probe zu verhindern. Schneiden Sie jedes Blattprimordium vorsichtig aus und entfalten Sie es mit einer Zahnsonde mit einer gebogenen Nadel oder einer feinen Pinzette, bis SAM, P1 und P2 sichtbar sind (Abbildung 1D). Notieren Sie das Aussehen und die Maße jedes Plastochrons.
HINWEIS: Ein Beispiel für die Stadieneinteilung der Blattentwicklung bei Maissämlingen finden Sie in der Ergänzungsdatei 1.
- Bestimmen Sie das Alter und das Entwicklungsstadium der Pflanzen, die für das Experiment verwendet werden sollen, und führen Sie eine detaillierte Inszenierung der Blattentwicklung durch.
- Planung des Experiments
HINWEIS: Eine sorgfältige Planung des Experiments kann die Effizienz verbessern. Bei der Querschnittsanalyse kann beispielsweise die Bestimmung der ungefähren zu schneidenden Position bei der Abbildung bestimmter Plastochronen oder Regionen die Präparierzeit verkürzen. Abbildung 2 zeigt ein Beispiel für eine standardisierte Stichprobenahme. Darüber hinaus kann die Optimierung der Bildgebungsparameter auf der Grundlage der Eigenschaften der FPs oder Fluoreszenzsonden die Effizienz der Experimente verbessern.- Verwenden Sie die Staging-Informationen als Leitfaden, um das Experiment auf bestimmte Plastochronen, Primordiumregionen und Gewebe zu konzentrieren. Verwenden Sie die FPs oder andere Fluoreszenzsonden von Interesse, um die Abschnitte 2 und/oder 3 unten an einigen Proben zu befolgen.
HINWEIS: Siehe verfügbare Saatgutvorräte der Mais-FP-Markierungslinie 36-42 unter https://www.maizegdb.org/data_center/stock. - Bestimmen Sie die optimalen Bildaufnahmeparameter für jeden FP-Reporter oder jede Sonde, einschließlich des geeigneten Detektors oder Filters, der Anregungs- und Emissionswellenlängen, der Lochblendengröße und anderer Einstellungen. Speichern Sie die Einstellungen, um die Arbeitsbedingungen für jedes der Experimente zu reproduzieren.
HINWEIS: Siehe Linh und Scarpella30 für einige technische Richtlinien für die konfokale Laser-Scanning-Mikroskopie von sich entwickelnden Blättern. - Planen Sie die Aufnahmezeit auf der Grundlage der Halbwertszeit und anderer Eigenschaften des FP-Reporters43.
HINWEIS: Bei der Planung eines Experiments muss auch die Zeit zwischen Präparation und Bildgebung berücksichtigt werden, da frische Pflanzenproben in diesem Zeitraum Austrocknung und zellulären Veränderungen ausgesetzt sind.
- Verwenden Sie die Staging-Informationen als Leitfaden, um das Experiment auf bestimmte Plastochronen, Primordiumregionen und Gewebe zu konzentrieren. Verwenden Sie die FPs oder andere Fluoreszenzsonden von Interesse, um die Abschnitte 2 und/oder 3 unten an einigen Proben zu befolgen.
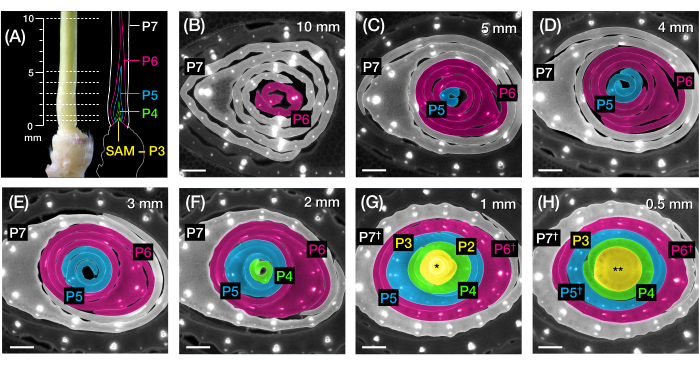
Abbildung 2: Probenahmeschema für die Querschnittsanalyse von Maisblattprimordien. (A, links) Proximaler Spross eines 7 DAG Maiskeimlings, der ein freiliegendes viertes Blatt (L4) bei P7 zeigt. Die gestrichelten Linien zeigen die sieben Probenahmepunkte entlang des Primordiums, von 0,5 mm bis 10 mm. (A, rechts) Schematische Darstellung der Blattprimordien mit der projizierten Größe und Position jedes Plastochronen: P7 (weiß); P6 (magenta); P5 (blau); P4 (grün); und P3 bis zum SAM (gelb). (B-H) Fluoreszenzbilder von Querschnitten, die die in A dargestellten Probenahmepunkte von 10 mm (B) bis 0,5 mm (H) darstellen. Die Primordien sind nach dem Plastochron-Farbschema in (A) pseudogefärbt. Die Schnitte wurden mit einem Epifluoreszenzmikroskop unter Verwendung eines Langpass-Emissions-UV-Filters für Autofluoreszenz abgebildet. Maßstabsbalken = 200 μm (B-H). Diese Abbildung wurde mit Genehmigung von Robil und McSteen24 modifiziert und reproduziert. Abkürzungen: DAG = Tage nach der Keimung; P = Plastochron; SAM = apikales Meristem schießen; † = Blattscheide oder Vorlighäut im eigentlichen Sinne; * = apikales Meristem schießen; ** = Stiel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
2. Darstellung von Querschnitten von Maisblatt-Primordien
- Abstreifen älterer umliegender Blätter und Vermessung des Blattprimordiums
- Sammeln Sie den Sämling, indem Sie ihn vorsichtig mit einem kleinen Messer oder einer Schere am Mesokotyl unter der Bodenoberfläche abschneiden, wie in den Schritten 1.1.3-1.1.4 beschrieben (Abbildung 1A).
- Bestimmen Sie die richtige Lochgröße für die Abisolierzange und die Stelle, an der der Schnitt entlang des Triebs vorgenommen werden soll. Stellen Sie sicher, dass das Loch groß genug ist, um in das Zielprimordium zu passen.
- Beginnen Sie mit dem Schneiden am distaleren Teil des Triebs und arbeiten Sie sich allmählich zur Basis vor, bis die Spitze des Primordiums freiliegt.
- Um den Schnitt zu machen, halten Sie die Abisolierzange so, dass die Backen zum Trieb zeigen. Positionieren Sie den Trieb auf das ausgewählte Loch und drücken Sie die Streiche des Abstreifers zusammen, um den Blattwirtel zu durchtrennen.
Anmerkungen: Um ein versehentliches Schneiden des Zielprimordiums zu vermeiden, richten Sie die Mitte des Triebs sorgfältig am Loch in der Abisolierzange aus. Bei größeren Pflanzen empfiehlt es sich, einige der umliegenden Blätter manuell von Hand zu entfernen, bevor man den Abstreifer verwendet.
VORSICHT: Abisolierzangen haben scharfe Backen, die bei unvorsichtiger Verwendung Schnitte oder andere Verletzungen verursachen können. - Während Sie die Griffe weiter zusammendrücken, schieben Sie den Abstreifer vom Trieb weg, um den oberen Teil der Blätter abzuschneiden. Achten Sie darauf, dass der Zielbereich im Primordium von den umgebenden Blättern umhüllt bleibt (ergänzende Abbildung S1C,D).
Anmerkungen: Nach dem Beschneiden der oberen Blätter bleibt der Trieb mit einer freiliegenden Primordiumspitze zurück (P5, P6 oder P7, abhängig von der Position des Schnitts und der Größe des verwendeten Abisolierlochs; Abbildung 1E). Dieses Primordium dient als Referenz für die Abschätzung der Größe und des Stadiums jüngerer Primordien. P6 ist aufgrund seines Größenbereichs und seiner stiftartigen Morphologie die praktischste Referenz für den Einsatz in Maissämlingen. - Verwenden Sie ein Lineal, um die Länge von der Spitze des Primordiums bis zur Basis des Triebs zu messen. Zeichnen Sie die Messung auf.
HINWEIS: Die Länge des Primordiums wird leicht überschätzt, da sich an der Basis der Primordien einige Millimeter Stiel befinden (Abbildung 2A).
- Freihandschnitt und Bildgebung des Blattprimordiums
- Halten Sie den Trieb unter einem Stereomikroskop bei etwa 0,8-facher Vergrößerung ruhig auf einer glatten Oberfläche, z. B. einem Glasschneidebrett oder einem anderen kratzfesten Material. Verwenden Sie eine Rasierklinge oder ein Skalpell, um dünne Abschnitte (0,25-0,8 mm) des Triebs an den gewünschten Stellen entlang der Länge des Primordiums zu schneiden.
- Machen Sie einen ersten Schnitt etwas oberhalb der Zielregion, um den distalen Teil des Triebs zu verwerfen, und erhalten Sie dann dünne Schnitte um die Zielregion. Um saubere Schnitte zu machen, halten Sie die Klinge senkrecht zum Trieb und schieben Sie sie mit einer sanften, gleichmäßigen Bewegung nach innen.
HINWEIS: Bei größeren Primordien (P6 oder P7) kann es zu einigen Millimetern Schnittspielraum kommen. Bei kleineren Primordien (P5 und früher) kann 1 mm jedoch einen signifikanten Unterschied bei der Probenahme ausmachen, da diese Primordien innerhalb der ersten Millimeter der Triebbasis gestapelt sind (siehe Abbildung 2A, F-H). Es wird empfohlen, eine zweischneidige Rasierklinge für feinere Schnitte zu verwenden.
VORSICHT: Seien Sie vorsichtig, wenn Sie Rasierklingen und Skalpelle verwenden, um versehentliche Schnitte oder Verletzungen zu vermeiden. - Tauschen Sie die Rasierklinge regelmäßig aus, da die Blattscheiden die Klinge leicht stumpf machen können. Decken Sie den Trieb nach jeder Schnittrunde mit feuchten Tüchern ab, um ein Austrocknen der Probe zu verhindern.
- Montieren Sie den Blattabschnitt auf einem sauberen Objektträger (75 mm x 25 mm), tragen Sie mit einer Pipette einen Tropfen Wasser (oder 50 % Glycerin) direkt auf den Abschnitt auf und legen Sie ein Deckglas (22 mm x 22 mm) darauf.
- Wenn Sie einen Fluoreszenzfarbstoff verwenden, tragen Sie die Farbstofflösung auf den Abschnitt auf, legen Sie ein Deckglas darauf und inkubieren Sie es nach Bedarf.
HINWEIS: Je nach Art des Farbstoffs können zusätzliche Schritte zur Verarbeitung der Blattabschnitte erforderlich sein, bevor mit dem nächsten Schritt fortgefahren wird. Diese Referenzen44,45,46,47 enthalten allgemeine Details und Verwendungen von Fluoreszenzfarbstoffen in der Bildgebung von Pflanzenzellen. Ein kernfärbender Fluoreszenzfarbstoff, 5-Ethinyl-2′-desoxyuridin (EdU), wurde effektiv auf Querschnitten von Maisblattprimordien48 eingesetzt. Im Gegensatz dazu liefern Plasmamembran-Färbefarbstoffe wie Fei Mao 4-64 (FM 4-64) und Propidiumiodid keine zufriedenstellenden Ergebnisse (Abbildung 3A-D). - Legen Sie den Objektträger auf den Tisch eines Epifluoreszenz- oder konfokalen Laser-Scanning-Mikroskops und passen Sie den Fokus und die Einstellungen nach Bedarf an, um den Fluorophor30 zu visualisieren. In Tabelle 1 sind die Beleuchtungs- und Bildaufnahmeeinstellungen aufgeführt, die für ausgewählte Mais-FP-Reporterlinien verwendet werden.
Anmerkungen: Dicke Schnitte können ein hohes Maß an Hintergrund-Autofluoreszenz erzeugen. Ziehen Sie einige Bildgebungs- und Probenvorbereitungsstrategien in Betracht, die dazu beitragen können, dieses Problem zu entschärfen30,49 (siehe Tabelle 2).
VORSICHT: Seien Sie vorsichtig und tragen Sie eine Schutzbrille, wenn Sie mit leistungsstarken Lichtquellen und Lasern arbeiten, um das Risiko von Augenschäden zu verringern. - Bild durch den Blattausschnitt mit unterschiedlichen Vergrößerungen und Detektionskanälen (einschließlich der Hellfeldbeleuchtung)30. Verwenden Sie eine 4-fache Vergrößerung, um den gesamten Querschnitt abzubilden, und eine 20- oder 40-fache Vergrößerung, um bestimmte Gewebe abzubilden. Reduzieren Sie die Belichtungszeit bei höheren Vergrößerungen für die Epifluoreszenzbildgebung, um die Proben vor schnellem Photobleichen zu schützen (siehe Tabelle 1).
- Erfassen Sie Bilder des gesamten Abschnitts oder bestimmter Interessenbereiche (ROIs). Erfassen Sie bei Bedarf einen Z-Stapel, bei dem es sich um eine Reihe von Bildern handelt, die mit unterschiedlichen Brennweiten30 aufgenommen wurden.
- Um einen Z-Stapel zu erhalten, bestimmen Sie zuerst die obere und untere Position in der Probe und dann die Anzahl der optischen Abschnitte, die zwischen diesen Positionen erfasst werden müssen. Weniger optische Abschnitte führen zu einer größeren z-Schrittgröße, d. h. dem Abstand zwischen den einzelnen Abschnitten. Abhängig von den Eigenschaften des FP und der verwendeten Vergrößerung können drei bis 25 optische Schnitte mit z-Schrittgrößen zwischen 1 und 12 μm für die Abbildung von Querschnitten von Maisblattprimordien erfasst werden.
ANMERKUNG: Um den Z-Stapel zu glätten, verwenden Sie eine geeignete Volumen-Rendering-Technik50 , die über die Mikroskop-Software oder durch Verwendung von ImageJ/FIJI34 durchgeführt werden kann (siehe Schritte 4.1 und 4.2). - Speichern Sie alle Bilddateien und die zugehörigen Metadaten, falls verfügbar.
Tabelle 1: Beleuchtungs- und Bildaufnahmeeinstellungen für die Fluoreszenz- und konfokale Bildgebung ausgewählter Mais-FP-Reporter. Abkürzungen: FP = fluoreszierendes Protein; TRITC = Tetramethylrhodamin; FITC = Fluorescein-Isothiocyanat; WLL = Weißlichtlaser; Ar-Ion = Argon-Ionen-Laser; HyD = Hybrid-Detektor; AU = Luftige Einheit; Hz = Hertz, Scanzeile pro Sekunde. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
3. Abbildung ganzer Mounts von Maisblatt-Primordien
- Präparieren der Primordien und Präparation des Objektträgers mit Nano-Tape
- Sammeln Sie den Sämling, indem Sie ihn vorsichtig mit einem kleinen Messer oder einer Schere am Mesokotyl unter der Bodenoberfläche abschneiden, wie in den Schritten 1.1.3-1.1.4 beschrieben (Abbildung 1A).
- Bereiten Sie den Objektträger (75 mm x 25 mm) mit dem durchsichtigen, doppelseitigen Nanoklebeband (ergänzende Abbildung S1H) vor, bevor Sie die Pflanze präparieren. Schneiden Sie ein rechteckiges Stück Nano-Klebeband ab und kleben Sie es in die Mitte eines sauberen Objektträgers. Entfernen Sie nicht die Schutzfolie auf der Oberseite des Klebebands.
HINWEIS: Bestimmen Sie die Größe des zu verwendenden Bandes entsprechend der Größe der abzubildenden Proben (siehe Beispiele für übergroße Proben in der ergänzenden Abbildung S1I,J). Das Nano-Klebeband ist entweder in Acryl- oder Polyurethan-Gelmaterial erhältlich. Beide Typen sind für die Fluoreszenz- und konfokale Bildgebung geeignet24. Um eine Verfärbung des Klebebandes zu vermeiden, schützen Sie es vor längerer Lichteinwirkung, indem Sie es in einem dunklen Behälter aufbewahren. - Beginnen Sie mit dem Sezieren des Triebs, indem Sie den oberen Teil der älteren Blätter mit einer Abisolierzange entfernen, wie in Schritt 2.1 beschrieben.
- Halten Sie den Trieb am Mesokotyl fest und entfernen Sie vorsichtig die umliegenden Blätter mit einer Zahnsonde, um die Primordien zu extrahieren. Entfernen Sie dazu die Blätter von der Basis des Triebs und entfalten Sie sie nacheinander, bis das interessierende Primordium freigelegt ist.
- Alternativ können Sie die Blattprimordien mit der Abisolierzange direkt extrahieren, indem Sie einen Schnitt in der Basalregion des Sprosses machen (ergänzende Abbildung S1E-G).
Anmerkungen: Diese Methode birgt ein höheres Risiko, dass die Primordien brechen.
- Montage und Abbildung des Primordiums.
- Entfernen Sie die Schutzfolie vom Klebeband. Legen Sie das freiliegende Primordium auf das Klebeband. Schneiden Sie das Primordium mit einer Rasierklinge an der Basis ab und entsorgen Sie den basalen Stamm und das Hypokotyl (Abbildung 1F).
- Das Primordium wird mit einer Dentalsonde mit einer gebogenen Nadel (Spitzendurchmesser 0,25-0,6 mm) abgerollt. Verwenden Sie eine Mikrosonde (Spitzendurchmesser von 0,15-0,2 mm) für Primordien, die kleiner als 3 mm sind (P3 bis frühes P4).
- Positionieren Sie die Nadelspitze parallel zur Oberfläche und entfalten Sie den äußeren Basalrand, indem Sie ihn sanft gegen das Band drücken.
- Für ein sanfteres Abrollen schmieren Sie die Innenfläche (adaxial) des Blattes, indem Sie die Nadelspitze in 100% Glycerin tauchen.
- Richten Sie die Nadelspitze mit dem äußeren Rand auf dem Band parallel zur Längsachse des Blattes aus. Schieben Sie die Nadel vorsichtig zur Seite, um das Blatt abzurollen und auf dem Klebeband flach zu drücken (Abbildung 1F).
Anmerkungen: Das Brechen oder Beschädigen des Blattes beim Abrollen ist häufig, insbesondere bei größeren Primordien mit einer robusten Mittelrippe (siehe Abbildung 3E-G). Das kräftige Abrollen des Blattes kann seine Oberfläche beschädigen und Artefakte während der Bildgebung erzeugen (Abbildung 3H). In Tabelle 2 und in der Diskussion finden Sie empfohlene Lösungen für diese Probleme. - Tragen Sie einen Tropfen Wasser auf das ausgerollte Primordium auf. Legen Sie sofort ein Deckglas auf den Wassertropfen und das Primordium. Drücken Sie die Kanten des Deckglases vorsichtig nach unten, damit sie am Klebeband haften.
HINWEIS: Es wird empfohlen, große rechteckige Deckgläser (50 mm x 24 mm oder 60 mm x 24 mm) mit zusätzlichen Flächen zum Aufkleben des Klebebandes zu verwenden. Minimieren Sie die Bildung von Luftblasen, die das Blatt verformen und eine ungleichmäßige Lichtbrechung verursachen können (siehe Abbildung 3I-K). Achten Sie darauf, dass das Deckglas vollständig auf dem Klebeband haftet, da die Ränder des montierten Primordiums unter einem locker aufgeklebten Deckglas zurückrollen können (siehe Abbildung 3L). - Legen Sie den Objektträger auf den Tisch eines Epifluoreszenzmikroskops (siehe ergänzende Abbildung S1K) oder eines konfokalen Laser-Scanning-Mikroskops und passen Sie den Fokus und die Einstellungen nach Bedarf an, um den Fluorophor30 zu visualisieren. In Tabelle 1 sind die Beleuchtungs- und Bildaufnahmeeinstellungen aufgeführt, die für die ausgewählten Mais-FP-Reporterlinien verwendet werden.
- Bilden Sie das gesamte Blatt oder einen bestimmten ROI über verschiedene Erkennungskanäle30 ab. Um das gesamte Blatt abzubilden, nehmen Sie entweder manuell Mosaikbilder des Blattes auf oder verwenden Sie den Kachelbetrieb des Mikroskops bei geringer Vergrößerung (4x oder 10x).
HINWEIS: Selbst bei der niedrigsten Vergrößerung können Maisblattprimordien zu groß sein, um sie mit einem Epifluoreszenz- oder konfokalen Laser-Scanning-Mikroskop abzubilden, was zu längeren Bildgebungszeiten führt, die zu Photobleichung (siehe Abbildung 3M) oder erhöhter Hintergrundautofluoreszenz führen können. Daher wird empfohlen, ein Fluoreszenz-Stereomikroskop für die Abbildung von Ganzblattproben zu verwenden, die größer als 5 mm sind. - Für die konfokale Bildgebung ist es notwendig, Z-Stapel zu erhalten, die die Dicke des Blattes umfassen (siehe Schritt 2.2.9).
- Speichern Sie alle Bilddateien und die zugehörigen Metadaten, falls verfügbar.
Tabelle 2: Fehlerbehebung bei häufigen Problemen bei der Bildgebung von Querschnitten und ganzen Mounts von Maisblattprimordien. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
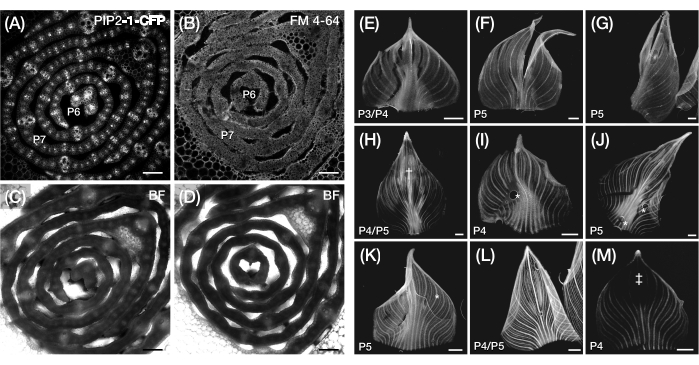
Abbildung 3: Suboptimaler Querschnitt und Whole-Mount-Präparation von Maisblattprimordien. (A-D) Repräsentative konfokale Aufnahmen von Querschnitten von Blattprimordien mit einem Plasmamembranmarker, PIP2-1-CFP (A), und einem plasmamembranbindenden Fluoreszenzfarbstoff, FM 4-64 (B), mit entsprechenden Hellfeldbildern (C,D). Im Vergleich zu PIP2-1-CFP zeigt FM 4-64 eine suboptimale Visualisierung der Zellumrisse. (E-M) Repräsentative Fluoreszenzbilder ganzer Halterungen von Blattprimordien, die das Vorhandensein von Rissen (E-G), gequetschten Oberflächen† (H), Luftblasen* (I-K), zurückgerollten Rändern (L) und photogebleichten Regionen‡ (M) zeigen. Die Blattprimordien exprimieren DII-Venus (E-G), GAR2-YFP (H-J), mDII-Venus (K), PIN1a-YFP (L) und DR5-RFP (M). Maßstabsbalken = 200 μm (A-D); 500 μm (E-M). Abbildung 3A wurde mit Genehmigung von Robil und McSteen24 modifiziert und reproduziert, während Abbildung 5B-M unveröffentlichte Daten der Autoren sind. Abkürzungen: P = Plastochron; YFP = gelb fluoreszierendes Protein; RFP = rot fluoreszierendes Protein; CFP = cyan fluoreszierendes Protein; PIP2-1-CFP = p Zm PIP2-1::ZmPIP2-1:CFP; DII-Venus = pZmUbi:DII:YFP-NLS; GAR2-YFP = p Zm GAR2::ZmGAR2:YFP; mDII-Venus = pZmUbi:mDII:YFP-NLS; PIN1a-YFP = p Zm PIN1a::ZmPIN1a:YFP; DR5-RFP = DR5rev::mRFPer; BF = Hellfeld. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
4. Verarbeiten der Bilder mit ImageJ/FIJI
- Abflachen von Z-Stapeln mit maximaler Intensitätsprojektion (MIP)
- Starten Sie ImageJ/FIJI und öffnen Sie den Stapel von Bildern, oder erstellen Sie einen neuen Stapel aus mehreren separaten Bildern, indem Sie Bild | Stapel | Zu stapelnde Bilder aus dem Menü.
- Wählen Sie den Bildstapel aus. Wählen Sie im Menü Bild | Stapel | Z-Projekt.... Wählen Sie im Dialogfeld "Z-Projekt" im Dropdown-Menü "Projektionstyp" die Option "Maximale Intensität" aus. Klicken Sie auf OK, um die Z-Projektion mit maximaler Intensität zu erstellen.
HINWEIS: Das reduzierte Bild ist eine 2D-Projektion der Pixel mit der höchsten Intensität über die Z-Stapel50. Diese Methode eignet sich für die Visualisierung des Fluorophors, nicht aber der anatomischen Merkmale in Hellfeldbildern von dicken Querschnitten und Ganzblattmontagen. - Speichern Sie das Bild als TIFF (Tagged Image File Format), indem Sie Datei | Speichern unter | tiff. Geben Sie einen Namen für die Datei ein und klicken Sie auf Speichern.
HINWEIS: Es wird empfohlen, TIFF zum Speichern von Fluoreszenz- und konfokalen Bildern zu verwenden, da die Qualität und Details beim Speichern des Bildes erhalten bleiben.
- Abflachen von Z-Stapeln mit dem EDF-Plugin (Extended Depth of Field)51
- Installieren Sie das EDF-Plugin52. Laden Sie die Extended_Depth_Field.jar Datei (Version 17.05.2021) von http://bigwww.epfl.ch/demo/edf/ herunter und legen Sie sie im Ordner "plugins" von ImageJ/FIJI ab.
- Starten Sie ImageJ/FIJI und öffnen Sie den Stapel von Bildern, oder erstellen Sie einen neuen Stapel aus mehreren separaten Bildern, indem Sie Bild | Stapel | Zu stapelnde Bilder aus dem Menü.
- Wählen Sie den Bildstapel aus. Wählen Sie im Menü Plugins | Erweiterte Schärfentiefe | EDF Easy Mode. Verwenden Sie im Dialogfeld " Erweiterte Schärfentiefe " die Schieberegler unter "Geschwindigkeit/ Qualität", um die gewünschte Qualität und Verarbeitungsgeschwindigkeit festzulegen, und die Height-Map-Reg , um den Glättungsgrad für die Topografie festzulegen. Klicken Sie auf Ausführen , um das rekonstruierte Bild zu erstellen.
HINWEIS: EDF kombiniert den Stapel zu einem scharfen, zusammengesetzten 2D-Bild, indem der beste Fokusprojiziert wird 51. Diese Technik ist nützlich, um die begrenzte Tiefenschärfe zu überwinden und eignet sich für die Rekonstruktion von Hellfeldbildern von dicken Querschnitten oder Ganzblattmontagen. Seien Sie vorsichtig, wenn Sie diesen Filter anwenden, um den Fluorophor zu visualisieren, da EDF im Gegensatz zu MIP den Kontrast des Bildes anpasst und sich auf die Pixelintensität auswirkt. - Speichern Sie das Bild als TIFF-Datei, indem Sie Datei | Speichern unter | tiff. Geben Sie einen Namen für die Datei ein und klicken Sie auf Speichern.
- Zusammenfügen von Bildern mit dem Grid/Collection Stitching-Plugin53
- Speichern Sie die Sammlung von Bildern oder Bildstapeln, die zusammengefügt werden sollen, in einem einzigen Verzeichnis.
- Starten Sie ImageJ/FIJI und wählen Sie Plugins | Nähte | Raster-/Sammlungs-Stitching aus dem Menü. Wählen Sie im Dialogfeld "Raster-/Sammlungszuordnung" im Dropdown-Menü "Typ" die Option "Unbekannte Position" und dann "Alle Dateien im Verzeichnis" aus der Reihenfolge aus. Klicken Sie auf OK.
- Geben Sie im Dialogfeld Rasterzusammenfügung: Unbekannte Position, Alle Dateien im Verzeichnis den Speicherort der Bilder an, indem Sie den Verzeichnispfad in das Textfeld Verzeichnis eingeben oder einfügen oder auf Durchsuchen klicken, um das Verzeichnis zu suchen. Klicken Sie auf OK, um den Stitching-Vorgang zu starten.
HINWEIS: Die Option "Unbekannte Position " im Grid/Collection Stitching-Plugin rekonstruiert ein zusammengesetztes Bild aus einer Reihe zusammenhängender Bilder, indem sie deren überlappende Bereiche analysiert. Daher ist diese Option nützlich, um Mosaike von Bildern von Querschnitten oder ganzseitigen Passepartouts zusammenzufügen, die nicht mit einem Kachel-Scanvorgang erhalten wurden. - Wenn das Zusammenfügen abgeschlossen ist, erscheint ein neues Fenster mit dem zusammengefügten Bild. Speichern Sie das Bild als TIFF-Datei, indem Sie Datei | Speichern unter | tiff. Geben Sie einen Namen für die Datei ein und klicken Sie auf Speichern.
HINWEIS: Das Zusammenfügen großer Montagen von 2D- und 3D-Bildern kann auch mit Bildrekonstruktionsbefehlen in der Mikroskopsoftware oder über die Importoptionen für Bioformate in ImageJ/FIJI durchgeführt werden.
- Kombinieren mehrerer Kanäle mit dem Befehl "Kanäle zusammenführen"
- Starten Sie ImageJ/FIJI und öffnen Sie die Bilder oder Bildstapel, die zusammengeführt werden sollen.
HINWEIS: Bildstapel können zuerst mit den Schritten 4.1 oder 4.2 reduziert werden. MIP wird im Allgemeinen für die Abflachung von Fluorophorkanälen empfohlen, während EDF besser für die Abflachung von Hellfeldkanälen oder anderen Kanälen mit unterschiedlicher Schärfentiefe geeignet ist. - Passen Sie die Helligkeit und den Kontrast der Bilder mit Bild | Anpassen | Helligkeit/Kontrast. Verwenden Sie die Schieberegler oder Eingabefelder, um die Parameter anzupassen, und klicken Sie dann auf "Anwenden". Alternativ können Sie eine lineare Histogramm-Streckungsmethode verwenden, indem Sie Prozess | Kontrast verstärken.... Legen Sie den Prozentsatz der gesättigten Pixel fest, wählen Sie "Normalisieren" und klicken Sie dann auf "OK".
HINWEIS: Bei der Quantifizierung oder dem Vergleich der Signalintensität von Fluorophoren zwischen Bildern ist es im Allgemeinen am besten, die Helligkeit oder den Kontrast nicht anzupassen, da dies die relativen Pixelintensitäten im Bild verändern kann, was sich auf die Messungen und Vergleiche auswirken kann. - Um zwischen den Kanälen zu unterscheiden, wenden Sie mithilfe einer Lookup-Tabelle (LUT) Pseudofarben auf bestimmte Bilder an. Wählen Sie dazu das Bild aus, wählen Sie dann im Menü ImageJ/FIJI die Option Image aus, klicken Sie auf Lookup-Tabellen und wählen Sie die entsprechende LUT aus der Dropdown-Liste aus.
- Wählen Sie im Menü Bild | Farbe | Kanäle zusammenführen. Verwenden Sie für jeden Kanal (C1, C2, C3...) die Dropdown-Liste, um das Bild auszuwählen, das diesem Kanal zugewiesen werden soll. Klicken Sie auf OK , um die Kanäle zusammenzuführen.
- Speichern Sie das neue Bild als TIFF, indem Sie Datei | Speichern unter | tiff. Geben Sie einen Namen für die Datei ein und klicken Sie auf Speichern.
- Starten Sie ImageJ/FIJI und öffnen Sie die Bilder oder Bildstapel, die zusammengeführt werden sollen.
Ergebnisse
Querschnittsanalyse von Maisblattprimordien
Wir verwendeten Protokollabschnitt 2, um die Anzahl der Venen zu quantifizieren und Hormonreaktionsmuster in Querschnitten von Maisblattprimordien mit FPs zu charakterisieren (Abbildung 4)24. Um die Rolle des Pflanzenhormons Auxin für das Blattwachstum und die Venenbildung zu untersuchen, haben wir die Anzahl der Adern in den Blattprimordien einer Auxin-defizienten Maismutante, der verschwindenden Quaste, quantifiziert254. In frühen Plastochronen zeigen sich entwickelnde Adern in der medianen Zellschicht des Maisblattprimordiumsdeutlich zellularisiert 21,22. Die Identifizierung und Zählung von Venen mit konventionellen histologischen Techniken22 kann jedoch arbeitsintensiv und zeitaufwändig sein. Um die Venen zu quantifizieren, verwendeten wir daher den Mais-Auxin-Efflux-Proteinmarker p Zm PIN1a::ZmPIN1a:YFP41 (PIN1a-YFP im Folgenden), der sich entwickelnde Venen und prokambiale Stränge (PCS; Abbildung 4A). Mit Hilfe des Protokollabschnitts 2 konnten wir die Querschnittsprobenahme standardisieren, indem wir die Primordien vor dem Schnitt vermessen (Abbildung 4B,C). Wir entdeckten einen Trend, bei dem vt2 ein breiteres Primordium und mehr Adern als normal aufweist (Abbildung 4D,E)24, was mit den Daten von vollständig ausgebreiteten Blättern55 übereinstimmt, was darauf hindeutet, dass der Defekt in vt2 früh in der Blattentwicklung begann. Mit Hilfe von Protokollabschnitt 2 konnten wir auch die Expressionsmuster von FP-Reportern der Hormonantwort in den Blattprimordien systematisch untersuchen (siehe Beispiele der Bilder in Abbildung 4F-I). Durch ein standardisiertes Querschnitts-Probenahmeschema kartierten wir die Verteilung von Auxin-, Cytokinin- (CK) und Gibberellinsäure (GA)-Reaktionen auf verschiedene Plastochronen und Regionen von Blattprimordien und entdeckten neue Reaktionsmuster, von denen wir annehmen, dass sie Auswirkungen auf das Blattwachstum und die Venenbildung haben24. Daher zeigen diese repräsentativen Ergebnisse die Nützlichkeit des Protokollabschnitts 2 für die Querschnittsanalyse von Maisblattprimordien.
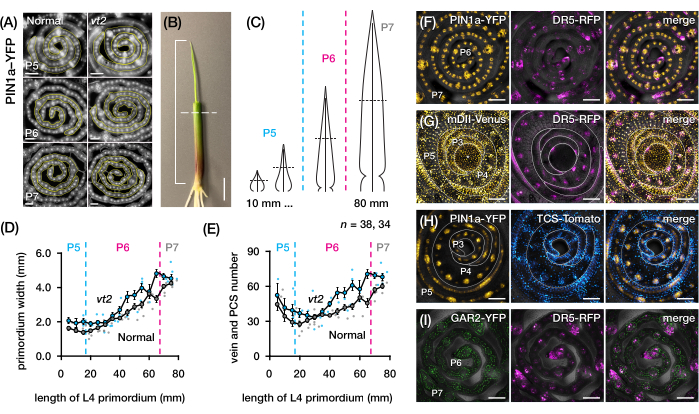
Abbildung 4: Repräsentative Ergebnisse für die Querschnittsanalyse von Maisblattprimordien. (A-E) Quantifizierung der Venenzahl und Primordiumbreite in Blattprimordien normaler und verschwindender Quasten2 mit dem Auxin-Efflux-Proteinmarker PIN1a-YFP. (A) Repräsentative Fluoreszenzbilder von Querschnitten von P5 bis P7, die PIN1a-YFP in sich entwickelnden Venen und prokambialen Strängen exprimieren. Die Querschnitte wurden mit einem Epifluoreszenzmikroskop unter Verwendung eines FITC-Filters (495-519 nm Anregung) abgebildet. Die Anzahl der Erzgänge und die Mächtigkeit des Primordiums wurden mit den Mehrpunkt- bzw. Freihandlinienwerkzeugen in FIJI/ImageJ quantifiziert. (B) Ein Maissämling, bei dem die oberen Blattquirle mit einer Abisolierzange entfernt wurden, um die Spitze des vierten Blattes freizulegen (L4). Die Klammer erstreckt sich über die projizierte Primordiumlänge, wobei die gestrichelte Linie die mittlere Länge anzeigt. (C) Schematische Darstellung der verschiedenen Primordiumformen von P5 bis P7, die veranschaulicht, wie die Aderzahl und die Primordiumbreite in der mittleren Länge (horizontale gestrichelte Linie) je nach Entwicklungsstadium des Primordiums variieren können. (D,E) Messung der Primordiumbreite (D) und der Venenzahl (E) im mittleren Längenabschnitt des L4 von normal und vt2. Die Trendlinie stellt gleitende 10-mm-Durchschnittswerte der Messungen ± Standardfehler des Mittelwerts (REM) dar. (F-I) Repräsentative konfokale Bilder von Querschnitten von Blattprimordien, die Kombinationen aus PIN1a-YFP, Auxin-Response-Reporter, DR5-RFP, Cytokinin-Response-Reporter, TCS-Tomate, Gibberellinsäure-responsivem Marker, GAR2-YFP und mDII-Venus, einer mutierten Version des Auxin-Signaleingangsreporters DII-Venus, exprimieren. Die FP-Kanäle werden in jedem Bild mit dem Hellfeldkanal überlagert. Maßstabsbalken = 200 μm (A); 10 mm (B); 100 μm (F-I). Diese Abbildung wurde mit Genehmigung von Robil und McSteen24 modifiziert und reproduziert. Abkürzungen: DAG = Tage nach der Keimung; P = Plastochron; VT2 = verschwindende Quaste2; PCS = prokambialer Strang; YFP = gelb fluoreszierendes Protein; FITC = Fluorescein-Isothiocyanat; RFP = rot fluoreszierendes Protein; PIN1a-YFP = p Zm PIN1a::ZmPIN1a:YFP; DR5-RFP = DR5rev::mRFPer; TCS-Tomate = TCSv2::NLS-tdTomato; GAR2-YFP = p Zm GAR2::ZmGAR2:YFP; mDII-Venus = pZmUbi:mDII:YFP-NLS. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Whole-Mount-Analyse von Maisblatt-Primordien
Wir folgten Protokollabschnitt 3, um die FP-Expression in ganzblättrigen Mounts von Maisblattprimordien zu visualisieren und zu analysieren (Abbildung 5). Durch die Visualisierung von Venenmustern mit PIN1a-YFP fanden wir heraus, dass die Bildung von Venen im gesamten Primordium während früher Plastochronen auftritt, dieser Prozess jedoch später in der Entwicklung in den proximalen Regionen eingeschränkt wird (Abbildung 5A)24. Komplementär zu den Querschnittsanalysen haben die Ganzblattanalysen gewebe- und stadienspezifische Muster der Hormonantwort während der Venenbildung aufgezeigt24. Ein Beispiel ist das Expressionsmuster des CK-Antwortreporters TCSv2::NLS-tdTomato37 (TCS-Tomate) relativ zur PIN1a-YFP-Expression (Abbildung 5B)24. In Anlehnung an Protokollabschnitt 3 konnten wir sowohl qualitative als auch quantitative Analysen der FP-Expression in den Blattprimordien durchführen (Abbildung 5D-F; unveröffentlichte Daten). Wir untersuchten die Expressionsmuster eines Auxin-Antwort-Reporters, DR5rev::mRFPer41 (DR5-RFP), und eines GA-responsiven Markers, p Zm GAR2::ZmGAR2:YFP39 (GAR2-YFP), in Ganzblatt-Montierungen von Einzel- und Doppelmutanten von vt2 und Zwergpflanze3 (d3), einer GA-defizienten Mutante56 (Abbildung 5D). Wir verglichen auch das relative Ausmaß der Zellproliferation zwischen normalen und vt2-Blattprimordien mit pZm Cyclin-D2B::ZmCyclin-D2B:YFP 42 (Cyclin-D2B-YFP), das ein Marker für den G1/S-Übergang im Zellzyklus 57 ist (Abbildung 5E,F). Während es keinen signifikanten Unterschied zwischen normal und vt2 gab, stimmte die Cyclin-D2B-YFP-Expression mit dem bekannten Zellproliferationsprofil der frühen Plastochronenüberein 31. Wir kommen zu dem Schluss, dass Protokollabschnitt 3 eine effektive Methode zur Analyse ganzer Mounts von Maisblattprimordien ist, die aufgrund ihrer gerollten Morphologie schwer abzubilden sind.
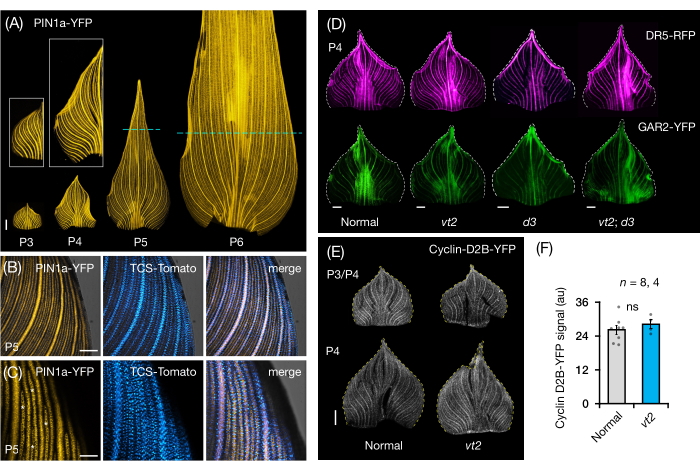
Abbildung 5: Repräsentative Ergebnisse für die Whole-Mount-Analyse von Maisblattprimordien. (A) Repräsentative Fluoreszenzbilder von Blattprimordien von 7 DAG-Maiskeimlingen, die sich entwickelnde Adern und prokambiale Stränge zeigen, die durch den Auxin-Efflux-Proteinmarker PIN1a-YFP markiert sind. P3-P6 Primordien wurden aus der Basis herausgeschnitten, abgerollt und mit der adaxialen Seite nach oben abgeflacht. Die Einschübe zeigen Nahaufnahmen von P3 und P4. In P5 und P6 markieren gestrichelte Linien das distale Ende der proliferativen Zonen, in denen sich die meisten prokambialen Stränge noch entwickeln und ausdehnen. (B,C) Repräsentative konfokale Bilder zeigen die Expression von PIN1a-YFP und Cytokinin-Response-Reporter, TCS-Tomato, in der proximalen Marginalregion eines P5-Primordiums. (D) Repräsentative Fluoreszenzbilder von P4-Primordia, die die Expression des Auxin-Antwort-Reporters DR5-RFP und des Gibberellinsäure-responsiven Markers GAR2-YFP in normalen und Einzel- und Doppelmutanten von verschwindenden Quasten2 und Zwergpflanzen3 zeigen. (E) Repräsentative konfokale Bilder von P3 und/oder P4, die die Expression von Cyclin-D2B-YFP, einem Reporter für den G1/S-Übergang im Zellzyklus, zeigen. (F) Relative Mengen der Zellproliferation in P3/P4-Blattprimordien von normal und vt2, quantifiziert durch Messung der integrierten Dichte des Cyclin-D2B-YFP-Signals über die Fläche des Primordiums mit ImageJ/FIJI. Die Balken stellen die Mittelwerte ± Standardfehler des Mittelwerts dar. Maßstabsbalken = 500 μm (A,D,E); 200 μm (B); 100 μm (C). Abbildung 4A-C wurde mit Genehmigung von Robil und McSteen24 modifiziert und reproduziert, während Abbildung 4D-F unveröffentlichte Daten der Autoren sind. Abkürzungen: DAG = Tage nach der Keimung; P = Plastochron; VT2 = Verschwindende Quaste2; D3 = Zwergpflanze3; YFP = gelb fluoreszierendes Protein; RFP = rot fluoreszierendes Protein; PIN1a-YFP = p Zm PIN1a::ZmPIN1a:YFP; TCS-Tomate = TCSv2::NLS-tdTomato; DR5-RFP = DR5rev::mRFPer; GAR2-YFP = p Zm GAR2::ZmGAR2:YFP; Cyclin-D2B-YFP = p Zm Cyclin-D2B::ZmCyclin-D2B:YFP; ns = kein signifikanter Unterschied; au = beliebige Einheit. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzungsdossier 1: Beispiele für Blattentwicklungsstadien bei Maiskeimlingen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S1: Pflanzenproben und Materialien, die in den Protokollen verwendet werden. (A,B) Ein Maissämling 7-8 DAG, bei dem der obere Blattquirl mit einer Abisolierzange entfernt wurde. (B) Der Einsatz zeigt eine Nahaufnahme des Triebes mit dem belichteten P6-Primordium. (C-G) Triebe von Maissämlingen, bei denen die oberen Quirle für die Querschnittsanalyse (C,D) und die umgebenden Blätter für die Analyse der gesamten Montage (E-G) vollständig entfernt wurden. (H) Eine Rolle Polyurethan-Gel-klares, doppelseitiges Nano-Klebeband. (I,J) P6-Blattprimordium (I) und proximaler Bereich des P8-Blattes (J) abgerollt und mit dem Nanoband auf Glasobjektträger montiert. (K) Ein Objektträger aus Glas mit einem abgerollten Blattprimordium, das auf dem Tisch eines Epifluoreszenzmikroskops montiert ist. Abkürzung: DAG = Tage nach der Keimung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Wir stellen zwei Methoden vor, um Maisblatt-Primordien für die zelluläre Bildgebung zu präparieren. Die erste Methode (Protokollteil 2) ermöglicht die Messung des Primordiums für die Querschnittsanalyse, während die zweite Methode (Protokollabschnitt 3) das Abrollen und Abflachen des Primordiums für die Analyse der gesamten Montierung ermöglicht. Diese Methoden erleichtern die zelluläre Bildgebung von FPs in Maisblattprimordien24 (wie in Abbildung 4 und Abbildung 5 dargestellt) und bieten einfache Lösungen für die Herausforderungen bei der Bildgebung von sich entwickelnden Maisblättern. Protokollabschnitt 2 reduziert die Präparierzeit und verbessert die Probenahmegenauigkeit, indem die Primordien vor dem Schneiden gemessen werden, anstatt sich nur auf die Staging-Parameterzu verlassen 9,16. Mit kommerziell erhältlichem Nanotape löst Protokollabschnitt 3 das seit langem bestehende Problem der Abbildung ganzblättriger Primordien in Mais. Dieses Protokoll verbessert die bisherige Methode, bei der Dialyseschläuche 31 verwendet wurden, und ist eine viel billigere Alternative zu CT und MRT11,32,33. Wenn es jedoch darum geht, anatomische Merkmale von Blättern zu visualisieren und optimale Ergebnisse zu erzielen, weisen beide Protokolle einige Einschränkungen auf, die in Tabelle 2 beschrieben und im Folgenden näher erläutert werden.
In Protokollabschnitt 2 hatten wir Schwierigkeiten, Zellumrisse in dicken Querschnitten der Blattprimordien sichtbar zu machen, und die Gegenfärbung mit Zellwand- oder Plasmamembran-bindenden Fluoreszenzfarbstoffen lieferte keine zufriedenstellenden Ergebnisse. Zum Beispiel lieferte FM 4-64 suboptimale Ergebnisse im Vergleich zum FP-Marker der Plasmamembran, p Zm PIP2-1::ZmPIP2-1:CFP39 (PIP2-1-CFP; Abbildung 3A-D). Um diese Einschränkung zu überwinden, empfehlen wir die Verwendung eines Vibratoms, um dünnere Gewebeschnitte (~0,1 mm)58 zu erzeugen, was eine lebendige Hellfeld-Abbildung von Zellumrissen oder die Optimierung des Gegenfärbeprotokolls ermöglicht 47,59.
In Protokollabschnitt 3 besteht die Haupteinschränkung darin, dass es schwierig ist, den Flügel ohne Riss, Beschädigung oder Luftblasen zu montieren, wie in den Protokollschritten 3.2.5-3.2.6 beschrieben (Abbildung 3E-K). Da das Maisblatt beidseitig symmetrisch ist, kann für die Visualisierung9 eine halbblättrige Halterung anstelle einer ganzblättrigen Halterung ausreichen. Dazu kann das Primordium nach dem Abrollen bis zur Mittelrippe mit einer Rasierklinge entlang der Längsachse geschnitten werden, so dass nur die Hälfte des Blattes montiert werden kann. Eine weitere Einschränkung des Protokollabschnitts 3 besteht darin, dass die Dicke des Blattes die optische Auflösung des Fluorophorsignals während der Tiefenbildgebung begrenzen kann. Um dieses Problem zu lösen, ist es möglich, eine Gewebereinigungstechnik60 anzuwenden. Wir stellten jedoch fest, dass ClearSee61, ein häufig verwendetes Clearing-Reagenz für die Bildgebung von Pflanzengewebe, nicht mit dem Protokoll kompatibel ist, da es dazu führt, dass sich die Probe und das Deckglas vom Nanoband lösen. Eine mögliche Lösung für dieses Problem könnte darin bestehen, eine semipermeable Membran31 über die Blattprobe aufzubringen, so dass sie mit der Clearing-Lösung behandelt werden kann, während sie durch das Nanoband an Ort und Stelle gehalten wird. Eine solche Methode, die es ermöglicht, flüssige Lösungen auf das abgerollte Blatt aufzubringen, könnte auch für Whole-Mount-RNA-in-situ-Hybridisierungs- und Immunlokalisierungstechniken verwendet werden, die bisher für die Entwicklung von Maisblütenständen, nicht aber für ganzblättrige Primordien optimiert wurden 62,63.
Wir haben Protokolle für Mais beschrieben, der bereits im Keimlingsstadium große Blattprimordien hat. Andere Grasarten mit viel kleineren Blattprimordien, wie Reis, Gerste, Weizen, Setaria und Brachypodium 16,23,64,65,66, erfordern möglicherweise den Einsatz zusätzlicher Präzisionswerkzeuge, um diese Protokolle effektiv anzuwenden. Darüber hinaus waren diese Protokolle nicht für die Bildgebung lebender Zellen gedacht, die dynamische Prozesse der Gewebebildung und zellulärer Reaktionen in Echtzeit erfasst. Da jedoch Fluoreszenzsonden, Bildgebungstechnologien und Rechenkapazitäten bei der Bildgebung lebender Zellen für Pflanzen67 weiter voranschreiten, könnte die zukünftige Forschung auf diesen Protokollen aufbauen, um Strategien zur Bildgebung lebender Zellen zu entwickeln, die auf die einzigartigen Eigenschaften von Grasblatt-Primordien zugeschnitten sind.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren danken der Maize Genetics Cooperation, dem Maize Cell Genomics Project, Dave Jackson (Cold Spring Harbor Laboratory, NY), Anne W. Sylvester (Marine Biological Laboratory, University of Chicago, IL), Andrea Gallavotti (Rutgers University, NJ) und Carolyn G. Rasmussen (University of California, Riverside) für die Bereitstellung der mutierten und transgenen Bestände sowie Robert F. Baker und Alexander Jurkevich vom Advanced Light Microscopy Core an der University of California. Missouri-Columbia für ihre Unterstützung bei der konfokalen Mikroskopie. JMR wurde durch das J. William Fulbright Fellowship, den Diane P. and Robert E. Sharp Fund und das Plant Genome Research Program (IOS-1546873) der National Science Foundation unterstützt. CDTC, CMRV, EDCDP und RJRR werden durch das Stipendienprogramm des Ateneo College unterstützt. CDTC, EDCDP und RJRR werden durch das DOST-SEI S&T Undergraduate-Stipendium unterstützt. Das DODL wird durch das Akademische Stipendium von P. Thomas Steinbugler SJ unterstützt. RJRR wird von Aiducation International – Pathways to Higher Education Scholarship unterstützt. Diese Arbeit wurde von der School of Science and Engineering und der Rizal Library der Ateneo de Manila University unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Acrylic Gel Clear Double Sided Nano Tape 16.5 ft x 1.2 in, 2 mm thick | EZlifego Store (Amazon) | B07YB1ZXG6 | 1 roll |
| Bellucci Pick Curved micro probe 16.8 cm, 6.6 in | Bausch & Lomb | N1692 9 | 1 pc |
| Clayman guide microprobe Sinskey hook angled shaft, 11.6 cm, 4.6 in | Storz Opthalmic Instruments | E0542 | 1 pc |
| Dental Probe, Bent Needle, 14 cm (5.5 in) | Ted Pella | 13553 | 1 pc |
| DOWELL 10-22 AWG Wire Stripper | Dowell Store (Amazon) | 10-22 AWG | 1 pc |
| Feather Double Edge Carbon Steel Blades | Ted Pella | 121-9 | pkg/10; for fine sectioning |
| Frosted End Glass Microscope Slides, 75 mm x 25 mm x 1-1.2 mm | Ted Pella | 260442 | pkg/144 |
| GEM Single Edge, Stainless Steel Uncoated Blades | Ted Pella | 121-1 | box/200; for general cutting/sectioning |
| Glycerol | Thermo Scientific | PI17904 | 1 liter |
| ImageJ/FIJI with EDF plugin (version 17.05.2021) and Grid/Collection Stitching plugin | National Institutes of Health (NIH) USA | version 2.9.0/1.54s | The EDF plugin was developed by Alex Prudencio, Jesse Berent, and Daniel Sage for the Biomedical Imaging Group, École Polytechnique Fédérale de Lausanne (EPFL; http://bigwww.epfl.ch/demo/edf/). The grid/collection stitching software was developed by Stephan Preibisch for the Max Planck Institute of Molecular Cell Biology and Genetics (MPI-CBG). |
| Kimwipes Ex-L Small 111.76 mm x 213.36 mm | Kimtech Science | 34155 | box/280 ply |
| Micro Cover Glasses, 22 mm x 22 mm x 0.13 - 0.16 mm thick | Ted Pella | 260140 | 1 ounce |
| PU Gel Clear Double Sided Nano Tape 29.5 ft x 1.18 in, 1 mm thick | Yecaye Store (Amazon) | L354 W1.18 | 2 rolls |
| Superslip Cover Glasses, 24 mm x 50 mm x 0.13 - 0.16 mm thick | Ted Pella | 260166 | 1 ounce |
| Superslip Cover Glasses, 24 mm x 60 mm x 0.13 - 0.16 mm thick | Ted Pella | 260168 | 1 ounce |
| Tempered Glass Cutting Board | Hacaroa (Amazon) | B09XMXBT5S | 4 pc |
Referenzen
- McSteen, P., Kellogg, E. A. Molecular, cellular, and developmental foundations of grass diversity. Science. 377 (6606), 599-602 (2022).
- Wang, P., Vlad, D., Langdale, J. A. Finding the genes to build C4 rice. Current Opinion in Plant Biology. 31, 44-50 (2016).
- Wang, P., et al. Candidate regulators of early leaf development in maize perturb hormone signalling and secondary cell wall formation when constitutively expressed in rice. Scientific Reports. 7 (1), 4535(2017).
- Perico, C., Tan, S., Langdale, J. A. Developmental regulation of leaf venation patterns: Monocot versus eudicots and the role of auxin. New Phytologist. 234 (3), 783-803 (2022).
- Wang, P., Kelly, S., Fouracre, J. P., Langdale, J. A. Genome-wide transcript analysis of early maize leaf development reveals gene cohorts associated with the differentiation of C4 Kranz anatomy. The Plant Journal. 75 (4), 656-670 (2013).
- Liu, W. Y., et al. Regulators of early maize leaf development inferred from transcriptomes of laser capture microdissection (LCM)-isolated embryonic leaf cells. Proceedings of the National Academy of Sciences. 119 (35), e2208795119(2022).
- Liu, W. Y., et al. Anatomical and transcriptional dynamics of maize embryonic leaves during seed germination. Proceedings of the National Academy of Sciences. 110 (10), 3979-3984 (2013).
- Strable, J., Nelissen, H. The dynamics of maize leaf development: Patterned to grow while growing a pattern. Current Opinion in Plant Biology. 63, 102038(2021).
- Reynolds, J. O., Eisses, J. F., Sylvester, A. W. Balancing division and expansion during maize leaf morphogenesis: Analysis of the mutant, warty-1. Development. 125 (2), 259-268 (1998).
- Richardson, A. E., et al. Evolution of the grass leaf by primordium extension and petiole-lamina remodeling. Science. 374 (6573), 1377-1381 (2021).
- Johnston, R., Leiboff, S., Scanlon, M. J. Ontogeny of the sheathing leaf base in maize (Zea mays). New Phytologist. 205 (1), 306-315 (2015).
- Sharman, B. C. Developmental anatomy of the shoot of Zea mays L. Annals of Botany. 6 (22), 245-282 (1942).
- Johnston, R., et al. Transcriptomic analyses indicate that maize ligule development recapitulates gene expression patterns that occur during lateral organ initiation. The Plant Cell. 26 (12), 4718-4732 (2014).
- Sharman, B. C. Leaf and bud initiation in the Gramineae. Botanical Gazette. 106 (3), 269-289 (1945).
- Nelissen, H., et al. A local maximum in gibberellin levels regulates maize leaf growth by spatial control of cell division. Current Biology. 22 (13), 1183-1187 (2012).
- Itoh, J., et al. Rice plant development: From zygote to spikelet. Plant and Cell Physiology. 46 (1), 23-47 (2005).
- Ritchie, S. W., Hanway, J. J., Benson, G. O. How a corn plant develops. Iowa State University of Science and Technology. , (1986).
- Freeling, M., Walbot, V. The Maize Handbook. , Springer Science & Business Media. (2013).
- Freeling, M., Hake, S. Developmental genetics of mutants that specify knotted leaves in maize. Genetics. 111 (3), 617-634 (1985).
- Durbak, A. R., et al. Transport of boron by the tassel-less1 aquaporin is critical for vegetative and reproductive development in maize. The Plant Cell. 26 (7), 2978-2995 (2014).
- Langdale, J. A., Lane, B., Freeling, M., Nelson, T. Cell lineage analysis of maize bundle sheath and mesophyll cells. Developmental Biology. 133 (1), 128-139 (1989).
- Bosabalidis, A. M., Evert, R. F., Russin, W. A. Ontogeny of the vascular bundles and contiguous tissues in the maize leaf blade. American Journal of Botany. 81 (6), 745-752 (1994).
- Junqueira, N. E. G., et al. Anatomy and ultrastructure of embryonic leaves of the C4 species Setaria viridis. Annals of Botany. 121 (6), 1163-1172 (2018).
- Robil, J. M., McSteen, P. Hormonal control of medial-lateral growth and vein formation in the maize leaf. New Phytologist. 238 (1), 125-141 (2023).
- Donnelly, P. M., Bonetta, D., Tsukaya, H., Dengler, R. E., Dengler, N. G. Cell cycling and cell enlargement in developing leaves of Arabidopsis. Developmental Biology. 215 (2), 407-419 (1999).
- Govindaraju, P., Verna, C., Zhu, T., Scarpella, E. Vein patterning by tissue-specific auxin transport. Development. 147 (13), (2020).
- Linh, N. M., Scarpella, E. Leaf vein patterning is regulated by the aperture of plasmodesmata intercellular channels. PLoS Biology. 20 (9), e3001781(2022).
- Verna, C., Ravichandran, S. J., Sawchuk, M. G., Linh, N. M., Scarpella, E. Coordination of tissue cell polarity by auxin transport and signaling. eLife. 8, e51061(2019).
- Sawchuk, M. G., Head, P., Donner, T. J., Scarpella, E. Time-lapse imaging of Arabidopsis leaf development shows dynamic patterns of procambium formation. New Phytologist. 176 (3), 560-571 (2007).
- Linh, N. M., Scarpella, E. Confocal imaging of developing leaves. Current Protocols. 2 (1), e349(2022).
- Poethig, R. S., Szymkowiak, E. J. Clonal analysis of leaf development in maize. Maydica. 40, 67-76 (1995).
- Sprangers, K., Thys, S., van Dusschoten, D., Beemster, G. T. S. Gibberellin enhances the anisotropy of cell expansion in the growth zone of the maize leaf. Frontiers in Plant Science. 11, 1163(2020).
- Tsuda, K., et al. KNOTTED1 cofactors, BLH12 and BLH14, regulate internode patterning and vein anastomosis in maize. The Plant Cell. 29 (5), 1105-1118 (2017).
- Schindelin, J., et al. FIJI: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Plant Care Protocols - Maize. The Donald Danforth Plant Science Center's Plant Growth Facility. , Available from: https://www.danforthcenter.org/our-work/core-facilities/plant-growth/ (2019).
- Mir, R., et al. A DII domain-based auxin reporter uncovers low auxin signaling during telophase and early G1. Plant Physiology. 173 (1), 863-871 (2017).
- DeBlasio, S. L., Sylvester, A. W., Jackson, D. Illuminating plant biology: Using fluorescent proteins for high-throughput analysis of protein localization and function in plants. Briefings in Functional Genomics. 9 (2), 129-138 (2010).
- Wu, Q., Luo, A., Zadrozny, T., Sylvester, A., Jackson, D. Fluorescent protein marker lines in maize: Generation and applications. The International Journal of Developmental Biology. 57 (6-8), 535-543 (2013).
- Mohanty, A., et al. Advancing cell biology and functional genomics in maize using fluorescent protein-tagged lines. Plant Physiology. 149 (2), 601-605 (2009).
- Baker, R. F., et al. Sucrose transporter ZmSut1 expression and localization uncover new insights into sucrose phloem loading. Plant Physiology. 172 (3), 1876-1898 (2016).
- Gallavotti, A., Yang, Y., Schmidt, R. J., Jackson, D. The relationship between auxin transport and maize branching. Plant Physiology. 147 (4), 1913-1923 (2008).
- Krishnakumar, V., et al. A maize database resource that captures tissue-specific and subcellular-localized gene expression, via fluorescent tags and confocal imaging (Maize Cell Genomics Database). Plant and Cell Physiology. 56 (1), 12(2015).
- Snapp, E. L. Fluorescent proteins: A cell biologist's user guide. Trends in Cell Biology. 19 (11), 649-655 (2009).
- Geng, Y., Zhou, Y. Confocal live imaging of shoot apical meristems from different plant species. Journal of Visualized Experiments. (145), e59369(2019).
- Stanislas, T., Hamant, O., Traas, J. In-vivo analysis of morphogenesis in plants. Methods in Cell Biology. 139, Elsevier. 203-223 (2017).
- Fodor, E., Ayaydin, F. Fluorescent probes and live imaging of plant cells. Advances in Plant Ecophysiology Techniques. , Springer. 241-251 (2018).
- Grandjean, O., et al. In vivo analysis of cell division, cell growth, and differentiation at the shoot apical meristem in Arabidopsis. The Plant Cell. 16 (1), 74-87 (2004).
- Conklin, P. A., Johnston, R., Conlon, B. R., Shimizu, R., Scanlon, M. J. Plant homeodomain proteins provide a mechanism for how leaves grow wide. Development. 147 (20), (2020).
- Shaw, S. L. Imaging the live plant cell. The Plant Journal. 45 (4), 573-598 (2006).
- Klaus, A. V., Schawaroch, V., Frischmann, K. J. Confocal imaging and three-dimensional visualization of thick autofluorescent specimens. Methods in Molecular Biology. 1075, 213-225 (2014).
- Forster, B., Van De Ville, D., Berent, J., Sage, D., Unser, M. Extended depth-of-focus for multi-channel microscopy images: a complex wavelet approach. 2004 2nd IEEE International Symposium on Biomedical Imaging: Nano to Macro (IEEE Cat No. 04EX821). IEEE. , 660-663 (2004).
- Extended Depth of Field. EPFL Biomedical Imaging Group. , Available from: http://bigwww.epfl.ch/demo/edf/ (2022).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Phillips, K. A., et al. vanishing tassel2 encodes a grass-specific tryptophan aminotransferase required for vegetative and reproductive development in maize. The Plant Cell. 23 (2), 550-566 (2011).
- Robil, J. M., et al. GrasVIQ: An image analysis framework for automatically quantifying vein number and morphology in grass leaves. The Plant Journal. 107 (2), 629-648 (2021).
- Helliwell, C. A., Chandler, P. M., Poole, A., Dennis, E. S., Peacock, W. J. The CYP88A cytochrome P450, ent-kaurenoic acid oxidase, catalyzes three steps of the gibberellin biosynthesis pathway. Proceedings of the National Academy of Sciences. 98 (4), 2065-2070 (2001).
- Gutierrez, R., Quiroz-Figueroa, F., Vazquez-Ramos, J. M. Maize cyclin D2 expression, associated kinase activity and effect of phytohormones during germination. Plant and Cell Physiology. 46 (1), 166-173 (2005).
- Atkinson, J. A., Wells, D. M. An updated protocol for high throughput plant tissue sectioning. Frontiers in Plant Science. 8, 1721(2017).
- Lux, A., Morita, S., Abe, J., Ito, K. An improved method for clearing and staining free-hand sections and whole-mount samples. Annals of Botany. 96 (6), 989-996 (2005).
- Heriche, M., Arnould, C., Wipf, D., Courty, P. E. Imaging plant tissues: Advances and promising clearing practices. Trends in Plant Science. 27 (6), 601-615 (2022).
- Kurihara, D., Mizuta, Y., Sato, Y., Higashiyama, T. ClearSee: A rapid optical clearing reagent for whole-plant fluorescence imaging. Development. 142 (23), 4168-4179 (2015).
- Chuck, G., Muszynski, M., Kellogg, E., Hake, S., Schmidt, R. J. The control of spikelet meristem identity by the branched silkless1 gene in maize. Science. 298 (5596), 1238-1241 (2002).
- Tran, T. M., et al. An optimized whole-mount immunofluorescence method for shoot apices. Current Protocols. 1 (4), e101(2021).
- O'Connor, D. L. PINs Lost and PINs Gained: Auxin-Transport Mediated Patterning in the Grasses. University of California, Berkeley. , Doctoral Dissertation (2012).
- Sharman, B. C., Hitch, P. A. Initiation of procambial strands in leaf primordia of bread wheat, Triticum aestivum L. Annals of Botany. 31 (2), 229-243 (1967).
- Serra, L., Tan, S., Robinson, S., Langdale, J. A. Flip-flap: A simple dual-view imaging method for 3D reconstruction of thick plant samples. Plants. 11 (4), 506(2022).
- Colin, L., et al. Imaging the living plant cell: From probes to quantification. The Plant Cell. 34 (1), 247-272 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten