Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Methodik zur metabolischen Inaktivierung von Bakterien für die Caenorhabditis elegans-Forschung
In diesem Artikel
Zusammenfassung
Die Nahrungsquelle für Caenorhabditis elegans im Labor sind lebende Escherichia coli. Da Bakterien metabolisch aktiv sind, stellen sie eine Störvariable in metabolischen und medikamentösen Studien an C. elegans dar. Ein detailliertes Protokoll zur metabolischen Inaktivierung von Bakterien mit Paraformaldehyd ist hier beschrieben.
Zusammenfassung
Caenorhabditis elegans ist ein gängiger Modellorganismus für die Erforschung von Genetik, Entwicklung, Alterung, Stoffwechsel und Verhalten. Da C. elegans sich von lebenden Bakterien ernährt, kann die Stoffwechselaktivität ihrer Nahrungsquelle Experimente verwirren, die nach den direkten Auswirkungen verschiedener Eingriffe auf den Wurm suchen. Um die verwirrenden Auswirkungen des bakteriellen Stoffwechsels zu vermeiden, haben die Forscher von C. elegans mehrere Methoden zur metabolischen Inaktivierung von Bakterien eingesetzt, darunter ultraviolette (UV)-Bestrahlung, Hitzeabtötung und Antibiotika. Die UV-Behandlung hat einen relativ geringen Durchsatz und kann nicht in der Flüssigkultur eingesetzt werden, da jede Platte auf eine erfolgreiche bakterielle Abtötung untersucht werden muss. Eine zweite Behandlungsmethode, die Hitzeabtötung, wirkt sich negativ auf die Textur und die Nährstoffqualität der Bakterien aus, was zum Entwicklungsstillstand von C. elegans führt. Schließlich kann die Behandlung mit Antibiotika die Physiologie von C. elegans direkt verändern und das Bakterienwachstum verhindern. Dieses Manuskript beschreibt eine alternative Methode zur metabolischen Inaktivierung von Bakterien mittels Paraformaldehyd (PFA). Bei der PFA-Behandlung werden Proteine in Bakterienzellen vernetzt, um die Stoffwechselaktivität zu verhindern und gleichzeitig die Zellstruktur und den Nährstoffgehalt zu erhalten. Diese Methode hat einen hohen Durchsatz und kann in Flüssigkulturen oder festen Platten verwendet werden, da das Testen einer Platte mit PFA-behandelten Bakterien auf Wachstum die gesamte Charge validiert. Die metabolische Inaktivierung durch PFA-Behandlung kann verwendet werden, um die verwirrenden Auswirkungen des bakteriellen Stoffwechsels auf Studien zur Arzneimittel- oder Metaboliten-Supplementierung, Stressresistenz, Metabolomik und Verhalten bei C. elegans zu eliminieren.
Einleitung
Caenorhabditis elegans wurde ursprünglich 1965 als Modellorganismusvorgeschlagen 1 und hat sich seitdem in Studien zu Genetik, Entwicklung, Verhalten, Altern und Stoffwechsel 2durchgesetzt. Aufgrund ihrer großen Brutgröße und transparenten Kutikula eignet sich C. elegans besonders gut für das Hochdurchsatz-Screening mit fluoreszierenden Reportern3. Ihr kurzer Lebenszyklus, ihre hermaphroditische Vermehrung und ihre genetische Homologie mit dem Menschen machen C. elegans auch zu einem wertvollen Modellsystem für Studien zur Entwicklung4 und zur Alterungsbiologie5. Darüber hinaus sind C. elegans relativ pflegeleicht. Würmer können in Flüssigkultur oder auf festen Agarplatten gezüchtet werden und ernähren sich von lebenden Escherichia coli OP50-Bakterien4.
Die Lebendnahrungsquelle von C. elegans kann jedoch Studien über den Stoffwechsel, die Einnahme von Medikamenten und das Verhalten verfälschen. Da lebende Bakterien ihren eigenen Stoffwechsel haben, verändern Versuchsbedingungen, die auf die Bakterien einwirken, auch die Nährstoffe und Metaboliten, die den Würmern zur Verfügung stehen. Zum Beispiel haben Unterschiede in den bakteriellen Eisen-, Aminosäure- und Folatkonzentrationen unterschiedliche Auswirkungen auf die Entwicklung, Physiologie und Lebensdauer von C. elegans 6. Viele gängige Laborpraktiken können solche Veränderungen in der Nährstoffzusammensetzung und den von OP50 produzierten Metaboliten hervorrufen. Insbesondere die Exposition gegenüber 5-Fluor-2'-Desoxyuridin (FUdR), einer Verbindung, die üblicherweise zur Verhinderung der Fortpflanzung bei C. elegans verwendet wird, löst breite Veränderungen in der OP50-Genexpression aus, einschließlich der Aminosäurebiosynthesewege7. Lebende Bakterien können auch Studien verfälschen, in denen C. elegans mit kleinen Molekülen ergänzt wird, da Bakterien die Wirkstoffe teilweise oder vollständig verstoffwechseln können. Darüber hinaus können die Auswirkungen dieser kleinen Moleküle auf die Bakterien wiederum die Physiologie von C. elegans verändern, wie mit dem lebensverlängernden Medikament Metformin8 berichtet wurde. Schließlich können lebende Bakterien die Umgebung des Wurms auf eine Weise verändern, die das Verhalten ändert, wie z. B. die Sekretion attraktiver Geruchsstoffe9, die Produktion exogener Neuromodulatoren10 und die Schaffung von Sauerstoffgradienten in einem dichten Bakterienrasen11.
Um die verwirrenden Auswirkungen des bakteriellen Stoffwechsels auf die Forschung an C. elegans abzuschwächen, wurden mehrere Methoden zur Abtötung von Bakterien entwickelt (Tabelle 1). Drei gängige Strategien zur Abtötung von OP50 sind UV-Bestrahlung, Hitzeabtötung und Antibiotikabehandlung. Obwohl sie unkompliziert und relativ kostengünstig sind, kann jede dieser Methoden unerwünschte Auswirkungen sowohl auf Bakterien als auch auf C. elegans haben. Die UV-Abtötung über einen UV-Vernetzer12 ist ein geringer Durchsatz, und die Geschwindigkeit ist durch die Anzahl der Platten begrenzt, die in den UV-Vernetzer passen können. Darüber hinaus kann die Wirksamkeit der UV-Abtötung von Platte zu Platte innerhalb einer Charge variieren, und das Testen auf Wachstum auf allen Platten kann in großen Experimenten schwierig werden. Die hitzeabtötende OP50 durch die Aussetzung der Kultur gegenüber Temperaturen von >60 °C bringt eine Reihe anderer Herausforderungen mit sich. Hohe Hitze kann die für den Wurm wichtigen Nährstoffe schädigen und die Zellstruktur von Bakterien zerstören, wodurch eine weichere Textur entsteht, die die Zeit, die Würmer mit dem Futter verbringen, verringert13. Diese Methode kann auch nicht während des gesamten Lebenszyklus von C. elegans angewendet werden, da Würmer, die mit hitzeabgetöteten Bakterien gefüttert werden, in einem frühen Entwicklungsstadium zum Stillstand kommen können13. Die Behandlung mit Antibiotika ist eine dritte gängige Methode zur Unterdrückung des bakteriellen Stoffwechsels14, aber Antibiotika können auch das Wachstum und den Stoffwechsel von Würmern verändern15.
Eine Lösung, um die metabolischen Auswirkungen lebender Bakterien zu eliminieren und gleichzeitig die Bakterienstruktur und die essentiellen Nährstoffe zu erhalten, besteht darin, OP50 mit Paraformaldehyd (PFA)16 abzutöten. PFA ist ein Polymer aus Formaldehyd, das Proteine innerhalb von Zellen17 vernetzen kann, um die bakterielle Replikation zu verhindern, ohne interne Zellstrukturen wie die innere Plasmamembran18 zu zerstören. Aufgrund dieser Erhaltung der inneren Zellstruktur zeigen PFA-behandelte Bakterien kein Wachstum oder Stoffwechselaktivität, bleiben aber eine essbare und nährstoffreiche Nahrungsquelle für C. elegans16. Hier wird ein detailliertes Protokoll zur Verfügung gestellt, das zeigt, wie Bakterien mit Paraformaldehyd metabolisch inaktiviert werden können.
| Methode | Benötigte Materialien | Skalierbar? | Ernährungs? | Auswirkungen auf Worm? | ||||
| UV | UV-Vernetzer | Begrenzt durch: | Ja | Unterschiedliche Auswirkungen auf die Lebensdauer von NGM12, 23, 24 | ||||
| Anzahl der Platten, die in den UV-Vernetzer passen | Unterschiedliche Auswirkungen auf die Lebensdauer von FUdR24, 26, 27 | |||||||
| Bestrahlungszeit pro Platte | Verminderte Lebensmittelpräferenz16 | |||||||
| Fähigkeit, jede Platte auf Wachstum zu überprüfen8 | ||||||||
| Wärme | >60 °C Inkubator | Ja | Nein: zerstört die Zellwand, vermindert den Nährwert | Entwicklungsstillstand 13 | ||||
| Verminderte Lebensmittelpräferenz13 | ||||||||
| Verlängert die Lebensdauer des NGM31 | ||||||||
| Antibiotika | Antibiotika (Kanamycin, Carbenicillin, etc.) | Ja | Ja | Verzögert Wachstum und Entwicklung15 | ||||
| Verlängert die Lebensdauer in flüssigen Medien19 | ||||||||
| Verlängert die Lebensdauer des NGM15 | ||||||||
| PFA | 0,5 % Paraformaldehyd | Ja | Ja | Verkleinerung der kleinen Brut16 | ||||
| Geringe Erhöhung der Entwicklungszeit16 | ||||||||
| Verminderte Lebensmittelpräferenz16 | ||||||||
Tabelle 1. Vergleiche von Methoden zur Abtötung von OP50. UV-Abtötung, Hitzeabtötung, Antibiotikabehandlung und PFA-Behandlung haben unterschiedliche Auswirkungen auf den Ernährungszustand der Bakterien und die Gesundheit von Würmern, die mit behandelten Bakterien gefüttert werden. Diese Methoden zur replikativen Inaktivierung von E. coli unterscheiden sich auch in ihren benötigten Materialien und ihrer Skalierbarkeit.
Protokoll
1. Bakterien-Inokulation
- Bereiten Sie Luria-Brühe (LB) zu, indem Sie 10 g Trypton, 5 g Hefeextrakt und 10 g Natriumchlorid (NaCl) in 950 ml destilliertem Wasser auflösen.
- Stellen Sie den pH-Wert des LB auf 7,0 ein, indem Sie 5 M Natriumhydroxid (NaOH) hinzufügen. Dies sollte nur etwa 0,2 ml NaOH erfordern.
- Autoklavieren Sie das pH-angepasste LB-Medium in einem Flüssigkeitszyklus für 45 Minuten bei 15 psi. Lassen Sie die Lösung abkühlen und lagern Sie sie bei Raumtemperatur.
- Eine einzelne Bakterienkolonie wird in 100 ml LB in einem 500-ml-Erlenmeyerkolben inokuliert. Kultivieren Sie die Bakterien über Nacht in einem 37 °C heißen Schüttel-Inkubator.
- Abhängig von der Gesundheit der Bakterienkolonie, der Größe des Kolbens und der Geschwindigkeit, auf die der Schüttler eingestellt ist, kann die Zeit, die das Wachstum der Bakterien benötigt, variieren. Nach ~14 h ist die optische Dichte (OD) der Bakterien bei 600 nm (OD600) zu überprüfen.
- Entfernen Sie die Bakterien aus dem Schüttler, wenn der OD600 3,0 beträgt (1 x 109 koloniebildende Einheiten (KBE)/ml). Wenn der Außendurchmesser600 kleiner als 3,0 ist, wird der Kolben wieder in den Schüttelinkubator gegeben, bis der gewünschte Außendurchmesser erreicht ist.
- Aliquotieren Sie die Bakterien in konische 50-ml-Röhrchen und lagern Sie sie bei 4 °C oder fahren Sie mit dem nächsten Schritt fort.
2. Arbeiten mit Paraformaldehyd
HINWEIS: Die verwendete Konzentration von Paraformaldehyd (PFA) und die Dauer der Exposition können je nach Klima, Standort und Art der zu behandelnden Bakterien etwas variieren. Ein guter Ausgangspunkt für OP50 ist die Exposition gegenüber 0,5 % PFA für 1 h, während 0,25 % PFA für 1 h für HT115 ausreichend sein können.
- Bereiten Sie 32 % PFA-Brühe vor oder verwenden Sie eine im Handel erhältliche 32 %ige PFA-Lösung. Verwenden Sie die richtige persönliche Schutzausrüstung (PSA), wenn Sie mit PFA arbeiten. Tragen Sie Handschuhe und Augenschutz.
- Fügen Sie PFA in einen chemischen Abzug mit ausreichender Belüftung ein. Entsorgen Sie PFA-haltige Waschmittel und Spitzen in geeigneten Behältern für chemische Gefahren im Abzug.
3. Bakterielle Behandlung mit Paraformaldehyd
- Sobald die Bakterien einen OD600 von 3,0 erreicht haben, verwenden Sie eine serologische Pipette, um 50 ml in einen neuen 250-ml-Erlenmeyerkolben zu überführen. Bewahren Sie den Rest für eine Live-Kontrolle, eine simulierte Kontrolle (siehe Schritt 4) oder für die Behandlung mit PFA nach Bedarf auf.
- Vermeiden Sie es, von einem Kolben in einen anderen zu gießen, und achten Sie darauf, die Seiten des neuen Kolbens nicht mit Bakterien zu bespritzen. Kolonien an der Seite des Kolbens können eine niedrigere Dosis von PFA erhalten.
- In der Chemikalienhaube 781 μl 32 % PFA auf 50 ml Bakterien geben, um die Endkonzentration auf 0,5 % zu bringen. Entsorgen Sie die verwendete Spitze in einem Behälter für feste chemische Abfälle.
- Decken Sie den Kolben mit Folie ab und stellen Sie ihn für 1 h in den 37 °C heißen Schüttelinkubator. Nach 1 h nehmen Sie den Kolben aus dem Inkubator und fahren Sie mit Schritt 5 fort.
4. Simulierte Kontrolle
- Sobald die Bakterien einen OD600 von 3,0 erreicht haben, verwenden Sie eine serologische Pipette, um 50 ml der Bakterien in ein konisches 50-ml-Röhrchen zu überführen.
- Fahren Sie mit Schritt 5.3 fort, um die Waschschritte ähnlich wie bei der PFA-behandelten Gruppe abzuschließen.
5. Waschen der Bakterien, um PFA-Reste zu entfernen
- In der Chemikalienhaube werden die behandelten Bakterien mit einer serologischen Pipette aus dem Erlenmeyerkolben in ein konisches 50-ml-Röhrchen überführt. Die Verwendung einer serologischen Pipette anstelle des Ausgießens der Bakterien verhindert eine Kontamination durch Bakterienkolonien am Rand des Kolbens, die möglicherweise eine direkte Behandlung mit PFA vermieden haben.
- Behandelte Bakterien bei ca. 3000 x g für 20 min zentrifugieren. Entfernen Sie den Überstand, indem Sie ihn in einem Behälter für chemische Gefahren aus flüssigen Abfällen in der Chemikalienhaube entsorgen.
- Fügen Sie 25 ml LB und Vortex hinzu, um das Bakterienpellet zu resuspendieren (Wenn Sie das Röhrchen vollständig füllen, wird es schwieriger, das Pellet zu resuspendieren). Zentrifugation und Pellet-Resuspension 4x wiederholen.
- Resuspendieren Sie das Pellet in Volumina, die für verschiedene Assays optimal sind. Für Lifelife-Assays werden 60-mm-Platten mit 200 μl Bakterien in 10 ml LB resuspendiert. Die Resuspendierung der Bakterien in 10 ml LB führt zu einer 5-fachen Konzentration gegenüber der ursprünglichen 50-ml-Kultur.
- Lagern Sie die Bakterien bei 4 °C.
6. Qualitätskontrolle des Bakterienwachstums
- Nach dem abschließenden Waschen und Resuspension wird eine LB-Platte (mit einer sterilen Pipettenspitze) mit den vorbereiteten Bakterien bestrichen. Es empfiehlt sich, das zum Waschen und Resuspendieren verwendete LB ebenfalls auf eine separate Platte zu streichen, um sicherzustellen, dass das verwendete LB nicht kontaminiert wurde.
- Stellen Sie die Platten über Nacht in einen 37 °C warmen Inkubator. Prüfen Sie, ob es Wachstum gibt. Die Bakterien gelten als replikativ tot, wenn auf der LB-Platte keine Kolonien wachsen.
7. Qualitätskontrolle des bakteriellen Stoffwechsels mittels Respirometer
- Vergewissern Sie sich nach dem abschließenden Waschen und der Resuspension aus Schritt 5.4, dass die Bakterien metabolisch abgestorben sind, indem Sie die verfügbaren Instrumente wie Respirometer19,20 verwenden und die basale Sauerstoffverbrauchsrate (OCR) messen.
- M9-Lösung vorbereiten: 3 g einbasisches Kaliumphosphat (KH2PO 4), 6 g dibasisches Natriumphosphat (Na2HPO4) und 5 g Natriumchlorid (NaCl) in 950 ml destilliertem Wasser lösen. Autoklavieren Sie in einem Flüssigkeitszyklus 45 Minuten lang bei 15 psi und lassen Sie die Lösung dann auf Raumtemperatur abkühlen. 1 ml 1 M Magnesiumsulfat (MgSO4) zugeben und bei Raumtemperatur lagern.
- Befeuchten Sie die Respirometerkartusche: Geben Sie 200 μl Kalibrant in alle Vertiefungen einer 96-Well-Platte. Legen Sie die Kartusche in die 96-Well-Platte und inkubieren Sie sie über Nacht in einem 37 °C warmen Inkubator.
- Assay-Kalibrierung: Legen Sie am nächsten Tag die hydratisierte Kartusche in das Gerät und beginnen Sie mit der Kalibrierung.
- Aufbau der Assay-Testplatte: Mit einer neuen 96-Well-Platte 160 μl M9 und 40 μl der vorbereiteten Bakterien (1 x 109 KBE/ml) in die Testwells geben. Geben Sie 200 μl M9 in die 4 Eckvertiefungen, um sie als Blindvertiefungen zu verwenden. Fügen Sie 160 μl M9 und 40 μl LB hinzu, die zum Waschen und Resuspendieren verwendet werden, um sie als Negativkontrollen zu verwenden. Geben Sie 200 μl M9 in die restlichen Vertiefungen, die nicht verwendet werden.
- Führen Sie den Assay durch: Sobald die Kartuschenkalibrierung abgeschlossen ist, setzen Sie die Assay-Platte aus Schritt 7.5 zur Analyse in das Gerät ein. Die Einstellungen umfassen Schritte zum Mischen, Warten, Messen und Loopen. Die Ergebnisse werden als Sauerstoffverbrauchsrate (OCR) angezeigt. Die Bakterien sind metabolisch tot und einsatzbereit, wenn die OCR Null ist.
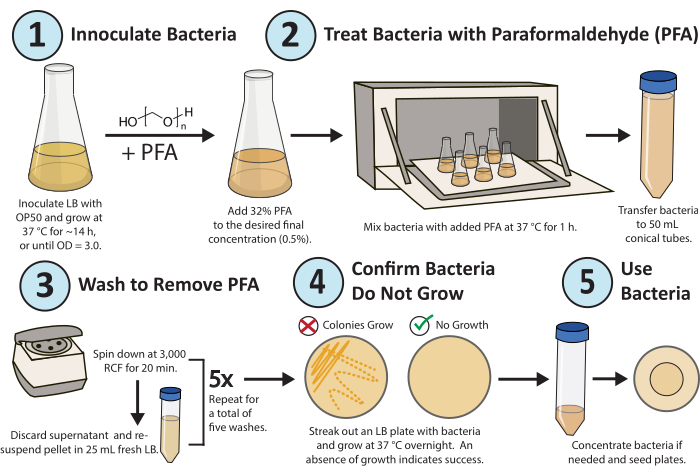
Abbildung 1. Arbeitsablauf für die Paraformaldehyd-Behandlung. Eine einzelne Kolonie von E. coli OP50-Bakterien wird über Nacht gezüchtet. PFA wird zu einer Endkonzentration von 0,5 % zugegeben und die PFA-behandelte Kultur wird 1 h bei 37 °C geschüttelt. Zum Schluss wird das PFA entfernt, indem die Kultur mit frischem LB 5x gewaschen wird. Um zu bestätigen, dass die behandelten Bakterien replizierend inaktiv sind, streifen Sie eine LB-Platte der behandelten Bakterien aus und wachsen Sie über Nacht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Ein detaillierter Workflow des Protokolls ist in Abbildung 1 dargestellt. Es wurde eine Hochdurchsatzmethode entwickelt und optimiert, um die bakterielle Replikation (Abbildung 2A) und den Stoffwechsel (Abbildung 2B) für Stoffwechsel- und Wirkstoffstudien in der C. elegans-Forschung unter Verwendung von Paraformaldehyd16 konsistent zu inaktivieren. Ziel war es, die niedrigste PFA-Konzentration und d...
Diskussion
Vorteile der PFA-Abtötung im Vergleich zu anderen bakterienabtötenden Methoden
Die PFA-Behandlung ist eine Hochdurchsatzmethode, um den bakteriellen Stoffwechsel zu verhindern und gleichzeitig eine nahrhafte Nahrungsquelle für C. elegans zu erhalten. Die Abtötung von Bakterien durch PFA-Behandlung hat mehrere Vorteile gegenüber anderen Methoden. Im Gegensatz zur UV-Behandlung, bei der jede Platte auf eine erfolgreiche Abtötung getestet werden muss, kann eine einzelne Platte aus einer Ch...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde von NIH R21AG059117 und den Paul F. Glenn Laboratories for Biology of Aging Research an der University of Michigan finanziert. SB wurde von T32AG000114 finanziert. ESK wurde von der NSF DGE 1841052 finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Aluminum Foil | Staples | 2549291 | |
| Bunsen burner | VWR | 470121-700 | |
| Cell Density Meter | Denville | 80-3000-45 | |
| Centrifuge | Eppendorg | 5430 | |
| Chemical fume hood | Labcono | 975050411384RG | |
| Conincal tubes (50 mL) | Fisher | 339652 | |
| Cuvettes | Fisher | 14-955-127 | |
| E. coli OP50 | CGC | OP50 | |
| Erlenmyer flasks | Fisher | 250 mL: FB501250 500 mL: FB501500 1000 mL: FB5011000 | |
| Inoculation loop | Fisher | 22-363-605 | |
| LB Agar | Fisher | BP1425500 | |
| Liquid waste collection bottle | Thomas Scientific | 1230G50 | |
| Magnesium Sulfate (MgSO4) | Sigma | M7506 | |
| Paraformaldehyde (32%) | Electron Microscopy Sciences | 15714-S | Paraformaldehyde – methanol free solution |
| Pipettor | Eppendorf | Eppendorf Easypet 3 | |
| Plastic dishes (100 mm) | Fisher | FB0875712 | |
| Potassium Phosphate Monobasic (KH2PO4) | Fisher | P2853 | |
| Seahorse XF Calibrant | Agilent | 100840-000 | |
| Seahorse XFe96 Extracellular Flux Assay Kit and Cell Culture Microplate | Agilent | 101085-004 | |
| Serological pipettes (50 mL) | Genesee Scientific | 12-107 | |
| Shaker incubator | Thermo | 11 676 083 | |
| Sodium Chloride (NaCl) | Fisher | S640-3 | |
| Sodium Hydroxide (NaOH) | Fisher | S318500 | |
| Sodium Phosphate Dibasic Anhydrous (Na2HPO4) | Sigma | S374-500 | |
| Solid waste collection bucket | M&M Industries | 5.0 Gallon M1 Traditional Pail | |
| Tryptone | Genesee Scientific | 20-251 | |
| Vortex | Thermo | 11676331 | |
| Weighing balance | C Goldenwall | HZ10K6B | |
| Yeast Extract | Genesee Scientific | 20-255 |
Referenzen
- Riddle, D. L., Blumenthal, T., Meyer, B. J., Priess, J. R. C. . Elegans II. 33, (1997).
- Corsi, A. K., Wightman, B., Chalfie, M. A transparent window into biology: A primer on caenorhabditis elegans. WormBook. , 1-31 (2015).
- Kaletta, T., Hengartner, M. O. Finding function in novel targets: C. elegans as a model organism. Nature reviews. Drug discovery. 5 (5), 387-398 (2006).
- Meneely, P. M., Dahlberg, C. L., Rose, J. K. Working with worms: Caenorhabditis elegans as a model organism. Current Protocols Essential Laboratory Techniques. 19 (1), (2019).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Frontiers in Endocrinology. 11, 554994 (2020).
- Feng, M., Gao, B., Garcia, L. R., Sun, Q. Microbiota-derived metabolites in regulating the development and physiology of Caenorhabditis elegans. Frontiers in Microbiology. 14, 1035582 (2023).
- McIntyre, G., Wright, J., Wong, H. T., Lamendella, R., Chan, J. Effects of FUdR on gene expression in the C. elegans bacterial diet OP50. BMC Research Notes. 14 (1), 207 (2021).
- Cabreiro, F., et al. Metformin retards aging in c. Elegans by altering microbial folate and methionine metabolism. Cell. 153 (1), 228-239 (2013).
- Worthy, S. E., et al. Identification of attractive odorants released by preferred bacterial food found in the natural habitats of c. Elegans. PLoS One. 13 (7), e0201158 (2018).
- Chen, Y. C., Seyedsayamdost, M. R., Ringstad, N. A microbial metabolite synergizes with endogenous serotonin to trigger C. elegans reproductive behavior. Proceedings of the National Academy of Sciences of the United States of America. 117 (48), 30589-30598 (2020).
- Kim, D. H., Flavell, S. W. Host-microbe interactions and the behavior of Caenorhabditis elegans. Journal of Neurogenetics. 34 (3-4), 500-509 (2020).
- Gems, D., Riddle, D. L. Genetic, behavioral, and environmental determinants of male longevity in Caenorhabditis elegans. Genetics. 154 (4), 1597-1610 (2000).
- Qi, B., Kniazeva, M., Han, M. A vitamin-b2-sensing mechanism that regulates gut protease activity to impact animal's food behavior and growth. eLife. 6, e26243 (2017).
- Garigan, D., et al. Genetic analysis of tissue aging in caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Virk, B., et al. Folate acts in E. coli to accelerate C. elegans aging independently of bacterial biosynthesis. Cell Reports. 14 (7), 1611-1620 (2016).
- Beydoun, S., et al. An alternative food source for metabolism and longevity studies in Caenorhabditis elegans. Communications Biology. 4 (1), 258 (2021).
- Thavarajah, R., Mudimbaimannar, V. K., Elizabeth, J., Rao, U. K., Ranganathan, K. Chemical and physical basics of routine formaldehyde fixation. Journal of Oral and Maxillofacial Pathology. 16 (3), 400-405 (2012).
- Felix, H. Permeabilized and immobilized cells. Methods in Enzymology. 137, 637-641 (1988).
- Lobritz, M. A., et al. Antibiotic efficacy is linked to bacterial cellular respiration. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8173-8180 (2015).
- Nadanaciva, S., et al. Assessment of drug-induced mitochondrial dysfunction via altered cellular respiration and acidification measured in a 96-well platform. Journal of Bioenergetics and Biomembranes. 44 (4), 421-437 (2012).
- Shtonda, B. B., Avery, L. Dietary choice behavior in Caenorhabditis elegans. The Journal of Experimental biology. 209 (Pt 1), 89-102 (2006).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. Diet-induced developmental acceleration independent of tor and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- Kumar, S., et al. Lifespan extension in C. elegans caused by bacterial colonization of the intestine and subsequent activation of an innate immune response. Developmental Cell. 49 (1), 100-117 (2019).
- Nakagawa, H., et al. Effects and mechanisms of prolongevity induced by Lactobacillus gasseri sbt2055 in Caenorhabditis elegans. Aging Cell. 15 (2), 227-236 (2016).
- Kaeberlein, T. L., et al. Lifespan extension in Caenorhabditis elegans by complete removal of food. Aging Cell. 5 (6), 487-494 (2006).
- Beaudoin-Chabot, C., et al. The unfolded protein response reverses the effects of glucose on lifespan in chemically-sterilized C. elegans. Nature Communication. 13 (1), 5889 (2022).
- Komura, T., Takemoto, A., Kosaka, H., Suzuki, T., Nishikawa, Y. Prolonged lifespan, improved perception, and enhanced host defense of Caenorhabditis elegans by Lactococcus cremoris subsp. cremoris.Microbiology Spectrum. 10 (3), e0045421 (2022).
- Ye, X., Linton, J. M., Schork, N. J., Buck, L. B., Petrascheck, M. A pharmacological network for lifespan extension in Caenorhabditis elegans. Aging Cell. 13 (2), 206-215 (2014).
- Hastings, J., et al. Wormjam: A consensus C. elegans metabolic reconstruction and metabolomics community and workshop series. Worm. 6 (2), e1373939 (2017).
- O'Donnell, M. P., Fox, B. W., Chao, P. H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Stuhr, N. L., Curran, S. P. Bacterial diets differentially alter lifespan and healthspan trajectories in C. elegans. Communications Biology. 3 (1), 653 (2020).
- Dirksen, P., et al. Cembio - the Caenorhabditis elegans microbiome resource. G3 (Bethesda). 10 (9), 3025-3039 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten