Method Article
Analyse der Aktivität humaner T-Zellen in einer allogenen Co-Kultur von vorbehandelten Tumorzellen
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt einen experimentellen Arbeitsablauf, der die ex vivo Analyse der Stimulation humaner T-Zellen in einem allogenen Co-Kultursystem mit vorbehandelten Tumorzellen ermöglicht.
Zusammenfassung
Zytotoxische T-Zellen spielen eine Schlüsselrolle bei der Eliminierung von Tumorzellen und werden daher in der Krebsimmunologie intensiv untersucht. Die Häufigkeit und Aktivität zytotoxischer T-Zellen in Tumoren und deren Tumormikroumgebung (TME) sind mittlerweile etablierte prognostische und prädiktive Biomarker für zahlreiche Tumorarten. Es ist jedoch bekannt, dass verschiedene Tumorbehandlungsmodalitäten, einschließlich Strahlentherapie, Chemotherapie, Immuntherapie und zielgerichtete Therapie, nicht nur die Immunogenität des Tumors, sondern auch das Immunsystem selbst modulieren. Folglich erfordert die Interaktion zwischen Tumorzellen und T-Zellen eine intensivere Untersuchung in verschiedenen therapeutischen Kontexten, um die komplexe Rolle von T-Zellen während der Tumortherapie vollständig zu verstehen. Um diesem Bedarf gerecht zu werden, wurde ein Protokoll entwickelt, um die Aktivität und proliferative Kapazität von humanen zytotoxischen (CD8+) T-Zellen in Co-Kultur mit vorbehandelten Tumorzellen zu analysieren. Konkret werden CD8+ T-Zellen von gesunden Spendern mit dem nicht-toxischen Proliferationsmarker Carboxyfluoresceindiacetat-Succinimidylester (CFSE) gefärbt und mit CD3/CD28-beschichteten Platten stimuliert. Anschließend werden T-Zellen mit vorbehandelten Tumorzellen co-kultiviert. Als Auslese wird die T-Zellproliferation quantifiziert, indem die CFSE-Signalverteilung gemessen und die Expression von Oberflächenaktivierungsmarkern mittels Durchflusszytometrie bewertet wird. Dies kann durch die Quantifizierung der Zytokinfreisetzung mittels Enzyme-linked Immunosorbent Assay (ELISA) weiter ergänzt werden. Diese Methode ermöglicht die Bewertung von behandlungsinduzierten Veränderungen in der Interaktion zwischen Tumorzellen und T-Zellen und bietet eine Grundlage für detailliertere Analysen der Tumorbehandlungsmodalitäten und ihrer Immunogenität in einem humanen ex vivo-Setting . Darüber hinaus trägt es zur Reduzierung präklinischer in vivo-Analysen bei.
Einleitung
Heutzutage wird immer deutlicher, dass das Entstehen und Fortschreiten von Tumoren stark von der effektiven Manipulation und Unterdrückung des Immunsystems des Wirts abhängt. Transformierte Zellen entstehen jeden Tag, auch in einem gesunden Organismus. Die Bildung von makroskopischen Tumoren ist jedoch ein eher seltenes Ereignis, da entstehende transformierte Zellen mit hoher Effizienz aus dem Organismus entfernt werden. Für die Entfernung bösartiger Zellen kommen verschiedene Immunzelltypen wie zytotoxische T-Zellen, natürliche Killerzellen (NK)T-Zellen, NK-Zellen oder Makrophagen zum Einsatz 1,2,3. Nichtsdestotrotz können manchmal transformierte Zellklone entstehen, die in einem Gleichgewichtszustand mit dem Immunsystem des Wirts überleben, was durch unterschiedliche immunsuppressive Strategien des Tumorzellklonsgekennzeichnet ist 4. Schließlich erwerben einige transformierte Zellen weitere Funktionen, die es den Tumorzellen ermöglichen, die Immunantwort aktiv zu unterdrücken, was in der Folge zum Auswachsen des Tumors führt. Diese Immunsuppression wird durch zahlreiche Mechanismen vermittelt, darunter die Expression immunsuppressiver Liganden auf den Tumorzellen oder die aktive Rekrutierung oder das Priming von immunregulatorischen oder immunsuppressiven Immunzellpopulationen. Dieses sogenannte Immun-Editing-Konzept zeigt die Schlüsselrolle der Immunmodulation während der Tumorbildung und -ausbreitung5.
So ist es nicht verwunderlich, dass das Immunsystem heutzutage nicht nur in der Krebstherapie, sondern auch als prädiktiver und prognostischer Faktor in vielen Tumorentitäten und Therapiesettings im Fokus steht. In den letzten Jahren haben sich Immun-Checkpoint-Inhibitoren (ICIs) als vielversprechende Therapieoptionen bei verschiedenen soliden Krebsarten, wie z.B. dem Plattenepithelkarzinom (HNSCC) oder dem nicht-kleinzelligen Lungenkrebs (NSCLC) erwiesen, mit dem Ziel, die Tumormikroumgebung (TME) hin zu einer effizienteren Anti-Tumor-Immunantwort und einer verminderten Immunsuppression der Tumorzellen zu modulieren6. Die Immun-Checkpoint-Inhibitoren zielen darauf ab, die T-Zell-vermittelte Abtötung von Tumorzellen zu verstärken, indem sie auf Immun-Checkpoint-Moleküle wie die des programmierten Todesproteins 1 (PD-1) und seiner Liganden-1-Achse (PD1/PD-L1) abzielen. Diese Tatsache unterstreicht die Schlüsselrolle von T-Zellen bei der Immunität gegen Tumore. Bei HNSCC wurden beispielsweise ICIs erfolgreich als Erstlinientherapie bei rezidivierendem und metastasiertem HNSCCzugelassen 7. Dementsprechend kann das Vorhandensein von zytotoxischen T-Zellen in der TME sowie die Expression von PD1 und PD-L1 auf den Tumorzellen und entsprechenden T-Zellen als prädiktive Biomarker in HNSCC dienen 8,9,10.
Auch wenn T-Zellen eine entscheidende Rolle in der Tumorimmunologie und Tumortherapie spielen, müssen noch viele offene Fragen zu ihren Wechselwirkungen mit dem Tumor geklärt werden. Heutzutage ist allgemein bekannt, dass die Immunantwort von Tumoren ein dynamischer Prozess ist und dass sich die Immunogenität von Tumoren im Verlauf der Erkrankung und der Behandlungen verändern kann. Verschiedene Behandlungsmodalitäten wie Chemotherapie (CT), Strahlentherapie (RT) oder zielgerichtete Therapie (TT) sind besonders bekannt, um den immunologischen Phänotyp von Tumorzellen zu modulieren. RT kann die Hochregulierung von Immun-Checkpoint-Molekülen im Tumorgewebe vorantreiben und die Häufigkeit tumorinfiltrierender Zellen verändern11,12. Andererseits kann TT auch günstige Veränderungen innerhalb des Tumors und der TME durch direkte Modulation der adaptiven Immunantwort befürworten 13,14,15,16. Diese Modulationen sind jedoch bei Patienten nur schwer zu untersuchen, da dies eine wiederholte Untersuchung des Tumorgewebes im Verlauf der Behandlung erfordern würde. Daher werden schlüssige experimentelle Modellsysteme benötigt, um den dynamischen immunologischen Phänotyp von Tumorzellen und T-Zellen und vor allem ihre Wechselwirkungen zu untersuchen.
Daher wird für die Analyse der T-Zellaktivität und der Wechselwirkungen von Tumorzellen und T-Zellen ein umfassender ex vivo Co-Kultur-Assay benötigt, der in jedem Labor auf der Grundlage einfacher Zellkulturarbeit und häufig verwendeter durchflusszytometrischer Analysen einfach zu implementieren ist. Basierend auf der vorhandenen und verfügbaren Literatur wurde bisher kein einfach zu handhabendes und häufig verwendetes Protokoll zur Co-Kultur von T-Zellen und Tumorzellen veröffentlicht. Auch wenn in letzter Zeit mehrere Co-Kultur-Assays für T-Zellen und Tumor-Organoide veröffentlicht wurden, ist die 3D-Zellkulturtechnik heute noch nicht als Standardtechnik in jedem Labor implementiert. Damit stellen wir ein Protokoll für den Einsatz in der 2D-Zellkultur zur Verfügung, das sich in Zukunft auch für die 3D-Zellkultur etablieren könnte. Andere 2D-Co-Kulturprotokolle sind oft komplexer, da sie z.B. die Transduktion von Tumorzellen mit Luciferaseerfordern 17 oder nur für hämatologische Malignome geeignet sind (Co-Kultur von nicht passenden T-Zellen mit Leukämiezellen)18. In dem hier beschriebenen Assay werden T-Zellen von normal gesunden Spendern aus peripherem Blut isoliert und mit Anti-CD3- und Anti-CD28-Antikörpern stimuliert. Anschließend werden die T-Zellen mit (vor-)behandelten Tumorzellen co-kultiviert, um die proliferative Kapazität der T-Zellen sowie deren Aktivität mittels Durchflusszytometrie zu analysieren. Dabei können die Auswirkungen verschiedener Behandlungsmodalitäten auf den Immunphänotyp der Tumorzelle, wie RT, CT oder TT, die wiederum die Aktivität und Proliferation der T-Zellen beeinflussen, leicht gescreent und als Grundlage für tiefergehende mechanistische Analysen und konsekutive ausgewählte präklinische in vivo-Analysen verwendet werden. Der hier beschriebene Assay bietet einen einfach zu bedienenden Aufbau, da keine unkonventionellen Geräte, Techniken oder Materialien erforderlich sind. Darüber hinaus kann der Assay leicht an verschiedene Tumorzelllinien oder spezifische T-Zell-Untergruppen (z.B. CD4+ T-Zellen) angepasst werden. Mit dieser Technik wird eine hohe Standardisierung und Reproduzierbarkeit erreicht.
Protokoll
Dieser Assay umfasst die Entnahme von Blut und die Kultivierung menschlicher Primärzellen. Daher ist für diese Analysen ein ethisches Votum zwingend erforderlich. Alle in diesem Manuskript vorgestellten Ergebnisse sind durch die ethische Genehmigung der IMMO-NHD-Studie abgedeckt, und es wurde eine schriftliche Einverständniserklärung aller Spender eingeholt. Die Bewilligung erfolgte am 9. November 2022 durch das Institutionelle Fachkollegium der Friedrich-Alexander-Universität Erlangen-Nürnberg (Antragsnummer 21-415-B). Die in dieser Studie verwendeten HSC4-Tumorzellen stammen aus einer kommerziell erhältlichen Zelllinie.
Dieser Assay veranschaulicht beispielhaft alle Schritte des T-Zell- und Tumorzell-Co-Kultur-Assays unter Verwendung der humanen Plattenepithelkarzinom-Zelllinie HSC4 (HNSCC) im Kopf- und Halsbereich in einer Behandlungsumgebung, die eine Strahlentherapie (RT) und zwei spezifische Kinase-Inhibitoren umfasst. Folglich sind Parameter wie Zellzahlen, Trypsinisierungszeiten und Behandlungsschemata spezifisch für diese Co-Kultur-Umgebung und sollten für andere Tumorzelllinien angepasst werden (siehe auch Abschnitt Diskussion). Alle Zentrifugationsschritte wurden bei Raumtemperatur durchgeführt. In dieser Studie wurden die Kinase-Inhibitoren AZD0156 und VE-822 verwendet, um das DNA-Schadensreparatursystem (DDR) von HSC4-Zellen zu bekämpfen. AZD0156 (Selleckchem) hemmt das Ataxia teleangiectasia mutated (ATM) Protein, während VE-822 (Selleckchem) auf die Ataxia teleangiectasia und das Rad3-related (ATR) Protein abzielt. Beide Inhibitoren wurden als potenzielle Wirkstoffe zur Erhöhung der Strahlenempfindlichkeit in Tumorzellen diskutiert. Sie werden in Dimethylsulfoxid (DMSO) gelöst und bei -20 °C gelagert. Abbildung 1 zeigt ein Flussdiagramm des ex vivo Assays, das die Co-Kultur von vorbehandelten Tumorzellen mit CD3/CD28-stimulierten humanen CD8+ T-Zellen zeigt. Die verwendeten Reagenzien und Geräte sind in der Materialtabelle aufgeführt.
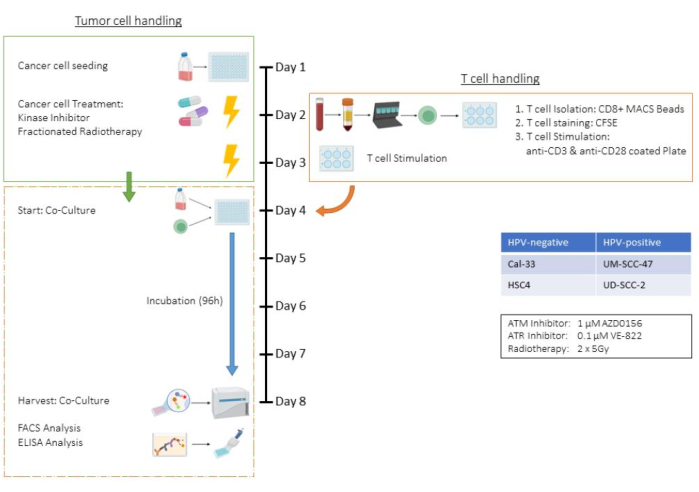
Abbildung 1: Flussdiagramm des ex vivo Assays von vorbehandelten Tumorzellen in Co-Kultur mit (CD3/CD28) stimulierten humanen CD8+ T-Zellen. Tag 1: Aussaat von HSC4-Tumorzellen. Tag 2: Behandlung von Tumorzellen und Isolierung, CFSE-Färbung und Aussaat in CD3/CD28-beschichtete Platten von humanen CD8+ T-Zellen. Tag 3: Behandlung der Tumorzellen. Tag 4: Zählung repräsentativer Vertiefungen von Tumorzellen. Ernte und Zählung aller T-Zellen. Co-Kultivierung von T-Zellen und HSC4-Tumorzellen im Verhältnis 1:1. Tag 5-Tag 8: Inkubation in Co-Kulturen. Tag 8: Ernte der Co-Kultur, Einfrieren der Überstände, Antikörper-basierte Färbung und Analyse der Zellen mittels Durchflusszytometrie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Aussaat von Tumorzellen (Tag 1)
HINWEIS: Zeit: 1 Std. Humane HSC4-Tumorzellen werden aus T75-Zellkulturflaschen in 96-Well-Platten ausgesät.
- Entsorgen Sie den Überstand (D10 Medium) der Zellkulturflasche.
- Waschen Sie die Tumorzellen mit 5 mL 37 °C warmem PBS. PBS verwerfen.
- Da HSC4-Tumorzellen dazu neigen, fest an Zellkulturflaschen zu haften, wird eine zweistufige Trypsinisierung empfohlen, um sicherzustellen, dass alle Zellen abgelöst werden. Fügen Sie dazu 3 mL Trypsin hinzu und stellen Sie die Flasche für 3 min auf eine Heizplatte bei 37 °C, dann entsorgen Sie das Trypsin mit einer Pipette.
- Fügen Sie weitere 3 ml Trypsin hinzu, stellen Sie die Flasche auf die Heizplatte und warten Sie, bis die einzellige Suspension erreicht ist. Mikroskopisch auf einzellige Suspension prüfen.
- Stoppen Sie das Trypsin, indem Sie das doppelte Volumen von D10 Medium (DMEM ergänzt mit 10 % fötalem Rinderserum und 1 % Penicillin-Streptomycin) hinzufügen, resuspendieren Sie es gründlich und übertragen Sie die Zellen in ein 50-ml-Zentrifugationsröhrchen.
- Bestimmen Sie die Zellzahl der Einzelzellsuspension und berechnen Sie dann die Gesamtmenge der Tumorzellen entsprechend dem Gesamtvolumen. Für die Bestimmung der Zellzahl empfiehlt sich eine Neubauer-Zellkammer.
- Zentrifugieren Sie das 50 mL Zentrifugationsröhrchen mit den Tumorzellen bei 300 x g für 5 min bei Raumtemperatur. Entsorgen Sie den Überstand.
- Resuspendieren Sie HSC4-Tumorzellen in einem geeigneten Volumen von D10-Medium, um eine Konzentration von 15.000 Zellen in einem 200-μl-Medium zu erreichen.
- Entsprechend den geplanten Behandlungen von Tumorzellen wird für jede Erkrankung mindestens 3 Vertiefungen mit 15.000 HSC4-Tumorzellen in 200 μl Medium in zwei 96-Well-Platten gesät (1x Probe, 1x Tumorzellkontrolle nur Kontrolle, 1x Zellzählvertiefung für den Co-Kulturtag).
HINWEIS: Die Zellzahl hängt von der Zelllinie und der Verdopplungszeit ab. Die Anzahl der Zellen für verschiedene Behandlungen muss in Vorexperimenten untersucht werden, um eine 100%ige Konfluenz während der Inkubation zu vermeiden. - Inkubieren Sie die Zellen 24 Stunden lang bei 37 °C und 5 % CO2 und gesättigter Luftfeuchtigkeit.
2. Behandlung von Tumorzellen (Tag 2)
HINWEIS: Zeit: 3-5 Stunden. Nach einer Inkubationszeit von 24 h können die zuvor ausgesäten HSC4-Zellen nach dem gewünschten Behandlungsschema behandelt werden. In diesem beispielhaften Fall werden die Tumorzellen mit einem ATM- oder ATR-Inhibitor behandelt. Hierbei wird zusätzlich eine von zwei 96-Well-Platten anschließend mit 2x 5 Gy bestrahlt.
- Bereiten Sie Kinase-Inhibitoren vor, um eine Konzentration von 1 μM AZD0156 (ATM-Inhibitor) und 0,1 μM VE-822 (ATR-Inhibitor) zur Behandlung von Tumorzellen zu erhalten.
- Behandeln Sie die Tumorzellen entsprechend, indem Sie beispielsweise eine Probenreihe mit 3,1 μl des ATM-Inhibitors und eine zweite mit 3,1 μl des ATR-Inhibitors in jeder Platte behandeln.
- Nach 3-5 h Inkubation bei 37 °C wird eine Platte mit 5 Gy bestrahlt.
- Nach einer weiteren Inkubation von 24 h bestrahlen Sie die gleiche Platte erneut mit 5 Gy.
3. T-Zell-Isolierung (Tag 2)
HINWEIS: Zeitdauer: 4 h. CD8+ T-Zellen werden magnetisch mit Anti-CD8-Mikrokügelchen aus PBMCs isoliert, nachdem peripheres Blut (PB) von gesunden erwachsenen Spendern mit Dichtegradienten zentrifugiert wurde. Die isolierten T-Zellen werden dann mit CFSE gefärbt und zur Stimulation in einer CD3/CD28-beschichteten Well-Platte inkubiert. Um in späteren Schritten ressourcen- und materialschonend arbeiten zu können, ist es wichtig, die Menge an T-Zellen abzuschätzen, die für die Co-Kultur-Experimente benötigt wird. Je nach Spender können zwischen 50.000.000 und 100.000.000 PBMCs aus ca. 45 ml EDTA-Blut isoliert werden. Etwa 10 % der gesamten PBMCs sind CD8+ T-Zellen.
- Dichtegradienten-Zentrifugation
- Übertragen Sie 3-5 EDTA-Blutröhrchen à 9 ml, die von einem gesunden Spender stammen, in zwei 50-ml-Zentrifugationsröhrchen.
- Füllen Sie beide Zentrifugationsröhrchen auf 50 mL mit PBS + 2 % FBS.
- Bereiten Sie sechs Zentrifugationsröhrchen (mit Kunststoffeinlage für die Trennung von PBMCs) vor und füllen Sie jedes mit 15 mL + 4 °C Kältegradientenmedium.
- Das Dichtegradientenmedium vorsichtig mit 12-15 ml des verdünnten Blutes aus Schritt 3.1.2 überlagern.
- Zentrifugieren Sie 10 Minuten lang bei 1200 x g (keine Verzögerung erforderlich).
- Vorbereitung einer 6-Well-Platte für die T-Zell-Stimulation
- Antikörper vorbereiten: Bereiten Sie eine Anti-CD3-Lösung (Klon OKT3) von 1 mg/ml mit PBS vor. Bereiten Sie eine Anti-CD28-Lösung (Klon 28.2) von 0,1 mg/ml mit PBS vor.
- Empfehlung: Die Zeit während der Zentrifugation (Schritt 3.1.5) für die Herstellung von Beschichtungslösungen und die Beschichtung der Vertiefungen selbst für die Stimulation der T-Zellen nutzen.
- Mischen Sie 5 μl CD3-Antikörperlösung mit 4,995 μl PBS und 50 μl CD28-Antikörperlösung mit 4,950 μl PBS (Endkonzentration: 1 μg/μl).
- Geben Sie 1.000 μl beider Antikörperlösungen in jede Vertiefung der 6-Well-Platte.
- Die 6-Well-Platte wird mit 2 oder 3 Vertiefungen, je nach erwarteter Menge an T-Zellen, beschichtet und mindestens 2 Stunden bei 37 °C inkubiert.
- Vorbereitung des T-Zell-Mediums
HINWEIS: Herstellung von 100x L-Lysin: 200 mg L-Lysinhydrochlorid in 50 mL destilliertem Wasser in einem 50 mL Zentrifugenröhrchen auflösen. Sterilisieren Sie die Lösung mit einem 0,2-μm-Spritzenvorsatzfilter und überführen Sie sie mit einer 50-ml-Perfusionsspritze in ein neues 50-ml-Röhrchen. Lagern Sie die Lösung bei 4-8 °C und verwenden Sie sie innerhalb von 3 Monaten. Zubereitung von 15 mM L-Arginin: Lösen Sie 26 mg L-Arginin in 10 mL DPBS. Sterilisieren Sie die Lösung mit einem 0,2-μm-Spritzenvorsatzfilter und überführen Sie sie mit einer 10-ml-Spritze in ein neues Röhrchen. Lagern Sie die Lösung bei 4-8 °C und verwenden Sie sie innerhalb von 3 Monaten.- Verwenden Sie die in Schritt 1.5 erwähnte Zentrifugationszeit, um das T-Zell-Medium vorzubereiten.
- Bereiten Sie etwa 10-30 ml T-Zellmedium vor, abhängig von der erwarteten Menge an T-Zellen.
- Mischen Sie das L-Arginin und L-Lysin-freie RPMI-Medium mit 10 % hitzeinaktiviertem FBS, 1 % Penicillin-Streptomycin, 1 % L-Arginin, 1 % L-Lysin und 1 % L-Glutamin. Beispiel: 25,8 ml RPMI-Medium + 3 ml FBS + 0,3 ml Pen/Strep + 0,3 ml L-Arginin + 0,3 ml L-Lysin + 0,3 ml L-Glutamin.
- Das restliche Medium wird in den nächsten zwei Tagen bei 4 °C gelagert, um es am Tag 4 - Beginn der Co-Kultur - zu verwenden. Für jeden neuen Durchlauf des gesamten Versuchs wird empfohlen, ein frisches Kulturmedium vorzubereiten.
- PBMC-Isolierung
- Nach der Zentrifugation (Schritt 3.1.5) wird der Überstand in vier neue 50-mL-Zentrifugationsröhrchen überführt und die gebrauchten Röhrchen entsorgt.
- Füllen Sie die Zentrifugationsröhrchen bis zu 50 mL mit PBS + 2% FBS.
- Zentrifugieren Sie bei 300 x g für 8 min bei Raumtemperatur.
- Der Überstand wird verworfen, das Zellpellet in je 1 ml PBS + 2 % FBS resuspendiert und in zwei Zentrifugationsröhrchen kombiniert.
- Füllen Sie die Röhrchen auf 50 mL mit PBS + 2% FCS auf.
- Bei 120 x g für 10 min bei Raumtemperatur zentrifugieren.
- Den Überstand verwerfen, das Zellpellet vorsichtig in 1 ml PBS +2 % FBS resuspendieren und die beiden Pellets in einem Falken kombinieren.
- Füllen Sie das Zentrifugationsröhrchen auf 50 mL mit PBS + 2% FBS.
- Zählen Sie die Gesamtzahl der PBMCs mit einer Neubauer-Zählkammer. Empfehlung: Die Zellsuspension für die Zählung 1:10 mit Trypanblau verdünnen.
- Trennung von CD8+ T-Zellen
HINWEIS: Verwenden Sie CD8 MicroBeads human zusammen mit magnetischen Trennsäulen (MS) und magnetisch aktiviertem Zellsortierpuffer (MACS+), um CD8+ T-Zellen gemäß dem Protokoll des Herstellers magnetisch zu trennen (siehe Materialtabelle).- Zentrifugieren Sie die PBMCs (nach Zellzählung) bei 300 x g für 10 min.
- Verwerfen Sie den Überstand und resuspendieren Sie das Zellpellet in 80 μl MACS+-Puffer (500 mL PBS, ergänzt mit 10 mM EDTA und 0,5 % BSA) pro 107 Zellen (z. B. werden 60.000.000 PBMCs in 6 x 80 μL = 480 μL Puffer resuspendiert).
HINWEIS: Beispiel: 465 mL PBS + 10 mL (0,5 M) EDTA und 25 mL BSA-Stammlösung. - Fügen Sie 20 μl CD8-Mikrokügelchen pro 107 Zellen hinzu und mischen Sie vorsichtig, indem Sie auf und ab pipettieren. 15 min bei 4 °C inkubieren.
- Waschen Sie die Zellen nach der Inkubation, indem Sie 2 ml MACS+-Puffer pro 107 Zellen hinzufügen.
- Bei 300 x g für 10 min bei Raumtemperatur zentrifugieren. Entsorgen Sie den Überstand und resuspendieren Sie die Zellen in 1.000 μl MACS+-Puffer.
- Setzen Sie zwei MS-Säulen in den Magneten ein und platzieren Sie zwei 15 mL Zentrifugationsröhrchen darunter.
- Bereiten Sie die Säulen mit 500 μL MACS+ Puffer vor. Der Puffer läuft durch die Säulen und kann in den darunter liegenden Zentrifugationsröhrchen aufgefangen werden. Dieser Schritt kann auch parallel zu Schritt 3.5.6 durchgeführt werden.
- Pipettieren Sie dann die Zellsuspension gleichmäßig in die vorbereiteten MS-Säulen. Der Durchfluss kann in den gleichen Zentrifugationsröhrchen aufgefangen werden. Es enthält nun alle unmarkierten, CD8-negativen Zellen.
- Spülen Sie die Säulen durch Zugabe von 3x 500 μl MACS+ Puffer. Fügen Sie erst dann einen neuen Puffer auf die Säulen hinzu, wenn diese trocken sind oder nicht mehr tropfen.
- Markieren Sie die fertig entnommenen Zellen als "Durchfluss" oder "CD8-negativ" und kombinieren Sie sie in einem einzigen Zentrifugationsröhrchen. Damit kann später die Reinheit der Isolierung mittels Durchflusszytometrie gemessen werden.
- Nehmen Sie ein neues 15-ml-Zentrifugationsröhrchen mit der Bezeichnung "CD8+ T-Zellen". Die Säulen werden vom Magneten entfernt und auf das Zentrifugationsröhrchen gelegt.
- Spülen Sie beide Säulen, die die magnetisch markierten CD8-positiven T-Zellen enthalten, mit jeweils 1.000 μl MACS+-Puffer. Pipettieren Sie daher den Puffer in die Säulen und beginnen Sie sofort, die Lösung vorsichtig mit dem vom Hersteller bereitgestellten Kolben aus den Säulen zu drücken.
HINWEIS: Führen Sie dies für beide Säulen durch und sammeln Sie 2 ml T-Zell-Gesamtsuspension in der Zentrifugation. - Zählen Sie die Gesamtzahl der T-Zellen mit einer Neubauer-Zählkammer. Empfehlung: Verwenden Sie eine 1:4-Verdünnung mit Trypanblau.
- Färbung von T-Zellen mit CFSE
- Zentrifugieren Sie die isolierten T-Zellen bei 300 x g für 5 min.
- Bereiten Sie während der Zentrifugation die CFSE-Färbelösung vor, indem Sie 1,1 μl CFSE-Lösung mit 10 ml PBS mischen. Die Endkonzentration muss 1 μM betragen.
- Entsorgen Sie den Überstand und resuspendieren Sie das Zellpellet in 1.000 μl PBS. Bei 300 x g 5 min zentrifugieren.
- Den Überstand verwerfen und das Zellpellet in 2000 μl CFSE-Färbelösung (1 μM) resuspendieren. 20 min bei 37 °C inkubieren.
- Zentrifugieren Sie die gefärbten Zellen bei 300 x g für 5 min. Entsorgen Sie den Überstand und resuspendieren Sie das Zellpellet in 1.000 μl PBS.
- Bei 300 x g 5 min zentrifugieren. Der Überstand wird verworfen und das Zellpellet in der entsprechenden Menge T-Zellmedium resuspendiert, um eine Konzentration von 1,5 bis 2 x 106 T-Zellen in etwa 3 bis 4 ml T-Zellmedium zu erreichen.
- Aussaat von T-Zellen in eine 6-Well-Platte
- Entsorgen Sie die Beschichtungslösung von der 6-Well-Platte.
- Keimen Sie 1,5-2 Millionen isolierte und gefärbte T-Zellen in 3-4 mL T-Zellmedium pro Well.
- Inkubieren Sie die T-Zellen für die nächsten 48 Stunden bei 37 °C und 5 % CO2
HINWEIS: Stellen Sie die Aussaatdichte immer auf 1,5 Mio. Zellen / 3 mL ein. Vermeiden Sie es, T-Zellen in einer geringeren Dichte zu säen. Eine höhere Dichte ist möglich, muss aber getestet werden.
4. Beginn der Co-Kultur (Tag 4)
HINWEIS: Zeit: 2 h. Nach der Bestimmung der Zellzahl der HSC4-Tumorzellen werden die T-Zellen im Verhältnis 1:1 auf die 96-Well-Platten gegeben.
- Entnahme einer Vertiefung pro Bedingung zur Bestimmung einer repräsentativen Zellzahl
- Entsorgen Sie den Überstand der Vertiefung, der exemplarisch für die Bestimmung der Zellzahl ausgesät wurde.
- Waschen Sie zuerst die Zellen mit 100 μL PBS, dann verwerfen Sie das PBS.
- 100 μl Trypsin zugeben und 5 min auf einer Heizplatte inkubieren. Dann resuspendieren und überprüfen Sie die Einzelzellsuspension unter dem Mikroskop; Wenn sich die Zellen nicht lösen, fügen Sie weitere 50 μl Trypsin hinzu.
- Sobald sich alle Zellen abgelöst haben, stoppen Sie die Trypsinreaktion, indem Sie 100 μl (oder 150 μl, wenn zuvor zusätzliche 50 μl Trypsin hinzugefügt wurden) des D10-Mediums hinzufügen und resuspendieren.
- Übertragen Sie das gesamte Volumen von 200 μl aus der Vertiefung in ein 1,5-ml-Probenröhrchen und verwenden Sie 100 μl zur Messung der Zellzahl. Denken Sie daran, die gemessene Zellzahl an das Probenvolumen anzupassen.
- Färbung von HSC4-Tumorzellen mit einem ungiftigen fluoreszierenden Zellverfolgungsfarbstoff
- Bereiten Sie den Zelltracker vor (siehe Materialtabelle), indem Sie 2 μL in 20 mL PBS auflösen (Endkonzentration 0,1 μM laut Hersteller).
- Pipettieren Sie das D10-Medium aus allen Vertiefungen, die Tumorzellen enthalten, und entsorgen Sie es.
- Geben Sie 200 μl der Cell-Tracker-Lösung in die Tumorzell-Wells. 20 min bei 37 °C inkubieren.
- Nach der Inkubation die Färbelösung verwerfen und mit 100 μl PBS waschen. Verwerfen Sie anschließend auch die PBS.
- Fügen Sie 200 μl frisches D10-Medium hinzu.
- Ernte von T-Zellen (6-Well-Platte)
- Resuspendieren Sie die T-Zellen vorsichtig in ihrem Medium. Die meisten T-Zellen befinden sich in Suspension und können geerntet werden, indem sie einfach aus ihrer Vertiefung pipettiert werden.
- Mikroskopisch prüfen, ob die Vertiefungen leer sind; Wenn nicht, ernten Sie mit 1.000 μl Trypsin, stellen Sie die Platte auf eine Heizplatte bei 37 °C, bis sich die T-Zellen abgelöst haben, und stoppen Sie dann die Reaktion, indem Sie 1.000 μl PBS- oder T-Zellmedium (optional) hinzufügen.
- Zählen Sie die T-Zellen mit Hilfe einer Neubauer-Zählkammer. Dann zentrifugieren Sie die T-Zellen bei 300 x g für 5 min.
- Resuspendieren Sie die T-Zellen in T-Zell-Medium (verwenden Sie das gleiche Medium, das an Tag 2 der T-Zell-Isolierung hergestellt wurde) auf eine Endkonzentration von 10.000 T-Zellen pro 20 μl.
- Hinzufügen von T-Zellen zu HSC4-Tumorzellen (96-Well-Platte)
- Geben Sie die gewünschte Menge an T-Zellen entsprechend der zuvor ermittelten Tumorzellzahl im Verhältnis 1:1 in die Tumorzellvertiefungen.
- Halten Sie eine Tumorzellvertiefung pro Zustand frei von T-Zellen als "nur Tumorzellen"-Kontrolle. Diese "nur Tumorzelle"-Steuerung ermöglicht das präzise Gating der Flussdaten.
- Geben Sie 200 μl der T-Zell-Suspension in eine leere Vertiefung als "nur T-Zelle"-Kontrolle (200.000 T-Zellen). Diese "only T-Cell"-Regelung ermöglicht das präzise Gating der Strömungsdaten.
- Inkubation der Co-Kultur für 96 h (37 °C, 5% CO2)
5. Quantifizierung der T-Zellproliferation durch Durchflusszytometrie (Tag 7)
HINWEIS: Zeit: 3 Stunden. Nach einer weiteren Inkubation der Co-Kultur von weiteren 96 Stunden werden die Vertiefungen geerntet und mit einem Antikörpermix gefärbt, der je nach Forschungshypothese verschiedene Antikörper enthält (z. B. Anti-CD3-, CD8-, HLA-DR- und CD25-Antikörper). Anschließend werden die Zellen mittels Mehrfarben-Durchflusszytometrie analysiert.
- Bereiten Sie eine Charge von FACS-Röhrchen und Mikrozentrifugenröhrchen für jede zu erntende Vertiefung vor und beschriften Sie sie.
- Ernte von Zellen
- Resuspendieren Sie die Zellen in ihrem Medium und überführen Sie sie in die FACS-Röhrchen.
- Waschen Sie die Zellen mit 100 μl PBS und überführen Sie diese dann in die FACS-Röhrchen.
- 100 μl Trypsin in die Vertiefungen geben und 5 min auf der Heizplatte (37 °C) inkubieren.
- Suspendieren Sie die Zellen wieder und kontrollieren Sie dann mikroskopisch, ob alle Zellen abgelöst sind. Wenn ja, überführen Sie die trypsinierte Zellsuspension in die FACS-Röhrchen.
- Mikroskopisch prüfen, ob alle Vertiefungen leer sind; Ist dies nicht der Fall, wiederholen Sie die Schritte 5.2.3-5.2.4.
- Sammeln des Überstands der Co-Kultur für weitere Experimente
- Zentrifugieren Sie die mit Zellen gefüllten Röhrchen bei 300 x g für 5 min.
- Etwa 300 μl des Überstands werden vorsichtig aus den Röhrchen in eine andere Charge von Mikrozentrifugenröhrchen pipetiert, die bei -20° C eingefroren werden sollen. Der Überstand kann später verwendet werden, um z.B. ELISA-Assays durchzuführen, um sezernierte Zytokine zu quantifizieren.
- Färbung der Zellen mit Antikörpern zur Durchführung der Durchflusszytometrie
- Antikörper-Mix vorbereiten: 5 μl anti-CD3-Krome Orange, 0,5 μl anti-CD8-PerCE-Cyp5.5, 1 μl anti-HLA-DR-APCVio770, 2,5 μl anti-CD25-PE-Dazzle zu 91 μl PBS/FACS-Puffer hinzufügen.
- Geben Sie 200 μl FACS-Puffer (PBS ergänzt mit 2 % FBS und 2 % EDTA) in jedes Röhrchen und resuspendieren Sie.
- Nochmals bei 300 x g für 5 min zentrifugieren. Der Überstand wird verworfen und die Zellen werden in 100 μl des zuvor hergestellten Antikörpermixes resuspendiert (Schritt 5.4.1). 30-45 min bei 4 °C im Kühlschrank lichtgeschützt inkubieren.
- Nach der Inkubation bei 300 x g für 5 min zentrifugieren. Den Überstand verwerfen und die Zellen in 100 μl FACS-Puffer resuspendieren.
- Führen Sie die Durchflusszytometrie an einem Zytometer durch, das in der Lage ist, alle oben genannten fluoreszierenden Antikörper und das CSFE-Signal zu unterscheiden.
6. Gating-Strategie und Datenanalyse
- Ausschluss von Dubletten und Identifizierung der korrekten T-Zellpopulation (Abbildung 2A).
- Schließen Sie die Dublettenpopulation basierend auf vorwärts vs. Seitenstreufläche (FSC-A vs. SSC-A) (Singuletts).
- Plot "Singuletts" zur Identifizierung von T-Zellen basierend auf Vorwärts- vs. Seitenstreufläche (FSC-A vs. SSC-A) (T-Zellen).
HINWEIS: Um den besten Größenausschluss zu erzielen, messen Sie die Proben "Nur T-Zellen" und "Nur Tumorzellen" parallel (Abbildung 2B). - Plotten Sie die CD3-Expression gegen die SSC-A, um die T-Zellen zu unterscheiden. Darüber hinaus können die CD8-positiven T-Zellen identifiziert werden, indem das CD8-Signal gegen das SSC-A aufgetragen wird. Alle T-Zellen sind CD3+/CD8+ (CD8+ T-Zellen) (Abbildung 2A).
- Plotten Sie das CFSE-Signal aller "CD8+ T-Zellen" als Histogramm.
HINWEIS: Gating der nicht-proliferativen Subpopulation (höchstes CFSE-Signal), der proliferativen Subpopulation (alle T-Zellen mit reduziertem CFSE-Signal) und der hochproliferativen Subpopulation (alle T-Zellen des 4. oder weniger intensiven CFSE-Peaks).
- Analyse von aktivitätsassoziierten Oberflächenmarkern auf der T-Zell-Population
- Befolgen Sie die Anschnittschritte (Schritt 6.1) (Abbildung 3A).
- Option A: Wählen Sie die "CD8+ T-Zellen" als Eingabe und analysieren Sie diese auf ihre CD25- oder HLA-DR-Expression, indem Sie die jeweilige Oberflächenmarker-Fluoreszenz gegen die SSC-A-Eigenschaften plotten.
HINWEIS: Wenn eine ausgeprägte CD25-hohe Subpopulation nachweisbar ist, wird ein Gating für die CD25-hohe Population empfohlen. Gleiches gilt für das Gating/die Analyse von HLA-DR an T-Zellen (Abbildung 3B). - Option B: Wählen Sie die "CD8+ T-Zellen" als Eingabe und analysieren Sie diese auf ihre CD25- oder HLA-DR-Expression, indem Sie die jeweilige Oberflächenmarkerfluoreszenz gegen die SSC-A-Eigenschaften plotten.
HINWEIS: Wenn keine eindeutige Subpopulation nachweisbar ist (abhängig vom analysierten Oberflächenmarker), wird die Analyse der Verschiebung der mittleren Fluoreszenzintensität (MFI) der gesamten T-Zellpopulation empfohlen (Abbildung 3C).
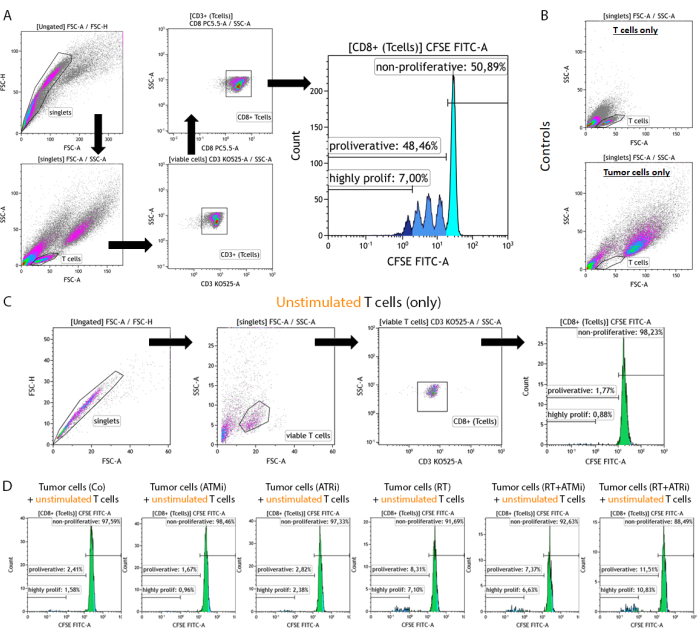
Abbildung 2: Gating-Strategie von CFSE-gefärbten, prästimulierten T-Zellen zur Analyse der Proliferation nach 96 h Co-Kultur mit vorbehandelten HNSCC-Tumorzellen. T-Zellen und Tumorzellen werden 96 Stunden nach der Co-Kultur von einer 96-Well-Platte geerntet, mit einem T-Zell-spezifischen Oberflächenmarker gefärbt und mit einem Durchflusszytometriegerät gemessen. (A) Dubletten wurden auf der Grundlage von FSC-A/FSC-H ausgeschlossen, und T-Zellen wurden zuerst nach Größe (FSC-A/SSC-A) begrenzt. T-Zellen wurden weiterhin auf ihre CD3- (anti-CD3 Krome Orange) und CD8 (anti-CD8 PerCP-Cy5.5) Expression analysiert. CD3+/CD8+ T-Zellen wurden in einem Histogramm dargestellt und die Intensität des CFSE-Signals analysiert. Das CFSE-Signal repräsentiert T-Zell-Untergruppen mit unterschiedlichem Proliferationsverhalten. T-Zellen mit dem höchsten CFSE-Signal wurden als "nicht-proliferativ" definiert. Alle Subpopulationen, die einen Verlust des CFSE-Signals zeigten, der durch Zellteilung verursacht wurde und zu einer Halbierung des Signals führte, wurden in deutlichen Peaks als "proliferativ" zusammengefasst. T-Zellen, die sich mehr als dreimal teilten (mehr als drei CSFE-Signalpeaks), wurden als "hochgradig proliferativ" definiert. (B) Als Kontrolle werden zusätzlich Proben gemessen, die nur aus vorstimulierten T-Zellen und nur aus vorbehandelten Tumorzellen bestehen. T-Zellen und HSC4-Tumorzellen können anhand von FSC-A vs. SCC-A-Signal. Des Weiteren wurden unstimulierte CD8+ T-Zellen analysiert, um die T-Zell-Aktivierung allein durch Co-Kultivierung mit allogenen Tumorzellen zu überprüfen. (C) Als Kontrolle wurde die Inkubation von unstimulierten CFSE-gefärbten T-Zellen für 96 h parallel zur Co-Kultur eingeschlossen. (D) Nicht stimulierte, CFSE-gefärbte T-Zellen wurden zusammen mit vorbehandelten Tumorzellen kultiviert. Nach 96 Stunden wurden die Zellen geerntet und das CFSE-Signal mit dem Standardverfahren gemessen. Unstimulierte T-Zellen zeigten weder allein noch nach Co-Kultivierung mit vorbehandelten Tumorzellen eine Proliferation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
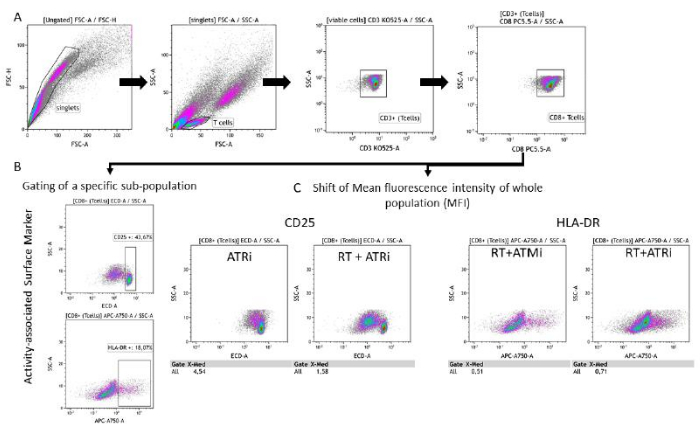
Abbildung 3: Gating-Strategie der Oberflächenmarkerexpression (CD25 und HLA-DR) im Zusammenhang mit der T-Zellaktivität nach 96 h Co-Kultivierung mit vorbehandelten HNSCC-Zellen. (A) Dubletten wurden aufgrund ihrer FSC-A/FSC-H-Merkmale ausgeschlossen, und T-Zellen wurden zuerst nach Größe (FSC-A/SSC-A) begrenzt. T-Zellen wurden weiterhin auf ihre CD3- (anti-CD3 Krome Orange) und CD8 (anti-CD8 PerCP-Cy5.5) Expression analysiert. Das Gating der Aktivitätsmarker CD25 (anti-CD25 PEDazzle594) und HLA-DR (anti-HLA-DR APC-Vio770) auf der T-Zelloberfläche kann in zwei verschiedenen Einstellungen durchgeführt werden. (B) Gating aller CD8+ T-Zellen für den Prozentsatz einerspezifischen CD25-High- oder HLA-DR-High-Subpopulation auf der Grundlage von Dichteplots. (C) Alternativ wird die Messung einer MFI-Verschiebung der gesamten CD8+ T-Zellpopulation für beide Aktivitätsmarker durchgeführt. Gezeigt werden repräsentative Bilder aus der Analyse der Expression von T-Zell-Oberflächenmarkern nach Co-Kultivierung mit HSC4-Tumorzellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
HSC4-Tumorzellen, die von einem HNSCC abgeleitet wurden, wurden über Nacht ausgesät und inkubiert. Nach 24 h wurden die Zellen mit einem Kinase-Inhibitor behandelt. Nach 3 Stunden wurden die ersten beiden Dosen von 5 Gy pro Fraktion verabreicht. Nach 24 Stunden wurde die zweite Dosis verabreicht und die Zellen wurden erneut über Nacht inkubiert. Parallel dazu wurden 48 h vor Beginn der Co-Kultur T-Zellen aus dem Blut eines gesunden Spenders isoliert. Zunächst wurden PBMCs mit Hilfe von Dichtegradientenzentrifugationsröhrchen und einem sterilen Trennmedium isoliert. PBMCs wurden mit einer Zellzählkammer gezählt, und CD8+ T-Zellen wurden mit dem CD8+ T-Zell-Isolationskit isoliert. Isolierte CD8+ T-Zellen wurden dann mit CFSE (1 μM) gefärbt und anschließend gezählt. T-Zellen wurden in CD3/CD28-vorbeschichtete Well-Platten mit einer Dichte von 1,5 x 106 Zellen/3 ml ausgesät. Nach 48 Stunden Stimulation wurden die T-Zellen geerntet, gezählt und in einer Dichte von 10.000 Zellen/10 μl resuspendiert. Zusätzlich wurden repräsentative Wells der ausgesäten Tumorzellen geerntet und gezählt, und das Medium wurde gegen alle verbleibenden Wells ausgetauscht. T-Zellen wurden den Tumorzellen im Verhältnis 1:1 zugesetzt. Die Co-Kultur wurde nach 96 h geerntet, der Überstand bei -20 °C gelagert und die Zellen wurden gefärbt und mittels Durchflusszytometrie gemessen (Abbildung 1).
T-Zellen wurden durch Größenausschluss und CD3/CD8-Positivität identifiziert. Das CFSE-Signal von CD3+/CD8+ positiven Zellen zeigte die Verteilung von nicht-proliferativen T-Zellen (hohes CFSE-Signal) und ausgeprägten Subpopulationen von proliferierenden T-Zellen (Verlust der CFSE-Signalintensität). Der Anteil proliferierender T-Zellen wurde exemplarisch für die HSC4 HNSCC-Zelllinie gemessen (Abbildung 4A). Alle T-Zellen, die mindestens eine Teilung zeigten, wurden als "proliferativ" definiert. T-Zellen, die mehr als 3 Teilungen zeigten, wurden als "hochgradig proliferativ" definiert (Abbildung 4B). Für die HPV-negative Zelllinie HSC4 wurde ein leichter Anstieg der Proliferationsrate festgestellt, wenn T-Zellen mit bestrahlten Tumorzellen kokultiviert wurden. RT und RT plus Hemmung von ATR führten zu einer signifikant erhöhten konsekutiven T-Zell-Proliferation im Vergleich zu Co-Kulturen von RT plus ATM-Hemmung behandelten HSC4-Zellen mit T-Zellen. In Bezug auf die "hochproliferative" Fraktion der T-Zellen war die Vorbehandlung von HSC4-Zellen mit RT plus Hemmung von ATR am effektivsten bei der Stimulierung der T-Zellproliferation (Abbildung 4B).
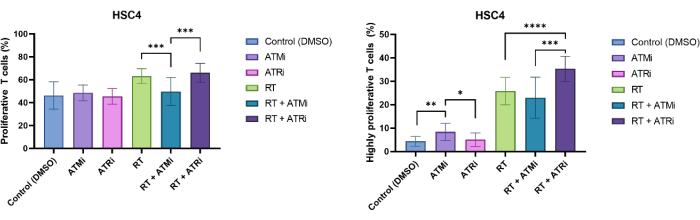
Abbildung 4: Proliferation von CFSE-gefärbten T-Zellen zur Analyse von HSC4-behandlungsabhängigen Veränderungen der T-Zellproliferation. (A) Anteil proliferierender T-Zellen nach 96 Stunden Co-Kultur mit vorbehandelten HPV-negativen HSC4-Tumorzellen. Die T-Zellproliferation war nach der Co-Kultur mit RT+ATMi-behandelten HSC4-Tumorzellen im Vergleich zu RT oder RT plus ATRi-Behandlung signifikant geringer. (B) Die RT von HSC4-Tumorzellen induzierte einen höheren Anteil an stark proliferierenden T-Zellen (mehr als drei Zellteilungen. Nach Co-Kultur von RT+ATRi vorbehandelten HSC4-Tumorzellen mit T-Zellen war der Anteil an hochproliferativen T-Zellen am höchsten. Die Balken zeigen Daten aus vier unabhängigen Experimenten mit den T-Zellen von vier unabhängigen, gesunden Spendern (n = 4; Mittelwert ± SD). (*p≤ 0,05, **p≤ 0,01, ***p≤ 0,001, ****p≤ 0,0001; statistische Signifikanz getestet durch Vergleich aller experimentellen Bedingungen unter Verwendung von zweiseitigem Mann-Whitney-U für nicht normalverteilte Daten). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Der proliferationsbasierte CFSE-Verlust kann zur Quantifizierung der T-Zell-Proliferationsrate verwendet werden. Darüber hinaus werden mehrere Zelloberflächenmarker beschrieben, die mit der T-Zell-Aktivität assoziiert sind, wie z. B. CD25 und HLADR. Daher wurde die Analyse von CD25 und HLA-DR auf der Oberfläche aller CD3+ und CD8+ T-Zellen mittels Durchflusszytometrie durchgeführt (Abbildung 5). Die Expression kann quantifiziert werden, wennunterschiedliche CD25-hohe- oder HLA-DR-hohe Subpopulationen nachweisbar sind und durch Gating dieser hochpositiven Zellen unterschieden werden können (Abbildung 3B). Wenn keine eindeutigen Subpopulationen nachweisbar sind, kann die mittlere Fluoreszenzintensität (MFI) der gesamten Population gemessen und zustandsbasierte Verschiebungen des MFI quantifiziert werden (Abbildung 3C).
Exemplarisch wurde die durch den MFI identifizierte Expression von CD25 und HLA-DR nach 96 h Co-Kultur mit vorbehandelten HSC4-Tumorzellen analysiert (Abbildung 5). Nach Co-Kultur von T-Zellen mit RT-behandelten HSC4-Tumorzellen war die Expression von CD25 auf den T-Zellen stark herunterreguliert. Bemerkenswert ist, dass die Vorbehandlung der Tumorzellen mit nur ATMi zu einer signifikant verringerten Expression von CD25 im Vergleich zur ATRi-Vorbehandlung führte. Obwohl die RT in der kombinierten Umgebung mit RT zu einer verminderten Expression von CD25 auf T-Zellen führte, führte die Kombination von RT mit ATMi zu einer erhöhten Expression von CD25 im Vergleich zu RT plus ATRi (Abbildung 5A). In Bezug auf die Expression von HLA-DR auf T-Zellen führte die RT im Allgemeinen zu einer Hochregulation von HLA-DR, aber auch hier wurde in Kombination mit ATMi oder ATRi ein anderes Verhalten beobachtet (Abbildung 5B). T-Zellen, die mit HSC4-Zellen kokultiviert wurden, die mit einer Kombination von RT+ATRi vorbehandelt wurden, erhöhten die Expression von HLA-DR im Vergleich zu RT allein oder RT+ATMi.
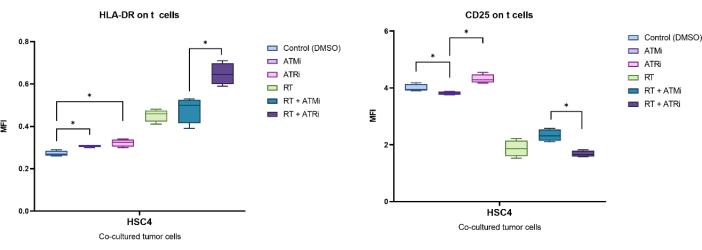
Abbildung 5: Expression der Aktivierungsmarker CD25 und HLA-DR auf der Oberfläche von T-Zellen nach 96 h Co-Kultur mit vorbehandelten HSC4-Tumorzellen. Die Expression von CD25 und HLA-DR wurde anhand der Verschiebung des MFI der gesamten T-Zell-Population analysiert. Die RT von HSC4 führte zu einer verminderten Expression von CD25 auf T-Zellen und einer erhöhten Expression von HLA-DR. Die Kombination von RT mit ATMi führte zu signifikant unterschiedlichen Expressionsmustern im Vergleich zu RT plus ATRi. Die Boxplots zeigen Daten aus vier unabhängigen Experimenten mit den T-Zellen von vier unabhängigen, gesunden Spendern (n = 4; Mittelwert ± SD). Die Daten wurden analysiert, indem alle experimentellen Bedingungen mit einem zweiseitigen Mann-Whitney-U-Test für nicht normalverteilte Daten verglichen wurden (*p≤ 0,050). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diese Daten deuten exemplarisch darauf hin, dass die Behandlung von Tumorzellen, in diesem Fall von HSC4 HNSCC-Zellen, die Immunogenität der Tumorzellen beeinflusst. Dies kann durch die Co-Kultivierung der Tumorzellen mit prästimulierten T-Zellen überwacht werden, was zu einem vielfältigen Proliferationsverhalten und der Expression von Aktivierungsmarkern auf humanen T-Zellen führt, basierend auf dem angewandten Tumorzellbehandlungsregime.
Diskussion
Das hier vorgestellte Protokoll bietet eine schnelle und einfache Methode, um die proliferative Kapazität von T-Zellen zusammen mit ihrem Aktivierungsstatus in einer Co-Kultur mit vorbehandelten Tumorzellen zu analysieren. Auf diese Weise können die Auswirkungen verschiedener Behandlungsmodalitäten wie RT, CT oder TT auf die Aktivität und Proliferation von T-Zellen leicht gescreent werden, was die Grundlage für spätere tiefergehende immunologische Analysen vielversprechender Ansätze bildet. Die repräsentativen Ergebnisse in diesem Manuskript belegen, dass dieser allogene T-Zell-Co-Kultur-Assay eine gute Leistung erbringt. Signifikante Unterschiede in der T-Zellproliferation sowie in der T-Zellaktivität wurden für die Co-Kultur mit differentiell vorbehandelten humanen HSC4 (HNSCC)-Zellen beobachtet (Abbildung 4 und Abbildung 5). Es zeigte sich, dass die Behandlung des HSC4 entweder mit RT allein oder in Kombination mit TT zu einer erhöhten Proliferation der T-Zellen führte, insbesondere im Hinblick auf den hochproliferativen Anteil der T-Zellen (Abbildung 4). Im Einklang mit der Quantifizierung der Proliferationsrate wurde auch der Aktivierungszustand der T-Zellen durch die verschiedenen Behandlungsschemata unterschiedlich beeinflusst. Zusammenfassend lässt sich sagen, dass die Anwendung von RT zu einer starken Herunterregulierung der Expression von CD25 auf T-Zellen führte, während die Expression von HLA-DR hochreguliert war. RT induziert DNA-Schäden in den bestrahlten Tumorzellen, was wiederum zu zellulären Stressreaktionen führt, die die Freisetzung von stress- und schadensassoziierten Molekülen und Zytokinen sowie die Expression von immunmodulierenden Zelloberflächenliganden umfassen19.
Die Kombination von RT mit TT in Form von DNA-Reparatur-Inhibitoren verstärkt und erhält diese Effekte weiter, da die Tumorzellen die RT-induzierten DNA-Schäden nicht effizient reparieren können. Dies könnte die Sekretion immunogener Faktoren und die Expression immunogener Zelloberflächenliganden weiter fördern und aufrechterhalten20. Dementsprechend haben wir bereits gezeigt, dass die Behandlung von Tumorzellen (HNSCC) mit RT und DNA-Schadenshemmern den Immunphänotyp auf der Tumorzelloberfläche verändert. Die Modulation des Immunphänotyps umfasste die Regulation des immunstimulierenden Moleküls ICOS-L sowie des immunsuppressiven Moleküls PD-L111. Als Reaktion auf diese Modulation des Immunphänotyps der Tumorzellen könnten T-Zellen auch die Expression von Aktivierungsmarkern auf ihrer Zelloberfläche regulieren. Darüber hinaus ist bekannt, dass RT die Freisetzung von proinflammatorischen Zytokinen wie IFNγ oder IL-6 stimuliert, die sich wiederum auf die Proliferation und Aktivität von T-Zellen auswirken21. Die Expression von Aktivierungsmarkern auf T-Zellen ist ein hochdynamischer Prozess. Daten von Zimmerman et al.22 zeigten, dass CD25 24 h nach der Stimulation stark exprimiert wird, aber nach 96 h nach der Stimulation wieder herunterreguliert wird. Dies steht im Einklang mit unseren Erkenntnissen. Im Gegensatz dazu handelt es sich bei der HLA-DR um einen Aktivierungsmarker in der Spätphase, dem in der Regel ein Anstieg von CD25 und CD69 vorausgeht23. Auffällig ist, dass die Kombination von RT + ATRi zu einer signifikanten Hochregulation von HLA-DR auf T-Zellen im Vergleich zu RT + ATMi führt. Dieser Befund steht im Einklang mit der Arbeit von Dillion et al., die synergistische Effekte von ATRi + RT bei der Induktion eines inflammatorischen TME zeigten24. Eine weniger ausgeprägte Modulation der T-Zellaktivität und -proliferation wurde durch die Tumorzellbehandlung allein mit den Kinase-Inhibitoren erreicht (Abbildung 5). Dies könnte daran liegen, dass die DNA-Reparaturinhibitoren ATMi und ATRi als Enhancer der RT-induzierten DNA-Schädigung fungieren und daher als Monotherapie nicht sehr immunogen sind. Wir haben bereits die minimale Toxizität von ATMi oder ATRi allein nachgewiesen11. Zusammenfassend bestätigen die Ergebnisse das immunogene Potenzial der RT (und der TT mit DNA-Schadensinhibitoren) und deuten darauf hin, dass dieses experimentelle System für das Screening der immunstimulierenden Fähigkeiten verschiedener Behandlungsmodalitäten in menschlichen Kopf- und Halskrebszellen geeignet ist.
Auch wenn die hier vorgestellte Methode einfach und robust ist, gibt es einige kritische Schritte im Protokoll, die im Vorfeld berücksichtigt werden müssen. Das Vorhandensein hoher EDTA-Konzentrationen während des Isolierungsprozesses sowie während der durchflusszytometrischen Analyse der T-Zellen ist erforderlich. Daher wird empfohlen, das Spenderblut in EDTA-beschichtete Entnahmeröhrchen zu entnehmen und einen mit 10 mM EDTA ergänzten MACS-Puffer zu verwenden, um eine effiziente T-Zell-Isolierung und die Erzeugung einer homogenen Einzelzellsuspension für die durchflusszytometrische Auswertung zu gewährleisten. Darüber hinaus ist die Zusammensetzung des T-Zell-Mediums von großer Bedeutung für die proliferative Kapazität von T-Zellen. Nach dem Testen verschiedener Zusammensetzungen von Kulturmedien ergibt ein Medium mit niedrigen Konzentrationen von L-Arginin und L-Lysin das beste T-Zell-Proliferationssignal, da L-Arginin für den T-Zell-Metabolismus und das Überleben entscheidend ist und in einer geeigneten Konzentration zugeführt werden sollte25,26. Nichtsdestotrotz sollte man bedenken, dass L-Arginin- und L-Lysin-Lösungen nicht langfristig stabil sind und daher innerhalb von 3 Monaten verwendet werden müssen. Für eine optimale Stimulation muss das mit den Aminosäuren angereicherte T-Zell-Medium für jedes T-Zell-Kultivierungsexperiment frisch zubereitet werden. Darüber hinaus sind sowohl die Zellzahlen der T-Zellen als auch die der Tumorzellen kritische Faktoren, die berücksichtigt werden müssen, da sie sich auf die Auswertung des Experiments auswirken.
Für die Tumorzellen ist zum einen eine Zellkonfluenz von 70% bis 80% am Ende des Co-Kultur-Experiments gewünscht. Ein Überwachsen der Tumorzellen könnte zur Sekretion von Faktoren führen, die das Zellwachstum hemmen und in der Folge auch die Proliferationsrate der T-Zellen beeinflussen würden. Da das Wachstum der Tumorzellen stark von der einzelnen Zelllinie abhängt, empfehlen wir, das Wachstumsverhalten der jeweiligen Tumorzelllinie in verschiedenen Well-Platten und für die verschiedenen Behandlungsschemata gründlich zu testen. Weiterhin sollten die Tumorzellen und die T-Zellen in einem Verhältnis von 1:1 co-kultiviert werden. Daher ist es obligatorisch, für jede Behandlungsbedingung eine zusätzliche Vertiefung mit Tumorzellen zu säen, die für die Bestimmung der Zellzahl am Tag des Beginns der Co-Kultur verwendet werden kann. Auf diese Weise kann sichergestellt werden, dass eine geeignete Anzahl von T-Zellen für die verschiedenen Behandlungsbedingungen ausgesät wird. Hinsichtlich der Zellzahl der T-Zellen ist zu berücksichtigen, dass die Anzahl der T-Zellen, die von einem gesunden Spender isoliert werden können, sehr individuell ist. Die T-Zellen machen etwa 45 bis 70 % der PBMCs eines gesunden Spenders aus27. Somit sollte die benötigte Menge an T-Zellen bereits zum Zeitpunkt der Blutentnahme grob abgeschätzt werden. Darüber hinaus ist die T-Zelldichte wichtig für die Effizienz der T-Zell-Aktivierung und ihr Überleben. Folglich sollten die T-Zellen in einer Konzentration von mindestens 1,5 Millionen Zellen pro Vertiefung einer 6-Well-Platte in 3 ml T-Zellmedium für die anfängliche Stimulation vor Beginn der Co-Kultur ausgesät werden. Eine höhere T-Zell-Dichte ist möglich, eine geringere Dichte sollte jedoch vermieden werden. Die Aktivierung der T-Zellen kann auch morphologisch unter dem Mikroskop beurteilt werden, da aktivierte T-Zellen dazu neigen, Zellcluster zu bilden.
Da es sich bei dieser Methode um ein vereinfachtes experimentelles System zur Untersuchung der immunstimulierenden Fähigkeiten verschiedener Tumorzelllinien und Behandlungsmodalitäten handelt, weist sie einige Einschränkungen auf, die berücksichtigt werden müssen. Erstens basiert dieser Assay auf einem allogenen System, was bedeutet, dass die Spender-T-Zellen nicht HLA-gematcht mit der jeweiligen Tumorzelllinie sind. Somit könnte dieses HLA-Mismatch bereits eine T-Zell-Stimulation und in der Folge eine T-Zell-Proliferation ohne weitere Stimulation durch die Tumorzellbehandlung induzieren28. Um diesen unerwünschten Effekt zu quantifizieren, wurden isolierte T-Zellen ohne vorherige Stimulation mit CD28- und CD3-Antikörpern mit der HSC4-Zelllinie co-kultiviert. Es zeigte sich, dass in diesem Setting fast keine Proliferation induziert wurde, was darauf hindeutet, dass das HLA-Mismatch in diesem speziellen Versuchssetting nur geringe Auswirkungen auf die T-Zell-Aktivierung hat. Darüber hinaus wurden signifikante Unterschiede in der T-Zell-Proliferation und -Aktivierung basierend auf der Tumorzellbehandlung trotz der allogenen Einstellung festgestellt. Nichtsdestotrotz sollte für die Etablierung dieses Assays die potentielle allogene induzierte Proliferation von T-Zellen einmalig zu Beginn der Experimente getestet und quantifiziert werden. Die empfohlenen Kontrollen "nur T-Zelle" und "nur Tumorzelle" sind für ein ausreichendes Gating erforderlich und bei jeder Wiederholung der Experimente obligatorisch. Nichtsdestotrotz muss man im Hinterkopf behalten, dass einem allogenen Co-Kultursystem eine tumorspezifische Antigenerkennung fehlt. Daher spiegelt dieses System möglicherweise nicht genau die spezifische Anti-Tumor-Reaktion wider, wie sie in vivo bei Patienten auftreten würde29,30. Für eine weitere Verbesserung wäre ein anspruchsvolleres Setting ein autologer Co-Kultur-Ansatz. In dieser Umgebung müssen von Patienten stammende Tumorbiopsien mit T-Zellen kultiviert und in Kultur gebracht werden, die aus dem peripheren Blut desselben Patienten isoliert wurden28. Dieser experimentelle Ansatz könnte jedoch nicht nur im Hinblick auf die Kultivierung von Primärtumorzellen, sondern auch in Bezug auf die Verfügbarkeit von patientenbezogenem Biomaterial eine Herausforderung darstellen. Eine weitere Einschränkung, die berücksichtigt werden muss, ist das Verhältnis der Tumorzellen zu den T-Zellen in der Co-Kultur. Da das empfohlene Verhältnis der Zellen 1:1 beträgt, spiegelt das Verhältnis nicht die physiologische Situation der TME bei Patientenwider 31. Diese Einschränkung muss jedoch in Kauf genommen werden, da bei niedrigeren T-Zellzahlen die Veränderungen der Proliferationsrate und des Aktivierungsstatus nicht quantifizierbar sind.
Im Bereich der Tumorimmunologie bietet dieser Assay die Möglichkeit, die Immunogenität verschiedener Behandlungsmodalitäten in einem einfachen und schnellen experimentellen in vitro-Setting zu untersuchen. Daher kann nicht nur durch das Pre-Screening der vielversprechendsten Ansätze in Bezug auf die Immunogenität, sondern auch durch In-vivo-Experimente Zeit gespart werden. Tiermodelle können reduziert werden, da in Tiermodellen nur vielversprechende Behandlungsschemata weiterverfolgt werden können, um das Immunsystem als Ganzes in einem Organismus abzubilden. Da dieser Assay auf menschlichen Primärzellen und menschlichen Krebszelllinien basiert, könnten die Ergebnisse in der Klinik besser übertragbar sein als Assays, die auf anderen Modellsystemen und Spezies basieren.
In Zukunft könnte dieser Assay moduliert und angepasst werden, um spezifischere Forschungsfragen zu beantworten. Man könnte zum Beispiel eine Behandlung der T-Zellen einbauen, um ein Behandlungsszenario widerzuspiegeln, das näher an der Situation bei den Patienten liegt. Da tumorreaktive T-Zellen hauptsächlich in der TME zu finden sind, sind sie gleichermaßen von lokalen Therapien wie RT betroffen und wahrscheinlich auch von systemischen Therapien wie CT oder TT32. Eine weitere Verbesserung des Assays ist die Co-Kultivierung von T-Zellen mit Tumorsphäroiden aus Tumorzelllinien oder auch die Co-Kultivierung mit patienteneigenen Tumororganoiden. Diese dreidimensionalen Kulturen sind eher vergleichbar mit der Struktur eines Tumors bei einem Patienten28. Schließlich kann auch die Auswertung des Experiments mittels Durchflusszytometrie leicht angepasst werden, um weitere Moleküle auf der Oberfläche von T-Zellen zu untersuchen oder die Expression von Immun-Checkpoint-Molekülen auf den Tumorzellen zu analysieren. Neben der Bestimmung des Immunphänotyps der T-Zellen könnte man die Zellkulturüberstände aus den Co-Kulturexperimenten zur Quantifizierung von sezernierten Zytokinen oder Chemokinen nutzen, um weitere Einblicke in die T-Zellaktivität zu gewinnen. Zusammenfassend lässt sich sagen, dass dieses Protokoll einen umfassenden, robusten und einfachen T-Zell- und Tumorzell-Co-Kultur-Assay liefert, der ein Screening auf die Immunogenität verschiedener Krebsbehandlungsmodalitäten ermöglicht. Da dieser Assay an spezifische Forschungsfragen angepasst werden kann, eignet er sich gut für den Einsatz im weiten Feld der Tumorimmunologie.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Forschung wurde teilweise vom Interdisziplinären Zentrum für Klinische Forschung Erlangen (IZKF Erlangen) und dem Bayerischen Zentrum für Krebsforschung (BZKF) gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL Cellstar tubes | Greiner Bio-One GmbH | 188271 | |
| 50 mL Cellstar tubes | Greiner Bio-One GmbH | 227261 | |
| 6 well cell culture plate sterile, with lid | Greiner Bio-One GmbH | 657160 | |
| 96 well cell culture plate sterile, F-bottom, with lid | Greiner Bio-One GmbH | 655180 | |
| AZD0156 | Selleck Chemicals GmbH | S8375 | |
| Berzosertib (VE-822) | Selleck Chemicals GmbH | S7102 | |
| CASYcups | OMNI Life Science GmbH & Co KG | 5651794 | |
| CASYton | OMNI Life Science GmbH & Co KG | 5651808 | |
| CD25a, PE-Dazzle594, Mouse IgG1 | Biolegend | 356126 | |
| CD28-UNLB | Beckmann Coulter, Inc. | IM1376 | |
| CD3a,Krome Orange, Mouse IgG1 | Beckmann Coulter, Inc. | B00068 | |
| CD3e Monoclonal Antibody | Thermo-Fisher Scientific, Inc. | MA1-10176 | |
| CD4, APC, Mouse Anti-Human Mouse IgG1 | BD Pharmingen | 555349 | |
| CD8 MicroBeads, human | Miltenyi Biotec, Inc. | 130-045-201 | |
| CD8a, PerCP-Cy5.5, Mouse IgG1 | Biolegend | 300924 | |
| CellTracker Deep Red Dye | Thermo Fisher Scientific, Inc. | C34565 | |
| CFSE | Merck KGaA (Sigma-Aldrich) | 21888 | |
| DMEM (Dulbecco´s Modified Eagle´s Medium) | PAN-Biotech GmbH | P04-02500 | |
| DxFlex Flow Cytometer (with Auto Loader) | Beckmann Coulter, Inc. | C44326, C02846 | |
| EDTA disodium salt dihydrate | Carl Roth GmbH + Co. KG | 8043.2 | |
| FBS superior | Merck KGaA (Sigma-Aldrich) | S0615-500ML | |
| FBS superior | Merck KGaA (Sigma-Aldrich) | S0615-100ML | For production of heat-inactivated FBS. Heat up for 30 min at 56 °C with mixing to inactivate complement proteins. |
| Graph Pad Prism (version number 9) | GraphPad Software | - | |
| HLA-DR, DP, DQ Antibody, anti-human, APC-Vio770 | Miltenyi Biotec, Inc. | 130-123-550 | |
| Kaluza (version number 2.1) | Beckmann Coulter, Inc. | - | |
| L-Arginin | Merck KGaA (Sigma-Aldrich) | A8094-25G | |
| L-Lysin-monohydrochloride | Merck KGaA (Sigma-Aldrich) | L5626-100G | |
| MACS BSA Stock Solution | Miltenyi Biotec, Inc. | 130-091-376 | |
| MS Columns | Miltenyi Biotec, Inc. | 130-042-201 | |
| Neubauer-improved counting chamber | Paul Marienfeld GmbH & Co. KG | 640010 | |
| PBS | Merck KGaA (Sigma-Aldrich) | D8537-500mL | |
| Penicillin-Streptomycin | Thermo-Fisher Scientific, Inc. | 1514-122 | |
| ROTISep 1077 | Carl Roth GmbH + Co. KG | 0642.2 | |
| RPMI-1640 Medium | Merck KGaA (Sigma-Aldrich) | R1790 | |
| SepMate 50mL tubes | Stemcell Technologies | 85450 | |
| Trypan blue | Merck KGaA (Sigma-Aldrich) | T6146-25G | |
| Trypsin | Thermo-Fisher Scientific, Inc. | 15400054 |
Referenzen
- Hiam-Galvez, K. J., Allen, B. M., Spitzer, M. H. Systemic immunity in cancer. Nat Rev Cancer. 21 (6), 345-359 (2021).
- Mantovani, A., Allavena, P., Marchesi, F., Garlanda, C. Macrophages as tools and targets in cancer therapy. Nat Rev Drug Discov. 21 (11), 799-820 (2022).
- Waldman, A. D., Fritz, J. M., Lenardo, M. J. A guide to cancer immunotherapy: From t cell basic science to clinical practice. Nat Rev Immunol. 20 (11), 651-668 (2020).
- Dunn, G. P., Old, L. J., Schreiber, R. D. The immunobiology of cancer immunosurveillance and immunoediting. Immunity. 21 (2), 137-148 (2004).
- Mittal, D., Gubin, M. M., Schreiber, R. D., Smyth, M. J. New insights into cancer immunoediting and its three component phases--elimination, equilibrium and escape. Curr Opin Immunol. 27, 6-25 (2014).
- Irianto, T., Gaipl, U. S., Ruckert, M. Immune modulation during anti-cancer radio(immuno)therapy. Int Rev Cell Mol Biol. 382, 239-277 (2024).
- Burtness, B., et al. Pembrolizumab alone or with chemotherapy versus cetuximab with chemotherapy for recurrent or metastatic squamous cell carcinoma of the head and neck (keynote-048): A randomized, open-label, phase 3 study. Lancet. 394 (10212), 1915-1928 (2019).
- Hecht, M., et al. Safety and efficacy of single cycle induction treatment with cisplatin/docetaxel/ durvalumab/tremelimumab in locally advanced HNSCC: First results of checkered-CD8. J Immunother Cancer. 8 (2), e001378(2020).
- Chen, J. A., Ma, W., Yuan, J., Li, T. Translational biomarkers and rationale strategies to overcome resistance to immune checkpoint inhibitors in solid tumors. Cancer Treat Res. 180, 251-279 (2020).
- Solomon, B., Young, R. J., Rischin, D. Head and neck squamous cell carcinoma: Genomics and emerging biomarkers for immunomodulatory cancer treatments. Semin Cancer Biol. 52 (Pt 2), 228-240 (2018).
- Meidenbauer, J., et al. Inhibition of atm or atr in combination with hypo-fractionated radiotherapy leads to a different immunophenotype on transcript and protein level in HNSCC. Front Oncol. 14, 1460150(2024).
- Kumari, S., et al. Immunomodulatory effects of radiotherapy. Int J Mol Sci. 21 (21), 8151(2020).
- Wimmer, S., et al. Hypofractionated radiotherapy upregulates several immune checkpoint molecules in head and neck squamous cell carcinoma cells independently of the HPV status while icos-l is upregulated only on HPV-positive cells. Int J Mol Sci. 22 (17), 9114(2021).
- Derer, A., et al. Chemoradiation increases pd-l1 expression in certain melanoma and glioblastoma cells. Front Immunol. 7, 610(2016).
- Schatz, J., et al. Normofractionated irradiation and not temozolomide modulates the immunogenic and oncogenic phenotype of human glioblastoma cell lines. Strahlenther Onkol. 199 (12), 1140-1151 (2023).
- Xu, M. M., Pu, Y., Zhang, Y., Fu, Y. X. The role of adaptive immunity in the efficacy of targeted cancer therapies. Trends Immunol. 37 (2), 141-153 (2016).
- Olivo Pimentel, V., Yaromina, A., Marcus, D., Dubois, L. J., Lambin, P. A novel co-culture assay to assess anti-tumor cd8(+) t cell cytotoxicity via luminescence and multicolor flow cytometry. J Immunol Methods. 487, 112899(2020).
- Kulp, M., Diehl, L., Bonig, H., Marschalek, R. Co-culture of primary human t cells with leukemia cells to measure regulatory t cell expansion. STAR Protoc. 3 (3), 101661(2022).
- Ruckert, M., et al. Immune modulatory effects of radiotherapy as basis for well-reasoned radioimmunotherapies. Strahlenther Onkol. 194 (6), 509-519 (2018).
- Samstein, R. M., Riaz, N. The DNA damage response in immunotherapy and radiation. Adv Radiat Oncol. 3 (4), 527-533 (2018).
- Meeren, A. V., Bertho, J. M., Vandamme, M., Gaugler, M. H. Ionizing radiation enhances il-6 and il-8 production by human endothelial cells. Mediators Inflamm. 6 (3), 185-193 (1997).
- Zimmerman, M., et al. Ifn-gamma upregulates survivin and ifi202 expression to induce survival and proliferation of tumor-specific T cells. PLoS One. 5 (11), e14076(2010).
- Saraiva, D. P., et al. Expression of HLA-dr in cytotoxic t lymphocytes: A validated predictive biomarker and a potential therapeutic strategy in breast cancer. Cancers (Basel). 13 (15), (2021).
- Dillon, M. T., et al. Atr inhibition potentiates the radiation-induced inflammatory tumor microenvironment. Clin Cancer Res. 25 (11), 3392-3403 (2019).
- Geiger, R., et al. L-Arginine modulates T cell metabolism and enhances survival and anti-tumor activity. Cell. 167 (3), 829-842.e13 (2016).
- Rodriguez, P. C., Quiceno, D. G., Ochoa, A. C. L-Arginine availability regulates t-lymphocyte cell-cycle progression. Blood. 109 (4), 1568-1573 (2007).
- Lozano-Ojalvo, D., López-Fandiño, R., López-Expósito, I., et al. The impact of food bioactives on health: In vitro and ex vivo models. Verhoeckx, K., et al. , Springer International Publishing. Cham. 169-180 (2015).
- Gronholm, M., et al. Patient-derived organoids for precision cancer immunotherapy. Cancer Res. 81 (12), 3149-3155 (2021).
- Perez, C., Gruber, I., Arber, C. Off-the-shelf allogeneic t cell therapies for cancer: Opportunities and challenges using naturally occurring "universal" donor t cells. Front Immunol. 11, 583716(2020).
- Martinez Bedoya, D., Dutoit, D., Migliorini, D. Allogeneic car t cells: An alternative to overcome challenges of car t cell therapy in glioblastoma. Front Immunol. 12, 640082(2021).
- Schnellhardt, S., et al. The prognostic value of FOXP3+ tumor-infiltrating lymphocytes in rectal cancer depends on immune phenotypes defined by CD8+ cytotoxic T cell density. Front Immunol. 13, 781222(2022).
- Zitvogel, L., Kroemer, G. Oncoimmunology: A practical guide for cancer immunotherapy. , Springer International Publishing. Cham. 23-39 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten