Method Article
Analisi dell'attività delle cellule T umane in un contesto di co-coltura allogenica di cellule tumorali pretrattate
In questo articolo
Riepilogo
Il presente protocollo descrive un flusso di lavoro sperimentale che consente l'analisi ex vivo della stimolazione di cellule T umane in un sistema di co-coltura allogenica con cellule tumorali pretrattate.
Abstract
Le cellule T citotossiche svolgono un ruolo chiave nell'eliminazione delle cellule tumorali e sono, quindi, intensamente studiate nell'immunologia del cancro. La frequenza e l'attività delle cellule T citotossiche all'interno dei tumori e del loro microambiente tumorale (TME) sono ora biomarcatori prognostici e predittivi ben consolidati per numerosi tipi di tumore. Tuttavia, è noto che varie modalità di trattamento dei tumori, tra cui la radioterapia, la chemioterapia, l'immunoterapia e la terapia mirata, modulano non solo l'immunogenicità del tumore ma anche il sistema immunitario stesso. Di conseguenza, l'interazione tra cellule tumorali e cellule T richiede uno studio più intensivo in diversi contesti terapeutici per comprendere appieno il complesso ruolo delle cellule T durante la terapia tumorale. Per rispondere a questa esigenza, è stato sviluppato un protocollo per analizzare l'attività e la capacità proliferativa delle cellule T citotossiche umane (CD8+) in co-coltura con cellule tumorali pretrattate. In particolare, le cellule T CD8+ provenienti da donatori sani vengono colorate con il marcatore di proliferazione non tossico carbossifluoresceina diacetato succinimidil estere (CFSE) e stimolate utilizzando piastre rivestite di CD3/CD28. Successivamente, le cellule T vengono co-coltivate con cellule tumorali pretrattate. Come lettura, la proliferazione delle cellule T viene quantificata misurando la distribuzione del segnale CFSE e valutando l'espressione dei marcatori di attivazione superficiale tramite citometria a flusso. Questo può essere ulteriormente integrato quantificando il rilascio di citochine utilizzando il saggio di immunoassorbimento enzimatico (ELISA). Questo metodo facilita la valutazione dei cambiamenti indotti dal trattamento nell'interazione tra cellule tumorali e cellule T, fornendo una base per analisi più dettagliate delle modalità di trattamento dei tumori e della loro immunogenicità in un contesto umano ex vivo . Inoltre, contribuisce alla riduzione delle analisi precliniche in vivo .
Introduzione
Al giorno d'oggi, sta diventando sempre più evidente che la crescita e il progresso dei tumori dipendono fortemente dall'efficace manipolazione e soppressione del sistema immunitario dell'ospite. Le cellule trasformate emergono ogni giorno, anche in un organismo sano. Tuttavia, la formazione di tumori macroscopici è un evento piuttosto raro, poiché le cellule trasformate emergenti vengono rimosse dall'organismo con un'elevata efficienza. Per la rimozione delle cellule maligne, entrano in azione diversi tipi di cellule immunitarie come le cellule T citotossiche, le cellule T natural killer (NK), le cellule NK o i macrofagi 1,2,3. Ciononostante, a volte possono emergere cloni cellulari trasformati che sopravvivono in uno stato di equilibrio con il sistema immunitario dell'ospite, che è caratterizzato da diverse strategie immunosoppressive del clone di cellule tumorali4. Alla fine, alcune cellule trasformate acquisiscono ulteriori funzioni che consentono alle cellule tumorali di sopprimere attivamente la risposta immunitaria, che di conseguenza porta alla crescita del tumore. Questa immunosoppressione è mediata da numerosi meccanismi, che includono l'espressione di ligandi immunosoppressivi sulle cellule tumorali o il reclutamento attivo o il priming di popolazioni di cellule immunitarie immunoregolatorie o immunosoppressive. Questo cosiddetto concetto di immunoediting dimostra il ruolo chiave della modulazione immunitaria durante la formazione e la crescita del tumore5.
Pertanto, non sorprende che il sistema immunitario sia oggi un obiettivo importante non solo nella terapia del cancro, ma anche come fattore predittivo e prognostico in molte entità tumorali e contesti terapeutici. Negli ultimi anni, gli inibitori del checkpoint immunitario (ICI) si sono presentati come opzioni terapeutiche promettenti in diverse entità tumorali solide, come il carcinoma a cellule squamose della testa e del collo (HNSCC) o il carcinoma polmonare non a piccole cellule (NSCLC) con l'obiettivo di modulare il microambiente tumorale (TME) verso una risposta immunitaria antitumorale più efficiente e una ridotta immunosoppressione delle cellule tumorali6. Gli inibitori del checkpoint immunitario mirano a potenziare l'uccisione delle cellule tumorali mediata dalle cellule T prendendo di mira le molecole del checkpoint immunitario come quelle della proteina di morte programmata 1 (PD-1) e del suo asse ligando 1 (PD1/PD-L1). Questo fatto sottolinea il ruolo chiave delle cellule T nell'immunità antitumorale. Nell'HNSCC, ad esempio, gli ICI sono stati approvati con successo come terapia di prima linea nell'HNSCC7 ricorrente e metastatizzato. In linea con ciò, la presenza di cellule T citotossiche nel TME, così come l'espressione di PD1 e PD-L1 sulle cellule tumorali e sulle cellule T di conseguenza, possono fungere da biomarcatori predittivi nell'HNSCC 8,9,10.
Anche se le cellule T hanno un ruolo cruciale nell'immunologia e nella terapia dei tumori, molte domande aperte sulle loro interazioni con il tumore devono ancora essere affrontate. Al giorno d'oggi, è noto che la risposta immunitaria tumorale è un processo dinamico e che l'immunogenicità dei tumori può cambiare nel corso della malattia e dei trattamenti. Diverse modalità di trattamento, come la chemioterapia (TC), la radioterapia (RT) o la terapia mirata (TT), sono particolarmente note per modulare il fenotipo immunologico delle cellule tumorali. La RT può guidare l'upregolazione delle molecole del checkpoint immunitario nel tessuto tumorale e alterare la frequenza delle cellule infiltranti il tumore11,12. La TT, d'altra parte, può anche approvare cambiamenti favorevoli all'interno del tumore e del TME attraverso la modulazione diretta della risposta immunitaria adattativa 13,14,15,16. Tuttavia, queste modulazioni sono difficili da studiare nei pazienti, in quanto ciò richiederebbe l'esame ripetitivo del tessuto tumorale durante il corso del trattamento. Pertanto, sono necessari sistemi modello sperimentali conclusivi per studiare il fenotipo immunologico dinamico delle cellule tumorali e delle cellule T e, soprattutto, le loro interazioni.
Pertanto, allo scopo di analizzare l'attività delle cellule T e le interazioni tra cellule tumorali e cellule T, è necessario un test completo di co-coltura ex vivo che sia facile da implementare in qualsiasi laboratorio basato su un semplice lavoro di coltura cellulare e analisi di citometria a flusso comunemente utilizzate. Sulla base della letteratura esistente e disponibile, finora non è stato pubblicato alcun protocollo di facile utilizzo e comunemente usato per quanto riguarda la co-coltura di cellule T e cellule tumorali. Anche se recentemente sono stati pubblicati diversi saggi di co-coltura per cellule T e organoidi tumorali, la tecnica di coltura cellulare 3D non è ancora implementata come tecnica standard in tutti i laboratori. Pertanto, forniamo un protocollo per l'uso nella coltura cellulare 2D, che potrebbe anche essere stabilito per la coltura cellulare 3D in futuro. Altri protocolli di co-coltura 2D sono spesso più complessi, in quanto richiedono, ad esempio, la trasduzione di cellule tumorali con luciferasi17 o sono adatti solo per neoplasie ematologiche (co-coltura di cellule T non compatibili con cellule leucemiche)18. Nel test qui descritto, le cellule T di donatori sani normali vengono isolate dal sangue periferico e stimolate con anticorpi anti-CD3 e anti-CD28. Successivamente, le cellule T vengono co-coltivate con cellule tumorali (pre)trattate al fine di analizzare la capacità proliferativa delle cellule T e la loro attività tramite citometria a flusso. In tal modo, gli effetti di diverse modalità di trattamento sul fenotipo immunitario delle cellule tumorali, come RT, TC o TT, che a loro volta influenzano l'attività e la proliferazione delle cellule T, possono essere facilmente sottoposti a screening e utilizzati come base per analisi meccanicistiche più approfondite e analisi precliniche in vivo selezionate consecutive. Il test qui descritto offre una configurazione facile da usare, in quanto non sono necessari dispositivi, tecniche o materiali non convenzionali. Inoltre, il test può essere facilmente adattato a diverse linee cellulari tumorali o a specifici sottogruppi di cellule T (ad esempio, cellule T CD4+). Utilizzando questa tecnica, si ottiene un'elevata standardizzazione e riproducibilità.
Protocollo
Questo test prevede prelievi di sangue e la coltivazione di cellule primarie umane. Pertanto, un voto etico è obbligatorio per queste analisi. Tutti i risultati presentati in questo manoscritto sono coperti dall'approvazione etica dello studio IMMO-NHD e il consenso informato scritto è stato ottenuto da tutti i donatori. L'approvazione è stata concessa dal comitato di revisione istituzionale della Friedrich-Alexander-Universität Erlangen-Nürnberg il 9 novembre 2022 (numero di domanda 21-415-B). Le cellule tumorali HSC4 utilizzate in questo studio provengono da una linea cellulare disponibile in commercio.
Questo test esemplifica tutte le fasi del test di co-coltura delle cellule T e delle cellule tumorali, utilizzando la linea cellulare HSC4 del carcinoma a cellule squamose della testa e del collo (HNSCC) umana in un contesto di trattamento che coinvolge la radioterapia (RT) e due inibitori specifici della chinasi. Di conseguenza, parametri come il numero di cellule, i tempi di tripsinizzazione e gli schemi di trattamento sono specifici per questo contesto di co-coltura e dovrebbero essere adattati per altre linee cellulari tumorali (vedere anche la sezione Discussione). Tutte le fasi di centrifugazione sono state eseguite a temperatura ambiente. In questo studio, gli inibitori della chinasi AZD0156 e VE-822 sono stati utilizzati per colpire il sistema di riparazione del danno al DNA (DDR) delle cellule HSC4. AZD0156 (Selleckchem) inibisce la proteina mutata dell'atassia teleangectasia (ATM), mentre VE-822 (Selleckchem) ha come bersaglio l'atassia teleangectasia e la proteina correlata a Rad3 (ATR). Entrambi gli inibitori sono stati discussi come potenziali agenti per aumentare la radiosensibilità nelle cellule tumorali. Vengono disciolti in dimetilsolfossido (DMSO) e conservati a -20 °C. La Figura 1 fornisce un diagramma di flusso del test ex vivo , che descrive in dettaglio la co-coltura di cellule tumorali pretrattate con cellule T CD8+ umane stimolate con CD3/CD28. I reagenti e le attrezzature utilizzate sono elencati nella Tabella dei Materiali.
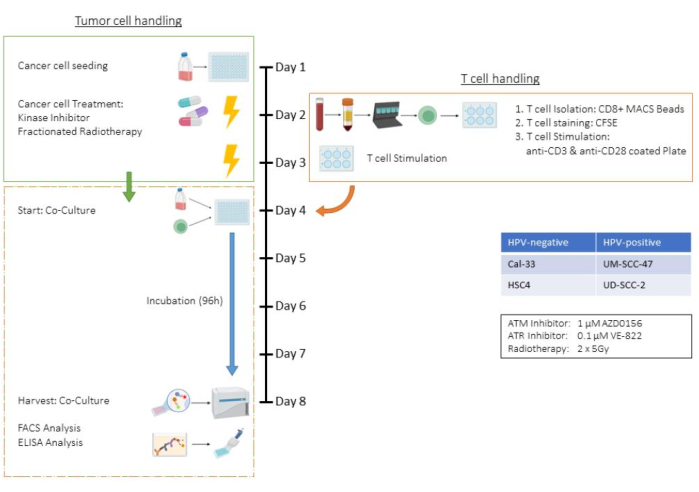
Figura 1: Diagramma di flusso del saggio ex vivo di cellule tumorali pretrattate in co-coltura con cellule T CD8+ umane stimolate (CD3/CD28). Giorno 1: Semina delle cellule tumorali HSC4. Giorno 2: Trattamento delle cellule tumorali e isolamento, colorazione CFSE e semina in piastre rivestite di CD3/CD28 di cellule T CD8+ umane. Giorno 3: Trattamento delle cellule tumorali. Giorno 4: Conteggio dei pozzetti rappresentativi delle cellule tumorali. Raccolta e conteggio di tutte le cellule T. Co-coltivazione di cellule T e cellule tumorali HSC4 in un rapporto 1:1. Giorno 5-Giorno 8: Incubazione in co-coltura. Giorno 8: Raccolta della co-coltura, congelamento dei surnatanti, colorazione a base di anticorpi e analisi delle cellule mediante citometria a flusso. Clicca qui per visualizzare una versione più grande di questa figura.
1. Semina delle cellule tumorali (Giorno 1)
NOTA: Temporizzazione: 1 h. Le cellule tumorali umane HSC4 vengono seminate da flaconi di coltura cellulare T75 in piastre a 96 pozzetti.
- Scartare il surnatante (D10 Medium) del flacone per colture cellulari.
- Lavare le cellule tumorali con 5 mL di PBS caldo a 37 °C. Scartare PBS.
- Poiché le cellule tumorali HSC4 tendono ad aderire strettamente alle fiasche di coltura cellulare, si raccomanda la tripsinizzazione in due fasi per garantire che tutte le cellule siano staccate. Per questo, aggiungere 3 ml di tripsina e posizionare il flacone su una piastra riscaldante a 37 °C per 3 minuti, quindi scartare la tripsina con una pipetta.
- Aggiungere altri 3 ml di tripsina, posizionare il flacone sulla piastra riscaldante e attendere fino a quando non si raggiunge la sospensione a cellula singola. Controllare al microscopio la sospensione di una singola cellula.
- Arrestare la tripsina aggiungendo il doppio volume di D10 Medium (DMEM integrato con il 10% di siero fetale bovino e l'1% di penicillina-streptomicina), risospendere accuratamente e trasferire le cellule in una provetta da centrifugazione da 50 mL.
- Determinare il numero di cellule della sospensione a cellula singola, quindi calcolare la quantità totale di cellule tumorali in base al volume totale. Per la determinazione della conta cellulare si consiglia una camera cellulare Neubauer.
- Centrifugare la provetta da centrifugazione da 50 ml contenente le cellule tumorali a 300 x g per 5 minuti a temperatura ambiente. Scartare il surnatante.
- Risospendere le cellule tumorali HSC4 in un volume adeguato di terreno D10 per raggiungere una concentrazione di 15.000 cellule in un terreno da 200 μl.
- In base ai trattamenti pianificati delle cellule tumorali, seminare per ogni condizione almeno 3 pozzetti contenenti 15.000 cellule tumorali HSC4 in terreno da 200 μL in due piastre da 96 pozzetti (1x campione, 1x controllo solo cellule tumorali, 1x pozzetto di conteggio delle cellule per la giornata di co-coltura).
NOTA: Il numero di celle dipende dalla linea cellulare e dal tempo di raddoppio. Il numero di cellule per i diversi trattamenti deve essere esaminato nei pre-esperimenti per evitare il 100% di confluenza durante l'incubazione. - Incubare le cellule per 24 ore a 37 °C e 5% di CO2 e umidità satura.
2. Trattamento delle cellule tumorali (Giorno 2)
NOTA: Temporizzazione: 3-5 h. Dopo un tempo di incubazione di 24 ore, le cellule HSC4 precedentemente seminate possono essere trattate secondo lo schema di trattamento desiderato. In questo caso esemplare, le cellule tumorali vengono trattate con un inibitore di ATM o ATR. Qui, inoltre, una delle due piastre da 96 pozzetti viene irradiata con 2x 5 Gy in seguito.
- Preparare gli inibitori delle chinasi per ottenere una concentrazione di 1 μM AZD0156 (inibitore ATM) e 0,1 μM VE-822 (inibitore ATR) per trattare le cellule tumorali.
- Trattare le cellule tumorali di conseguenza, ad esempio, trattando una fila di campioni con 3,1 μL dell'inibitore ATM e una seconda con 3,1 μL dell'inibitore ATR in ciascuna piastra.
- Dopo 3-5 ore di incubazione a 37 °C, irradiare una piastra con 5 Gy.
- Dopo l'incubazione per altre 24 ore, irradiare nuovamente la stessa piastra con 5 Gy.
3. Isolamento delle cellule T (giorno 2)
NOTA: Tempistica: 4 ore. Le cellule T CD8+ vengono isolate magneticamente con microsfere anti-CD8 da PBMC dopo centrifugazione in gradiente di densità di sangue periferico (PB) derivato da donatori adulti sani. Le cellule T isolate vengono quindi colorate con CFSE e incubate in una piastra a pozzetti rivestita di CD3/CD28 per la stimolazione. Per essere in grado di lavorare in modo rispettoso delle risorse e dei materiali nelle fasi successive, è importante stimare la quantità di cellule T necessarie per gli esperimenti di co-coltura. A seconda del donatore, da circa 45 ml di sangue EDTA possono essere isolate tra 50.000.000 e 100.000.000 di PBMC. Circa il 10% del totale delle PBMC sono cellule T CD8+.
- Centrifugazione in gradiente di densità
- Trasferire 3-5 provette di sangue EDTA da 9 mL derivate da un donatore sano in due provette da centrifugazione da 50 mL.
- Riempire entrambe le provette da centrifugazione fino a 50 mL con PBS + 2% FBS.
- Preparare sei provette da centrifugazione (con inserto in plastica per la separazione delle PBMC) e riempire ciascuna con 15 mL di terreno a gradiente di densità a freddo + 4 °C.
- Sovrapporre con cura il terreno di gradiente di densità con 12-15 mL di sangue diluito dal passaggio 3.1.2.
- Centrifugare a 1200 x g per 10 min (senza decelerazione).
- Preparazione di una piastra a 6 pozzetti per la stimolazione delle cellule T
- Preparare gli anticorpi: preparare una soluzione anti-CD3 (clone OKT3) di 1 mg/mL con PBS. Preparare una soluzione anti-CD28 (clone 28.2) di 0,1 mg/mL con PBS.
- Raccomandazione: utilizzare il tempo durante la centrifugazione (passaggio 3.1.5) per la preparazione delle soluzioni di rivestimento e il rivestimento dei pozzetti stessi per la stimolazione delle cellule T.
- Miscelare 5 μL di soluzione di anticorpi CD3 con 4,995 μL di PBS e 50 μL di soluzione di anticorpi CD28 con 4,950 μL di PBS (concentrazione finale: 1 μg/μL).
- Aggiungere 1.000 μl di entrambe le soluzioni anticorpali a ciascun pozzetto della piastra a 6 pozzetti.
- Rivestire 2 o 3 pozzetti, a seconda della quantità prevista di cellule T, della piastra a 6 pozzetti e incubare per almeno 2 ore a 37 °C.
- Preparazione del terreno delle cellule T
NOTA: Preparazione di 100x L-Lisina: Sciogliere 200 mg di L-Lisina cloridrato in 50 ml di acqua distillata in una provetta da centrifuga da 50 ml. Sterilizzare la soluzione utilizzando un filtro per siringa da 0,2 μm e trasferirla in una nuova provetta da 50 mL utilizzando una siringa da perfusione da 50 mL. Conservare la soluzione a 4-8 °C e utilizzarla entro 3 mesi. Preparazione di 15 mM di L-Arginina: Sciogliere 26 mg di L-Arginina in 10 mL di DPBS. Sterilizzare la soluzione utilizzando un filtro per siringa da 0,2 μm e trasferirla in una nuova provetta utilizzando una siringa da 10 mL. Conservare la soluzione a 4-8 °C e utilizzarla entro 3 mesi.- Utilizzare il tempo di centrifugazione come indicato al punto 1.5 per preparare il terreno delle cellule T.
- Preparare circa 10-30 ml di terreno di coltura delle cellule T, a seconda della quantità prevista di cellule T.
- Miscelare il terreno RPMI privo di L-Arginina e L-Lisina con il 10% di FBS inattivato termicamente, l'1% di penicillina-streptomicina, l'1% di L-Arginina, l'1% di L-Lisina e l'1% di L-Glutammina. Esempio: 25,8 mL di terreno RPMI + 3 mL di FBS + 0,3 mL di Pen/Strep + 0,3 mL di L-Arginina + 0,3 mL di L-Lisina + 0,3 mL di L-Glutammina.
- Conservare il terreno rimanente a 4 °C per i due giorni successivi per essere utilizzato il giorno 4 - Inizio della co-coltura. Per ogni nuova esecuzione dell'intero esperimento, si raccomanda di preparare un terreno di coltura fresco.
- Isolamento PBMC
- Dopo la centrifugazione (punto 3.1.5), trasferire il surnatante in quattro nuove provette da centrifugazione da 50 mL ed eliminare le provette usate.
- Riempire le provette da centrifugazione fino a 50 mL con PBS + 2% FBS.
- Centrifugare a 300 x g per 8 min a temperatura ambiente.
- Scartare il surnatante, risospendere il pellet cellulare in 1 mL di PBS + 2% FBS ciascuno e combinarli in due provette da centrifugazione.
- Riempire le provette a 50 mL con PBS + 2% FCS.
- Centrifugare a 120 x g per 10 min a temperatura ambiente.
- Scartare il surnatante, risospendere con cura il pellet cellulare in 1 mL di PBS +2% FBS e combinare i due pellet in un falco.
- Riempire la provetta da centrifugazione fino a 50 mL con PBS + 2% FBS.
- Conta il numero totale di PBMC utilizzando una camera di conteggio Neubauer. Raccomandazione: diluire la sospensione cellulare 1:10 con blu di tripano per il conteggio.
- Separazione delle cellule T CD8+
NOTA: Utilizzare CD8 MicroBeads human insieme a colonne di separazione magnetica (MS) e tampone MACS+ (Magnetic Activated Cell Sorting Plus) per separare magneticamente le cellule T CD8+ secondo il protocollo del produttore (vedere la Tabella dei materiali).- Centrifugare le PBMC (dopo il conteggio delle cellule) a 300 x g per 10 minuti.
- Scartare il surnatante e risospendere il pellet cellulare in 80 μL di tampone MACS+ (500 mL di PBS integrati con 10 mM di EDTA e 0,5% di BSA) per 10-7 cellule (ad esempio, 60.000.000 di PBMC vengono risospese in 6 x 80 μL = 480 μL di tampone).
NOTA: Esempio: 465 mL di PBS + 10 mL (0,5 M) di EDTA e 25 mL di soluzione BSA-Stock. - Aggiungere 20 μl di microsfere CD8 per 10-7 cellule e mescolare accuratamente pipettando verso l'alto e verso il basso. Incubare per 15 minuti a 4 °C.
- Dopo l'incubazione, lavare le cellule aggiungendo 2 mL di tampone MACS+ per 10-7 cellule.
- Centrifugare a 300 x g per 10 min a temperatura ambiente. Scartare il surnatante e risospendere le cellule in 1.000 μL di tampone MACS+.
- Inserire due colonne MS nel magnete e posizionare due provette da centrifugazione da 15 mL al di sotto.
- Preparare le colonne con 500 μL di tampone MACS+. Il tampone scorre attraverso le colonne e può essere raccolto nelle provette di centrifugazione sottostanti. Questo passaggio può essere eseguito anche parallelamente al passaggio 3.5.6.
- Quindi, pipettare la sospensione cellulare in modo uniforme nelle colonne MS preparate. Il flusso può essere raccolto nelle stesse provette da centrifugazione. Ora contiene tutte le cellule CD8-negative non marcate.
- Lavare le colonne aggiungendo 3 x 500 μL di tampone MACS+. Aggiungere un nuovo buffer sopra le colonne solo non appena si sono asciugate o hanno smesso di gocciolare.
- Etichettare le cellule finali raccolte come "Flow-through" o "CD8 negative" e combinarle in un'unica provetta da centrifugazione. Questo può essere successivamente utilizzato per misurare la purezza dell'isolamento tramite citometria a flusso.
- Prendi una nuova provetta da centrifugazione da 15 ml etichettata come "cellule T CD8+". Le colonne vengono rimosse dal magnete e posizionate sulla provetta da centrifugazione.
- Lavare entrambe le colonne contenenti le cellule T CD8 positive marcate magneticamente con 1.000 μL di tampone MACS+ ciascuna. Pertanto, pipettare il tampone nelle colonne e iniziare immediatamente a spingere con cautela la soluzione fuori dalle colonne utilizzando lo stantuffo fornito dal produttore.
NOTA: Eseguire questa operazione per entrambe le colonne, raccogliendo 2 mL di sospensione totale di cellule T in centrifugazione. - Contare il numero totale di cellule T utilizzando una camera di conteggio Neubauer. Raccomandazione: Utilizzare una diluizione 1:4 con il blu di tripano.
- Colorazione delle cellule T con CFSE
- Centrifugare le cellule T isolate a 300 x g per 5 minuti.
- Durante la centrifugazione, preparare la soluzione di colorazione CFSE mescolando 1,1 μL di soluzione CFSE in 10 mL di PBS. La concentrazione finale deve essere di 1 μM.
- Scartare il surnatante e risospendere il pellet di cella in 1.000 μl di PBS. Centrifugare a 300 x g per 5 min.
- Scartare il surnatante e risospendere il pellet di cella in 2000 μL di soluzione colorante CFSE (1 μM). Incubare per 20 minuti a 37 °C.
- Centrifugare le cellule colorate a 300 x g per 5 minuti. Scartare il surnatante e risospendere il pellet di cella in 1.000 μl di PBS.
- Centrifugare a 300 x g per 5 min. Scartare il surnatante e risospendere il pellet cellulare nella quantità appropriata di terreno di coltura delle cellule T per raggiungere una concentrazione di 1,5-2 x 106 cellule T in circa 3-4 mL di terreno di coltura delle cellule T.
- Semina di cellule T in una piastra a 6 pozzetti
- Eliminare la soluzione di rivestimento dalla piastra a 6 pozzetti.
- Seminare 1,5-2 milioni di cellule T isolate e colorate in terreno di cellule T da 3-4 mL per pozzetto.
- Incubare le cellule T a 37 °C e 5% di CO2 per le successive 48 ore
NOTA: Regolare la densità di semina sempre a 1,5 milioni di cellule / 3 mL. Evita di seminare le cellule T a una densità inferiore. Una densità più elevata è possibile, ma deve essere testata.
4. Insorgenza della co-cultura (Giorno 4)
NOTA: Temporizzazione: 2 h. Dopo aver determinato la conta cellulare delle cellule tumorali HSC4, le cellule T vengono aggiunte alle piastre a 96 pozzetti in un rapporto 1:1.
- Raccolta di un pozzetto per condizione per determinare una conta cellulare rappresentativa
- Scartare il surnatante del pozzetto che è stato seminato in modo esemplare per la determinazione della conta cellulare.
- Innanzitutto, lavare le celle con 100 μL di PBS, quindi scartare il PBS.
- Aggiungere 100 μL di tripsina e incubare per 5 minuti su una piastra riscaldante. Quindi risospendere e controllare la sospensione di una singola cellula al microscopio; se le cellule non si staccano, aggiungere altri 50 μL di tripsina.
- Non appena tutte le cellule si sono staccate, interrompere la reazione della tripsina aggiungendo 100 μL (o 150 μL se sono stati aggiunti altri 50 μL di tripsina in precedenza) del terreno D10 e risospendere.
- Trasferire l'intero volume di 200 μl dal pozzetto in una provetta da 1,5 mL e utilizzare 100 μl per misurare la conta cellulare. Ricordarsi di regolare il numero di celle misurato in base al volume del campione.
- Colorazione delle cellule tumorali HSC4 con un colorante fluorescente non tossico per il tracciamento delle cellule
- Preparare il tracciatore di cellule (vedere la tabella dei materiali) sciogliendo 2 μl in 20 mL di PBS (concentrazione finale 0,1 μM secondo il produttore).
- Pipettare il terreno D10 da tutti i pozzetti contenenti cellule tumorali e scartarlo.
- Aggiungere 200 μl della soluzione di tracciatore cellulare ai pozzetti delle cellule tumorali. Incubare per 20 minuti a 37 °C.
- Dopo l'incubazione, scartare la soluzione colorante e lavare aggiungendo 100 μL di PBS. Successivamente, scartare anche il PBS.
- Aggiungere 200 μl di terreno D10 fresco.
- Raccolta di cellule T (piastra a 6 pozzetti)
- Risospendere con cura le cellule T nel loro mezzo; la maggior parte delle cellule T sono in sospensione e possono essere raccolte semplicemente pipettando fuori dal loro pozzetto.
- Controllare al microscopio se i pozzetti sono vuoti; in caso contrario, raccogliere utilizzando 1.000 μL di tripsina, posizionare la piastra su una piastra riscaldante a 37 °C fino a quando le cellule T non si sono staccate, quindi interrompere la reazione aggiungendo 1.000 μL di PBS o terreno di coltura delle cellule T (opzionale).
- Contare le cellule T utilizzando una camera di conteggio Neubauer. Quindi centrifugare le cellule T a 300 x g per 5 minuti.
- Risospendere le cellule T nel terreno di coltura delle cellule T (utilizzare lo stesso terreno preparato per l'isolamento delle cellule T del giorno 2) fino a una concentrazione finale di 10.000 cellule T per 20 μl.
- Aggiunta di cellule T alle cellule tumorali HSC4 (piastra a 96 pozzetti)
- Aggiungere la quantità desiderata di cellule T ai pozzetti delle cellule tumorali in base alla conta delle cellule tumorali precedentemente determinata in un rapporto di 1:1.
- Mantenere un pozzetto di cellule tumorali per condizione libero da cellule T come controllo "solo cellule tumorali". Questo controllo "solo cellula tumorale" consente il gating preciso dei dati di flusso.
- Aggiungere 200 μl della sospensione di cellule T a un pozzetto vuoto come controllo "solo cellule T" (200.000 cellule T). Questo controllo "solo cellula T" consente il gating preciso dei dati di flusso.
- Incubazione della co-coltura per 96 ore (37 °C, 5% CO2)
5. Quantificazione della proliferazione delle cellule T mediante citometria a flusso (Giorno 7)
NOTA: Cronometraggio: 3h. Dopo aver incubato la co-coltura per altre 96 ore, i pozzetti vengono raccolti e colorati con una miscela di anticorpi contenente anticorpi diversi a seconda dell'ipotesi di ricerca (ad esempio, anticorpi anti-CD3, CD8, HLA-DR e CD25). Quindi, le cellule vengono analizzate mediante citometria a flusso multicolore.
- Preparare e iscrivere un lotto di provette FACS e provette per microcentrifuga per ogni pozzetto da raccogliere.
- Raccolta di cellule
- Risospendere le cellule nel loro terreno e trasferirle nelle provette FACS.
- Lavare le cellule utilizzando 100 μl di PBS, quindi trasferirlo nelle provette FACS.
- Aggiungere 100 μL di tripsina ai pozzetti e incubare sulla piastra riscaldante (37 °C) per 5 minuti.
- Risospendere le cellule, quindi controllare al microscopio se tutte le cellule sono staccate. In tal caso, trasferire la sospensione cellulare tripsinata nelle provette FACS.
- Controllare al microscopio se tutti i pozzetti sono vuoti; In caso contrario, ripetere i passaggi 5.2.3-5.2.4.
- Raccolta del surnatante della co-coltura per ulteriori esperimenti
- Centrifugare le provette riempite con le cellule a 300 x g per 5 min.
- Pipettare con cautela circa 300 μl di surnatante dalle provette e inserirli in un altro lotto di provette per microcentrifuga da congelare a -20 °C. Il surnatante può essere successivamente utilizzato per eseguire, ad esempio, saggi ELISA al fine di quantificare le citochine secrete.
- Colorazione delle cellule con anticorpi per eseguire la citometria a flusso
- Preparare la miscela di anticorpi: aggiungere 5 μL di anti-CD3-Krome Orange, 0,5 μL di anti-CD8-PerCE-Cyp5.5, 1 μL di anti-HLA-DR-APCVio770, 2,5 μL di anti-CD25-PE-Dazzle a 91 μL di tampone PBS/FACS.
- Aggiungere 200 μl di tampone FACS (PBS integrato con FBS al 2% e EDTA al 2%) in ciascuna provetta e risospendere.
- Centrifugare nuovamente a 300 x g per 5 min. Scartare il surnatante e risospendere le cellule in 100 μl della miscela di anticorpi precedentemente preparata (fase 5.4.1). Incubare per 30-45 minuti a 4 °C in frigorifero al riparo dalla luce.
- Dopo l'incubazione, centrifugare a 300 x g per 5 min. Scartare il surnatante e risospendere le cellule in 100 μL di tampone FACS.
- Eseguire la citometria a flusso su un citometro in grado di discriminare tutti i suddetti anticorpi fluorescenti e il segnale CSFE.
6. Strategia di gating e analisi dei dati
- Escludendo i doppietti e identificando la corretta popolazione di cellule T (Figura 2A).
- Escludere la popolazione di doppietti in base a forward vs. area di dispersione laterale (FSC-A vs. SSC-A) (canottiere).
- Grafico "Singoletti" per l'identificazione delle cellule T in base alla funzione diretta vs. area di dispersione laterale (FSC-A vs. SSC-A) (cellule T).
NOTA: Per l'esclusione delle dimensioni ottimali, misurare in parallelo i campioni "Solo cellule T" e "Solo cellule tumorali" (Figura 2B). - Tracciare l'espressione di CD3 contro l'SSC-A per discriminare le cellule T. Inoltre, le cellule T CD8-positive possono essere identificate tracciando il segnale CD8 contro l'SSC-A. Tutte le cellule T sono CD3+/CD8+ (cellule T CD8+) (Figura 2A).
- Traccia il segnale CFSE di tutte le "cellule T CD8+" come istogramma.
NOTA: Gating della sottopopolazione non proliferativa (segnale CFSE più alto), della sottopopolazione proliferativa (tutte le cellule T con segnale CFSE ridotto) e della sottopopolazione altamente proliferativa (tutte le cellule T del 4° picco CFSE o meno intensivo).
- Analisi dei marcatori di superficie associati all'attività sulla popolazione di cellule T
- Seguire i passaggi di iniezione (passaggio 6.1) (Figura 3A).
- Opzione A: Scegliere le "cellule T CD8+" come input e analizzarle per la loro espressione CD25 o HLA-DR tracciando la rispettiva fluorescenza del marcatore di superficie rispetto alle caratteristiche SSC-A.
NOTA: Se è rilevabile una sottopopolazione distinta con CD25-alto, si raccomanda il gating per la popolazione CD25-alta. Lo stesso vale per il gating/analisi di HLA-DR su cellule T (Figura 3B). - Opzione B: Scegliere le "cellule T CD8+" come input e analizzarle per la loro espressione CD25 o HLA-DR tracciando la rispettiva fluorescenza del marcatore di superficie rispetto alle caratteristiche SSC-A.
NOTA: Se non è rilevabile alcuna sottopopolazione distinta (a seconda del marcatore di superficie analizzato), si raccomanda l'analisi dello spostamento dell'intensità media di fluorescenza (MFI) dell'intera popolazione di cellule T (Figura 3C).
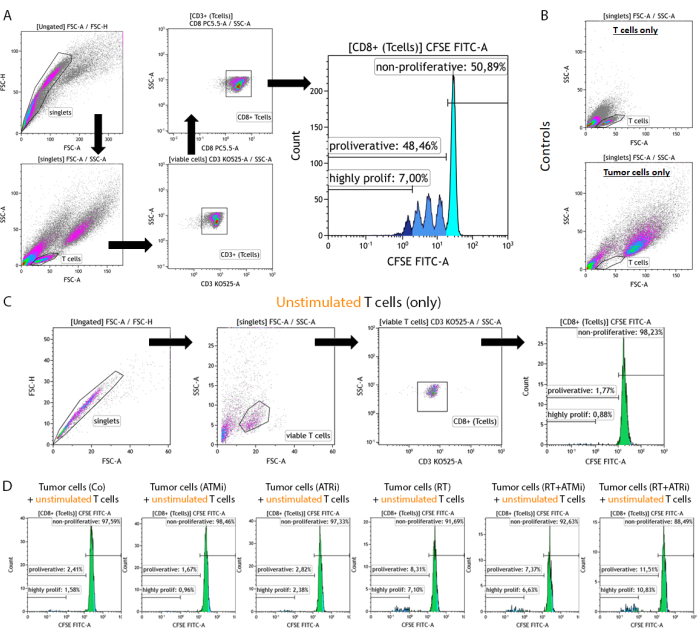
Figura 2: Strategia di gating di cellule T pre-stimolate e colorate con CFSE per l'analisi della proliferazione dopo 96 ore di co-coltura con cellule tumorali HNSCC pretrattate. Le cellule T e le cellule tumorali vengono raccolte 96 ore dopo la co-coltura da una piastra a 96 pozzetti, colorate utilizzando un marcatore di superficie specifico per le cellule T e misurate utilizzando un dispositivo di citometria a flusso. (A) I doppietti sono stati esclusi in base a FSC-A/FSC-H e le cellule T sono state controllate per prime in base alle dimensioni (FSC-A/SSC-A). Le cellule T sono state ulteriormente analizzate per la loro espressione di CD3 (anti-CD3 Krome Orange) e CD8 (anti-CD8 PerCP-Cy5.5). Le cellule T CD3+/CD8+ sono state tracciate in un istogramma ed è stata analizzata l'intensità del segnale CFSE. Il segnale CFSE rappresenta sottogruppi di cellule T con diverso comportamento di proliferazione. Le cellule T con il più alto segnale CFSE sono state definite "non proliferative". Tutte le sottopopolazioni che mostravano una perdita del segnale CFSE, causata dalla divisione cellulare che porta alla metà del segnale, in picchi distinti sono state riassunte come "proliferative". Le cellule T che si sono divise più di tre volte (più di tre picchi di segnale CSFE) sono state definite "altamente proliferative". (B) Come controllo, vengono misurati in aggiunta campioni costituiti solo da cellule T prestimolate e solo cellule tumorali pretrattate. Le cellule T e le cellule tumorali HSC4 possono essere discriminate in base alle dimensioni utilizzando FSC-A vs. Segnale SCC-A. Inoltre, le cellule T CD8+ non stimolate sono state analizzate per verificare l'attivazione delle cellule T sulla base della co-coltivazione con le sole cellule tumorali allogeniche. (C) Come controllo, è stata inclusa l'incubazione di cellule T colorate con CFSE non stimolate per 96 ore in parallelo alla co-coltura. (D) Le cellule T non stimolate e colorate con CFSE sono state co-coltivate con cellule tumorali pretrattate. Dopo 96 ore, le cellule sono state raccolte e il segnale CFSE è stato misurato utilizzando la procedura standard. Le cellule T non stimolate non hanno mostrato alcuna proliferazione né da sole né dopo co-coltivazione con cellule tumorali pretrattate. Clicca qui per visualizzare una versione più grande di questa figura.
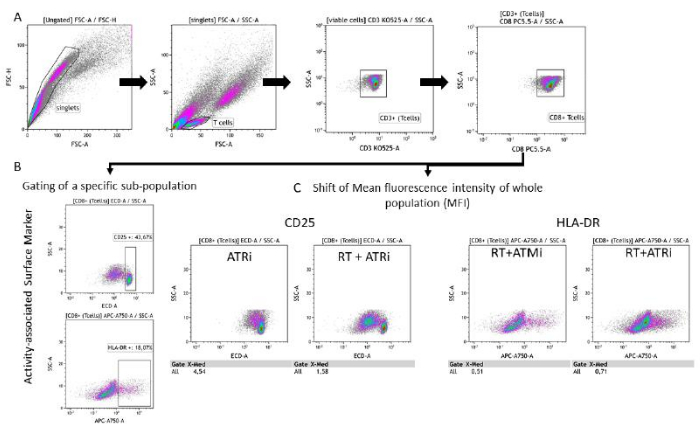
Figura 3: Strategia di gating dell'espressione del marcatore di superficie (CD25 e HLA-DR) associata all'attività delle cellule T dopo 96 ore di co-coltivazione con cellule HNSCC pretrattate. (A) I doppietti sono stati esclusi in base alle loro caratteristiche FSC-A/FSC-H e le cellule T sono state controllate per prime in base alle dimensioni (FSC-A/SSC-A). Le cellule T sono state ulteriormente analizzate per la loro espressione di CD3 (anti-CD3 Krome Orange) e CD8 (anti-CD8 PerCP-Cy5.5). Il gating dei marcatori di attività CD25 (anti-CD25 PEDazzle594) e HLA-DR (anti-HLA-DR APC-Vio770) sulla superficie delle cellule T può essere eseguito in due diversi contesti. (B) Gating di tutte le cellule T CD8+ per la percentuale di una specifica sottopopolazioneCD25 alta o HLA-DRalta in base ai grafici di densità. (C) In alternativa, viene eseguita la misurazione di uno spostamento MFI dell'intera popolazione di cellule T CD8+ per entrambi i marcatori di attività. Vengono mostrate immagini rappresentative dell'analisi dell'espressione dei marcatori di superficie delle cellule T dopo la co-coltivazione con cellule tumorali HSC4. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Le cellule tumorali HSC4 derivate da un HNSCC sono state seminate e incubate durante la notte. Dopo 24 ore, le cellule sono state trattate con un inibitore della chinasi. Dopo 3 ore, sono state applicate le prime due dosi di 5 Gy per frazione. Dopo 24 ore, è stata applicata la seconda dose e le cellule sono state nuovamente incubate durante la notte. Parallelamente, 48 ore prima dell'inizio della co-coltura, le cellule T sono state isolate dal sangue di un donatore sano. In primo luogo, le PBMC sono state isolate utilizzando provette per centrifugazione a gradiente di densità e un mezzo di separazione sterile. Le PBMC sono state contate utilizzando una camera di conteggio delle cellule e le cellule T CD8+ sono state isolate utilizzando il kit di isolamento delle cellule T CD8+. Le cellule T CD8+ isolate sono state quindi colorate con CFSE (1 μM) e successivamente contate. Le cellule T sono state seminate in piastre a pozzetti pre-rivestite CD3/CD28 con una densità di 1,5 x 106 cellule/3 mL. Dopo 48 ore di stimolazione, le cellule T sono state raccolte, contate e risospese in una densità di 10.000 cellule/10 μL. Inoltre, sono stati raccolti e contati pozzetti rappresentativi delle cellule tumorali seminate e il terreno è stato sostituito con tutti i pozzetti rimanenti. Le cellule T sono state aggiunte alle cellule tumorali in un rapporto 1:1. La co-coltura è stata raccolta dopo 96 ore, il surnatante è stato conservato a -20 °C e le cellule sono state colorate e misurate mediante citometria a flusso (Figura 1).
Le cellule T sono state identificate in base all'esclusione dimensionale e alla positività CD3/CD8. Il segnale CFSE delle cellule CD3+/CD8+ positive ha mostrato la distribuzione delle cellule T non proliferative (alto segnale CFSE) e sottopopolazioni distinte di cellule T proliferanti (perdita dell'intensità del segnale CFSE). La proporzione di cellule T proliferanti è stata misurata in modo esemplare per la linea cellulare HSC4 HNSCC (Figura 4A). Tutte le cellule T che mostravano almeno una divisione sono state definite "proliferative". Le cellule T che mostravano più di 3 divisioni sono state definite "altamente proliferative" (Figura 4B). Per la linea cellulare HPV-negativa HSC4, è stato rilevato un leggero aumento del tasso di proliferazione quando le cellule T sono state co-coltivate con cellule tumorali irradiate. L'inibizione di RT e RT plus di ATR ha determinato un aumento significativo della proliferazione consecutiva delle cellule T rispetto alle co-colture di cellule HSC4 trattate con RT più inibizione ATM con cellule T. Per quanto riguarda la frazione "altamente proliferativa" delle cellule T, il pretrattamento delle cellule HSC4 con RT e inibizione di ATR è stato più efficace nello stimolare la proliferazione delle cellule T (Figura 4B).
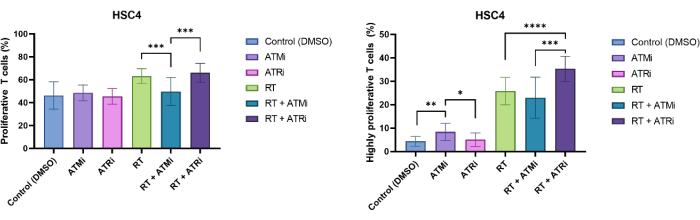
Figura 4: Proliferazione di cellule T colorate con CFSE per l'analisi delle variazioni dipendenti dal trattamento delle HSC4 nella proliferazione delle cellule T. (A) Proporzione di cellule T proliferanti dopo 96 ore di co-coltura con cellule tumorali HSC4 HPV-negative pretrattate. La proliferazione delle cellule T è risultata significativamente più bassa dopo la co-coltura con cellule tumorali HSC4 trattate con RT+ATMi rispetto al trattamento con RT o RT più ATRi. (B) La RT delle cellule tumorali HSC4 ha indotto una frazione più elevata di cellule T altamente proliferanti (più di tre divisioni cellulari. Dopo la co-coltura di cellule tumorali HSC4 pretrattate RT+ATRi con cellule T, la percentuale di cellule T altamente proliferative era la più alta. Le barre mostrano i dati di quattro esperimenti indipendenti con le cellule T di quattro donatori indipendenti e sani (n = 4; Mean ± SD). (*p≤ 0,05, **p≤ 0,01, ***p≤ 0,001, ****p≤ 0,0001; significatività statistica testata confrontando tutte le condizioni sperimentali con ciascuna di esse utilizzando Mann-Whitney-U a due code per dati non distribuiti normalmente). Clicca qui per visualizzare una versione più grande di questa figura.
La perdita di CFSE basata sulla proliferazione può essere utilizzata per quantificare il tasso di proliferazione delle cellule T. Inoltre, diversi marcatori della superficie cellulare sono descritti come associati all'attività delle cellule T, come CD25 e HLADR. Pertanto, è stata eseguita l'analisi di CD25 e HLA-DR sulla superficie di tutte le cellule T CD3+ e CD8+ mediante citometria a flusso (Figura 5). L'espressione può essere quantificata se sono rilevabili sottopopolazioni distintedi CD25 oHLA-DR alte e può essere discriminata mediante gating di queste cellule altamente positive (Figura 3B). Se non sono rilevabili sottopopolazioni distinte, è possibile misurare l'intensità media di fluorescenza (MFI) dell'intera popolazione e quantificare gli spostamenti dell'MFI basati sulle condizioni (Figura 3C).
A titolo esemplificativo, l'espressione identificata dall'MFI di CD25 e HLA-DR è stata analizzata dopo 96 ore di co-coltura con cellule tumorali HSC4 pretrattate (Figura 5). Dopo la co-coltura di cellule T con cellule tumorali HSC4 trattate con RT, l'espressione di CD25 sulle cellule T è risultata fortemente sottoregolata. In particolare, il pretrattamento delle cellule tumorali con solo ATMi ha determinato una significativa riduzione dell'espressione di CD25 rispetto al pretrattamento con ATRi. Nel contesto combinato con RT, anche se RT ha determinato una diminuzione dell'espressione di CD25 sulle cellule T, la combinazione di RT con ATMi ha determinato un aumento dell'espressione di CD25 rispetto a RT più ATRi (Figura 5A). Per quanto riguarda l'espressione di HLA-DR sulle cellule T, la RT ha generalmente portato a una sovraregolazione di HLA-DR, ma ancora una volta, in combinazione con ATMi o ATRi, è stato osservato un comportamento diverso (Figura 5B). Le cellule T co-coltivate con cellule HSC4 che sono state pre-trattate con una combinazione di RT+ATRi hanno aumentato l'espressione di HLA-DR rispetto a RT da solo o RT+ATMi.
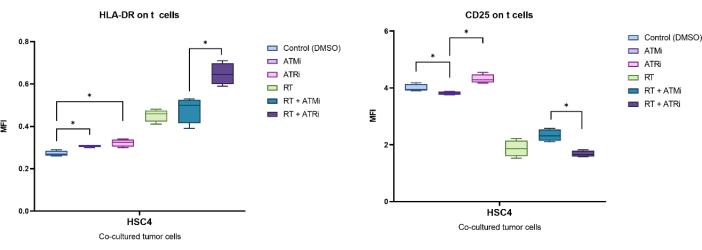
Figura 5: Espressione dei marcatori di attivazione CD25 e HLA-DR sulla superficie delle cellule T dopo 96 ore di co-coltura con cellule tumorali HSC4 pretrattate. L'espressione di CD25 e HLA-DR è stata analizzata in base allo spostamento dell'MFI dell'intera popolazione di cellule T. La RT di HSC4 ha determinato una diminuzione dell'espressione di CD25 sulle cellule T e un aumento dell'espressione di HLA-DR. La combinazione di RT con ATMi ha portato a modelli di espressione significativamente diversi rispetto a RT più ATRi. I boxplot mostrano i dati di quattro esperimenti indipendenti con le cellule T di quattro donatori sani indipendenti (n = 4; Mean ± SD). I dati sono stati analizzati confrontando tutte le condizioni sperimentali tra loro utilizzando un test di Mann-Whitney-U a due code per dati non distribuiti normalmente (*p≤ 0,050). Clicca qui per visualizzare una versione più grande di questa figura.
Questi dati indicano in modo esemplare che il trattamento delle cellule tumorali, in questo caso delle cellule HSC4 HNSCC, influisce sull'immunogenicità delle cellule tumorali. Questo può essere monitorato mediante la co-coltivazione delle cellule tumorali con le cellule T pre-stimolate, il che porta a un comportamento di proliferazione diversificato e all'espressione di marcatori di attivazione sulle cellule T umane in base al regime di trattamento delle cellule tumorali applicato.
Discussione
Il protocollo qui presentato offre un metodo semplice e veloce per analizzare la capacità proliferativa delle cellule T insieme al loro stato di attivazione in un contesto di co-coltura con cellule tumorali pretrattate. In tal modo, gli effetti di diverse modalità di trattamento, come RT, CT o TT, sull'attività e sulla proliferazione delle cellule T possono essere facilmente analizzati, gettando le basi per successive analisi immunologiche più approfondite di approcci promettenti. I risultati rappresentativi mostrati in questo manoscritto dimostrano che questo test allogenico di co-coltura di cellule T sta funzionando bene. Differenze significative nella proliferazione delle cellule T, così come nell'attività delle cellule T, sono state osservate per la co-coltura con cellule HSC4 umane (HNSCC) pretrattate in modo differenziato (Figura 4 e Figura 5). È stato riscontrato che il trattamento delle HSC4 con RT da solo o in combinazione con TT ha determinato un aumento della proliferazione delle cellule T, in particolare per quanto riguarda la frazione altamente proliferativa delle cellule T (Figura 4). In linea con la quantificazione del tasso di proliferazione, anche lo stato di attivazione delle cellule T è stato influenzato in modo differenziale dai diversi regimi di trattamento. In sintesi, l'applicazione della RT ha portato ad una forte sottoregolazione dell'espressione di CD25 sulle cellule T, mentre l'espressione di HLA-DR è stata sovraregolata. La RT induce danni al DNA nelle cellule tumorali irradiate, che a loro volta portano a risposte cellulari allo stress che includono il rilascio di molecole e citochine associate allo stress e al danno, nonché l'espressione di ligandi di superficie cellulare immunomodulanti19.
La combinazione di RT con TT sotto forma di inibitori della riparazione del DNA sta ulteriormente migliorando e mantenendo tali effetti, poiché le cellule tumorali non possono riparare efficacemente il danno al DNA indotto dalla RT. Ciò potrebbe promuovere e mantenere ulteriormente la secrezione di fattori immunogenici e l'espressione di ligandi di superficie cellulare immunogenici20. In accordo con ciò, abbiamo precedentemente dimostrato che il trattamento delle cellule tumorali (HNSCC) con RT e inibitori del danno al DNA altera il fenotipo immunitario sulla superficie delle cellule tumorali. La modulazione del fenotipo immunitario includeva la regolazione della molecola immunostimolante ICOS-L e della molecola immunosoppressiva PD-L111. In risposta a questa modulazione del fenotipo immunitario delle cellule tumorali, le cellule T potrebbero regolare l'espressione dei marcatori di attivazione anche sulla loro superficie cellulare. Inoltre, la RT è nota per stimolare il rilascio di citochine pro-infiammatorie, come l'IFNγ o l'IL-6, che a loro volta influiscono sulla proliferazione e sull'attività delle cellule T21. L'espressione di marcatori di attivazione sulle cellule T è un processo altamente dinamico. I dati di Zimmerman et al.22 hanno mostrato che il CD25 è altamente espresso 24 ore dopo la stimolazione, ma è nuovamente sottoregolato dopo 96 ore dopo la stimolazione. Ciò è in linea con i nostri risultati. Al contrario, l'HLA-DR è un marcatore di attivazione in fase avanzata ed è generalmente preceduto da un aumento di CD25 e CD6923. Da notare che la combinazione di RT + ATRi porta a una significativa sovraregolazione di HLA-DR sulle cellule T rispetto a RT + ATMi. Questa scoperta è in linea con il lavoro di Dillion et al. che hanno dimostrato gli effetti sinergici di ATRi + RT nell'indurre un TME24 infiammatorio. Una modulazione meno pronunciata dell'attività e della proliferazione delle cellule T è stata ottenuta dal trattamento delle cellule tumorali con i soli inibitori delle chinasi (Figura 5). Ciò potrebbe essere dovuto al fatto che gli inibitori della riparazione del DNA ATMi e ATRi funzionano come potenziatori del danno al DNA indotto dalla RT e di conseguenza non sono molto immunogenici quando applicati in monoterapia. Abbiamo già dimostrato la tossicità minima di ATMi o ATRi da soli11. In sintesi, i risultati confermano il potenziale immunogenico della RT (e della TT con inibitori del danno al DNA) e indicano inoltre che questo sistema sperimentale è adatto per lo screening delle capacità immunostimolatorie di diverse modalità di trattamento nelle cellule tumorali umane della testa e del collo.
Anche se il metodo qui presentato è semplice e robusto, ci sono alcuni passaggi critici nel protocollo che devono essere considerati in anticipo. È necessaria la presenza di alte concentrazioni di EDTA durante il processo di isolamento, nonché durante l'analisi citofluorimetrica delle cellule T. Pertanto, si consiglia di raccogliere il sangue del donatore in provette di raccolta rivestite di EDTA e di utilizzare un tampone MACS integrato con 10 mM di EDTA per garantire un isolamento efficiente delle cellule T e la generazione di una sospensione omogenea di singole cellule per la valutazione citofluorimetrica. Inoltre, la composizione del mezzo delle cellule T è di grande importanza per la capacità proliferativa delle cellule T. Dopo aver testato diverse composizioni di terreni di coltura, un terreno con basse concentrazioni di L-Arginina e L-Lisina produce il miglior segnale di proliferazione delle cellule T, poiché la L-Arginina è fondamentale per il metabolismo e la sopravvivenza delle cellule T e dovrebbe essere fornita in una concentrazione adeguata25,26. Tuttavia, si dovrebbe tenere presente che le soluzioni di L-Arginina e L-lisina non sono stabili a lungo termine e quindi devono essere utilizzate entro 3 mesi. Per una stimolazione ottimale, il terreno delle cellule T integrato con gli aminoacidi deve essere preparato fresco per ogni esperimento di coltivazione delle cellule T. Inoltre, il numero di cellule delle cellule T, così come quello delle cellule tumorali, sono fattori critici che devono essere considerati in quanto influiscono sulla lettura dell'esperimento.
Per le cellule tumorali, da un lato, si desidera una confluenza cellulare compresa tra il 70% e l'80% alla fine dell'esperimento di co-coltura. Una crescita eccessiva delle cellule tumorali potrebbe provocare la secrezione di fattori che inibiscono la crescita cellulare e successivamente influenzerebbe anche il tasso di proliferazione delle cellule T. Poiché la crescita delle cellule tumorali dipende fortemente dalla singola linea cellulare, si consiglia di testare accuratamente il comportamento di crescita della rispettiva linea cellulare tumorale in diverse piastre a pozzetti e per i diversi regimi di trattamento. Inoltre, le cellule tumorali e le cellule T dovrebbero essere co-coltivate in un rapporto di 1:1. Pertanto, è obbligatorio seminare un pozzetto extra di cellule tumorali per ogni condizione di trattamento, che può essere utilizzato per la determinazione della conta cellulare il giorno dell'inizio della co-coltura. In questo modo, si può garantire che venga seminato un numero adeguato di cellule T per le diverse condizioni di trattamento. Per quanto riguarda il numero di cellule delle cellule T, è necessario considerare che la quantità di cellule T che possono essere isolate da un donatore sano è molto individuale. Le cellule T rappresentano circa il 45-70% delle PBMC di un donatore sano27. Pertanto, la quantità richiesta di cellule T dovrebbe essere già ampiamente stimata al momento del prelievo di sangue. Inoltre, la densità delle cellule T è importante per l'efficienza dell'attivazione delle cellule T e la loro sopravvivenza. Di conseguenza, le cellule T devono essere seminate in una concentrazione di almeno 1,5 milioni di cellule per pozzetto di una piastra a 6 pozzetti in 3 mL di terreno di coltura T per la stimolazione iniziale prima dell'inizio della co-coltura. È possibile una densità di cellule T più elevata, ma una densità inferiore dovrebbe essere evitata. L'attivazione delle cellule T può anche essere valutata morfologicamente al microscopio, poiché le cellule T attivate tendono a formare cluster cellulari.
Poiché questo metodo è un sistema sperimentale semplificato per lo studio delle capacità immunostimolatorie di diverse linee cellulari tumorali e modalità di trattamento, presenta alcune limitazioni che devono essere prese in considerazione. Innanzitutto, questo test si basa su un sistema allogenico, il che significa che le cellule T del donatore non sono HLA-compatibili con la rispettiva linea cellulare tumorale. Pertanto, questo disallineamento HLA potrebbe già indurre la stimolazione delle cellule T e, successivamente, la proliferazione delle cellule T senza ulteriore stimolazione dal trattamento con cellule tumorali28. Per quantificare questo effetto indesiderato, le cellule T isolate sono state co-coltivate con la linea cellulare HSC4 senza previa stimolazione con anticorpi CD28 e CD3. È stato riscontrato che quasi nessuna proliferazione è stata indotta in questo contesto, il che indica che il mismatch HLA ha solo effetti minori sull'attivazione delle cellule T in questo specifico contesto sperimentale. Inoltre, sono state riscontrate differenze significative nella proliferazione e nell'attivazione delle cellule T in base al trattamento delle cellule tumorali nonostante l'impostazione allogenica. Tuttavia, per la messa a punto di questo test, la potenziale proliferazione allogenica indotta delle cellule T dovrebbe essere testata e quantificata una volta all'inizio degli esperimenti. I controlli raccomandati "solo cellule T" e "solo cellule tumorali" sono necessari per un gating sufficiente e sono obbligatori in ogni replicazione degli esperimenti. Tuttavia, è necessario tenere presente che un sistema di co-coltura allogenica manca del riconoscimento dell'antigene tumore-specifico. Pertanto, questo sistema potrebbe non riflettere accuratamente la risposta antitumorale specifica come si verificherebbe in vivo nei pazienti29,30. Per un ulteriore miglioramento, un ambiente più sofisticato sarebbe un approccio di co-cultura autologa. In questo contesto, le biopsie tumorali derivate dal paziente devono essere coltivate e portate in coltura con cellule T isolate dal sangue periferico dello stesso paziente28. Questo approccio sperimentale, tuttavia, potrebbe essere impegnativo non solo in termini di coltura di cellule tumorali primarie, ma anche in termini di disponibilità di biomateriale derivato dal paziente. Un'ulteriore limitazione che deve essere considerata è il rapporto tra le cellule tumorali e le cellule T nella co-coltura. Poiché il rapporto raccomandato tra le cellule è di 1:1, il rapporto non riflette la situazione fisiologica del TME nei pazienti31. Tuttavia, questa limitazione deve essere accettata, poiché con un numero inferiore di cellule T, le variazioni del tasso di proliferazione e dello stato di attivazione non sono quantificabili.
Nel campo dell'immunologia dei tumori, questo test offre l'opportunità di eseguire lo screening dell'immunogenicità di diverse modalità di trattamento in un contesto sperimentale in vitro facile e veloce. Pertanto, non solo è possibile risparmiare tempo pre-selezionando gli approcci più promettenti in termini di immunogenicità, ma anche sperimentando in vivo . I modelli animali possono essere ridotti, poiché solo schemi di trattamento promettenti potrebbero essere ulteriormente perseguiti nei modelli animali per riflettere il sistema immunitario nel suo insieme in un organismo. Inoltre, poiché questo test si basa su cellule primarie umane e linee cellulari tumorali umane, i risultati potrebbero essere più traducibili in clinica rispetto ai test basati su altri sistemi e specie modello.
In futuro, questo test potrebbe essere modulato e adattato per rispondere a domande di ricerca più specifiche. Ad esempio, si potrebbe includere un trattamento delle cellule T per riflettere uno scenario di trattamento più vicino alla situazione dei pazienti. Poiché le cellule T reattive al tumore si trovano principalmente nel TME, sono ugualmente influenzate da terapie locali come la RT e probabilmente anche da terapie sistemiche come la TC o la TT32. Un ulteriore miglioramento del test è la co-coltivazione di cellule T con sferoidi tumorali da linee cellulari tumorali o anche la co-coltivazione con organoidi tumorali derivati da pazienti. Queste colture tridimensionali sono più paragonabili alla struttura di un tumore in un paziente28. Infine, anche la lettura dell'esperimento tramite citometria a flusso può essere facilmente adattata al fine di studiare ulteriori molecole sulla superficie delle cellule T o analizzando l'espressione di molecole di checkpoint immunitario sulle cellule tumorali. Oltre alla determinazione del fenotipo immunitario delle cellule T, si potrebbero utilizzare i surnatanti di coltura cellulare provenienti dagli esperimenti di co-coltura per la quantificazione di citochine o chemochine secrete per ottenere maggiori informazioni sull'attività delle cellule T. In sintesi, questo protocollo fornisce un test di co-coltura completo, robusto e facile per le cellule T e le cellule tumorali che consente lo screening dell'immunogenicità di diverse modalità di trattamento del cancro. Poiché questo test è adattabile a specifiche domande di ricerca, è adatto per l'applicazione nell'ampio campo dell'immunologia dei tumori.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questa ricerca è stata in parte finanziata dal Centro interdisciplinare per la ricerca clinica di Erlangen (IZKF Erlangen) e dal Bayerisches Zentrum für Krebsforschung (BZKF).
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL Cellstar tubes | Greiner Bio-One GmbH | 188271 | |
| 50 mL Cellstar tubes | Greiner Bio-One GmbH | 227261 | |
| 6 well cell culture plate sterile, with lid | Greiner Bio-One GmbH | 657160 | |
| 96 well cell culture plate sterile, F-bottom, with lid | Greiner Bio-One GmbH | 655180 | |
| AZD0156 | Selleck Chemicals GmbH | S8375 | |
| Berzosertib (VE-822) | Selleck Chemicals GmbH | S7102 | |
| CASYcups | OMNI Life Science GmbH & Co KG | 5651794 | |
| CASYton | OMNI Life Science GmbH & Co KG | 5651808 | |
| CD25a, PE-Dazzle594, Mouse IgG1 | Biolegend | 356126 | |
| CD28-UNLB | Beckmann Coulter, Inc. | IM1376 | |
| CD3a,Krome Orange, Mouse IgG1 | Beckmann Coulter, Inc. | B00068 | |
| CD3e Monoclonal Antibody | Thermo-Fisher Scientific, Inc. | MA1-10176 | |
| CD4, APC, Mouse Anti-Human Mouse IgG1 | BD Pharmingen | 555349 | |
| CD8 MicroBeads, human | Miltenyi Biotec, Inc. | 130-045-201 | |
| CD8a, PerCP-Cy5.5, Mouse IgG1 | Biolegend | 300924 | |
| CellTracker Deep Red Dye | Thermo Fisher Scientific, Inc. | C34565 | |
| CFSE | Merck KGaA (Sigma-Aldrich) | 21888 | |
| DMEM (Dulbecco´s Modified Eagle´s Medium) | PAN-Biotech GmbH | P04-02500 | |
| DxFlex Flow Cytometer (with Auto Loader) | Beckmann Coulter, Inc. | C44326, C02846 | |
| EDTA disodium salt dihydrate | Carl Roth GmbH + Co. KG | 8043.2 | |
| FBS superior | Merck KGaA (Sigma-Aldrich) | S0615-500ML | |
| FBS superior | Merck KGaA (Sigma-Aldrich) | S0615-100ML | For production of heat-inactivated FBS. Heat up for 30 min at 56 °C with mixing to inactivate complement proteins. |
| Graph Pad Prism (version number 9) | GraphPad Software | - | |
| HLA-DR, DP, DQ Antibody, anti-human, APC-Vio770 | Miltenyi Biotec, Inc. | 130-123-550 | |
| Kaluza (version number 2.1) | Beckmann Coulter, Inc. | - | |
| L-Arginin | Merck KGaA (Sigma-Aldrich) | A8094-25G | |
| L-Lysin-monohydrochloride | Merck KGaA (Sigma-Aldrich) | L5626-100G | |
| MACS BSA Stock Solution | Miltenyi Biotec, Inc. | 130-091-376 | |
| MS Columns | Miltenyi Biotec, Inc. | 130-042-201 | |
| Neubauer-improved counting chamber | Paul Marienfeld GmbH & Co. KG | 640010 | |
| PBS | Merck KGaA (Sigma-Aldrich) | D8537-500mL | |
| Penicillin-Streptomycin | Thermo-Fisher Scientific, Inc. | 1514-122 | |
| ROTISep 1077 | Carl Roth GmbH + Co. KG | 0642.2 | |
| RPMI-1640 Medium | Merck KGaA (Sigma-Aldrich) | R1790 | |
| SepMate 50mL tubes | Stemcell Technologies | 85450 | |
| Trypan blue | Merck KGaA (Sigma-Aldrich) | T6146-25G | |
| Trypsin | Thermo-Fisher Scientific, Inc. | 15400054 |
Riferimenti
- Hiam-Galvez, K. J., Allen, B. M., Spitzer, M. H. Systemic immunity in cancer. Nat Rev Cancer. 21 (6), 345-359 (2021).
- Mantovani, A., Allavena, P., Marchesi, F., Garlanda, C. Macrophages as tools and targets in cancer therapy. Nat Rev Drug Discov. 21 (11), 799-820 (2022).
- Waldman, A. D., Fritz, J. M., Lenardo, M. J. A guide to cancer immunotherapy: From t cell basic science to clinical practice. Nat Rev Immunol. 20 (11), 651-668 (2020).
- Dunn, G. P., Old, L. J., Schreiber, R. D. The immunobiology of cancer immunosurveillance and immunoediting. Immunity. 21 (2), 137-148 (2004).
- Mittal, D., Gubin, M. M., Schreiber, R. D., Smyth, M. J. New insights into cancer immunoediting and its three component phases--elimination, equilibrium and escape. Curr Opin Immunol. 27, 6-25 (2014).
- Irianto, T., Gaipl, U. S., Ruckert, M. Immune modulation during anti-cancer radio(immuno)therapy. Int Rev Cell Mol Biol. 382, 239-277 (2024).
- Burtness, B., et al. Pembrolizumab alone or with chemotherapy versus cetuximab with chemotherapy for recurrent or metastatic squamous cell carcinoma of the head and neck (keynote-048): A randomized, open-label, phase 3 study. Lancet. 394 (10212), 1915-1928 (2019).
- Hecht, M., et al. Safety and efficacy of single cycle induction treatment with cisplatin/docetaxel/ durvalumab/tremelimumab in locally advanced HNSCC: First results of checkered-CD8. J Immunother Cancer. 8 (2), e001378 (2020).
- Chen, J. A., Ma, W., Yuan, J., Li, T. Translational biomarkers and rationale strategies to overcome resistance to immune checkpoint inhibitors in solid tumors. Cancer Treat Res. 180, 251-279 (2020).
- Solomon, B., Young, R. J., Rischin, D. Head and neck squamous cell carcinoma: Genomics and emerging biomarkers for immunomodulatory cancer treatments. Semin Cancer Biol. 52 (Pt 2), 228-240 (2018).
- Meidenbauer, J., et al. Inhibition of atm or atr in combination with hypo-fractionated radiotherapy leads to a different immunophenotype on transcript and protein level in HNSCC. Front Oncol. 14, 1460150 (2024).
- Kumari, S., et al. Immunomodulatory effects of radiotherapy. Int J Mol Sci. 21 (21), 8151 (2020).
- Wimmer, S., et al. Hypofractionated radiotherapy upregulates several immune checkpoint molecules in head and neck squamous cell carcinoma cells independently of the HPV status while icos-l is upregulated only on HPV-positive cells. Int J Mol Sci. 22 (17), 9114 (2021).
- Derer, A., et al. Chemoradiation increases pd-l1 expression in certain melanoma and glioblastoma cells. Front Immunol. 7, 610 (2016).
- Schatz, J., et al. Normofractionated irradiation and not temozolomide modulates the immunogenic and oncogenic phenotype of human glioblastoma cell lines. Strahlenther Onkol. 199 (12), 1140-1151 (2023).
- Xu, M. M., Pu, Y., Zhang, Y., Fu, Y. X. The role of adaptive immunity in the efficacy of targeted cancer therapies. Trends Immunol. 37 (2), 141-153 (2016).
- Olivo Pimentel, V., Yaromina, A., Marcus, D., Dubois, L. J., Lambin, P. A novel co-culture assay to assess anti-tumor cd8(+) t cell cytotoxicity via luminescence and multicolor flow cytometry. J Immunol Methods. 487, 112899 (2020).
- Kulp, M., Diehl, L., Bonig, H., Marschalek, R. Co-culture of primary human t cells with leukemia cells to measure regulatory t cell expansion. STAR Protoc. 3 (3), 101661 (2022).
- Ruckert, M., et al. Immune modulatory effects of radiotherapy as basis for well-reasoned radioimmunotherapies. Strahlenther Onkol. 194 (6), 509-519 (2018).
- Samstein, R. M., Riaz, N. The DNA damage response in immunotherapy and radiation. Adv Radiat Oncol. 3 (4), 527-533 (2018).
- Meeren, A. V., Bertho, J. M., Vandamme, M., Gaugler, M. H. Ionizing radiation enhances il-6 and il-8 production by human endothelial cells. Mediators Inflamm. 6 (3), 185-193 (1997).
- Zimmerman, M., et al. Ifn-gamma upregulates survivin and ifi202 expression to induce survival and proliferation of tumor-specific T cells. PLoS One. 5 (11), e14076 (2010).
- Saraiva, D. P., et al. Expression of HLA-dr in cytotoxic t lymphocytes: A validated predictive biomarker and a potential therapeutic strategy in breast cancer. Cancers (Basel). 13 (15), (2021).
- Dillon, M. T., et al. Atr inhibition potentiates the radiation-induced inflammatory tumor microenvironment. Clin Cancer Res. 25 (11), 3392-3403 (2019).
- Geiger, R., et al. L-Arginine modulates T cell metabolism and enhances survival and anti-tumor activity. Cell. 167 (3), 829-842.e13 (2016).
- Rodriguez, P. C., Quiceno, D. G., Ochoa, A. C. L-Arginine availability regulates t-lymphocyte cell-cycle progression. Blood. 109 (4), 1568-1573 (2007).
- Lozano-Ojalvo, D., López-Fandiño, R., López-Expósito, I., Verhoeckx, K. . The impact of food bioactives on health: In vitro and ex vivo models. , 169-180 (2015).
- Gronholm, M., et al. Patient-derived organoids for precision cancer immunotherapy. Cancer Res. 81 (12), 3149-3155 (2021).
- Perez, C., Gruber, I., Arber, C. Off-the-shelf allogeneic t cell therapies for cancer: Opportunities and challenges using naturally occurring "universal" donor t cells. Front Immunol. 11, 583716 (2020).
- Martinez Bedoya, D., Dutoit, D., Migliorini, D. Allogeneic car t cells: An alternative to overcome challenges of car t cell therapy in glioblastoma. Front Immunol. 12, 640082 (2021).
- Schnellhardt, S., et al. The prognostic value of FOXP3+ tumor-infiltrating lymphocytes in rectal cancer depends on immune phenotypes defined by CD8+ cytotoxic T cell density. Front Immunol. 13, 781222 (2022).
- Wang, W., Green, M., Rebecca Liu, J., Lawrence, T. S., Zou, W., Zitvogel, L., Kroemer, G. . Oncoimmunology: A practical guide for cancer immunotherapy. , 23-39 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon