Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Linealización del ensayo de proteínas Bradford
En este artículo
Resumen
La precisión y la sensibilidad de la determinación de proteínas mediante el ensayo de Bradford rápida y conveniente se ve comprometida por la no linealidad intrínseca. Se muestra un procedimiento de linealización simple que aumenta en gran medida la precisión, mejora la sensibilidad de la prueba unas 10 veces, y reduce significativamente la interferencia de los detergentes.
Resumen
Determinación de las cantidades de microgramos de proteína en el ensayo de Bradford azul brillante de Coomassie se lleva a cabo mediante la medición de la absorbancia a 590 nm. Este ensayo más común permite la cuantificación de proteínas rápida y sencilla en lisados de células, las fracciones celulares, o muestras de proteínas recombinantes, con el propósito de la normalización de las mediciones bioquímicas. Sin embargo, una no-linealidad intrínseca compromete la sensibilidad y la precisión de este método. Se demuestra que bajo condiciones de ensayo estándar, la proporción de las mediciones de absorbancia a 590 nm y 450 nm es estrictamente lineal con la concentración de proteínas. Este sencillo procedimiento aumenta la precisión y mejora la sensibilidad de la prueba unas 10 veces, permitiendo la cuantificación de hasta 50 ng de albúmina sérica bovina. Además, la interferencia común introducido por los detergentes que se utilizan para crear los lisados celulares se reduce considerablemente por el nuevo protocolo. Una ecuación lineal desarrollado sobre la base de la acción de masas y la ley de Beer se adapta perfectamente a los datos experimentales.
Protocolo
Linealización de la curva de calibración Bradford Proteínas:
El Coomassie brillante ensayo de la proteína azul, conocido comúnmente como el ensayo de Bradford 1, se utiliza ampliamente debido a su protocolo rápido y conveniente, así como de su sensibilidad relativa. Desafortunadamente, no existe un alto grado de curvatura en un amplio rango de concentraciones de proteína (Fig. 1). Por lo tanto, sólo una estrecha gama de concentraciones de proteína relativamente alto, 10.2 mg / ml de BSA, se utiliza para la curva de calibración, la cual se ajusta mejor a la regresión lineal (Fig. 1, color verde). Sin embargo, la no linealidad requiere la concentración de proteínas de las muestras desconocidas a caer dentro de la gama limitada de la curva de calibración con el fin de evitar un gran error, y también reduce la precisión en el alcance limitado. La no linealidad se presenta un problema serio, en particular, cuando las cantidades de microgramos de proteína no están disponibles, y con frecuencia requiere múltiples diluciones de las muestras desconocidas.
Como se señaló en el documento original de Bradford, "la fuente de la no linealidad es el reactivo en sí mismo, ya hay un solapamiento en el espectro de las dos formas de diferente color de la tintura." 1. De hecho, las tres formas del colorante azul brillante de Coomassie se encuentran en equilibrio ácido-base en el pH ácido normal de la prueba 2. Las formas de color rojo, azul y verde han absorbancia máxima a 470, 590 y 650 nm, respectivamente (Fig. 2). El azul es la forma que se une a la proteína, formando un complejo que absorbe la luz intensa a 594 nm 3, 4 (Fig. 2). Bradford también señaló que "el valor de fondo para el reactivo disminuye constantemente a medida que más colorante se une a proteínas" 1 (Fig. 3). Por lo tanto, tratamos de calcular la reducción del fondo de 590 nm como el aumento de cantidades de la proteína se agregan, midiendo el cambio de absorbancia a 450 nm, donde el complejo proteína-colorante no absorbe. Hemos encontrado que la disminución parcial de fondo, pero no totalmente, da cuenta de la no linealidad (Fig. 4).
A continuación, la hipótesis de que la disminución de la concentración de colorante libre produce otra distorsión de la respuesta lineal, porque a medida que la proteína de fijación de colorantes está en equilibrio 5, la formación de complejos no sólo depende de la concentración de la proteína libre, sino también en el de la tintura libre (Fig. 5). Teniendo en cuenta tanto cuestiones relacionadas con la concentración variable del colorante libre, se desarrolló una ecuación matemática que describe una relación lineal entre la concentración de proteínas y la relación de medidas de absorbancia, 590 nm más de 450 nm (Fig. 6). Una descripción detallada del estudio teórico y experimental se puede encontrar en nuestra publicación en 1996 de Analytical Biochemistry 6. La ecuación matemática que fue probado experimentalmente y se encontró que el rendimiento de una curva de calibración lineal en la proteína de toda la gama de concentraciones (Fig. 7). Por otra parte, la ecuación también fue validado por una determinación independiente de la correcta depende del pH valor de la intersección del eje 6.
Protocolo detallado para el ensayo de proteína mejorada Bradford, utilizando un lector de microplacas Absorbancia:
- Preparar un 0,1 mg / ml solución madre de la norma, la albúmina sérica bovina. Cualquier otra norma puede ser elegido, pero tenga en cuenta que la misma norma debe ser utilizado en todos los experimentos.
- Diluir las muestras desconocidas en agua destilada. Objetivo de 50 a 50 mg / ml. Sin embargo, mayor o menor concentración de proteínas son aceptables, ya que no existe un límite claro para el rango lineal del ensayo. Sin embargo, la medición debe estar dentro del rango lineal del lector de absorbancia.
- Diluir el reactivo de Bradford (Bio-Rad) de 2,5 veces en agua destilada.
- Añadir 0 y 10 a 50 l de solución stock de BSA para tres pozos (la creación de un 0-5 mg curva de calibración BSA). Complementar con agua destilada para llegar a 100 l / pocillo.
- En diferentes pozos, añadir 100 ml de la muestra problema por triplicado. Varias concentraciones de la muestra desconocida puede ser utilizado para aumentar la precisión.
- Añadir 100 ml de los gases de reactivo de Bradford a todos los pocillos. El volumen total es de 200 l / pocillo.
- Espere por lo menos 5 minutos, pero no más de 60 minutos para que el color.
- El blanco debe ser de 200 l de agua destilada (y no el tinte de proteínas y cero).
- Medir la absorbancia a 590 nm y 450 nm.
- Preparar una curva de calibración dividiendo los valores de absorbancia neta a 590 nm y 450 nm. Tenga en cuenta que la proteína cero (tinte solamente) el valor debe ser incluido como un punto de datos (Fig. 8).
- Calcular la concentración de la muestra problema sobre la base de la ecuación lineal de la curva de calibración (Fig. 9).
Los resultados representativos:
A diferencia de la absorbancia a una longitud de onda de 590 nm, la proporción de valores de absorción, 590 nm más de 450 nm, es lineal wconcentración de proteína i (Fig. 10).
La concentración de proteína de la muestra desconocida puede ser simplemente calculó mediante la ecuación lineal de la curva de calibración (Fig. 10, la ecuación).
Sin embargo, una mayor precisión se obtiene mediante la medición de varias diluciones de la muestra desconocida. Con este fin, preparar dos gráficas. La primera es una curva de calibración para el estándar de proteínas g en el eje X (Fig. 11, derecha). El segundo gráfico es que la muestra desconocida, con l de la muestra original sin diluir en el eje X (Fig. 11, izquierda). El único valor medio de contraste debe ser incluido en ambos gráficos (Fig. 11). La concentración de proteína de la muestra desconocida se obtiene dividiendo las laderas de la muestra desconocida y el estándar (Fig. 11, la ecuación).
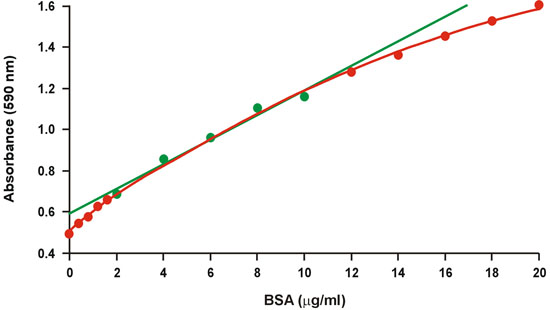
Figura 1. Gráfica convencional de calibración de Bradford. El rango lineal es representada por símbolos de color verde.

Figura 2. Espectros del complejo proteína-colorante y el colorante solo.

Figura 3. Proteína-colorante equilibrio.
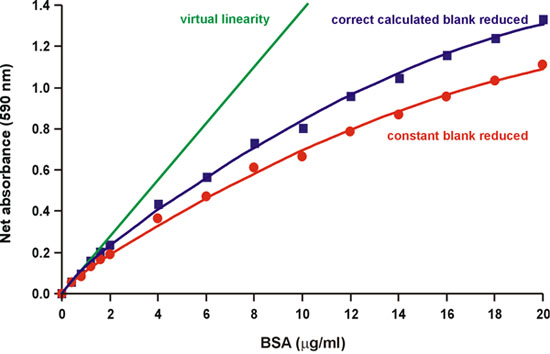
Figura 4. Reducción de los antecedentes correctos calculado disminuye parcialmente la no linealidad.
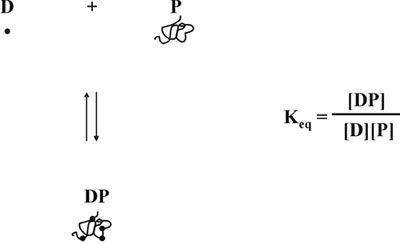
Figura 5. Proteína-colorante equilibrio.

Figura 6. Base matemática para la linealización de la proteína de ensayo Bradford.
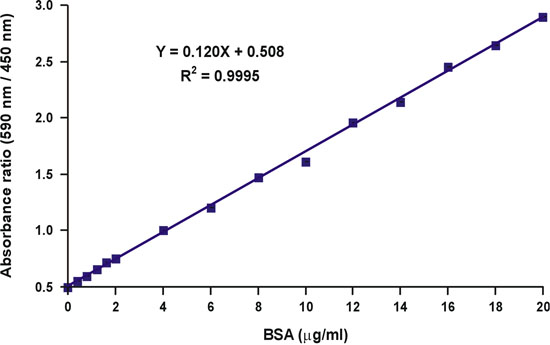
Figura 7. Linealización de la curva de calibración Bradford.

Figura 8. Cálculo de los valores de absorbancia relación.

Figura 9. Un gráfico lineal Bradford calibración.
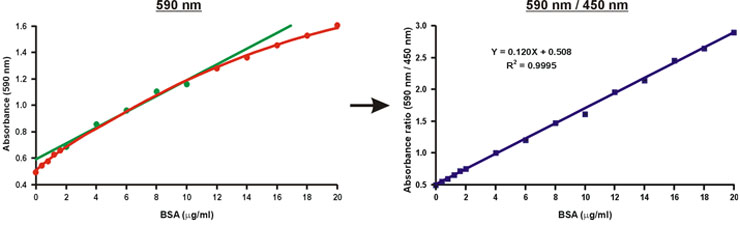
Figura 10. Linealización de la curva de calibración Bradford.
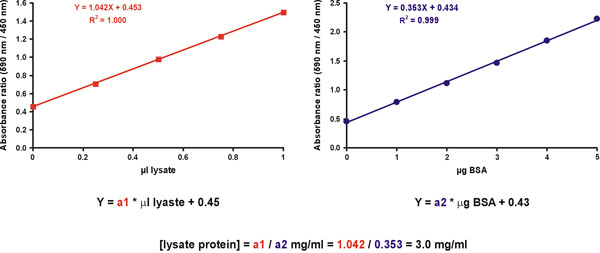
Figura 11. Desconocido muestra la concentración de cálculo.
Discusión
El ensayo de proteínas Bradford es muy popular debido a su facilidad de realización y la sensibilidad relativa. La linealización sobre las concentraciones de proteína completa gama que se obtenga el protocolo que aquí se presenta simplifica aún más el ensayo, ya que las muestras desconocidas no tiene que estar dentro del rango de la curva de calibración.
Es importante destacar que el protocolo mejorado aún ofrece las siguientes ventajas sobre el protocolo original de Bradford:
<...Agradecimientos
Este trabajo está dedicado a la memoria del fallecido Dr. Zvi Selinger, que fue sede de la investigación original se describe aquí.
Materiales
| Name | Company | Catalog Number | Comments |
| Bradford reagent | Bio-Rad | 500-0006 | |
| Bovine Serum Albumin | Amersco | 0332 | |
| Multiplate absorbance reader | BioTek | Synergy2 |
Referencias
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie blue G. Anal Biochem. 209, 258-266 (1993).
- Chial, H. J., Splittgerber, A. G. A comparison of the binding of Coomassie brilliant blue to proteins at low and neutral pH. Anal Biochem. 213, 362-369 (1993).
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Anal Biochem. 151, 369-374 (1985).
- Congdon, R. W., Muth, G. W., Splittgerber, A. G. The binding interaction of Coomassie blue with proteins. Anal Biochem. 213, 407-413 (1993).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: theoretical and experimental studies. Anal Biochem. 236, 302-308 (1996).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados